Method Article
Metodo per identificare piccole molecole inibitori della interazione proteina-proteina tra HCN1 e TRIP8b
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
L'interazione tra i canali HCN e la loro subunità accessoria è stato identificato come un obiettivo terapeutico nel Disturbo Depressivo Maggiore. Qui, un metodo di polarizzazione basato fluorescenza per identificare piccole molecole inibitrici di questa interazione proteina-proteina, è presentato.
Abstract
canali nucleotide-gated cicliche iperpolarizzazione attivato (HCN) sono espressi ubiquitariamente in tutto il cervello, dove funzionano per regolare l'eccitabilità dei neuroni. La distribuzione subcellulare di questi canali nei neuroni piramidali dell'ippocampo CA1 di superficie è regolata da tetratricopeptide Rab8b proteina repeat-contenenti interagenti (TRIP8b), subunità accessoria. knockout genetica di HCN poro formare subunità o TRIP8b, sia portare ad un aumento dei comportamenti antidepressivo-like, suggerendo che la limitazione della funzione dei canali HCN può essere utile come trattamento per il disturbo depressivo maggiore (MDD). Nonostante rilevante interesse terapeutico, canali HCN sono anche espressi nel cuore, dove regolano ritmicità. Per aggirare i problemi fuori bersaglio associati con il blocco canali HCN cardiaci, il nostro laboratorio ha recentemente proposto mira l'interazione proteina-proteina tra HCN e TRIP8b al fine di interrompere specificamente la funzione di canale HCN nel cervello.TRIP8b lega al HCN pori formando subunità a due siti di interazione distinti, anche se qui l'attenzione è rivolta all'interazione tra la ripetizione tetratricopeptide (TPR) domini di TRIP8b e la coda del terminale C di HCN1. In questo protocollo, una descrizione estesa di un metodo per purificare TRIP8b e l'esecuzione di uno schermo ad alta produttività per identificare piccole molecole inibitrici della interazione fra HCN e TRIP8b, è descritto. Il metodo di screening ad alto rendimento utilizza un fluorescenza polarizzata (FP) a base di test per monitorare il legame di un grande frammento TRIP8b ad un undici fluoroforo-tag amino peptide acido corrispondente alla coda terminale HCN1 C. Questo metodo permette composti HIT per essere identificati sulla base della variazione della polarizzazione della luce emessa. test di convalida vengono poi eseguiti per garantire che 'colpiscono' composti non sono artefatta.
Introduzione
(HCN) canali nucleotide-gated cicliche iperpolarizzazione attivato sono espressi nel cuore e sistema nervoso centrale dove giocano un ruolo importante nella regolazione dell'eccitabilità di membrana 1. Canali HCN sono stati implicati nella patogenesi del Disturbo Depressivo Maggiore (MDD) 2, che ha portato diversi gruppi di proporre che la limitazione HCN funzione del canale farmacologicamente può essere efficace come un trattamento innovativo per MDD 3. Tuttavia, il targeting direttamente canali HCN non è praticabile a causa del loro ruolo importante nella cardiaca potenziale d'azione 4. Ivabradina, l'unico approvato dalla FDA antagonista dei canali HCN, viene utilizzato per il trattamento dell'insufficienza cardiaca per produrre un effetto bradicardia 5. Come tale, vi è la necessità di agenti farmacologici che limitano la funzione del canale HCN esclusivamente nel sistema nervoso centrale.
Tetratricopeptide ripetere contenenti Rab8b proteine che interagiscono (TRIP8b) è una specifica del cervellofic subunità accessoria dei canali HCN che controlla l'espressione di superficie e la localizzazione dei canali HCN 6,7. Knockout genetica di TRIP8b provoca una riduzione del cervello canali HCN 7 senza influenzare l'espressione HCN nel cuore 8. È interessante notare che, topi knockout TRIP8b passano meno tempo immobile sul forzata compito nuotata e sospensioni di coda compito 7, due test di screening comunemente usati per efficacia antidepressiva 9-11. Questi risultati suggeriscono che, piuttosto che prendere di mira direttamente i canali HCN con una piccola molecola di antagonisti funzione del canale HCN, interrompendo l'interazione tra TRIP8b e HCN può essere sufficiente a produrre un comportamento antidepressivo-simile.
TRIP8b lega al HCN a due siti di legame. Il dominio di legame ciclico nucleotide (CNBD) di HCN interagisce con un dominio conservato di terminale TRIP8b situata N per i domini TPR di TRIP8b 12,13. Sebbene i residui della CNBD che sono coinvolti in questointerazione sono stati mappati 14, la regione di TRIP8b che è coinvolto non è stato ristretto al di là di un-80-amino frammento di acido 13. Una seconda interazione avviene tra i tetratricopeptide ripetizione (TPR) domini di TRIP8b e del tripeptide terminal C di HCN ( 'SNL' in HCN1, HCN2, e HCN4, ma 'ANM' in HCN3) 3,12. La struttura cristallina di recente risolto 15 di questa interazione C coda rivelato notevole somiglianza strutturale con l'interazione tra il recettore importazione peroxisomal, peroxin 5 (PEX5), e la sua interazione partner, contenente tipo 1 perossisomiali di targeting sequenze (PTS1) 16.
Sebbene entrambi i siti di interazione sono necessari per la funzione del canale HCN, l'interazione tra i domini TPR di TRIP8b e terminale tripeptide C di HCN1 serve come sito di legame dominante e regola l'espressione di superficie HCN. Pertanto, questa interazione è stata scelta come sito di targeting in questo studio.Per il resto del manoscritto, quando si fa riferimento all'interazione tra TRIP8b e HCN, è questa interazione che viene indicato. Questa interazione è ricapitolato da un frammento altamente solubile di TRIP8b corrispondente al suo terminale conservata C contenente i domini TPR richiesti per il legame della coda C terminale HCN (residui 241-602 della 1A-4 isoforme di TRIP8b) 3.
Per sviluppare uno schermo ad alta produttività per identificare piccole molecole in grado di interrompere questa interazione, una polarizzazione di fluorescenza (FP) a base di dosaggio è stato impiegato 17. Fluorescenza polarizzata è basata sull'eccitazione di un ligando fluoroforo-contrassegnate con luce polarizzata, e misurando il grado di polarizzazione della fluorescenza emessa 18. In presenza di un partner legante, il moto di rotazione del ligando fluorescente viene vincolata e la luce polarizzata viene emessa 19. In assenza di un partner legante, tegli moto di rotazione del ligando porta alla emissione di luce depolarizzata.
Nel protocollo allegato, un metodo per la purificazione di N terminale-tag (6xHis) TRIP8b (241-602) con acido nitrilotriacetico nichel (Ni-NTA) perline è presentato. Un protocollo simile è stato impiegato per purificare il glutatione-S-transferasi (GST) -tagged C terminale di 40 amminoacidi di HCN1 (HCN1 C40) utilizzato nel passaggio 7 del protocollo. Per ragioni di spazio, una descrizione dettagliata di tale procedura è stata omessa.
Nei passaggi da 2 a 7 del protocollo, un flusso di lavoro screening ad alto rendimento è presentato (vedi Figura 1). Interazioni proteina-proteina sono un obiettivo notoriamente difficile per lo screening ad alto rendimento ed i lettori sono invitati a cercare ulteriori risorse sul tema 20.
I punti 2 e 3 della procedura caratterizzano l'affinità in vitro della TRIP8b purificato (241-602) costruire fora fluoresceina isotiocianato (FITC) -tagged undici peptide amminoacido corrispondente alla coda terminal C di HCN1 (HCN1 FITC). Sulla base della struttura cristallina del TRIP8b-HCN complesso 15, questo segmento acido undici aminoacidi è sufficiente a produrre vincolante con TRIP8b (241-602). Nella fase 2, la K D dell'interazione viene misurata per titolazione TRIP8b (241-602) in una concentrazione fissa di HCN1 FITC. Al punto 3, una versione senza etichetta del peptide HCN utilizzata al punto 2 viene titolato in una concentrazione fissa di entrambi TRIP8b (241-602) e HCN1 FITC di esaminare se il tag FITC interferisce con il legame. Questi esperimenti sono essenziali per la selezione delle concentrazioni adeguate di TRIP8b (241-602) e HCN1 FITC utilizzati nella schermata di throughput elevato.
La premessa dello schermo throughput elevato è che una piccola molecola in grado di interrompere l'interazione tra TRIP8b (241-602) e HCN1 FITC produrrà un DECRease in luce polarizzata. Nel passo 4, il fattore Z del dosaggio è calcolata 21 per una data concentrazione di TRIP8b (241-602) e HCN1 FITC per assicurare che il saggio è appropriato per screening ad alta (fase 5). I punti 6 e 7 sono saggi di convalida per confermare che i colpi individuati nella schermata principale throughput elevato agiscono interrompendo l'interazione tra TRIP8b (241-602) e HCN1 FITC piuttosto che attraverso un meccanismo non specifico. Al punto 6, carboxytetramethylrhodamine (TAMRA) -labeled HCN1 peptide (HCN1 TAMRA) è utilizzato in un polarizzazione di fluorescenza altrimenti identica a filtrare i composti fluorescenti che compromettono il test FP utilizzando il tag FITC. Fase 7 utilizza un frammento più ampio terminale HCN1 C (HCN1 C40) e impiega un saggio di prossimità bead-based, che si basa sul 'tunneling' di ossigeno singoletto da un tallone donatore per tallone accettore avvicinato tra loro da proteine interagenti 22.
Protocollo
1. Purificazione di TRIP8b (241-602) Proteine
- Trasformare il plasmide contenente TRIP8b (241-602) nella espressione della proteina batterica vettore pGS21 3 in E. competente coli per l'espressione della proteina in base alle istruzioni del produttore. Piastra 300 ml di cultura su Luria Broth (LB) -agar con 5 mg / ml di cloramfenicolo e ampicillina. Incubare la piastra a 37 ° C per 16 h.
- Il giorno successivo, scegliere una singola colonia per inoculare 50 ml LB con 50 mg / ml di cloramfenicolo e ampicillina. Incubare la coltura a 37 ° C per 16 ore (con agitazione).
- Aggiungere cultura 50 ml di 1 L di LB con 50 ug / ml di ampicillina. Incubare a 37 ° C con agitazione.
- Una volta che la coltura ha raggiunto un OD 600 lettura di 0,8-1,2, cambiare la temperatura dell'incubatore a 18 ° C e aggiungere IPTG (isopropil-Beta-D-tiogalattoside) ad una concentrazione finale di 1 mM. Consentire l'espressione della proteina di procedere per 16 ore.
- Spin giù per la E. coli a 6.000 xg per 15 min a 4 ° C per sedimentare i batteri. Dopo aver rimosso i tubi dalla centrifuga, mantenere i batteri in ghiaccio per il restante della procedura. Risospendere i batteri in 36 ml di tampone A con 0,25 mm di phenylmethanesulfonyl fluoruro (PMSF).
- Sonicare i batteri risospeso su ghiaccio con una vite piatta per 5-10 minuti, alternando 30 sec 'on' e 30 sec 'off' ad alta potenza.
- Centrifugare a 12.000 xg per 15 min a 4 ° C. Centrifugare per un ulteriore 15 minuti se il surnatante non è ancora chiaro.
- Applicare il surnatante in una colonna di 2 perle ml Ni-NTA. Incubare per 60 minuti a 4 ° C con dolce dondolio.
- Lasciare che il materiale non legato di fluire attraverso la colonna e lavare con 500 ml di tampone A.
- Lavare la colonna con 250 ml di tampone B.
- Lavare la colonna con 125 ml di tampone A integrati con 5 mM imidazolo.
- Eluire la proteina from la colonna con 20 ml di tampone di eluizione.
- Aggiungere la proteina eluita ad una dialisi cassetta (cutoff Peso molecolare - 10 kDa) usando una siringa. Dializzare la proteina per 60 min a 4 L di freddo Phosphate Buffered Saline (PBS) a 4 ° C.
- Spostare la cassetta di dialisi in un nuovo 4 L secchio di PBS freddo. Dializzare proteine per 16 ore a 4 ° C.
- La mattina seguente, controllare la concentrazione di proteine utilizzando un kit di analisi delle proteine Coomassie, seguendo le istruzioni del produttore. Concentrare la proteina utilizzando un concentratore proteine, seguendo le istruzioni del produttore, se si osserva una concentrazione inferiore a 40 micron. Aliquotare e congelare a -80 ° C per la conservazione.
2. Small Scale fluorescenza polarizzata test per caratterizzare l'interazione dei due frammenti di proteine
- Scongelare 200 ml di 40 mM TRIP8b (241-602) e aggiungerlo a una provetta da 1,5 ml. Aggiungere 100 ml di soluzione salina in 11 ulteriori tubi da 1,5 ml.
- Eseguire diluizioni seriali trasferendo 100 ml di TRIP8b (241-602) dal tubo originale al tubo successivo nella serie, pipettando su e giù, e ripetendo il processo. Ciò produrrà una serie di 12 punti di diluizioni di 2 volte di TRIP8b (241-602) che vanno 0,01-40 micron.
- Preparare una master mix 650 ml di 0,1 micron HCN1 FITC (6,5 ml di una aliquota di 10 micron) e 2 mm ditiotreitolo (DTT) in PBS.
Nota: Il master mix è 2x. - Impostare 12 nuovi tubi da 1,5 ml. Aggiungere 50 microlitri della master mix contenente HCN1 FITC a ciascuna provetta, e quindi si aggiungono 50 ml di ciascuna delle 12 diluizioni seriali.
Nota: Questo genererà 12 tubi, ciascuna contenente 0,05 mM HCN1 FITC e metà della concentrazione di TRIP8b (241-602) dalla diluizione seriale originale. - Aggiungere la serie di diluizioni di un piatto di test, 30 ml per pozzetto, in triplice copia. Utilizzare una solida piastra a 384 pozzetti nero vincolante basso.
- Spin la piastra Fo 2 min a 900 xg a RT.
- Leggere la piastra con un lettore per micropiastre, seguendo le istruzioni del produttore. Misurare fluorescenza polarizzata utilizzando i seguenti parametri di misura: 485 nm di eccitazione, 530 nm di emissione, 505 specchio dicroico nm, 100 msec tempo di integrazione.
- Tracciare i valori di polarizzazione (MP) rispetto al TRIP8b (241-602) la concentrazione su una scala logaritmica. Montare i dati con l'equazione Hill per determinare l'affinità (K d) di TRIP8b (241-602) per il peptide HCN1 etichettati.
Nota: Per la preparazione delle diluizioni seriali, una piastra multi-pozzo può essere usato al posto dei tubi.
3. esaminare l'interazione proteina-proteina utilizzo di un controllo positivo
- Aggiungere 200 ml di peptide non marcato HCN1 (200 mM) in una provetta da 1,5 ml. Aggiungere 100 ml di soluzione salina in 11 ulteriori tubi da 1,5 ml. Eseguire diluizioni seriali trasferendo 100 microlitri dalla provetta contenente 200 ml di peptide al primo tubodi 100 microlitri di PBS. Ripetere il processo per generare 12 tubi con 2 diluizioni del peptide non marcato HCN1.
- Preparare una master mix 650 ml di 0,2 micron HCN1 FITC e 4 mM TRIP8b (241-602).
Nota: Questa è una soluzione 2x. - Aggiungere 50 ml di master mix per 12 nuovi tubi da 1,5 ml.
- Aggiungere 50 ml di ogni diluizione seriale a una provetta da 1,5 ml.
- Caricare la banda nera microtiter da 384 pozzetti in triplice copia, l'aggiunta di 30 ml per pozzetto.
- Centrifugare la piastra per 2 min a 900 xg a RT.
- Leggere la piastra con un lettore di micropiastre in grado di FP. Utilizzare le stesse impostazioni come descritto al punto 2.7.
- Calcolare l'affinità del peptide non marcato per TRIP8b (241-602) utilizzando l'equazione di Cheng-Prusoff: K d = 1 IC 50 / (1 + [L] / K d 2), dove K d 1 rappresenta l'affinità del marcato peptide per TRIP8b (241-602), IC 50 è determinato nel passo precedentee rappresenta la capacità del ligando non marcato per spostare il legante marcato, [L] è la concentrazione del legante marcato nel passo 3.2 e K d 2 è l'affinità di TRIPb (241-602) per il legante marcato.
4. Valutare Assay Performance (Calcolare Z Factor)
- Preparare una master mix facendo una soluzione 12 ml di tampone FP con 2 mM TRIP8b (241-602), 50 nm HCN1 FITC, e 1 mm DTT in FP tampone.
Nota: Questo richiederà 60 ml di 40 mM TRIP8b (241-602), 60 ml di 10 micron HCN1 FITC, e 1.080 ml di FP tampone. - Aggiungere 30 ml di master mix per ciascun pozzetto di una 384 pozzetti micropiastra nero.
- Aggiungere 1 ml di 1 mM non marcato peptide HCN1 a 192 pozzetti per servire come controllo positivo.
- Aggiungere 1 ml di tampone FP a 192 pozzetti per servire come controllo negativo.
- Centrifugare la piastra per 2 min a 900 xg a RT.
- Leggere la piastra con un miLettore croplate.
- Calcolare il fattore Z per il saggio utilizzando la seguente formula: Z = 1 - 3 * (σ pos -σ neg) / (μ neg -μ pos) dove σ pos e neg σ sono le deviazioni standard dei pozzetti di controllo positivi e negativi e pos μ e neg μ rappresentano i segnali medi nei pozzetti di controllo positivi e negativi.
Schermo 5. High Throughput
- Scongelare aliquote di TRIP8b (241-602) e HCN1 FITC e tenerli in ghiaccio.
- Preparare 10 ml di TRIP8b (2 micron) e HCN1 FITC (50 nm) in tampone FP.
- Dispensare 25 ml della miscela in ciascun pozzetto di un basso legame 384 pozzetti micropiastra nero con una pipetta.
- Per lo screening della libreria, aggiungere composti in tutti i pozzetti di colonne 3 a 22 (40 micron concentrazione finale per ciascuna), utilizzando un gestore di liquido acustica.
Nota: A causa della relatively basso tasso di successo osservato con questo test, due composti diversi sono stati riuniti in un unico bene. I due composti da ciascun pozzetto attiva sono stati successivamente testati singolarmente per identificare che ha conferito l'attività. - Aggiungere 100 nl di dimetilsolfossido (DMSO) in ciascun pozzetto di colonne 1 e 23 di ogni piatto come controlli negativi.
- Aggiungere senza etichetta peptide HCN1 in ciascun pozzetto di colonne 2 e 24 di ogni piatto come controlli positivi.
- Incubare le piastre a temperatura ambiente per 2 ore. Leggere le piastre o incubare a 4 ° C per 16 h prima della lettura.
- Misurare fluorescenza polarizzata su un lettore di piastre. Utilizzare le stesse impostazioni come descritto al punto 2.7.
- Calcolare inibizione percentuale normalizzando i segnali utilizzando i comandi medi positivi e negativi da ciascuna piastra come 100%. Usare l'equazione XN = 100% * ((X neg - X) / (X neg - X pos)) dove X N rappresenta l'inibizione percentuale normalizzata corrispondente al segnale X, X pos È il segnale media dai pozzetti di controllo positivi, e X neg è il segnale dai pozzetti di controllo negativo.
6. Conferma della Hits Utilizzando TAMRA-tag HCN Peptide
- Convalida colpisce con inibizione oltre il 50% utilizzando TAMRA marcato peptide HCN1 (HCN1 TAMRA).
- Preparare una master mix di 12 ml di 2 mM TRIP8b (241-602) e 50 nm HCN1 TAMRA in FP buffer.
- Dispensare 25 microlitri della master mix in ciascun pozzetto di una 384 pozzetti micropiastra nero.
- Trasferire diluizioni seriali di ciascuna composti attivi nei rispettivi pozzetti. Fare due diluizioni tali che la concentrazione di ogni colpo varia da 0,2 micron a 200 micron.
- Per le reazioni di controllo negativo, trasferire 100 nl di DMSO in ciascun pozzetto. Per il controllo positivo aggiungere 100 nl di serialmente diluiti peptide HCN1 senza etichetta con concentrazione finale che vanno da 200μM a 0.1μM.
- Incubare la piastra a RTper 2 ore. Leggi piastre o incubare per 16 ore a 4 ° C prima di leggere.
- Misurare fluorescenza polarizzata (MP) utilizzando i seguenti parametri di misura: 535 nm di eccitazione; 580 nm di emissione; 535 nm specchio dicroico, 20 msec tempo di integrazione.
- Calcolare IC50 valori inserendo i dati di concentrazione dipendenti FP di ogni composto utilizzando un modello di regressione lineare a quattro parametri 23.
7. Convalida dose-risposta di dosaggio di prossimità Bead-based
- Scongelare 1 aliquota di His-tag TRIP8b (241-602) (His-TRIP8b) e proteine di fusione GST-tagged HCN1 (GST- HCN1 C40) e tenerli in ghiaccio.
- Combinare His-TRIP8b con GST-HCN1 C40 in tampone a concentrazioni di 400 Nm His-TRIP8b e 40 Nm GST HCN1 C40. Preparare 300 microlitri della miscela per ogni composto da testare.
Nota: I composti vengono eseguiti in triplicato a 11 differenti concentrazioni più un DMSO di controllo (non composto). - Per pozzetti di controllo, di preparare 30 ml di His-TRIP8b (200 nm) senza GST-HCN1C40 e quindi preparare separatamente 30 ml di GST-HCN1C40 (20 nM) senza il suo-TRIP8b in tampone.
- Per ogni composto da testare, aggiungere 7 ml di His-TRIP8b: miscela GST-HCN1 C40 (preparata al punto 7.2) a 36 pozzetti di una piastra con una pipetta a 16 canali. Per testare un singolo composto, disporre i 12 differenti concentrazioni in righe AL e le tre repliche in colonne 1-3. Aggiungere 7 ml di soluzione di His-TRIP8b (preparato in 7.3) alle colonne 1-3 di fila M e 7 ml di soluzione di GST-HCN1 C40 (preparato in 7.3) alle colonne 1-3 di fila N. centrifugare brevemente la piastra per assicurano i liquidi sono in fondo a ciascun pozzetto e per rimuovere eventuali bolle.
- Dispensare i composti hit da testare nella piastra tale che la concentrazione di ogni colpo varia da 200 mM (in fila A) fino a 0,1 mM (nella riga K). Erogare un volume diDMSO in righe LN che corrisponde alla quantità di composto dispensati in righe AK.
- Agitare la piastra per 2 minuti e incubare per 2,5 ore a temperatura ambiente.
- Diluire l'accettante perle anti-GST 1:50 con il tampone.
- Aggiungere 3,5 microlitri di perle Acceptor diluito a ciascun pozzetto della piastra con una pipetta a 16 canali e mescolare pipettando delicatamente per evitare di creare bolle.
- Incubare la piastra per 1 ora a RT al buio.
- Diluire il donatore perline nichel chelati 1:50 con il tampone. Non esporre la miscela alla luce.
- Aggiungere 3,5 microlitri di perle donatori diluito a ciascun pozzetto della piastra con una pipetta a 16 canali e mescolare pipettando delicatamente per evitare di creare bolle.
- Incubare la piastra per 1,5 ore a RT al buio.
- Leggere su un lettore di piastre. Utilizzare un lettore di piastre con i seguenti parametri di misura: 40 msec eccitazione tempo, 100 msec tempo di emissione, 0,1 millimetri di altezza di rilevamento.
- Calcolare IC 50 valori da dati adatta per EACh composti utilizzando un modello di regressione lineare a quattro parametri 23.
Risultati
Per evitare problemi di copyright con la nostra precedente pubblicazione, una sonda TAMRA-tag HCN1 TAMRA è stato utilizzato per generare figure 2 e 3. Si noti che questa sostituzione non ha fatto una differenza apprezzabile nei risultati, ed i protocolli sono identiche a quelle descritte sopra, con HCN1 FITC. Per valutare l'interazione con HCN1 TAMRA, TRIP8b (241-602) è stato titolato in una concentrazione fissa di HCN1 TAMRA usando il protocollo descritto nella fase 2 (Figura 2). Quindi, l'esperimento descritto nel passaggio 3 è stato eseguito e non marcato HCN1 peptide è stato titolato in una concentrazione fissa di TRIP8b (241-602) e HCN1 TAMRA (Figura 3).
Per verificare che il test era adatto per lo screening ad alto rendimento, la sua performance è stata prossima esaminata dal protocollo al punto 4; e un fattore Z di 0.89 è ottenuto, indicando che il test era pronto per screening ad alta (Figura 4). Poi, una libreria piccola molecola 20.000 composto è stato proiettato con la procedura descritta al punto 5. Tutti i composti che hanno l'inibizione percentuale superiore al 50% sono stati poi testati nel secondo test FP con HCN1 TAMRA (punto 6). Infine, i risultati confermati sono stati analizzati con il test tallone a base di prossimità (passaggio 7, Figura 5). Un composto hit, NUCC-5953, è stato identificato come un risultato di questi esperimenti.

Figura 1: HTS Workflow schematica che descrive il flusso di lavoro per lo screening ad alto rendimento.. Procede Diagramma dalla cima a fondo, con ogni triangolo che rappresenta un passo nel protocollo. Cliccate qui per visualizzare ungrande versione di questa figura.
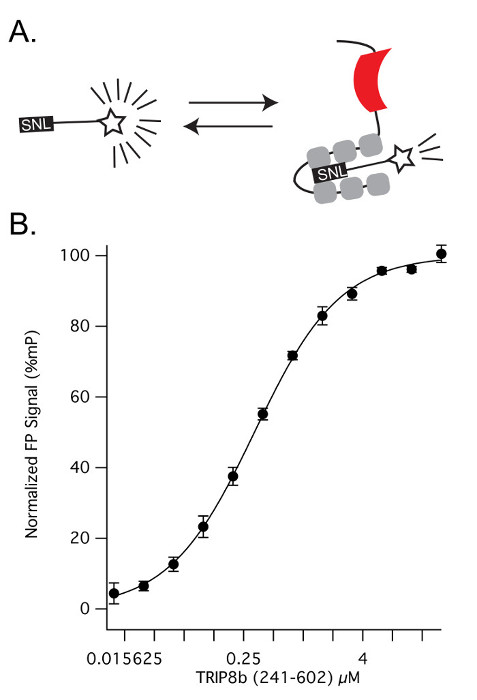
Figura 2:. K D di interazione proteina-proteina (A) Schema che mostra l'esperimento descritto al punto 2. Come TRIP8b (241-602) è titolato in una concentrazione fissa di HCN1 TAMRA, i domini TPR di TRIP8b (in grigio) si legano al peptide di acido 11 aminoacidi (in nero, con 'SNL' terminale evidenziato). (B) come la concentrazione di TRIP8b (241-602) aumenta, più molecole HCN1 Tamra essere vincolati e polarizzazione aumenta (K D = 0.320.01μM). Le barre di errore indicano la deviazione standard. Cliccate qui per vedere una versione più grande di questa figura.

Figura 3:. IC50 di controllo positivo (A) Schema dimostrando paradigma sperimentale per il passo 3. Per quanto privo di etichetta peptide HCN1 è titolato in una concentrazione fissa di TRIP8b (241-602) e HCN1 TAMRA, il peptide marcato viene spostato. (B) Si noti che il segnale diminuisce con l'aumento della concentrazione di HCN1 senza etichetta, indicando spostamento HCN1 TAMRA (IC 50 = 1.070.08μM). Le barre di errore indicano la deviazione standard. Cliccate qui per vedere una versione più grande di questa figura.
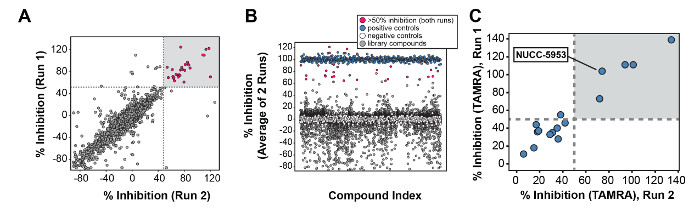
Figura 4:. Rappresentante dei risultati da HTS (A) può derivare dalla elevata produttività sc reen eseguita utilizzando il protocollo descritto nel passaggio 5. Ogni punto sul grafico rappresenta un singolo composto. Le coordinate X e Y sono determinate dalla percentuale di inibizione in ogni seduta. (B) I risultati dello schermo tracciati con controlli positivi e negativi (vedi legenda). l'inibizione percentuale media di ciascun composto (su due corse del saggio) è tracciata sull'asse Y. (C) I risultati di esperimenti di conferma FP utilizzando un peptide HCN1 TAMRA marcato. Composti che mostrano maggiore del 50% di inibizione in passaggio 5 sono stati poi utilizzati nel passaggio 6. Come in (A), le coordinate X e Y sono determinate dalla percentuale di inibizione in due corse del saggio. Tratto da Han et al. 3. Clicca qui per vedere una versione più grande di questa figura.
40 / 54540fig5.jpg "/>
Figura 5:. Basata branello Proximity Assay (A) Schema che mostra il saggio di prossimità bead-based. TRIP8b (241-602) contiene un tag terminale hexahistidine N che lega perline donatori Nickel chelati (cerchio con strisce). HCN1 C40 contiene un tag GST terminale N che lega perline accettore. Quando portato in prossimità dall'interazione dei domini TPR di TRIP8b e del tripeptide terminale C di HCN1, eccitazione del tallone donatore da 680 nm luce produce ossigeno singoletto. Questo singoletto trasferimento dell'ossigeno energia accettore perline in un raggio definito e porta alla emissione di luce. (B) Titolazione di concentrazioni crescenti del composto hit (NUCC-5953) in una concentrazione fissa di TRIP8b (241-602) e HCN1 C40. Tratto da Han et al. 3. Le barre di errore indicano la deviazione standard.target = "_ blank"> Clicca qui per vedere una versione più grande di questa figura.
Discussione
A causa della sua potenziale come un bersaglio terapeutico in MDD 24, c'è stato un considerevole interesse per gli approcci farmacologici che antagonizzano funzione del canale HCN nel sistema nervoso centrale 4. Tuttavia, questi sforzi sono stati in fase di stallo per l'importante ruolo dei canali HCN in pacemaking cardiaco e il rischio di aritmia 25. Abbiamo ragionato che interrompere l'interazione tra HCN e la sua subunità ausiliaria specifica del cervello, TRIP8b 8, potrebbe essere sufficiente a produrre effetti antidepressivi simili senza influenzare i canali cardiaci HCN 3. Questa ipotesi è stata sostenuta dall'osservazione che i topi privi TRIP8b mostrare un comportamento antidepressivo-simile 7. Targeting questa interazione potrebbe diventare un nuovo paradigma per il trattamento del disturbo depressivo maggiore.
Un protocollo dettagliato per identificare piccole molecole inibitrici della interazione tra i domini TPR di TRIP8b e il terminale tripeptide C di HCN1 èdi cui sopra. Per facilitare la sua applicazione generale, noi qui descriviamo il nostro ragionamento che ha portato allo sviluppo di questo test. Anche se TRIP8b lega alla subunità HCN in due sedi, ci siamo concentrati sull'interazione tra i domini TPR di TRIP8b e la coda del terminale C di HCN. La delucidazione strutturale di questa interazione mediante cristallografia a raggi x rivelato una tasca profonda formata dai domini TPR di TRIP8b intorno terminale C di HCN 15. La seconda interazione TRIP8b-HCN avviene tra un tratto di 80 aminoacidi di TRIP8b (che si trova terminale N per i domini TPR) e il ciclico nucleotide dominio di legame di HCN. Questa interazione si verifica in molti aminoacidi diversi di ogni proteina e costituisce una superficie diffusa con un minor numero di interazioni intermolecolari ben definiti 26. Sulla base di queste osservazioni strutturali, abbiamo concluso che l'interazione tra i domini TPR di TRIP8b e il terminale C di HCN è più soggetta a interruzioni da una piccola molecola INHIBITOR.
Inizialmente, un frammento più grande di TRIP8b stato usato nel saggio basato sul presupposto che un costrutto più lungo può avere siti regolatori allosterici e aumentare la probabilità di successo. Lunghezza totale TRIP8b è stato inizialmente utilizzato, ma questo approccio è stata limitata dalla aggregazione di proteine, il degrado, e la sensibilità di gelo e disgelo cicli. Successivamente, due intermedia dimensioni TRIP8b costruisce, a più uno (residui 219-602) e uno più corto (residui 259-602), sono stati testati prima di un costrutto intermedia (residui 241-602) è stato selezionato. Sebbene ciascuno dei quattro troncamenti sopra descritti contengono i domini TPR rilevanti per il legame al terminale C di HCN, solo la lunghezza clone intermedia (241-602) era sufficientemente stabile per l'uso in saggi di screening. In particolare, siamo stati in grado di ottenere grandi quantità di proteina dopo purificazione per Ni 2+ cromatografia di affinità senza fasi di separazione aggiuntivi.
dopo chooszione un adeguato frammento di TRIP8b e HCN, abbiamo successivamente calcolato l'affinità del peptide HCN1 etichettato per TRIP8b (241-602). Come regola generale, una misura precisa può essere fatta solo se la concentrazione del legante è sostanzialmente inferiore al K D del partner di legame 28. Nel nostro caso, abbiamo utilizzato 50 Nm di peptide HCN1 etichettato per l'esperimento nella fase 2, e ha ottenuto una K D di 0.320.01μM. Per considerazioni di progettazione più sperimentali in materia di interazioni proteina-proteina, si rimanda a una delle numerose ottime recensioni sul tema 27-30.
Una volta che abbiamo determinato la K D del peptide HCN1 etichettato per TRIP8b (241-602), abbiamo quindi determinato se l'etichetta si interferisce con il legame. Al punto 3, abbiamo ottenuto un valore di IC 50 di 1.070.08 micron titolando un peptide HCN1 senza etichetta in una concentrazione fissa di TRIP8b (241-602) per spostare il peptide etichettati. In combinazione con la K D della peptide marcato per TRIP8b (241-602), e applicando l'equazione di Cheng-Prusoff, abbiamo quindi stimato l'affinità del peptide non marcato per TRIP8b (241-602) come K D = 0.93μM. Questo è in stretto accordo con l'affinità di TRIP8b (241-602) per il peptide etichettati, e suggerisce che l'etichettatura del peptide non ha influenzato sostanzialmente la sua affinità con TRIP8b. Una caratteristica importante della telematica polarizzazione di fluorescenza è l'eccellente rapporto segnale-rumore, come indicato da un fattore Z di 0.89. Con altri parametri stesso, l'entità della variazione del segnale FP è dettata dalla dimensione del partner di legame (TRIP8b (241-602)) e la dimensione del legante fluoroforo-marcato. Un cambiamento di polarizzazione (44-224 MP) osservata con il 11 amino peptide di acido legante tra i suoi Stati liberi e legati con il ~ 42 kDa TRIP8b (241-602) proteina è sufficiente per riprodurre l'interazione mirata e fornisce una gamma dinamica sufficiente per lo screening della libreria.
S copi "> Una delle sfide di qualsiasi test di screening ad alto rendimento è distinguere vero 'colpo' composti da cambiamenti artefatti di intensità del segnale. Nelle schermate di polarizzazione a base di fluorescenza, è comune per molti composti a uno interagire direttamente con il fluoroforo o fluorescenza su loro. per aggirare questi problemi, una procedura di screening in due fasi che utilizza due fluorofori distinti è descritto sopra. Questa procedura riduce la probabilità che i composti fluorescenti e composti che interagiscono direttamente con il fluoroforo avanzerà attraverso il processo di screening. Oltre al legame il fluoroforo , alcuni composti agiscono come "aggregatori" in vitro e portare a cambiamenti non specifici nel segnale a fluorescenza polarizzata. questi composti sono pensati per formare strutture micellari detergenti sensibili e di inibire le interazioni proteina-proteina 31,32. per attenuare questi effetti, è importante che il buffer a fluorescenza polarizzata utilizzato in altail throughput protocollo di screening sopra descritto comprende un detergente (Triton), anche se un'ulteriore convalida con altri detergenti dovrebbe essere considerato.Va notato che ci sono diversi importanti limitazioni della procedura di screening descritto in precedenza. Anche se TRIP8b lega al HCN in due sedi, la schermata di cui sopra prende in esame solo un sito di interazione e non identificherà piccole molecole di targeting l'interazione CNBD. Allo stesso modo, composti che modulano allostericamente TRIP8b legame HCN interagendo con TRIP8b nella regione terminale N non saranno identificati come risultati. Entrambe queste considerazioni sono il risultato di utilizzare un frammento TRIP8b più piccolo (vedi sopra), che garantisce la riproducibilità nei saggi di screening descritti nella procedura. Per evitare queste limitazioni, sforzi futuri possono essere dirette ad utilizzare uno schermo basato cellule incorporanti piena HCN lunghezza e TRIP8b costruisce 33,34. Questo tipo di schermo può fare affidamento su di alta throughput metodi elettrofisiologici per identificare composti capaci di interrompere l'interazione fra HCN e TRIP8b, e limitando l'espressione dei canali HCN sulla superficie cellulare. Da segnalare, approcci come questi dovrebbero includere contro-schermi per garantire che le piccole molecole non limitavano direttamente la funzione di canale HCN come antagonisti, in quanto ciò potrebbe portare ad effetti di destinazione off in vivo.
Anche se HCN1, HCN2, e HCN4 tutti hanno un conservata 'SNL' peptide terminal C, i residui terminali N della presente tripeptide variare in modo sostanziale e probabilmente influenzare l'affinità di legame TRIP8b. Ciò solleva la possibilità che una piccola molecola colpito ottenuta tramite schermo o progettato sulla base di altri composti hit possono fornire HCN isoforma specificità a interrompere l'interazione TRIP8b-HCN. Nella schermata iniziale, NUCC-5953 è stata identificata come la prima piccola molecola in grado di interrompere l'interazione tra TRIP8b e HCN1 3. wor FutureK con questo test può individuare ulteriori piccole molecole inibitrici con desiderabili proprietà chimiche per lo sviluppo di farmaci.
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
This work was supported by National Institutes of Health Grant R21MH104471 and R01MH106511 (D.M.C.), Brain Research Foundation SG 2012-01 (D.M.C.), Northwestern University Clinical and Translational Sciences Institute 8UL1TR000150 (Y.H.), Chicago Biomedical Consortium HTS-004 (Y.H. and D.M.C.), and National Institutes of Health Grant 2T32MH067564 (K.L.). A part of this work was performed by the Northwestern University Medicinal and Synthetic Chemistry Core (ChemCore) at the Center for Molecular Innovation and Drug Discovery (CMIDD), which is funded by the Chicago Biomedical Consortium with support from the Searle Funds at the Chicago Community Trust and Cancer Center Support Grant P30 CA060553 from the National Cancer Institute awarded to the Robert H. Lurie Comprehensive Cancer Center. The high throughput screen work was performed in the High Throughput Analysis Laboratory which is also a core facility of the Robert H. Lurie Comprehensive Cancer Center.
Materiali
| Name | Company | Catalog Number | Comments |
| Ni-NTA agarose | Qiagen | 30210 | |
| Dialysis cassette | ThermoFisher | 66456 | |
| Isopropyl b-D-1-thiogalactopyranoside | Sigma-Aldrich | I5502 - 1 gr | |
| 384-Well Black plate | Corning | 3820 | |
| Proxiplate | Perkin-Elmer | 6008289 | |
| Anti-GST Acceptor beads | Perkin-Elmer | 6760603C | |
| NiChelate | Perkin-Elmer | AS101D | |
| pGS21-a | Genscript | SD0121 | |
| PMSF | Sigma-Aldrich | 10837091001 | |
| Coomassie Kit | ThermoFisher | 23200 | |
| Protein concentrator | ThermoFisher | 88527 | |
| Perkin Elmer Enspire Multimode Plate reader | Perkin-Elmer | #2300-001M | |
| BL21 (DE3) Competent Cells | Agilent | 200131 |
Riferimenti
- Biel, M., Wahl-Schott, C., Michalakis, S., Zong, X. Hyperpolarization-activated cation channels: from genes to function. Physiol Rev. 89 (3), 847-885 (2009).
- Shah, M. M. HCN1 channels: a new therapeutic target for depressive disorders?. Sci Signal. 5 (244), pe44 (2012).
- Han, Y., et al. Identification of Small-Molecule Inhibitors of Hyperpolarization-Activated Cyclic Nucleotide-Gated Channels. J Biomol Screen. 20 (9), 1124-1131 (2015).
- Postea, O., Biel, M. Exploring HCN channels as novel drug targets. Nat Rev Drug Discov. 10 (12), (2011).
- Stieber, J., Wieland, K., Stöckl, G., Ludwig, A., Hofmann, F. Bradycardic and proarrhythmic properties of sinus node inhibitors. Mol Pharmacol. 69 (4), 1328-1337 (2006).
- Santoro, B., Wainger, B. J., Siegelbaum, S. A. Regulation of HCN channel surface expression by a novel C-terminal protein-protein interaction. J Neurosci. 24 (47), 10750-10762 (2004).
- Lewis, A. S., et al. Deletion of the hyperpolarization-activated cyclic nucleotide-gated channel auxiliary subunit TRIP8b impairs hippocampal Ih localization and function and promotes antidepressant behavior in mice. J Neurosci. 31 (20), 7424-7440 (2011).
- Heuermann, R. J., et al. Reduction of thalamic and cortical Ih by deletion of TRIP8b produces a mouse model of human absence epilepsy. Neurobiol Dis. 85, 81-92 (2016).
- Steru, L., Chermat, R., Thierry, B., Simon, P. The tail suspension test: A new method for screening antidepressants in mice. Psychopharmacology. 85 (3), 367-370 (1985).
- Petit-Demouliere, B., Chenu, F., Bourin, M. Forced swimming test in mice: a review of antidepressant activity. Psychopharmacology. 177 (3), 245-255 (2004).
- Cryan, J. F., Mombereau, C., Vassout, A. The tail suspension test as a model for assessing antidepressant activity: Review of pharmacological and genetic studies in mice. Neurosci Biobehav Rev. 29 (4-5), 571-625 (2005).
- Han, Y., et al. Trafficking and gating of hyperpolarization-activated cyclic nucleotide-gated channels are regulated by interaction with tetratricopeptide repeat-containing Rab8b-interacting protein (TRIP8b) and cyclic AMP at distinct sites. J Biol Chem. 286 (23), 20823-20834 (2011).
- Hu, L., Santoro, B., Saponaro, A., Liu, H., Moroni, A., Siegelbaum, S. Binding of the auxiliary subunit TRIP8b to HCN channels shifts the mode of action of cAMP. J Gen Physiol. 142 (6), 599-612 (2013).
- Saponaro, A., et al. Structural basis for the mutual antagonism of cAMP and TRIP8b in regulating HCN channel function. Proc Natl Acad Sci U S A. 111 (40), 14577-14582 (2014).
- Bankston, J. R., Camp, S. S., DiMaio, F., Lewis, A. S., Chetkovich, D. M., Zagotta, W. N. Structure and stoichiometry of an accessory subunit TRIP8b interaction with hyperpolarization-activated cyclic nucleotide-gated channels. Proc Natl Acad Sci U S A. 109 (20), 7899-7904 (2012).
- Gatto, G. J., Geisbrecht, B. V., Gould, S. J., Berg, J. M. Peroxisomal targeting signal-1 recognition by the TPR domains of human PEX5. Nat Struct Biol. 7 (12), 1091-1095 (2000).
- Owicki, J. C. Fluorescence polarization and anisotropy in high throughput screening: perspectives and primer. J Biomol Screen. 5 (5), 297-306 (2000).
- Smith, D. S., Eremin, S. A. Fluorescence polarization immunoassays and related methods for simple, high-throughput screening of small molecules. Anal Bioanal Chem. 391 (5), 1499-1507 (2008).
- Roehrl, M. H. A., Wang, J. Y., Wagner, G. A general framework for development and data analysis of competitive high-throughput screens for small-molecule inhibitors of protein-protein interactions by fluorescence polarization. Biochemistry. 43 (51), 16056-16066 (2004).
- Arkin, M. R., Wells, J. A. Small-molecule inhibitors of protein-protein interactions: progressing towards the dream. Nat Rev Drug Discov. 3 (4), 301-317 (2004).
- Zhang, J., Chung, T., Oldenburg, K. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. J Biomol Screen. 4 (2), 67-73 (1999).
- Eglen, R. M., et al. The use of AlphaScreen technology in HTS: current status. Curr Cheml Genomics. 1 (1), 2-10 (2008).
- Lehninger, A. L., Nelson, D. L., Cox, M. M. . Lehninger Principles of Biochemistry. , (2000).
- Kim, C. S., Chang, P. Y., Johnston, D. Enhancement of dorsal hippocampal activity by knockdown of HCN1 channels leads to anxiolytic- and antidepressant-like behaviors. Neuron. 75 (3), 503-516 (2012).
- Wahl-Schott, C., Biel, M. HCN channels: structure, cellular regulation and physiological function. Cell Mol Life Sci. 66 (3), 470-494 (2009).
- DeBerg, H. A., Bankston, J. R., Rosenbaum, J. C., Brzovic, P. S., Zagotta, W. N., Stoll, S. Structural Mechanism for the Regulation of HCN Ion Channels by the Accessory Protein TRIP8b. Structure. 23 (4), 734-744 (2015).
- Weiss, J. N. The Hill equation revisited: uses and misuses. FASEB J. 11 (11), 835-841 (1997).
- Prinz, H. Hill coefficients, dose-response curves and allosteric mechanisms. J Chem Biol. 3 (1), 37-44 (2009).
- Pollard, T. D. A Guide to Simple and Informative Binding Assays. Mol Biol Cell. 21 (23), 4061-4067 (2010).
- Rossi, A. M., Taylor, C. W. Analysis of protein-ligand interactions by fluorescence polarization. Nat Protoc. 6 (3), (2011).
- Ryan, A. J., Gray, N. M., Lowe, P. N., Chung, C. -. W. Effect of Detergent on "Promiscuous" Inhibitors. J Med Chem. 46 (16), 3448-3451 (2003).
- McGovern, S. L., Caselli, E., Grigorieff, N., Shoichet, B. K. A common mechanism underlying promiscuous inhibitors from virtual and high-throughput screening. J Med Chem. 45 (8), 1712-1722 (2002).
- Hertzberg, R. P., Pope, A. J. High-throughput screening: new technology for the 21st century. Curr Opin Chem Biol. 4 (4), 445-451 (2000).
- Sundberg, S. A. High-throughput and ultra-high-throughput screening: solution-and cell-based approaches. Curr Opin Biotechnol. 11 (1), (2000).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon