Method Article
Visualizzazione di miniSOG Tagged proteine di riparazione del DNA, in combinazione con Electron spettroscopica Imaging (ESI)
In questo articolo
Riepilogo
Electron spectroscopic imaging can image and distinguish nucleic acid from protein at nanometer resolution. It can be combined with the miniSOG system, which is able to specifically label tagged proteins in transmission electron microscopy samples. We illustrate the use of these technologies using double-strand break repair foci as an example.
Abstract
I limiti della risoluzione ottica e la sfida di identificare popolazioni di proteine specifiche in microscopia elettronica a trasmissione sono stati gli ostacoli in biologia cellulare. Molti fenomeni non possono essere spiegati mediante analisi in vitro in sistemi semplificati e necessitano di ulteriori informazioni strutturali in situ, in particolare nel campo tra 1 nm e 0,1 micron, per essere pienamente compreso. Qui, electron spettroscopica di imaging, una tecnica di microscopia elettronica di trasmissione che permette la mappatura simultanea della distribuzione delle proteine e acidi nucleici, e un tag espressione, miniSOG, vengono combinati per studiare la struttura e l'organizzazione di DNA a doppio filamento break riparazione foci.
Introduzione
Nonostante i progressi significativi nella microscopia ottica negli ultimi anni 1, cell-biologi soffrono ancora di una lacuna nella risoluzione. Questo limita comprensione delle relazioni struttura-funzione nei processi cellulari fondamentali che coinvolgono l'interazione coordinata tra complessi macromolecolari (ad esempio, nel rimodellamento della cromatina, la riparazione del DNA, RNA trascrizione e replicazione del DNA). Sebbene microscopi elettronici a trasmissione (TEM) s fornire la risoluzione richiesta, è stato difficile da definire questi processi strutturalmente a causa della incapacità di etichettare specifiche proteine pur essendo in grado di determinare la composizione biochimica delle strutture visualizzate. In assenza di membrane interne per aiutare a differenziare le strutture nucleari, il nucleo è stato particolarmente impegnativo. Electron spettroscopica di imaging (ESI) risolve alcune di queste limitazioni, consentendo la determinazione e differenziazione di DNA, RNA, e protein-based strutture nucleari 2-5.
Elettrone di imaging spettroscopico:
Per mappare distribuzioni elementari ad alta sensibilità e risoluzione al microscopio elettronico, si può usare uno spettrometro ad immagine che seleziona elettroni che sono stati anelasticamente dispersi attraverso le interazioni con elettroni guscio interno di un elemento nel campione 6. Poiché specifici dell'elemento quantità di energia vengono persi come conseguenza della ionizzazione degli atomi nel campione, questi elettroni possono essere separati e visualizzati usando uno spettrometro che è collegato al microscopio elettronico. Pertanto, l'analisi dello spettro degli elettroni che hanno interagito con il campione rivela informazioni qualitative e quantitative sulla composizione elementare del campione 7. Gli elettroni che non perdono energia quando passa attraverso il campione si trovano nella "picco perdita zero" della perdita di energia degli elettronispettro. L'abbondanza di questi elettroni è correlato alla massa, densità e spessore del campione e comprende elettroni che passano attraverso il campione senza urtare il campione o perdere energia durante il passaggio attraverso il campione. Queste informazioni possono essere utili per la quantificazione assoluta dei numeri di atomi di un elemento specifico presente nel campione 8.
Poiché campioni biologici consistono principalmente di elementi leggeri che mal deviano gli elettroni nel fascio incidente del TEM, metodi di colorazione utilizzando metallo pesante sali devono essere applicate in modo da generare contrasto nel campione. La mancanza di specificità della maggior parte di questi agenti di contrasto e l'incapacità di visualizzare più di una macchia in cui la specificità è possibile ha limitato il valore di microscopia elettronica convenzionale nello studio del nucleo. ESI ha vantaggi significativi rispetto TEM convenzionali, in particolare per lo studio delle strutture del nucleo cellulare.È possibile sfruttare la natura ricca fosforo di DNA e complessi macromolecolari-RNA contenente distinguere complessi nucleoproteici da complessi proteici e risolvere diversi complessi nucleoproteici basate sulla loro densità di acidi nucleici. Il materiale biologico residuo può essere ripreso in base alla sua abbondanza di azoto. Mappatura solo questi due elementi e analisi della loro distribuzione e abbondanza relativa all'interno di diverse strutture anatomiche ci fornisce un sacco di informazioni sul nucleo. Ad esempio, è facile identificare cromatina e ribosomi nella mappa rappresentano fosforo abbondanza. Lo spazio interchromatin, complessi del poro nucleare, e corpi nucleari, invece, possono essere facilmente individuati nell'immagine azoto mappa.
Mini sistema di generatore di ossigeno singoletto (miniSOG)
Mentre ESI rappresenta una potente tecnica per studiare in situ struttura della cromatina, perché ci vuoles vantaggio dei rapporti caratteristici di composizione elementare tra il fosforo e l'azoto, la composizione elementare non può essere normalmente utilizzati per discriminare tra le diverse popolazioni di complessi proteici. Anticorpi marcati con piccole particelle d'oro nella gamma nanometri sono stati ampiamente utilizzati per mappare la posizione delle singole molecole. Poiché la particella oro viene normalmente collegato a un anticorpo secondario, apparirà entro una circonferenza di circa 20 nm circa l'epitopo rilevato dal anticorpo primario. In campioni successivi-embedding, anticorpi possono rilevare solo epitopi che sono esposte alla superficie delle sezioni. Mentre è possibile dimostrare la presenza di un antigene e si riferiscono ad una certa struttura anatomica della cellula, le informazioni che si ottiene è incompleta, poiché la maggior parte degli epitopi sono oscurati da resina. Tecniche, che utilizzano protocolli simili a quelli utilizzati per la microscopia a fluorescenza Pre-embedding, consentire l'accesso agli antigeni tuttol'intera profondità del campione ma i passi permeabilizzazione necessari per consentire l'anticorpo di penetrare cella tipicamente richiede la rimozione di membrane lipidiche e rimuovere i componenti che non possono essere risolti con aldeidi. Inoltre, il fissativo aldeide preferito per la conservazione ultrastruttura, glutaraldeide, comunemente distrugge epitopi e, di conseguenza, è in genere necessario paraformaldeide. Questo è meno efficace a proteina-proteina reticolazione. Un altro svantaggio di anticorpi che sono etichettati con una nanoparticella di oro è che l'oro è un materiale molto elettron-denso che crea un forte contrasto che possono oscurare interessanti dettagli strutturali del campione, che ha più debole contrasto.
L'emergere della proteina verde fluorescente (GFP) come un tag di proteina espressa ha trasformato l'uso di microscopia a fluorescenza per rispondere alle domande di biologia cellulare. Tagging una proteina con un piccolo dominio fluorescente permette mappatura della sua distribuzione in vivo senza la necessità di procedure di permeabilizzazione che possono alterare la struttura. Per tradurre il principio elegante di usare i tag proteine per mappare distribuzioni cellulari delle proteine di microscopia elettronica, un sistema era necessario che sia in grado di produrre un segnale in prossimità della struttura di interesse e generare contrasto nella TEM. Diaminobenzidina polimerizzato è una macchia che viene spesso utilizzato in istologia per rilevare legame degli anticorpi. Questa macchia è di solito depositato con perossidasi (HRP) coniugata con un anticorpo secondario. Sebbene la reazione produce un risultato affidabile, HRP non è cataliticamente attivo nel citosol delle cellule 9. I prodotti di reazione di HRP possono anche diffondono lontano dal sito di generazione in modo che la loro risoluzione è peggiore rispetto al metodo nanogold 10. Per bypassare questi problemi, il sistema / Reash Flash è stato sviluppato 11. È costituita da proteine di fusione ricombinanti che possiedono un motivo tetracysteine. Questo motivo permette vincolanteun fluoroforo biarsenic. Quando eccitato, il flash associato o Reash è in grado di generare l'ossigeno singoletto altamente reattivo e quindi photoconvert diaminobenzidina (DAB) in un polimero che precipita immediatamente al sito delle proteine tag. Il polimero DAB può essere colorato con tetrossido di osmio, che è denso di elettroni e quindi può essere utilizzato per mappare la distribuzione della proteina di fusione ricombinante nel TEM.
Nel 2011, Shu et al. 10 presentato il sistema miniSOG, che consiste in un piccolo 106 amino acidi tag fusione derivato da una flavoproteina di Arabidopsis che è fluorescente e in grado di creare molti radicali ossigeno singoletto eccitato con 448 quando nm luce blu. Tali radicali dell'ossigeno singoletto possono essere utilizzati per foto-diaminobenzidina ossidare per formare polimeri sopra e vicino alla superficie della proteina marcata, che è notevolmente più vicino di anticorpi marcati immunogold 11. Mentre il sistema di flash / Reash richiede portare il fluorocromo e la diaminobenzidina nelle cellule prima di fare il fotoconversione, il sistema richiede solo miniSOG diaminobenzidina e luce e in aggiunta è circa due volte più efficace a polimerizzazione diaminobenzidina. Qui, miniSOG è impiegato in combinazione con ESI per mappare l'ultrastruttura di riparazione del DNA focolai.
Riparazione del DNA focolai (DRF)
DNA non riparato doppie rotture dei filamenti rappresentano una grave minaccia alla cella in quanto possono portare a traslocazioni e la perdita di informazioni genetiche. A sua volta, questo può portare a senescenza, il cancro, e la morte cellulare. Molte proteine che sono coinvolte nella riparazione del DNA a doppia interruzione filo accumulano nei focolai che assemblare intorno a un DNA a doppio filamento pausa 12-14. Anche se la loro funzione non è nota, rappresentano il sito nel nucleo che contiene il DNA a doppio filamento pausa ed è il sito di DNA a doppio filamento break riparazione.
Riparazione del DNA foci (DRF) sono stati caratterizzati da microscopio a fluorescenza e servono come biomarcatori per danno al DNA 12,15. Sono massiccio rispetto alle dimensioni della pausa doppio filamento e sono stati considerati relativamente omogenea fino recenti studi di microscopia super-risoluzione rivelato alcune prove di sub-compartimentalizzazione delle molecole all'interno di ogni fuoco 16. Per comprendere come sono organizzati questi siti, è necessario visualizzare tutte le strutture biologiche sottostanti rispetto all'altro. Ciò non può essere ottenuto mediante microscopia a fluorescenza, ma è possibile attraverso microscopia elettronica 17,18. Qui, viene descritto un metodo che combina elettroni spettroscopica con il metodo miniSOG per illustrare il potenziale di questo approccio combinato di esplorare l'ultrastruttura di DNA a doppio filamento break riparazione.
Protocollo
1. Generazione di linee cellulari miniSOG
- Grow U2OS (osteosarcoma umano) cellule in un piatto da 35 mm contenente 2 ml di glucosio basso modificata mezzo di Eagle Dulbecco (DMEM) che è integrato con 10% di siero fetale bovino (FBS) a 37 ° C in un incubatore umidificato con 5% di CO 2 atmosfera.
- Trasfezione le cellule quando sono l'80% confluenti da lipofezione con qualità trasfezione purificato (A269 / 280 rapporto 1,8-2,0) costrutti plasmidici contenenti le sequenze per miniSOG e mCherry contrassegnati proteine di riparazione secondo il protocollo del produttore del reagente trasfezione 19. I plasmidi di espressione miniSOG possono essere ordinati dalla Tsien-Lab: http://www.tsienlab.ucsd.edu/Samples.htm
- Ordinare le cellule che esprimono la proteina ricombinante con il tag miniSOG e mCherry per fluorescenza delle cellule attivate ordinamento due giorni dopo la trasfezione. Raccogliere le cellulecon intensità di fluorescenza intermedio per evitare cellule che sono overexpressing 20.
- Cultura le cellule positive su un piatto 10 cm in 10 ml di terreno DMEM che viene supplementato con 10% FBS e 0,6 mg / ml dell'agente selezione G418 (geneticina).
- Dopo colonie visibili sono emersi sulle piastre, schermo per mCherry colonie positive utilizzando un microscopio invertito a fluorescenza e disegnare cerchi intorno a loro (sul fondo del piatto) con un pennarello indelebile. Utilizzare una lente obiettiva aria basso ingrandimento (ad esempio, 10x) e selezionare i cloni che usano una tecnica sterile grattando attentamente le colonie fuori e lentamente li succhia in una sterile, punta della pipetta 1000 ml.
- Espandere le colonie selezionate e congelare parti di essi verso il basso con il mezzo di crescita descritto al punto 1.1 supplementato con 10% dimetilsolfossido. Metti alla prova le cellule per la formazione DRF da loro crescente su sterili 18 x 18 mm 2 vetrini e esponendoli a2 Gy di radiazioni. Le cellule che esprimono stabilmente irradiati un miniSOG mCherry taggati proteina riparazione deve mostrare il motivo caratteristica tipica focale di DSB quando visualizzati con un microscopio a fluorescenza utilizzando un filtro-set per l'imaging mCherry.
Cultura 2. Cella
- Cultura cellule U2OS esprimono stabilmente la miniSOG e mCherry contrassegnati proteine di riparazione alle condizioni descritte al punto 1.1. Crescere le cellule a 80% di confluenza in un piatto fondo di vetro di diametro 35 mm contenente 2 ml di DMEM. Assicurarsi che il collante che attacca il vetrino per la plastica e la plastica utilizzata è resistente alle soluzioni acquose e alcoliche e resistente alla resina utilizzata!
- Prima di effettuare l'esperimento, delineare una piccola superficie di circa 1 mm 2 che contiene cellule che esprimono proteine ectopiche graffiando con una penna di diamante sterile utilizzando un microscopio a fluorescenza per identificare la regione di interesse. In alternativa, utilizzare un piatto fondo di vetro tcappello contiene un vetrino con un sistema a griglia di pre-inciso integrato nel vetrino.
NOTA: Se si utilizza la microscopia correlativa di alta qualità che combina ESI e fluorescenza immagini microscopiche 21, assicurarsi che lo spessore del vetrino è compatibile con gli obiettivi ad immersione olio. Dovrebbe avere spessore No. 1.5 (0,16-0,18 mm). Scegliere una zona vicino al centro del piatto per la regione da esaminare nella TEM per evitare problemi con la polimerizzazione della resina che può verificarsi in prossimità dei bordi (vedi punto 4,10-4,13).
3. DNA Damage induzione
- Irradiare le cellule con radiazioni gamma, utilizzare un farmaco radiomimetico, o indurre il danno da micro irradiazione laser su un microscopio confocale (ad esempio, come descritto in 18,22 o 23).
- In questo esempio, irradiare le cellule con 2 e 6 Gy di raggi gamma in un microirradiate cesio 137-sorgente di radiazione laser o utilizzando il sol 405 nmid laser a stato di un microscopio confocale dopo sensibilizzare le cellule per 20 minuti con 0,5 mg / ml Hoechst.
Preparazione del campione 4.
- Fissare le cellule con 1 ml di 4% paraformaldeide cautela in tampone cacodilato 0.1 M di sodio o in 0,1 M tampone fosfato pH 7,4 per 30 minuti a RT al buio.
ATTENZIONE: Paraformaldeide e cacodilato sodio sono tossici! Indossare guanti e smaltire in modo adeguato le sostanze tossiche. - Lavare le cellule due volte con 4 ml di 0,1 M tampone sodio cacodilato pH 7,4 o tampone fosfato pH 7,4.
- Trattare le cellule fissate con glicina 2 ml di 50 mM, 5 mM amminotriazolo e 10 mM cianuro di potassio cautela in 0,1 M tampone sodio cacodilato o tampone fosfato 0,1 M (pH 7,4) (controllare il pH!) Per 30 minuti per bloccare non reagito gruppi aldeidici e per sopprimere le specie reattive dell'ossigeno generati da gruppi eme, che può aumentare il fondo.
ATTENZIONE: cianuro di potassio è estremamente tossico! Wguanti orecchio, lavorano sotto un cappuccio e smaltire adeguatamente le sostanze tossiche. La reazione è molto sensibile al pH, quindi è importante che il pH verificato e preciso. - Registrare la zona che è stata selezionata al passo 2.2 in 2D o 3D su un microscopio a fluorescenza invertito, se la microscopia correlativa si desidera.
- Preparare una / ml di soluzione di cloridrato ATTENZIONE diaminobenzidina 1 mg ponendo un 10 mg diaminobenzidina cloridrato in una provetta da 2 ml. Aggiungere 980 ml di H 2 O distillata e 20 ml di acido cloridrico concentrato (11,65 M) e miscelare fino a che la soluzione è marrone chiaro e trasparente. Diluire questa soluzione 0,1 M di fosfato di sodio 0,1 M o tampone cacodilato ad una concentrazione finale di 1 mg / ml mentre regolando il pH con idrossido di sodio fino a pH tra 7,0 e 7,6 (pH 7.4 è raccomandato).
ATTENZIONE: diamminobenzidina è tossico! Indossare guanti e smaltire in modo adeguato le sostanze tossiche. - Per fotoossidazione, replace buffer con 2 ml di una / ml soluzione di cloridrato diaminobenzidina 1 mg in 0,1 M tampone sodio cacodilato o tampone fosfato. Protect questa soluzione dalla luce e raffreddarlo in ghiaccio a 4 ° C al fine di aumentare la solubilità di ossigeno. Saturare la soluzione con facendovi gorgogliare ossigeno attraverso la soluzione (utilizzare un tubo che viene inserito nella soluzione e collegato ad una bombola di ossigeno). Controllare di nuovo il pH e regolare se necessario.
NOTA: E 'fondamentale per la reazione fotossidazione che il pH è compreso tra pH 7,0 e pH 7,6. - Montare il piatto con le cellule fissate con cura su un microscopio a fluorescenza invertito dotato di una lente obiettivo 40x olio di immersione e spostarlo l'area di interesse. Excite il tag miniSOG con luce blu utilizzando cubi filtro per GFP o PCP. MiniSOG ha un massimo di eccitazione a 448 nm con una spalla a 473 nm 10.
- Continuare con l'illuminazione, anche dopo il verde miniSOG fluorescenza ettaris completamente scomparsi e finché il prodotto fotoossidazione marrone della diaminobenzidina polimerizzata emerge nel canale luce trasmessa. Quando il prodotto di ossidazione è visibile nella maggior parte delle cellule, disattivare l'illuminazione a fluorescenza per arrestare la foto-ossidazione.
NOTA: Foto-ossidazione può richiedere diversi minuti a seconda della lente dell'obiettivo, le lunghezze d'onda di trasmissione filtro e l'efficienza, e la sorgente di illuminazione. - Postfix le cellule con 1 ml di 2% glutaraldeide in 0,1 M di sodio cacodilato pH 7,4 tampone o tampone fosfato pH 7,4 per 30 min.
- Fissare le membrane delle cellule con 0,1% -0,5% tetrossido di osmio per 20 min in 0,1 M tampone sodio cacodilato pH 7,4 o 0,1 M tampone fosfato a pH 7,4.
NOTA: si consiglia di utilizzare la concentrazione più bassa possibile di tetrossido di osmio necessario per stabilizzare le membrane. Poiché i precipitati DAB polimerizzate presentano un'alta densità di azoto, che può essere rilevata direttamente da ESI, un forteosmio colorazione non è necessario per identificare la proteina miniSOG-tag e potrebbe essere dannoso per la ESI. Tetrossido di osmio è molto tossica! Lavorare in una cappa aspirante e smaltire adeguatamente i rifiuti tossici. - Disidratare le cellule attraverso una serie di etanolo usando 30%, 50%, 70%, 90%, 98% 100% etanolo (passaggi ogni 5 min). Successivamente, incubare le cellule in una miscela 1: 1 di etanolo al 100% e resina acrilica (LR bianco) e mettere il piatto su un agitatore per 4 ore per facilitare l'infiltrazione nelle cellule prima incubando le cellule per almeno un altro 4 ore a 100 resina acrilica% (LR bianco).
- Per polimerizzare la resina, tagliare il coperchio di una etichetta provetta 2 ml con una lama di rasoio e rivestire il cerchio con acceleratore resina acrilica. Assicurarsi che l'acceleratore copre solo il cerchio e non scorre nel tubo. Successivamente, compilare accuratamente il tubo di circa due terzi con resina LR bianco con una pipetta Pasteur. Fare attenzione che la resina non entrare in contatto with l'acceleratore sul cerchio!
- Rimuovere la resina che infiltrato le cellule nel piatto e posizionare il piatto capovolto sul tubo microcentrifuga permanente posizione verticale in modo che la larghezza del tubo riempie quasi completamente la finestra di osservazione di vetro rivestita del piatto.
- Attendere 1 per 2 min per l'acceleratore per sigillare il tubo e il vetrino, poi invertire il tubo in modo che la resina nel tubo ora copre le cellule.
- Porre la capsula con il tubo in un forno a 60 ° C e curare per 12 hr.
- Quando il blocco viene guarito, rimuovere il piatto con la resina-riempito provetta attaccata dal forno e separare la provetta dal piatto. Facilitare la separazione dai cicli di gelo e disgelo in azoto liquido e acqua calda.
- Eliminare il piatto fondo di vetro e tagliare con attenzione aprire la provetta con una lametta per rimuovere il blocco.
- Etichettare il blocco con un pennarello indelebile.
- Utilizzare una lama di rasoio per trim il blocco in modo che solo la regione 1 mm 2 che contiene l'area precedentemente segnato con la penna diamante o tungsteno (o contiene l'area con le cellule di interesse) rimane.
- Montare il blocco in un ultramicrotomo, tagliare il blocco con un coltello taglio e tagliare sezioni ultrasottili di circa 50 nm con un coltello di diamante.
- Raccogliete le sezioni ad alta trasmissione 300 reti mesh. Queste griglie sono molto piccoli bar griglia e coprono quindi un minor numero di cellule nella zona di interesse.
- Cappotto sezioni sulle griglie con circa 0,2-0,4 nm di carbonio utilizzando un dispositivo a induzione di carbonio per aiutarli a stabilizzare sotto il fascio di elettroni del TEM.
5. Microscopia Elettronica
- Caricare le griglie in un TEM che è dotato di un filtro di energia. Dopo aver sintonizzato il microscopio e il filtro di energia in base alle istruzioni del fabbricante, controllare le celle delle sezioni in modalità basso ingrandimento (trasmissione normale) e confrontareloro di fluorescenza dei dati quando opportuno (ottenuto nel passaggio 4.4).
- Una volta che un nucleo con caratteristiche interessanti (danni al DNA delle tracce o riparazione del DNA foci) viene trovato, passare alla modalità di filtraggio energia e registrare una mappa di spessore.
NOTA: Questa procedura include la registrazione di una perdita zero e un'immagine non filtrata. Spessori fino a 0,3 cammino libero medio (30% degli elettroni del fascio incidente viene dispersa dal campione) sono abbastanza sottile per generare buone mappe elementari. - Mappe rapporto record di fosforo e azoto elementi utilizzando il software che controlla il filtro energia del microscopio utilizzato. Registrare le immagini successive fosforo mappa giuntati a 175 eV perdita di energia con una larghezza di 20 eV feritoia e pre-edge immagini a 120 eV perdita di energia, anche con una larghezza di 20 eV feritoia. Per le mappe di azoto, registrare immagini successive giuntati a 447 eV con fenditura di 35 eV e pre-edge immagini a 358 eV, anche con una larghezza di 35 eV feritoia.
NOTA: Poiché il contenuto degli elementi di immagine (phosphOrus e azoto) è relativamente bassa (circa. 1%), scegliere "Rapporto Immagine" (qualitativa mappa elementale) sopra il "metodo 3 finestra" (quantificare mappa elementale) al fine di generare immagini con un segnale migliore di rumore.
Elaborazione 6. Immagine
- Aprire le mappe rapporto elementari in un software di elaborazione delle immagini in grado di gestire il formato file delle immagini acquisite (ad esempio, Digital Micrograph). Copiare la mappa azoto nel canale rosso e la mappa di fosforo nel canale verde di un'immagine RGB, sovrapporre e allineare le mappe.
- Esportare l'immagine composita come un file di formato di file di immagine con tag (TIFF). Apri l'immagine con un software di elaborazione di immagini che può utilizzare strati (ad esempio, Photoshop).
- Regolare la gamma dinamica di ciascun canale di riscalare i valori minimo e nell'immagine di un set di dati a 8 bit (da 0 a 255).
- Sottrarre il fosforo dalla mappa di azoto (utilizzando laFinestra "livelli") al fine di generare una mappa qualitativa che mostra la distribuzione delle proteine.
- Convertire le mappe del acido nucleico (fosforo) e proteine (azoto) creato nella fase precedente in "scala di colore" e creare una tabella di ricerca gialla nello spazio colore CMYK per la mappa che mostra la distribuzione degli acidi nucleici e un tavolo ciano di ricerca per visualizzare la carta non nucleoproteina. I colori sono scelti per generare il massimo contrasto tra le due mappe elementari.
- Creare una nuova immagine e importare i mappe che mostrano le distribuzioni di fosforo e proteine come diversi strati. Utilizzare la modalità di trasparenza "schermo" per vedere entrambi gli strati sovrapposti l'uno sull'altro.
- Aprite l'immagine a perdita zero acquisito nel passaggio 5.2. Questa immagine rappresenta un'immagine dell'area registrata che assomiglia un'immagine TEM convenzionale, ma contiene solo gli elettroni che sono passati attraverso il campione senza urtare il campione. Esportazionequesta immagine come immagine TIFF.
- Aprite l'immagine perdita pari a zero esportato in un editor di foto e copiarlo nella strato superiore della immagine che mostra la mappa elementare. Allineare l'immagine utilizzando la modalità di trasparenza "schermo".
- Facoltativamente soglia dell'immagine perdita zero per segmentare il segnale che proviene dal miniSOG. Aggiungere l'immagine risultante come livello separato come descritto sopra.
Risultati
ESI
Confrontando le immagini ESI del nucleo (figura 1) con le immagini TEM convenzionali (ad esempio, la Figura 7-1) rivela un drammatico aumento strutture anatomiche che possono essere facilmente distinguibili. Le creste cromatina appaiono in giallo ed è abbastanza facile identificare le chromocenters in cellule di topo. Nucleoli può essere facilmente distinta dalla cromatina dalla loro struttura rotonda e colore diverso, dal momento che i ribosomi premontati già contengono elevate quantità di proteine, che è ricco di azoto, oltre al RNA, che è ricco di fosforo. La cromatina periferica risiede in uno strato sottile al confine tra nucleo e pori nucleari può essere visto come strutture ricchi di azoto che interrompono la cromatina periferica. Spesso la lamina può essere vista come uno strato molto sottile blu di fuori della cromatina periferica. Nel citoplasma, molte piccole particelle ricche di fosforo puòessere visto. Questi possono essere identificati come ribosomi base alla loro dimensione, l'abbondanza e la posizione al di fuori del nucleo. Lo spazio interchromatin può essere vista come la zona ricca di proteine situato tra la cromatina. Questo contiene corpi e zone ricche di piccoli segnali gialli, come particelle ribonucleoproteiche e grappoli di granuli ribonucleoproteiche (Figura 2) nucleari. Questi ultimi sono definiti interchromatin cluster granuli e sono tipicamente arricchiti in proteine necessarie per la pre-mRNA splicing. Figura 1B, C e D mostrano la comparsa di altre strutture note come cromosomi mitotici, centrosomi, mitocondri e centromeri. Nella figura 2, i passaggi utilizzati per generare immagini a colori compositi dalle prime mappe rapporto elementari sono riassunti. Unire la mappa di fosforo (verde) e la mappa di azoto (rosso) in spazio colore RGB (riga superiore) consente una migliore valutazione degli importi di questi elementi nel campione. HoweveR, sottrarre il segnale di fosforo per esaurire il contributo delle strutture contenenti acidi nucleici dalla mappa di azoto per produrre la mappa proteine (ciano) e la fusione con la mappa di fosforo (giallo) nello spazio colore CYMK fornisce una rappresentazione visiva più distinta di nucleoproteina e non nucleoproteina (fila in basso). Questo permette ai dati di essere più facilmente interpretati da individui non hanno familiarità con la tecnologia.
Laser cellule che esprimono microirradiated MDC1, 53BP1 e RAD52
Grazie al definita dall'utente dimensioni, forma e posizione delle tracce danni laser, la regione di DAB deposito nelle cellule laser microirradiated sono molto facili da trovare nel TEM quando si utilizza il sistema miniSOG. Le foto scattate in modalità di trasmissione a basso ingrandimento (vale a dire, campo chiaro convenzionale TEM) rivelano le caratteristiche tracce di danno e possono essere confrontati con i dati che sono stati registrati con un microscopio a fluorescenza prima di incorporamento.Ciò è particolarmente utile se microscopia correlativa è destinato, in quanto consente una facile identificazione e trasferimento di singoli nuclei. (Figura 2-4).
Il primo esempio (figura 3) mostra una linea di cellule che esprimono stabilmente MDC1 U2OS miniSOG-mCherry. In un esperimento microirradiation laser, il reclutamento del MDC1 contenente i tag miniSOG e mChrerry era molto simile alle versioni GFP-tagged di questa proteina (dati non riportati). Che fissa le cellule 1 ora dopo l'induzione danni al DNA e preparazione del campione per il TEM, le tracce di danno MDC1 erano facilmente identificati in sezioni ultrasottili in modo TEM normale come strisce scure che attraversano i nuclei, che corrispondeva molto bene ai dati di fluorescenza ( Figura 3-5). Osservando la distribuzione della proteina, un chiaro aumento del segnale di azoto può essere osservato nei siti danneggiati laser. Va qui ricordato che questo è causato principalmente dal diami polimerizzato nobenzidine, che è ricco di azoto e amplifica il segnale del miniSOG tag proteine di riparazione, e non soltanto attraverso l'accumulo di proteine di riparazione nella traccia danni. Confrontando la struttura della cromatina nel resto del nucleo con la struttura nelle tracce danni mostra una chiara differenza. La cromatina danneggiato (che è mostrato all'interno delle linee tratteggiate in figura 3) appare più decondensazione in contrasto con la cromatina non irradiati, che si verifica in spessi creste nucleoplasma. All'interno dei percorsi di danno, potrebbero anche essere osservati corpi nucleari (200-300 nm) (evidenziate da frecce in figura 3 II2, IIb, IIc e IIIa), che sono ricchi di proteine MDC1-miniSOG ma privo di acidi nucleici. Sarebbe molto difficile definire questi organismi nucleari come strutture senza cromatina senza questa tecnica. Suggerisce anche un ulteriore livello di complessità per i siti di danno che non è previsto dalla biochimica noto di MDC1.
S copi "> Guardando le tracce danni di 53BP1 (figura 4) che sono stati creati utilizzando le stesse condizioni del danno precedentemente descritto in cellule MDC1-trasfettate, risultati molto simili può essere osservato. Il segnale che è stato prodotto dalla foto-ossidazione la miniSOG era affollato e riempito lo spazio tra le pieghe della cromatina. corpi nucleari macchiata pesantemente potuto essere osservate (indicata da una freccia in figura 4 D, E, F).Da esperimenti con costrutti RAD52-GFP, è noto che RAD52 formerà piccole microcompartements luminose 24 lungo un danno pista indotta da laser. A causa del limite di risoluzione dei microscopi luminose convenzionali, è stato chiaro quanto grande queste strutture sono davvero e il loro rapporto con il DNA danneggiato. Guardando l'immagine TEM di irradiate laser nuclei U2OS costitutivamente esprimenti RAD52-miniSOG-mCherry, le dimensioni di questi corpi sono stati misurati per essere in media (150-250 nm). esso èancora più sorprendente un'occhiata a questi focolai con ESI. In contrasto con gli esempi precedenti, le foci RAD52-miniSOG-mCherry sembrano essere organizzato in modo diverso lungo la pista danni e sembrano essere le strutture del corpo come nucleari compatti che sono costituiti da proteine e non hanno acidi nucleici percepibili loro interno. In base a guardare il rapporto / proteine-acidi nucleici nelle aree danneggiate ottenuti con il nostro approccio miniSOG e ESI combinato, suggeriamo che la riparazione del DNA può avvenire sulla superficie dei fuochi, piuttosto che al suo interno.
L'immagine inferiore Figura 5 mostra i risultati simile alla Figura 3-III. La cromatina nella zona danneggiata sembra essere più decondensazione rispetto alla cromatina nelle zone non irradiate (vedi area delineata da trattini rossi).
Cellule irradiate Gamma
Mentre laser micro-irradiazione è uno strumento utile per quicklproteine di prova y che contengono un tag fluorescenti per l'assunzione a siti di lesioni del DNA, che crea grandi quantità di danni complesso (rotture del doppio filamento, rotture singolo filamento, danno base, e dimeri cyclopyrimidine) 25. Per studiare i vani che si formano più direttamente intorno DNA rotture del doppio filamento, abbiamo esaminato DRF che si formano intorno individuali DSB esponendo le cellule a raggi gamma.
Cellule che esprimono stabilmente U2OS 53BP1-miniSOG-mCherry sono stati esposti a 2 Gy di raggi gamma e fissati mezz'ora dopo l'irradiazione. Il modello caratteristico di grandi focolai distribuiti su tutto il nucleoplasma potrebbe essere osservato (Figura 6Ia). Dopo fotossidazione di diaminobenzidina, macchie scure emergenti nelle posizioni in cui si trovavano i segnali fluorescenti in mCherry micrografie fluorescenza potevano vedere nelle micrografie elettroniche (Figura 6iB e c). Queste macchie possono essere correlati a TEM immaginiche sono stati presi a basso ingrandimento (Figura 6Id ed e). Le immagini ESI di questi focolai, che sembrava essere circa 1-1,5 micron di diametro, ha mostrato una struttura interessante. Fili sottili di cromatina sembravano essere intervallati da proteine 53BP1 di circa il doppio dello spessore.
In altri esperimenti, le cellule sono state esposte ad una maggiore quantità di radiazioni e fissati a successivi punti temporali (dopo 3 ore e 6 ore, rispettivamente) per generare foci grandi e facilitare localizzarli nelle sezioni ultra-sottili (Figura 7). Poiché siamo in grado di mappare separatamente nucleoproteina e proteine, ESI rivela che la struttura dei foci sembrano riorganizzazione nel tempo. La quantità relativa di proteine 53BP1 nel fuoco sembra aumentare mentre la cromatina assume una posizione più periferica.
Poiché 53BP1 foci appaiono anche in cellule come "focolai criptico" in cellule G1 in siti di sotto-replica non irradiatid DNA 26,27, i focolai sono stati anche ripreso. Queste criptici foci sembrano nutrire elevate quantità di proteine rispetto ai fuochi radiazioni indotta e mostrano una interessante struttura regolare della cromatina.
Così, la visualizzazione di riparazione del DNA costituenti foci utilizzando il metodo miniSOG aiuta a identificare regioni di danno al DNA e per mappare la distribuzione della proteina riparazione tag rispetto alla cromatina.

Figura 1. Panoramica delle strutture che possono essere osservati in un'immagine ESI Clicca qui per vedere una versione più grande di questa figura.
(A) immagine ESI di un topo fibroblasti embrionali. Creste cromatina (giallo) sono circondate da nucleoplasma (ciano) compilando il sp interchromatinasso. Il nucleo è circondato da eterocromatina periferica e chromocenters (in basso a sinistra), che sono interrotti da complessi pori nucleari (ciano). Il nucleoli può essere chiaramente separato dal loro colore. Ciò è dovuto al fatto che la quantità di proteine rispetto ad acido nucleico è superiore nella struttura nucleolare quanto lo sia nella cromatina. Al di fuori del nucleo, mitocondri e ribosomi (ricchi di acido nucleico) possono essere facilmente identificati.
(B) L'immagine di un nucleo e centrosomi (vedi frecce), che può essere visto come particelle proteiche cavi in alto a sinistra dell'immagine.
Sezione (C) Croce attraverso una piastra di metafase. I cromosomi in metafase altamente condensati sono allineati su un disco che si estende da un angolo in alto a sinistra verso l'angolo in basso a sinistra. Elevate concentrazioni di azoto sono visibili sulla superficie di alcuni cromosomi. Queste aree sono i cinetocori (vedi frecce).
D: Parti mitocondri (vedi frecce superiori) nel citoplasma, circondato dai ribosomi (piccole macchie gialle - vedi frecce inferiori). La membrana interna dei mitocondri con la sua invaginazioni tipici (creste) è visibile.

Figura 2. Creazione di un ESI quadro multicolore
(Riga superiore) Le prime due immagini della fila mostrano le mappe rapporto di fosforo e azoto. L'ultima immagine nella riga superiore mostra queste mappe elementari sovrapposti nello spazio colore RGB. Strutture giallo rappresentano nucleoproteine, riflette la presenza di fosforo e azoto.
(Fila inferiore) La prima immagine mostra la mappa di fosforo, che rivela soprattutto la distribuzione di acido nucleico nel campione. L'immagine centrale mostra la distribuzione di nucleico / stru negativo acido impoverito ctures. E 'stato calcolato sottraendo la mappa fosforo dalla mappa azoto. L'immagine in basso a destra mostra una fusione della mappa acidi nucleici e la mappa delle proteine nello spazio colore sottrattivo. Questo composito di colore dovrebbe essere utilizzato per contribuire all'identificazione delle singole strutture e di classificarli come nucleoproteina o meno nucleoproteici. Le informazioni quantitative sul fosforo e dell'azoto abbondanza dovrebbero essere raccolti visivamente dal fosforo e azoto rete rete le mappe in scala di grigi o da composti generati utilizzando colori additivi, come ad esempio nella riga superiore.

Figura 3. Laser micro irradiazione di cellule U2OS esprimono stabilmente MDC1-miniSOG-mCherry. Cliccate qui per vedere una versione più grande di questa figura.
(II) L'area, evidenziata da un quadrato verde a Ic è stato ingrandito e visualizzato in modalità normale trasmissione (IIa) e ESI (IIb). IIc mostra una sovrapposizione dell'immagine ESI con il segnale della traccia danni. Il segnale pista danno è stato ottenuto thresholding dell'immagine superiore.
(III) Esempi della struttura di micro lasercromatina -irradiated e cromatina di fuori dell'area danneggiata laser.
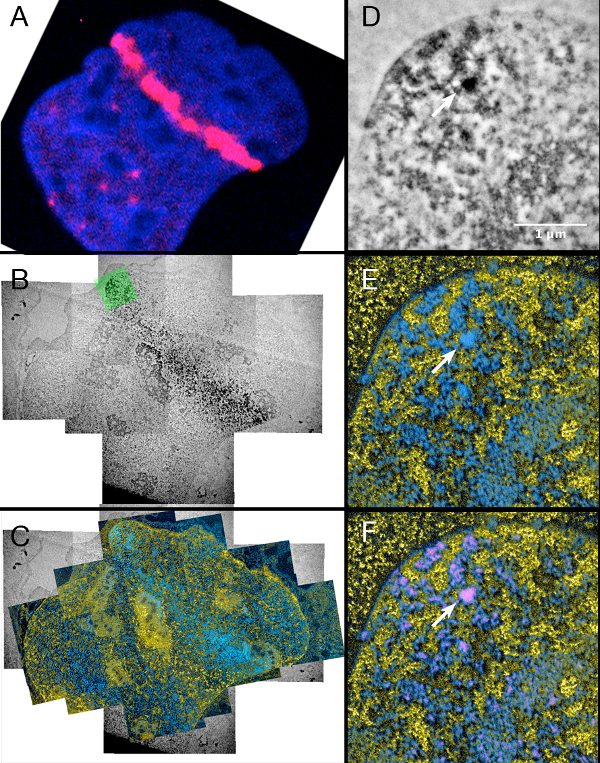
Figura 4. Laser micro irradiazione di cellule che esprimono stabilmente 53BP1-miniSOG-mCherry. Cliccate qui per vedere una versione più grande di questa figura.
Colonna sinistra: (A) fluorescenza, (B) e TEM convenzionale (C) ESI immagini con lo stesso micro laser irradiati nucleo.
Colonna destra: (D) Convenzionale TEM, (E) ESI e (F) Sovrapposizione dell'immagine ESI con il segnale miniSOG che è stata segmentata da soglia di (D) (vedi punto 6.10)

Figura linea cellulare 5. U2OS esprimono stabilmente RAD52-miniSOG-mCherry e danneggiato da laser micro-irradiazione. Cliccate qui per vedere una versione più grande di questa figura.
(A) Sovrapposizione di un'immagine di fluorescenza un'immagine TEM basso ingrandimento che mostra un laser microirradiated U2OS nucleo con una pista danno in basso a sinistra con.
(B) Rappresentante immagine microscopia confocale a fluorescenza di un micro laser irradiato nucleo Hoechst macchiati (blu) che esprime RAD52-miniSOG-mCherry che mostra il modello caratteristico di piccoli focolai lungo la (segnale rosso) traccia danni.
(C) convenzionale TEM, (D) ESI, (E) fosforo mappa, (F) e il segnale ESI segmentato da (C) Alto ingrandimento del nucleo mostrato in alto a sinistra.
(G) ESI immagine di un altro nucleo U2OS esprimono stabilmente RAD52-miniSOG-mCherry mostrando un danno traccia indotta da laser nel contesto dell'intero nucleo.

Figura 6. cellule U2OS esprimono stabilmente 53BP1-miniSOG-mCherry irradiate con 2 Gy e fissati dopo 30 min. Cliccate qui per vedere una versione più grande di questa figura.
(I) a) immagine di fluorescenza che mostra i fuochi 53BP1 raggi gamma-indotta. b) immagine luce trasmessa dopo fotopolimerizzazione. c) Overlay della fluorescenza con l'immagine luce trasmessa. d) Immagine in bassa ingrandimento TEM (la striscia nera è una barra griglia TEM). e) Sovrapposizione dell'immagine fluorescente l'immagine basso ingrandimento con TEM.
Messa a fuoco (II) danno marcato con un asterisco nell'immagine luce trasmessa registrato nel TEM normale e in modalità ESI. a) zero-perdita, b) ESI, c) fosforo, d) Proteine e) Overlay ESI e il segnale segmentato dall'immagine perdita zero.
Fuoco (III) danno marcato con una croce l'immagine di fluorescenza registrata in TEM normale e in modalità ESI in. a) zero-perdita, b) ESI, c) fosforo, d) Proteine e) Overlay ESI e il segnale segmentato dall'immagine perdita zero.
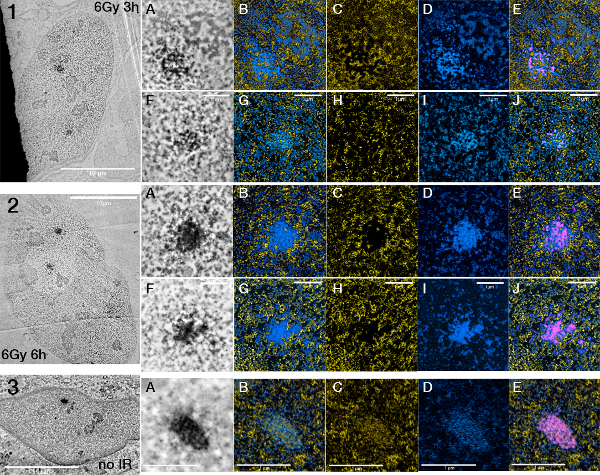
Figura 7. Cambiamenti nella struttura focolai tra foci dopo diversi momenti irradiazione. < / strong> Clicca qui per vedere una versione più grande di questa figura.
Cellule U2OS 53BP1 miniSOG mCherry esposti a 6 Gy di raggi gamma fisso dopo 3h (1A-E, FJ) e 6h (2A-E, FJ), rispettivamente. 3 mostra un nucleo non irradiato U2OS 53BP1 miniSOG mCherry mostrando una particolare attenzione criptico (AE). Quadri ESI sono presentati nell'ordine ESI (acidi nucleici e proteine), l'acido nucleico (alone), proteine (alone), ESI (acidi nucleici e proteine) + segmentati segnale miniSOG.

Figura 8. foci di riparazione del DNA sono ancora visibili in modalità di trasmissione convenzionale, quando dopo fissazione con tetrossido di osmio viene omesso. arge.jpg "target =" _ blank "> Clicca qui per vedere una versione più grande di questa figura.
Sezione ultrasottile (50 nm) di un nucleo U2OS mostra due 53BP1 foci da un campione che non è stato trattato con tetrossido di osmio. (A) In modalità TEM convenzionale, è possibile vedere i siti macchiate dal polimero DAB anche senza migliorare contrasto con il macchia l'heavy metal. (B) Il contrasto dei fuochi può essere ulteriormente migliorata prendendo immagini zero perdita (filtrare via tutti gli elettroni che origine da eventi di scattering plurale).
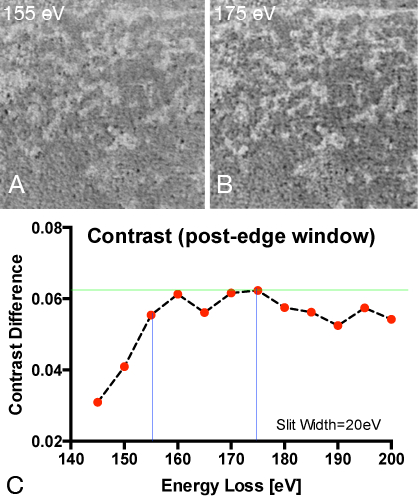
Figura 9. Determinazione delle impostazioni della finestra di energia ottimali per il fosforo di mappatura
(A) Elemental rapporto map registrato per il fosforo con il post bordo a 155 eV e pre-bordo a 120 eV.
contenuto "> (B) Rapporto Elemental mappa registrato per il fosforo con il post bordo a 175 eV e pre-bordo a 120 eV.(C) Calcolo mappe fosforo rapporto da una finestra pre-bordo costante a 120 eV perdita di energia e una finestra post-bordo variabile da perdita di energia 140-200eV per determinare la combinazione che è meglio in contrasto. La differenza intensità dei più brillanti e dimmestpixel in una linea di scansione che coprono oltre la stessa fosforo ricco e fosforo regione povera mostra valori di contrasto più alti (range dinamico) quando è stato scelto un quadro post-bordo di 175 eV perdita di energia.
Discussione
ESI può servire come un ottimo strumento per l'esame diversi stati di cromatina e ribonucleoproteine nel nucleo, perché è in grado di mappare in particolare le aree che sono ricchi di fosforo. Essa aumenta profondamente la quantità di dettagli che può essere ottenuta con un microscopio elettronico e non dipende opposti metodi non specifici. Uno svantaggio di ESI è che richiede sezioni sottili di TEM convenzionale alla stessa tensione di accelerazione. Questo può essere superato lavorando con sezioni seriali o con metodi tomografici 28.
Anche se un sacco di informazioni può essere ottenuto guardando le distribuzioni di fosforo e azoto, che è di interesse per essere in grado di mappare anche la distribuzione di alcune proteine. Con il metodo presentato in questo documento, è stato dimostrato che ESI potrebbe essere combinata con i metodi che si basano sulla polimerizzazione sito-specifica di diaminobenzidina. Anche se questo metodo non aggiunge un full nuovo colore a ESI, è chiaramente utile per mappare la distribuzione di una proteina. Ciò vale in particolare per proteine che interagiscono con la cromatina. Se uno studio ha bisogno di visualizzare più popolazioni proteina, dei metodi di etichettatura utilizzando immunogold possono comunque essere utilizzati quando applicato come colorazione pre-embedding, prima fotoossidazione 9. Rispetto alle linee cellulari stabili, trasfezioni transienti hanno lo svantaggio che i livelli di espressione possono variare notevolmente tra le cellule e quindi rendere difficile trovare più cellule che esprimono simili quantità di proteina tag. Pertanto, si raccomanda la creazione di linee cellulari stabili. Tuttavia, è possibile valutare la quantità relativa di espressione della proteina mediante fluorescenza e selezionare cellule con un'espressione intermedia seguenti trasfezione transiente.
Anche se il tag di fusione miniSOG si è fluorescente, la sua resa quantistica è significativamente peggiore rispetto GFP (0,37 vs 0.6) 10. Per il purpose della microscopia correlativa, l'aggiunta di un tag aggiuntivo proteina fluorescente come mCherry permette l'imaging in cellule vive, senza la produzione di ossigeno singoletto che si verifica con miniSOG eccitazione. Il livello di espressione delle proteine miniSOG-tag, la quantità di ossigeno nel buffer-DAB contenente, e il pH può anche influire sulla velocità di fotoossidazione. A volte può essere molto problematico per ottenere omogenea fotoossidazione nella regione di interesse a causa della dipendenza luce della reazione di fotoossidazione. Prolungato fotossidazione può portare a colorazione aspecifica e della deposizione di troppo metallo pesante nel campione, che può complicare il rilevamento di segnali specifici degli elementi da ESI. Il processo di fotossidazione richiede molto tempo in modo che di solito solo una piccola regione contenente poche cellule sul vetrino sono normalmente macchiato.
Abbiamo dimostrato la combinazione di proteine miniSOG etichettato con ESI utilizzando un monostrato di un aderente hulinea di cellule di cancro nell'uomo. In linea di principio, il metodo può essere applicato a qualsiasi altro campione (lievito, batteri, tessuti, embrione) fintanto che è possibile esprimere la proteina miniSOG-tag a livelli ragionevoli e fintanto traslucidità e la diffusione di ossigeno e DAB è sufficientemente elevato per consentire una corretta fotoossidazione e quindi la colorazione omogenea. Per cellule in sospensione, sarebbe vantaggioso per fissare la posizione di cellule su un vetrino con un adesivo, come poli-L-lisina. Per i campioni più spessi, gli obiettivi con apertura numerica inferiore raggiungeranno un'illuminazione più omogenea ma richiedere più tempo per foto-ossidare.
Abbiamo presentato l'uso di proteine di fusione miniSOG-tag più vicino al protocollo originale pubblicato da Shu et al 10 possibile -. Compreso l'uso di tetrossido di osmio. Ci si può osservare che, oltre preferenzialmente depositarsi sulla DAB-polimeri e membrane, tetrossido di osmio mostra alcuni reattività edeposizione su gran parte del materiale biologico nel campione (10 Figura 3). Tetrossido di osmio ha un vantaggio elementare a 45 eV. Questo segnale ei suoi plasmoni possono essere sovrapposti sullo spettro perdita di energia degli elettroni come lo spessore del provino aumenta e singoli elettroni subiscono più eventi di perdita di energia durante il passaggio attraverso il campione. Ciò può limitare la qualità delle mappe elementari fosforo se vengono utilizzate alte OsO 4 concentrazioni nella preparazione dei campioni. Le concentrazioni che abbiamo usato qui, che sono significativamente inferiori a quello che è stato utilizzato in precedenza 10, ha permesso la generazione di buone mappe fosforo qualità. In un esperimento utilizzando miniSOG tagged proteine che esige maggiori OsO 4 concentrazioni, la migliore concentrazione che consente ancora una buona fosforo creazione di mappa dovrà essere determinato empiricamente.
Abbiamo trovato che il polimero DAB che si forma alla foci riparazione del DNA è sufficiente denso per essere visto in modo TEM convenzionale (o in modalità a perdita zero con ancora maggior contrasto) anche quando post-fissazione con tetrossido di osmio è omesso (vedere Figura 8). La segmentazione dei segnali che sono stati registrati in questo modo è così robusta come con i campioni che sono stati trattati con l'osmio. A seconda della proteina nucleare che deve essere visualizzato, le dimensioni delle strutture forma e la necessità di visualizzare i sistemi a membrana, l'uso di tetrossido di osmio potrebbero non essere necessarie e la sua omissione rimuoverà il potenziale di mascherare la firma elementale di fosforo.
Rispetto ad altre pubblicazioni che utilizzano ESI 18,29,30 abbiamo utilizzato diverse impostazioni per le immagini pre e post-edge per generare mappe rapporto elementare con la minor quantità di rumore. Abbiamo determinato empiricamente le impostazioni presentati nel manoscritto (vedi figura 9). Le impostazioni scelte intorno ai bordi dovrebbero portare a correggere elemappe mentali meno che le finestre di raccolta si sovrappongono con altri elementi presenti nel campione. Nel nostro caso, lo zolfo può essere un tale elemento. Poiché la concentrazione di zolfo (che si verifica nel aminoacidi metionina e cisteina) è molto bassa e contribuisce alla mappa calcolato solo in quantità trascurabile, riteniamo che il migliore rapporto S / N che abbiamo ottenuto utilizzando le impostazioni utilizzate qui superino gli svantaggi che potrebbe derivare da tracce di zolfo nel campione.
Abbiamo rivelato l'ultrastruttura di DRF a risoluzioni nanometri utilizzando le proteine di riparazione del DNA MDC1, 53BP1 e RAD52 in combinazione con tecniche di ESI e TEM. L'organizzazione della cromatina e individuale localizzazione delle proteine di riparazione DSB è stata risolta utilizzando questo approccio. È stato mostrato loro localizzazione in domini ricchi di lesioni del DNA seguenti laser micro-irradiation DNA e la tendenza di proteine di riparazione per riempire lo spazio tra le creste cromatina. Nel caso di RAD52, che plays un ruolo nella ricombinazione omologa, la formazione di strutture sferiche nucleari corpo simile lungo il binario danneggiata laser potrebbe essere osservato e questo era in contrasto con gli altri due proteine testate. Quando il danno è applicata con raggi gamma, 53BP1 formò focolai intorno alla cromatina danneggiata. La composizione e la posizione relativa della cromatina e proteine nel foci sembra cambiare nel tempo. Il rapporto tra queste proteine e la cromatina danneggiata così come i cambiamenti nell'organizzazione della cromatina e la composizione degli organi nucleari può essere facilmente ottenuto con la tecnica della ESI, mentre la microscopia in campo chiaro convenzionale utilizzando uranio e piombo come agenti contrastanti non possono valutare direttamente queste proprietà. Così, questa tecnica mostra un elevato potenziale per lo studio delle relazioni struttura-funzione, in particolare per processi che agiscono sul DNA.
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
We thank Dr. Roger Tsien for providing us with the miniSOG constructs. Dr. Xuejun Sun for help with the TEM. Lisa Lem, and Peter Shipple from the Cross Cancer Institute for supplying oxygen. Hilmar Strickfaden holds a postdoctoral fellowship by the Alberta Cancer foundation and was supported by the Bayrische Forschungsallianz. This work was supported by grants from the Canadian Institutes of Health Research and Alberta Cancer Foundation.
Materiali
| Name | Company | Catalog Number | Comments |
| 35 mm glass bottom dishes | MatTek | P35G-1.5-14-C | |
| Gridded 35 mm glass bottom dishes | MatTek | P35G-2-14-CGRD | not made for immersion objectives |
| LR White: acryl resin | emsdiasum | 14381 | |
| LR White accelerator | emsdiasum | 14385 | |
| 3,3′-Diaminobenzidine tetrahydrochloride 10 mg tablets | Sigma | D5905 | |
| Slim Bar Grids 300 Mesh | SPI | 1161123 | |
| Tungsten-Point Lab Pen | emsdiasum | 41148 | |
| Osmium Tetroxide | emsdiasum | 19100 | |
| Carbon Coater 208 carbon | Cressington | ||
| Ultra microtome Leica EM UC6 | Leica | ||
| Photoshop CS5 | Adobe | might even work with older versions | |
| Digital Micrograph V.2.30 | Gatan | might even work with older versions | |
| Hoechst 33342 | Sigma | H-1399 | |
| Effectene | Quiagen | ||
| MiniSOG-Constructs | Tsien-Lab | tsienlab@yahoo.com | |
| MDC1 miniSOG mCherry | |||
| 53BP1 miniSOG mCherry | |||
| Rad52 miniSOG mCherry | |||
| cesium 137 radiation source "MARK 1" | (J.L. Shepherd & Associated) | ||
| ImageJ/FiJi | open source | http://fiji.sc/Fiji | |
| 2 ml Eppendorf tubes | Fisherbrand | 05-408-146 | |
| Diamond Knife ultra 35° | Diatome | ||
| Trimming Knife ultratrim | Diatome | ||
| Sodium cacodylate trihydrate | emsdiasum | 12300 | Caution Toxic! |
| Glutaraldehyde EM Grade 8% | emsdiasum | 16020 | Caution Toxic! |
| Sodium phosphate dibasic | emsdiasum | 21180 | |
| Sodium phosphate monobasic | emsdiasum | 21190 | |
| Paraformaldehyde | emsdiasum | 19202 | |
| Osmium tetroxide 4% solution | emsdiasum | 19150 | |
| Inverted Fluorescence Micoscope Axiovert 200M | Zeiss | ||
| Hydrochloric Acid | Fisherbrand | A142-212 | |
| Sodium Hydroxide Solution 10M | Fluka | 72068 | |
| Oxygen | Medigas | ||
| 3-Amino-1,2,4-triazole | Sigma | A8056 | |
| Potassium cyanide | Sigma | 207810 | Caution Toxic! |
| Ethanol | emsdiasum | 15058 | Caution Toxic! |
| Razor blade Single Edge Carbon Steel | emsdiasum | 71960 | Caution Sharp! |
| DMEM | Sigma | D 5546 | |
| FBS | Life Technologies | 16000-044 | |
| G418 | Life Technologies | 11811-023 | |
| DMSO | Sigma | D2650 | |
| Transmission Electron Microscope 200 kV | JEOL | 2100 | |
| GIF Tridiem 863 Energy filter | Gatan |
Riferimenti
- Schermelleh, L., Heintzmann, R., Leonhardt, H. A guide to super-resolution fluorescence microscopy. The Journal of Cell Biology. 190, 165-175 (2010).
- Bazett-Jones, D. P., Hendzel, M. J., Kruhlak, M. J. Stoichiometric analysis of protein- and nucleic acid-based structures in the cell nucleus. Micron (Oxford, England: 1993). 30, 151-157 (1999).
- Michael J Hendzel, F. M. B., Bazett-Jones, D. P. Direct Visualization of a Protein Nuclear Architecture. Molecular biology of the cell. 10, 2051(1999).
- Ottensmeyer, F. P., Andrew, J. W. High-resolution microanalysis of biological specimens by electron energy loss spectroscopy and by electron spectroscopic imaging. Journal of ultrastructure research. 72, 336-348 (1980).
- Leapman, R. D., Ornberg, R. L. Quantitative electron energy loss spectroscopy in biology. Ultramicroscopy. 24, 251-268 (1988).
- Simon, G. T. Electron spectroscopic imaging. Ultrastructural pathology. 11, 705-710 (1987).
- Egerton, R. Electron Energy-Loss Spectroscopy in the Electron Microscope. , Springer. (2011).
- Aronova, M. A., Kim, Y. C., Zhang, G., Leapman, R. D. Quantification and thickness correction of EFTEM phosphorus maps. Ultramicroscopy. 107, 232-244 (2007).
- Hopkins, C., Gibson, A., Stinchcombe, J., Futter, C. Chimeric molecules employing horseradish peroxidase as reporter enzyme for protein localization in the electron microscope. Methods in enzymology. 327, 35-45 (2000).
- Shu, X., et al. A Genetically Encoded Tag for Correlated Light and Electron Microscopy of Intact Cells, Tissues, and Organisms. PLoS biology. 9, e1001041(2011).
- Gaietta, G., et al. Multicolor and electron microscopic imaging of connexin trafficking. Science (New York, NY). 296, 503-507 (2002).
- Fernandez-Capetillo, O., Celeste, A., Nussenzweig, A. Focusing on foci: H2AX and the recruitment of DNA-damage response factors. Cell cycle (Georgetown, Tex). 2, 426-427 (2003).
- Haaf, T., Golub, E. I., Reddy, G., Radding, C. M., Ward, D. C. Nuclear foci of mammalian Rad51 recombination protein in somatic cells after DNA damage and its localization in synaptonemal complexes. Proceedings of the National Academy of Sciences of the United States of America. 92, 2298-2302 (1995).
- Maser, R. S., Monsen, K. J., Nelms, B. E., Petrini, J. H. hMre11 and hRad50 nuclear foci are induced during the normal cellular response to DNA double-strand breaks. Molecular and Cellular Biology. 17, 6087-6096 (1997).
- Bekker-Jensen, S., Mailand, N. Assembly and function of DNA double-strand break repair foci in mammalian cells. DNA repair. 9, 1219-1228 (2010).
- Chapman, J. R., Sossick, A. J., Boulton, S. J., Jackson, S. P. BRCA1-associated exclusion of 53BP1 from DNA damage sites underlies temporal control of DNA repair. Journal of cell science. 125, 3529-3534 (2012).
- Dellaire, G., Kepkay, R., Bazett-Jones, D. P. High resolution imaging of changes in the structure and spatial organization of chromatin, gamma-H2A.X and the MRN complex within etoposide-induced DNA repair foci. Cell cycle (Georgetown, Tex). 8, 3750-3769 (2009).
- Kruhlak, M. J., et al. Changes in chromatin structure and mobility in living cells at sites of DNA double-strand breaks. The Journal of Cell Biology. 172, 823-834 (2006).
- Goodson, H. V., Dzurisin, J. S., Wadsworth, P. Generation of stable cell lines expressing GFP-tubulin and photoactivatable-GFP-tubulin and characterization of clones. Cold Spring Harbor protocols. 2010, (2010).
- Zeyda, M., Borth, N., Kunert, R., Katinger, H. Optimization of sorting conditions for the selection of stable, high-producing mammalian cell lines. Biotechnology progress. 15, 953-957 (1999).
- Dellaire, G., Nisman, R., Bazett-Jones, D. P. Correlative light and electron spectroscopic imaging of chromatin in situ. Methods in enzymology. , 375-456 (2004).
- Campbell, S., Ismail, I. H., Young, L. C., Poirier, G. G., Hendzel, M. J. Polycomb repressive complex 2 contributes to DNA double-strand break repair. Cell cycle. 12, 2675-2683 (2013).
- Mortusewicz, O., et al. Recruitment of RNA polymerase II cofactor PC4 to DNA damage sites. J Cell Biol. 183, 769-776 (2008).
- Bekker-Jensen, S., et al. Spatial organization of the mammalian genome surveillance machinery in response to DNA strand breaks. The Journal of Cell Biology. 173, 195-206 (2006).
- Ferrando-May, E., et al. Highlighting the DNA damage response with ultrashort laser pulses in the near infrared and kinetic modeling. Frontiers in genetics. 4, 135(2013).
- Lukas, C., et al. 53BP1 nuclear bodies form around DNA lesions generated by mitotic transmission of chromosomes under replication stress. Nature. 13, 243-253 (2011).
- Harrigan, J. A., et al. Replication stress induces 53BP1-containing OPT domains in G1 cells. The Journal of Cell Biology. 193, 97-108 (2011).
- Aronova, M. A., et al. Reprint of 'Three-dimensional elemental mapping of phosphorus by quantitative electron spectroscopic tomography (QuEST). J. Struct. Biol. 161, 322-335 (2007).
- Fussner, E., et al. Constitutive heterochromatin reorganization during somatic cell reprogramming. The EMBO journal. 30, 1778-1789 (2011).
- Kepkay, R., Attwood, K. M., Ziv, Y., Shiloh, Y., Dellaire, G. KAP1 depletion increases PML nuclear body number in concert with ultrastructural changes in chromatin. Cell cycle. 10, Georgetown, Tex. 308-322 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon