Method Article
In vitro Sintesi di mRNA modificata per l'induzione di espressione della proteina nelle cellule umane
In questo articolo
Riepilogo
In questo articolo video, si descrive la sintesi in vitro di mRNA modificata per l'induzione dell'espressione proteica nelle cellule.
Abstract
La consegna esogena di codifica messenger RNA sintetico (mRNA) per l'induzione della sintesi proteica nelle cellule desiderate ha un enorme potenziale nel campo della medicina rigenerativa, biologia cellulare di base, il trattamento di malattie e riprogrammazione delle cellule. Qui, descriviamo un passo per passo protocollo per la produzione di mRNA modificato con ridotto potenziale di attivazione immunitaria e una maggiore stabilità, controllo di qualità di prodotti mRNA, trasfezione di cellule con mRNA e la verifica della espressione proteica indotta mediante citometria a flusso. Fino a 3 giorni dopo una singola trasfezione con eGFP mRNA, le cellule HEK293 trasfettate producono eGFP. In questo articolo video, la sintesi di mRNA eGFP è descritto come esempio. Tuttavia, la procedura può essere applicata per la produzione di altri mRNA desiderati. Utilizzando il sintetico modificato mRNA, le cellule possono essere indotte ad esprimere transitoriamente le proteine desiderate, che normalmente non avrebbero esprimere.
Introduzione
Nelle cellule, la trascrizione di RNA messaggero (mRNA) e la seguente traduzione di mRNA in proteine desiderate assicurare il corretto funzionamento delle cellule. Malattie genetiche ereditarie o acquisite possono portare a insufficiente e disfunzionale sintesi delle proteine e causare gravi malattie. Pertanto, un nuovo approccio terapeutico per indurre la produzione di proteine mancanti o difettosi è la consegna esogena di sintetica modificata mRNA in cellule, che codifica per la proteina desiderata. In tal modo, le cellule vengono attivate per sintetizzare le proteine funzionali, che normalmente non possono produrre o sarebbe naturalmente non hanno bisogno. Usando questo approccio, malattie genetiche possono essere corretti dalla introduzione di mRNA che codifica per la proteina difettosa o mancante 1. La terapia mRNA può essere utilizzato anche per la vaccinazione di sintetizzare antigeni proteici che sono espressi dalle cellule tumorali o patogeni. In tal modo, il sistema immunitario dell'ospite può essere attivato per eliminare efficacemente le cellule tumorali o impedire infeZIONI 2,3. Inoltre, negli ultimi anni, mRNA è stato usato con successo per generare cellule staminali pluripotenti indotte (iPSCs). A tal fine, fibroblasti sono state trasfettate con mRNA di indurre l'espressione di fattori di riprogrammazione 4-6 e convertirli in iPSCs con un enorme potenziale in medicina rigenerativa.
In precedenza, l'uso di mRNA convenzionale è stata associata con scarsa stabilità e forte immunogenicità. In questo modo, le applicazioni cliniche di mRNA convenzionali erano limitate. Tuttavia, la sostituzione di citidina e uridina da 5-methylcytidine e pseudouridina all'interno della molecola di mRNA mediante Kariko e colleghi reso molecole di mRNA stabili nei fluidi biologici e drammaticamente ridotto attivazione immunitaria 7-10, che ora permette l'applicabilità clinica di mRNA modificati.
Utilizzando mRNA modificati sinteticamente prodotti, le trascrizioni del gene desiderato può essere recapitato temporaneamente in vivo 11o in vitro per indurre l'espressione della proteina. L'mRNA introdotto è tradotto in condizioni fisiologiche dalla macchina di traduzione cellulare. A causa della mancanza di integrazione nel genoma della cellula ospite, rispetto ai vettori di terapia genica virale, il rischio di oncogenesi è impedito 12,13. Pertanto, la terapia con mRNA sintetico modificato andrà meglio accettazione clinica nel futuro.
Qui, descriviamo un protocollo dettagliato per la produzione di mRNA modificato (Figura 1), trasfezione delle cellule con mRNA e la valutazione di espressione della proteina in cellule trasfettate.
Protocollo
1. Aumento di plasmidi contenenti richiesto codifica sequenze di DNA (CDS)
- Medio pre-SOC calda, che è incluso nel kit di trasformazione, a temperatura ambiente e le piastre di agar LB contenenti 100 ug / ml di ampicillina a 37 ° C. Equilibrare il bagno d'acqua a 42 ° C.
- Scongelare un flacone di chimicamente componente E. coli su ghiaccio.
- Aggiungere 1-5 ml di plasmide (10 pg a 100 ng) contenente i CDS nel flaconcino di chimicamente componente E. coli e mescolare delicatamente. Non mescolare le cellule pipettando. Dopo aver aggiunto plasmidi, Mescolare agitando delicatamente il tubo.
- Incubare la miscela in ghiaccio per 30 min.
- Heat-shock E. coli per 30 sec a 42 ° C in un bagno d'acqua senza agitazione.
- Posizionare la fiala in ghiaccio per 2 min.
- Aggiungere 250 microlitri di media SOC pre-riscaldato a E. coli e agitare i batteri orizzontalmente a 300 rpm per 1 ora a 37 ° C in un incubatore batterica.
- Distribuire 100 ml e 150 ml di trasformazione mix su piastre di agar LB pre-riscaldato, capovolgere le piastre ed incubare per una notte a 37 ° C.
- Dopo colonie diventano visibili, inoculare una singola colonia di ciascuna piastra in una provetta da 15 ml contenente 5 ml di terreno LB con 100 ug / ml di ampicillina. Incubare durante la notte a 37 ° C con agitazione a 300 rpm fino a quando la cultura è alla fine del registro o fase stazionaria.
- Per la conservazione a lungo termine di trasformare E. coli, pipetta 75 ml di 100% glicerolo nel esageratamente. Aggiungere 225 ml di coltura batterica (magazzino congelato conterrà 25% glicerolo), mescolare bene e conservare il esageratamente a -80 ° C.
- Isolare i plasmidi contenenti i CDS richiesti utilizzando un kit di purificazione plasmide secondo le istruzioni del produttore.
- Determinare la concentrazione plasmide utilizzando uno spettrofotometro.
- Preparare aliquote di 20 ml e conservare a -20 ° C per un tempo prolungato.
- NOTA: Per ottenere il DNA stampo per la trascrizione in vitro (IVT), il CDS di interesse, qui eGFP, viene amplificato mediante PCR. Contemporaneamente, un poli T-coda 120 thymidines (T) viene aggiunto l'inserto utilizzando un primer reverse con T 120 estensione. In tal modo, gli mRNA generati ottenere un poli A-coda con una lunghezza fissa dopo l'IVT.
- Preparare la miscela di PCR come indicato nella tabella 1.
- Mescolare accuratamente la miscela di reazione. NOTA: prodotto della PCR può essere utilizzato in 4-5 reazioni IVT. Per aumentare la quantità di DNA stampo FPI, il volume totale della miscela PCR può essere aumentata. Diversi provette PCR contenenti ciascuna 100 microlitri miscela di PCR possono essere utilizzati per ottenere una resa sufficiente di prodotto di PCR.
- Mettere le provette per PCR in un termociclatore ed eseguire il PCR utilizzando il protocollo ciclismo PCR (Tabella 2).
- Pulire il PReazione CR utilizzando un kit di purificazione PCR secondo le istruzioni del produttore ed eluire il DNA con 20 di acqua priva di nucleasi microlitri.
- Misurare la concentrazione del DNA usando uno spettrofotometro. NOTA: La concentrazione di DNA rilevato è di circa 300 mg / ml. Pertanto, l'importo complessivo previsto dovrebbe essere ~ 6 mg. La quantità di DNA per altre proteine possono differire a seconda della lunghezza dei CDS, quantità di plasmide usato per la PCR, temperatura di appaiamento dei primer, e il numero di cicli.
- Congelare il DNA a -20 ° C per un lungo periodo o utilizzare direttamente per IVT.
3. Controllo Passo: Qualità della PCR prodotto
- Controllare la qualità del prodotto di PCR mediante elettroforesi su gel di DNA.
- Miscelare la quantità desiderata di agarosio con 1x TBE in un pallone. Per un gel di agarosio all'1%, aggiungere 0,5 g di agarosio a 50 ml di TBE 1x.
- Forno a microonde fino l'agarosio è sciolto. Non lasciate bollire. Assicurarsi che l'agarosio è completamente in soluzione.
- Aggiungere 5 ml macchia gel di acido nucleico a 50 ml di gel di agarosio.
- Versare il gel in una scatola di gel di agarosio, impostare il pettine, e attendere circa 1 ora fino a quando il gel solidificato.
- Rimuovere il pettine e posizionare la scatola gel in una vaschetta per gel.
- Mescolare 2 ml (200 ng) di 80-10,000 bp DNA-Ladder e 200 ng di prodotto di PCR ciascuna con 2 ml di tampone di caricamento 6x in un volume totale di 12 ml.
- Caricare con cautela campioni di DNA nei pozzetti del gel di agarosio.
- Utilizzando TBE 1x come tampone di corsa, eseguire il gel di agarosio a 100 V per 1 ora.
- Visualizzare le bande di DNA su un sistema di imaging (Figura 2).
4. Trascrizione in vitro (IVT)
- NOTA: Dopo la PCR, gli inserti plasmide sono amplificati e viene aggiunto un poli T-coda. Durante la IVT, informazione genetica è trascritto dal DNA all'RNA. L'mRNA trascritto generato viene utilizzato per produrre proteine nelle cellule.
- Pulire l'area di lavoro e le pipette con RNasi dsoluzione di decontaminazione. Usa PCR punte pulito e sterile a doppio filtro per pipette e cambiare frequentemente i guanti per minimizzare la contaminazione di reazione si mescola con RNasi.
- Preparare la miscela analogico NTP / cap come descritto nella Tabella 3.
- Mescolare la miscela analogico NTP / cap accuratamente su vortex e centrifugare brevemente.
- Montare la miscela di reazione IVT, come descritto nella tabella 4.
- Mescolare la miscela di reazione IVT accuratamente pipettando gentilmente su e giù. Poi centrifugare la provetta PCR brevemente per raccogliere la miscela sul fondo della provetta.
- Incubare a 37 ° C per 3 ore in un thermomixer.
NOTA: Il tempo di incubazione ottimale dipende dalla lunghezza degli inserti e l'efficienza trascrizionale dato modello. Per inserti brevi (<500 nt), un tempo di incubazione più lungo può essere vantaggioso. Un esperimento temporale corso può essere fatto per determinare il tempo ottimale di incubazione per il massimo rendimento. Pertanto, impostare le reazioni FPI, per example, per 1, 2, 3, 4, 6 ore, e notte di incubazione e determinare la quantità di mRNA. La cinetica della trascrizione in vitro per la generazione di eGFP mRNA è illustrato nella figura 3. - Per rimuovere il DNA stampo, aggiungere 1 ml di DNasi (2 U / mL) alla miscela di reazione IVT, mescolare bene e incubare 15 minuti a 37 ° C.
- Purificare la miscela di reazione utilizzando un kit di purificazione RNA secondo le istruzioni del fabbricante. Eluire l'mRNA modificato dalla membrana Spin Column due volte con 40 di acqua priva di nucleasi microlitri. NOTA: La concentrazione di RNA rilevato è di circa 1.500 mcg / ml. Pertanto, l'importo complessivo previsto dovrebbe essere ~ 120 mg. La quantità di mRNA sintetizzato per altre proteine possono differire a seconda della lunghezza del CDS e la quantità di prodotto di PCR utilizzati per IVT.
5. Il trattamento di mRNA purificato con Antartico fosfatasi
- NOTA: L'mRNA generata viene trattato con fosfatasi per rimuovere 5 'trifosfati, che possono essere riconosciuti da RIG-I e portano alla attivazione immunitaria. Inoltre, il trattamento con fosfatasi evita re-circularization in una reazione auto-ligazione.
- Aggiungere 9 ml di tampone di reazione 10x fosfatasi Antartico a 79 ml di soluzione di mRNA purificata. Successivamente, aggiungere 2 ml di fosfatasi Antartico (5 U / ml) e mescolare delicatamente il campione. Incubare la miscela di reazione a 37 ° C per 30 min.
- Purificare la miscela di reazione utilizzando un kit di purificazione RNA secondo le istruzioni del fabbricante. Eluire l'mRNA modificato dalla membrana Spin Column due volte con 50 di acqua priva di nucleasi microlitri.
- Misurare la concentrazione degli mRNA modificati utilizzando uno spettrofotometro. Verificare che il rapporto di assorbanza a 260 nm / 280 nm (A 260 / A 280) è ≥ 1,8 e il rapporto A 260 / A 230 è 2,0, che indica la purezza.
NOTA: Il rendimento totale atteso dovrebbe essere di circa 90 mg, che è sufficient per circa 36 trasfezioni mRNA. - Regolare la concentrazione di mRNA di 100 ng / ml per aggiunta di acqua priva di nucleasi.
- Aliquota della mRNA modificato in monouso aliquote necessarie per transfezioni e conservare a -80 ° C. Evitare ripetuti cicli di congelamento e scongelamento. Adeguatamente preparati e conservati mRNA è stabile per molti anni. Durante il lavoro, tenere sempre l'mRNA modificato sul ghiaccio.
6. Controllo Passo: Qualità di mRNA sintetizzato Modificato
- Controllare la qualità del mRNA modificato mediante elettroforesi su gel di RNA.
- Miscelare la quantità desiderata di agarosio con 1x TBE in un pallone. Per un gel di agarosio all'1%, aggiungere 0,5 g di agarosio a 50 ml di TBE 1x.
- Forno a microonde fino l'agarosio è sciolto. Non lasciate bollire. Assicurarsi che l'agarosio è completamente in soluzione.
- Aggiungere 5 ml macchia gel di acido nucleico a 50 ml di gel di agarosio.
- Versare il gel in una scatola gel, impostare il pettine, e attendere circa 1 ora fino a quando il gel è solisolidificati.
- Rimuovere il pettine e posizionare la scatola gel in una vaschetta per gel.
- Mescolare 3 microlitri (3 mg) di 0,5-10 kb RNA Ladder e 200 ng di sintetizzati modificato mRNA ciascuna con 7 ml di tampone di caricamento in un volume totale di 10 microlitri. La composizione del tampone di caricamento sono descritte nella Tabella 5.
- Denaturare la scala e campioni di mRNA a 70 ° C per 10 min.
- Sistemare con cura la scala ed i campioni di mRNA sul gel di agarosio 1%.
- Eseguire elettroforesi a 100 V per 1 ora con TBE 1x come tampone di corsa.
- Visualizzare la scala e bande di mRNA su un transilluminatore UV e fotografia mediante un sistema di documentazione gel (Figura 4).
7. Preparazione delle cellule per la trasfezione
- Piastra 2 x 10 5 cellule (cellule HEK293) per pozzetto della piastra 12 pozzetti.
- Incubare le cellule per una notte a 37 ° C in un incubatore cella. NOTA: Il giorno della trasfezione, le cellule devono avere reazionehed 80-90% di confluenza.
8. L'esecuzione di mRNA trasfezione di cellule
- Scongelare il mRNA modificato.
- Generare i lipoplexes per trasfezione.
- Aggiungere 25 ml (2,5 mg) di mRNA modificato e 2 ml di reagente di lipide cationico trasfezione a 473 microlitri Opti-MEM (mezzo essenziale minimo) ho ridotto medio siero per generare la miscela di trasfezione per la trasfezione di un pozzetto di una piastra 12 pozzetti. Scala i volumi in base al numero di pozzetti da transfettate. Come controllo negativo, preparare una miscela di trasfezione, senza mRNA.
- Miscelare i componenti delicatamente con una pipetta. Incubare la miscela di trasfezione a temperatura ambiente per 20 min per generare lipoplexes per trasfezione.
NOTA: La miscela di trasfezione non contiene antibiotici. Quindi, fare attenzione a garantire la sterilità durante la manipolazione delle cellule. - Lavare le cellule con 500 DPBS microlitri / bene.
- Aggiungere 500 microlitri miscela di trasfezione di un pozzetto di un pla 12 pozzettite.
- Incubare le cellule per 4 ore a 37 ° C e 5% di CO 2.
- Aspirare la miscela di trasfezione e aggiungere 1 ml di mezzo completo di coltura cellulare per le cellule.
- Incubare le cellule per 24 ore nell'incubatrice cellulare.
- Eseguire fluorescenza microscopica analisi usando un microscopio a fluorescenza (Figura 5). NOTA: Per esaminare l'espressione di altre proteine nelle cellule, che sono prodotti da trasfezione con altri mRNA che codifica eGFP mRNA, le cellule possono essere coltivate su vetrini. Poi la trasfezione può essere eseguito e le cellule può essere trattata con anticorpi appropriati e analizzato al microscopio a fluorescenza.
9. Analisi di citometria a flusso eGFP espressione in cellule
- Rimuovere terreno di coltura cellulare dai pozzetti e lavare i pozzetti con 1 ml DPBS (senza calcio e magnesio). Aggiungere 500 microlitri tripsina / EDTA per pozzetto per staccare le cellule dalla superficie delle piastre di coltura cellulare. Successivamente, aggiungere 500TNS ml per pozzetto per inattivare la tripsina.
- Centrifugare le cellule staccate per 5 min a 300 xg a temperatura ambiente. Rimuovere il surnatante lasciando solo il pellet. Risospendere il pellet di cellule in 150 microlitri soluzione di fissazione delle cellule e trasferire la sospensione cellulare in un tubo di citometria a flusso.
- Analizzare la percentuale di cellule che esprimono eGFP e l'intensità di fluorescenza utilizzando Geo medio (media geometrica dell'intensità di fluorescenza) (Figura 6).
10. Misura di espressione della proteina nel tempo
- Eseguire mRNA trasfezione di cellule in 3 pozzetti di una piastra a 12 pozzetti come descritto nel passaggio 8. Inoltre, incubare 3 pozzetti di cellule HEK293 con la miscela di trasfezione senza l'aggiunta di mRNA.
- Misurare l'espressione di eGFP 1, 2, e 3 giorni dopo la trasfezione per valutare la durata di espressione proteica (Figura 7).
Risultati
Utilizzando una pcDNA 3.3 plasmide contenente il CDS di eGFP, è stato stabilito la sintesi di mRNA modificato eGFP (Figura 1). Dopo l'inserimento di amplificazione mediante PCR e poli T-tailing, una banda chiara con una lunghezza di circa 1.100 bp viene rilevata (Figura 2). Aumentando il tempo IVT aumentata la resa di mRNA (Figura 3). Dopo l'IVT, una fascia mRNA chiaro con una lunghezza di circa 1.100 bp è stato rilevato, che corrisponde alla lunghezza di eGFP mRNA da produrre (Figura 4).
La funzionalità del generato eGFP mRNA è stato testato mediante trasfezione di cellule HEK293. A questo scopo, complessi trasfezione (lipoplexes) sono stati generati utilizzando un lipide cationico reagente di trasfezione. La trasfezione è stata effettuata con 2 x 10 5 cellule per pozzetto della piastra 12 pozzetti. La produzione di eGFP nelle cellule è stata rilevata 24 ore dopo la trasfezione tramite microscopia a fluorescenza (Figuri 5) e citometria a flusso (Figura 6).
Cellule HEK293 sono state trasfettate con eGFP mRNA. espressione di eGFP è stata determinata 1, 2, e 3 giorni dopo la trasfezione per valutare la durata di espressione proteica nelle cellule (Figura 7). Dopo 24 ore, l'espressione della proteina è stata più elevata nelle cellule. L'importo è stato ridotto di 1,6 volte ogni giorno successivo. Anche dopo 3 giorni, le cellule contenute eGFP.
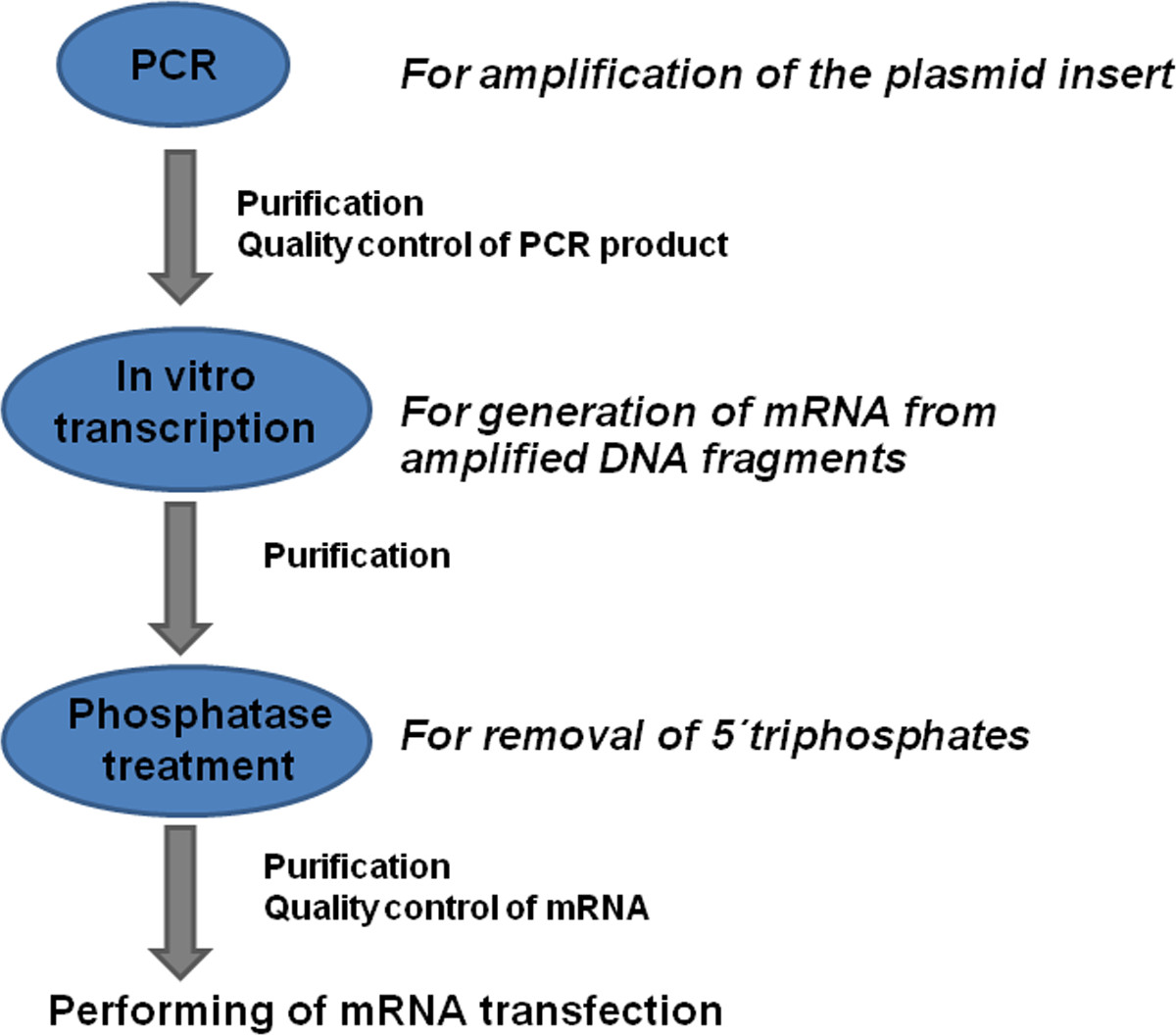
Figura 1:. Panoramica del processo di produzione di mRNA modificato codifica sequenze di DNA (CDS) con sequenze fiancheggianti note sono amplificati mediante PCR utilizzando primer specifici. Il prodotto di PCR è purificato e la qualità del DNA generato è determinata. L'mRNA è generato dal prodotto DNA usando il processo di trascrizione in vitro. Il prodotto è purifie d e trattati con fosfatasi per rimuovere 5'-trifosfati. Dopo la purificazione aggiuntiva e controllo di qualità dei generato mRNA, le trasfezioni mRNA possono essere eseguite.

Figura 2:. Analisi del prodotto di DNA dopo la PCR DNA ladder e prodotti di PCR sono stati analizzati su un gel di agarosio 1%. Una banda di DNA libero con una lunghezza di circa 1.100 bp deve essere rilevato.

Figura 3:. Cinetica di trascrizione in vitro per la produzione di mRNA eGFP 1) RNA scaletta e IVT prodotti dopo 2 min) 0, 3) 10 min, 4) 30 min, 5) 180 min, 6) 360 min, e 7) solo stampo di DNA sono stati analizzati su un gel di agarosio 1%.
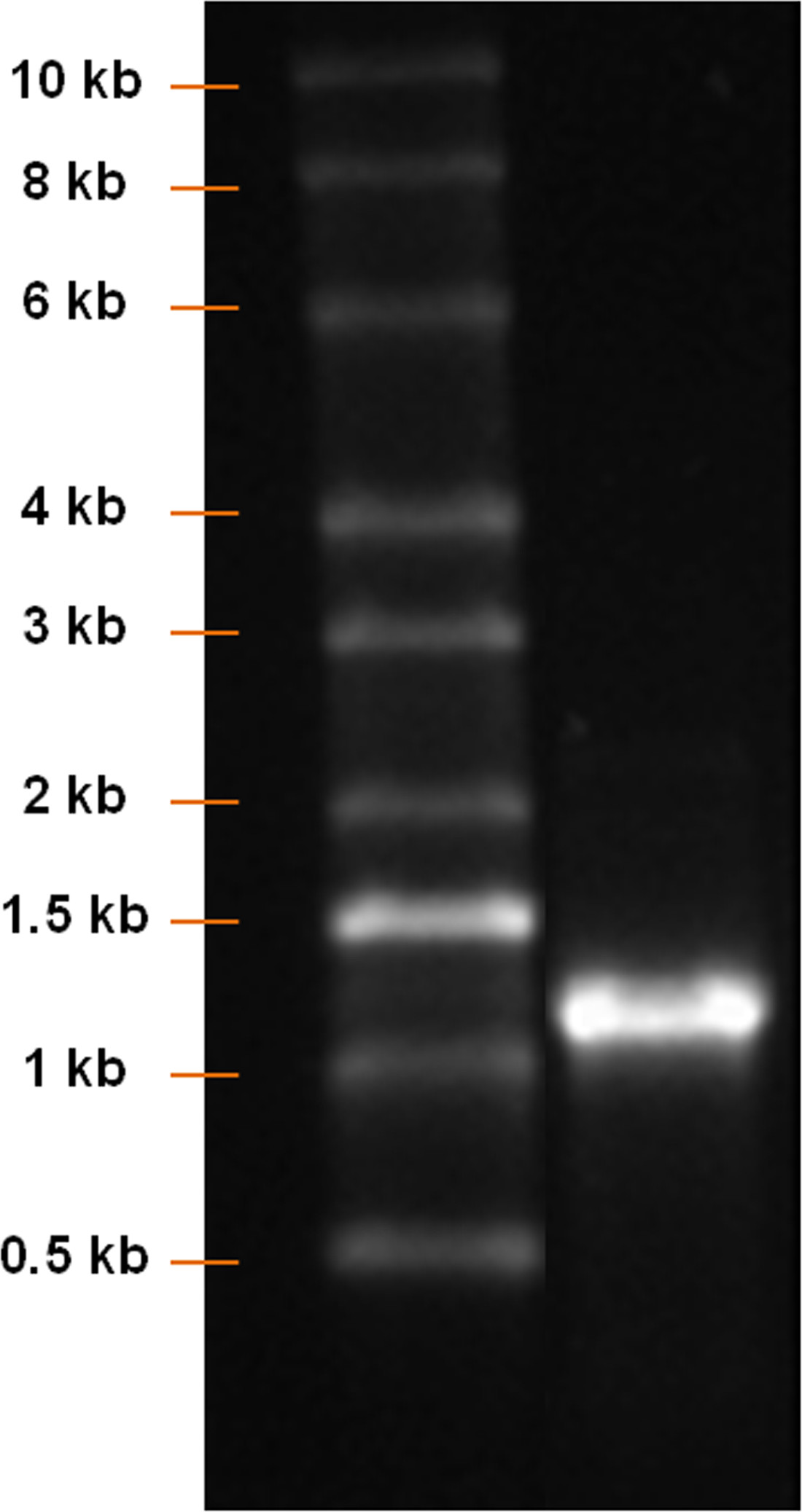
Figura 4:. Analisi del prodotto mRNA dopo IVT prodotto RNA scala e IVT sono stati analizzati su un gel di agarosio 1%. Una banda mRNA chiaro con una lunghezza di circa 1.100 bp deve essere rilevato.
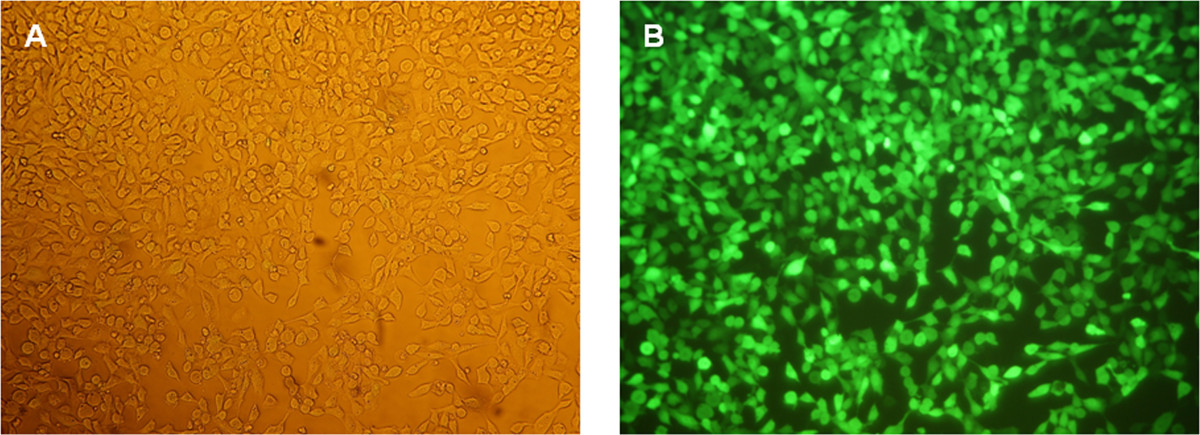
Figura 5: fluorescenza analisi microscopiche di cellule HEK293 24 ore dopo eGFP mRNA trasfezione. Immagine (A) Fase contrasto delle cellule con un ingrandimento di 100X. (B) immagini di fluorescenza di cellule con un ingrandimento di 100X.
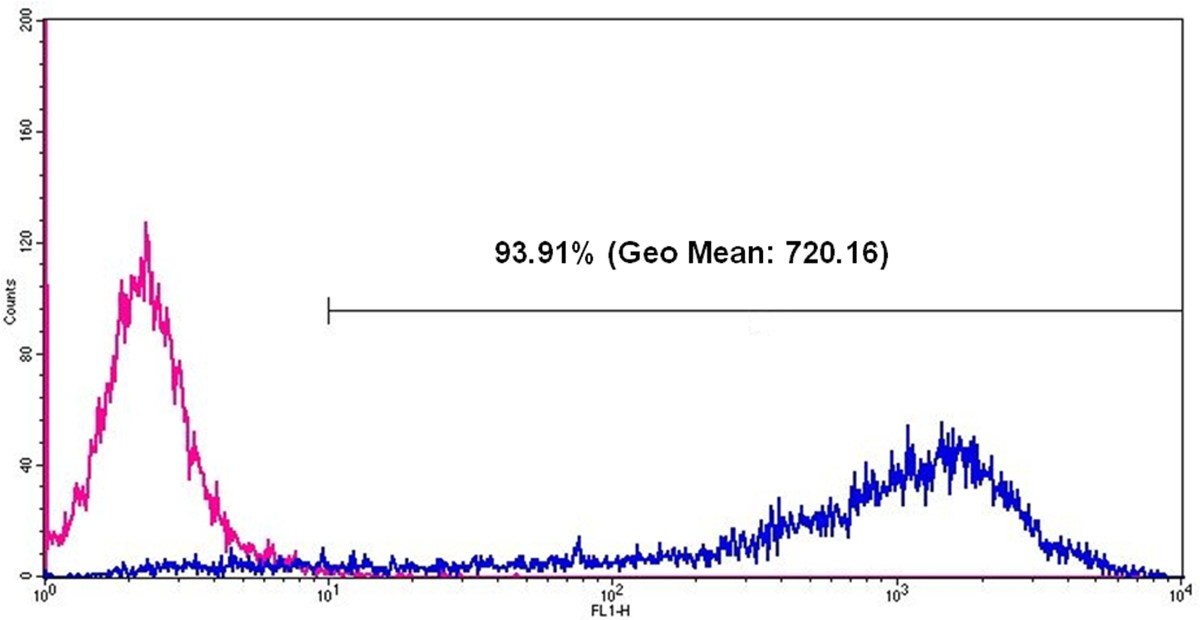
Figura6: citometria a flusso analisi di espressione eGFP in cellule HEK293 24 ore dopo eGFP mRNA trasfezione La linea rosa rappresenta cellule senza mRNA trasfezione e la linea blu rappresenta le cellule positive eGFP dopo eGFP mRNA trasfezione.. Dopo eGFP mRNA trasfezione, 93.91% di tutte le cellule misurati sono positivi e il Geo Media (media geometrica di intensità di fluorescenza) è 720,16.

Figura 7: citofluorimetrica analisi di espressione eGFP in cellule HEK293 1, 2, e 3 giorni dopo eGFP mRNA trasfezione L'espressione proteica è massima 24 ore dopo la trasfezione mRNA.. Successivamente, l'importo è ridotto di 1,6 volte ogni giorno (n = 3).
Tabella 1: Composizione della miscela di PCR.
| Componente | Concentrazione finale | Importo (ml) |
| Forward Primer | 0,7 mM | 7 |
| Primer Reverse | 0,7 mM | 7 |
| 5x Q-Solution | 1x | 20 |
| 5x HotStar HiFidelity PCR Buffer | 1x | 20 |
| DNA plasmidico | 50 ng / 100ul | Variabile |
| HotStar HiFidelity DNA polimerasi (2,5 U / ml) | 2.5 U | 1 |
| Acqua priva di nucleasi | Variabile | |
| Volume totale | 100 |
Tabella 1: Composizione della miscela di PCR.
| Numero ciclo | Tempo | Temperatura (° C) | |
| Fase di denaturazione iniziale | 1 | 5 min | 95 |
| 3-step ciclismo | 2-25 | ||
| · Denaturazione | 45 sec | 95 | |
| · Ricottura | 1 min | 55 | |
| · Estensione | 1 min | 72 | |
| Ultimo passo estensione | 26 | 10 min | 72 |
| Fine della PCR ciclismo | Indefinito | 4 |
Tabella 2: PCR ciclismo protocol.
| Componente | Concentrazione della (mm) | Concentrazione finale (mM) | Volume (ml) |
| ATP (da MEGAscript T7 Kit) | 75 | 7.5 | 4 |
| GTP (da MEGAscript T7 Kit) | 75 | 1.875 | 1 |
| Me-CTP (da Trilink) | 100 | 7.5 | 3 |
| Pseudo-UTP (da Trilink) | 100 | 7.5 | 3 |
| 3'-O-Me-m 7 G (5') ppp (5') G RNA struttura cap analogico | 10 | 2.5 | 10 |
| Volume totale | 23 |
Tabella 3: Composizione della miscela analogico NTP / cap.
| Componente | Concentrazione finale | Importo (ml) |
| Acqua priva di nucleasi | Variabile | |
| RNase Inhibitor | 40 U | 1 |
| NTP / miscela analogico cap (dal punto 4.3) | 23 | |
| Prodotto di PCR | 1 mg | Variabile |
| Tampone di reazione 10x | 1x | 4 |
| 10x T7 RNA polimerasi mix enzima | 1x | 4 |
| Volume totale | 40 |
Tabella 4: Composizione della trascrizione in vitro (IVT) miscela di reazione.
| Componente | Importo (ml) |
| Formammide | 3.3 |
| 37% di formaldeide | 1 |
| Buffer di MEN (10x) | 1 |
| Tampone di caricamento 6x (in dotazione con il peqGOLD gamma Mix DNA-Ladder) | 1.7 |
| Volume totale | 7 |
Tabella 5: Preparazione del tampone di caricamento per elettroforesi su gel di RNA.
| Terreno di coltura cellulare e Buffer | |
| HEK-293 mezzo di coltura cellulare | Aggiungere 25 ml di FCS, 2,5 ml di penicillina / streptomicina, 2.5 ml di L-glutamine in 220 ml di DMEM alte concentrazioni di glucosio. Conservare il mezzo a 4 ° C e utilizzarlo entro 2 settimane. |
| Tampone TBE (10x) | Sciogliere 0,9 M Tris base, acido borico 0,9 M e 20 mM EDTA in 1 L di acqua (Ampuwa). Il pH del tampone è 8. |
| Buffer di MEN (10x) | Sciogliere MOPS 200 mm, 50 NaOAc mM, EDTA 10 mM in 1 L di acqua (Ampuwa). Regolare il valore di pH con NaOH a 7. |
Tabella 6: Cell terreno di coltura e tamponi.
Discussione
La terapia mRNA ha un enorme potenziale nel campo della medicina rigenerativa, il trattamento di malattie e vaccinazione. In questo video, dimostriamo la produzione di un mRNA modificato stabilizzata per l'induzione dell'espressione proteica nelle cellule. Usando questo protocollo, altri mRNA desiderati possono essere generati. La sintesi in vitro di mRNA modificato consente trasfezione di cellule con mRNA desiderati per indurre l'espressione di proteine bersaglio. In tal modo, la proteina desiderata è espressa transitoriamente in condizioni fisiologiche fino mRNA esogenamente erogata è completamente degradata.
In questo video, l'espressione di eGFP è stata dimostrata per 3 giorni dopo una singola trasfezione di cellule HEK293 con eGFP mRNA. molecole di mRNA che codificano altre proteine potrebbe portare a un periodo più breve espressione della proteina. L'espressione della proteina diminuisce a causa della degradazione dell'mRNA esogenamente consegnato. Pertanto, la limitazione di questa tecnica per somapplicazioni e potrebbe essere l'induzione transitoria di espressione della proteina. Pertanto, per mantenere l'espressione della proteina in cellule per un periodo più lungo, è necessaria la consegna ripetuta di mRNA. Sebbene, mRNA trasfezione ha il vantaggio di non integrazione nel genoma dell'ospite, che impedisce mutagenesi inserzionale e lo sviluppo del cancro e la leucemia rispetto a vettori virali, l'efficienza di trasfezione in vivo potrebbe essere inferiore utilizzando vettori virali.
La concentrazione di mRNA richiesta e la quantità di reagente di trasfezione dovrebbero essere ottimizzati per ogni differente tipo di cellula 14, che sono le cellule bersaglio per la consegna esogena di mRNA per produrre la proteina mancante.
Cicli ripetuti di congelamento e scongelamento di mRNA deve essere evitata per mantenere la stabilità del prodotto mRNA. Pertanto, aliquote di lavoro possono essere preparati. Dopo la PCR e IVT, una sola banda specifica dovrebbe essere rilevata. Altrimenti, il numero di cicli di PCR, Annea Primertemperatura e / o la quantità di DNA plasmidico ling dovrebbero essere ottimizzati per ottenere il prodotto di DNA specifico per IVT. Inoltre, il tempo IVT e la quantità del DNA stampo per FPI possono essere ottimizzati per ottenere mRNA di una lunghezza specifica in quantità sufficienti.
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari in competizione.
Riconoscimenti
Questo progetto è stato finanziato dal Fondo Sociale Europeo in Baden-Wuerttemberg, Germania.
Materiali
| Name | Company | Catalog Number | Comments |
| Cell culture | |||
| DMEM, high glucose | PAA | E15-009 | |
| FBS | Life Technologies | 10500 | |
| Penicillin/Streptomycin | PAA | P11-010 | 100x |
| L-glutamine | PAA | M11-004 | 200 mM |
| DPBS without calcium and magnesium | PAA | E15-002 | |
| 0.04% Trypsin / 0.03% EDTA | Promocell | C-41020 | |
| TNS | Promocell | C-41120 | Trypsin Neutralizing Solution, 0.05% trypsin inhibitor in 0.1% BSA |
| HEK-293 cells | ATCC | CRL-1573 | |
| Consumables | |||
| Tissue culture plates, 12-well | Corning | 3512 | |
| Cell culture flask (75 cm2) | Corning | 430641 | |
| DNase-and RNase-free 1.5 ml sterile microcentrifuge tubes | Eppendorf | 0030 121.589 | Safe-Lock, Biopur |
| 15 ml conical tubes | greiner bio-one | 188271 | |
| PCR clean and sterile epT.I.P.S. dualfilter pipette tips | Eppendorf | 10 µl: 022491202; 100 µl: 022491237; 1,000 µl: 022491253 | |
| Cryovial | greiner bio-one | 122279-128 | Cryo.s |
| 14 ml polypropylene round bottom tube for bacterial culture | BD Falcon | 352059 | |
| Plasmid amplification and purification | |||
| pcDNA 3.3_eGFP Plasmid | Addgene | 26822 | |
| One Shot Top10 chemically component Escherichia coli | Invitrogen | C4040-10 | |
| Sterile water (Ampuwa) | Fresenius Kabi | 1636071 | |
| LB medium (Luria/Miller) | Carl Roth | X968.1 | Dissolve 25 g L-1 in sterile water. |
| LB agar (Luria/Miller) | Carl Roth | X969.1 | Dissolve 40 g L-1 in sterile water. |
| Ampicillin Ready Made Solution | Sigma Aldrich | A5354 | 100 mg/ml |
| Glycerol | Sigma Aldrich | G2025 | |
| QIAprep Spin Miniprep Kit | Qiagen | 27104 | |
| mRNA production | |||
| HotStar HiFidelity Polymerase Kit | Qiagen | 202602 | |
| QIAquick PCR Purification Kit | Qiagen | 28106 | |
| MEGAscript T7 Kit | Life Technologies | AM1334 | |
| 5-Methylcytidine-5´-triphosphate | Trilink | N1014 | 5-Methyl-CTP |
| Pseudouridine-5´-triphosphate | Trilink | N1019 | Pseudo-UTP |
| 3´-O-Me-m7G(5´)ppp(5´)G RNA cap structure analog | New England Biolabs | S1411L | |
| RiboLock RNase Inhibitor | Thermo Scientific | EO0381 | 40 U/µl |
| TURBO DNase | Life Technologies | AM1334 | 2 U/µl (from MEGAscript T7 Kit) |
| Antarctic phosphatase | New England Biolabs | MO289S | |
| RNeasy mini kit | Qiagen | 74104 | |
| RNaseZap solution | Life Technologies | AM9780 | |
| Transfection | |||
| Lipofectamine 2000 Transfection Reagent | Invitrogen | 11668-019 | Cationic lipid transfection reagent |
| Opti-MEM I Reduced Serum Media | Invitrogen | 11058-021 | Improved Minimal Essential Medium (MEM) that allows a reduction of Fetal Bovine Serum (FBS) supplementation |
| Gel electrophoresis | |||
| Agarose | Sigma-Aldrich | A9539 | |
| Gelred Nucleic Acid Gel Stain | Biotium | 41003 | 10,000x in water |
| peqGOLD Range Mix DNA-Ladder | Peqlab | 25-2210 | |
| Flow cytometry analyses | |||
| CellFIX (1x) | BD Biosciences | 340181 | 10x concentrate |
| Primer for insert amplification and poly (T) tail PCR | |||
| Forward Primer (HPLC-grade) 10 µM | Ella Biotech | 5´-TTGGACCCTCGTAC AGAAGCTAATACG-3´ | |
| Reverse Primer (HPLC-grade) 10 µM | Ella Biotech | 5´- T120-CTTCCTACT CAGGCTTTATTCAA AGACCA-3´ | |
| Equipment | |||
| Cell incubator | Binder | CO2 (5%) and O2 (20%) | |
| CASY cell counter | Schärfe System | ||
| Sterile workbench | BDK Luft-und Reinraumtecknik GmbH | ||
| Bacterial incubator | Incutec | ||
| Water bath | |||
| ScanDrop spectrophotometer | Analytic Jena | ||
| PCR thermocycler | Eppendorf | ||
| Microcentrifuge | Eppendorf | ||
| Vortex | peqlab | ||
| Thermomixer | Eppendorf | ||
| Gel apparatus for electrophoresis | Bio-Rad | ||
| Gel documentation system | Bio-Rad | ||
| FACScan System | BD Biosciences | ||
| Fluorescence microscope | Nikon | ||
| Phase-contrast microscope | Zeiss | ||
Riferimenti
- Bangel-Ruland, N., et al. CFTR-mRNA delivery: a novel alternative for cystic fibrosis "gene therapy". The journal of gene medicine. , (2013).
- Benteyn, D., et al. Design of an Optimized Wilms" Tumor 1 (WT1) mRNA Construct for Enhanced WT1 Expression and Improved Immunogenicity In Vitro and In Vivo. Molecular therapy Nucleic acids. 2, 134(2013).
- Petsch, B., et al. Protective efficacy of in vitro synthesized, specific mRNA vaccines against influenza A virus infection. Nature. 30, 1210-1216 (2012).
- Mandal, P. K., Rossi, D. J. Reprogramming human fibroblasts to pluripotency using modified mRNA. Nature protocols. 8, 568-582 (2013).
- Warren, L., et al. Highly efficient reprogramming to pluripotency and directed differentiation of human cells with synthetic modified mRNA. Cell stem cell. 7, 618-630 (2010).
- Yakubov, E., Rechavi, G., Rozenblatt, S., Givol, D. Reprogramming of human fibroblasts to pluripotent stem cells using mRNA of four transcription factors. Biochemical and biophysical research communications. 394, 189-193 (2010).
- Anderson, B. R., et al. Incorporation of pseudouridine into mRNA enhances translation by diminishing PKR activation. Nucleic acids research. 38, 5884-5892 (2010).
- Kariko, K., Buckstein, M., Ni, H., Weissman, D. Suppression of RNA recognition by Toll-like receptors: the impact of nucleoside modification and the evolutionary origin of RNA. Immunity. 23, 165-175 (2005).
- Kariko, K., et al. Incorporation of pseudouridine into mRNA yields superior nonimmunogenic vector with increased translational capacity and biological stability. Molecular therapy : the journal of the American Society of Gene Therapy. 16, 1833-1840 (2008).
- Kariko, K., Weissman, D. Naturally occurring nucleoside modifications suppress the immunostimulatory activity of RNA: implication for therapeutic RNA development. Current opinion in drug discovery & development. 10, 523-532 (2007).
- Kormann, M. S., et al. Expression of therapeutic proteins after delivery of chemically modified mRNA in mice. Nature biotechnology. 29, 154-157 (2011).
- Hacein-Bey-Abina, S., et al. Insertional oncogenesis in 4 patients after retrovirus-mediated gene therapy of SCID-X1. The Journal of clinical investigation. 118, 3132-3142 (2008).
- Hacein-Bey-Abina, S., et al. Efficacy of gene therapy for X-linked severe combined immunodeficiency. The New England journal of medicine. 363, 355-364 (2010).
- Avci-Adali, M., et al. Optimized conditions for successful transfection of human endothelial cells with in vitro synthesized and modified mRNA for induction of protein expression. Journal of biological engineering. 8, 8(2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon