Method Article
Cromatina Analisi Interazioni con Tag Sequencing appaiati-End (Chia-PET) per la mappatura Interazioni cromatina e comprensione del regolamento Trascrizione
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Interaction Analysis cromatina per tag Sequencing paired-End (Chia-PET) è un metodo per De novo delle interazioni della cromatina, per una migliore comprensione del controllo trascrizionale.
Abstract
Genomi sono organizzati in strutture tridimensionali, adottando ordine superiore conformazioni all'interno degli spazi nucleari dimensioni micron 7, 2, 12. Architetture di questo tipo non sono casuali e comportano interazioni tra geni promotori e gli elementi normativi 13. Il legame di fattori di trascrizione specifici per sequenze regolatrici determina una rete di regolazione della trascrizione e coordinamento 1, 14.
Interaction Analysis cromatina per tag Sequencing paired-End (Chia-PET) è stato sviluppato per identificare queste strutture sovra-ordinate cromatina 5,6. Cellule vengono fissate e loci interagenti sono catturati dal covalenti DNA-proteina legami incrociati. Per minimizzare il rumore non specifico e ridurre la complessità, così come per aumentare la specificità del analisi dell'interazione cromatina, immunoprecipitazione della cromatina (ChIP) viene usato contro fattori proteici specifici per arricchire frammenti cromatina di interesse prima legatura prossimità. Legatura coinvolgendo mezze linkers successivamente forma legami covalenti tra coppie di frammenti di DNA legati insieme all'interno di complessi cromatina individuali. I accompagnamento MmeI siti di restrizione degli enzimi nella mezza linkers permettono l'estrazione di coppie di terminali tag-linker-tag costrutti (PET) su MmeI digestione. Poiché le mezze linkers sono biotinilati, questi costrutti PET sono purificati utilizzando streptavidina-perle magnetiche. Le PET purificati vengono legati con la prossima generazione di adattatori di sequenziamento e un catalogo di frammenti interagenti viene generato tramite sequencer di nuova generazione come il Illumina Genome Analyzer. Mappatura e analisi bioinformatica viene quindi eseguita per identificare ChIP arricchito vincolante siti e ChIP arricchiti interazioni cromatina 8.
Abbiamo prodotto un video per dimostrare aspetti critici del Chia-PET protocollo, in particolare la preparazione del chip come la qualità di ChIP gioca un ruolo importante nel risultato di una Chia-PET libreria. Poiché i protocolli sonomolto lungo, solo i punti critici sono mostrati nel video.
Protocollo
A. immunoprecipitazione della cromatina (ChIP) (vedi Figura 1)
1. La reticolazione doppio della cromatina proteine legate e raccolta di cellule
(Al 02:10 del video)
Immunoprecipitazione della cromatina (ChIP) è il primo passo critico coinvolti nella costruzione di una Chia-PET biblioteca. Questo passaggio è importante per ridurre il livello di complessità, rumore di fondo e aggiungere specificità. La preparazione di ChIP dovrà essere ottimizzata per tipo di cellula e fattore di interesse. Questo protocollo è basato sul RNA polimerasi II Chia-PET libreria preparato da cellule MCF-7 9 costruiti nel nostro laboratorio. Per garantire che la biblioteca che ne risulta è di complessità sufficiente, si consiglia di utilizzare 1 x 10 8 celle per preparare il materiale ChIP. A seconda della linea cellulare di destinazione e, la resa può essere ottenuto tra 100 ng a 300 ng. Si consiglia un minimo di 100 ng ChIP deve essere utilizzato per construct uno Chia-PET biblioteca con meno di 20 cicli di PCR per ridurre al minimo la ridondanza durante la sequenza. Grandi quantità di materiale ChIP consentirà di ulteriori riduzioni di ridondanza, migliorare la qualità delle librerie.
- Lavare 1 x 10 8 cellule MCF-7 (equivalente a cinque 500 cm 2 piatti quadrati di 2 x 10 7 cellule) due volte con soluzione tampone fosfato (PBS) pre-riscaldato a 37 ° C.
- Reticolazione cellule MCF-7 con 1,5 mM preparata EthylGlycol bis (SuccinimidylSuccinate) (EGS)) per 45 minuti a temperatura ambiente (22 ° C) con rotazione in una cappa di laboratorio.
- Aggiungere 37% formaldeide ad una concentrazione finale di 1% per 20 minuti a temperatura ambiente (22 ° C) con rotazione in una cappa di laboratorio.

- Quench i reticolanti aggiungendo 2 M glicina ad una concentrazione finale di 200 mM. Incubare per 10 minuti a temperatura ambiente(22 ° C) con rotazione.
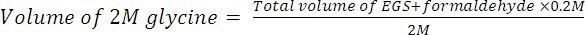
- Gettare reticolanti temprati in un pallone di rifiuti e lavare le cellule due volte con PBS freddo.
- Eliminare PBS e le cellule raccolto raschiando. Conservare le cellule raccolte su ghiaccio. Sciacquare la piastra con 5ml PBS (con inibitori della proteasi ("+ PI"), ha aggiunto, secondo le istruzioni del produttore) per massimizzare la raccolta di cellule.
- Agglomerare le cellule a 1.800 xg (3.000 rpm) per 10 minuti a 4 ° C. Gettare il surnatante e procedere alla lisi cellulare. In alternativa, memorizzare il pellet a -80 ° C.
2. Lisi cellulare
(Alle 04:15 del video)
- Risospendere il pellet in 15 ml 0,1% SDS tampone di lisi (50 mM HEPES pH7.5, 150 mM NaCl, 1 mM EDTA, 1% Triton X-100, sodio deossicolato 0,1%, 0,1% SDS + PI) a fondo pipettando. Ruota per 15 minuti a 4 ° C.
- Pellet lisate le cellule a 800 xg (2.000 rpm) per 10 minuti a 4 ° C.
- Gettare il surnatante e ripetere lisi cellulare una volta come per passo 2.1 e 2.2.
- L'isolato nucleare pellet è pronto per lisi nucleare. In alternativa, negozio di pellet a -80 ° C.
3. Lysis nucleare
(Alle 05:00 del video)
Lisi nucleare viene eseguita per rilasciare cromatina reticolato prima di frammentazione della cromatina. In assenza della membrana nucleare, la cromatina reticolato può essere sonicata utilizzando dolci condizioni. Talvolta, la forza sonicazione può non essere sufficiente a rompere la membrana nucleare in questo caso, meno cromatina si ottiene poiché i nuclei intatti sarà scartata dopo centrifugazione. Tuttavia, diversi tipi di cellule può richiedere condizioni diverse.
- Risospendere isolato nuclei pellet in 15 ml 1% SDS Tampone di lisi (50 mM HEPES pH7.5, 150 mM NaCl, 1 mM EDTA, 1% Triton X-100, 0,1% di sodio Deoxycholate, 1% SDS + PI). Trasferire la sospensione nuclei ad una centrifuga ad alta velocità tubo (tubo centrifuga Oak Ridge (Polipropilene)) e ruotare per 15 minuti a 4 ° C.
- Pellet lisate nuclei pellet a 47.810 xg (20.000 rpm) per 30 minuti a 4 ° C.
- Decantare il surnatante e pellet rottura con una pipetta P1000.
- Lavare pellet con 30 ml 0,1% SDS Lysis Buffer (+ PI).
- Ruota per 15 minuti a 4 ° C.
- Pellet cromatina a 47.810 xg (20.000 rpm) per 30 minuti a 4 ° C.
- Gettare il surnatante e ripetere lavare una volta, come per step 3,4-3,6 prima di procedere ad una frammentazione della cromatina. In alternativa, negozio di cromatina pellet a -80 ° C.
4. La frammentazione della cromatina
(Alle 07:03 del video)
- Trasferimento cromatina pellet ad un 14 ml tubo tondo di prova polistirene fondo.
- Aggiungere 1 ml 0,1% SDS Lysis Buffer (+ PI) alla cromatina peLLET. Assicurarsi che non vi siano bolle nella miscela come questo influirà efficienza sonicazione.
- Shear cromatina-DNA a una dimensione di 200 a 600 coppie di basi con una Digital Branson Sonifer cellulare Disrupter (ampiezza 35%, 9 minuti (30 secondi, 30 secondi off)) e mantenere i campioni freddo per tutto il tempo per evitare il surriscaldamento eseguendo l' sonicazione in una stanza fredda (vedi Figura 2).
- Reverse-reticolare un'aliquota della cromatina e verificare l'efficienza frammentazione con i seguenti passi. (I seguenti passaggi non vengono mostrati nel video).
- Aliquotare 10 pl della cromatina dopo sonicazione.
- Centrifugare sonicata cromatina a 16.110 xg (13.200 rpm) per 5 minuti a 4 ° C.
- Trasferire il surnatante in una nuova provetta e aggiungere 2 pl di soluzione di proteinasi K.
- Incubare per 30 minuti a 50 ° C.
- Risolvere il reverse reticolato cromatina su un gel di agarosio 1,5%.
- Ripett sonicazione se le dimensioni di DNA sono maggiori del previsto.
- Centrifugare lisato rimanendo a 16.110 xg (13.200 rpm) per 30 minuti a 4 ° C e trasferimento sonicata cromatina (surnatante) in una nuova provetta prima preclearing con perline magnetiche. In alternativa, negozio sonicata cromatina a -80 ° C fino cromatina sufficiente viene raccolto per avviare un chip.
5. Lavaggio, Preclearing e rivestimento di Anticorpi anti-Beads
(Ore 08:17 del video)
- Preclearing della cromatina
- Utilizzare 300 ml di biglie magnetiche per una proteina G IP.
- Lavare perline tre volte con 5 ml Beads Tampone lavaggio (PBS, 0,1% Triton X-100). Questo lavaggi e future coinvolgono perline magnetiche proteina G costituiti i seguenti passaggi. Assicurarsi biglie magnetiche non si seccano.
- Reclaim perline usando un concentratore di particelle magnetiche.
- Discard supernatante.
- Risospendere le sfere con tampone.
- Ruota per 5 minuti a 4 ° C.
- Centrifugare a 129 xg (800 rpm) per 1 minuto a 4 ° C.
- Reclaim perline usando un concentratore di particelle magnetiche.
- Gettare il surnatante.
- Combina cromatina sonicato con perline lavata, per rimuovere lo sfondo vincolante della cromatina alle perle.
- Tenere 10 pl della cromatina sonicato come "input" per il controllo successivo arricchimento mediante PCR quantitativa (qPCR). Conservare a 4 ° C.
- Ruotare la notte a 4 ° C.
- Centrifugare sonicata cromatina a 129 xg (800 rpm) per 1 minuto a 4 ° C.
- Posizionare il tubo concentratore di particelle magnetiche.
- Anticorpi anti-Coating sfere magnetiche
- Aliquotare 300 pl di perline magnetiche proteina G in una nuova provetta.
- Lavare perline tre volte con 5ml Wash Buffer Beads (PBS, 0,1% Triton X-100).
- Aggiungere un volume equivalente (come passaggio 5.1.3) di Wash Buffer Beads.
- Aggiungere 35 microgrammi di RNA polimerasi II (8WG16) anticorpo monoclonale.
- Ruotare la notte a 4 ° C.
- Lavare anticorpo perle rivestite due volte con 5 ml di tampone di lavaggio Perline.
- Reclaim anticorpo perle rivestite usando un concentratore di particelle magnetiche.
6. Immunoprecipitazione della cromatina
(Alle 10:03 del video)
Per ridurre il livello di complessità e di rumore di fondo, anticorpi contro fattori proteici specifici vengono utilizzati per arricchire cromatina frammenti specifici di interesse prima legatura prossimità 6.
Qui, abbiamo utilizzato un topo monoclonale RNA polimerasi IIanticorpo (8WG16) che riconosce il modulo di avvio della proteina. Per frammenti di DNA arricchimento che sono associati con RNA polimerasi II, specificità della libreria può essere aumentata, consentendo l'identificazione di interazioni a lungo raggio cromatina tra promotori attivi e le loro regioni corrispondenti regolamentazione 9.
- Scartare tampone di lavaggio da anticorpi perle rivestite con l'aiuto di un concentratore particella magnetica.
- Transfer sonicata cromatina (surnatante dal punto 5.1.6) con l'aiuto del concentratore particelle magnetiche all'anticorpo perle rivestite (dal punto 5.2.7).
- Ruotare la notte a 4 ° C.
7. Lavaggio e eluizione di immunoprecipitate complessi DNA-proteine
(Alle 11:13 del video)
- Lavare cromatina-immunoprecipitate perle tre volte con 5 ml 0,1% tampone di lisi SDS.
- Lavare una volta con 5 perline tampone sali Wash ml (50 mM pH 7,5 HEPES, 350 mM NaCl, 1 mM EDTA, 1% Triton X-100, desossicolato di sodio 0,1%, 0,1% SDS).
- Lavare perline una volta con 5 ml di tampone di lavaggio litio cloruro (10 mM Tris pH 8,0, 250 mM LiCl, 1 mM EDTA, 0,5% Nonidet P-40, sodio deossicolato 0,5%).
- Scartare il tampone di lavaggio e risospendere le sfere lavate con 1 ml di tampone TE.
- Ruota per 5 minuti a 4 ° C. Il campione è pronto per la quantificazione e l'arricchimento controllo ChIP. Una volta che sufficiente materiale ChIP è stato raccolto, il campione è pronto per essere costruito in una Chia-PET biblioteca. Questo passaggio è fondamentale, e deve essere fatto per garantire il successo Chia-PET costruzione della libreria. ChIP arricchiti di perle possono essere conservati per un massimo di 2 settimane a 4 ° C, mentre in fase di quantificazione, i controlli di arricchimento di chip, e l'accumulo di materiale sufficiente.
- Eluire e reverse-reticolazione DNA "input" e ChIP arricchiti con perline con le seguenti operazioni. (I seguenti passaggi non vengono mostrati nel video.)
- Eluire 20% di ChIP arricchiti di perle con 200 microlitri tampone di eluizione ChIP (50 mM Tris pH 8,0, 10 mM EDTA, 1% SDS). Ruota per 30 minuti a 37 ° C. Centrifugare perline eluiti a 6.100 xg (800 rpm) per 1 minuto a 4 ° C. Trasferimento eluiti complessi Chip (surnatante) in una nuova provetta con l'aiuto del concentratore di particelle magnetiche.
- Complessi ChIP Reverse-reticolazione "ingresso" e eluita con 2 pl Proteinasi K (concentrazione finale di 0,2 g / ml) per 2 ore a 50 ° C.
- Trasferimento inverso reticolato DNA "input" ChIP e DNA eluito in una densità 2 ml MaXtract alta (rispettivamente). Aggiungere 200 ul 25:24:1 fenolo-cloroformio-alcol Isoamyl pH 7,9 per i tubi in una cappa chimica per fumi e capovolgere per mescolare bene. Centrifugare le provette per 5 minuti a 16.110 xg (13.200 rpm) a temperatura ambiente.
- Trasferire superiore fase acquosa in una nuova provetta e precipitare invertire spirito DNA reticolatoh 20 pl 3 M acetato di sodio pH 5,5, 200 microlitri isopropanolo e 1 pl 15 mg / ml Glycoblue. Incubare a -80 ° C per 30 minuti e pellet di DNA per 30 minuti a 16.110 xg (13.200 rpm) a 4 ° C.
- Dopo la centrifugazione di isopropanolo DNA precipitato, lavare DNA pellet con il 75% ghiacciato etanolo due volte e risospendere il DNA pellet in 20 microlitri di buffer TE.
- Quantificare DNA "input" (dal punto 5.1.3) e il DNA Chip picogreen test 10.
- Eseguire un controllo di arricchimento mediante PCR qualitativa (qPCR).
- Eluire e reverse-reticolazione DNA "input" e ChIP arricchiti con perline con le seguenti operazioni. (I seguenti passaggi non vengono mostrati nel video.)
B. cromatina analisi dell'interazione con appaiati-End Tag Sequencing (Chia-PET)
La seconda metà del video metterà in evidenza i passaggi chiave nella costruzione di una Chia-PET biblioteca.
1. End-ottundimento di frammenti di DNA di ChIP
Questo capitolo del video mette in evidenza due main punti, un passo-passo la procedura di lavaggio biglie magnetiche per rimuovere gli enzimi e il buffering sale della reazione precedente (Step 1.1, 12:22-0:54 del video) e la procedura di creazione di reazioni enzimatiche che coinvolge sfere magnetiche nella miscela di reazione (Fase 1,2, 12:54-13:25 del video).
- Lavare ChIP arricchiti di perle di una volta con tampone ghiacciato TE. Questo lavaggi e future coinvolgono perline magnetiche sono costituiti i seguenti passaggi.
- Centrifugare a 6100 xg (800 rpm) per 1 minuto a 4 ° C.
- Reclaim perline usando un concentratore di particelle magnetiche.
- Gettare il surnatante.
- Aggiungi buffer perline.
- Mescolare perline con tampone da sfogliando azione.
- Centrifugare a 6100 xg (800 rpm) per 1 minuto a 4 ° C.
- Reclaim perline usando un concentratore di particelle magnetiche.
- Gettare il surnatante.
- Per riempire-in sonicato overhaNGS, preparare un master-mix con 70 microlitri 10 x Buffer per DNA polimerasi T4, 7 pl 10 mM dNTPs e 615,8 ul-acqua priva di nucleasi. Mescolare accuratamente e risospendere le sfere lavati con il maestro-mix. Aggiungere 7,2 microlitri 9,7 U / microlitro di T4 DNA polimerasi in un volume finale di 700 microlitri. Mescolare e incubare per 40 minuti a 37 ° C con rotazione (usando Palico Biotech Intelli-Mixer RM-2L, F8, 30 rpm, U = 50, u = 60). Eseguire tutte le successive reazioni enzimatiche con i seguenti passaggi.
- Diluire buffer di reazione con acqua priva di nucleasi.
- Aggiungere tutti gli altri reagenti (tranne enzimi) al buffer diluito.
- Mescolare accuratamente. Risospendere le sfere / pellet con la miscela di reazione.
- Aggiungi enzima per microsfere in sospensione / pellet (Keep enzimi sulla scatola da banco fresco in ogni momento per garantire la massima attività enzimatica).
- Sigillare il tubo con parafilm.
- Assicurarsi che le microsfere sono ben mescolati in tutta la incubatio interan.
- Scartare miscela di reazione e lavare via i sali tampone residue ed enzimi tre volte con tampone ghiacciato di lavaggio (10 mM Tris-Cl pH 7,5, 1 mM EDTA, 500 mM NaCl).
2. Legatura di Biotinylated mezze Linkers al DNA ChIP
Questo capitolo del video evidenzia le caratteristiche dei semi-linker oligonucleotidi utilizzati nella costruzione di Chia-PET e l'uso di codici a barre composizione nucleotidica di distinguere tra prodotti di ligazione non-specifici e specifici (13:28-14:24) dei video). Il capitolo mostra anche la step-by-step procedura di creazione di un half-linker reazione di ligazione (Step 2.2, 14:24-15:54 del video).
Due tipi di biotinilati mezze linkers sono introdotte nel protocollo e sono progettati con un codice a barre interno di quattro nucleotidi (TAAG o ATGT) e un sito di riconoscimento per l'enzima di restrizione MmeI tipo di IIS (TCCAAC). Dopo ChIP arricchirezione, sonicato frammenti cromatina sono divise in due aliquote e vengono prima ligato con un eccesso di due semi-linker A o semi-linker B 5,9.
- Dividere ChIP arricchiti di perle in due aliquote per mezzo del DNA-linker legatura di biotinilati mezze linkers A o mezze linkers B rispettivamente. Queste due mezze linkers hanno le stesse sequenze nucleotidiche, tranne quattro nucleotidi nel mezzo che servono (Half-linker A con TAAG, la metà-linker B con ATGT) come codici a barre nucleotidi. Quando combinate durante ligazione prossimità, casuale e non-specifici legature tra due complessi ChIP differenti possono essere identificati dalla popolazione di sequenze con eterodimero linker AB, rispetto legature specifici che possono essere identificate dalla popolazione di sequenze con omodimero AA o linker BB .
- Legare ChIP arricchiti di perle con 140 microlitri Buffer 5 T4 DNA ligasi x con PEG, 3,5 pl 200 ng / ul biotinilati mezze linker A o B, 553,5 ul nucleasiacqua libera e 3 microlitri 30 U / microlitro di DNA ligasi T4 (T4 DNA ligasi Tenere su una scatola da banco fresco in ogni momento come ligasi sono instabili anche su ghiaccio). Sigillare il tubo con parafilm e incubare durante la notte a 16 ° C con rotazione.
3. Eluizione e legatura di prossimità di frammenti di DNA di ChIP
Questo capitolo del video spiega il ruolo di tampone EB, SDS e Triton X-100 durante l'eluizione dei complessi cromatina da perle e mostra anche la step-by-step procedura di creazione di una reazione circolarizzazione (Step 3.9, 15:54 di 17:10 del video).
Dopo legando le mezze linkers ai frammenti di cromatina, entrambe le parti sono combinate e eluite al largo delle perline. Frammenti di DNA interagenti verranno poi collegati da una sequenza completa linker durante ligazione prossimità.
Utilizzando la composizione di codici a barre nucleotide, le sequenze possono essere classificati in tre categorie, vale a dire sequenze wi° eterodimero linkers AB (codice a barre ATGT / TAAG) e le sequenze con omodimero AA o BB linkers (codice a barre TAAG / o TAAG ATGT / ATGT) per distinguere i prodotti legatura non specifici e specifici, rispettivamente, 9.
Quindi, l'uso di due mezze linkers con diversi codici a barre nucleotidiche consente di specificare diversi esperimenti o repliche, nonché al monitoraggio della non-specifica ligazione chimerico tra diversi complessi ChIP 5.
- Lavare mezzo-linker-legato perline tre volte con tampone ghiacciato di lavaggio (10 mM Tris-Cl pH 7,5, 1 mM EDTA, 500 mM NaCl).
- Combinare entrambi i tubi di mezzo-linker-legato perle nel modo seguente.
- Reclaim uno dei tubi di mezzo-linker-legato perline usando un concentratore di particelle magnetiche ("Tubo A").
- Mantenere l'altro tubo di mezzo-linker-legato perline su ghiaccio ("Tubo B").
- Gettare il tampone di lavaggio (dal punto 3.2.1) fUn tubo rom e trasferire mezzo-linker-legato perline (dal punto 3.2.2) da B tubo nel tubo Un po 'di tubo A è ancora sul collettore di particelle magnetiche.
- Aggiungere 700 ul ghiacciata tampone di lavaggio B tubo per raccogliere eventuali residui half-linker-legato perline e assicurare entrambi i tubi di mezzo-linker-legato perle sono combinati in modo corretto. Trasferire il tampone di lavaggio in A. Eliminare il tubo vuoto tubo B.
- Fosforilano mezzo-linker-legato microsfere con 70 microlitri 10 T4 DNA ligasi x tampone (NEB), 616 microlitri di acqua priva di nucleasi e 14 microlitri 10 U / microlitro di T4 polinucleotide chinasi DNA. Incubare per 50 minuti a 37 ° C con rotazione.
- Reclaim fosforilati half-linker-legato perline usando un concentratore di particelle magnetiche ed eliminare miscela di reazione.
- Eluire mezzo-linker-ligato cromatina-DNA complesso con 200 microlitri tampone di eluizione preparata (tampone TE, 1% SDS). Incubare per 30 minuti a temperatura ambiente (22 °C) con rotazione.
- Trasferire eluato ad una nuova provetta e lavare con perline 900 EB tampone pl. Trasferire il surnatante al tubo stesso per combinare i eluizioni.
- Trasferimento eluato raccolto per le tazze filtro di filtri centrifuga due tubi e centrifugare a 16.110 xg (13.200 rpm) per 1 minuto a temperatura ambiente (22 ° C).
- Sequestrare SDS con l'aggiunta di 90 microlitri 20% Triton X-100 e capovolgere per mescolare bene. Incubare per 1 ora a 37 ° C.
- Circolarizzazione viene effettuata in condizioni estremamente diluita in modo da favorire eventi ligazione nei singoli complessi reticolati cromatina minimizzando eventi ligazione tra diversi complessi cromatina che darebbero luogo ad indesiderabili molecole di DNA chimeriche. Lavorando su ghiaccio, il trasferimento eluato bonificato ad un tubo da 50 ml Falcon e aggiungere 7776 microlitri di acqua priva di nucleasi, 1000 ml 10 x Buffer T4 DNA ligasi (NEB) e 33,33 microlitri 30 U / microlitro di DNA ligasi T4. Incubare per una notte a 16 anni° C.
4. Reverse-reticolazione e di purificazione del DNA
Questo capitolo del video mostra passo per passo la procedura di fenolo: estrazione con cloroformio (Step 4.3, 17:11-17:59) e il close-up del DNA pellet dopo centrifugazione al punto 4.4.
- Reverse-reticolazione e proteina degrado aggiungendo 100 pl 20 mg / ml proteinasi K soluzione. Incubare per 2 ore a 50 ° C.
- Trasferire DNA cromatina in una densità di 50 ml MaXtract alta e aggiungere 9ml priva di nucleasi acqua per ottenere un volume finale di 19 ml.
- Aggiungere 19 ml 25:24:1 fenolo-cloroformio-alcol Isoamyl pH 7,9 al tubo in una cappa chimica per fumi e capovolgere per mescolare bene. Centrifugare le provette per 5 minuti a 1.800 xg (3.000 rpm) a temperatura ambiente.
- Trasferire fase acquosa superiore ai 50 ml Tubes centrifuga Oak Ridge (Teflon FEP) e il DNA precipitato cromatina con 1,9 ml 3 M acetato di sodio pH 5.5, 19 ml isopropanolo e 5 Glycoblue pl. Glycoblue viene aggiunto per migliorare la visibilità del pellet.
- Incubare a -80 ° C per almeno un'ora. Lasciare la soluzione ghiacciata di scongelare prima centrifugazione a 38.720 xg (18.000 rpm) per 30 minuti a 4 ° C.
- Dopo la centrifugazione di isopropanolo DNA precipitato, lavare DNA pellet con il 75% ghiacciato etanolo due volte e risospendere il DNA pellet in 34 microlitri EB Buffer. Trasferire miscela di DNA in una provetta 1,7 ml LoBind DNA.
- Risospendere DNA pellet in 34 microlitri EB Buffer.
- (Trattamento RNasi è facoltativa successivo lavaggio e purificazione della Chia-PET protocollo servono per rimuovere la contaminazione RNA e le ispezioni manuali dei Chia-PET sequenze dai nostri in-house biblioteche hanno mostrato alcuna traccia di contaminazione RNA).
- Eseguire la digestione RNasi con l'aggiunta di 10 microlitri RNase ONE 10 x tampone di reazione, 55 microlitri priva di nucleasi acqua e 1 pl 10 U / microlitro RNAsi ONE ribonucleasi. Incubare a 37° C per un'ora.
- Trasferire il DNA della cromatina in una densità 2 ml MaXtract alta. Aggiungere 100 ul 25:24:1 fenolo-cloroformio-alcol Isoamyl pH 7,9 al tubo in una cappa chimica per fumi e capovolgere per mescolare bene. Centrifugare la provetta per 5 minuti a 16.110 xg (13.200 rpm) a temperatura ambiente (22 ° C).
- Trasferire superiore fase acquosa in una nuova provetta e precipitato inversa reticolato DNA con 10 microlitri 3 M acetato di sodio pH 5,5 e 100 pl isopropanolo. Incubare a -80 ° C per 30 minuti e pellet di DNA per 30 minuti a 16.110 xg (13.200 rpm) a 4 ° C.
- Dopo la centrifugazione di isopropanolo DNA precipitato, lavare il pellet di DNA con il 75% ghiacciato etanolo due volte e risospendere il DNA pellet in 34 microlitri EB Buffer.
5. Immobilizzazione di Chia-PET DNA Beads streptavidina
L'introduzione di questo capitolo parla della presenzadel sito di riconoscimento MmeI e la T biotinilato presente nei semi-linker oligonucleotidi per facilitare l'estrazione di tag-linker-tag costrutti ("PET", 18:02-19:10 del video). Inoltre, questo capitolo del video offre anche una rapida visione globale del Passo 5.2, step-by-step procedura di creazione di una reazione PCR (Step 6.1, 19:10-20:00 del video) ed ablazione con successo Chia-PET DNA (Step 6,9, 20:00 alle 20:30).
Mezze linkers A e B contengono siti di riconoscimento MmeI di accompagnamento, che permette questo tipo di enzima di restrizione IIS di tagliare 18/20 paia di basi a valle dei loro siti obiettivo vincolante per generare brevi "tag" del frammento cromatina, producendo coppie di tag-linker-tag costrutti ("PET").
Per consentire la purificazione di costrutti PET da grani magnetici streptavidina rivestite, sia un mezzo-linker A e B sono modificati con biotina, permettendo la cattura e la purificazione dei Chia-PET costrutti.
- Uscita captured Chia-PET DNA aggiungendo 5 microlitri 10 x NEBuffer 4, 5 microlitri preparata 500 pM (10 x) S-adenosilmetionina (SAM) e 1 pl 2 U / pl MmeI al DNA risospeso. Quench MmeI eccesso con l'aggiunta di 5 pl non-biotinilati mezze linkers a miscela di reazione per MmeI può essere auto-inibitrice in eccesso. Incubare per 2 ore a 37 ° C.
- Per catturare rilasciati Chia-PET DNA, 50 pl di trasferimento di sedimento M-280 perline streptavidina ad un tubo LoBind DNA. Lavare due volte con perline 2 x Legatorie tampone di lavaggio (2 x B & W: 10 mM Tris-HCl pH 7.5, 1 mM EDTA, 2 M NaCl). Risospendere le sfere in 50 microlitri 2 x B & W Buffer e trasferire 50 microlitri di miscela di digestione (dal punto 5.1) al tubo stesso. Mescolare bene e incubare per 45 minuti a temperatura ambiente con rotazione.
- Reclaim perline utilizzando un concentratore di particelle magnetiche ed eliminare il surnatante. Lavare perle due volte con 150 ml 1 x Legatorie tampone di lavaggio (1 x B & W: 5 mM Tris-HCl pH 7.5, 0.5 mM EDTA, 1 M NaCl).
- Legare 454 GS20 Adattatori a Chia-PET DNA con 5 pl 10 Buffer T4 DNA ligasi x (Si noti che il Buffer ligasi utilizzata in questa fase è diversa dal punto 3.9, il Buffer ligasi utilizzata in questo passaggio è dalla società stessa che fornisce la DNA ligasi T4), 4 microlitri 200 ng / pl 454 GS20 adattatore A, 4 pl 200 ng / pl 454 GS20 B adattatore, 36 microlitri di acqua priva di nucleasi e 1 pl 30 U / microlitro di T4 DNA ligasi. Incubare per una notte a 16 ° C con rotazione. Chia-PET DNA può anche essere legato con Illumina-NN adattatori, nel qual caso si consiglia di amplificazione della libreria con PCR Primer PE1.0 e PCR Primer PE 2.0 acquistato da Illumina.
- Reclaim perline utilizzando un concentratore di particelle magnetiche ed eliminare il surnatante. Lavare tre volte con perle di 150 ml 1 x B & W Buffer.
- Nick translate Chia-PET DNA con 5 pl 10 x NEBuffer 2, 2,5 pl 10 mM, dNTPs 38,5 ul priva di nucleasi acqua e 4 pl 10 U / microlitro Escherichia coli DNA polimerasi I. Incubare per una notte atemperatura ambiente (22 ° C) con rotazione.
- Reclaim perline utilizzando un concentratore di particelle magnetiche ed eliminare il surnatante. Lavare tre volte con perle di 150 ml 1 x B & W Buffer.
- Risospendere le sfere con 50 EB Buffer ul.
6. Amplificazione di Chia-PET DNA
- Impostare reazioni PCR con due sospensioni perline, ul 21 microlitri di nucleasi acqua libera, 1 pl 10 pM Illumina 1-454 primer, 1 pl 10 pM Illumina 2-454 primer e 25 pl Phusion High Fidelity Mix Master.
- Per determinare il numero di cicli necessari per generare prodotti PCR sufficiente per il sequenziamento, costituito prova tre reazioni PCR con cicli di 16, 18 e 20. Non superare i 20 cicli che abbiamo trovato che facendo così i risultati in termini di complessità biblioteca molto bassa.
| Fase iniziale | 30 secondi | 98 ° C | (Denaturazione) |
| Da 18 a 25 cicli di | 10 secondi | 98 ° C | (Denaturazione) |
| 30 secondi | 65 ° C | (Annealing) | |
| 30 secondi | 72 ° C | (Estensione) | |
| Fase Finale | 5 minuti | 72 ° C | (Estensione finale) |
- Determinare il numero di ciclo più basso possibile eseguendo le reazioni di PCR su un gel gradiente 4-20% TBE e colorazione con SYBR I verde, assicurando la presenza di una banda di 223 paia di basi, che è la lunghezza del Chia-PET DNA.
- Amplifica il resto dei Chia-PET DNA-bound perle streptavidina in una grande scala PCR con il minor numero possibile di cicli di PCR, pur di ottenere rendimento sufficiente per il sequenziamento.
- Reperle di reclamo da tutte le reazioni PCR utilizzando un concentratore di particelle magnetiche e piscina surnatante a due tubi freschi LoBind DNA. Precipitare ogni tubo di supernatante con l'aggiunta di 60 pl 3 M acetato di sodio, 2 GlycoBlue ul e 600 ul isopropanolo alle reazioni messe in comune.
- Incubare a -80 ° C per 30 minuti. Centrifugare a 16.110 xg (13.200 rpm) per 30 minuti a 4 ° C.
- Dopo la centrifugazione di isopropanolo DNA precipitato, lavaggio DNA pellet con il 75% ghiacciato etanolo due volte pellet e risospendere il DNA in 100 microlitri di buffer TE e 5 pl 6 x loading dye.
- Distribuire prodotti PCR uniformemente nei pozzetti di un gel TBE 6%. Eseguire il gel in 1 x tampone TBE a 200 V per 35 minuti e macchia con SYBR Green I. Visualizza il gel con un Transilluminatore Reader Dark.
- Accuratamente asportare il gruppo 223 bp dal gel ed eseguire "crush gel" DNA protocollo di estrazione come segue:
- Posizionare frammenti gel asportati in 0,6 ml MicroCentrifuge tubi che sono stati forati al fondo con un ago 21-G.
- Collocare ciascuna provetta in un tubo 1,5 ml tappo a vite e centrifugare a 16.110 xg (13.200 rpm) per 5 minuti a 4 ° C.
- Aggiungere 200 microlitri TE tampone a pH 8,0 in ciascuna provetta e garantire i pezzi di gel sono completamente immerso nel buffer.
- Congelare a -80 ° C per un'ora e incubare a 37 ° C per una notte.
- Trasferire pezzi gel con il tampone in ciascuna provetta per la tazza del filtro di un filtro provetta da centrifuga e centrifugare a 16.110 xg (13.200 rpm) per 10 minuti a 4 ° C.
- Lavare ogni provetta 1,5 ml con 200 microlitri tampone TE pH 8,0 e buffer di trasferimento risciacquo a ciascuna unità filtro al termine della prima rotazione.
- Pool filtro attraverso e precipitato con isopropanolo.
- Risospendere DNA pellet con 15 microlitri di buffer TE.
7. Controllo della qualità unoAmplificazione d di Chia-PET DNA
- Procedere con il controllo di qualità con Agilent 1000 DNA test o test del DNA Agilent ad alta sensibilità su Agilent Bioanalyzer 2100.
- Il elettroferogramma del test DNA Agilent dovrebbe mostrare un solo picco insieme a una linea di base piatta prima di procedere alla Illumina Genome Analyzer IIx sequenziamento.
- Prima di Illumina Genome Analyzer IIx sequenziamento, Chia-PET DNA ideale dovrebbe avere una concentrazione minima di 10 nM. Eseguire a 4 punti PCR quantitativa in base alla Guida Illumina protocollo Quantificazione qPCR per determinare la quantità di campione da caricare sulla cella di flusso. In alternativa, effettuare un punto quantitativa PCR come segue:
- Diluire il modello di controllo a 100 pM, 10:00 e 13:00. Come descritto nel protocollo Illumina quantificazione qPCR, un modello di controllo è definito come qualsiasi libreria preparato per sequenziamento sulla piattaforma Illumina e dovrebbe essere il più possibile simile in termini ditemplate dimensioni, contenuto di GC e tipo di libreria.
- Diluire il Chia-PET DNA alle 10 di sera.
- Impostare PCR triplica di ciascuna diluizione con 0,1 Primer qPCR ul 1,1, 0,1 Primer qPCR ul 2.1, 5 pl LightCycler480 DNA SYBR Green I Master Mix, 3,8 microlitri priva di nucleasi acqua e 1 ml di modello con Roche Applied Science LightCycler 480 Real-Time PCR System. Includere due controlli negativi con 1 ml di acqua priva di nucleasi come modello.
- Denaturazione iniziale
- 95 ° C 5 minuti 4,8 ° C / s
- Amplificazione
- 95 ° C 10 secondi 4.8 ° C / s
- 60 ° C 1 minuto 2,5 ° C / s
- 72 ° C 30 ° C secondo 4.8/ S
- Curve di fusione
- 95 ° C 5 secondi 4.8 ° C / s
- 65 ° C 1 minuto 2,5 ° C / s
- 95 ° C 5 ° C per acquisizioni
- Raffreddamento
- 40 ° C 10 secondi 2,0 ° C / s
- Denaturazione iniziale
- Assicurarsi che i controlli negativi mostrano alcuna amplificazione e che la deviazione standard dei valori di Ct è inferiore a 0,1 prima di calcolare la concentrazione dei modelli di raccolta basati sulla curva standard generata da diluizioni del modello di controllo.
- Generare cluster sulla superficie delle cellule di flusso Illumina caricando int 01:05Illumina o CBOT Generation System Cluster secondo le istruzioni visualizzate sullo schermo.
- Procedere alla sequenza di Chia-PET DNA Illumina Genome Analyzer IIx con 3-454 fondo Illumina la sequenza e il fondo Illumina 4-454 sequenziamento in una sola corsia per vedere la qualità della biblioteca. Conservare restante Chia-PET DNA a -20 ° C. Se la libreria dispone di buoni dati, preparare il campione per corse supplementari di 4 a 8 corsie, se necessario in base ai risultati corsia di esempio per ottenere 18-20000000 letture unico.
- La quantità di Chia-PET DNA caricata sulle celle di flusso dipende molto dalla versione del software di analisi dei dati di sequenziamento, Studio di controllo Sequencing Illumina. Per la versione 2.9, gli intervalli di concentrazione di carico da un terzo alla metà della capacità massima di ogni piastrella, il che equivale a circa 21 milioni filtrato passa legge con circa 9,5 milioni legge delle tecnologie PET.
- Carico ridotto è necessaria per garantire la corretta base chiamata fo le sequenze di codici a barre dei linker. Questo errore si verifica quando un elevato numero di nucleotidi simili vengono sequenziati, come è il caso se il linker è sequenziato, per ottenere informazioni di codici a barre. Se le informazioni di codici a barre non è desiderato, e le linker non vengono lette, quindi concentrazione pieno carico può essere utilizzato.
- I file convertiti qSeq dal chiamante in linea di base può essere ulteriormente analizzato utilizzando il Chia-PET strumento secondo il Chia-PET utensile manuale di installazione (versione 4.1) 8.
C. rappresentative Chia-PET Risultati
Abbiamo costruito con successo Chia-PET librerie utilizzando RNA polimerasi II anticorpo (8WG16) in cellule MCF-7 (CHM160 e CHM163) 9 usando 672 ng di materiale ChIP come descritto sopra. Il gel diagnostica iniziale, eseguire di questa PCR-amplificato biblioteca, come indicato al punto 6.2 del Chia-PET protocollo visualizzata una banda luminosa e ben definita ala dimensione prevista di 223 coppie di basi per tutto il ciclismo PCR usato (vedi figura 4).
16 cicli di PCR è stato utilizzato per amplificare il Chia-PET libreria e una resa totale di 17,1 ng è stata ottenuta. Un unico picco intenso elettroferogramma è stata osservata alla dimensione prevista di 223 coppie di basi del DNA tramite analisi Agilent 1000 come indicato nella Fase 7,1 del Chia-PET protocollo (vedi figura 5).

Figura 1. Panoramica ChIP. Cellule MCF-7 sono doppio reticolato con EthylGlycol bis (SuccinimidylSuccinate (EGS) e formaldeide in sequenza, con conseguente legami covalenti tra cromatina spazialmente adiacenti. Il reticolato cromatina sono ottenute dai fissi cellule MCF-7 da lisi cellulare e lisi nucleare. La cromatina stata quindi sottoposta a frammentazione per un intervallo di dimensioni di 200-600 coppie di basi. Dopo il pre-compensazione della cromatina sonicato con ProteiG n perline magnetiche per rimuovere il DNA non specifico, il pre-eliminato cromatina stato immunoprecipitato notte con anticorpo perle rivestite per catturare cromatina di interesse.

Figura 2. L'analisi su gel di frammenti cromatina sonicati. Un DNA ladder 100 bp è mostrato nella corsia del primo e dell'ultimo riferimento dimensioni. La cromatina sonicato visualizzata forte intensità tra 200 a 600 bp, che è ideale per catturare interazioni a lungo raggio della cromatina.

Figura 3. Chia-PET visione d'insieme. Frammenti di cromatina sono frammentati end-smussati e legato al biotinilati mezze linkers di accompagnamento contenenti siti di restrizione MmeI. Complessi cromatina intatte vengono poi eluiti al largo delle perline e sottoposti a legatura di prossimità in condizioni estremamente diluita, in modo tale che i frammenti di DNA che interagiscono sono preferentially ligato l'uno all'altro. Dopo l'inversione di reticolazione per rimuovere il DNA proteine associate, digestione MmeI viene effettuata per rilasciare tag-linker-tag (PET) costrutti, che vengono quindi purificato mediante legame selettivo di perline streptavidina. I costrutti PET vengono legati con adattatori per l'high-throughput sequencing.

Figura 4. Analisi Gel di Chia-PET dopo amplificazione PCR. A 25 bp DNA ladder è mostrato in corsia 1 e 5 per la dimensione di riferimento. Le corsie 2 a 4 sono prodotti PCR generati dopo 16, 18 e 20 cicli di amplificazione PCR da 2 pl di template tallone-immobilizzato, rispettivamente. Questa è una libreria di successo, come indicato dalle luminose e ben definite bande con la dimensione prevista di 223 bp. Il non-specifico striscio viene generato quando il numero di cicli PCR è aumentata mentre la banda più bassa di ciascuna reazione PCR è costituita da dimeri di primer.

Figura 5. Agilent 2100 Bioanalyzer analisi del purificata Illumina-454 adattatore legato Chia-PET. Schermo cattura di Agilent 2100 elettroferogrammi Bioanalyzer profilazione una biblioteca di successo, con un singolo picco intensa la dimensione prevista di 223 bp. Si noti che la Agilent Bioanalyzer saggio genere segnala una dimensione leggermente maggiore di quella prevista, in questo caso, il picco desiderato è visualizzato a 237 bp invece di 223 bp. Questo è entro l'intervallo di errore 10% del dosaggio Agilent.
Discussione
Chia-PET è un metodo sviluppato per identificare le interazioni a lungo raggio di regolazione della trascrizione. Uno dei fattori critici che determinano la qualità di un Chia-PET libreria è la qualità del materiale ChIP.
Il protocollo mostrato nel video incorpora l'uso di EGS e formaldeide per reticolare le cellule. L'uso di formaldeide combinato con un secondo reagente di reticolazione che porta un braccio più lungo distanziatore può aiutare nel legame di proteine che non poteva essere vincolati dalla sola formaldeide 3,11,15. Abbiamo costruito librerie con questo metodo, che hanno dimostrato robuste siti di legame e interazioni a lungo raggio 9. Tuttavia, la reticolazione e condizioni chip dovrebbe essere ottimizzato per ogni fattore di interesse, ed è importante non troppo reticolazione come troppo reticolazione comporta difficoltà di frammentazione per sonicazione, e può eventualmente provocare interazioni cromatina spuri . Le interazioni della cromatinaidentificato da Chia-PET dovrebbe essere convalidato con un metodo diverso, come fluorescenza ibridazione in situ 4.
Si consiglia un minimo di 100 ng di materiale cromatina. Mentre abbiamo costruito librerie di buona qualità da 50 ng di materiale cromatina, abbiamo osservato che grandi quantità di materiale di partenza ha permesso la costruzione di Chia-PET librerie con meno di 16 cicli di PCR, amplificati in modo da minimizzare e la ridondanza di ogni libreria. Questa ridondanza inferiore correlato con alti tag unici mappati e anche una percentuale elevata di dati utilizzabili, consentendo così una più completa mappa interazione cromatina con meno corsie di sequenziamento. Il volume finale ricco di perle in ogni tubo dovrebbe essere di 50 ul e 100 microlitri per biglie magnetiche e Sepharose rispettivamente. Se il volume ricco di cordone è inferiore a quella indicata, portare a volume minimo imballato con biglie magnetiche o Sepharose simile pre-autorizzato in bianco per ridurre al minimo la perdita di DNA-cuscinetto tallones nei passaggi successivi. Sawed-off consigli o suggerimenti di grandi dimensioni-core dovrebbe essere usato per pipettaggio perline Sepharose.
Le modifiche seguenti sono stati inseriti a seguito della precedentemente pubblicato con il Chia-PET protocollo 5. In primo luogo, magnetici perle G sono stati utilizzati per ridurre al minimo la perdita del campione durante i lavaggi. Inoltre, abbiamo identificato non-specifici gruppi con dimensioni approssimative di 100 bp e 138 bp per essere ampliconi di auto-legato mezze linkers o / e adattatori. Pertanto, si riduce la concentrazione di biotinilati mezze linkers e 454 GS20 adattatori per ridurre al minimo non-specifiche bande durante l'amplificazione PCR. Il volume vicinanza ligazione è stato ridotto da 50 ml a 10 ml per minimizzare la perdita di campione durante successive fasi di purificazione e di risparmiare sui costi dei reagenti. Abbiamo anche aumentato il tempo di incubazione di immobilizzare Chia-PET DNA a microsfere per garantire la cattura massima di Chia-PET DNA sulle perline streptavidina.
Durante la fase di legatura di prossimità, legature chimerici that non rappresentano vero in vivo interazioni cromatina sono inevitabilmente generate in modo non specifico e casuale. Quindi, per valutare la qualità dei dati da qualsiasi Chia-PET esperimento, il tasso di chimerismo viene stimata dalla uso di due differenti mezze linkers nucleotidica specifica con codici a barre e TAAG ATGT 5. Dopo alta sequenziamento, il Chia-PET sequenze sono dapprima analizzati per la composizione barcode linker e sequenze derivate da prodotti di ligazione specifici e non specifici prodotti di ligazione si possono distinguere 8. La percentuale di chimere conosciute (ad esempio eterodimeri linkers AB) presenti nella nostra società in-house MCF-7 RNA polimerasi II Chia-PET librerie è inferiore al 15%.
Chia-PET sequenze sono poi classificati in due categorie, vale a dire auto-legatura tra PET e PET-legatura. Self-legatura PET sono ottenuti da self-circolarizzazione legatura dei frammenti di cromatina, mentre inter-legatura PET sono derivati da inter-ligazione tra due frammenti di DNA differenti. Quest'ultimo è quindi suddiviso in tre differenti categorie a seconda della distanza genomica di ciascuna etichetta sul cromosoma stesso (intracromosomica inter-ligazione PET) o che entrambi i tag vengono mappati a due cromosomi diversi (interchromosomal inter-ligazione PET). Abbiamo sviluppato una Chia-PET pacchetto software strumento per risolvere le diverse categorie 8. Questo si basa sui frammenti di DNA che sono nella libreria. In generale, i frammenti più piccoli ChIP darà una maggiore risoluzione e la tagliata di questi RNA polimerasi II Chia-PET librerie è di circa 4 kb.
Inoltre, le interazioni cromatina reali possono essere distinte dal rumore casuale contando il numero di inter-ligazione PET in un cluster interazione, in altre parole, un cluster di conteggio PET alto è detto avere una probabilità maggiore di essere una interazione cromatina reale 8 .
Per filtrare i falsi positivi unopassare da ancore altamente arricchito, che può formare inter-legatura PET per caso a caso, un quadro di analisi statistica è stata formulata per spiegare la formazione casuale di qualsiasi inter-legatura animali da compagnia tra due ancore 8.
In conclusione, il Chia-PET tecnica permette di mappare le reti di interazione cromatina su scala globale. L'implementazione di ChIP in Chia-PET consente la riduzione della complessità della libreria e rumore di fondo. Inoltre, aggiunge ChIP specificità per le interazioni della cromatina, consentendo l'esame delle interazioni della cromatina specifici associati a particolari fattori di trascrizione 5.
Divulgazioni
Non ci sono conflitti di interesse dichiarati.
Riconoscimenti
Gli autori sono supportati da A * STAR di Singapore. Inoltre, MJF è supportata da una borsa di studio STAR A * National Science, uno Oreal For Women in Science Fellowship nazionale e Lee Kuan Yew Post-Doctoral Fellowship. YR è supportato da sovvenzioni NIH Codifica HG004456 (R01-01 e R01 HG003521-01). Gli autori riconoscono anche la squadra videografia di 8 Pixel Productions, Singapore, in particolare il signor Kelvin Issey, Mr. Chang Kai Xiang e il signor Sherwin Gan per girare le scene, la signora Siti Rahim per il video editing e la signora Michelle Teo per la voice-over.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reattivo | Azienda | Numero di catalogo | Comments |
| 4-20% di pendenza TBE gel | Invitrogen | EC6225BOX | Fase B, 6.3 |
| 5 x T4 DNA ligasi Buffer con PEG | Invitrogen | 46300018 | Fase B, 2.2 |
| 6% TBE gel | Invitrogen | EC6263BOX | Fase B, 6.8 |
| Agilent DNA 100 Assay | Agilent Technologies | 5067-1504 | Fase B, 7.1 |
| Agilent ad alta sensibilità del test del DNA | Agilent Technologies | 5067-4626 | |
| Agilent Bioanalyzer 2100 | Agilent Technologies | G2940CA | |
| Centrifugare Filtri tubi (Spin-X) | Corning | CLS8160 | Fase B, 3.7 e 6.9 |
| Reader Transilluminatore scuro | Clare Chemical Research | DR46B | Fase B, 6.8 |
| Digital Sonifier cella Disrupter | Branson | 450D-0101063591 | Fase A, 4,3 |
| DynaMag-2 magnete (Concentrator particelle magnetiche) | Invitrogen | 123-21D | Fase A, Fase B 7.5.1, per tutte le biglie magnetiche lavaggio passi |
| DynaMag-15 Magnet (Concentrator particelle magnetiche) | Invitrogen | 123.01D | Fase A, 5 e 6 |
| DynaMag-PCR (Concentrator particelle magnetiche) | Invitrogen | 49-2025 | Fase B, 6.5 |
| Escherichia coli DNA polimerasi I | NEB | M0209 | Fase B, 5,6 |
| GlycoBlue | Ambion | AM9516 | Fase A, Fase B 7.5.1, 4.3 e 6.5 |
| Illumina CBOT System Cluster Generation | Illumina | SY-301-2002 | Fase B, 7.4 |
| Illumina Genome Analyzer IIx | Illumina | SY-301-1301 | Fase B, 7,5 |
| Illumina PE primer | Illumina | PE-102-1004 | Fase B, 5,4 (se necessario) |
| Intelli-Mixer | Palico Biotech | RM-2L | Fase B, le incubazioni con rotazione |
| LightCycler 480 Real-Time PCR System | Roche | 04 640 268 001 | Fase A, Fase B 7.5.3, 7.3 |
| LightCycler480 DNA SYBR Green I MasterMix | Roche | 03 752 186 001 | Fase B, 7.5.3 |
| M-280 streptavidina Dynabeads | Invitrogen | 11206D | Fase B, 5.2 |
| Magnetic (Dynabeads) Proteine G | Invitrogen | 100.03D | Fase A, 5.1.1 e 5.2.1 |
| MaXtract ad alta densità (2 ml) | Qiagen | 129056 | Fase A, 7.5.1 |
| MaXtract ad alta densità (50ml) | Qiagen | 129073 | Fase B, 4.2 |
| MmeI | NEB | R0637 | R0637 |
| RNAsi ONE ribonucleasi | Promega | M426C | Fase B, 4.8 |
| RNA polimerasi II (8WG16) anticorpo monoclonale | Covance | MMS-126R | Fase A, 5.2.4 |
| Provette da centrifuga Oak Ridge (polipropilene) | Nalgene | 3119-0050 | Fase A, 3,1 |
| Provette da centrifuga Oak Ridge (Teflon FEP) | Nalgene | 3114-0050 | Fase B, 4.3 |
| PHUsion ad alta fedeltà Master Mix | Finnzymes | F-531 | Fase B, 6 |
| Picogreen (Quant-iT) reagente dsDNA | Invitrogen | P11495 | Fase A, 7.5.2 |
| Polistirolo Provetta a fondo sferico | BD Biosciences | 352057 | Fase A, 4,1 |
| Cocktail di inibitori della proteasi compresse (completo, EDTA-free) | Roche | 11873580001 | Fase A, 1.6 in poi |
| Proteinasi K Solution (20mg/ml) | Fermentas | E00491 | Fase A, 4.4, 7.5.1 Fase B, 4.1 |
| DNA ligasi T4 | Fermentas | EL0013 | Fase B, 2.2, 3.9 e 5.4 |
| T4 DNA ligasi Buffer (NEB) | NEB | B0202S | Fase B, 3.3 e 3.9 |
| DNA polimerasi T4 | Promega | M4215 | Fase B, 1.2 |
| DNA T4 polinucleotide chinasi | NEB | M0201 | Fase B, 3.3 |
| TruSeq SBS Kit v5-GA | Illumina | FC-104-5001 | Reggenti per Illumina Genome Analyzer IIx sistema |
| TruSeq PE Cluster Kit v2-CBOT-GA | Illumina | PE-300-2001 | per essere utilizzato con il sistema Illumina Generation Cluster CBOT |
| SYBR Green I | Invitrogen | S-7585 | Fase B, 6.3 e 6.8 |
| XCell SureLock Mini-Cell Electrophoresis sistema | Invitrogen | EI0001 | Fase B, 6.3 e 6.8 |
| Nome | Sequenza | Comments | |
| Biotinilato mezze linkers A (200ng/μl) | Top | 5 'GG CCG CGA / iBiodT / ATC TTA TCC AAC 3' | 250 nmoli scala HPLC purificato interno Biotin dT (9) |
| Bot | 5 'GTT GGA TAA GAT ATC GC 3' | 250 nmoli scala HPLC purificato | |
| Biotinilato mezze linkers B (200ng/μl) | Top | 5 'GGC CGC GA / iBiodT / ATA CAT TCC AAC 3' | 250 nmoli scala HPLC purificato interno Biotin dT (9) |
| Bot | 5 'GTT GGA ATG TAT ATC GC 3' | 250 nmoli scala HPLC purificato | |
| Non biotinilati mezze linkers (200ng/μl) | Top | 5 'GGC CGC GAT ATC GGA TCC AAC 3' | 250 nmoli scala PCR Grade |
| Bot | 5 'GTT GGA TCC GAT ATC GC 3' | 250 nmoli scala PCR Grade | |
| GS20 Adaptor A (200ng/μl) | Top | 5 'CAT TCT CCA CCC TGC GTG TCC CATCTG TTC CCT CCC TGT CTC AGN N 3 ' | 250 nmoli scala PCR Grade |
| Bot | 5'CTG AGA CAG GGA GGG AAC AGA TGG GAC ACG CAG GGA TGA GAT GG 3 ' | 250 nmoli scala PCR Grade | |
| GS20 Adattatore B (200ng/μl) | Top | 5 'CTG AGA CAC GCA ACA GGG GAT AGG CAA GGC ACA CAG GGG ATA GG 3' | 250 nmoli scala PCR Grade |
| Bot | 5 'ATC CCC CCT TGT GTG CCT TGC CTA GTT TCC CCT GCG TGT CTC AGN N 3' | 250 nmoli scala PCR Grade | |
| Illumina-NN adattatori | Top | 5 'ACA CTC TTT CCC TAC ACG ACG CTC TTC CGA TC 3' | 250 nmoli scala HPLC purificato Vedi Fase B, 5.4 |
| Bot | 5 'phos - GAT CGG AAG AGC GGT TCA GCA GGA ATG CCG AG 3' | 250 nmoli scala HPLC purificato fosforilata 5 'Vedere B Step, 5,4 | |
| Illumina 1-454 (in avanti) primer (10 pM) | 5 'AAT GAT ACG GCG ACC ACC ACC GAG ATC TAC CTA TCC CCT GTG TGC CTT G 3' | 250 nmoli scala PCR Grade | |
| Illumina 2-454 (reverse) primer (10 micron) | 5 'CAA GCA GAA GAC GGC ATA CGA GAT CGG TCC ATC TCA TCC CTG CGT GTC 3' | 250 nmoli scala PCR Grade | |
| qPCR Primer 1.1 (10 pM) | 5 'AAT GAT ACG GCG ACC ACC GAG A 3' | 10nmole scala PCR Grade | |
| qPCR Primer 2.1 (10 pM) | 5 'CAA GCA GAA GAC GGC ATA CGA 3' | 10nmole scala PCR Grade | |
| Illumina 3-454 sequenziamento primer (100 um) | 5'TGC GTG TCC CAT CTG TTC CCT CCC TGT CTC AG 3 ' | 100nmole scala HPLC purificato | |
| Illumina 4-454 sequenziamento primer (100 um) | 5'GTG CCT TGC CTA GTT TCC CCT GCG TGT CTC AG 3 ' | Scala 100nmoleHPLC purificato | |
Oligo ordine Tabella di oligo e adattatori. Da Integrated DNA Technologies (IDT) e preparare linkers e adattatori nello stesso modo descritto in precedenza 10. Mezze linker e adattatori possono essere preparate in anticipo e conservati per diversi mesi a -20 ° C.
Riferimenti
- Cai, S., Lee, C. C. SATB1 packages densely looped, transcriptionally active chromatin for coordinated expression of cytokine genes. Nat. Genet. 38, 1278-1288 (2006).
- Cook, P. R. The organization of replication and transcription. Science. 284, 1790-1795 (1999).
- Das, P. M., Ramachandran, K. Chromatin immunoprecipitation assay. Biotechniques. 37, 961-969 (2004).
- Deng, W., Blobel, G. A. Do chromatin loops provide epigenetic gene expression states. Curr. Opin. Genet. Dev. 20, 548-554 (2010).
- Fullwood, M. J., Han, Y. Chromatin interaction analysis using paired-end tag sequencing. Curr. Protoc. Mol. Biol. Chapter 21, 21-25 (2010).
- Fullwood, M. J., Liu, M. H. An oestrogen-receptor-alpha-bound human chromatin interactome. Nature. 462, 58-64 (2009).
- Jackson, D. A., Hassan, A. B. Visualization of focal sites of transcription within human nuclei. EMBO J. 12, 1059-1065 (1993).
- Li, G., Fullwood, M. J. ChIA-PET tool for comprehensive chromatin interaction analysis with paired-end tag sequencing. Genome Biol. 11, R22 (2010).
- Li, G., Ruan, X. Extensive Promoter-Centered Chromatin Interactions Provide a Topological Basis for Transcription Regulation. Cell. 148, 84-98 (2012).
- Ng, P., Wei, C. L. Paired-end diTagging for transcriptome and genome analysis. Curr. Protoc. Mol. Biol. Chapter. Chapter 21, 12 (2007).
- Nowak, D. E., Tian, B. Two-step cross-linking method for identification of NF-kappaB gene network by chromatin immunoprecipitation. Biotechniques. 39, 715-725 (2005).
- Osborne, C. S., Chakalova, L. Active genes dynamically colocalize to shared sites of ongoing transcription. Nat. Genet. 36, 1065-1071 (2004).
- Rippe, K., von Hippel, P. H. Action at a distance: DNA-looping and initiation of transcription. Trends Biochem. Sci. 20, 500-506 (1995).
- Schoenfelder, S., Sexton, T. Preferential associations between co-regulated genes reveal a transcriptional interactome in erythroid cells. Nat. Genet. 42, 53-61 (2010).
- Zeng, P. Y., Vakoc, C. R. In vivo dual cross-linking for identification of indirect DNA-associated proteins by chromatin immunoprecipitation. Biotechniques. 41, 694-698 (2006).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon