Method Article
Chromatin Interaction Analysis gepaart mit End-Tag-Sequenzierung (Chia-PET) zur Abbildung von Chromatin-Wechselwirkungen zu verstehen und Transkriptionsregulation
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Chromatin Interaction Analysis von Paired-End-Tag-Sequenzierung (Chia-PET) ist eine Methode zur De-novo- Nachweis von Chromatin-Wechselwirkungen, zum besseren Verständnis der transkriptionellen Kontrolle.
Zusammenfassung
Genome werden in dreidimensionale Strukturen auf, die zur Annahme höherer Ordnung Konformationen innerhalb der Mikrometergröße nuklearen Räumen 7, 2, 12. Solche Architekturen sind nicht zufällig und beinhalten Wechselwirkungen zwischen Gen-Promotoren und regulatorische Elemente 13. Die Bindung von Transkriptionsfaktoren an regulatorische Sequenzen bewirkt ein Netzwerk der Transkriptionssteuerung und Koordinierung 1, 14.
Chromatin Interaction Analysis von Paired-End-Tag-Sequenzierung (Chia-PET) wurde entwickelt, um erkennen diese höherer Ordnung Chromatinstrukturen 5,6. Die Zellen werden fixiert und wechselwirkenden Loci durch kovalente DNA-Protein-Vernetzungen erfasst. Um nicht-spezifische Geräusche zu minimieren und die Komplexität zu reduzieren, sowie um die Spezifität des Chromatins Interaktionsanalyse zu erhöhen, wird Chromatin-Immunopräzipitation (ChIP) gegen spezifische Protein-Faktoren verwendet, um Chromatin-Fragmente von Interesse vor Ligationssysteme bereichern. Ligation mit Halb-Linker bildet anschließend kovalente Verbindungen zwischen Paaren von DNA-Fragmenten zusammen innerhalb einzelner Chromatin-Komplexe gebunden. Die flankierenden MmeI Restriktionsenzymstellen in den Halb-Linker erlauben Extraktion von gepaarten End-Tag-Linker-Tag-Konstrukte (PET) auf MmeI Verdauung. Da die halbe Linker biotinyliert werden, werden diese PET-Konstrukte unter Verwendung Streptavidin-magnetischen Kügelchen. Die gereinigten Haustiere sind mit Next-Generation-Sequencing-Adapter und einem Katalog von wechselwirkenden Fragmente erfolgt über Next-Generation-Sequenzern wie dem Illumina Genome Analyzer generiert ligiert. Mapping und bioinformatische Analyse wird dann durchgeführt, um ChIP angereicherte Bindungsstellen und Chip-angereicherten Chromatin-Wechselwirkungen 8 zu identifizieren.
Wir haben ein Video produziert, um kritische Aspekte des Chia-PET-Protokoll, insbesondere die Vorbereitung der ChIP demonstrieren, wie die Qualität von CHIP spielt eine wichtige Rolle für das Ergebnis eines CHIA-PET-Bibliothek. Da die Protokollesehr lang, werden nur die kritischen Schritte in dem Video gezeigt.
Protokoll
A. Chromatin Immunopräzipitation (ChIP) (siehe Abbildung 1)
1. Dual-Vernetzung von Chromatin-gebundenen Proteine und Zellernte
(Um 02:10 Uhr des Videos)
Chromatin Immunopräzipitation (ChIP) ist der erste kritische Schritt bei der Konstruktion eines CHIA PET-Bibliothek beteiligt. Dieser Schritt ist wichtig, um den Grad der Komplexität, Hintergrundrauschen reduzieren und sich Spezifität. Die Aufstellung von ChIP müssen für Zelltyp und den Faktor Interesse optimiert werden. Dieses Protokoll basiert auf der RNA-Polymerase II CHIA-PET-Bibliothek aus MCF-7 Zellen 9 in unserem Labor hergestellt vorbereitet wird. Um sicherzustellen, dass die resultierende Bibliothek von ausreichender Komplexität ist, empfehlen wir 1 x 10 8 Zellen, um die Chip-Material vorzubereiten. Je nach Ziel und die Zelllinie, kann die Ausbeute von 100 ng bis 300 ng sein. Wir empfehlen, einen Mindestabstand von 100 ng-Chip verwendet werden sollten, um gemeinsamAnwe eine CHIA PET-Bibliothek mit weniger als 20 PCR-Zyklen, um Redundanz bei der Sequenzierung zu minimieren. Höhere Mengen an Chip-Material wird für weitere Senkungen der Redundanz erlauben, die Verbesserung der Qualität der Bibliotheken.
- Waschen Sie 1 x 10 8 MCF-7 Zellen (entspricht fünf 500 cm 2 quadratischen Platten von 2 x 10 7 Zellen) zweimal mit Phosphat-gepufferte Kochsalzlösung (PBS) bei 37 vorgewärmt ° C.
- Crosslink MCF-7 Zellen mit 1,5 mM frisch Ethylglykol Bis (succinimidylsuccinat) (EGS)) für 45 Minuten bei Raumtemperatur (22 ° C) mit Rotation in einem chemischen Abzug bereitet.
- In 37% Formaldehyd in einer Endkonzentration von 1% für 20 Minuten bei Raumtemperatur (22 ° C) mit der Drehung in einem Abzug.

- Quench der Vernetzer durch Zugabe von 2 M Glycin zu einer Endkonzentration von 200 mM. Inkubieren für 10 Minuten bei Raumtemperatur(22 ° C) mit der Drehung.
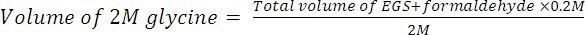
- Entsorgen abgeschreckt Vernetzer in einen Abfallbehälter Kolben und waschen die Zellen zweimal mit kaltem PBS.
- Entsorgen PBS und Ernte Zellen durch Schaben. Halten Sie geernteten Zellen auf Eis. Spülen Sie die Platte mit 5 ml PBS (mit Protease-Inhibitoren ("+ PI") hinzugefügt nach Herstellerangaben) auf die Sammlung von Zellen zu maximieren.
- Pellet die Zellen bei 1800 × g (3000 UpM) für 10 Minuten bei 4 ° C Überstand verwerfen und fahren Sie mit Zell-Lyse. Alternativ speichern Sie das Pellet bei -80 ° C
2. Cell Lysis
(Um 04:15 des Videos)
- Pellet in 15 ml 0,1% SDS Lysepuffer (50 mM HEPES pH 7,5, 150 mM NaCl, 1 mM EDTA, 1% Triton X-100, 0,1% Natriumdesoxycholat, 0,1% SDS + PI) gründlich durch Pipettieren. Drehen für 15 Minuten bei 4 ° C
- Pellet lysierten Zellen bei 800 xg (2000 UpM) für 10 Minuten bei 4 ° C
- Überstand verwerfen und wiederholen Zelllyse einmal gemäß Schritt 2.1 und 2.2.
- Die isolierte Kernpellet ist bereit für nukleare Lyse. Alternativ speichern Pellet bei -80 ° C
3. Nuclear Lysis
(Um 05:00 Uhr des Videos)
Nuclear Lyse durchgeführt wird, um vernetzte Chromatin Chromatin vor Zersplitterung zu lösen. In Abwesenheit der Kernmembran, kann das vernetzte Chromatin Ultraschall behandelt unter Verwendung milder Bedingungen werden. Manchmal, Ultraschallbehandlung Festigkeit kann nicht ausreichend sein, zum Aufbrechen der Kernmembran in diesem Fall weniger Chromatin erhalten, da die Kerne nach der Zentrifugation erhalten werden verworfen werden. Es können jedoch verschiedene Zelltypen erfordern unterschiedliche Bedingungen.
- Resuspendieren isolierten Kernen Pellet in 15 ml 1% SDS-Lysepuffer (50 mM HEPES pH 7,5, 150 mM NaCl, 1 mM EDTA, 1% Triton X-100, 0,1% Natrium Deoxycholate, 1% SDS + PI). Übertragen Kerne Suspension auf eine hohe Geschwindigkeit-Zentrifugenröhrchen (Oak Ridge Zentrifugenröhrchen (Polypropylen)) und drehen für 15 Minuten bei 4 ° C
- Pellet lysiert Kerne Pellet bei 47.810 × g (20.000 UpM) für 30 Minuten bei 4 ° C
- Dekantieren des Überstandes und Pause Pellet mit einem P1000 Pipettenspitze.
- Waschen Sie Pellet mit 30 ml 0,1% SDS-Lysepuffer (+ PI).
- Drehen für 15 Minuten bei 4 ° C
- Pellet Chromatin bei 47.810 × g (20.000 UpM) für 30 Minuten bei 4 ° C
- Überstand verwerfen und wiederholen waschen einmal pro Schritt als 3,4 bis 3,6, bevor Sie mit Chromatin-Fragmentierung. Alternativ speichern Chromatin Pellet bei -80 ° C
4. Die Fragmentierung von Chromatin
(Um 07:03 des Videos)
- Transfer-Chromatin-Pellets in ein 14 ml Polystyrol Rundboden Reagenzglas.
- 1 ml 0,1% SDS-Lysepuffer (+ PI) an Chromatin PElet. Stellen Sie sicher, es gibt keine Blasen in der Mischung, da dies Beschallung Effizienz beeinträchtigt wird.
- Schere Chromatin-DNA zu einer Größe von 200 bis 600 Basenpaaren mit einem Branson Digital Sonifer Zelle Disrupter (Amplitude 35%, 9 Minuten (30 Sekunden, 30 Sekunden aus)) und zu halten Proben kalten jederzeit zu einer Überhitzung zu verhindern, indem die Beschallung in einem kalten Raum (siehe Abbildung 2).
- Reverse-Vernetzung eine aliquote Menge von Chromatin und überprüfen Fragmentierung Effizienz mit den folgenden Schritten. (Die folgenden Schritte sind nicht im Video gezeigt).
- 10 ul Aliquot des Chromatins nach der Beschallung.
- Zentrifuge beschallt Chromatin bei 16.110 × g (13.200 rpm) für 5 Minuten bei 4 ° C
- Überstand in ein neues Gefäß legen und 2 ul Proteinase K-Lösung.
- Inkubieren für 30 Minuten bei 50 ° C
- Auflösen Reverse-vernetzten Chromatin auf einem 1,5% Agarosegel unterworfen.
- REPEAt Beschallung, wenn die Größen der DNA größer als erwartet sind.
- Zentrifuge verbleibenden Lysat bei 16.110 × g (13.200 UpM) für 30 Minuten bei 4 ° C mit Ultraschall behandelt und Übertragung Chromatin (Überstand) in ein neues Röhrchen vor preclearing mit magnetischen Kügelchen. Alternativ beschallt Shop Chromatin bei -80 ° C bis genügend Chromatin gesammelt, um einen Chip zu starten.
5. Waschen, Preclearing und Beschichtung von Antikörpern gegen Perlen
(Um 08:17 Uhr des Videos)
- Preclearing von Chromatin
- Verwenden Sie 300 ul magnetische Protein G-Kügelchen für ein IP.
- Waschen Kügelchen dreimal mit 5 ml Perlen Waschpuffer (PBS, 0,1% Triton X-100). Dieses und zukünftige Waschungen mit magnetischen Protein G-Kügelchen bestehen aus den folgenden Schritten. Stellen Sie sicher, magnetische Kügelchen nicht austrocknen.
- Reclaim Perlen unter Verwendung eines magnetischen Partikels Konzentrator.
- Discard Überstand.
- Perlen mit Puffer resuspendieren.
- Drehen für 5 Minuten bei 4 ° C
- Zentrifuge bei 129 xg (800 rpm) für 1 Minute bei 4 ° C
- Reclaim Perlen unter Verwendung eines magnetischen Partikels Konzentrator.
- Überstand verwerfen.
- Kombinieren Sie beschallten Chromatin mit Perlen vorgewaschen, um Hintergrund Bindung von Chromatin, um Perlen zu entfernen.
- Halten Sie 10 ul beschallten Chromatin als "Input" für die anschließende Prüfung durch die Anreicherung quantitativer PCR (qPCR). Lagerung bei 4 ° C.
- Drehen Sie über Nacht bei 4 ° C.
- Zentrifuge beschallt Chromatin bei 129 xg (800 rpm) für 1 Minute bei 4 ° C
- Platzieren Sie auf dem Rohr Magnetpartikelkonzentrator.
- Coating Antikörper an magnetische Kügelchen
- Aliquotieren 300 ul magnetische Protein G-Kügelchen in ein frisches Röhrchen.
- Waschen Kügelchen dreimal mit 5 ml Waschpuffer Perlen (PBS, 0,1% Triton X-100).
- Fügen sie eine gleichwertige Menge (wie in Schritt 5.1.3) von Perlen Waschpuffer.
- In 35 ug der RNA Polymerase II (8WG16) monoklonaler Antikörper.
- Drehen Sie über Nacht bei 4 ° C.
- Waschen Sie Antikörper-beschichteten Beads zweimal mit 5 ml Waschpuffer Perlen.
- Reclaim Antikörper-beschichteten Kügelchen mit einem Magnetpartikelkonzentrator.
6. Chromatin Immunopräzipitation
(Um 10:03 des Videos)
Um den Grad der Komplexität und Hintergrundgeräusche zu reduzieren, werden Antikörper gegen spezifische Protein-Faktoren verwendet, um spezifische Chromatin-Fragmente von Interesse vor Ligationssysteme 6 bereichern.
Hier haben wir eine Maus RNA-Polymerase II monoklonaleAntikörper (8WG16), die die Einleitung Form des Proteins erkennen. Durch die Anreicherung von DNA-Fragmenten, die mit RNA-Polymerase II, die Spezifität der Bibliothek kann erhöht werden, damit verbunden sind, werden Kennzeichnung von Langstrecken-Chromatin-Wechselwirkungen zwischen aktiven Promotoren und ihre entsprechenden regulatorischen Regionen 9.
- Verwerfen des Waschpuffers von Antikörper-beschichteten Kügelchen mit Hilfe eines magnetischen Partikels Konzentrator.
- Übertragen beschallt Chromatin (Überstand aus Schritt 5.1.6) mit Hilfe der magnetischen Teilchen Konzentrator, um Antikörper-beschichteten Kügelchen (aus Schritt 5.2.7).
- Drehen Sie über Nacht bei 4 ° C.
7. Waschen und Elution von immunpräzipitierten DNA-Protein-Komplexe
(Um 11:13 des Videos)
- Waschen Sie Chromatin-immunpräzipitierten Perlen dreimal mit 5 ml 0,1% SDS-Lysepuffer.
- Waschen Perlen einmal mit 5 ml hohe Salz-Waschpuffer (50 mM HEPES pH 7,5, 350 mM NaCl, 1 mM EDTA, 1% Triton X-100, 0,1% Natriumdesoxycholat, 0,1% SDS).
- Waschen Kügelchen einmal mit 5 ml Lithiumchlorid Waschpuffer (10 mM Tris pH 8,0, 250 mM LiCl, 1 mM EDTA, 0,5% Nonidet P-40, 0,5% Natriumdesoxycholat).
- Entsorgen Waschpuffer resuspendieren und gewaschenen Kügelchen mit 1 ml TE-Puffer.
- Drehen für 5 Minuten bei 4 ° C Die Probe ist bereit für die Quantifizierung und ChIP Bereicherung Scheck. Sobald genügend Chip-Material gesammelt worden ist, die Probe ist bereit, in einen CHIA-PET-Bibliothek aufgebaut werden. Dieser Schritt ist wichtig und muss getan werden, um erfolgreich CHIA PET-Bibliothek Konstruktion zu gewährleisten. ChIP angereicherte Kügelchen können für bis zu 2 Wochen bei 4 ° C gelagert werden, während die Quantifizierung unterzogen, ChIP Bereicherung Schecks und Anhäufung von ausreichend Material.
- Eluieren und Reverse-vernetzt "Eingang" DNA und Chip-angereicherten Partikel mit den folgenden Schritten. (Die folgenden Schritte sind nicht in dem Video gezeigt.)
- Eluieren 20% der ChIP angereicherten Perlen mit 200 ul ChIP Elutionspuffer (50 mM Tris pH 8,0, 10 mM EDTA, 1% SDS). Drehen für 30 Minuten bei 37 ° C Zentrifugieren eluierten Perlen bei 6.100 xg (800 rpm) für 1 Minute bei 4 ° C Übertragen eluierten ChIP Komplexe (Überstand) in ein neues Röhrchen mit Hilfe der magnetischen Teilchen Konzentrator.
- Reverse-Vernetzung "Eingang" und eluiert ChIP Komplexe mit 2 ul Proteinase K (Endkonzentration von 0,2 g / ml) für 2 Stunden bei 50 ° C
- Übertragen Sie Reverse-vernetzte "Input" und eluierten DNA Chip-DNA in ein 2 ml MaXtract High Density (jeweils). Geben Sie 200 ul 25:24:1 Phenol-Chloroform-Isoamylalkohol pH 7,9 zu den Rohren in einer chemischen Abzugshaube und schwenken gut mischen. Drehen Sie die Röhrchen für 5 Minuten bei 16.110 xg (13.200 rpm) bei Raumtemperatur.
- Die obere wässrige Phase in ein neues Röhrchen und fällt rückwärts vernetzten DNA-Witzh 20 ul 3 M Natriumacetat pH 5,5, 200 ul Isopropanol und 1 ul 15 mg / ml Glycoblue. Inkubation bei -80 ° C für 30 Minuten und Pellet DNA für 30 Minuten bei 16.110 × g (13.200 UpM) bei 4 ° C
- Nach der Zentrifugation von Isopropanol gefällte DNA, waschen DNA-Pellet mit 75% eiskaltem Ethanol zweimal und resuspendieren DNA-Pellet in 20 ul TE-Puffer.
- Quantifizierung von "input"-DNA (aus Schritt 5.1.3) und Chip-DNA-Test durch PicoGreen 10.
- Führen Sie eine Bereicherung Scheck über qualitative PCR (qPCR).
- Eluieren und Reverse-vernetzt "Eingang" DNA und Chip-angereicherten Partikel mit den folgenden Schritten. (Die folgenden Schritte sind nicht in dem Video gezeigt.)
B. Chromatin Interaction Analysis mit Paired-End-Tag-Sequenzierung (Chia-PET)
Die zweite Hälfte des Videos beleuchten die wichtigsten Schritte bei der Konstruktion eines CHIA-PET-Bibliothek.
1. End-Abstumpfung der ChIP DNA-Fragmenten
Dieses Kapitel des Video zeigt zwei main Punkte, ein Schritt-für-Schritt-Verfahren des Waschens magnetischen Kügelchen, Enzyme zu entfernen und Puffersalzes der vorhergehenden Reaktion (Schritt 1.1, 12.22 bis 12.54 Uhr des Videos) und dem Verfahren der Einrichtung enzymatische Reaktionen mit magnetischen Kügelchen in der Reaktionsmischung (Schritt 1.2, 12.54 bis 13.25 Uhr von Video).
- Waschen Sie ChIP angereicherten Perlen einmal mit eiskaltem TE-Puffer. Dieses und zukünftige Waschungen mit magnetischen Kügelchen bestehen aus den folgenden Schritten.
- Zentrifugieren bei 6.100 xg (800 rpm) für 1 Minute bei 4 ° C
- Reclaim Perlen unter Verwendung eines magnetischen Partikels Konzentrator.
- Überstand verwerfen.
- Add-Puffer, um Perlen.
- Mix Perlen mit Puffer von Schlenzaktionsdaten.
- Zentrifugieren bei 6.100 xg (800 rpm) für 1 Minute bei 4 ° C
- Reclaim Perlen unter Verwendung eines magnetischen Partikels Konzentrator.
- Überstand verwerfen.
- Um Fill-in beschallten overhaNGS, bereiten Sie einen Master-Mix mit 70 ul 10 x Puffer für T4 DNA-Polymerase, 7 ul 10 mM dNTPs und 615,8 ul Nuklease-freies Wasser. Nach gründlichem Mischen und resuspendieren gewaschenen Kügelchen mit Master-Mix. In 7,2 ul 9,7 U / ul T4 DNA-Polymerase in einem Endvolumen von 700 ul. Mischen und Inkubieren für 40 Minuten bei 37 ° C unter Rotation (mit Palico Biotech Intelli-Mixer RM-2L, F8, 30 UpM, U = 50, a = 60). Führen Sie alle nachfolgenden enzymatischen Reaktionen mit den folgenden Schritten.
- Verdünnen Sie Reaktionspuffer mit Nuklease-freiem Wasser.
- Fügen Sie alle anderen Reagenzien (außer Enzyme) gegen verdünnte Puffer.
- Gründlich mischen. Resuspendieren Perlen / Pellet mit Reaktionsmix.
- Fügen Sie Enzym zu resuspendierten Beads / Pellet (Halten Enzyme auf Benchtop Kühlbox jederzeit die maximale Enzymaktivität zu gewährleisten).
- Verschließen Sie die Röhrchen mit Parafilm.
- Sicherstellen, dass die Perlen gut gemischt sind während der gesamten incubation.
- Entsorgen Sie Reaktionsgemisch und abwaschen Rest Puffersalze und Enzyme dreimal mit eiskaltem Waschpuffer (10 mM Tris-Cl pH 7,5, 1 mM EDTA, 500 mM NaCl).
2. Ligation von biotinylierter Half-Linker an Chip-DNA
In diesem Kapitel des Video zeigt die Merkmale der halben Linker-Oligonukleotide in der Konstruktion von Chia-PET und die Verwendung von Nukleotid-Barcode Zusammensetzung auf zwischen unspezifischen und spezifischen Ligationsprodukte (13.28 bis 14.24 Uhr) der deutlich gemacht werden Video). Das Kapitel zeigt auch die Schritt-für-Schritt-Verfahren für die Einrichtung einer halben Linkerligation Reaktion (Schritt 2.2, 14.24 bis 15.54 Uhr des Videos).
Zwei Arten von biotinylierten halb Linker sind in diesem Protokoll eingeführt und mit einem Barcode aus vier Nukleotiden (TAAG oder ATGT) und einer Erkennungsstelle für die Typ IIS Restriktionsenzym MmeI (TCCAAC) ausgebildet ist. Nach dem Chip zu bereichernment, beschallt Chromatinfragmenten gleichmäßig in zwei Aliquots aufgeteilt und werden zunächst mit einem Überschuss an entweder die Hälfte-Linker ein Linker oder halb-B 5,9 ligiert.
- Teilen Sie ChIP angereicherten Perlen in zwei Aliquots für die DNA-Halb-Linker-Ligation von biotinylierten Halb-Linker A oder Halb-Linker B ein. Diese beiden Halb-Linker die gleichen Nucleotidsequenzen, mit Ausnahme von vier Nukleotiden in der Mitte, (Halb-Linker A mit TAAG, die Hälfte-Linker B mit ATGT) dienen als Nucleotid Barcodes. Wenn während Ligationssysteme, zufällig und nicht-spezifische Ligationen zwischen zwei verschiedenen Chip-Komplexen kombiniert werden aus der Population von Sequenzen mit Heterodimer AB Linker identifiziert werden, wie mit spezifischen Ligationen, die aus der Population von Sequenzen mit Homodimer AA bzw. BB Linker identifiziert werden kann im Vergleich .
- Ligieren ChIP angereicherte Kügelchen mit 140 ul 5 x T4-DNA-Ligase-Puffer mit PEG, 3,5 ul 200 ng / ul biotinylierten halb Linker A oder B, 553,5 ul Nuklease-freies Wasser und 3 ul 30 U / ul T4 DNA-Ligase (Keep T4 DNA-Ligase auf einem Tisch-Kühlbox jederzeit als Ligasen instabil sind sogar auf Eis). Verschließen Sie die Röhrchen mit Parafilm und Inkubation über Nacht bei 16 ° C mit Rotation.
3. Elution und Proximity Ligation von DNA-Fragmenten ChIP
Dieses Kapitel des Video erläutert die Rolle der Puffer EB, SDS und Triton X-100 während der Elution von Chromatin-Komplexe aus Perlen und zeigt auch die Schritt-für-Schritt-Verfahren für die Einrichtung einer Zirkularisierung Reaktion (Schritt 3.9, 15.54 bis 17.10 des Videos).
Nach Ligation der halben Linker an die Chromatin-Fragmente, sind beide Fraktionen vereinigt und die Kügelchen eluiert. Die Interaktion von DNA-Fragmenten wird dann durch eine komplette Linker-Sequenz während Ligationssysteme angeschlossen werden.
Mit dem Barcode-Nukleotid-Zusammensetzung, die Sequenzen in drei Kategorien klassifiziert werden können, nämlich Sequenzen with AB Heterodimer Linker (Barcode ATGT / TAAG) und Sequenzen mit Homodimer AA oder BB-Linker (Barcode TAAG / TAAG oder ATGT / ATGT), um unspezifische und spezifische Ligationsprodukte bzw. 9 zu unterscheiden.
Daher erlaubt die Verwendung von zwei Halb-Linker mit unterschiedlichen Nukleotid Barcodes Spezifikation verschiedener Experimente oder repliziert, sowie zur Überwachung der nicht-spezifische chimäre Ligation Rate zwischen unterschiedlichen Chip-Komplexe 5.
- Waschen Sie Halb-Linker-ligierten Perlen dreimal mit eiskaltem Waschpuffer (10 mM Tris-Cl pH 7,5, 1 mM EDTA, 500 mM NaCl).
- Kombinieren der beiden Rohre halb Linker-ligierten Kügelchen in der folgenden Weise.
- Reclaim eines der Rohre der Hälfte-Linker-ligierten Kügelchen mit einem Magnetpartikelkonzentrator ("Tube A").
- Halten des anderen Rohrs von Halb-Linker-ligierten Kügelchen auf Eis ("Tube B").
- Entsorgen Waschpuffer (aus Schritt 3.2.1) fROM Röhrchen A und übertragen ein Halb-Linker-ligierten Perlen (aus Schritt 3.2.2) von Röhrchen B in Röhrchen A, während Tube A ist immer noch auf der magnetischen Partikel Sammler.
- Fügen Sie 700 ul eiskaltem Waschpuffer Röhrchen B, um alle restlichen halben Linker-ligierten Perlen zu sammeln und sicherzustellen, dass beide Rohre von Halb-Linker-ligierten Perlen richtig kombiniert sind. Übertragen Sie den Waschpuffer in das Rohr A. Entsorgen der leeren Hülse B.
- Phosphorylieren Halb-Linker-ligierten Perlen mit 70 ul 10 x T4-DNA-Ligase-Puffer (NEB), 616 ul Nuklease-freies Wasser und 14 ul 10 U / ul T4 DNA-Polynukleotid-Kinase. Inkubieren für 50 Minuten bei 37 ° C unter Rotation.
- Reclaim phosphorylierten Halb-Linker-ligierten Perlen unter Verwendung eines magnetischen Partikels Konzentrator und entsorgen Reaktionsmix.
- Eluiert halb Linker-ligierten Chromatin-DNA-Komplex mit 200 ul frisch zubereitet Elutionspuffer (TE Puffer, 1% SDS). Inkubieren für 30 Minuten bei Raumtemperatur (22 °C) mit der Drehung.
- Übertragen Eluat in ein frisches Röhrchen und spülen Perlen mit 900 ul Puffer EB. Überstand in dem gleichen Rohr, um die Elution zu kombinieren.
- Übertragen auf die gesammelten Eluat Filtertassen von zwei Zentrifugenröhrchen Filter und Zentrifuge bei 16.110 xg (13.200 rpm) für 1 Minute bei Raumtemperatur (22 ° C).
- Sequestrieren SDS durch Zugabe von 90 ul 20% Triton X-100 und schwenken gut mischen. Inkubation für 1 Stunde bei 37 ° C
- Zirkularisierung unter extrem verdünnten Bedingungen durchgeführt, um Ligation Ereignissen innerhalb der einzelnen vernetzten Chromatin-Komplexe zu bevorzugen, während Minimierung Ligation Ereignisse zwischen verschiedenen Chromatin-Komplexe, die zu unerwünschten chimäre DNA-Moleküle würden. Arbeiten auf Eis, übertragen abgeschreckten Eluat in einem 50 ml Falcon-Röhrchen und fügen 7776 ul Nuklease-freies Wasser, 1000 ul 10 x T4-DNA-Ligase-Puffer (NEB) und 33,33 ul 30 U / ul T4 DNA-Ligase. Inkubieren über Nacht bei 16° C
4. Reverse-Vernetzung und DNA-Reinigung
Dieses Kapitel des Video zeigt die Schritt-für-Schritt-Verfahren von Phenol: Chloroform-Extraktion (Schritt 4.3, 17.11 bis 17.59 Uhr) und die Nahaufnahme des pelletierten DNA nach der Zentrifugation in Schritt 4.4.
- Reverse-Vernetzung und zur Funktionsbeeinträchtigung von Protein durch Zugabe von 100 ul 20 mg / ml Proteinase K-Lösung. Inkubieren für 2 Stunden bei 50 ° C
- Übertragen Chromatin-DNA in einem 50 ml MaXtract High Density und fügen 9ml Nuklease-freiem Wasser auf ein Endvolumen von 19 ml zu erhalten.
- Fügen Sie 19 ml 25:24:1 Phenol-Chloroform-Isoamylalkohol pH 7,9 auf das Rohr in einer chemischen Abzugshaube und schwenken gut mischen. Drehen Sie die Röhrchen für 5 Minuten bei 1.800 xg (3.000 rpm) bei Raumtemperatur.
- Die obere wässrige Phase in ein 50 ml Oak Ridge Zentrifugenröhrchen (Teflon FEP) und Niederschlag Chromatin DNA mit 1,9 ml 3 M Natriumacetat pH 5,5, 19 ml isopropanol und 5 ul Glycoblue. Glycoblue zugegeben, um die Sichtbarkeit des Pellets zu verbessern.
- Inkubation bei -80 ° C für mindestens eine Stunde. Lassen Sie die gefrorene Lösung zum Auftauen vor dem Zentrifugieren bei 38.720 × g (18.000 UpM) für 30 Minuten bei 4 ° C
- Nach der Zentrifugation von Isopropanol gefällte DNA, waschen DNA-Pellet mit 75% eiskaltem Ethanol zweimal und resuspendieren DNA-Pellet in 34 ul Puffer EB. Übertragen DNA-Gemisch zu einem 1,7 ml DNA LoBind Rohr.
- DNA-Pellet resuspendieren in 34 ul Puffer EB.
- (RNase-Behandlung ist optional als anschließendes Waschen und Reinigungsschritte in der Chia-PET-Protokoll dienen zur Entfernung kontaminierender RNA und manuelle Kontrollen des Chia-PET-Sequenzen aus unserer hauseigenen Bibliotheken haben keine Spuren von Verunreinigungen durch RNA angezeigt).
- Führen RNase-Verdau durch Zugabe von 10 ul RNase ONE 10 x Reaktionspuffer, 55 ul Nuklease-freies Wasser und 1 ul 10 U / ul RNase ONE Ribonuclease. Bei 37° C für eine Stunde.
- Übertragen Chromatin DNA in ein 2 ml MaXtract High Density. Geben Sie 100 ul 25:24:1 Phenol-Chloroform-Isoamylalkohol pH 7,9 auf das Rohr in einer chemischen Abzugshaube und schwenken gut mischen. Drehen des Rohrs für 5 Minuten bei 16.110 × g (13.200 UpM) bei Raumtemperatur (22 ° C).
- Die obere wässrige Phase in ein neues Röhrchen und Niederschlag Reverse-vernetzten DNA mit 10 ul 3 M Natriumacetat pH 5,5 und 100 ul Isopropanol. Inkubation bei -80 ° C für 30 Minuten und Pellet DNA für 30 Minuten bei 16.110 × g (13.200 UpM) bei 4 ° C
- Nach der Zentrifugation von Isopropanol gefällte DNA, waschen Sie die DNA-Pellet mit 75% eiskaltem Ethanol zweimal und resuspendieren DNA-Pellet in 34 ul Puffer EB.
5. Immobilisierung von Chia-PET-DNA an Streptavidin Beads
Die Einleitung zu diesem Kapitel spricht über die Präsenzder MmeI Erkennungsstelle und dem biotinylierten T in den halb-Linker-Oligonukleotide, um die Extraktion von Tag-Linker-Tag-Konstrukte ("Privatsphäre", 18.02 bis 19.10 Uhr des Videos) zu erleichtern. Darüber hinaus dieses Kapitel des Videos auch gibt einen schnellen Überblick über Step 5.2, die Schritt-für-Schritt-Verfahren für die Einrichtung einer PCR-Reaktion (Schritt 6.1, 19.10 bis 20.00 Uhr des Videos) und Ausschneiden eines erfolgreichen CHIA PET-DNA (Schritt 6.9, 20.00 bis 20.30 Uhr).
Half-Linker A und B enthalten flankierenden MmeI Erkennungsstellen, mit dem dieser Typ IIS Restriktionsenzym bis 18/20 Basenpaare stromabwärts von ihrer Zielbindesequenz Stellen geschnitten, um kurze "Tags" der Chromatin-Fragment zu erzeugen, Erzeugen paired tag-Linker-Tag-Konstrukte ("Haustiere").
Um die Reinigung von PET-Konstrukte von Streptavidin-beschichteten magnetischen Kügelchen, sowohl Halb-Linker A und B sind mit Biotin modifiziert aktivieren, so dass Abscheidung und Reinigung des Chia-PET-Konstrukte.
- Release Captured CHIA PET-DNA durch Zugabe von 5 ul 10 x NEB-Puffer 4, 5 ul frisch zubereitet 500 uM (10 x) S-Adenosylmethionin (SAM) und 1 ul 2 U / ul MmeI zu resuspendierten DNA. Quench überschüssige MmeI durch Zugabe von 5 ul nicht biotinylierte Halb-Linker an Reaktionsmix für MmeI können selbst-hemmend im Übermaß. Inkubieren für 2 Stunden bei 37 ° C
- Um freigegeben CHIA PET-DNA, Transfer-50 ul des resuspendierten M-280 Streptavidin-Beads mit einem DNA LoBind Tube zu erfassen. Waschen Perlen zweimal mit 2 x Bindungs-und Waschpuffer (2 x B & W: 10 mM Tris-HCl pH 7,5, 1 mM EDTA, 2 M NaCl). Resuspendieren Sie Perlen in 50 ul 2 x B & W Puffer und 50 ul übertragen Verdauung Mix (aus Schritt 5.1) auf die gleiche Röhre. Gut mischen und inkubieren für 45 Minuten bei Raumtemperatur unter Rotation.
- Reclaim Perlen mit einem magnetischen Konzentrator Teilchen und Überstand verwerfen. Waschen Perlen zweimal mit je 150 ul 1 x Bindungs-und Waschpuffer (1 x B & W: 5 mM Tris-HCl pH 7,5, 0,5 mM EDTA, 1 M NaCl).
- Ligieren 454 GS20 Adapter nach Chia-PET-DNA mit 5 ul 10 × T4-DNA-Ligase-Puffer (Beachten Sie, dass die Ligase-Puffer in diesem Schritt verwendet verschieden von Schritt 3.9 ist, ist die Ligase-Puffer in diesem Schritt verwendet der Firma als das, was liefert die T4 DNA Ligase), 4 ul 200 ng / ul 454 GS20 Adapter A, 4 ul 200 ng / ul 454 GS20 Adapter B, 36 ul Nuklease-freies Wasser und 1 ul 30 U / ul T4 DNA-Ligase. Inkubieren über Nacht bei 16 ° C unter Rotation. CHIA-PET DNA kann auch mit Illumina-NN-Adapter ligiert werden, wobei wir Amplifikation der Bibliothek unter Verwendung PCR-Primer und PCR-Primer PE1.0 PE 2.0 von Illumina gekauft empfohlen.
- Reclaim Perlen mit einem magnetischen Konzentrator Teilchen und Überstand verwerfen. Waschen Perlen dreimal mit 150 ul 1 x B & W-Buffer.
- Nick übersetzen CHIA PET-DNA mit 5 ul 10 x NEB-Puffer 2, 2,5 ul 10 mM dNTPs, 38,5 ul Nuklease-freies Wasser und 4 ul 10 U / ul Escherichia coli DNA-Polymerase I Inkubieren über Nacht beiRaumtemperatur (22 ° C) mit der Drehung.
- Reclaim Perlen mit einem magnetischen Konzentrator Teilchen und Überstand verwerfen. Waschen Perlen dreimal mit 150 ul 1 x B & W-Buffer.
- Resuspendieren Perlen mit 50 ul Puffer EB.
6. Verstärkung von Chia-PET-DNA
- Richten Sie PCR-Reaktionen mit 2 ul Perlen Federung, 21 ul Nuclease freies Wasser, 1 ul 10 uM Illumina 1-454 Primer, 1 ul 10 uM Illumina 2-454 Primer und 25 ul Phusion High-Fidelity-Master Mix.
- Um die Anzahl der Zyklen notwendig, um genug PCR-Produkte für die Sequenzierung generieren zu bestimmen, Einrichtung von drei Test-PCR-Reaktionen mit 16, 18 und 20 Zyklen. Nicht mehr als 20 Zyklen, wie wir fanden, dass dies führt zu sehr niedrigen Bibliothek Komplexität haben.
| Erste Schritt | 30 Sekunden | 98 ° C | (Denaturierung) |
| 18 bis 25 Zyklen | 10 Sekunden | 98 ° C | (Denaturierung) |
| 30 Sekunden | 65 ° C | (Annealing) | |
| 30 Sekunden | 72 ° C | (Erweiterung) | |
| Letzter Schritt | 5 Minuten | 72 ° C | (Endausbau) |
- Bestimmen der niedrigsten Zykluszahl, indem Sie die PCR-Reaktionen auf einem 4-20% Gradienten TBE-Gel und Färbung mit SYBR Green I, Sicherstellen der Anwesenheit eines Bandes von 223 Basenpaaren, die die Länge des Chia-PET DNA ist.
- Erhöhen der Rest des Chia-PET DNA-gebundene Streptavidin-Kügelchen in einem großen PCR mit so wenig wie möglich PCR-Zyklen, während immer noch ausreichende Ausbeute zu erhalten für die Sequenzierung.
- ReAnspruch Perlen aus allen PCR-Reaktionen mit Hilfe eines magnetischen Partikels Konzentrator und Pool Überstand in zwei frische DNA LoBind Röhrchen. Man fällt jedes Rohr der Überstand durch Zugabe von 60 ul 3 M Natriumacetat, 2 ul GlycoBlue und 600 ul Isopropanol auf gepoolten Reaktionen.
- Inkubation bei -80 ° C für 30 Minuten. Zentrifuge bei 16.110 × g (13.200 UpM) für 30 Minuten bei 4 ° C
- Nach der Zentrifugation von Isopropanol gefällte DNA-, Wasch-DNA-Pellet mit 75% eiskaltem Ethanol zweimal und resuspendieren DNA-Pellet in 100 ul TE-Puffer und 5 ul 6 x Loading-Farbstoff.
- Verteilen PCR-Produkte gleichmäßig in die Wells einer 6% TBE-Gel. Führen Sie das Gel in 1 x TBE-Puffer bei 200 V für 35 Minuten und Flecken mit SYBR Green I. Visualisieren Sie das Gel mit einem dunklen Reader Transilluminator.
- Sorgfältig Verbrauchsteuern die 223 bp Bande aus dem Gel und führen Sie "Gel Crush" DNA-Extraktions-Protokoll wie folgt:
- Platzieren Sie exzidiert Gelfragmente in 0,6 ml MicroCentrifuge Rohre, die an der Unterseite wurden mit einer 21-G-Nadel durchbohrt.
- Platzieren Sie jede Röhre innerhalb einer 1,5-ml-Tube und Schraubverschluss Zentrifuge bei 16.110 xg (13.200 rpm) für 5 Minuten bei 4 ° C
- Geben Sie 200 ul TE-Puffer pH 8,0 zu jedem Röhrchen und sicherzustellen, dass die Gel-Stücke sind voll im Puffer eingetaucht.
- Einfrieren bei -80 ° C für eine Stunde und bei 37 ° C über Nacht.
- Übertragen Gelstücke zusammen mit dem Puffer in jedem Röhrchen zu dem Filter Tasse ein Zentrifugenröhrchen Filter und Zentrifuge bei 16.110 × g (13.200 UpM) für 10 Minuten bei 4 ° C
- Spülen je 1,5 ml-Röhrchen mit 200 ul TE-Puffer pH 8,0 und Übertragung Spülpuffer jeder Filtereinheit nach Abschluss der ersten Spin.
- Pool-Filter und durch Niederschlag mit Isopropanol.
- Resuspendieren DNA-Pellet mit 15 ul TE-Puffer.
7. Quality Check einD-Verstärkung von Chia-PET-DNA
- Fahren Sie mit dem Qualitäts-Check mit Agilent DNA-Test 1000 oder Agilent High Sensitivity DNA-Test auf Agilent Bioanalyzer 2100.
- Das Elektropherogramm der Agilent DNA-Test sollte nur einen Peak zusammen mit einer flachen Basislinie angezeigt, bevor Sie mit Illumina Genome Analyzer IIx-Sequenzierung.
- Vor dem Illumina Genome Analyzer IIx-Sequenzierung, Chia-PET-DNA im Idealfall sollte eine minimale Konzentration von 10 nM. Führen Sie eine 4-Punkt-quantitative PCR nach Anspruch Illumina qPCR Quantifizierung Protocol Guide, um die Menge der Probe auf die Durchflusszelle Last zu bestimmen. Alternativ führen eine Ein-Punkt-quantitative PCR wie folgt:
- Verdünnen Sie die Steuerelementvorlage bis 100 pM, 10.00 Uhr und 01.00 Uhr. Wie in Illumina qPCR Quantifizierung Protokoll beschrieben, wird eine Kontrolle Vorlage als jeder Bibliothek für die Sequenzierung auf die Illumina-Plattform vorbereitet definiert und es sollte so ähnlich wie möglich in Bezug aufVorlage Größe, GC-Gehalt und eine Bibliothek Typ.
- Verdünnen Sie die Chia-PET-DNA, um 10.00 Uhr.
- Richten Sie PCR Triplikate jeder Verdünnung mit 0,1 ul qPCR Primer 1,1, 0,1 ul qPCR Primer 2,1, 5 ul LightCycler480 DNA SYBR Green I Master Mix, 3,8 ul Nuklease-freies Wasser und 1 ul Template mit Roche Applied Science LightCycler 480 Real-Time PCR-System. Gehören zwei negative Kontrollen mit 1 ul Nuklease-freies Wasser als Vorlage.
- Initiale Denaturierung
- 95 ° C 5 Minuten 4,8 ° C / s
- Verstärkung
- 95 ° C 10 Sekunden 4,8 ° C / s
- 60 ° C 1 Minute 2,5 ° C / s
- 72 ° C 30 Sekunden 4,8 ° C/ S
- Schmelzkurvenanalyse
- 95 ° C 5 Sekunden 4,8 ° C / s
- 65 ° C 1 Minute 2,5 ° C / s
- 95 ° C 5 ° C pro Akquisitionen
- Kühlung
- 40 ° C 10 Sekunden 2,0 ° C / s
- Initiale Denaturierung
- Stellen Sie sicher, dass die negativen Kontrollen keine Verstärkung und zeigen, dass die Standardabweichung für die CT-Werte kleiner als 0,1 vor der Berechnung der Konzentration der Bibliothek Vorlagen auf der Standardkurve aus den Steuerelementvorlage Verdünnungen erzeugt werden.
- Generieren Cluster auf der Oberfläche der Illumina Fließzellen durch Laden 1.05 into Illumina CBOT Cluster-Generation System nach Anweisungen am Bildschirm.
- Gehen Sie zur Folge CHIA PET-DNA auf Illumina Genome Analyzer IIx mit Illumina 3-454 Sequenzierprimers und Illumina 4-454 Sequenzierprimers in einer einzigen Spur, um die Qualität der Bibliothek zu sehen. Bewahren verbleibenden CHIA PET-DNA bei -20 ° C Wenn die Bibliothek verfügt über gute Daten, bereiten Sie die Probe für weitere Läufe von 4 bis 8 Bahnen wie auf der Grundlage der Ergebnisse benötigte Probe Fahrspur auf 18 bis 20 Millionen Unique mal gelesen zu erhalten.
- Der Betrag von Chia-PET-DNA auf den Fluss Zellen geladen hängt stark von der Version der Datenanalyse-Software-Sequenzierung, Illumina Sequencing Control Studio. Für Version 2.9 liest die Be-Konzentration im Bereich von einem Drittel bis der Hälfte der maximalen Kapazität jeder Fliese, die auf rund 21 Millionen hochpaßgefilterte entspricht mit etwa 9,5 Millionen liest von Haustieren.
- Reduzierter Belastung ist notwendig, um korrekte Basis-calling f gewährleistenoder die Barcode-Sequenzen der Linker. Dieser Fehler tritt auf, wenn eine große Anzahl von ähnlichen Nukleotide sequenziert werden, wie es der Fall, wenn der Linker sequenziert, um Strichcodes zu erhalten. Wenn der Barcode Informationen nicht erwünscht ist, und die Linker nicht gelesen werden, dann beladener Konzentration verwendet werden.
- Die qSeq Dateien, die vom Anrufer offline Basis umgewandelt werden weiter analysiert werden über die Chia-PET-Tool nach der Chia-PET-Tool Installationshandbuch (Version 4.1) 8.
C. Repräsentative CHIA-PET-Ergebnisse
Wir haben erfolgreich CHIA-PET-Bibliotheken unter Verwendung von RNA Polymerase II Antikörper (8WG16) in MCF-7-Zellen (CHM160 und CHM163) 9 mit 672 ng des Chip-Material, wie oben beschrieben aufgebaut. Der erste diagnostische Gel dieser PCR-amplifizierten Bibliothek laufen, wie in Schritt 6.2 des Chia-PET-Protokoll erwähnt, erscheint eine helle und gut definierte Bande beidie erwartete Größe von 223 Basenpaaren für alle PCR-Cycling verwendet werden (siehe Abbildung 4).
16 PCR-Zyklen wurde verwendet, um die Chia-PET-Bibliothek amplifiziert und eine Gesamtausbeute von 17,1 ng erhalten. Eines einzigen, intensiven Elektropherogramm Peak bei der erwarteten Größe von 223 Basenpaaren über Agilent DNA-Analyse 1000 beobachtet, wie in Schritt 7.1 des Chia-PET-Protokoll (siehe Abbildung 5) erwähnt.

Abbildung 1. ChIP Übersicht. MCF-7-Zellen sind mit zwei Ethylglykol Bis (succinimidylsuccinat (EGS) und Formaldehyd sequentiell, was zu kovalenter Verbindungen zwischen räumlich benachbarten Chromatin vernetzt sind. Die vernetzte Chromatin aus den festen MCF-7-Zellen wurde durch Zell-Lyse erhaltenen und nukleare Lyse. Das Chromatin wurde dann zu einer Fragmentierung zu einer Größe von 200-600 Basenpaaren unterzogen. Nach vor dem Löschen des beschallten Chromatin mit Protein G magnetische Kügelchen, um nicht-spezifische DNA zu entfernen, die bereits geklärten Chromatin wurde über Nacht mit Antikörper-beschichteten Beads immunpräzipitiert an Chromatin von Interesse zu erfassen.

Abbildung 2. Gelanalyse von beschallten Chromatinfragmenten. Ein 100 bp DNA-Leiter in dem ersten und letzten Bahn für eine Größe Referenz gezeigt. Das beschallte Chromatin angezeigt starke Intensität zwischen 200 bis 600 bp, die ideal auf Langstrecken-Chromatin-Wechselwirkungen zu erfassen ist.

Abbildung 3. CHIA-PET-Übersicht. Fragmentierte Chromatin-Fragmente sind Ende abgestumpft und ligiert, um biotinylierte Halb-Linker, die flankierenden MmeI Restriktionsstellen. Intakte Chromatin-Komplexe werden dann von den Kügelchen eluiert und Ligationssysteme unter extrem verdünnten Bedingungen, so dass die Interaktion von DNA-Fragmenten sind preferentially miteinander ligiert. Nach Umgekehrte Vernetzung an DNA-assoziierte Proteine zu entfernen, wird MmeI Verdau tag-Linker-Tag (PET)-Konstrukten, die dann durch selektive Bindung an Streptavidin-Kügelchen gereinigt Freigabe durchgeführt. Die PET-Konstrukte werden mit Adaptern für Hochdurchsatz-Sequenzierung ligiert.

Abbildung 4. Gel-Analyse von Chia-Haustiere nach PCR-Amplifikation. Ein 25 bp DNA-Leiter ist in Spur 1 und 5 für die Größe Referenz gezeigt. Die Spuren 2 bis 4 sind PCR-Produkte nach 16 erzeugt wird, 18 und 20 Zyklen PCR-Amplifikation von 2 ul Kügelchen immobilisierte Templat bzw.. Dies ist eine erfolgreiche Bibliothek, wie sie von den hellen, gut definierten Banden bei der erwarteten Größe von 223 bp angegeben. Die unspezifische Schmierschicht erzeugt wird, wenn die Anzahl der PCR-Zyklen erhöht wird, während das unterste Band jeder PCR-Reaktion wird von Primer-Dimeren aus.

Abbildung 5. Agilent 2100 Bioanalyzer Analyse der gereinigten Illumina-454 Adapter-ligierten CHIA-Pets. Screen Capture von Agilent 2100 Bioanalyzer Elektropherogramme Profilerstellung für eine erfolgreiche Bibliothek, mit einem einzigen intensiven Peak bei der erwarteten Größe von 223 bp. Beachten Sie, dass die Agilent Bioanalyzer Assay Regel meldet einen etwas höher als erwartet groß, in diesem Fall die gewünschte Peak bei 237 bp anstelle von 223 bp angezeigt. Dies ist innerhalb der 10% Fehlerbereich des Agilent-Assay.
Diskussion
CHIA-PET ist eine Methode entwickelt, um weitreichende Wechselwirkungen in Transkriptionsregulation identifizieren. Einer der kritischen Faktoren, die die Qualität eines CHIA PET-Bibliothek bestimmen, ist die Qualität der Chip-Material.
Das Protokoll, in dem Video gezeigt, beinhaltet die Verwendung von EGS und Formaldehyd zu vernetzen die Zellen. Die Verwendung von Formaldehyd mit einem zweiten Vernetzungsmittel mit längerer Spacer-Arm, kann die Bindung von Proteinen, die nicht allein durch Formaldehyd gebunden 3,11,15 könnte helfen. Wir haben Bibliotheken mit dieser Methode, die robust Bindungsstellen und weitreichenden Wechselwirkungen 9 gezeigt haben, gebaut. Jedoch kann die Vernetzung und ChIP Bedingungen sollen für jeden Faktor von Interesse optimiert werden, und es ist wichtig, nicht zu stark vernetzt, wie viel der Vernetzung in Schwierigkeiten bei der Fragmentierung durch Beschallung führt und möglicherweise zu falschen Chromatin-Wechselwirkungen führen . Chromatin-Wechselwirkungenidentifiziert von Chia-PET sollte nach einem anderen Verfahren, wie die Fluoreszenz-in situ-Hybridisierung 4 validiert werden.
Wir empfehlen einen Mindestabstand von 100 ng von Chromatin Material. Während wir gute Qualität Bibliotheken aus 50 ng von Chromatin Material konstruiert haben, haben wir beobachtet, dass große Mengen an Ausgangsmaterial erlaubt Bau von Chia-PET-Bibliotheken mit weniger als 16 PCR-Zyklen und minimiert dadurch Amplikons und Redundanz der einzelnen Bibliotheken. Diese geringere Redundanz mit höheren einmaligen abgebildet Tags und auch einem hohen Anteil an verwertbaren Daten korreliert, wodurch eine umfassendere Interaktion Chromatin Karte mit weniger Fahrspuren der Sequenzierung. Das endgültige Volumen der Kugeln verpackt in jedem Rohr sollte 50 ul und 100 ul für magnetische und Sepharosekügelchen betragen. Wenn die Raupe gepackten Volumen ist kleiner als angegeben, auf das Minimum verpackt Volumen mit ähnlich bereits geklärten unbespielten magnetischen oder Sepharosekügelchen bringen, um den Verlust von DNA-tragenden Wulst minimierens in den nachfolgenden Schritten. Abgesägte Tipps oder großen Kern-Tipps sollten zum Pipettieren Sepharosekügelchen verwendet werden.
Folgende Modifikationen wurden nach dem zuvor veröffentlichten CHIA-PET-Protokoll 5 eingearbeitet. Erstens wurden magnetische Beads G verwendet werden, um durch Verluste während Wäschen zu minimieren. Darüber hinaus haben wir festgestellt, unspezifische Banden mit den ungefähren Größen von 100 bp und 138 bp bis Amplikons von selbst ligiert, Halb-Linker oder / und Adapter sein. Daher konnten wir die Konzentration von biotinyliertem halb Linker und 454 GS20 Adapter unspezifische Banden während der PCR Amplifikation zu minimieren. Die Ligationssysteme Volumen von 50 ml bis 10 ml eingeengt, um durch Verluste während nachfolgenden Reinigungsschritte zu minimieren und auch an Reagenz zu sparen. Wir erhöhte auch die Inkubationszeit nach Chia-PET-DNA zu immobilisieren Perlen, um eine maximale Aufnahme von Chia-PET-DNA auf den Streptavidinbeads zu gewährleisten.
Während der Nähe Ligationsschritt, chimäre Ligierungen that nicht die wahre in vivo Chromatin-Wechselwirkungen zwangsläufig in einer nicht-spezifischen und zufällige Weise erzeugt. Um daher die Qualität der Daten von jedem CHIA-PET Erfahrung zu evaluieren, wird die Geschwindigkeit des Chimärismus aus der Verwendung von zwei unterschiedlichen Halbwertszeiten Linker mit spezifischen Nukleotid Barcodes TAAG und ATGT 5 geschätzt. Nach dem Hochdurchsatz-Sequenzierung, werden die Chia-PET-Sequenzen zunächst für Barcode-Linker Zusammensetzung und spezifische Sequenzen aus Ligationsprodukte und unspezifische Ligationsprodukte abgeleitet analysiert unterschieden werden 8. Der Prozentsatz der bekannten Chimären (dh Heterodimere AB Linker e) in der hauseigenen MCF-7-RNA-Polymerase II CHIA-PET-Bibliotheken weniger als 15% ist.
CHIA PET-Sequenzen anschließend in zwei Kategorien, nämlich Selbst-Ligation Privatsphäre und inter-Ligation Tierbörse. Selbst-Ligation HAUSTIERE sind aus selbständiger Zirkularisierung Ligation der Chromatin-Fragmente erhalten werden, während inter-Ligation Haustiere von i abgeleitet werdennter-Ligation zwischen zwei verschiedenen DNA-Fragmente. Letzteres ist dann in drei verschiedene Kategorien auf der genomischen Abstand von jedem Tag auf dem gleichen Chromosom (intrachromosomale inter-Ligation PET) oder dass beide Variablen werden auf zwei verschiedenen Chromosomen (interchromosomale inter-Ligation PETs) abgebildet basierend unterteilt. Wir haben eine CHIA-PET-Tool-Software-Paket zum Aussortieren der verschiedenen Kategorien 8 entwickelt. Dies wird auf der DNA-Fragmente, die in der Bibliothek gründen. Im Allgemeinen werden kleinere Chip-Fragmente geben eine höhere Auflösung und die sich für diese RNA Polymerase II CHIA-PET-Bibliotheken zugeschnitten ist ca. 4 kb.
Darüber hinaus realen Chromatin-Wechselwirkungen von Rauschen kann durch Zählen der Anzahl von inter-Ligation Privatsphäre in einer Interaktion Cluster zu unterscheiden, mit anderen Worten, wird eine Gruppe von PET hohen Anzahl der eine höhere Wahrscheinlichkeit hat, eine echte Wechselwirkung Chromatin 8 weisen .
Um unsere Fehlalarme Filter Asteigen von hoch angereichertem Anker, die inter-Ligation Privatsphäre durch Zufall entstehen kann, ist eine statistische Analyse Rahmen auch formuliert worden, um für zufällige Bildung aller zwischenzeitlich Ligation PETs zwischen zwei Ankern 8 ausmachen.
Im Ergebnis ermöglicht das Chia-PET-Technik die Abbildung Chromatin Interaktion Netzwerke auf globaler Ebene. Die Umsetzung der ChIP in Chia-PET ermöglicht die Reduzierung der Komplexität und der Bibliothek Hintergrundrauschen. Darüber hinaus fügt ChIP Spezifität für Chromatin-Wechselwirkungen, so dass die Untersuchung von Wechselwirkungen mit spezifischen Chromatin bestimmte Transkriptionsfaktoren 5 zugeordnet.
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Die Autoren werden von A * STAR Singapur unterstützt. Darüber hinaus wird durch eine MJF A * STAR National Science Scholarship, ein L'Oreal For Women in Science Fellowship Nationale und Lee Kuan Yew Postdoc-Stipendium unterstützt. YR ist durch NIH ENCODE Zuschüsse (R01 HG004456-01 und R01 HG003521-01) unterstützt. Die Autoren erkennen die Videografie Team von 8 Pixel Productions, Singapur, insbesondere Herr Kelvin Issey, Mr. Chang Xiang Kai und Herr Sherwin Gan für die Dreharbeiten der Szenen, Frau Siti Rahim für die Videobearbeitung und Frau Michelle Teo für die Voice-Over.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | Kommentare |
| Gradienten von 4-20% TBE-Gel | Invitrogen | EC6225BOX | Schritt B, 6.3 |
| 5 x T4 DNA-Ligase-Puffer mit PEG | Invitrogen | 46300018 | Schritt B, 2.2 |
| 6% TBE-Gel | Invitrogen | EC6263BOX | Schritt B, 6.8 |
| Agilent DNA-Test 100 | Agilent Technologies | 5067-1504 | Schritt B, 7,1 |
| Agilent High Sensitivity DNA-Test | Agilent Technologies | 5067-4626 | |
| Agilent Bioanalyzer 2100 | Agilent Technologies | G2940CA | |
| Reaktionsgefäßständer Filter (Spin-X) | Corning | CLS8160 | Schritt B, 3,7 und 6,9 |
| Dunkle Reader Transilluminator | Clare Chemical Research | DR46B | Schritt B, 6.8 |
| Digitale Sonifier Zelle Disrupter | Branson | 450D-0101063591 | Schritt A, 4.3 |
| Dynamag-2 Magneten (Magnetic Particle Concentrator) | Invitrogen | 123-21D | Schritt A, 7.5.1 Schritt B für alle magnetischen Kügelchen Waschschritte |
| Dynamag-15 Magnet (Magnetic Particle Concentrator) | Invitrogen | 123.01D | Schritt A, 5 und 6 |
| Dynamag-PCR (Magnetic Particle Concentrator) | Invitrogen | 49-2025 | Schritt B, 6,5 |
| Escherichia coli-DNA-Polymerase I | NEB | M0209 | Schritt B, 5.6 |
| GlycoBlue | Ambion | AM9516 | Schritt A, 7.5.1 Schritt B, 4,3 und 6,5 |
| Illumina CBOT Cluster-Generation System | Illumina | SY-301-2002 | Schritt B, 7.4 |
| Illumina Genome Analyzer IIx | Illumina | SY-301-1301 | Schritt B, 7,5 |
| Illumina PE-Primer | Illumina | PE-102-1004 | Schritt B, 5,4 (falls erforderlich) |
| Intelli-Mixer | Palico Biotech | RM-2L | Schritt B, keine Inkubationen mit Rotation |
| LightCycler 480 Real-Time PCR-System | Roche | 04 640 268 001 | Schritt A, 7.5.3 Schritt B, 7.3 |
| LightCycler480 DNA SYBR Green I MasterMix | Roche | 03 752 186 001 | Schritt B, 7.5.3 |
| M-280 Streptavidin Dynabeads | Invitrogen | 11206D | Schritt B, 5.2 |
| Magnetische (Dynabeads) Protein G | Invitrogen | 100.03D | Schritt A, 5.1.1 und 5.2.1 |
| MaXtract High Density (2 ml) | Qiagen | 129056 | Schritt A, 7.5.1 |
| MaXtract High Density (50ml) | Qiagen | 129073 | Schritt B, 4.2 |
| MmeI | NEB | R0637 | R0637 |
| RNase ONE Ribonuclease | Promega | M426C | Schritt B, 4,8 |
| RNA-Polymerase II (8WG16) monoklonaler Antikörper | Covance | MMS-126R | Schritt A, 5.2.4 |
| Oak Ridge Zentrifugenröhrchen (Polypropylen) | Nalgene | 3119-0050 | Schritt A, 3.1 |
| Oak Ridge Zentrifugenröhrchen (Teflon FEP) | Nalgene | 3114-0050 | Schritt B, 4.3 |
| PHUsIonen-High-Fidelity-Master Mix | Finnzymes | F-531 | Schritt B, 6 |
| PicoGreen (Quant-iT) dsDNA Reagenz | Invitrogen | P11495 | Schritt A, 7.5.2 |
| Polystyrol Rundboden Test Tube | BD Biosciences | 352057 | Schritt A, 4.1 |
| Protease Inhibitor Cocktail Tablets (komplett, EDTA-frei) | Roche | 11873580001 | Schritt A, 1,6 ab |
| Proteinase K-Lösung (20mg/ml) | Fermentas | E00491 | Schritt A, 4.4, 7.5.1 Schritt B, 4.1 |
| T4 DNA-Ligase | Fermentas | EL0013 | Schritt B, 2.2, 3.9 und 5.4 |
| T4-DNA-Ligase-Puffer (NEB) | NEB | B0202S | Schritt B, 3.3 und 3.9 |
| T4 DNA-Polymerase | Promega | M4215 | Schritt B, 1,2 |
| T4-DNA-Polynukleotidkinase | NEB | M0201 | Schritt B, 3.3 |
| TruSeq SBS Kit v5-GA | Illumina | FC-104-5001 | Regents für Illumina Genome Analyzer IIx-System |
| TruSeq PE Cluster Kit v2-CBOT-GA | Illumina | PE-300-2001 | auf den Einsatz mit Illumina CBOT Cluster Generation System sein |
| SYBR Green I | Invitrogen | S-7585 | Schritt B, 6.3 und 6.8 |
| XCell SureLock Mini-Cell Elektrophorese-System | Invitrogen | EI0001 | Schritt B, 6.3 und 6.8 |
| Name | Reihenfolge | Kommentare | |
| Biotinyliertes halben Linker A (200ng/μl) | Top- | 5 'GG CCG CGA / iBiodT / ATC TTA TCC AAC 3' | 250 nmol Maßstab HPLC gereinigte Interne Biotin dT (9) |
| Bot | 5 'GTT GGA TAA GAT ATC GC 3' | 250 nmol Maßstab HPLC-gereinigten | |
| Biotinyliertes halben Linker B (200ng/μl) | Top- | 5 'CGC GGC GA / iBiodT / ATA-CAT TCC AAC 3' | 250 nmol Maßstab HPLC gereinigte Interne Biotin dT (9) |
| Bot | 5 'ATG GTT GGA TAT ATC GC 3' | 250 nmol Maßstab HPLC-gereinigten | |
| Nicht biotinylierte Halb-Linker (200ng/μl) | Top- | 5 'GGC CGC GGA TCC ATC GAT AAC 3' | 250 nmol Maßstab PCR Grade |
| Bot | 5 'GTT GGA TCC ATC GAT GC 3' | 250 nmol Maßstab PCR Grade | |
| GS20 Adapter A (200ng/μl) | Top- | 5 'CCA TCT CAT CCC TGC GTG TCC CATCTG CCT CCC TTC TGT CTC AGN N 3 ' | 250 nmol Maßstab PCR Grade |
| Bot | 5'CTG AGA CAG GGA GGG AAC TGG AGA GAC ACG CAG GGA TGA GAT GG 3 ' | 250 nmol Maßstab PCR Grade | |
| GS20 Adaptor B (200ng/μl) | Top- | 5 'CTG AGA CAC ACA GCA GGG GAT AGG CAA CAG ACA GGC GGG ATA GG 3' | 250 nmol Maßstab PCR Grade |
| Bot | 5 'CCT ATC CCC GTG TGT CCT TGC CTA TCC CCT GTT TGT CTC GCG AGN N 3' | 250 nmol Maßstab PCR Grade | |
| Illumina-NN-Adaptern | Top- | 5 'ACA CTC TTT ACG ACG CCC TAC CTC TTC CGA TC 3' | 250 nmol Maßstab HPLC gereinigte Siehe Schritt B, 5,4 |
| Bot | 5 'Phosphat - GAT CGG AAG AGC TCA GCA GGT GGA CCG ATG AG 3' | 250 nmol Maßstab HPLC gereinigten phosphorylierten 5'-Ende Siehe Schritt B, 5,4 | |
| Illumina 1-454 (Vorwärts)-Primer (10 pM) | 5 'AAT GCG GAT ACG ACC ACC ATC GAG TAC CTA ACC TCC TGC CTT CCT GTG G 3' | 250 nmol Maßstab PCR Grade | |
| Illumina 2-454 (Reverse)-Primer (10 pM) | 5 'CAA GCA GAA GAC GGC ATA CGA GAT CGG TCC ATC TCA TCC CTG CGT GTC 3' | 250 nmol Maßstab PCR Grade | |
| qPCR Primer 1.1 (10 pM) | 5 'AAT GCG GAT ACG ACC ACC GAG bei 3' | 10nmole Skala PCR Grade | |
| qPCR Primer 2.1 (10 pM) | 5 'CAA GCA GAA GAC GGC ATA CGA 3' | 10nmole Skala PCR Grade | |
| Illumina 3-454 Sequenzierprimers (100 uM) | 5'TGC GTG TCC CAT CTG CCT CCC TTC TGT CTC AG 3 ' | 100nmole Maßstab HPLC-gereinigten | |
| Illumina 4-454 Sequenzierprimers (100 uM) | 5'GTG CCT TGC CTA TCC CCT GTT TGT GCG CTC AG 3 ' | 100nmole SkalaHPLC-gereinigten | |
Tabelle von Oligos und Adapter. Auftrag Oligos von Integrated DNA Technologies (IDT) und bereiten Linker und Adapter in der gleichen Weise wie zuvor beschrieben 10. Half-Linker und Adapter kann vorher hergestellt werden und für mehrere Monate bei -20 ° C gelagert
Referenzen
- Cai, S., Lee, C. C. SATB1 packages densely looped, transcriptionally active chromatin for coordinated expression of cytokine genes. Nat. Genet. 38, 1278-1288 (2006).
- Cook, P. R. The organization of replication and transcription. Science. 284, 1790-1795 (1999).
- Das, P. M., Ramachandran, K. Chromatin immunoprecipitation assay. Biotechniques. 37, 961-969 (2004).
- Deng, W., Blobel, G. A. Do chromatin loops provide epigenetic gene expression states. Curr. Opin. Genet. Dev. 20, 548-554 (2010).
- Fullwood, M. J., Han, Y. Chromatin interaction analysis using paired-end tag sequencing. Curr. Protoc. Mol. Biol. Chapter 21, 21-25 (2010).
- Fullwood, M. J., Liu, M. H. An oestrogen-receptor-alpha-bound human chromatin interactome. Nature. 462, 58-64 (2009).
- Jackson, D. A., Hassan, A. B. Visualization of focal sites of transcription within human nuclei. EMBO J. 12, 1059-1065 (1993).
- Li, G., Fullwood, M. J. ChIA-PET tool for comprehensive chromatin interaction analysis with paired-end tag sequencing. Genome Biol. 11, R22 (2010).
- Li, G., Ruan, X. Extensive Promoter-Centered Chromatin Interactions Provide a Topological Basis for Transcription Regulation. Cell. 148, 84-98 (2012).
- Ng, P., Wei, C. L. Paired-end diTagging for transcriptome and genome analysis. Curr. Protoc. Mol. Biol. Chapter. Chapter 21, 12 (2007).
- Nowak, D. E., Tian, B. Two-step cross-linking method for identification of NF-kappaB gene network by chromatin immunoprecipitation. Biotechniques. 39, 715-725 (2005).
- Osborne, C. S., Chakalova, L. Active genes dynamically colocalize to shared sites of ongoing transcription. Nat. Genet. 36, 1065-1071 (2004).
- Rippe, K., von Hippel, P. H. Action at a distance: DNA-looping and initiation of transcription. Trends Biochem. Sci. 20, 500-506 (1995).
- Schoenfelder, S., Sexton, T. Preferential associations between co-regulated genes reveal a transcriptional interactome in erythroid cells. Nat. Genet. 42, 53-61 (2010).
- Zeng, P. Y., Vakoc, C. R. In vivo dual cross-linking for identification of indirect DNA-associated proteins by chromatin immunoprecipitation. Biotechniques. 41, 694-698 (2006).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten