Method Article
Radioattivo In situ Ibridazione per il rilevamento Diversi schemi di espressione genica nei tessuti
In questo articolo
Riepilogo
Questo protocollo è utilizzato con successo per rilevare quantitativamente i livelli e dei modelli spaziali di espressione di mRNA in diversi tipi di tessuto in tutta specie di vertebrati. Il metodo può rilevare trascrizioni poco abbondanti e permette l'elaborazione di centinaia di diapositive simultaneamente. Vi presentiamo questo protocollo con profilo di espressione di aviaria formazione del cervello dell'embrione come esempio.
Abstract
Conoscendo il tempo, livello, localizzazione cellulare, e tipo di cellula che un gene viene espresso in contribuisce alla nostra comprensione della funzione del gene. Ciascuna di queste caratteristiche può essere realizzato con ibridazione in situ di mRNA all'interno delle cellule. Qui vi presentiamo un metodo radioattivo in ibridazione in situ modificato da Clayton et al. (1988) 1, che opera con successo nel nostro laboratorio da molti anni, soprattutto per adulti cervelli vertebrati 2-5. Le lunghe RNA complementari (cRNA) sonde alla sequenza bersaglio permette di individuare trascritti poco abbondanti 6,7. L'incorporazione di nucleotidi radioattivi nelle sonde cRNA consente di sensibilità di rivelazione ulteriore trascrizioni poco abbondanti e analisi quantitative, sia dalla luce sensibile alla pellicola a raggi X o emulsione rivestita sul tessuto. Questi metodi di rilevamento forniscono un lungo periodo di registrazione di espressione del gene bersaglio. Rispetto ai non-probe radioattivo methods, come DIG-etichettatura, il metodo di ibridazione della sonda radioattiva non richiede fasi di amplificazione multipli utilizzando HRP-anticorpi e / o TSA kit per rilevare trascritti poco abbondanti. Pertanto, questo metodo fornisce una relazione lineare fra l'intensità del segnale e la quantità di mRNA bersaglio per l'analisi quantitativa. Esso consente l'elaborazione 100-200 vetrini simultaneamente. Funziona bene per varie fasi di sviluppo di embrioni. Maggior parte degli studi di sviluppo di espressione genica utilizzare gli embrioni interi e non-radioattivi approcci 8,9, in parte perché il tessuto embrionale è più fragile tessuto adulto, con meno della coesione tra le cellule, il che rende difficile vedere confini tra popolazioni di cellule con sezioni di tessuto. Al contrario, il nostro approccio radioattivo, a causa della più ampia gamma di sensibilità, è in grado di ottenere una maggiore risoluzione di contrasto in genica tra le regioni di tessuto, rendendo più facile vedere confini tra le popolazioni. Usando questo metodo, i ricercatori potrebbero rivelare lapossibile significato di un gene appena identificato, e ulteriormente prevedere la funzione del gene di interesse.
Protocollo
1. Preparazione del tessuto
- Raccogliere i tessuti freschi. Per embrioni di uccelli, aprire con cautela le uova e pulire l'embrione in un piatto con 1xPBS, due volte. Per il cervello adulto, rimuovere rapidamente il cervello e lavare delicatamente con 1 x PBS.
- Incorpora embrioni o cervello adulto in uno stampo incorporato piena di ottobre tessuto tek, orientando il tessuto come necessario per il sezionamento, e rapidamente congelare il blocco ponendolo in una miscela di etanolo e ghiaccio secco, facendo attenzione a non ottenere la miscela all'interno del blocco .
- Tagliare il campione congelato in un criostato in sezioni di 10-12 micron di spessore. Per tessuto cerebrale e embrionale, la migliore temperato taglio è nell'intervallo di -18 ° C a -20 ° C.
- Montare le sezioni congelate di super scivoli più vetro.
- Conservare le sezioni in una casella di scorrimento a -80 ° C.
2. Generazione di ribosonde radioattivi (Utilizzare le procedure di radiazione di sicurezza del vostro istituto)
- Generare un DN purificata lineareUn modello del cDNA di interesse mediante enzima ristretta digest di un frammento clonato circondato da siti promotore polimerasi RNA da un DNA plasmidico o mediante PCR dell'inserto allegato ai siti RNA polimerasi promotore di legame. E 'anche possibile generare frammenti di PCR con i promotori polimerasi RNA come parte del 3' e 5 'primer PCR. Gel purificare i frammenti ristrette o PCR generati con il kit di purificazione GeneClean gel.
- Per una provetta Eppendorf da 0,5 ml, aggiungere 0,5-1 microgrammi di DNA purificato modello lineare, buffer di trascrizione, DTT, RNasin soluzione mix AGC nucleotide, e RNasi acqua libera per portare il volume fino al livello desiderato. Quindi aggiungere S 35-UTP e la RNA polimerasi appropriata per rendere sia ribosonde antisenso o senso.
- Incubare la miscela per 1 ora in un bagno di acqua a 37 ° C. Aggiungi un'altra aliquota di RNA polimerasi e incubare per un altro 1 hr.
- Aggiungere la soluzione di sodio acetato 3M (0,1 volte del volume totale) e 100% EtOH (2,5 FOld del volume totale) nel tubo Eppendorf per precipitare il ribosonda RNA sintetizzato.
- Incubare in ghiaccio secco o -80 ° C per 15 minuti o più di 3 ore per assistere precipitazione.
- RNA pellet mediante centrifugazione a 4 ° C e 15.000 rpm in un tavolo centrifuga Eppendorf per 30 min.
- Rimuovere il surnatante e lavare il pellet con il 70% EtOH, toccare con le dita per mescolare bene il pellet.
- Pellet RNA nuovamente mediante centrifugazione a 4 ° C e 15000 rpm per 30 min.
- Rimuovere il surnatante completamente dell'uso lasciare equilibrare e quindi aggiungere 40 ul di soluzione di ibridazione. Mescolare bene Pipette dentro e fuori.
- Mettere 1 ml della soluzione in 3 ml di sicurezza-Solve soluzione nel flacone scintillazione, mescolare bene e misurare i conteggi del contatore a scintillazione.
- Posizionare il Eppendorf con la ribosonda a -20 ° C per l'uso entro una settimana.
3. Trattamento dei tessuti
- In un cappuccio, preparare PBS fresco tamponata al 4% paraformaldehysoluzione de a circa 3-5 ° C sopra la temperatura ambiente. Collocare i vetrini da -80 ° C in stoccaggio in un rack metallico in ghiaccio secco, portare a spazio di lavoro sotto il cofano, e poi mettere la griglia nella soluzione di paraformaldeide al 4%, e incubare per 5 minuti a temperatura ambiente.
- Lavare 3 volte in 1 x PBS circa 15 tuffi ciascuno (circa 1 secondo per dip).
- Nel cofano, fare buffer di acetilazione e scuoterlo energicamente entro 10 secondi. Versare immediatamente in un cassetto contenente il rack dei vetrini e incubare per 10 min, per ridurre il fondo legame di ribosonda.
- Lavare 3 volte in 2 x SSPE per 15 tuffi.
- Disidratare in 70%, 95% EtOH e seriale 100% per 2 min in ogni passaggio (non deve essere nella cappa).
- Asciugare i vetrini sotto il cofano per almeno 10-15 min.
4. Ibridazione
- Calcolare la quantità di ribosonda (0,5-1x10 6 cpm per slide) e la soluzione di ibridazione (100 pl per vetrino) necessari per tutte le diapositive. Prewarm l'ibridazione ribosonda-mix a 65 ° C per 5 minuti per denaturare il ribosonda.
- Dispensare 100 ul di ribosonda-ibridazione soluzione in una linea attraverso la diapositiva, e utilizzare un vetrino di vetro per diffondere uniformemente il tessuto e coprioggetto. Luogo scorre orizzontalmente, rivolto in alto in un rack in metallo e porta molto lentamente in posizione verticale in bagno d'olio a 65 ° C per un minimo di 4 ore e un massimo di 16 ore. L'olio crea una chiusura ermetica intorno i coprioggetti.
- Rimuovere scaffalature metalliche dal bagno d'olio e pulire l'olio in eccesso intorno rack con carta velina.
- Lavare l'olio dalle diapositive coperti e telaio metallico in vassoi di vetro o metallo contenenti cloroformio, due volte. La 2 cloroformio lavaggio può essere utilizzato come il primo per l'esperimento successivo.
- Trasferire il rack con scivoli coperti in un vassoio con nello 0,1% β-mercaptoetanolo + 2 x soluzione SSPE e spostare su e giù per un paio di tuffi per allentarla coprioggetto e rimuovere sciogliere eccesso di soluzione di ibridazionezione.
- Trasferire il rack con scivoli coperti in soluzione fresca del 0,1% β-mercaptoetanolo + 2 x SSPE, e quindi rimuovere coprioggetto con una pinza RNasi libere in soluzione per evitare di graffiare sezioni di tessuto. Trasferire i vetrini scoperte nel rack fresca in un supporto orizzontale scorrivassoio cremagliera, in una soluzione di 0,1% β-mercaptoetanolo in 2 x SSPE.
- Incubare la rastrelliera con le diapositive in fresco 0,1% β-mercaptoetanolo + 2 x SSPE soluzione a temperatura ambiente per 1 ora per rimuovere l'eccesso non legato RNA sonda. Scartare questa e le precedenti soluzioni acquose di lavaggio, così come coprioggetti, come scorie radioattive.
- Trasferire la cremagliera con scivoli ad una soluzione preriscaldata 2x SSPE a 65 ° C, aggiungere β-mercaptoetanolo ad una finale di 0,1% di concentrazione, e incubare a 65 ° C per 1 ora. Smaltire come rifiuto radioattivo.
- Trasferimento e incubare la rastrelliera con le diapositive due volte a 0,1 x SPPE preriscaldato a 65 ° C per 30 minuti ciascuno. La quantità di radioattività in rimossoquesto passaggio è molto piccolo e non è più considerato eccessivo rifiuti radioattivi dopo questo passo.
- Disidratare il rack e vetrini in 70%, 95% e 100% EtOH per 2 min ciascuna.
- Asciugare i vetrini nella cappa per almeno 30 min.
5. Visualizzazione del segnale radioattivo
- Collocare i vetrini a secco in una cassetta del film e in una stanza buia, posizionare il x-ray film (Kodak BioMax MR film) nel corso degli scivoli, e chiudere il cassetto. Assicurarsi che i vetrini siano di fronte al lato emulsione della pellicola di raggi x. Esporre i vetrini per ~ 1-7 giorni a seconda abbondanza atteso delle trascrizioni.
- Sviluppare la x-ray film in standard e sviluppatore di fissaggio. Il segnale di ibridazione si presenta come nero (esposto granuli di argento nella emulsione) sulla pellicola (Fig. 1).
- (Facoltativo) Per determinare la risoluzione del segnale cellulare e vedere sul tessuto, le diapositive devono essere immersi in emulsione fotografica e di contrasto.Se avete intenzione di di contrasto eventualmente con violetto cresolo, poi delipidize sezioni incubando in xilene per 5 minuti a temperatura ambiente per due volte, reidratare 1 min ciascuna in 100%, 100%, 95%, 95%, 70% e 50% EtOH e poi in acqua deionizzata. Se non si ha intenzione di macchiare con un colorante che non richiede delipidizzazione, allora delipidizzazione non è necessario. Diapositive a secco e sotto una cappa per almeno 2-3 ore.
- Nella stanza buia con una luce sicura, scavare abbastanza NTB emulsione Kodak per coprire la metà della slitta longitudinale (cioè del tessuto) in contenitore di vetro immersione, e poi si fondono in un bagno di acqua 42 ° C per 20-30 min. Poi diluire con acqua distillata per 1:1. Il livello della emulsione dovrebbe coprire tutte le sezioni su un vetrino quando la slitta è immerso in esso. Se hai un sacco di diapositive, potrebbe essere necessario per preparare un'emulsione extra.
- Immergere i vetrini nella emulsione diluita nel 42 ° C bagnomaria a secco e scivoli immersi in un contenitore chiuso a tenuta di luce overnight in camera oscura, o in un forno a 37 ° C per 2-3 ore, con luci spente.
- Trasferire i vetrini in slot rack in scatole nere contenenti essiccatori, facendo attenzione per le diapositive di non toccare l'altro e creare così artefatti. Sigillare i bordi delle scatole con nastro isolante nero lentamente per evitare statica indotte scintille leggeri e quindi avvolgere le scatole in foglio di alluminio. Conservare le cassette a 4 ° C da diversi giorni a settimana (segnale da 1 giorno su x-ray del film è simile a 5 giorni in emulsione).
- Riscaldare le caselle di scorrimento a temperatura ambiente per 1 ora.
- Nella camera oscura, rimuovere la rastrelliera con le diapositive (o porre i vetrini in un rack metallico se si utilizza scatole con asole scorrevoli) dalle scatole e svilupparle nel D-19 Kodak sviluppatore a 16 ° C per 3.5 min.
- Lavare le diapositive sviluppate in acqua di rubinetto a temperatura ambiente per 1 min.
- Incubare i vetrini due volte in fissatore a 19 ° C per 6 minuti ciascuna. Le luci possono essere acceso durante l'incubazione fissatore secondo.
- Lavare i vetrini in acqua corrente a temperatura ambiente per almeno 30 min e raschiare l'emulsione dal lato posteriore della slitta mentre 'wet' con una lama di rasoio per evitare di graffiare il vetro.
- Colorare tessuti con lo 0,3% violetto cresolo in acqua corrente per 5 min.
- Lavare ulteriore soluzione violetto cresolo in acqua potabile fresca per ~ 15 tuffi.
- Disidratare i vetrini per ~ 15 immerge in ogni soluzione alcolica: 50%, 70%, 95%, 95%, 100% e 100% EtOH.
- Incubare i vetrini in xilene per 5 min a temperatura ambiente per due volte.
- Coverslip con il mezzo Permount sul vetrino e asciugare il vetrino coperto il cofano per tutta la notte (> 16 ore), ci vorranno diversi giorni prima che la colla sia abbastanza solido per pulire le diapositive ulteriormente.
6. Generazione di immagini a colori campo oscuro
- Se necessario, ulteriormente emulsione pulita eccesso sul retro dei vetrini (non-tessuto senza coversliped lato) bagnando con acqua e raschiando con una lama di rasoio.
- Sciacquare i vetrini con una soluzione di EtOH 80% e strofinare delicatamente 1-2 volte per sbarazzarsi di detriti e polvere.
- Scatta foto in campo scuro o brightfield illuminazione. Passaggi 6.2 e 6.3 possono essere ripetuto 2-3 volte al fine di ottenere un buone immagini senza particelle di polvere, che sono facilmente visibili in campo scuro sotto un microscopio da dissezione.
7. Risultati rappresentativi
Ci sono due modi principali di visualizzazione nei risultati di ibridazione in situ su sezioni di tessuto ibridati con sonde radioattive S 35: 1) x-ray film che è stato collocato sopra le diapositive o 2) emulsione che è stato rivestito nelle diapositive. Un terzo approccio utilizza uno schermo phosphorimager posta sopra le diapositive, ma non sono stati soddisfatti con la risoluzione di questo approccio. Pellicole radiografiche forniscono un risultato rapido e analisi della condizione generale del ibridazione. Il x-ray film dati rivela anche l'ampia risoluzione anatomica e può essere utilizzato per analys quantitativiè 10. Esempi di immagini a raggi X di film del tardo capi embrioni aviaria ibridati con sonde antisenso per FoxP1 e CoupTF2 espressione genica sono in Figure 1A e B. Entrambi i geni sono molto abbondanti nelle sottodivisioni specifiche del cervello. Una buona qualità x-ray risultato film dovrebbe essere acuta (non sfocata) e hanno un alto rapporto segnale-background ratio. Un'immagine offuscata può essere dovuto al contatto irregolare tra un x-ray film e il vetrino con il tessuto ibridate.
Per le diapositive emulsione anabbaglianti, l'emulsione contiene sali d'argento sensibili alla luce rivestiti in tessuto come apposto ad essere sulla plastica della x-ray film. Durante lo sviluppo, la S 35 esposti al sali d'argento vengono convertiti in grani d'argento metallico, proprio come nel x-ray film. Tuttavia, i depositi d'argento sono direttamente visibili sulle celle che rappresentano espressione genica che può essere osservato e misurata qualitativamente sotto un microscopio. I grani d'argento metallico bloccare la luce diretta attraversoe appaiono come i punti neri sotto vista brightfield. Il colorante di contrasto violetto cresolo appare di colore viola (Fig. 3A, 3C, e la fig. 4). In campo scuro, i granuli di argento riflettono la luce proveniente dal lato e appaiono come punti bianchi (Fig. 1C, 1D, 3B e 3D). In questa situazione, il violetto cresolo macchia appare di colore rosso. In brightfield, il segnale di ibridazione è facile vedere ad alti ingrandimenti a risoluzione cellulare, mentre in campo scuro, inoltre, il segnale di ibridazione possono essere visualizzati in basso ingrandimento su tutto il tessuto. La vista in campo oscuro è l'approccio che comunemente utilizzato per mostrare il complesso pattern di espressione genica. Tuttavia, rispetto al risultato ottenuto dalla pratica radiografie, le diapositive emulsione anabbaglianti richiede più tempo (una o più settimane) ed è più sensibile per ottenere sfondo.
Ci sono quattro fonti comuni di forte background: 1) Background in tutto il x-ray del film è di solito fare perproblemi con lo sviluppatore o il fissaggio, o pellicola parzialmente esposta, 2) Background sui vetrini di solito è a causa di problemi con scivolo di preparazione di lavaggio o di vetro, come silination improprio dei vetrini dalla fonte commerciale o auto-preparata, 3) Emulsione esposizione e sviluppo sfondo, e 4) di base sulla sezione dovuta alla mancanza di post-ibridazione accurate operazioni di lavaggio, troppo bassa di una temperatura di ibridazione, scarsa qualità della soluzione di ibridazione, la contaminazione paraformaldeide in stoviglie causando sonde a permanentemente reticolare la degradazione dei tessuti, ribosonda portando a piccole molecole etichettatura del tessuto non-specifico, DTT inattivo o β-mercaptoetanolo conseguente reticolazione di S 35-sonde a RNA-solfuro di legami al tessuto, e attendere troppo lungo per acetilazione. E 'fondamentale avere le diapositive nella soluzione di acetilazione in pochi secondi di miscelazione della anidride acetica e trietanolammina. Se alcuni minuti passano without aggiunta della soluzione alle diapositive, quindi i gruppi acetile non sarà eliminata in modo efficace e quindi associare a RNA non-specifico. Altri fattori includono ibridazione oltre 20 ore, che può generare troppo forte di un segnale, e goccioline di olio in eccesso sui vetrini, che sequestrare soluzione di ibridazione sulle slitte durante i lavaggi acquosi, con conseguente radioattive macchie sul tessuto e scivola dando segnali di fondo scuro. Se si lavora con molte diapositive (più di 100 sezioni), aggiungere un 3 ° cloroformio lavare o modificare i lavaggi cloroformio, per evitare che particelle di olio in eccesso di rimanere nelle diapositive. Careless trattamento del tessuto, vale a dire il congelamento non abbastanza in fretta (entro 5-10 minuti dopo la dissezione) o lo scongelamento e ri-aumenta anche il congelamento del fondo dovuta alla degradazione del mRNA. Fare attenzione a non confondere sfondo per sovraesposizione.
Per informazioni generali emulsione sulle slitte anabbaglianti è possibile perché è altamente sensibile alla luce e richiede una lunga esposizione al buio. Com problemi di fondo comuni sono troppo alti di temperatura per lo sviluppatore e fissatore. Quando la temperatura è superiore a 19 ° C, in prossimità o più caldo temperatura ambiente, più fondo grano argento si ottiene. L'esposizione a bassi livelli di luce che perde in una camera oscura causerà sfondo emulsione. Non lavando fixer abbastanza a lungo (almeno 30 min in acqua corrente), lascerà fixer che reagisce poi con violetto cresolo per generare un precipitato brunastro tutta l'emulsione. Tuttavia, se i vetrini vengono lavati in acqua più di 90 min dopo la fissazione prima della colorazione violetto cresolo, questo può causare l'emulsione si allenti e le sezioni a macchiarsi male. Se non c'è abbastanza tempo per colorare i vetrini in una finestra di 30-90 min dopo la fissazione e il lavaggio, dopo il lavaggio 30 min, lasciare asciugare il vetrino per tutta la notte e procedere con la colorazione violetto cresolo il giorno successivo. Generalmente, la maggior parte mRNA hanno specifici schemi di espressione genica, mentre il segnale di fondo è più uniforme.
e_content "> tessuto piegato Potrebbe essere fuorviante per i risultati dell'espressione genica su x-ray film, portando ad una regione con più scuro del segnale. Per determinare se il tessuto è ripiegato, esaminare non macchiate di sezioni in campo scuro o violetto cresolo sezioni colorate in campo chiaro. cresile di contrasto viola fornisce un modo migliore per esaminare la condizione dei tessuti.Per una migliore interpretazione della specificità della sonda, sonde di rilevamento di controllo dovrebbe essere applicato in diverse sezioni adiacenti. Sonde più senso non mostrano un segnale, ma alcuni lo fanno, e quando lo fanno, ci accorgiamo che è spesso diverso da quello del segnale antisenso. Noi crediamo che questo potrebbe essere correlato a antisenso sintesi o un altro gene sul filamento antisenso del genoma. Vi presentiamo un esempio Pax6 senso e antisenso sonde (Fig. 2A e B). Il filamento antisenso rivela etichettatura lungo la zona ventricolare del cervelletto prosencefalo, e occhio come atteso (Fig. 2A), ma il senso rivela labeling nello strato di pigmento retina (Fig. 2B).
Per dimensioni delle sonde, usiamo sonde di cDNA qualsiasi nell'intervallo 300-5000 bps. Sonde di lavoro inferiore a 300 bps, ma i segnali sono generalmente più deboli. Non abbiamo provato le sonde più grandi di 5000 bps. È preferibile utilizzare sonde sul tessuto dalla stessa specie, se possibile. In caso contrario, si attraversa-sonde ibridare su sezioni di altre specie e ridurre l'ibridazione e lavare le temperature in 3-5 ° C con incrementi basati su identità di sequenza, se conosciuto. Se non è nota, quindi eseguiamo ibridazione tentativi ed errori e lavare temperature. Se la temperatura si abbassa troppo, la sonda può attraversare cDNA-mRNA ibridarsi con altre sequenze simili in tutto o in specie stessa specie 11. In pratica, si trova che i cDNA pari ~ 95% identico o superiore al bersaglio mRNA nel tessuto, l'ibridazione stringenti e temperatura di lavaggio (65 ° C) condizioni funziona bene. Per le sequenze che sono squillòes di ~ 85 al 94% identiche, l'ibridazione e la temperatura di lavaggio può essere necessario ridurre nella gamma di ~ 50 a 60 ° C.
La ragione per usare un rack metallo nella maggior parte dei passi è l'uso di lavaggi cloroformio e xilene. Entrambe le sostanze organiche sciogliere molti tipi di plastiche. Vetro e alcuni tipi di plastiche sono resistenti a questi organici. Ma il vetro è più facile da rompere, e alcune materie plastiche, che in un primo momento sono resistenti si scioglierà per lunghi periodi di esposizione alle sostanze organiche.

Figura 1. Autoradiografia di immagini in-situ di ibridazione di x-ray film e diapositive emulsione anabbaglianti. (AB) X-ray film immagini di sezioni sagittali tutta la testa del diamante mandarino songbird al giorno embrionale 10, ibridata con ribosonde antisenso a (A) FoxP1 o (B) CoupTF2, assunto con illuminazione brightfield sotto un microscopio da dissezione. Nero, grani esposti in film che mostra mRNA expressione. Scala bar = 500 micron. (CD) Emulsione immerso immagini delle diapositive di sezioni sagittali tutta la testa del diamante mandarino al giorno dopo portello 6, ibridata con ribosonde antisenso a (C) FoxP1 e (D) CoupTF2, assunto con illuminazione campo scuro sotto un microscopio dissezione. Bianco, esposti i grani d'argento in emulsione al di sopra del tessuto mostrando l'espressione di mRNA. Rosso, violetto cresolo macchia. Scala bar = 200 micron. Per tutti immagine, il becco è rostrale a sinistra. Le immagini radiografiche pellicola sono stati esposti per un giorno, diapositive anabbaglianti per 3 giorni. La sonda è FoxP1 178 bps alla parte 1544-1711 bp del mRNA; CoupTF2 è 545 bps alla parte 1-545 bp del mRNA. Come si vede, nel mRNA proencefalo FoxP1 si arricchisce in mesopallium (M), striato (St) e talamo dorsale (DT), mentre CoupTF2 si arricchisce in nidopallium (N), arcopallium (A), e talamo più ventrale . C'è la coerenza di espressione tra i tipi di esposizione (ed età). Con i vetrini anabbaglianti, tuttavia, uno etichettatura risoluzione maggiore è visto, unoconfini e le suddivisioni dei tessuti d sono direttamente identificati. Queste e tutte le altre immagini mostrate nel documento sono da sezioni che utilizzano lo standard a 65 ° C ad alta stringenza di ibridazione.
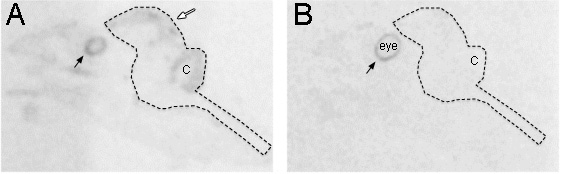
Figura 2. Confronto tra antisenso ed etichettatura senso che mostra diversi modelli. (A) Il filamento antisenso di Pax6 stata espressa nel cervello, in particolare la zona ventricolare (freccia bianca). Mostrato è autoradiografia su x-ray film di fette sagittali intera testa presi da diamante mandarino al giorno embrionale 12. (B) sezione adiacente ibridato con il filamento senso di Pax6 rivela nessuna espressione di fondo in tutta la testa dell'embrione, ma l'espressione evidente nello strato di pigmento della retina (frecce nere) come il filamento antisenso. La linea tratteggiata indica il contorno del tutto il cervello. C: cervelletto.

rong> Figura 3. In situ segnali di espressione genica nelle diapositive emulsione anabbaglianti prelevati dal cervello di diamante mandarino durante le fasi finali embrionali sotto vista chiaro e campo scuro. (A) Brightfield immagine di espressione D1B al giorno embrionale 10 da una esposizione normale alla emulsione. L'etichetta (nero) può essere intravista in questo ingrandimento. Probe è di 625 bps alla parte 1-625 bp del mRNA. (B) Sezione design e l'ingrandimento come in (A), ma passato a campo oscuro che mostra la vista sull'etichetta (bianco) nello striato (St) e il talamo (TH). (C) image Brightfield Slit3 di espressione in 12 giorni embrionale da un'esposizione verso l'emulsione. Label (nero) può essere visto facilmente. Sonda è 779 bps alla parte 1243-2021 del mRNA. (B) Sezione design e l'ingrandimento come in (A) passato a campo oscuro dell'etichetta vista che mostra (bianco) nel midollo spinale (SC), che corrisponde all'immagine campo chiaro. Rostrale è orientata a sinistra. Cb: cervelletto.
/ "Alt =" 3764/3764fig4.jpg Figura 4 "/>
Figura 4. Risoluzione di grano d'argento a livello cellulare. Mostrato è FoxP1 etichetta mRNA nel proencefalo diamante mandarino con grani d'argento (punti neri) al di sopra delle cellule (violetto cresolo) in differenti regioni del cervello ed età rispetto con le immagini di potenza inferiore della figura 1A e 1C. (A) espressione abbondanza Altissimo su singole cellule (frecce nere) negli adulti diamante mandarino mesopallium. (B) espressione bassa abbondanza sulle cellule singole (frecce nere) nel nidopallium adiacente della stessa sezione. (C) High FoxP1 espressione di mRNA in cellule (frecce nere) nel giorno 12 mesopallium embrionale. (D) espressione bassa abbondanza sulle celle (frecce nere) nel nidopallium adiacente della stessa sezione. Cellule esempio sono cerchiati da una linea gialla. Le cellule embrionali (C e D) sono più piccole e più ravvicinate rispetto alle cellule adulte (A e B). Il poiché non vi è spazio tra le cellule e emulsione, l'area di esposta SIgrani LVER dalla sonda 35 S sono leggermente grande rispetto all'area dei corpi cellulari. Scala bar = 10 micron.
Discussione
Radioattivo ibridazione in situ di mRNA è ampiamente usato per molteplici scopi, compresi per lo studio dell'organizzazione regionale tessuto, tipi di cellule, e l'attività cerebrale funzionale 2-5,10,12-14. L'uso successivo è sui geni la cui espressione mRNA nel cervello dipende da una maggiore attività neurale, spesso chiamati attività-dipendente geni o geni precoci immediati. Con questi usi, il nostro metodo è stato applicato tra le specie più, anche in uccelli, mammiferi (ad esempio umano), pesci e anfibi, in diversi tessuti, compreso il cervello, pelle e muscoli, ed età diversi, tra cui hatchlings / neonati, minori , gli adulti, e qui nelle sezioni embrioni interi 2,3,5,15-17. Le caratteristiche speciali del nostro protocollo sono: (1) produce un equilibrio tra specificità anatomica e specificità quantitativa. Per quantificare l'espressione del gene sul x-ray film, scattare foto digitali delle immagini (es:. Fig. 1A e 1B), utilizzare il Photoshop (Adobe) funzione istogramma per misurare la densità di pixel nelle regioni di interesse e sottrarre i livelli di fondo del film al di fuori del tessuto, ma ancora sul vetrino 2,4. Per quantificare l'espressione a livello cellulare, prendiamo immagini di granuli di argento su cellule in alto ingrandimento (40-100X; Fig. 4). Abbiamo quindi utilizzare la soglia e le funzioni di misura J Immagine Wayne Rasband a NIH per contare il numero di granuli di argento nell'immagine, sottrarre il conteggio di fondo in una zona simile senza cellule sul vetrino, dividere per il numero di celle, per ottenere un valore di espressione per cella. 4,18 (2) può essere velocità relativamente elevata, permettendo l'elaborazione di 100-200 diapositive simultaneamente, a causa della tenuta coverslip tenuta creata dal bagno d'olio minerale. Standard in-situ di ibridazione metodi di richiedere più tempo per sigillare i vetrini con parafilm, smalto, ed altri mezzi, dove scorre prendono un sacco di spazio, (3) E 'moltosensibile per trascritti poco abbondanti a causa di destrano solfato e la soluzione di Denhardt nel tampone di ibridazione 2,13; (4) L'approccio di imaging in campo scuro produce immagini ad alto contrasto a causa fotografare sotto illuminazione a campo scuro un microscopio da dissezione 4,5. Inoltre, permette di rivelazione sensibile piccoli cambiamenti nell'espressione genica, come in attività dipendente espressione genica per identificare regioni specifiche del cervello attivate durante la percezione e la produzione di specifici comportamenti 19. Le limitazioni relative non radioattivi sono protocolli che questi ultimi sono più chiare nella risoluzione cellulare e la posizione del mRNA all'interno della cellula, e lavorando con emulsione è molto sensibile alla manipolazione e alla luce. È possibile combinare il nostro metodo con altri metodi, come la non-radioattivo ibridazione in situ per etichettare l'espressione mRNA di più di un gene nel tessuto stesso 10,17,20. Esso può essere combinato con l'immunocitochimicaetichettare sia RNA e proteica sullo stesso campione al fine di localizzare la co-mRNA con certi tipi di cellule 10. Le modifiche necessarie per il protocollo tali esperimenti doppia marcatura sono descritti nei riferimenti citati.
In sintesi, il nostro approccio facilita la comprensione dei tempi e localizzazione cellulare di espressione genica, al fine di comprendere l'organizzazione regione, l'attività funzionale dei tessuti, e la funzione del gene.
Divulgazioni
Non abbiamo nulla da rivelare.
Riconoscimenti
Gli autori desiderano ringraziare tutti i membri di laboratorio Jarvis che hanno migliorato il protocollo nel corso degli anni.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reattivo | Azienda | Numero di catalogo | Comments |
| Anidride acetica | VWR | MK242002 | |
| Cloroformio | VWR | BDH1109 | |
| Viola acetato cresolo | Sigma | C5042 | |
| Criostato | Thermo Scientific | Microm HM550 | |
| Formammide deionizzata | Sigma | F9037 | |
| DTT | Promega | P1171 | 100mM |
| EDTA | Sigma | ED | |
| Incorporare stampo | VWR | 15160-215 | |
| Fisher marchio Superfrost più scorrevole | Fisher Scientific | 22-034-979 | |
| Formaldeide | VWR | BDH0506-4LP | |
| Formammide | Sigma | F7508 | |
| GeneClean kit | Q-Bio gene | 1001-200 | |
| Kodak BioMax MR cinema | Sigma | Z350370 | |
| Kodak NTB Emulsione | Carestream Health | 8895666 | |
| Kodak Professional developer D19 | Kodak | 1462593 | |
| Kodak Professional Fixer | Kodak | 1971746 | |
| β-mercaptoetanolo | Calbiochem | 444203 | |
| Olio minerale | VWR | IC15169491 | |
| NaOH | VWR | SX0600-1 | |
| Paraformaldeide | Sigma | 76240 | |
| Poly A | Invitrogen | POLYA.GF | |
| RATP | Promega | P1132 | 10mM |
| rCTP | Promega | P1142 | 10mM |
| rGTP | Promega | P1152 | 10mM |
| RNasin | Promega | N2111 | 40Units/μl |
| S 35 UTP | PerkinElmer | NEG039C001MC | |
| Safety-Solve soluzione | Sicurezza Risolvere Research Products International | 111177 | |
| Acetato di sodio | Sigma | S7899 | 3M |
| Fosfato di sodio bibasico | Sigma | S3264 | |
| Sodio fosfato monobasico | Sigma | S3139 | |
| SP6RNA polimerasi | Promega | P1085 | |
| Colorazione telaio metallico | Scienze della microscopia elettronica | 70312-54 | |
| T7 RNA polimerasi | Promega | P2075 | |
| Tissue-Tek ottobre | Sakura | 4583 | |
| Trascrizione del buffer 5x | Promega | P1181 | |
| Trietanolammina | VWR | IC15216391 | |
| Tris-HCl (1 M, pH 8,0) | VWR | 101449-446 | |
| tRNA | Roche | 10109509001 |
Soluzioni:
- Reagenti per la produzione di ribosonde: 1,5 microlitri modello DNA (0.2-0.3 mg / pl), 2 microlitri di tampone 5x trascrizione ottimizzato, 1 ml di DTT 100mm (viene fornito con Polymerase da Promega, mixbene a temperatura ambiente), 0,3 pl di RNasin (40 unità / mL), 1,5 pl di soluzione di miscela AGC ribonucleotide, 3,5 pl di S 35 UTP, e 1 pl RNA polimerasi. Portare a 10 ml con acqua priva di nucleasi.
- AGC mix soluzione ribonucleotide: miscelare la stessa quantità di 10mM ATP, GTP e CTP insieme.
- Soluzione di sodio acetato per EtOH precipitazione per rimuovere libera S 35 UTP: 40 microlitri e acqua RNase DNase free, 5 pl di acetato di sodio 3M, e 125 microlitri di EtOH al 100%.
- Ibridazione tampone (10 ml stock): 5 ml del 100% formammide deionizzata, 600 pl di 5M NaCl, 1M Tris-HCl (pH = 8,0), 240 pl di 0,5 M EDTA (pH = 8,0), 100 pl di soluzione di Denhart 100x, 100 pl di 1M DTT, 250 pl di 20mg/ml tRNA, 125 pl di 20 mg / ml di poli A, e 1 g di solfato di sodio destrano. Aggiungere acqua trattata con DEPC per portare il volume totale di 10 ml. Agitare vigorosamente e incubare a 55 ° C fino a quando tutti destrano solfato di sodio viene sciolto. Conservare il tampone di ibridazione in-20 ° C, che è buono per ~ 6 mesi.
- 4% soluzione di paraformaldeide tamponata: Aggiungere 40 g di paraformaldeide in 760 ml di acqua distillata in un pallone designato per paraformaldeide, il calore a 50 ° C su una piastra bollente sotto agitazione. Aggiungere 320 microlitri di NaOH 10N per sciogliere il paraformaldeide. Dopo aver dissolto (~ 10 min), aggiungere 100 ml di PBS 10x, e portare il volume al massimo con acqua distillata fino a 1 litro. Mescolare e riscaldare la soluzione fino a quando paraformaldeide si scioglie. Il pH dovrebbe essere 7.4.
- L'acetilazione tampone: 13,6 ml di trietanolammina più 2,52 ml di anidride acetica in 1 litro di acqua distillata.
- 20x SSPE soluzione: 3M NaCl, 200 mM NaH 2 PO 4-H 2 O, e 200 mM EDTA in acqua distillata. Regolare la soluzione a pH 7,4 con NaOH 10N.
- 10x tampone PBS: 80 g di NaCl, 2,0 g di KCl, 14.4g di Na 2 HPO 4, e 2,4 g di KH 2 PO 4 in acqua distillata. Regolare la soluzione a pH 7,0 e portare il volume totale a 1 liter.
- Soluzione secondo lavaggio: 0,1% β-mercaptoetanolo e 50% formammide a 2x SSPE
- Cresolo acetato soluzione viola: 3% acetato violetto cresolo in acqua di rubinetto (acqua distillata impedisce una buona colorazione), disciolto notte con agitazione in un pallone a temperatura ambiente. Filtrare con aspirazione a vuoto attraverso un filtro di carta Whatman 1 millimetro e imbuto Buchner.
Riferimenti
- Clayton, D. F., Huecas, M. E., Sinclair-Thompson, E. Y., Nastiuk, K. L., Nottebohm, F. Probes for rare mRNAs reveal distributed cell subsets in canary brain. Neuron. 1, 249-261 (1988).
- Wada, K., Sakaguchi, H., Jarvis, E. D., Hagiwara, M. Differential expression of glutamate receptors in avian neural pathways for learned vocalization. J. Comp. Neurol. 476, 44-64 (2004).
- Haesler, S. FoxP2 expression in avian vocal learners and non-learners. J. Neurosci. 24, 3164-3175 (2004).
- Jarvis, E. D., Nottebohm, F. Motor-driven gene expression. Proc. Natl. Acad. Sci. U.S.A. 94, 4097-4102 (1997).
- Holzenberger, M. Selective expression of insulin-like growth factor II in the songbird brain. J. Neurosci. 17, 6974-6987 (1997).
- Mahmood, R., Mason, I. In-situ hybridization of radioactive riboprobes to RNA in tissue sections. Methods Mol. Biol. 461, 675-686 (2008).
- Wilkinson, D. G., Nieto, M. A. Detection of messenger RNA by in situ hybridization to tissue sections and whole mounts. Methods Enzymol. 225, 361-373 (1993).
- Acloque, H., Wilkinson, D. G., Nieto, M. A. In situ hybridization analysis of chick embryos in whole-mount and tissue sections. Methods Cell Biol. 87, 169-185 (2008).
- Moorman, A. F., Houweling, A. C., de Boer, P. A., Christoffels, V. M. Sensitive nonradioactive detection of mRNA in tissue sections: novel application of the whole-mount in situ hybridization protocol. J. Histochem. Cytochem. 49, 1-8 (2001).
- Horita, H., Wada, K., Rivas, M. V., Hara, E., Jarvis, E. D. The dusp1 immediate early gene is regulated by natural stimuli predominantly in sensory input neurons. J. Comp. Neurol. 518, 2873-2901 (2010).
- Braissant, O., Wahli, W. A simplified in situ hybridization protocol using non-radioactively labelled probes to detect abundant and rare mRNAs on tissue sections. Biochemica. 1, 10-16 (1998).
- Kubikova, L., Wada, K., Jarvis, E. D. Dopamine receptors in a songbird brain. J. Comp. Neurol. 518, 741-769 (2010).
- Wada, K. A molecular neuroethological approach for identifying and characterizing a cascade of behaviorally regulated genes. Proc. Natl. Acad. Sci. U.S.A. 103, 15212-15217 (2006).
- Jarvis, E. D. Avian brains and a new understanding of vertebrate brain evolution. Nat. Rev. Neurosci. 6, 151-159 (2005).
- Hoke, K. L., Ryan, M. J., Wilczynski, W. Social cues shift functional connectivity in the hypothalamus. Proc. Natl. Acad. Sci. U.S.A. 102, 10712-10717 (2005).
- Burmeister, S. S., Jarvis, E. D., Fernald, R. D. Rapid behavioral and genomic responses to social opportunity. PLoS. Biol. 3, e363 (2005).
- Jarvis, E. D., Schwabl, H., Ribeiro, S., Mello, C. V. Brain gene regulation by territorial singing behavior in freely ranging songbirds. Neuroreport. 8, 2073-2077 (1997).
- Jarvis, E. D., Scharff, C., Grossman, M. R., Ramos, J. A., Nottebohm, F. For whom the bird sings: context-dependent gene expression. Neuron. 21, 775-788 (1998).
- Feenders, G. Molecular mapping of movement-associated areas in the avian brain: a motor theory for vocal learning origin. PLoS One. 3, e1768 (2008).
- Chen, C. C., Fernald, R. D. Distributions of two gonadotropin-releasing hormone receptor types in a cichlid fish suggest functional specialization. J. Comp. Neurol. 495, 314-323 (2006).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon