Method Article
שיטת צ'יפ (DAP-שבב) מטוהרת-DNA-זיקה לקביעת מטרות גן לחיידקית שתי מערכות רגולטוריות רכיב
In This Article
Summary
מאמר וידאו זה מתאר שיטת microarray מבוסס במבחנה כדי לקבוע את מטרות הגן ואתרי קישור לשני רגולטורים תגובת מערכת רכיב.
Abstract
בשיטות vivo כגון שבב שבב טכניקות מבוססות היטב המשמשות לקביעת מטרות גן הגלובליות לגורמי שעתוק. עם זאת, הם של שימוש מוגבל בחקר שתי מערכות רגולציה מרכיב חיידקים עם תנאי הפעלת uncharacterized. מערכות כאלה להסדיר שעתוק רק כאשר מופעלים בנוכחות אותות ייחודיים. מאז האותות הללו הם לעתים קרובות לא ידועים, חוץ גופית בשיטה microarray מבוססת שבתוארה במאמר זה וידאו יכול לשמש כדי לקבוע מטרות גן ואתרי קישור לרגולטורי תגובה. שיטה מטוהרת-DNA-זיקת שבב זה עשויה לשמש לכל רגולטור מטוהר בכל אורגניזם עם הגנום רצף. הפרוטוקול כרוך מאפשר מטוהר החלבון מתויג להיקשר הדנ"א הגנומי טעון ולאחר מכן טיהור זיקת DNA הנכנס החלבון, ואחריו ניאון תיוג של ה-DNA וכלאה למערך ריצוף מותאם אישית. שקדמו לצעדים שעשויים לשמש כדי לייעל את assay לספציפיהרגולטורים ג גם מתוארים. הפסגות שנוצרו על ידי ניתוח נתוני מערך משמשות כדי לחזות מוטיבים אתר קישור, אשר לאחר מכן הם תוקף באופן ניסיוני. תחזיות המוטיב יכולות לשמש נוספת כדי לקבוע מטרות גן של רגולטורים תגובת orthologous במינים קרובים. אנו מדגימים את תחולתה של שיטה זו על ידי קביעת מטרות הגן ומחייב מוטיבים אתר ובכך לחזות את הפונקציה עבור DVU3023 רגולטור תגובת sigma54 תלוי בחיידק הסביבתי Desulfovibrio vulgaris Hildenborough.
Introduction
היכולת של חיידקים לשרוד ולשגשג תלויה באופן קריטי על כמה טוב הם מסוגלים לקלוט ולהגיב להפרעות בסביבתם, וזה בתורו הוא תלוי במערכות העברת אותותיהם. מספר מערכות אזעקת encodes חיידק כבר נקרא "IQ של חיידקים" שלה ויכול להיות אינדיקציה של שתי השתנות של סביבתו ואת יכולתו לחוש אותות מרובים ולכוונן את תגובתה 1. שתי מערכות רכיב אות התמרה (TCS) הן מערכות האיתות הנפוצות ביותר בשימוש על ידי חיידקים, והם מורכבים מקינאז היסטידין (HK) שחש את האות החיצונית ומעביר באמצעות זירחון לרגולטור תגובת מפעיל (RR) 2. RRs יכול להיות מגוון של תחומים פלט ומצבים שונים ובכך מפעיל, אבל התגובה הנפוצה ביותר היא תקנת תעתיק באמצעות DNA מחייב תחום 1. האותות חשו ופונקציות המקבילה vasלא רוב TCSs אינו ידוע.
למרות in vivo שיטות כגון שבב שבב משמשות באופן שיגרתי לקביעת אתרי קישור הגנומי של שעתוק גורמי 3, הם יכולים לשמש רק לRRs שתי מערכת מרכיב חיידקים אם התנאים להפעלה או אותות ידועים. לעתים קרובות הרמזים הסביבתיים המפעילים TCS הם יותר קשים לקבוע ממטרות הגן שלהם. במבחנה microarray assay המבוסס המתואר כאן ניתן להשתמש ביעילות ובמהירות כדי לקבוע את מטרות הגן ולחזות פונקציות של TCSs. assay זה מנצל את העובדה שניתן phosphorylated RRs וכך הופעל במבחנה באמצעות תורמי מולקולה קטנים כמו פוספט אצטיל 4.
בשיטה זו, בשם DAP-שבבים עבור ה-DNA זיקה מטוהר שבב (איור 1), גן RR של עניין הוא משובט עם התג שלו בE. coli, וטהר לאחר מכן חלבון מתויג מותר דוnd לטעון הדנ"א הגנומי. ה-DNA חייב החלבון מועשר לאחר מכן על ידי זיקה לטיהור, ה-DNA המועשר וקלט הם מוגברות, שכותרתו fluorescently, אספו יחד והכלאה למגוון ריצוף כי הוא עשה מותאם אישית לאורגניזם של עניין (איור 1). ניסויי microarray כפופים לחפצים, ולכן צעדים נוספים מועסקים כדי לייעל את assay. צעד אחד כזה הוא לנסות ולקבוע יעד אחד לRR תחת מחקר באמצעות מבחני משמרת ניידות electrophoretic (EMSA) (ראה זרימת עבודה באיור 2). לאחר מכן, בעקבות מחייב הדנ"א הגנומי וצעדי DAP, ה-DNA חייב החלבון והקלט נבדקים על ידי qPCR כדי לראות אם היעד החיובי הוא מועשר בשבריר מאוגד החלבון ביחס לשבריר הקלט, ובכך אישרה תנאים אופטימליים מחייב עבור RR (איור 2). לאחר הכלאת מערך, הנתונים מנותחים למצוא פסגות של אות בעוצמה גבוהה המעידות על לוקוסים הגנומי שבו שעות החלבוןמודעה קשורה. פונקציות יכולות להיות חזויה עבור RR מבוסס על מטרות הגן שהושגו. הלוקוסים הגנומי היעד משמשים כדי לחזות מוטיבים אתר מחייב, אשר לאחר מכן בניסוי תוקף באמצעות EMSAs (איור 2). התחזיות פונקציונליות ומטרות גן לRR יכולים אז להיות מורחבות למינים קשורות באופן הדוק המקודדים RRs orthologous ידי סריקת הגנום אלה למוטיבים המחייבים דומים (איור 2). שיטת DAP-השבב יכולה לספק שפע של מידע לTCS בהם בעבר לא היה כלום. גם השיטה יכולה לשמש לכל רגולטור תעתיק אם החלבון יכול להיות מטוהרת וניתן לקבוע תנאים המחייבים DNA, ולכל אורגניזם של עניין עם רצף הגנום זמין.

איור 1. מטוהר-DNA-זיקת השבב (DAP-אסטרטגית שבב) 7. גן RR מהאורגניזם של עניין הוא משובט עם התג שלו carboxy-מסוף לE. זן ביטוי coli. חלבון שלו מתויג מטוהר מופעל על ידי זירחון עם פוספט אצטיל, ומעורבב עם הדנ"א הגנומי טעון. Aliquot של התגובה מחייבת נשמר כDNA קלט, ואילו השאר הוא נתון לטיהור זיקה באמצעות שרף Ni-נ.ת. ע. הקלט ואת ה-DNA הנכנס RR הוא הגנום כולו מוגבר, ושכותרתו עם Cy3 וCy5, בהתאמה. ה-DNA שכותרתו היא אספו יחד והכלאה למגוון ריצוף, אשר לאחר מכן נותח על מנת לקבוע מטרות הגן. איור שונה והודפס מחדש באמצעות הרישיון של Creative Commons מ7.

איור 2. סיכום של זרימת עבודה. לכל מטוהר protei מתויגn, להתחיל על ידי קביעת יעד באמצעות EMSA. אפשר לחלבון לקשור הדנ"א הגנומי ולאחר מכן ה-DNA זיקה לטהר (DAP) והגנום כולו להגביר את ה-DNA והקלט המועשר (WGA). אם יעד גן ידוע, השתמש qPCR כדי להבטיח כי היעד הידוע הוא מועשר בשבריר מאוגד החלבון. אם לא ניתן לקבוע יעד, להמשיך ישירות לתיוג ה-DNA וכלת מערך. אם העשרה על ידי qPCR לא יכולה להיות שנצפתה, ולאחר מכן לחזור על החלבון מחייב-gDNA וצעדי DAP-WGA המשתמשים בכמויות חלבון שונות. השתמש בניתוח מערך למצוא פסגות ולמפות אותם כדי למקד את הגנים. השתמש באזורים במעלה הזרם של גנים המטרה לחזות מוטיבים אתר קישור. לאמת את המוטיבים בניסוי באמצעות EMSAs. השתמש במוטיב לסרוק את הגנום של מינים קרובים קידוד orthologs של RR תחת מחקר, ולחזות גנים ממוקדים במינים אלה גם כן. בהתבסס על מטרות הגן השיגו, התפקוד הפיזיולוגי של RR וorthologs ניתן לחזות. איור שונה והודפס מחדש באמצעות ג יצירתייםommons רישיון מ7.
Protocol
הערה: הפרוטוקול להלן מותאם לקביעת מטרות גן של DVU3023 RR מחיידק Desulfovibrio vulgaris Hildenborough. זה יכול להיות מותאם לכל רגולטור תעתיק אחר של עניין.
1. Clone ולטהר RR
- לשבט את גן RR, במיוחד DVU3023, מד vulgaris Hildenborough לתוך וקטור ביטוי חיידקי Escherichia כך שהגן הוא C-סופני מתויג וביטוי הוא תחת השליטה של אמרגן T7.
הערה: שיטות שיבוט כמה עשויות לשמש והוא נקבע על ידי החוקר. עשויים לשמש גם תגי זיקה חלופי. - להפוך את הביטוי לבנות לתוך E. coli BL21 (DE3) מתח ביטוי.
- תתבגר 1 ליטר של זן ביטוי BL21 ב 37 ° C. בשלב אמצע יומן, להוסיף IPTG ל0.5 מ"מ לגרום ביטוי חלבון. המשך צמיחה בטמפרטורת חדר למשך 24 שעות.
- תאי צנטריפוגה ב XG 5,000 ל10 מייליםn ב 4 ° C. תאי resuspend במאגר תמוגה (פוספט נתרן 20 מ"מ, pH 7.4 500 mM NaCl, imidazole 40 מ"מ, 1 מ"ג / מיליליטר ליזוזים, 1x Benzonase nuclease). תאי Lyse באמצעות עיתונות צרפתית ב 4 ° C.
- lysate צנטריפוגה ב 15,000 XG במשך 30 דקות ב 4 ° C. לסנן באמצעות מסנן מזרק 0.45 מיקרומטר.
- שטוף עמודת Ni Sepharose 1 מיליליטר במכשיר FPLC באמצעות 10 מיליליטר של חיץ לשטוף (פוספט נתרן 20 מ"מ, pH 7.4 500 mM NaCl, 40 imidazole מ"מ).
- טען lysate על הטור. לשטוף טור עם 20 מיליליטר של חיץ לשטוף.
- Elute DVU3023 באמצעות שיפוע של 0-100% חיץ elution (פוספט 20 מ"מ נתרן, pH 7.4 500 mM NaCl, 500 מ"מ imidazole).
- טען שברי eluted על עמודת desalting, ולשטוף עם חיץ desalting (pH 20 מ"מ נתרן פוספט 7.4, 100 מ"מ NaCl).
- מדגם חלבון צנטריפוגה במסנן גבוה מולקולרי משקל הפסקת צנטריפוגות כדי לרכז את החלבון. הוספת DTT ל0.1 מ"מ וגליצרול 50% וחלבון חנות ב -20 ° C.
הערה: שיטות טיהור צריכים להיות מותאמות באופן אינדיבידואלי לכל חלבון הנחקר.
2. לקבוע יעד גן לRR באמצעות Shift Electrophoretic ניידות Assay (EMSA)
- PCR להגביר 400 מעלה זרם אזור נ"ב של DVU3025 גן יעד מועמד, באמצעות ד ה-DNA וולגריס Hildenborough הגנומי כתבנית, ויחל ללא תווית קדימה ו5'-שכותרתו ביוטין פריימר ההפוכה.
הערה: טיפים לבחירת גן יעד מועמד: לעתים קרובות RRs לאגד האזורים במעלה הזרם של הגן / אופרון שלהם, או שהם עשויים לווסת את הגנים מקודדים proximally. אם יש RR orthologs במינים אחרים, לחפש גנים, כי הם נשמרים בשכונה. שיטות חלופית כוללות גנים מועמדים בבחירה המבוססים על תחזיות regulon (למשל, RegPrecise 5), או יזמים sigma54 תלוי ניבוי 6 לRRs כי הם עצמם sigma54 תלויים. - הפעל את המוצר ה-PCR על ג'ל agarose 1%, לגזור 400 במוצר בגודל p, ולטהר את ה-DNA באמצעות מיצוי ג'ל לנקות את הערכה.
- מערבבים את חלבון DVU3023 (0.5 pmol) עם מצע של ה-DNA biotinylated 100 fmol ב10 מ"מ טריס HCl, pH 7.5, 50 מ"מ KCl, 5 מ"מ MgCl 2, 1 מ"מ DTT, גליצרול 25% ומיקרוגרם / פולי מיליליטר 1 dI.dC (לא ספציפי DNA מתחרה) בהיקף כולל של 20 μl. גם להגדיר תגובה ללא כל חלבון כביקורת. דגירה התגובות בטמפרטורת חדר על הספסל במשך 20 דקות.
- שים לב: אלה הם תנאים סטנדרטיים המפורטים ומרכיבי תגובה אחרים ניתן להוסיף בהתאם לRR של עניין. אם ההפעלה היא רצויה, להוסיף פוספט אצטיל 50 מ"מ לתגובה (לעתים קרובות RRs יהיה לאגד DNA במבחנה ללא הפעלה).
- טרום להפעיל 6% ג'ל טרומי מיני polyacrylamide-0.5x TBE במאגר TBE 0.5x ל30 דקות 100 V. הוסף 5 μl של חיץ טעינת 5x (bromophenol 0.1% הכחולים, 0.1% cyanol קסילן, גליצרול 30% ב1x TBE) לתגובות מחייבות ו18 μl העומס של התגובות על gאל. לרוץ לשעה 2 100 V.
הערה: על מנת למנוע התחממות יתר, מה שעלול להוביל להתפוררותו של קומפלקסי החלבונים ה-DNA, למלא את תא החיץ החיצוני במערכת אלקטרופורזה עם ריצת חיץ כזה שרוב ג'ל מבודד במאגר. לחלופין, ג'ל ניתן להפעיל ב 4 ° C. - חותכים קרום ניילון מחויב לגודל של ג'ל ולהשרות אותו בTBE 0.5x לפחות עשר דקות. להסיר את הג'ל מהקלטת. לחתוך כל רכסים את הג'ל וכריך ג'ל וקרום בין שני ניירות סינון עבים ספוגים בTBE 0.5x, ומניח בתוך מנגנון סופג חצי יבש ולרוץ ב20 V ל30 דקות.
- מניחים את הקרום בתוך מכשיר crosslinker אור UV מסחרי ולהגדיר את הזמן עד 3 דקות.
הערה: הקרום עשוי כעת להיות מאוחסן יבש בטמפרטורת חדר במשך כמה ימים לפני שתמשיך לשלב הבא. - השתמש בערכת זיהוי chemiluminescent זמינה מסחרי שמעסיקה peroxida צנון streptavidin סוסיםהמצומד se לפתח את הכתם לפי הוראות יצרן.
- תמונת הכתם באמצעות מחשב שמחובר למצלמת CCD המאובזר ולחפש שינוי בניידות מצע DNA בנוכחות RR, אשר מציין כי RR נקשר ה-DNA שנבדק.
הערה: הספציפיות של המשמרת-DNA החלבון, ניתן לבדוק על ידי הכללה בתגובה עודפת של ה-DNA מתחרה ללא תווית, שאמורה למנוע או להקטין את המשמרת ראתה.
3. ודא יעד העשרה לאחר חלבון דנ"א הגנומי העקידה
- תגובה מחייבת חלבון דנ"א גנומי
- הדנ"א הגנומי גזירה לגודל ממוצע של 500 נ"ב ידי sonicating 100 הדנ"א הגנומי μl (בריכוזים 100-200 ng / μl) בצינור 1.5 מיליליטר microfuge באמצעות microtip עם 9 פולסים משרעת נמוכים של 1 שניות, עם 2 שניות בפער בין כל אחד.
הערה: אם ה-DNA משפריץ לצדדים של הצינור, ספין למטה את התוכן לאחר כל 3 פעימות. התנאים המדויקים בשימושישתנה עם מכשיר sonicator בשימוש. שברי DNA טעון עשויים לנוע בין 100-1000 נ"ב, אבל הגודל הממוצע צריך להיות בתוך 400-600 נ"ב. יותר מדי גז עלול לגרום לאתרי קישור בשלמות פחות, וגם יותר מדי או מעט מדי גז ישפיע על הכנת ספרייה במהלך שלבי הגברה הגנום כולו במורד הזרם. - מערבבים 2-3 מיקרוגרם של הדנ"א הגנומי הטעון עם חלבון RR (0.5 pmol של DVU3023) ב10 מ"מ טריס-HCl pH 7.5, 1 מ"מ DTT, 50 מ"מ KCl, 5 מ"מ MgCl 2, גליצרול 25%, ו50 מ"מ פוספט אצטיל. דגירה תגובות ב25 מעלות צלזיוס במשך 30 דקות. ההעברה 10 μl של התגובה הזו לצינור 1.5 מיליליטר ותווית כמו DNA קלט.
הערה: פוספט אצטיל מתווסף כדי להפעיל את החלבון על ידי זירחון במבחנה. זירחון מגרה DNA המחייב, אבל RRs רבים מחייב גם DNA במבחנה ללא הפעלה 7. כמות החלבון שנוספה לתגובה תהיה תלויה בפעילות של הכנת החלבון. EMSA ניתן להשתמש כדי לבחורimize סכום זה.
- הדנ"א הגנומי גזירה לגודל ממוצע של 500 נ"ב ידי sonicating 100 הדנ"א הגנומי μl (בריכוזים 100-200 ng / μl) בצינור 1.5 מיליליטר microfuge באמצעות microtip עם 9 פולסים משרעת נמוכים של 1 שניות, עם 2 שניות בפער בין כל אחד.
- זיקה לטהר את ה-DNA חייב החלבון
- הוסף 30 μl של שרף agarose Ni-נ.ת. ע לצינור 0.6 מיליליטר microfuge. צנטריפוגה בדקות 1 100 XG כדי לאסוף את השרף בתחתית, ולהסיר את supernatant. הוספה של חיץ לשטוף (10 מ"מ טריס-HCl, pH 7.5, 5 מ"מ MgCl 2, 50 מ"מ KCl, גליצרול 25%) 100 μl, קפיצי הצינור לערבב, ו צנטריפוגות ב 2 דקות 100 XG. הסר supernatant.
- הוסף את 90 μl הנותר של התגובה מחייב את שרף Ni-נ.ת. ע שטף ודגירה שבייקרה רוטרי 30 דקות.
- צנטריפוגה ב 2 דקות 100 XG, ולהסיר את supernatant (DNA הכרוך). הוספה של חיץ לשטוף 100 μl, קפיצי הצינור לערבב את תוכן, ו צנטריפוגות ב 2 דקות 100 XG. הסר supernatant. חזור על השלב לשטוף פעמיים נוספות.
- הוסף 35 μl של חיץ elution (pH חיץ פוספט נתרן 20 8 מ"מ, 500 mM NaCl, 500 מ"מ imidazole) לשרף ומערבבים על ידי vortexing. דגירה על הספסל ב RT למשך 5 דקות. צנטריפוגה ב 2 דקות 100 XG. מעביר את supernatant לצינור חדש 1.5 מיליליטר ותווית כחלק-DNA קשור חלבון.
- גם להוסיף 35 μl של חיץ elution ל-DNA הקלט.
- לטהר את הקלט ושברי דנ"א הנכנס חלבון באמצעות ערכת טיהור PCR.
- הגנום כולו להגביר את הקלט ואת דגימות ה-DNA חייב חלבון.
- הוסף 10 μl של הקלט וה-DNA חייב חלבון על מנת להפריד בין צינורות PCR. הוספת μl 1 של חיץ 10x פיצול, 2 μl של חיץ ספריית הכנה, ו1 μl של פתרון ספריית ייצוב מדגם זה. מערבבים היטב על ידי vortexing. חום בthermocycler ב-C ° 95 למשך 2 דקות, ומקרר על קרח.
הערה: הסכום ההתחלתי של ה-DNA צריך להיות לפחות 10 ng כדי למנוע החדרת הטיה הגברה. - הוסף 1 μl של ספריית הכנת אנזימים, לערבב ידי pipetting ו דגירה בCycler תרמית על 16 מעלות C/20 דקות, 24 ° C/20 דקות, 37 מעלות צלזיוס במשך 20 דקות, 75 ° C / 5 דקות, ויחזיק ב 4 ° C.
- על צינור אחד, להוסיף 47.5 μl מים, 7.5 μl של 10x תערובת הורים הגברה, ו5 μl של פולימראז. מערבבים היטב וחום על 95 מעלות דקות C / 3, ואחריו 20 מחזורים של 94 מעלות שניות C/15, 65 ° דקות C / 5. החזק תגובות ב 4 ° C.
- לטהר את דגימות ה-DNA המוגבר באמצעות ערכת טיהור PCR ולמדוד את ריכוז ה-DNA spectrophotometrically.
- הוסף 10 μl של הקלט וה-DNA חייב חלבון על מנת להפריד בין צינורות PCR. הוספת μl 1 של חיץ 10x פיצול, 2 μl של חיץ ספריית הכנה, ו1 μl של פתרון ספריית ייצוב מדגם זה. מערבבים היטב על ידי vortexing. חום בthermocycler ב-C ° 95 למשך 2 דקות, ומקרר על קרח.
- ודא העשרת היעד ב-DNA הנכנס החלבון באמצעות qPCR
- פריימרים qPCR העיצוב כדי להגביר 200 נ"ב באזור במעלה הזרם של גן המטרה לאמת-EMSA (DVU3025) באמצעות כל תוכנת עיצוב פריימר זמינה באופן חופשי.
- הגדרת תגובות qPCR שלושה עותקים עבור כל תבנית ה-DNA בכל קבוצת פריימר. הכן תערובת אב כל קבוצת פריימר. תערובת ההורים 1x מכילה 10 μl תמהיל 2x Sybr הירוק qPCR, 0.5 מיקרומטר של כל צבע יסוד ומים לסך של 18 μl. 18 μl aliquot של מיקס מאסטר לכל טוב של צלחת PCR 96 היטב.
- Dilutדואר מוגבר וקלט מטוהרים ודגימות ה-DNA קשור לחלבון 5 ng / μl עם מים ולהשתמש בו כתבנית ה-DNA. הוסף 2 μl של תבנית ה-DNA לבארות.
- חותם את הצלחת עם סרט איטום qPCR אולטרה ברור, ספין למטה צלחת בצנטריפוגה ב XG 200/1 דקות. מניחים צלחת במכונה qPCR זמן אמת. מחזור כדלקמן באמצעות התוכנה הנלווית qPCR: 95 ° C / 1 דקות, ו40 מחזורים של 95 מעלות C/10 שניות, 59 ° C/15 שניות, ו70 ° C/35 שניות.
- גם להגדיר תגובות qPCR שלושה העתקים עם פריימרים כדי להגביר אזורים במעלה הזרם של גנים שאינם קשורים (בקרה שלילית).
- חישוב ΔC T על ידי הפחתת ערך T C של ה-DNA הנכנס חלבון מזה של ה-DNA הקלט. חישוב העשרת קיפול של גן המטרה בדנ"א הנכנס חלבון כמו 2 ΔC T.
הערה: אם באזור במעלה הזרם היעד הועשר ב-DNA הנכנס לעומת חלבון ה-DNA הקלט, זה מצביע על כך שהתגובות מחייבות וטיהור זיקה היו successful. המשך לשלב 4. אם העשרת האזור במעלה הזרם היעד לא נצפתה, חזור על התגובות מחייבות (שלב 3.1) עם כמות החלבון שונה.
4. סימון ה-DNA וכלת מערך
- ה-DNA קלט תווית עם DNA Cy3 והמועשר עם Cy5
OTE: צבעי Cy3 וCy5 הם רגישים לאור ויש להקפיד לשמור על חשיפה לאור למינימום.- DNA מיקרוגרם מערבבים 1 עם 40 μl Cy3/Cy5-labeled 9 מרס ולהתאים את עוצמת קול ל80 μl עם מים.
- חום לפגל על 98 מעלות צלזיוס למשך 10 דקות בחושך (בthermocycler). צינה מהירה על קרח במשך 2 דקות.
- הוסף 2 פולימראז μl Klenow (3'-5'-Exo -, 50,000 U / ml), 5 dNTPs מ"מ, ו -8 מים μl לכל תגובה, ומערבב היטב, ולדגור על 37 מעלות צלזיוס במשך שעה 2 בחושך (ב thermocycler).
- להוסיף EDTA ל50 מ"מ כדי לעצור את התגובה ופתרון NaCl ל0.5 מ '
- העברת דגימות ל1.5 צינורות מיליליטר המשךaining 0.9 נפח isopropanol, דגירה בחושך במשך 10 דקות, ו צנטריפוגות ב XG 12,000 עבור 10 דקות. גלולה צריכה להיות ורוד עבור ה-DNA שכותרתו Cy3 וכחול עבור ה-DNA שכותרתו Cy5.
- לשטוף עם גלולה 80% אתנול (500 μl) ו צנטריפוגות ב XG 12,000 למשך 2 דקות. כדורי אוויר יבשים ל5-10 דקות בחושך.
הערה: פלטות עשויות להיות מאוחסנות ב -20 ° C. - גלולה גלולה ב25 מים μl. למדוד את ריכוז ה-DNA spectrophotometrically.
- בריכה ביחד 6 מיקרוגרם כל אחת של ה-DNA שכותרתו Cy5 Cy3-ויבש 1.5 צינור מיליליטר וואקום בצנטריפוגה על אש נמוכה בחושך (לכסות את המכסה צנטריפוגות אם הוא שקוף).
הערה: פלטות עשויות להיות מאוחסנות ב -20 ° C עד מוכן להכלאה.
- הכלאה microarray
- הפעל את מערכת ההכלאה על 3-4 שעה לפני שימוש ולהגדיר את הטמפרטורה ל 42 ° C.
- הכן תערובת אב פתרון הכלאה כזאת, כי פתרון 1x מכיל 11.8 μl 2x Hybridiחיץ zation, 4.7 μl הכלאה רכיב, ו0.5 אוליגו יישור μl.
- Resuspend את כדורים במים 5 μl. הוסף 13 μl זה תמהיל למדגם. מערבולת 15 שניות, דגירה על 95 מעלות צלזיוס באמבט יבש למשך 5 דקות. שמור דגימות על 42 מעלות צלזיוס במערכת ההכלאה עד מוכן להעמסה.
- הכן את ההרכבה שקופית מיקסר microarray, על פי הפרוטוקול של היצרן.
- הנח את מכלול מיקסר שקופיות בתוך מערכת ההכלאה. טען 16 μl של מדגם ליציאת המילוי, לאטום את היציאות עם סרט דבק, להפוך ערבוב במערכת, ולהכליא ל16-20 שעות ב42 ° C.
- הכן את המאגרים לשטוף 1x אני (250 מיליליטר), השני (50 מיליליטר) ו-III (50 מיליליטר) על ידי דילול 10x המאגרים הזמינים מסחרי במים, ולכל אחד מוסיף DTT עד 1 מ"מ. הצפת חמה אני 42 ° C.
- חלק את מיקסר השקופית לתוך כלי פירוק, ומקום בתוך צלחת המכילה מאגר חם I. פיל המיקסר את, ואילו במרץ shaking כלי הפירוק ביד.
- מניחים את השקופית לתוך מיכל עם 50 מיליליטר של חיץ לשטוף אני ולנער במרץ למשך 2 דקות ביד.
- העברת שקופיות למכל שני עם חיץ 50 מיליליטר לשטוף השני ולנער במרץ דקות 1 ביד.
- העברת שקופיות לתוך מיכל שלישי עם 50 מיליליטר חיץ לשטוף שלישי, ולנער במרץ ל15 שניות ביד.
- מהר למחוק את הקצוות של השקופית על מגבת נייר, ומניח במדף שקופיות. צנטריפוגה ב XG 200 במשך 2 דקות לייבוש השקף. הנח בתוך מקרה שקופיות, לעטוף אותו בנייר כסף, ולאחסן בתא ייבוש.
- סריקת המערך
- מניחים את השקף בתוך סורק microarray על פי ההוראות של המכשיר.
- השתמש בתוכנת סורק כדי לקבוע את אורכי הגל כ532 ננומטר = Cy3 ו635 ננומטר = Cy5, והרווחים מכפיל הראשוניים בין 350 ו400.
- תצוגה מקדימה של השקופיות כדי לאתר את המערך בשקופית. בחר את אזור שהמערך לסריקה.
- סרוק את המערך ולהתאים את ההגדרות מכפיל כך שתכונות המערך הן בעיקר צהובות. היסטוגרמה צריכה להראות עקומות האדומות וירוקות על גבי זו או קרובה זה לזה ככל האפשר. העקומות צריכה להסתיים מעל 10 -5 הספירה המנורמלת ברוויה.
- להציל את שני 532 ו635 תמונות ננומטר בנפרד.
- ניתוח נתוני מערך
- לייבא את התמונות לתוך תוכנת ניתוח מערך. שימוש בתוכנה, ליצור שני דוחות זוג (. זוג) לכל מערך (לתמונות Cy3 וCy5). יצירת קבצי יחס מדורגים יומן 2 באמצעות קבצי הזוג. השתמש ביומן 2 קבצי יחס לחפש פסגות באמצעות חלון הזזה של 500 נ"ב. מפת לוקוסי השיא לאזורים במעלה הזרם של מטרות גן.
- בדוק אם המטרה החיובית (נקבע באמצעות EMSA ואושר על ידי qPCR, DVU3025 בדוגמא זו) מופיעה בתוך הפסגות העליונה.
הערה: אם המטרה החיובית היא לא בין הלהיטים העליונים, זה אפשרי, כי RR תחת consiיש deration כמה מטרות גן ולשכפל DAP-שבבים יכול להתנהל ופסגות משותפות לכל משכפל יכולות לשמש כדי ליצור רשימת יעד.
5. חיזוי מוטיב אתר הקישור ואימות
- אחזר רצפים לאזורים במעלה הזרם (400 נ"ב) למטרות גן העליונים שכנוצר על ידי ניתוח DAP-שבב. החל MEME על רצפים אלה באמצעות חיידקים מקוונים (meme.nbcr.net) לחזות מוטיבים.
הערה: Enhancer מחייבים חלבונים כגון רגולטורים sigma54 תלויים ניתן לאגד כמה מאה זוגות בסיסים במעלה הזרם של אתר ההתחלה. לרגולטורי תעתיק אחרים, אזור קצר יותר כגון 200 נ"ב במעלה הזרם יהיה מספיק. - עליון עיצוב וoligomers גדיל DNA התחתון המכיל את מוטיב אתר הקישור חזה מוקף 10 בסיסים בשני קצותיו. הזמן את הגדיל העליון 5 'biotinylated.
- מערבבים את oligos הגדיל העליון ותחתון ביחס 1:1.5 ב10 מ"מ טריס HCl, pH 8.0, 1 mM EDTA, ו50 מ"מ NaCl בבסך הכל נפח תגובה של 20 μl בצינורות ה-PCR. חום ל-C ° 95 למשך 5 דקות בCycler תרמית, ואחריו קירור איטי ל25 ° C. לדלל פי עשרה ולהשתמש בו כמצע dsDNA סוג בר לEMSA.
- הכן את מצעים שונה באמצעות אותם השלבים כאמור לעיל, אבל oligomers העיצוב לשאת 4-6 החלפות בבסיסי שימור של המוטיב המחייב.
- הגדרת תגובות מחייבות עם RR והסוג בר או מצעים שונה ולבחון באמצעות EMSA כמתואר בשלב 2.
- הערה: המוטיב ניבא הוא תוקף אם RR נקשר מצע הסוג בר, אבל לא שונה אחד.
6. שימור של מוטיב במיני חיידקים קשורים אחרים
- בחר הגנום של עניין המכיל orthologs של DVU3023. השג לגנומים מאתר צמח השדה קבצי רצף וביאור.
- השתמש בשפת תכנות כמו Perl לכתוב תסריטים שישתמשו רצפי מוטיב ממטרות DAP-השבב לבנות posמטריצה המשוקללת ition ולהשתמש במטריצה להבקיע מוטיבים דומים קיימים בגנומים רצף אחרים.
תוצאות
השיטה הנ"ל יושמה כדי לקבוע את מטרות הגן הגלובליות של RRs בסולפט מודל הפחתת חיידק Desulfovibrio vulgaris 7 Hildenborough. יש אורגניזם זה מספר גדול של TCSs מיוצג על ידי מעל 70 RRs, המעיד על המגוון הרחב של אותות אפשריים שהוא חש ומגיב ל. בvivo מנתח על הפונקציות של מערכות אזעקה אלה הם קשים לביצוע שכן האותות שלהם ובכך התנאים להפעלתם אינם ידועים. הנה שיטת DAP-השבב שימשה כדי לקבוע את מטרות גן ובכך לחזות פונקציות אפשריות עבור DVU3023 RR נציג.
DVU3023 הוא-54 תלויים סיגמא RR מקודד באופרון עם (איור 3 א) HK מאותו המקור שלה. הגן שלו מתויג בטרמינל C-שובט ולמטוהר מן E. coli. לקביעת יעד ראשונית, RR המטוהרים נבדק על ההתקשרות לאופרון במורד הזרם שלה, אשר הוא ג עשרה גני אופרון (DVU3025-3035)onsisting של גנים ספיגה וחמצון חומצת החלב. RR DVU3023 העביר את האזור במעלה הזרם של DVU3025 (איור 3 ב). הבא RR DVU3023 הורשה להיקשר לד הטעון הדנ"א הגנומי vulgaris Hildenborough. למרות זירחון לא נדרש על התקשרות לאזור המקדם של DVU3025, פוספט אצטיל התווסף לתגובה במקרה זה נדרש לכריכה ליזמים אחרים. בעקבות טיהור זיקה של שבריר-DNA קשור חלבון, qPCR שימש כדי להראות שהאזור במעלה הזרם של DVU3025 מועשר (8.45 פי) בשבריר מאוגד החלבון (C ט = 6.9) ביחס ל-DNA הקלט (t C = 9.98 ) (איור 3 ג), ובכך מצביעים על כך שהתנאים המחייבים שימוש היו מתאימים לDVU3023 RR. חוסר ההעשרה של האזור המקדם של גן שנבחר באקראי (DVU0013) שימש כביקורת שלילית.
הדגימות הנכנס חלבונים ו-DNA הקלט אז תויגו fluorescently והכלאהד מערך ריצוף vulgaris Hildenborough שצפיפות בדיקה גבוהה באזורי intergenic. ארבע פסגות העליונים נבחרו כמטרות הסבירה ביותר לDVU3023 (איור 3D). ארבע פסגות אלה ואחרי שזוהו גם בDAP-שבב מנתח במשך כמה RRs אחר ומופיעים ומכאן להיות DNA הדביק (טבלת 1). איור 3E הוא ייצוג סכמטי של גנים מוסדרים על ידי DVU3023, עוד כמה. DVU3025 היעד החיובי היה השיא הראשון שהושג עם הציון הגבוה ביותר. שתי מטרות גן הן שתי permeases אחר מקודד ביחידים חומצת החלב (DVU2451 וDVU3284). יעד הגן הרביעי אינו טמון באזור נגד הזרם, אבל באזור intergenic בין שני גנים / operons עיבד מתכנס (DVU0652 וDVU0653). זהו אזור intergenic גדול, ובנוסף גם מקודדים אמרגן sigma54 תלוי חזה. יתכן כי יש Srna ידועות מקודד באזור זה שהוא regulated ידי RR DVU3023.
שימוש באזורים במעלה הזרם של המטרות שהושגו על ידי DAP-שבב, ממה שימש לחזות מוטיב אתר קישור (איור 3F, טבלת 2). מצעי EMSA נושאים במעלה הזרם מוטיב המסוים של DVU3025 נועדו לאשר כי DVU3023 RR מזהה ונקשר המוטיב חזה. המוטיב קבל תוקף נוסף על ידי ביצוע החלפות בבסיסים נשמרים בתוך המוטיב אשר ביטל את השינוי המחייב (איור 3G). מוטיב תוקף זה שימש לאחר מכן בסקריפטים שנוצרו פרל כדי לסרוק את רצפי הגנום של חיידקי סולפט הפחתה קשורים באופן הדוק אחרים שהיו לי orthologs לDVU3023. לוקוסים נבחרו כמטרות גן אפשריות כאשר המוטיב היה ממוקם באזורים במעלה הזרם של מסגרות פתוחות קריאה (לוח 3). שימוש ברצפי המוטיב חזו לRRs orthologous, מוטיב אתר מחייב קונסנסוס נוצר (איור 3F) שדמה מאוד לone מתקבל עבור ד vulgaris Hildenborough לבד.

איור 3. קביעת מטרות הגנומי לד רגולטור תגובת vulgaris DVU3023. א DVU3023 מקודד באופרון עם HK מאותו המקור שלה. יש אופרון מורד הזרם אמרגן sigma54 תלוי (חץ שחור כפוף) ושימש כגן יעד מועמד. ב מטוהר RR DVU3023 מחויב והעביר את האזור במעלה הזרם של DVU3025 7. ג Q-PCR של אזורים במעלה הזרם של DVU3025 (חיובי היעד) וDVU0013 (שנבחר כביקורת שלילית). E הוא שבריר DNA מועשר בכריכת חלבון, אני הוא DNA קלט. ד למעלה ארבע פסגות המתקבלות לאחר ניתוח DAP-שבב. התחלה וסיום מתייחס ל-DNA קואורדינטות בהתחלה והסיום של PEAKS; ציון מתייחס ליחס יומן 2 R של החללית הרביעית הגבוהה ביותר בשיא; רוזוולט = שיעור גילוי שווא; cutoff_p הוא אחוז הפסקת שבשיא היה מזוהה. ייצוג סכמטי א 'של מטרות הגן לDVU3023 מבוססות על פסגות DAP-השבב. מספרים בתיבות מצביעים על מספר השיא בגנים HK-RR ד נמצא בגנים ירוקים, יעד בכחול ואחרים בגנים אפורים. חיצים מכופפים שחורים הם יזמים sigma54 תלויים, עיגולים מלאים ירוקים הם חזו מוטיבים אתר קישור. שמות ג'ין הם כדלקמן: por - oxidoreductase ferredoxin פירובט; LLP - חומצת החלב permease; מונואמין GLCD-glycolate; glpC-Fe-S אשכול חלבון מחייב; Pta - phosphotransacetylase; קינאז ack-אצטט; lldE - מקטע מונואמין חומצת החלב; lldF / G - מקטע מונואמין חומצת החלב; MCP -. חלבון chemotaxis קבלה-מתיל פ Weblogo 8 תמונות של predicted מוטיב אתר קישור. למעלה - נגזר ממטרות DAP-שבב; תחתון - נבע מאתרי קישור קיימים בגנומים עם orthologs של 7 DVU3023 ג תיקוף מוטיב אתר קישור חזה באמצעות EMSA.. DVU3023 העביר את מוטיב הסוג בר (w) אך לא את המוטיב שונה (מ '). רצפים למוטיבי w מ 'מוצגים בצד הימין 7. איור שונה והודפס מחדש באמצעות הרישיון של Creative Commons מאל Rajeev ואח' 7.

טבלת 1. 20 פסגות למעלה מניתוח מערך DAP-שבב. הטבלה מחולקת לשלושה חלקים. תכונות שיא מראות פרטים של הפסגות שכנוצרו על ידי תוכנת ניתוח מערך, שבו מיקום מתייחס לשאלה האם השיא נמצא בגנום או פלסמיד extrachromosomal, התחלה וסיום מתייחסים ליםטארט ולוקוסי הסוף לפסגה, ציון מתייחס ליחסי יומן 2 R לבדיקה הרביעית הגבוהה ביותר בשיא, רוזוולט מתייחס לערך שיעור גילוי השווא, וcutoff_p הוא אחוז הפסקת שבשיא היה מזוהה. שני סעיפים האחרים התחילו לתאם מיפוי וסיים לתאם מפת מיפוי לוקוסי ההתחלה וסיום, בהתאמה, של השיא לגנים. בקטעים אלה גדיל ג'ין מתייחס לגדיל קידוד הגנטי, אופסט מציין מרחק מההתחלה של הגן (ערכים חיוביים מצביעים על הלוקוסים הוא במעלה הזרם של גן, בעוד שערכים שליליים מצביעים על לוקוסים הוא בתוך הגן), ערך גן החפיפה הוא TRUE אם מוקד חופף גן, DVU מתייחס ל# ^ ש של הגן שקואורדינטות מפה ולתיאור מציין את ביאור הגן. הותאם שולחן והודפס מחדש באמצעות הרישיון של Creative Commons מאל Rajeev ואח' 7. אנא לחץ כאן לצפייה באלגרסת arger של השולחן הזה.

שונה רצפי טבלה 2. משמשים לבניית יעד DAP-שבב הקונצנזוס מבוסס מוטיב באיור 3. שולחן והודפס מחדש באמצעות הרישיון של Creative Commons מאל Rajeev ואח' 7.
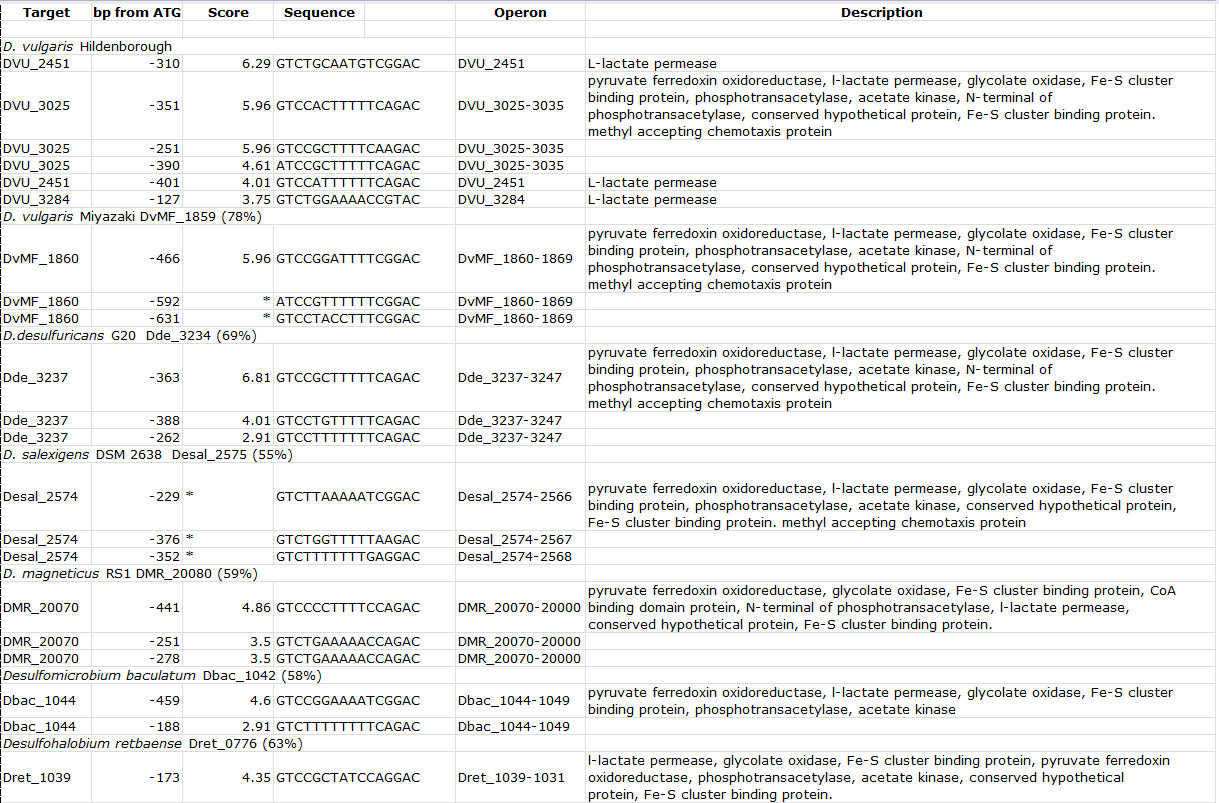
לוח 3. מוטיבים אתר קישור לDVU3023 orthologs ההווה בDesulfovibrio רצף אחר ומינים קרובים. עבור כל הגנום הסרוק, שם האורגניזם מצויינים, ואחריו את תג המוקד לortholog DVU3023 וזהות אחוזים לDVU3023 בסוגריים. ציון מציין את הערך שהוקצה על ידי תכנית פרל על סמך דמיון לsequen הקלטCES. תיאור קובע את ההסברים גנטיים לגנים באופרון היעד. הותאם שולחן והודפס מחדש באמצעות הרישיון של Creative Commons מאל Rajeev ואח' 7. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של השולחן הזה.
Discussion
שיטת DAP-השבב שתוארה כאן הייתה הצלחה בשימוש כדי לקבוע את מטרות הגן במשך כמה RRs בDesulfovibrio vulgaris 7 Hildenborough של איזה מהם מוצג כאן כתוצאה נציג. לDVU3023 RR, בחירת יעד גן מועמד הייתה פשוטה. DVU3025 ממוקם מייד במורד הזרם של גן RR, וגני RR והיעד הם נשמרים בכמה מיני Desulfovibrio, ובנוסף יש DVU3025 אמרגן sigma54 תלוי חזה. EMSA מספק שיטה פשוטה לבדיקת מהירות RR לכריכה לגן יעד המועמד, וגם מאפשר ההערכה של הפעילות של מדגם החלבון מטוהר, כמו גם לקבוע תנאים המחייבים-DNA החלבון אופטימליים.
הפעילות מחייבת DNA יכולה גם להיבדק בנוכחות והיעדרות של פוספט אצטיל כדי לראות אם זירחון הוא הכרחי עבור ה-DNA מחייבת. לא כל RRs הם phosphorylated על ידי תורמי מולקולה קטנים9, במקרים אלה קינאז החיישן מאותו המקור מטוהר, אם הוא זמין, ניתן להשתמש בו כדי להפעיל את החלבון. ישנם גם RRs טיפוסי ידוע כי חוסר שאריות אתר פעילים מרכזיות בתחום המקלט ואינם מופעל על ידי זירחון 10. עבור רוב RRs למד, זירחון מגרה-DNA מחייבת 11. עם זאת יש דוגמאות שבי זירחון אינו משפיע ב-DNA המבחנה מחייב 12, וגם יש דוגמאות שבי זירחון נדרש לכריכת 13, ומקרים שבם קבוצת המשנה של יזמים מחויבים עולה עם זירחון 14. שיטת DAP-השבב יכולה להתבצע עם ובלי זירחון כדי לקבוע אם יש הבדלים בקבוצה של יזמים מחויבים RR. עם זאת, ראוי גם לציין כי חלק RRs ניתן יהיה לטהר בצורה פונקציונלית פוספורילציה מE. coli 13.
הפרוטוקול המתואר כאן הוא לprotei מתויג-n כך כי ה-DNA הנכנס החלבון היא הזיקה מטוהרת באמצעות שרף agarose Ni-נ.ת. ע, אבל זה יכול בקלות להיות מותאם לכל סוג של חלבון מתויג על ידי שימוש בשרף הזיקה המתאים לנפתח. בעקבות צעדי DAP-WGA, צעד qPCR מספק שליטה נוספת על מנת להבטיח כי התנאים מחייבים חלבון gDNA היו מתאימים וכי ה-DNA הנכנס החלבון מועשר בהצלחה על ידי טיהור הזיקה. העשרה גדולה יותר פי 3 היא מספיק כדי להמשיך לשלב ההכלאה. בניגוד לתגובת EMSA, אין DNA מתחרה שאינו ספציפי כמו פולי dI.dC הוסיף לgDNA מחייב תגובה מאחר והוא משבש את צעד WGA. בגלל זה אם מדגם החלבון יש פעילות מחייבת DNA שאינו ספציפית אז העשרה ברורה של ה-DNA היעד אישר לא נצפתה על ידי qPCR. לכן צעד שליטת qPCR ניתן להשתמש כדי לייעל את כמות החלבון המשמשת בתגובה המחייבת gDNA. ערכות זמינות מסחרי עבור ההגברה הגנום כולו טוענות לתנטרנהoduce אין הטיה הגברה כאשר בעקבות ההנחיות שלהם לחומר מינימאלי ההתחלה ה-DNA ומספר נמוך של מחזורי הגברה. ההטיה ההגברה ניתן לבדוק באמצעות qPCR עם ערכות פריימר שונות על מוגבר לעומת DNA לא מוגבר. כל השפעה על הכלאת מערך מורד הזרם יכולה להיות גם שנקבעה על ידי באופן דיפרנציאלי תיוג וההכלאה נקווה מוגברת ולא מוגבר הדנ"א הגנומי.
הרשימה של פסגות שנוצרו בעקבות ניתוח נתוני מערך היא בדרך כלל ארוכה עם כמה פסגות בשווי שיעור גילוי שווא של 0. לכן שילוב של תכונות שיא אחרות כגון גבוה יומן 2 ציוני R וערכי cutoff_p (כפי שנוצר על ידי ניתוח המערך תוכנה המשמשת במחקר זה) משמשות כדי לברור את הרשימה עד למטרות גן האמון הגבוהות ביותר. הנוכחות של יעד הגן נקבע מראש בין חמשת הלהיטים העליונים מאוד מחזקת את האמון בערכת נתונים. ביצוע assay זה עבור מספר החלבונים רגולטוריים לכאן ולכאןמ 'באותו אורגניזם גם יעזור לזהות רצפי DNA המופיעים בכמה נתונים "דביקים" קובע 7. בנוסף, אם מוטיב אתר קישור ניתן לחזות ומאומתים אז הנוכחות של המוטיב בפסגות נמוכה יותר למטה ברשימה עשויה לשמש גם לבחירת רשימת גן יעד שמרנית. העשרה של רצפי DNA אחרים מאשר אזורי אמרגן עשויה להיות חפצים של הכלאת מערך או עשוי להצביע על הסדרת מסגרות קריאה פתוחה מזוהות קודם לכן או RNA הקטן 7. לרגולטורים שבו יעד גן לא יכול להיות קבוע מראש באמצעות EMSA, assay DAP-השבב ניתן לבצע "עיוור" 7. במקרים כאלה גם, זיהוי מוטיב אתר קישור ישפר את הבחירה של מטרות גן. קביעת ריכוז החלבון כדי לשמש assay יהיה תלוי בפעילות מחייבת שאינו הספציפית של החלבון, אשר עשוי להיות מוערך על ידי EMSA באמצעות מצעי DNA שנבחרו באקראי. ריכוזים נמוכים יותר לעבוד טוב יותר עבור tחלבוני צינור עם פעילות ספציפי מחייב גבוה. האמינות של DAP-שבב עיוור ניתן לשפר על ידי ביצוע מבחני לשכפל עם ריכוזי חלבון שונים. לRRs עם כמה מטרות, רק שניים או אפילו assay אחד עשוי להיות מספיק כדי ליצור רשימת יעד שעשוי להיות מאומתת באמצעות EMSAs שלאחר מכן. נתוני DAP-השבב לRRs כאלה בדרך כלל להראות קפיצה בולטת בציוני יומן 2 R או ערכי cutoff_p מעבר לכמה פסגות הראשוניות. לRRs עם כמה מטרות, נתונים משלושה משכפל עשויים להיות מנותחים כדי ליצור רשימה של פסגות משותפות, שחלקם עשוי להיבחר לאימות EMSA.
היכולת לחזות מוטיבים אתר קישור המבוססים על מטרות DAP-השבב מוסיפה לערך של שיטה זו ומגדיל בהרבה את המידע שנצבר. EMSA מספק שוב פרוטוקול יעיל כדי לוודא בניסוי את התחזיות. תסריטי תוכנה בפרל או שפת תכנות אחרת עשויים לשמש כדי לחפש במהירות הדרך seque גנום זמיןnces. התוצאות תהיה לזהות שני גנים orthologous היעד, כמו גם גנים המטרה מוסדרים באופן ייחודי בגנומים חיפשו. בתוצאת הנציג המוצגת כאן, שלוש מתוך ארבעת אזורי היעד במעלה הזרם המזוהים עבור DVU3023 לי מבוארות פונקציות גן הקשור לספיגה וחמצון חומצת החלב. בנוסף מאז מוטיב אתר הקישור לDVU3023 קבל תוקף, גנומים אחרים יכולים להיות חיפשו אתרי מוטיב דומים. יחד תוצאות המחקר מראים כי DVU3022-3023 TCS נשמרת היטב בDesulfovibrio והפחתת חיידקים-סולפט נלווים אחרים, וכי הוא מסדיר גנים להובלת חומצת החלב וחמצון ליצטטו. מאז חומצת החלב הוא מקור פחמן ואלקטרונים העיקרי בשימוש על ידי אורגניזמים אלה, DVU3023 סביר ממלא תפקיד מרכזי בפיסיולוגיה שלהם. בין פסגות DAP-השבב העליונה לRR DVU3023, היה גם אזור intergenic. למרות מוטיב אתר קישור לא נמצא באזור זה, הנוכחות של-54 תלויים סיגמא אמרגן חזה מרמזת tכובע ייתכנו ORF לא מזוהה או Srna מקודד, וכי היא יכולה להיות מטרה אמיתית לDVU3023. ממצא זה מדגיש את הערך של גישת DAP-שבב הניסוי בשילוב עם תחזיות אתר קישור בניגוד לקביעת אתרי יעד המבוססים על תחזיות חישובית לבד.
שיטות דומות במבחנה כמו זה המתואר כאן, היו בשימוש רק בכמה מקרים 15-17 ולעתים רחוקות מאוד לרגולטור לא נחקר בעבר. אופטימיזציה EMSA וqPCR צעדים לפני הכלאת המערך יהיה משמעותי לסייע בניתוח התוצאות כאשר השיטה היא לשמש לרגולטור רומן. אם הדפסת מערך הופכת להיות מכשול, צעדי DAP עשויים להיות משולבים עם רצף הדור הבא כדי לקבל אתרי קישור 18,19. ככל שיותר שיטות המבוסס על מערך להיות מוחלפות עם אסטרטגיות רצף מבוססים, הסתגלות עתיד כזה של השיטה עוקפת את הצורך לעצב microarrays ריצוף המותאם אישית עבור organism של עניין. טכנולוגיות שבב Seq לגרום לרגישות רבה יותר וספציפיות של זיהוי של פסגות בהשוואה לשיטות שבב שבב, וגם הופכות להיות יותר 20 חסכוניים במהירות. למרות שמאמר זה התמקד בשני רגולטורים תגובת רכיב, שיטה זו יכולה לשמש לכל גורם שעתוק פרוקריוטים או אוקריוטים 15. לעתים קרובות יש תפוסות הנוקלאוזום השפעה על מטרות שמוסדרות ומשווה את יעד אתרי קישור לגורמי שעתוק אוקריוטים ידי in vivo ו במבחנה ניתוחים יכול לחשוף את ההשפעות הללו 21.
Disclosures
יש הסופרים שאין ניגוד עניינים לחשוף.
Acknowledgements
אנו מודים איימי חן על עזרתה בהכנה לצילומי הווידאו ולהפגין את הטכניקה. עבודה זו שנערכה על ידי ENIGMA: מערכות אקולוגיות ורשתות משולבות עם גנים והרכבות מולקולריות (http://enigma.lbl.gov), תכנית אזור המוקד מדעי במעבדה הלאומית לורנס ברקלי, נתמכת על ידי משרד מדע, המשרד הביולוגי ו מחקר סביבתי, של משרד אנרגיה של ארה"ב תחת חוזה מס 'DE-AC02-05CH11231.
Materials
| Name | Company | Catalog Number | Comments |
| HisTrapFF column (Ni-Sepharose column) | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5255-01 | |
| Akta explorer (FPLC instrument) | GE Lfe Sciences, Pittsburgh, PA, USA | ||
| HiPrep 26/10 Desalting column | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5087-01 | |
| Qiaquick Gel extraction kit | Qiagen Inc, Valencia, CA, USA | 28704 | |
| Biotin-labeled oligonucleotides | Integrated DNA Technologies | N/A | |
| 6% polyacrylamide-0.5x TBE precast mini DNA retardation gel | Life Technologies, Grand Island, NY, USA | EC63652BOX | Alternately, you can pour your own gel. |
| Nylon membrane | EMD Millipore, Billerica, MA, USA | INYC00010 | |
| Trans-Blot SD Semi-dry electrophoretic transfer cell | Biorad, Hercules, CA, USA | 170-3940 | |
| Extra thick blot paper, 8 x 13.5 cm | Biorad, Hercules, CA, USA | 170-3967 | |
| UV crosslinker Model XL-1000 | Fisher Scientific | 11-992-89 | |
| Nucleic Acid chemiluminescent detection kit (Pierce) | Thermo fisher Scientific, Rockford, IL, USA | 89880 | |
| Ni-NTA agarose resin | Qiagen Inc, Valencia, CA, USA | 30210 | |
| GenomePlex Whole genome amplification kit (Fragmentation buffer, library preparation buffer, library stabilization solution, library preparation enzyme, 10x amplification master mix, WGA polymerase) | Sigma-Aldrich, St. Louis, MO, USA | WGA2-50RXN | |
| Nanodrop ND-1000 | Thermo Scientific, Wilmington, DE, USA | For quantitation of DNA | |
| Perfecta Sybr Green SuperMix, with ROX | Quanta biosciences | 95055-500 | Any Sybr Green PCR mix may be used |
| PlateMax Ultra clear heat sealing film for qPCR | Axygen | ||
| [header] | |||
| 96-well clear low profile PCR microplate | Life Technologies, Grand Island, NY, USA | PCR-96-LP-AB-C | |
| Applied Biosystems StepOne Plus Real time PCR system | Life Technologies, Grand Island, NY, USA | 4376600 | Any real time PCR system may be used |
| Qiaquick PCR purification kit | Qiagen Inc, Valencia, CA, USA | 28104 | Any PCR clean up kit may be used |
| Cy3/Cy5-labeled nonamers | Trilink biotechnologies, San Diego, CA, USA | N46-0001, N46-0002 | |
| Klenow polymerase 50,000 U/ml, 3'-5' exo- | New England Biolabs, Ipswich, MA | M0212M | |
| Hybridization system | Roche-Nimblegen, Madison, WI, USA | N/A | This company no longer makes arrays or related items, so alternate sources such as Agilent or Affymetrix will need to be used. |
| Custom printed microarrays and mixers | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Hybridization kit (2x Hybridization buffer, Hybridization component A, Alignment oligo) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Wash buffer kit (10x Wash buffer I, II, III, 1 M DTT) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| GenePix 4200A microarray scanner | Molecular Devices, Sunnyvale CA, USA | This model has been replaced by superior ones | |
| GenePix Pro microarray software | Molecular Devices, Sunnyvale CA, USA | ||
| Nimblescan v.2.4, ChIP-chip analysis software | Roche-Nimblegen, Madison, WI, USA | N/A | |
References
- Galperin, M. Y. Diversity of structure and function of response regulator output domains. Curr Opin Microbiol. 13, 150-159 (2010).
- Casino, P., Rubio, V., Marina, A. The mechanism of signal transduction by two-component systems. Curr Opin Struct Biol. 20, 763-771 (2010).
- Aparicio, O., Geisberg, J. V., Struhl, K. Chromatin immunoprecipitation for determining the association of proteins with specific genomic sequences in vivo. Curr Protoc Cell Biol. 17 (17), (2004).
- McCleary, W. R., Stock, J. B. Acetyl phosphate and the activation of two-component response regulators. J Biol Chem. 269, 31567-31572 (1994).
- Novichkov, P. S., et al. RegPrecise web services interface: programmatic access to the transcriptional regulatory interactions in bacteria reconstructed by comparative genomics. Nucleic Acids Res. 40, 604-608 (2012).
- Studholme, D. J., Buck, M., Nixon, T. Identification of potential sigma(N)-dependent promoters in bacterial genomes. Microbiology. 146 (12), 3021-3023 (2000).
- Rajeev, L., et al. Systematic mapping of two component response regulators to gene targets in a model sulfate reducing bacterium. Genome Biol. 12, 99(2011).
- Crooks, G. E., Hon, G., Chandonia, J. M., Brenner, S. E. WebLogo: a sequence logo generator. Genome Res. 14, 1188-1190 (2004).
- Barbieri, C. M., Mack, T. R., Robinson, V. L., Miller, M. T., Stock, A. M. Regulation of response regulator autophosphorylation through interdomain contacts. J Biol Chem. 285, 32325-32335 (2010).
- Bourret, R. B. Receiver domain structure and function in response regulator proteins. Curr Opin Microbiol. 13, 142-149 (2010).
- Gao, R., Stock, A. M. Molecular strategies for phosphorylation-mediated regulation of response regulator activity. Curr Opin Microbiol. 13, 160-167 (2010).
- Hung, D. C., et al. Oligomerization of the response regulator ComE from Streptococcus mutans is affected by phosphorylation. J Bacteriol. 194, 1127-1135 (2012).
- Ladds, J. C., et al. The response regulator Spo0A from Bacillus subtilis is efficiently phosphorylated in Escherichia coli. FEMS Microbiol Lett. 223, 153-157 (2003).
- Wen, Y., Feng, J., Scott, D. R., Marcus, E. A., Sachs, G. Involvement of the HP0165-HP0166 two-component system in expression of some acidic-pH-upregulated genes of Helicobacter pylori. J Bacteriol. 188, 1750-1761 (2006).
- Liu, X., Noll, D. M., Lieb, J. D., Clarke, N. D. DIP-chip: rapid and accurate determination of DNA-binding specificity. Genome Res. 15, 421-427 (2005).
- Gossett, A. J., Lieb, J. D. DNA Immunoprecipitation (DIP) for the Determination of DNA-Binding Specificity. CSH Protoc. 2008, (2008).
- Mascher, T., et al. The Streptococcus pneumoniae cia regulon: CiaR target sites and transcription profile analysis. J Bacteriol. 185, 60-70 (2003).
- Park, P. J. ChIP-seq: advantages and challenges of a maturing technology. Nat Rev Genet. 10, 669-680 (2009).
- Lefrancois, P., Zheng, W., Snyder, M. ChIP-Seq using high-throughput DNA sequencing for genome-wide identification of transcription factor binding sites. Methods Enzymol. 470, 77-104 (2010).
- Ho, J. W., et al. ChIP-chip versus ChIP-seq: lessons for experimental design and data analysis. BMC Genomics. 12, 134(2011).
- Liu, X., Lee, C. K., Granek, J. A., Clarke, N. D., Lieb, J. D. Whole-genome comparison of Leu3 binding in vitro and in vivo reveals the importance of nucleosome occupancy in target site selection. Genome Res. 16, 1517-1528 (2006).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved