Method Article
تنقية الحمض النووي تقارب رقاقة (DAP رقاقة) طريقة لتحديد الأهداف الجيني للبكتيريا نظامان التنظيمية المكون
In This Article
Summary
توضح هذه المقالة الفيديو على ميكروأري يستند أسلوب في المختبر لتحديد أهداف الجينات ومواقع الربط لمدة المنظمين استجابة النظام المكون.
Abstract
في الأساليب فيفو مثل رقاقة رقاقة هي تقنيات راسخة المستخدمة لتحديد الأهداف الجين العالمية لعوامل النسخ. ومع ذلك، فإنها ليست ذات فائدة محدودة في استكشاف البكتيرية نظامين التنظيمية المكون مع الظروف تفعيل uncharacterized. هذه النظم تنظيم النسخ فقط عند تفعيلها في وجود إشارات فريدة من نوعها. منذ هذه الإشارات غالبا ما تكون غير معروفة، في المختبر ميكروأري تستند الطريقة الموضحة في هذه المقالة الفيديو يمكن استخدامها لتحديد الأهداف الجينات ومواقع الربط للمنظمين الاستجابة. هذا الأسلوب تنقية الحمض النووي تقارب رقاقة يمكن أن تستخدم لأي منظم تنقيته في أي كائن حي مع تسلسل الجينوم. ينطوي على بروتوكول يسمح البروتين الموسومة تنقيته لربط الحمض النووي الجيني المنفصمة ثم تنقية تقارب الحمض النووي المرتبط بالبروتين، يليه وضع العلامات الفلورية من الحمض النووي والتهجين لمجموعة وتبليط مخصصة. السابقة الخطوات التي يمكن استخدامها لتحسين مقايسة لspecifiكما وصف المنظمين ج. وتستخدم القمم الناتجة عن تحليل البيانات للتنبؤ مجموعة زخارف موقع الملزمة، والتي يتم بعد ذلك التحقق من صحة تجريبيا. التنبؤات عزر يمكن استخدامها لتحديد المزيد من الأهداف الجينات المنظمين استجابة orthologous في الأنواع ذات الصلة عن كثب. ونحن لشرح تطبيق هذه الطريقة من خلال تحديد أهداف ملزمة الجينات والزخارف الموقع وبالتالي توقع وظيفة لمنظم استجابة DVU3023 تعتمد على sigma54 في البكتيريا البيئية منتزعة الكبريت الشائع Hildenborough.
Introduction
قدرة البكتيريا على البقاء على قيد الحياة وتزدهر اعتمادا كبيرا على مدى وهم قادرون على إدراك والرد على الاضطرابات في بيئاتهم، وهذا بدوره يعتمد على أنظمتها نقل الإشارة. وقد دعا عدد من النظم يدل على تشفير بكتيريا في "الذكاء الميكروبي"، ويمكن أن يكون مؤشرا لكلا تنوع بيئتها وقدرتها على الإحساس إشارات متعددة وتهذيب ردها 1. نظامان مكون نقل الإشارة (TCS) هي أنظمة الإشارات الأكثر انتشارا التي تستخدمها البكتيريا، وأنها تتكون من كيناز الحامض الاميني (هونج كونج) تستشعر إشارة خارجية وينقل عبر الفسفرة إلى منظم استجابة المستجيب (RR) 2. يمكن سجلات المورد دينا مجموعة متنوعة من المجالات الانتاج وسائط المستجيب مما مختلفة، لكن الرد الأكثر شيوعا هو التنظيم النسخي عن طريق الحمض النووي نطاق 1 ملزمة. إشارات لمست وظائف المقابلة من خدمات القيمة المضافةر غالبية TCSs لا تزال مجهولة.
على الرغم من أن في الجسم الحي أساليب مثل رقاقة رقاقة تستخدم بشكل روتيني لتحديد مواقع الربط الجيني لعوامل النسخ 3، فإنها يمكن أن تستخدم إلا لاثنين من الممثلين الإقليميين النظام المكون الجرثومي إذا من المعروف أن الظروف تفعيل أو الإشارات. غالبا ما تكون العظة البيئية التي تنشيط TCS هي أصعب لتحديد الأهداف من الجينات الخاصة بهم. في المختبر ميكروأري مقايسة على أساس وصفها هنا يمكن أن تستخدم لتحديد بفعالية وبسرعة الأهداف الجينات والتنبؤ ظائف TCSs. هذا الاختبار يستفيد من حقيقة أن الممثلين الإقليميين يمكن فسفرته وبالتالي تفعيلها في المختبر باستخدام جزيء صغير المانحين مثل الاسيتيل الفوسفات 4.
في هذه الطريقة، واسمه DAP رقاقة الحمض النووي للتقارب منقى رقاقة (الشكل 1)، يتم استنساخ الجين RR من الفائدة مع صاحب العلامة في E. القولونية، ويسمح للبروتين الموسومة تنقيته بعد ذلك إلى ثنائيةالثانية لالمنفصمة الحمض النووي الجيني. ثم يتم إثراء الحمض النووي ملزمة البروتين تقارب تنقية، وتتضخم الحمض النووي المخصب والمدخلات، fluorescently المسمى، المجمعة معا، وتهجين لمجموعة والتبليط الذي هو العرف لكائن من الفائدة (الشكل 1). تجارب ميكروأري تخضع لالتحف، وبالتالي يعملون خطوات إضافية لتحسين الفحص. واحدة من هذه الخطوة هو محاولة لتحديد هدف واحد لRR قيد الدراسة باستخدام فحوصات الكهربي التحول التنقل (EMSA) (انظر سير العمل في الشكل 2). ثم، بعد الملزمة لالحمض النووي الجيني والخطوات مديرية إدارة السجون، ويتم فحص الحمض النووي المرتبط بالبروتين والمدخلات التي كتبها QPCR لمعرفة ما إذا تم تخصيب الهدف الإيجابي في جزء محدد من البروتين بالنسبة للجزء المدخلات، مؤكدة بذلك الشروط الملزمة الأمثل ل RR (الشكل 2). بعد مجموعة التهجين، يتم تحليل البيانات للعثور على قمم أعلى كثافة إشارة تشير إلى مواضع الجيني حيث البروتين حالإعلان ملزمة. يمكن توقع وظائف للRR على أساس أهداف الجينات التي تم الحصول عليها. يتم استخدام مواضع الجيني المستهدف للتنبؤ الزخارف موقع ملزمة، والتي يتم بعد ذلك التحقق من صحة تجريبيا باستخدام EMSAs (الشكل 2). ثم قد يتم تمديد التوقعات الوظيفية والأهداف الجينات لRR إلى الأنواع التي تكود الممثلين الإقليميين orthologous عن طريق مسح الجينوم لتلك الزخارف ملزمة مماثلة (الشكل 2) ترتبط ارتباطا وثيقا. يمكن طريقة DAP رقاقة توفر ثروة من المعلومات عن TCS حيث سبق وكان هناك لا شيء. ويمكن أيضا أن تستخدم أسلوب لأي منظم النسخي إذا كان البروتين يمكن تنقيته ويمكن تحديد شروط ملزمة الحمض النووي، ولأي كائن من الفائدة مع تسلسل الجينوم المتاحة.

الشكل 1. ورقاقة تنقية الحمض النووي تقارب (DAP-رقاقة) استراتيجية 7. يتم استنساخ الجين RR من كائن من الفائدة مع كربوكسي محطة صاحب العلامة إلى E. سلالة التعبير القولونية. يتم تنشيط البروتين تنقية صاحب الموسومة من قبل الفسفرة مع الاسيتيل الفوسفات، ومختلطة مع الحمض النووي الجيني المنفصمة. يتم حفظ قسامة من رد فعل ملزم ال DNA المدخلات، في حين يخضع الباقي لتنقية تقارب باستخدام الراتنج ني NTA. المدخلات والحمض النووي محددة RR هي الجينوم تضخيمها، والمسمى مع CY3 وCy5، على التوالي. يتم تجميع الحمض النووي المسمى معا، وتهجين لمجموعة وتبليط، والتي يتم بعد ذلك تحليلها لتحديد الأهداف الجين. الرقم تعديل وطبعها باستخدام رخصة المشاع الإبداعي من 7.

الشكل 2. ملخص سير العمل. للحصول على أي البروتينات الموسومة تنقيتهن، تبدأ من خلال تحديد الهدف باستخدام EMSA. تسمح البروتين لربط الحمض النووي الجيني ومن ثم الحمض النووي تقارب تنقية (DAP) والجينوم تضخيم (WGA) والحمض النووي والمدخلات المخصب. إذا كان من المعروف هدفا الجين، استخدم QPCR لضمان أن الهدف معروف غير المخصب في جزء محدد من البروتين. إذا لم يتسن تحديد الهدف، الانتقال مباشرة إلى وضع العلامات الحمض النووي ومجموعة التهجين. إذا التخصيب بحلول QPCR لا يمكن ملاحظة، ثم كرر ملزمة البروتين gDNA والخطوات DAP-WGA باستخدام كميات البروتين المختلفة. استخدام تحليل مجموعة للعثور على القمم ورسم خريطة لها لاستهداف الجينات. استخدام مناطق المنبع من الجينات المستهدفة للتنبؤ الزخارف موقع ملزمة. التحقق من صحة الزخارف تجريبيا باستخدام EMSAs. استخدام الحافز لمسح الجينوم من الأنواع ذات الصلة ترميز orthologs من لوائح الراديو قيد الدراسة، والتنبؤ الجينات المستهدفة في تلك الأنواع كذلك. استنادا إلى أهداف الجينات التي تم الحصول عليها، ويمكن توقع وظيفة فسيولوجية من لوائح الراديو وorthologs لها. الرقم تعديل وطبعها باستخدام ج الإبداعيةترخيص ommons من 7.
Protocol
ملاحظة: بروتوكول الواردة أدناه مصممة لتحديد الأهداف الجين من DVU3023 RR من البكتيريا الشائع Hildenborough منتزعة الكبريت. ويمكن أن تتكيف مع أي منظم النسخي الأخرى ذات الاهتمام.
1. استنساخ وتنقية RR
- استنساخ الجين RR، وتحديدا DVU3023، من D. الشائع Hildenborough إلى القولونية التعبير ناقلات من هذا القبيل أن الجين هو C-عضال صاحب الموسومة والتعبير هو تحت سيطرة المروج T7.
ملاحظة: عدة طرق الاستنساخ ويمكن استخدام ويتم تحديدها من قبل الباحث. ويمكن أيضا استخدام علامات تقارب بديل. - تحويل التعبير إلى بناء E. القولونية BL21 (DE3) سلالة التعبير.
- يكبر 1 L من سلالة التعبير BL21 في 37 درجة مئوية. في مرحلة منتصف السجل، إضافة إلى 0.5 ملي IPTG للحث على البروتين التعبير. يستمر النمو في درجة حرارة الغرفة لمدة 24 ساعة.
- خلايا الطرد المركزي في 5000 x ج لمدة 10 ميلن في 4 درجات مئوية. خلايا resuspend في تحلل العازلة (20 ملي فوسفات الصوديوم، ودرجة الحموضة 7.4، 500 مم كلوريد الصوديوم، و 40 ملي إيميدازول، 1 ملغ / مل الليزوزيم، 1x أداة benzonase نوكلياز). ليز خلايا باستخدام الصحافة الفرنسية في 4 درجات مئوية.
- المحللة أجهزة الطرد المركزي في 15،000 x ج لمدة 30 دقيقة في 4 درجات مئوية. تصفية باستخدام 0.45 ميكرون تصفية حقنة.
- غسل 1 مل ني سيفاروز العمود على صك FPLC باستخدام 10 مل من غسل العازلة (20 ملي فوسفات الصوديوم، ودرجة الحموضة 7.4، 500 مم كلوريد الصوديوم، و 40 ملي إيميدازول).
- تحميل المحللة على العمود. غسل العمود مع 20 مل من غسل العازلة.
- أزل DVU3023 باستخدام التدرج من 0-100٪ شطف العازلة (20 ملي فوسفات الصوديوم، ودرجة الحموضة 7.4، 500 مم كلوريد الصوديوم، و 500 ملي إيميدازول).
- تحميل كسور مزال على عمود تحلية، ويغسل مع العازلة تحلية (20 ملي فوسفات الصوديوم ودرجة الحموضة 7.4، 100 مم كلوريد الصوديوم).
- الطرد المركزي عينة البروتين في الوزن الجزيئي قطع تصفية الطرد المركزي عالية لتركيز البروتين. إضافة إلى 0.1 ملي DTT والجلسرين إلى 50٪ والبروتين تخزينها في -20 درجة مئوية.
ملاحظة: ستكون بحاجة الى طرق تنقية ليكون الأمثل فردي لكل بروتين قيد الدراسة.
2. تحديد الجينات المستهدفة لRR طريق التحول الكهربي التنقل الفحص (EMSA)
- PCR تضخيم المنطقة 400 سنة مضت المنبع من DVU3025 مرشح الجينات المستهدفة، وذلك باستخدام D. الشائع Hildenborough الحمض النووي الجيني كقالب، وغير المسماة التمهيدي إلى الأمام وعكس 5'-البيوتين المسمى التمهيدي.
ملاحظة: نصائح لاختيار مرشح الجينات المستهدفة: في كثير من الأحيان الممثلين الإقليميين ربط مناطق المنبع من الجينات الخاصة بهم / الاوبرون، أو أنها قد تنظم الجينات المشفرة قريب. إذا كان لديه RR orthologs في الأنواع الأخرى، والبحث عن الجينات التي يتم حفظها في الحي. وتشمل الطرق البديلة اختيار الجينات المرشحة استنادا إلى التنبؤات regulon (على سبيل المثال، RegPrecise 5)، أو التنبؤ التي تعتمد على sigma54 المروجين 6 لسجلات المورد التي هي نفسها التي تعتمد sigma54. - تشغيل المنتج PCR على 1٪ agarose هلام، وقطع ال 400 بع المنتج الحجم، وتنقية الحمض النووي باستخدام استخراج جل تنظيف عدة.
- خلط البروتين DVU3023 (0.5 بمول) مع 100 fmol من الركيزة الحمض النووي البيروكسيديز في 10 ملي تريس حمض الهيدروكلوريك، ودرجة الحموضة 7.5، 50 ملي بوكل، 5 ملي MgCl 2، 1 ملم DTT، الجلسرين 25٪ و 1 ميكروغرام / مل بولي dI.dC (غير محددة الحمض النووي منافس) في حجم ما مجموعه 20 ميكرولتر. أيضا إعداد رد فعل من دون أي بروتين كمجموعة تحكم. احتضان ردود الفعل في درجة حرارة الغرفة على مقاعد البدلاء لمدة 20 دقيقة.
- ملاحظة: هذه هي الشروط القياسية المدرجة ويمكن إضافة مكونات أخرى اعتمادا على رد فعل لوائح الراديو من الفائدة. إذا كان المطلوب تفعيل، إضافة 50 ملي فوسفات الاسيتيل إلى رد فعل (غالبا الممثلين الإقليميين وربط الحمض النووي في المختبر دون تفعيل).
- قبل تشغيل الجاهزة مصغرة 6٪ بولكرلميد-0.5X هلام TBE في 0.5X العازلة TBE على 100 فولت لمدة 30 دقيقة. إضافة 5 ميكرولتر من العازلة 5X التحميل (0.1٪ برموفينول الزرقاء، 0.1٪ الزيلين cyanol، 30٪ الجلسرين في 1X TBE) إلى ردود فعل ملزمة وتحميل 18 ميكرولتر من ردود الفعل إلى زش. تشغيل في 100 فولت لمدة 2 ساعة.
ملاحظة: من أجل تجنب ارتفاع درجة الحرارة، والتي يمكن أن تؤدي إلى تفارق من المجمعات الحمض النووي البروتين، وملء الغرفة العازلة الخارجي في النظام الكهربائي مع تشغيل العازلة مثل أن غالبية من هلام معزول عن طريق العازلة. بالتناوب، قد يتم تشغيل هلام في 4 درجات مئوية. - قطع غشاء النايلون المحملة على حجم هلام ونقع عليه في 0.5X TBE لمدة عشر دقائق على الأقل. إزالة هلام من الكاسيت. قطع أي التلال قبالة هلام ساندويتش والجل والغشاء بين اثنين من أوراق الترشيح سميكة غارقة في 0.5X TBE، ووضع داخل الجهاز النشاف شبه الجافة وتشغيلها في 20 V لمدة 30 دقيقة.
- وضع غشاء داخل الصك ضوء الأشعة فوق البنفسجية crosslinker التجارية وضبط الوقت إلى 3 دقائق.
ملاحظة: قد الآن الغشاء خزنها الجاف في درجة حرارة الغرفة لعدة أيام قبل الانتقال إلى الخطوة التالية. - استخدام مجموعة الكشف chemiluminescent المتاحة تجاريا التي توظف الفجل peroxida streptavidin الحصانحد ذاتها المترافقة لتطوير وصمة عار وفقا لتعليمات الشركة الصانعة.
- صورة لطخة باستخدام الكمبيوتر التوصيل إلى كاميرا CCD مجهزة وابحث عن تحول في التنقل الركيزة DNA في وجود لوائح الراديو، مما يدل على أن RR يربط الحمض النووي التي يجري اختبارها.
ملاحظة: خصوصية التحول الحمض النووي البروتين يمكن اختبارها من قبل بما في ذلك في رد فعل وجود فائض من الحمض النووي منافس غير المسماة، والتي ينبغي القضاء عليها أو إنقاصها التحول ينظر إليها.
3. تحقق الهدف التخصيب بعد الجينوم الحمض النووي والبروتينات ملزم
- رد فعل ملزمة الجيني DNA البروتين
- الحمض النووي الجيني القص بمتوسط حجم 500 سنة مضت من قبل sonicating 100 ميكرولتر الحمض النووي الجيني (بتركيزات 100-200 نانوغرام / ميكرولتر) في أنبوب 1.5 مل microfuge باستخدام microtip مع 9 البقول السعة المنخفضة من 1 ثانية، مع 2 ثانية الفجوة بين لكل منهما.
ملاحظة: إذا كان يرش الحمض النووي على الجانبين من الأنبوب، وتدور باستمرار محتويات بعد كل 3 البقول. الظروف الدقيقة المستخدمةسوف تختلف مع الصك sonicator المستخدمة. شظايا الحمض النووي المنفصمة قد تتراوح بين 100-1000 سنة مضت، ولكن يجب أن يكون متوسط حجم 400-600 ضمن شركة بريتيش بتروليوم. الكثير من القص قد تؤدي إلى مواقع أقل ملزمة سليمة، وإما أكثر من اللازم أو قص القليل جدا سوف تؤثر على إعداد مكتبة خلال المصب الخطوات الجينوم التضخيم كله. - مزيج 2-3 ميكروغرام من الحمض النووي الجيني المنفصمة مع البروتين RR (0.5 بمول من DVU3023) في 10 ملي تريس، حمض الهيدروكلوريك درجة الحموضة 7.5، 1 ملم DTT، 50 ملي بوكل، 5 ملي MgCl 2، 25٪ الجلسرين، و 50 ملي فوسفات الاسيتيل. احتضان ردود الفعل عند 25 درجة مئوية لمدة 30 دقيقة. نقل 10 ميكرولتر من رد الفعل هذا إلى أنبوب 1.5 مل والتسمية كما DNA المدخلات.
ملاحظة: يتم إضافة الفوسفات الاسيتيل لتنشيط البروتين في المختبر الفسفرة. الفسفرة يحفز الحمض النووي ملزمة، ولكن العديد من الممثلين الإقليميين أيضا ربط الحمض النووي في المختبر دون تفعيل 7. فإن كمية البروتين وأضاف أن رد الفعل تعتمد على نشاط البروتين الإعدادية. وEMSA يمكن استخدامها لاختيارimize هذا المبلغ.
- الحمض النووي الجيني القص بمتوسط حجم 500 سنة مضت من قبل sonicating 100 ميكرولتر الحمض النووي الجيني (بتركيزات 100-200 نانوغرام / ميكرولتر) في أنبوب 1.5 مل microfuge باستخدام microtip مع 9 البقول السعة المنخفضة من 1 ثانية، مع 2 ثانية الفجوة بين لكل منهما.
- تقارب تنقية الحمض النووي المرتبط بالبروتين
- إضافة 30 ميكرولتر من ني NTA الراتنج الاغاروز إلى أنبوب 0.6 مل microfuge. أجهزة الطرد المركزي في 100 x ج لمدة 1 دقيقة لجمع الراتنج في الجزء السفلي، وإزالة طاف. إضافة 100 ميكرولتر من غسل العازلة (10 ملي تريس، حمض الهيدروكلوريك، ودرجة الحموضة 7.5، 5 ملي MgCl 2، 50 ملي بوكل، 25٪ الجلسرين)، ونفض الغبار الأنبوب إلى المزيج، وأجهزة الطرد المركزي في 100 x ج لمدة 2 دقيقة. إزالة طاف.
- إضافة 90 ميكرولتر المتبقية من رد فعل ملزم لغسلها راتنج ني NTA واحتضان في شاكر دوارة لمدة 30 دقيقة.
- أجهزة الطرد المركزي في 100 x ج لمدة 2 دقيقة، وإزالة طاف (DNA غير منضم). إضافة 100 ميكرولتر من غسل العازلة، ونفض الغبار أنبوب لخلط محتويات، وأجهزة الطرد المركزي في 100 x ج لمدة 2 دقيقة. إزالة طاف. كرر الخطوة غسل مرتين أكثر من ذلك.
- إضافة 35 ميكرولتر من شطف العازلة (20 ملي العازلة الفوسفات الصوديوم الهيدروجيني 8، 500 مم كلوريد الصوديوم، و 500 ملي إيميدازول) إلى الراتنج وتخلط من قبل vortexing. احتضان على مقاعد البدلاء في RT لمدة 5 دقائق. أجهزة الطرد المركزي في 100 x ج لمدة 2 دقيقة. نقل طاف لأنبوب جديد 1.5 مل والتسمية باسم الحمض النووي جزء محدد من البروتين.
- أيضا إضافة 35 ميكرولتر من شطف العازلة على الحمض النووي المدخلات.
- تنقية المدخلات والكسور الحمض النووي المرتبط بالبروتين استخدام عدة تنقية PCR.
- الجينوم تضخيم المدخلات وعينات من الحمض النووي المرتبط بالبروتين.
- إضافة 10 ميكرولتر من المدخلات ومحددة بروتين الحمض النووي لفصل أنابيب PCR. إضافة 1 ميكرولتر من 10X العازلة تجزئة، 2 ميكرولتر من العازلة إعداد المكتبة، و 1 ميكرولتر من المكتبة لتحقيق الاستقرار حل لكل عينة. مزيج جيد من قبل vortexing ل. الحرارة في thermocycler في 95 درجة مئوية لمدة 2 دقيقة، والبرد على الجليد.
ملاحظة: يجب أن يكون مقدار بدءا من الحمض النووي لا يقل عن 10 نانوغرام من أجل تجنب إدخال تحيز التضخيم. - إضافة 1 ميكرولتر من مكتبة تحضير الانزيم، ومزيج من قبل pipetting، واحتضان في cycler الحرارية في 16 ° C/20 دقيقة، 24 ° C/20 دقيقة، 37 درجة مئوية لمدة 20 دقيقة، 75 ° C / 5 دقيقة، وعقد في 4 درجات مئوية.
- إلى كل أنبوب، إضافة 47.5 ميكرولتر من المياه، 7.5 ميكرولتر من 10X التضخيم مزيج الرئيسي، و 5 ميكرولتر من البلمرة. تخلط جيدا والحرارة في 95 درجة مئوية / 3 دقائق، تليها 20 دورات من 94 درجة C/15 ثانية، 65 ° C / 5 دقائق. عقد ردود الفعل في 4 درجات مئوية.
- تنقية عينات من الحمض النووي تضخيمها باستخدام طقم تنقية PCR وقياس تركيز الحمض النووي طيفيا.
- إضافة 10 ميكرولتر من المدخلات ومحددة بروتين الحمض النووي لفصل أنابيب PCR. إضافة 1 ميكرولتر من 10X العازلة تجزئة، 2 ميكرولتر من العازلة إعداد المكتبة، و 1 ميكرولتر من المكتبة لتحقيق الاستقرار حل لكل عينة. مزيج جيد من قبل vortexing ل. الحرارة في thermocycler في 95 درجة مئوية لمدة 2 دقيقة، والبرد على الجليد.
- تحقق تخصيب الهدف في الحمض النووي ملزمة البروتين باستخدام QPCR
- الاشعال تصميم QPCR لتضخيم 200 نقطة أساس في المنطقة المنبع من الجين المستهدف (DVU3025) التحقق EMSA باستخدام أي التمهيدي تصميم البرمجيات المتاحة مجانا.
- إعداد ردود الفعل QPCR ثلاث نسخ لكل قالب الحمض النووي مع كل مجموعة التمهيدي. إعداد مزيج الرئيسي لكل مجموعة التمهيدي. 1X مزيج الرئيسي يحتوي على 10 ميكرولتر من 2X SYBR الأخضر QPCR المزيج، 0.5 ميكرومتر لكل التمهيدي والمياه إلى ما مجموعه 18 ميكرولتر. قسامة 18 ميكرولتر من مزيج الرئيسي لكل بئر من لوحة PCR 96 جيدا.
- مخففه وتضخيم وتنقية مدخلات وعينات من الحمض النووي المرتبط بالبروتين إلى 5 نانوغرام / ميكرولتر مع المياه واستخدامها كقالب الحمض النووي. إضافة 2 ميكرولتر من الحمض النووي القالب إلى الآبار.
- ختم لوحة واضحة جدا مع QPCR ختم الفيلم، وتدور أسفل لوحة في أجهزة الطرد المركزي في 200 x ج / 1 دقيقة. وضع لوحة في الوقت الحقيقي QPCR الجهاز. دورة على النحو التالي باستخدام البرمجيات المرتبطة QPCR: 95 ° C / 1 دقيقة، و 40 دورات من 95 درجة C/10 ثانية، 59 ° C/15 ثانية، و 70 درجة C/35 ثانية.
- أيضا إعداد ردود الفعل QPCR ثلاث نسخ مع الاشعال لتضخيم مناطق المنبع من الجين لا علاقة لها (مراقبة سلبية).
- حساب ΔC T عن طريق طرح قيمة T C من الحمض النووي المرتبط بالبروتين عن ذلك من الحمض النووي المدخلات. حساب تخصيب أضعاف من الجينات المستهدفة في الحمض النووي المرتبط بالبروتين ك 2 ΔC T.
ملاحظة: إذا تم التخصيب المنطقة المنبع الهدف في الحمض النووي المرتبط بالبروتين مقابل الحمض النووي المدخلات، فإنه يشير إلى أن ردود الفعل ملزمة وتنقية تقارب كانت التالىessful. انتقل إلى الخطوة 4. إذا لم يكن لوحظ إثراء المنطقة المنبع الهدف، كرر ردود فعل ملزمة (الخطوة 3.1) مع كمية مختلفة من البروتين.
4. صفها الحمض النووي والتهجين صفيف
- تسمية الحمض النووي DNA الإدخال مع CY3 والمخصب مع Cy5
OTE: CY3 والأصباغ Cy5 خفيفة حساسة وينبغي توخي الحذر للحفاظ على التعرض للضوء إلى أدنى حد ممكن.- مزيج 1 ميكروغرام الحمض النووي مع 40 ميكرولتر Cy3/Cy5-labeled 9-السائدة وضبط مستوى الصوت إلى 80 ميكرولتر مع الماء.
- الحرارة تفسد عند 98 درجة مئوية لمدة 10 دقيقة في الظلام (في thermocycler). البرد السريع على الجليد لمدة 2 دقيقة.
- إضافة 2 ميكرولتر Klenow البلمرة (3'-5'-إكسو -، 50،000 U / مل)، و 5 ملي dNTPs، و 8 ميكرولتر المياه إلى كل رد فعل، وتخلط جيدا، واحتضان عند 37 درجة مئوية لمدة 2 ساعة في الظلام (في وthermocycler).
- إضافة إلى 50 ملي EDTA لوقف رد الفعل وحل كلوريد الصوديوم إلى 0.5 م.
- عينات نقل إلى 1.5 مل أنابيب تابعaining 0.9 حجم الأيزوبروبانول، واحتضان في الظلام لمدة 10 دقيقة، وأجهزة الطرد المركزي في 12،000 x ج لمدة 10 دقيقة. يجب أن يكون بيليه وردي لDNA CY3 المسمى DNA والأزرق للCy5 المسمى.
- غسل بيليه مع 80٪ من الإيثانول (500 ميكرولتر) وأجهزة الطرد المركزي في 12،000 x ج لمدة 2 دقيقة. الكريات الهواء الجاف لمدة 5-10 دقيقة في الظلام.
ملاحظة: الكريات يمكن تخزينها في -20 درجة مئوية. - بيليه resuspend في 25 ميكرولتر المياه. قياس تركيز الحمض النووي طيفيا.
- تجمع معا 6 ميكروغرام كل من الحمض النووي CY3 وCy5 المسمى في أنبوب 1.5 مل والفراغ الجاف في جهاز للطرد المركزي على نار هادئة في الظلام (تغطية غطاء الطرد المركزي إذا كان شفافا).
ملاحظة: الكريات يمكن تخزينها في -20 درجة مئوية حتى جاهزة للتهجين.
- ميكروأري التهجين
- تحويل نظام التهجين في 3-4 ساعة قبل استخدامها وضبط درجة الحرارة إلى 42 درجة مئوية.
- إعداد التهجين الحل مزيج الرئيسي بحيث 1X الحل يحتوي 11.8 ميكرولتر 2X Hybridiاالنترنت العازلة، 4.7 ميكرولتر التهجين مكون A، و 0.5 ميكرولتر المحاذاة بنسبة ضئيلة.
- resuspend والكريات في 5 ميكرولتر المياه. إضافة 13 ميكرولتر من هذا الخليط للعينة. دوامة لمدة 15 ثانية، واحتضان عند 95 درجة مئوية في حمام جافة لمدة 5 دقائق. الحفاظ على العينات عند 42 درجة مئوية في نظام التهجين حتى جاهزة للتحميل.
- إعداد ميكروأري التجمع الشريحة خلاط، وفقا لبروتوكول الشركة المصنعة.
- وضع التجمع خلاط الشريحة داخل منظومة التهجين. تحميل 16 ميكرولتر من العينة إلى ميناء التعبئة، وختم الموانئ مع الفيلم لاصقة، بدوره على الخلط في النظام، وهجن ل 16-20 ساعة عند 42 درجة مئوية.
- إعداد مخازن يغسل 1X الأول (250 مل)، والثاني (50 مل) والثالث (50 مل) من خلال تمييع 10X مخازن المتاحة تجاريا في الماء، وإلى كل DTT إضافة إلى 1 ملم. العازلة الدافئة أنا إلى 42 درجة مئوية.
- حرك خلاط الانزلاق إلى أداة التفكيك، ومكان داخل صحن يحتوي العازلة الدافئة أولا قشر خلاط حالا، في حين ركلات الترجيح بقوةتخاذ أداة التفكيك باليد.
- ضع الشريحة في وعاء مع 50 مل من غسل العازلة أنا ويهز بقوة لمدة 2 دقيقة باليد.
- نقل الشرائح في وعاء الثانية مع 50 مل غسل عازلة الثاني ويهز بقوة لمدة 1 دقيقة باليد.
- نقل الشرائح في وعاء ثالث مع 50 مل غسل عازلة الثالث، ويهز بقوة لمدة 15 ثانية باليد.
- وصمة عار بسرعة حواف الشريحة على منشفة ورقية، ووضع في رفوف الشريحة. أجهزة الطرد المركزي في 200 x ج لمدة 2 دقيقة حتى يجف الشريحة. وضع ضمن حالة الانزلاق، ألفه مع احباط، وتخزينها في مجفف.
- مسح صفيف
- وضع الشريحة داخل الماسح الضوئي ميكروأري وفقا لتعليمات الصك.
- استخدام برنامج الماسح الضوئي لضبط موجات 532 نانومتر كما = 635 نانومتر CY3 و= Cy5، والمكاسب مضخم الأولي بين 350 و 400.
- معاينة الشريحة لتحديد موقع مجموعة على الشريحة. تحديد المنطقة مجموعة للمسح الضوئي.
- مسح مجموعة وضبط إعدادات مضخم بحيث ميزات مجموعة هي في معظمها الأصفر. الرسم البياني يجب أن تظهر منحنيات الأحمر والأخضر فرضه أو أقرب إلى بعضها البعض قدر الإمكان. يجب منحنيات إنهاء فوق 10 -5 التهم تطبيع في التشبع.
- حفظ كل 532 و 635 نانومتر الصور بشكل منفصل.
- مجموعة تحليل البيانات
- استيراد الصور إلى برنامج تحليل مجموعة. باستخدام البرمجيات، وإنشاء تقريرين الزوج (. الزوج) لكل مجموعة (لCY3 وCy5 الصور). إنشاء سجل تحجيم 2 ملفات نسبة استخدام الملفات الزوج. استخدام تسجيل 2 ملفات نسبة للبحث عن قمم باستخدام نافذة انزلاق 500 سنة مضت. خريطة ذروة مواضع إلى مناطق المنبع من الأهداف الجينات.
- تحقق من ظهور الهدف الإيجابي (تحديد باستخدام EMSA وأكدته QPCR، DVU3025 في هذا المثال) ضمن أفضل القمم.
ملاحظة: إذا كان الهدف إيجابيا ليس بين أكبر الفعالية، فمن الممكن أن RR تحت consiالتخفيض المقنن لديه عدة أهداف الجينات وتكرار DAP-رقائق يمكن أن تجرى وقمم مشتركة بين جميع مكررات يمكن استخدامها لإنشاء قائمة الهدف.
5. ربط الحافز تنبؤ الموقع والتحقق من صحة
- استرداد تسلسل للمناطق المنبع (400 بي بي) للأهداف الجينات أعلى كما تم إنشاؤها بواسطة التحليل DAP رقاقة. MEME على تطبيق هذه التسلسلات من خلال الميكروبات عبر الإنترنت (meme.nbcr.net) للتنبؤ الزخارف.
ملاحظة: يمكن محسن البروتينات ملزمة مثل المنظمين sigma54 التي تعتمد على ربط عدة مئات من أزواج قاعدة المنبع من موقع البداية. لالمنظمين النسخي الأخرى، وهي منطقة أقصر مثل 200 سنة مضت المنبع سوف يكون كافيا. - أعلى وأسفل تصميم الأوليغومرات الحمض النووي حبلا التي تحتوي على توقع عزر موقع ملزم يحيط بها 10 قواعد على حد سواء. تأمر أعلى حبلا 5 'المعقدة البيروكسيديز.
- خلط oligos حبلا أعلى وأسفل في نسبة 1:1.5 في 10 ملي تريس حمض الهيدروكلوريك، ودرجة الحموضة 8.0، 1 ملم EDTA، و 50 ملي مول كلوريد الصوديوم فيوحجم رد الفعل مجموعه 20 ميكرولتر في أنابيب PCR. الحرارة إلى 95 درجة مئوية لمدة 5 دقائق في cycler الحرارية، تليها التبريد البطيء إلى 25 درجة مئوية. تمييع عشرة أضعاف واستخدام ذلك النوع البري dsDNA والركيزة لEMSA.
- إعداد ركائز تعديلها باستخدام نفس الخطوات المذكورة أعلاه، ولكن الأوليغومرات تصميم لتنفيذ 4-6 بدائل في قواعد الحفظ من عزر ملزمة.
- إعداد ردود الفعل ملزم مع لوائح الراديو والنوع البري أو تعديل ركائز ودراسة باستخدام EMSA كما هو موضح في الخطوة 2.
- ملاحظة: يتم التحقق من صحة عزر توقع إذا كان RR يربط نوع الركيزة البرية ولكن ليست واحدة تعديلها.
6. حفظ الحافز في ذات الأنواع البكتيرية الأخرى
- حدد العوامل الوراثية ذات الأهمية التي تحتوي على orthologs من DVU3023. الحصول على ملفات تسلسل والشرح عن الجينوم من موقع NCBI.
- استخدام لغة برمجة مثل بيرل لكتابة البرامج النصية التي سوف تستخدم تسلسل عزر من الأهداف DAP رقاقة لبناء نقاط البيعition مصفوفة المرجح واستخدام مصفوفة ليسجل زخارف مماثلة موجودة في الجينوم التسلسل الأخرى.
النتائج
تم تطبيق الأسلوب أعلاه لتحديد الأهداف الجين العالمية من الممثلين الإقليميين في الحد من كبريتات نموذج بكتيريا منتزعة الكبريت الشائع Hildenborough 7. هذا الكائن لديها عدد كبير من TCSs يمثلها أكثر من 70 سجلات المورد، مما يدل على تشكيلة واسعة من إشارات الممكن أنه يستشعر ويستجيب ل. في الجسم الحي يحلل على وظائف هذه النظم مما يشير يصعب أداء منذ إشاراتها وبالتالي تفعيل الظروف الخاصة غير معروفة. هنا تم استخدام أسلوب DAP رقاقة لتحديد أهداف الجينات، وبالتالي التنبؤ الوظائف المحتملة لRR DVU3023 ممثل.
DVU3023 هو المعتمد على 54 سيغما RR المشفرة في الاوبرون مع هونج كونج وما شابه ذلك (الشكل 3A) لها. تم استنساخ الجين صاحب الموسومة C-محطة في وتنقيته من E. القولونية. للتقرير الأولي الهدف، تم اختبار RR تنقية للربط إلى الاوبرون المصب والذي هو بمثابة عشرة الجينات الاوبرون (DVU3025-3035) جonsisting اللاكتات في امتصاص والأكسدة الجينات. RR DVU3023 تحولت المنطقة المنبع من DVU3025 (الشكل 3B). سمح المقبل RR DVU3023 لربط المنفصمة D. الشائع Hildenborough الحمض النووي الجيني. على الرغم من أن الفسفرة لم يكن مطلوبا للربط إلى منطقة المروج من DVU3025، تم إضافة الاسيتيل الفوسفات إلى رد فعل في حال كان مطلوبا منها للربط إلى المروجين الأخرى. التالية تقارب تنقية الحمض النووي جزء محدد من البروتين، وكان يستخدم QPCR لاظهار ان المنطقة المنبع من DVU3025 غير المخصب (8.45 أضعاف) في جزء محدد بروتين (C T = 6.9) نسبة إلى الحمض النووي المدخلات (C = 9.98 طن ) (الشكل 3C)، مما يدل على أن الشروط الملزمة المستخدمة كانت مناسبة للRR DVU3023. وقد استخدم نقص إثراء منطقة المروج من الجينات اختيارها عشوائيا (DVU0013) كعنصر تحكم السلبية.
كانت عينات محددة من البروتين والحمض النووي المدخلات ثم fluorescently المسمى والمهجنة لوD. الشائع Hildenborough مجموعة تبليط التي كان لها كثافة عالية التحقيق في المناطق بين الجينات. وقد تم اختيار أعلى أربعة قمم مثل الأهداف الأكثر احتمالا لDVU3023 (الشكل 3D). وتلت هذه القمم الأربعة من قبل العديد من الآخرين، والتي تم تحديدها أيضا في DAP-رقاقة تحليلات لعدة سجلات المورد أخرى، وبالتالي يبدو أن الحمض النووي لزجة (الجدول 1). الشكل 3E هو تمثيل التخطيطي للجينات التي تنظمها DVU3023. كان الهدف DVU3025 إيجابية الذروة الأولى التي تم الحصول عليها وفقا لأعلى نقاط. أهداف الجينات اثنين هما permeases اللاكتات المشفرة منفردة أخرى (DVU2451 وDVU3284). لا يكمن الهدف الرابع الجين في منطقة المنبع، ولكن في المنطقة بين الجينات بين اثنين كتب convergently الجينات / operons (DVU0652 وDVU0653). هذه هي منطقة كبيرة بين الجينات، وبالإضافة إلى ذلك بترميز أيضا تعتمد على sigma54 المروج توقع. فمن الممكن أن هناك الحمض الريبي النووي الذواب المشفرة غير المكتشفة في هذه المنطقة التي هي reguذا الصلة من قبل RR DVU3023.
باستخدام مناطق المنبع من الأهداف التي حصلت عليها مديرية إدارة السجون رقاقة، وكان يستخدم للتنبؤ MEME موقع عزر ملزمة (الشكل 3F، الجدول 2). وقد صممت ركائز EMSA تحمل المنبع عزر محددة من DVU3025 للتأكد من أن DVU3023 RR تعترف ويربط عزر توقع. تم التحقق من صحة عزر مزيد بجعل بدائل في قواعد الحفظ داخل عزر الذي قضى على التحول ملزم (الشكل 3G). ثم تم استخدام هذا الحافز التحقق من صحتها في البرامج النصية ولدت بيرل لمسح متواليات جينوم أخرى ترتبط ارتباطا وثيقا كبريتات الحد من البكتيريا التي كان orthologs لDVU3023. وقد تم اختيار مواضع الجينات كأهداف ممكنة عندما كانت تقع عزر في مناطق المنبع من إطارات القراءة المفتوحة (الجدول 3). باستخدام تسلسل عزر توقع لسجلات المورد orthologous، وقد ولدت فكرة موقع ملزم الآراء (الشكل 3F) والتي تشبه إلى حد كبير سشمال شرق الحصول عليها لD. الشائع Hildenborough وحدها.

الشكل 3. تحديد الأهداف الجيني لD. منظم استجابة الشائع DVU3023. A. يتم ترميز DVU3023 في الاوبرون مع هونج كونج في وما شابه ذلك. والاوبرون المصب لديه المروج التي تعتمد على sigma54 (السهم الأسود عازمة)، وكان يستخدم بمثابة الجين المستهدف المرشح. B. تنقية RR DVU3023 ملزمة وتحولت المنطقة المنبع من DVU3025 7. C. Q-PCR من مناطق المنبع من DVU3025 (إيجابية استهداف) وDVU0013 (اختار كعنصر تحكم السلبية). E هو محدد البروتين المخصب جزء الحمض النووي، والحمض النووي هو الإدخال. D. أعلى قمم الأربعة التي تم الحصول عليها بعد التحليل DAP رقاقة. بدء وإنهاء الرجوع إلى الحمض النووي ينسق في بداية ونهاية PEاس؛ تشير النتيجة إلى نسبة R سجل 2 من رابع أعلى التحقيق في الذروة؛ روزفلت = معدل اكتشاف كاذبة؛ cutoff_p هو النسبة المئوية في قطع التي تم تحديدها الذروة. E. تمثيل تخطيطي للأهداف الجينات لDVU3023 استنادا إلى قمم DAP رقاقة. أرقام في صناديق تشير إلى عدد الذروة في D. الجينات HK-RR هي باللون الأخضر، الجينات المستهدفة في الجينات الزرقاء وغيرها في الرمادي. السهام السوداء هي عازمة المروجين sigma54 التي تعتمد، ومن المتوقع شغلها الدوائر الخضراء الزخارف موقع ملزمة. أسماء الجينات هي كما يلي: البرتغال - البيروفات فيريدوكسين مؤكسدة مختزلة؛ محاماة - لاكتات بيرمياز؛ أوكسيديز GLCD-غليكولات؛ glpC الحديد-S الكتلة بروتين ملزمة؛ منطقة التجارة التفضيلية - ناقلة أسيتيل الفسفات؛ كيناز ACK-خلات؛ lldE - اللاكتات أوكسيديز الوحيدات؛ lldF / G - لاكتات أوكسيديز الوحيدات؛ MCP - قبول-الميثيل البروتين الكيميائي F. Weblogo 8 صور من PREDicted موقع عزر ملزمة. الأعلى - المستمدة من أهداف DAP رقاقة؛ أسفل - المستمدة من مواقع الربط موجودة في الجينوم مع orthologs من DVU3023 7 G. التحقق من تنبأ موقع عزر ملزمة باستخدام EMSA. DVU3023 تحول نوع عزر البرية (ث) ولكن ليس عزر تعديل (م). يتم عرض تسلسل للزخارف وث م على الحق 7. الرقم تعديل وطبعها باستخدام رخصة المشاع الإبداعي من راجيف وآخرون 7.

الجدول 1. أعلى 20 قمم من تحليل مجموعة DAP رقاقة. ينقسم الجدول إلى ثلاثة أقسام. تظهر سمات ذروة تفاصيل القمم كما تم إنشاؤها بواسطة برامج تحليل مجموعة، حيث يشير إلى ما إذا كان الموقع تم العثور على الذروة في الجينوم أو البلازميد خارج الصبغي، بداية ونهاية الرجوع إلى لياليلاذع ونهاية مواضع للذروة، ويشير إلى نقاط نسب R 2 سجل للرابع أعلى التحقيق في الذروة، وفرانكلين روزفلت يشير إلى قيمة معدل اكتشاف كاذبة، وcutoff_p هو النسبة المئوية في قطع التي تم تحديدها الذروة. بدء تشغيل أخرى قسمين تنسيق رسم الخرائط ووضع حد للتنسيق ورسم الخرائط خريطة بداية ونهاية مواضع، على التوالي، من الذروة إلى الجينات. في هذه المقاطع يشير إلى جين حبلا حبلا ترميز الجين، أوفست يشير إلى المسافة من بداية الجين (القيم الإيجابية تدل على مواضع هو المنبع من الجين، بينما تشير القيم السلبية مواضع ضمن الجينات)، وقيمة التداخل الجينات هي TRUE إذا كانت موضع يتداخل جين، حدة مكافحة العنف العائلي يشير إلى حدة مكافحة العنف العائلي # من الجين الذي إحداثيات الخريطة ل، والوصف يدل على الشرح الجينات. جدول تعديل وأعيد طبعه باستخدام رخصة المشاع الإبداعي من راجيف وآخرون 7. الرجاء انقر هنا لعرض آلنسخة arger من هذا الجدول.

الجدول 2. متواليات تستخدم لبناء الهدف DAP رقاقة إجماع عالمي قائم عزر في الشكل 3. الجدول تعديل وطبعها باستخدام رخصة المشاع الإبداعي من راجيف وآخرون 7.
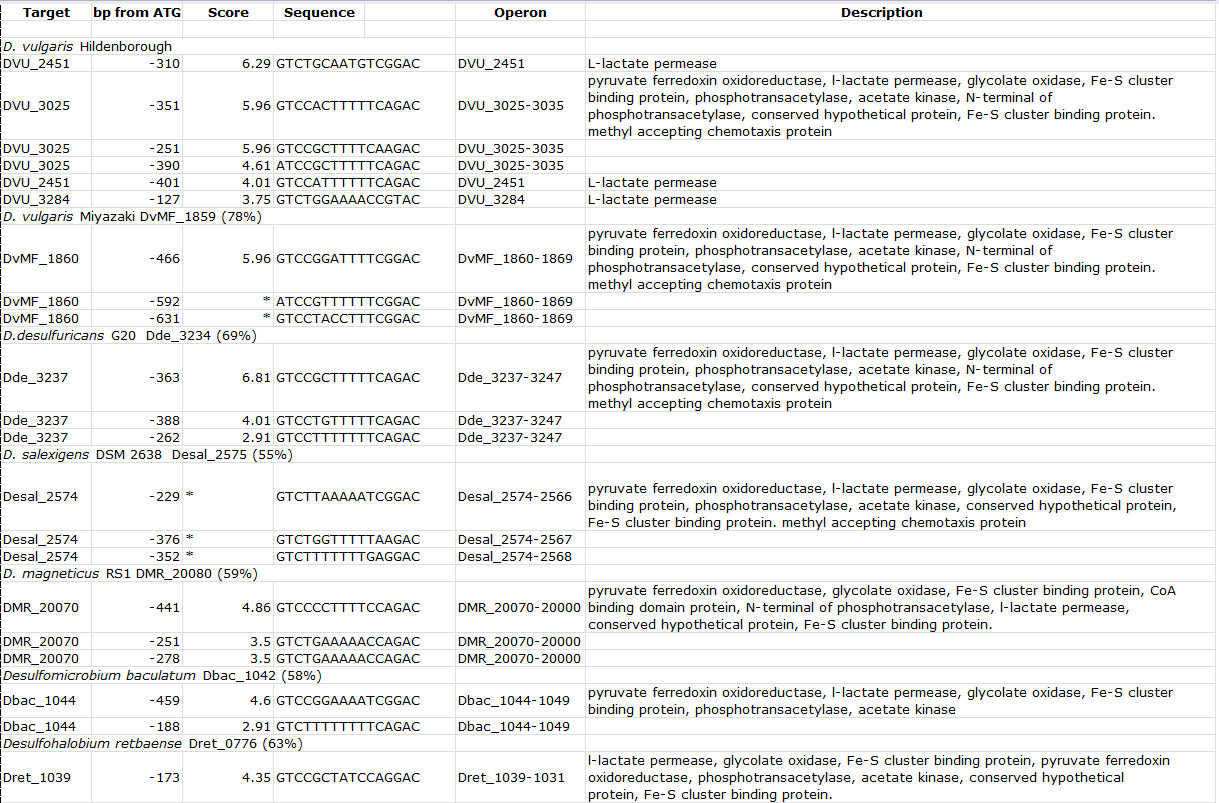
الجدول 3. الزخارف موقع ملزم لDVU3023 orthologs موجودة في غيرها منتزعة الكبريت متسلسلة والأنواع ذات الصلة. لكل الجينوم الممسوحة ضوئيا، يشار إلى اسم الحي، تليها علامة موضع لortholog DVU3023 وهويتها في المئة الى DVU3023 بين قوسين. النتيجة تشير القيمة التي تم تعيينها من قبل برنامج بيرل على أساس التشابه إلى sequen مدخلاتالمجال الاقتصادي الموحد. وصف تنص شروح الجين لجينات في الاوبرون الهدف. جدول تعديل وأعيد طبعه باستخدام رخصة المشاع الإبداعي من راجيف وآخرون 7. الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الجدول.
Discussion
تم استخدام أسلوب DAP رقاقة الموصوفة هنا بنجاح لتحديد الأهداف الجين لعدة سجلات المورد في منتزعة الكبريت الشائع Hildenborough 7 من الذي يظهر واحد هنا نتيجة ممثل. لRR DVU3023، واختيار الهدف الجين مرشح كان مباشرة. يقع DVU3025 المصب على الفور من الجين RR، ويتم حفظها لوائح الراديو والهدف الجينات في العديد من الأنواع منتزعة الكبريت، وبالإضافة إلى ذلك DVU3025 لديه تعتمد على sigma54 المروج توقع. يوفر EMSA طريقة بسيطة لاختبار بسرعة لوائح الراديو للربط إلى المرشح الجينات المستهدفة، ويسمح أيضا تقييم النشاط من العينة البروتين النقي، وكذلك تحديد شروط ملزمة الحمض النووي البروتين الأمثل.
ويمكن أيضا النشاط ملزمة الحمض النووي يتم اختبارها في حضور وغياب الاسيتيل الفوسفات لمعرفة ما إذا الفسفرة ضروري لالحمض النووي ملزمة. لا يتم فسفرته جميع الممثلين الإقليميين من المانحين جزيء صغير9، في هذه الحالات المنقى وما شابه ذلك كيناز أجهزة الاستشعار، إن وجدت، يمكن أن تستخدم لتنشيط البروتين. هناك أيضا سجلات المورد شاذة المعروفة التي تفتقر بقايا موقع نشط رئيسي في مجال الاستقبال وعدم تفعيلها من خلال الفسفرة 10. بالنسبة لغالبية الممثلين الإقليميين درس، الفسفرة يحفز الحمض النووي ملزمة 11. ولكن هناك أمثلة حيث لا يؤثر الفسفرة في الحمض النووي في المختبر ملزمة 12، وهناك أمثلة حيث هو مطلوب للربط الفسفرة 13، وكذلك الحالات التي يكون فيها فرعية من المروجين ملزمة الزيادات مع الفسفرة 14. طريقة DAP رقاقة يمكن أداؤها مع وبدون الفسفرة لتحديد ما إذا كان هناك اختلافات في مجموعة من المروجين ملزمة RR. ومع ذلك، تجدر الإشارة أيضا إلى أن بعض الممثلين الإقليميين يمكن تنقيته في شكل فسفرته وظيفيا من E. القولونية 13.
بروتوكول الموصوفة هنا هو لصاحب البروتينات الموسومةن مثل أن الحمض النووي ملزمة البروتين هو تقارب تنقيتها باستخدام ني NTA الراتنج الاغاروز، ولكن يمكن بسهولة أن تتكيف لأي نوع من البروتين الموسومة باستخدام راتنج تقارب المناسبة لالمنسدلة. الخطوات التالية DAP-WGA، ويوفر الخطوة QPCR عنصر تحكم إضافية لضمان أن الشروط ملزمة البروتين gDNA كانت المناسبة والتي تم التخصيب الحمض النووي المرتبط بالبروتين بنجاح من قبل تنقية تقارب. تخصيب أكبر من 3 أضعاف ما يكفي للمضي قدما في الخطوة التهجين. على عكس رد الفعل EMSA، لا يوجد منافس الحمض النووي غير محددة مثل وأضاف بولي dI.dC إلى gDNA رد فعل ملزم لأنه يتداخل مع خطوة WGA. ونتيجة لهذا إذا كان له نشاط البروتين عينة الحمض النووي ملزم غير محددة ثم تخصيب اضحة من الحمض النووي المستهدف أكد لن يتم مراعاتها من قبل QPCR. وبالتالي فإن خطوة السيطرة QPCR يمكن استخدامها لتحسين كمية البروتين المستخدمة في رد فعل ملزم gDNA. مجموعات المتاحة تجاريا لكامل الجينوم التضخيم يدعون INTRالعامة oduce أي تحيز التضخيم عند اتباع المبادئ التوجيهية من أجل الحد الأدنى المواد الحمض النووي البداية وانخفاض عدد دورات التضخيم. يمكن مراجعة التحيز التضخيم باستخدام QPCR مع مختلف مجموعات التمهيدي على تضخيم الحمض النووي مقابل unamplified. ويمكن أيضا أن تحدد أي تأثير على مجرى النهر مجموعة التهجين عن طريق وضع العلامات تفاضلي والتهجين المجمعة تضخيمها وunamplified الحمض النووي الجيني.
قائمة من القمم التي تم إنشاؤها بعد تحليل بيانات مجموعة عادة طويلة مع عدة قمم وجود قيمة معدل اكتشاف كاذبة من 0. ولذلك مزيج من سمات ذروة أخرى مثل سجل ارتفاع 2 عشرات R والقيم cutoff_p (كما ولدت من خلال تحليل مجموعة وتستخدم البرمجيات المستخدمة في هذه الدراسة) لاعدام القائمة أسفل إلى أعلى الأهداف الجينات الثقة. وجود هدف محدد مسبقا الجينات بين أفضل خمسة إصابات يعزز إلى حد كبير من الثقة في مجموعة البيانات. أداء هذا الاختبار لعدد من البروتينات التنظيمية جيئة وذهابام سوف نفس الحي يساعد أيضا على تحديد "لزجة" تسلسل الحمض النووي التي تظهر في العديد من مجموعات البيانات 7. بالإضافة إلى ذلك إذا كان من الممكن توقع موقع عزر ملزمة والتحقق من صحة ثم وجود الحافز في قمم خفض أسفل في القائمة يمكن أن تستخدم أيضا لاختيار قائمة الجينات المستهدفة المحافظة. إثراء تسلسل الحمض النووي بخلاف مناطق المروج قد تكون القطع الأثرية من مجموعة التهجين أو قد تشير إلى تنظيم إطارات القراءة المفتوحة مجهولة سابقا أو الرنا صغيرة 7. للمنظمين حيث لا يمكن سلفا هدف الجينات باستخدام EMSA، قد يتم تنفيذ الفحص DAP رقاقة "العمياء" 7. في مثل هذه الحالات أيضا، تحديد موقع عزر ملزمة من شأنها تحسين اختيار الأهداف الجينات. سوف تحديد تركيز البروتين لاستخدامها في فحص تعتمد على النشاط غير محددة وملزمة من البروتين، والتي يمكن تقييمها من قبل EMSA باستخدام الحمض النووي ركائز اختيارها عشوائيا. تركيزات أقل تعمل على نحو أفضل لرالبروتينات خرطوم مع ارتفاع النشاط ملزمة غير محددة. موثوقية أعمى DAP رقاقة يمكن أن يتحسن عن طريق إجراء فحوصات تكرار مع تركيزات بروتين مختلفة. لسجلات المورد مع عدد قليل من الأهداف، قد يكون اثنين فقط أو حتى فحص واحد كافية لإنشاء قائمة المستهدفة التي يمكن التحقق من صحتها باستخدام EMSAs اللاحقة. البيانات DAP رقاقة لمثل سجلات المورد عادة ما تظهر قفزة واضحة في عشرات R سجل 2 أو القيم cutoff_p وراء قمم القليلة الأولى. لسجلات المورد مع العديد من الأهداف، ويمكن تحليل البيانات من ثلاثة مكررات لإنشاء قائمة من القمم المشتركة، والبعض منها قد يكون محددا للمصادقة EMSA.
القدرة على التنبؤ الزخارف موقع ملزمة استنادا إلى الأهداف DAP رقاقة يضيف إلى قيمة هذا الأسلوب ويزيد بشكل كبير من المعلومات المكتسبة. يوفر EMSA مرة أخرى بروتوكول للتحقق من كفاءة تجريبيا التوقعات. مخطوطات برامج في بيرل أو لغة برمجة أخرى يمكن استخدامها للبحث بسرعة عن طريق المتاحة seque الجينومالامتحانات التنافسية الوطنية. فإن نتائج تحديد كل الجينات المستهدفة orthologous وكذلك الجينات المستهدفة ينظم فريد في الجينوم البحث. في النتيجة ممثل هو موضح هنا، وثلاثة من المناطق المستهدفة المنبع الأربعة المحددة لDVU3023 والمشروح وظائف الجينات المتعلقة امتصاص اكتات والأكسدة. بالإضافة إلى ذلك منذ أن تم التحقق من صحة موقع عزر ملزمة لDVU3023، يمكن البحث عن المواقع الأخرى الجينوم عزر مماثلة. معا تشير النتائج إلى أن DVU3022-3023 TCS يتم حفظها جيدا في منتزعة الكبريت وغيرها من البكتيريا والحد كبريتات ذات الصلة، وأنه ينظم الجينات للنقل اللاكتات والأكسدة لخلات. منذ اللاكتات هو الكربون والإلكترون المصدر الرئيسي المستخدمة من قبل هذه الكائنات، DVU3023 من المرجح ان يلعب دورا رئيسيا في علم وظائف الأعضاء الخاصة بهم. من بين أعلى قمم DAP-رقاقة لRR DVU3023، كان هناك أيضا منطقة بين الجينات. على الرغم من أن موقع عزر ملزمة لم يتم العثور في هذه المنطقة، وجود سيغما تعتمد على 54 مروجا وتوقع يوحي رقبعة قد تكون هناك ORF مجهولين أو الحمض الريبي النووي الذواب المشفرة، وأنه يمكن أن يكون هدفا صحيحا لDVU3023. هذه النتيجة يسلط الضوء على قيمة النهج DAP رقاقة التجريبية جنبا إلى جنب مع التنبؤات موقع ملزم بدلا من تحديد المواقع المستهدفة على أساس التنبؤات الحسابية وحدها.
أساليب مماثلة في المختبر مثل واحد هو موضح هنا تم استخدامها فقط في حالات قليلة جدا، ونادرا ما 15-17 لمنظم غير مدروسة مسبقا. التحسين EMSA وQPCR الخطوات قبل التهجين مجموعة سوف تساعد إلى حد كبير في تحليل النتائج عند هذه الطريقة التي ستستخدم لمنظم الرواية. إذا أصبح عائقا مجموعة الطباعة، ويمكن الجمع بين الخطوات DAP مع الجيل القادم التسلسل للحصول على مواقع الربط 18،19. كما مزيد من الأساليب المستندة إلى مجموعة تصبح استبداله مع الاستراتيجيات القائمة على التسلسل، مثل التكيف المستقبلي للطريقة تلتف على الحاجة لتصميم ميكروأرس تبليط مخصصة لسrganism من الفائدة. التكنولوجيات رقاقة تسلسل يؤدي إلى مزيد من حساسية وخصوصية كشف عن القمم بالمقارنة مع الأساليب رقاقة رقاقة، وأيضا أصبحت بسرعة أكثر فعالية من حيث التكلفة 20. على الرغم من أن هذه المادة تركز على اثنين من المنظمين استجابة عنصر، وهذه الطريقة يمكن أن تستخدم في أي عامل النسخ حقيقية النواة بدائية النواة أو 15. إشغال النيوكليوسومات غالبا ما يكون لها تأثير على الأهداف التي تنظم ومقارنة الهدف المواقع لعوامل النسخ حقيقية النواة من قبل في الجسم الحي في المختبر ملزمة وتحليلات يمكن أن تكشف عن هذه الآثار 21.
Disclosures
والكتاب ليس لديهم تضارب المصالح في الكشف عنها.
Acknowledgements
نشكر ايمي تشن لمساعدتها في التحضير لتصوير الفيديو ولإظهار هذه التقنية. هذا العمل الذي قامت به ENIGMA: النظم البيئية والشبكات المتكاملة مع الجينات الجزيئية وجمعيات (http://enigma.lbl.gov)، وهو برنامج مجال التركيز العلمي في مختبر لورنس بيركلي الوطني، وبدعم من مكتب العلوم ومكتب البيولوجية و البحوث البيئية، من وزارة الطاقة في الولايات المتحدة بموجب العقد رقم DE-AC02-05CH11231.
Materials
| Name | Company | Catalog Number | Comments |
| HisTrapFF column (Ni-Sepharose column) | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5255-01 | |
| Akta explorer (FPLC instrument) | GE Lfe Sciences, Pittsburgh, PA, USA | ||
| HiPrep 26/10 Desalting column | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5087-01 | |
| Qiaquick Gel extraction kit | Qiagen Inc, Valencia, CA, USA | 28704 | |
| Biotin-labeled oligonucleotides | Integrated DNA Technologies | N/A | |
| 6% polyacrylamide-0.5x TBE precast mini DNA retardation gel | Life Technologies, Grand Island, NY, USA | EC63652BOX | Alternately, you can pour your own gel. |
| Nylon membrane | EMD Millipore, Billerica, MA, USA | INYC00010 | |
| Trans-Blot SD Semi-dry electrophoretic transfer cell | Biorad, Hercules, CA, USA | 170-3940 | |
| Extra thick blot paper, 8 x 13.5 cm | Biorad, Hercules, CA, USA | 170-3967 | |
| UV crosslinker Model XL-1000 | Fisher Scientific | 11-992-89 | |
| Nucleic Acid chemiluminescent detection kit (Pierce) | Thermo fisher Scientific, Rockford, IL, USA | 89880 | |
| Ni-NTA agarose resin | Qiagen Inc, Valencia, CA, USA | 30210 | |
| GenomePlex Whole genome amplification kit (Fragmentation buffer, library preparation buffer, library stabilization solution, library preparation enzyme, 10x amplification master mix, WGA polymerase) | Sigma-Aldrich, St. Louis, MO, USA | WGA2-50RXN | |
| Nanodrop ND-1000 | Thermo Scientific, Wilmington, DE, USA | For quantitation of DNA | |
| Perfecta Sybr Green SuperMix, with ROX | Quanta biosciences | 95055-500 | Any Sybr Green PCR mix may be used |
| PlateMax Ultra clear heat sealing film for qPCR | Axygen | ||
| [header] | |||
| 96-well clear low profile PCR microplate | Life Technologies, Grand Island, NY, USA | PCR-96-LP-AB-C | |
| Applied Biosystems StepOne Plus Real time PCR system | Life Technologies, Grand Island, NY, USA | 4376600 | Any real time PCR system may be used |
| Qiaquick PCR purification kit | Qiagen Inc, Valencia, CA, USA | 28104 | Any PCR clean up kit may be used |
| Cy3/Cy5-labeled nonamers | Trilink biotechnologies, San Diego, CA, USA | N46-0001, N46-0002 | |
| Klenow polymerase 50,000 U/ml, 3'-5' exo- | New England Biolabs, Ipswich, MA | M0212M | |
| Hybridization system | Roche-Nimblegen, Madison, WI, USA | N/A | This company no longer makes arrays or related items, so alternate sources such as Agilent or Affymetrix will need to be used. |
| Custom printed microarrays and mixers | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Hybridization kit (2x Hybridization buffer, Hybridization component A, Alignment oligo) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Wash buffer kit (10x Wash buffer I, II, III, 1 M DTT) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| GenePix 4200A microarray scanner | Molecular Devices, Sunnyvale CA, USA | This model has been replaced by superior ones | |
| GenePix Pro microarray software | Molecular Devices, Sunnyvale CA, USA | ||
| Nimblescan v.2.4, ChIP-chip analysis software | Roche-Nimblegen, Madison, WI, USA | N/A | |
References
- Galperin, M. Y. Diversity of structure and function of response regulator output domains. Curr Opin Microbiol. 13, 150-159 (2010).
- Casino, P., Rubio, V., Marina, A. The mechanism of signal transduction by two-component systems. Curr Opin Struct Biol. 20, 763-771 (2010).
- Aparicio, O., Geisberg, J. V., Struhl, K. Chromatin immunoprecipitation for determining the association of proteins with specific genomic sequences in vivo. Curr Protoc Cell Biol. 17 (17), (2004).
- McCleary, W. R., Stock, J. B. Acetyl phosphate and the activation of two-component response regulators. J Biol Chem. 269, 31567-31572 (1994).
- Novichkov, P. S., et al. RegPrecise web services interface: programmatic access to the transcriptional regulatory interactions in bacteria reconstructed by comparative genomics. Nucleic Acids Res. 40, 604-608 (2012).
- Studholme, D. J., Buck, M., Nixon, T. Identification of potential sigma(N)-dependent promoters in bacterial genomes. Microbiology. 146 (12), 3021-3023 (2000).
- Rajeev, L., et al. Systematic mapping of two component response regulators to gene targets in a model sulfate reducing bacterium. Genome Biol. 12, 99(2011).
- Crooks, G. E., Hon, G., Chandonia, J. M., Brenner, S. E. WebLogo: a sequence logo generator. Genome Res. 14, 1188-1190 (2004).
- Barbieri, C. M., Mack, T. R., Robinson, V. L., Miller, M. T., Stock, A. M. Regulation of response regulator autophosphorylation through interdomain contacts. J Biol Chem. 285, 32325-32335 (2010).
- Bourret, R. B. Receiver domain structure and function in response regulator proteins. Curr Opin Microbiol. 13, 142-149 (2010).
- Gao, R., Stock, A. M. Molecular strategies for phosphorylation-mediated regulation of response regulator activity. Curr Opin Microbiol. 13, 160-167 (2010).
- Hung, D. C., et al. Oligomerization of the response regulator ComE from Streptococcus mutans is affected by phosphorylation. J Bacteriol. 194, 1127-1135 (2012).
- Ladds, J. C., et al. The response regulator Spo0A from Bacillus subtilis is efficiently phosphorylated in Escherichia coli. FEMS Microbiol Lett. 223, 153-157 (2003).
- Wen, Y., Feng, J., Scott, D. R., Marcus, E. A., Sachs, G. Involvement of the HP0165-HP0166 two-component system in expression of some acidic-pH-upregulated genes of Helicobacter pylori. J Bacteriol. 188, 1750-1761 (2006).
- Liu, X., Noll, D. M., Lieb, J. D., Clarke, N. D. DIP-chip: rapid and accurate determination of DNA-binding specificity. Genome Res. 15, 421-427 (2005).
- Gossett, A. J., Lieb, J. D. DNA Immunoprecipitation (DIP) for the Determination of DNA-Binding Specificity. CSH Protoc. 2008, (2008).
- Mascher, T., et al. The Streptococcus pneumoniae cia regulon: CiaR target sites and transcription profile analysis. J Bacteriol. 185, 60-70 (2003).
- Park, P. J. ChIP-seq: advantages and challenges of a maturing technology. Nat Rev Genet. 10, 669-680 (2009).
- Lefrancois, P., Zheng, W., Snyder, M. ChIP-Seq using high-throughput DNA sequencing for genome-wide identification of transcription factor binding sites. Methods Enzymol. 470, 77-104 (2010).
- Ho, J. W., et al. ChIP-chip versus ChIP-seq: lessons for experimental design and data analysis. BMC Genomics. 12, 134(2011).
- Liu, X., Lee, C. K., Granek, J. A., Clarke, N. D., Lieb, J. D. Whole-genome comparison of Leu3 binding in vitro and in vivo reveals the importance of nucleosome occupancy in target site selection. Genome Res. 16, 1517-1528 (2006).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved