Method Article
Triphosphorylation chimique des oligonucléotides
Dans cet article
Résumé
Les oligonucléotides 5′-triphosphates sont des composants omniprésents dans les voies biologiques essentielles et ont été de plus en plus utilisés dans les applications biotechnologiques. Nous décrivons ici des techniques de synthèse et de purification de routine des oligonucléotides 5′-triphosphates, à partir d’oligonucléotides préparés par des techniques de synthèse automatisées standard.
Résumé
Le 5′-triphosphate est une modification essentielle de l’acide nucléique que l’on trouve tout au long de la vie et qui est de plus en plus utilisée comme modification fonctionnelle des oligonucléotides en biotechnologie et en biologie synthétique. Les oligonucléotides 5′-triphosphates ont toujours été préparés in vitro par des méthodes enzymatiques. Cependant, ces méthodes sont limitées aux oligonucléotides naturels de l’ARN, ont de fortes préférences de séquence et ont tendance à produire des produits hétérogènes. De nouvelles méthodes de triphosphorylation chimique complètent à la fois le coût réduit de la synthèse automatisée des oligonucléotides par la chimie du phosphoramidite et la gamme variée de modifications des nucléotides maintenant disponibles. Ainsi, la synthèse d’oligonucléotides triphosphates de séquence et de longueur arbitraires, et contenant éventuellement diverses modifications non naturelles, est désormais accessible.
Cet article présente les méthodes et techniques appropriées pour la triphosphorylation chimique des oligonucléotides à l’aide de salicyl phosphorochloridite et de pyrophosphate. Cette méthode utilise des réactifs disponibles dans le commerce, est compatible avec la plupart des oligonucléotides préparés par des méthodes standard de synthèse en phase solide, et peut être complétée en 2 h après la synthèse des oligonucléotides, avant la déprotection et la purification. Deux utilisations d’oligonucléotides chimiquement triphosphorylés comme substrats pour les enzymes d’ARN catalytique sont démontrées, y compris la synthèse d’une version en image miroir du ribozyme à tête de marteau à partir de triphosphates d’ARN L non biologiques.
Introduction
La forme 5′-triphosphorylée de l’ARN est omniprésente en biologie car elle est générée par la transcription de l’ARN dans tous les domaines de la vie et par la réplication de l’ARN au cours du cycle de vie de nombreux virus à ARN. Ces triphosphates servent de substrat pour la formation d’ARNm coiffé de 7-méthylguanylate chez les eucaryotes et, par conséquent, jouent un rôle essentiel dans l’expression des protéines1. En revanche, le triphosphate est retenu dans les bactéries et les virus; ainsi, les ARN 5′-triphosphates sont reconnus par les régulateurs de la réponse immunitaire innée chez les eucaryotes 2,3,4,5,6,7. En dehors de la biologie, une foule de ribozymes d’ARN ligase ont été développés pour utiliser le 5′-triphosphate in vitro8 et modifiés pour une utilisation dans les tests diagnostiques 9,10,11,12,13,14,15. L’un de ces ribozymes peut être utilisé pour la synthèse dépendante du modèle de l’ARN-L, l’énantiomère non biologique « image miroir » de l’ARN D naturel, à partir de petits oligonucléotides d’ARN L 5′-triphosphates 16,17,18. La préparation de routine d’oligonucléotides triphosphorylés de séquence et de composition dorsale variables est essentielle à l’étude de ces systèmes.
La méthode la plus courante et la plus accessible pour préparer l’ARN 5′-triphosphates en laboratoire est la transcription in vitro. Cependant, l’ARN produit par cette méthode est limité dans la séquence et la taille par les exigences du promoteur et du substrat de l’enzyme ARN polymérase. L’ARN polymérase T7 et ses dérivés spécialisés sont les polymérases les plus couramment utilisées à cette fin 19,20,21,22. L’ARN transcrit in vitro préparé avec ces enzymes doit être initié avec une purine 5′-terminale et est fortement biaisé vers les purines dans les 10 premiers nucléotides23,24. De plus, l’incorporation enzymatique de nucléotides modifiés par la base ou l’épine dorsale est au mieux inefficace et plus souvent impossible avec les polymérases naturelles, limitant la possibilité de produire des oligonucléotides 5′-triphosphates composés de tout sauf de l’ARN D naturel. Un autre facteur limitant est que l’ARN généré par transcription in vitro peut contenir une hétérogénéité substantielle de 5′ et 3′ et est produit sous forme de produits extrêmement hétérogènes lorsqu’il est inférieur à20 nt 23,24,25,26,27.
En revanche, la triphosphorylation chimique d’oligonucléotides préparés par synthèse de phosphoramidite en phase solide 28,29,30,31,32,33,34,35 peut être utilisée pour préparer des oligonucléotides 5′-triphosphates de 3-50 nt de long, de n’importe quelle séquence. De plus, une vaste gamme de modifications des acides nucléiques accessibles à la synthèse du phosphoramidite peut être ajoutée aux oligonucléotides avant la 5′-triphosphorylation 14,15,16,17,18,29,36. Beaucoup de ces méthodes utilisent le réactif de phosphitylation salicyl phosphorochloridite, qui a été développé par Ludwig et Eckstein pour la triphosphorylation en phase solution des mononucléosides37. La triphosphorylation des oligonucléotides avec ce réactif est réalisée sur la phase solide par phosphitylation de l’oligonucléotide 5′-hydroxyle, conversion en triphosphate par réaction avec pyrophosphate et oxydation, suivie de procédures standard de clivage de l’oligonucléotide du support solide, de protection et de purification (Figure 1)28.
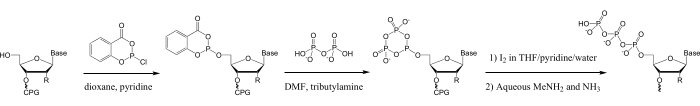
Figure 1 : Schéma de triphosphorylation des oligonucléotides synthétiques. Dans la première étape, l’oligonucléotide 5ʹ-hydroxyle est phosphitélé avec SalPCl. Dans l’étape suivante, le phosphite de 5ʹ-salicyle réagit avec tbAP pour former le métaphosphite cyclique, puis dans la troisième étape oxydé pour générer le 5ʹ-trimétaphosphate cyclique dans une solution d’oxydation de synthétiseur ADN/ARN (0,1 M d’iode/pyridine/H2O/THF), qui est rapidement hydrolysé pour obtenir le 5ʹ-triphosphate linéaire dans la même solution28,33, 37. Le clivage alcalin ultérieur du support solide du CPG et la déprotection de l’oligonucléotide dans la MeNH2 aqueuse/ammoniac hydrolyseront tout trimétaphosphate cyclique résiduel sous forme linéaire. Abréviations : SalPCl = phosphorochloridite de salicyle; TBAP = pyrophosphate de tributylammonium; THF = tétrahydrofurane; CPG = verre à pores contrôlés; MeNH2 = méthylamine. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Bien que les premiers rapports publiés utilisant cette méthode aient souvent souffert de faibles rendements et de produits secondaires indésirables 28,37,38, un entretien minutieux des conditions anhydres est tout ce qui est nécessaire pour obtenir régulièrement des rendements élevés. Cela peut être réalisé par une préparation minutieuse des réactifs et l’utilisation d’un dispositif de réaction simple assemblé à partir de composants en plastique standard. Ici, nous démontrons les étapes appropriées pour la triphosphorylation chimique des oligonucléotides, y compris la préparation des réactifs, l’assemblage de la chambre de réaction, la réaction de triphosphorylation, et la déprotection et la purification ultérieures des oligonucléotides triphosphorylés. L’utilisation représentative d’oligonucléotides 5′-triphosphorylés comme substrats pour les ribozymes ligase pour la synthèse de produits d’acides nucléiques plus importants avec de l’ARN D naturel et des épines dorsales abiotiques de l’ARN-L est également incluse.
Protocole
1. Synthèse automatisée en phase solide d’oligonucléotides 5′-hydroxyles sur un support solide
- Préparer le synthétiseur AUTOMATISÉ ADN/ARN avec des réactifs et des phosphoramidites selon la composition oligonucléotidique cible et les instructions de l’instrument.
- Chargez une colonne de synthèse contenant un support solide sur le synthétiseur et synthétisez des oligonucléotides selon les protocoles de l’instrument du synthétiseur.
REMARQUE: La procédure de triphosphorylation a été optimisée pour les oligonucléotides préparés à l’échelle de 1 μmole. - Retirer le groupe protecteur 5′-diméthoxytrityle pour obtenir l’oligonucléotide 5′-hydroxyle solide dans le cadre de la synthèse des oligonucléotides à l’étape précédente, ou en effectuant une étape de détritylation terminale selon les protocoles de l’instrument synthétiseur.
- Retirer la colonne contenant l’oligonucléotide 5′-hydroxyle sur support solide du synthétiseur, sécher sous vide domestique pendant 10 min pour éliminer le solvant résiduel, et procéder à la triphosphorylation (sections 3 et 4) une fois que les matériaux pour la triphosphorylation sont préparés (section 2).
REMARQUE: Si elle n’est pas utilisée immédiatement, la colonne séchée peut être stockée sous une atmosphère normale dans un récipient en plastique scellé avec un dessiccant à -20 ° C. Un séchage supplémentaire n’est pas nécessaire à ce stade car la colonne est soigneusement séchée avant la triphosphorylation à la rubrique 3.
2. Préparation des matériaux pour la triphosphorylation
- Fixez une source d’argon sec avec pression réglable à un collecteur de gaz avec au moins deux lignes et connectez-vous à un barboteur. Assurez-vous que les lignes se terminent dans des seringues en plastique de 1 mL pour faciliter la connexion à l’appareil de réaction.
- Rassemblez l’équipement à utiliser pendant la triphosphorylation, y compris des seringues en plastique de 1 mL, un robinet d’arrêt en polypropylène à trois voies, des aiguilles non carcibles, des tubes en polypropylène de 1,5 mL et une petite spatule en métal. Conservez-les dans un récipient scellé ou un dessiccateur avec du dessiccant à température ambiante pendant au moins 1 jour avant utilisation.
- Préparer 30 mL chacun de 1,4-dioxane anhydre, de dioxane:pyridine 3:1 en volume, de N,N-diméthylformamide (DMF) et d’acétonitrile (ACN) dans des bouteilles en verre séchées de 30 mL avec pièges à séchage (4 tamis moléculaires Å dans des sachets à membrane scellés) au moins 1 jour avant utilisation. Sceller avec des septa en caoutchouc et stocker dans un dessiccateur avec un dessiccant.
- Conserver le 2-chloro-4H-1,3,2-benzodioxaphosphorin-4-one solide (phosphorochloridite de salicyle, SalPCl) dans son contenant d’origine dans un bocal scellé avec un dessiccant à 4 °C. Rincez toujours le récipient avec de l’argon entre les utilisations.
- Préparer une solution de pyrophosphate de tributylammonium (TBAP) au moins 5 jours avant la réaction de triphosphorylation :
- Peser 1 à 5 g de TBAP solide dans une bouteille en verre séchée de 30 mL et dissoudre dans 1 mL de DMF et 0,5 mL de tributylamine par g de TBAP.
- Ajouter trois pièges de séchage, sceller la bouteille avec un septum en caoutchouc sous argon et faire une bulle avec de l’argon pendant 30 min pour dégazer.
- Conserver dans un bocal scellé avec un dessiccant à 4 °C pendant 5 jours pour permettre aux tamis moléculaires d’absorber toute trace d’eau. Conserver le pot à -20 °C et préparer frais après 6 mois.
3. Assemblage et utilisation de l’appareil de triphosphorylation
- Laisser la colonne de synthèse se réchauffer à la température ambiante si elle est récupérée du stockage à -20 °C.
- Assemblez la chambre de réaction illustrée à la figure 2 :
- Préparez l’antichambre : retirez le piston d’une seringue sèche de 1 mL, coupez le haut de la seringue à l’aide de ciseaux ou d’une lame de rasoir et fixez la seringue à la colonne de synthèse. Fixez le robinet d’arrêt à trois voies au sommet de la seringue et fixez l’entrée latérale du robinet d’arrêt à la source d’argon sec avec le barboteur, de sorte que l’entrée supérieure du robinet d’arrêt soit l’orifice d’injection du réactif.
- Fixez cet appareil à un support avec des pinces et scellez tous les joints en amont avec un film d’étanchéité à la cire. Réglez le robinet d’arrêt de sorte que l’orifice d’injection soit fermé et que l’appareil soit ouvert à la source d’argon. Fermez le barboteur et laissez l’argon à basse pression (<10 psi) circuler dans la chambre de réaction pendant 5 min.
REMARQUE: Plusieurs chambres de réaction peuvent être réglées parallèlement aux oligonucléotides triphosphorylates 2-4. Cependant, une ligne du collecteur doit être réservée à l’approvisionnement en argon des flacons de réactif. - Rouvrez le barboteur et fixez une seringue au bas de la colonne de synthèse, qui sera la seringue à déchets. Tirez l’argon à travers la colonne à plusieurs reprises à l’aide de la seringue à déchets; ensuite, reconnectez la seringue avec le piston enfoncé complètement.
REMARQUE: Sauf si les réactifs de chargement sont chargés, le robinet d’arrêt doit être réglé de manière à ce que l’orifice d’injection soit fermé et que l’appareil soit ouvert à la source d’argon, comme illustré à la figure 2A. De même, la seringue à déchets doit être fixée et le joint à la colonne de synthèse scellé avec un film d’étanchéité à la cire, à moins d’éliminer activement les réactifs.
- Pour ajouter un réactif ou un solvant :
- Fixez une aiguille à la source d’argon sec et insérez-la dans le septum du réactif ou du flacon de solvant, en prenant soin de ne pas immerger l’aiguille dans le contenu du flacon.
- Assemblez une seringue sèche avec une aiguille et insérez-la dans le septum du réactif ou du flacon de solvant, sans l’immerger dans le contenu du flacon. Remplissez la seringue avec de l’argon, retirez l’aiguille du septum et expulsez l’argon. Remplissez la seringue avec de l’argon et expulsez à nouveau; ensuite, remplissez la seringue avec le volume requis de solvant ou de réactif sous pression d’argon.
- Réglez le robinet d’arrêt de l’appareil de sorte que la source d’argon soit fermée et que l’orifice d’injection soit ouvert (Figure 2B). Retirez rapidement la seringue et l’aiguille remplies du flacon source, essuyez tout solvant collé sur le côté ou à l’extrémité de l’aiguille et insérez l’aiguille dans l’orifice d’injection. Expulsez le réactif dans l’antichambre de l’appareil, retirez l’aiguille et fermez l’orifice d’injection, rouvrant l’appareil à la source d’argon.
- Aspirez doucement le liquide de l’antichambre tout au long de la colonne de synthèse à l’aide de la seringue à déchets afin que tout le liquide soit maintenant maintenu dans la seringue à déchets. Maintenant, repoussez lentement la solution dans la colonne de synthèse, en vous assurant qu’aucune bulle de gaz n’est dans la colonne. Pour mélanger ou agiter, tirez doucement la solution de haut en bas sur la colonne avec la seringue à déchets.
REMARQUE: Déplacez toujours le liquide lentement et doucement à travers la chambre de réaction pour vous assurer qu’aucun joint n’est brisé, ce qui permet à l’air de pénétrer dans l’appareil.
- Pour retirer un réactif ou un solvant de la colonne :
- Tirez lentement la solution dans la seringue à déchets. Une fois que la majeure partie de la solution est passée dans la seringue à déchets, tirez l’argon pour rincer le solvant restant de la colonne.
- Retirez le film d’étanchéité à la cire autour du joint de la seringue usagée, puis retirez la seringue et jetez la solution de déchets. Remplacez la seringue usagée par une nouvelle seringue sèche et refermez le joint avec un film d’étanchéité à la cire.

Figure 2 : Appareil de triphosphorylation. Pendant le mélange ou les réactions, le dispositif (A) est ouvert à la source d’argon (i) et fermé à l’air en ajustant le robinet d’arrêt à trois voies (ii). Les réactifs sont extraits de l’antichambre iii) dans la colonne de synthèse iv) au moyen de la seringue à déchets v). Les réactifs sont éliminés en aspirant tout le liquide dans la seringue à déchets (v) et en les jetant. Lors du chargement des réactifs (B), le robinet d’arrêt à trois voies (ii) est ouvert à l’atmosphère et le réactif est chargé dans l’antichambre (iii) au moyen d’une seringue et d’une aiguille (vi). C) Une photographie de l’appareil assemblé réglé comme au point A) pour le mélange et la réaction des réactifs. Veuillez cliquer ici pour voir une version agrandie de cette figure.
4. Triphosphorylation sur colonne d’oligonucléotides synthétiques de 5′-hydroxyle
- Retirer la solution SalPCl et TBAP du stockage à -20 °C et les laisser chauffer à température ambiante avant utilisation.
- Ajouter 200 μL de pyridine/dioxane à l’antichambre, conformément aux étapes 3.3.1-3.3.3. Toutefois, ne chargez pas de solvant sur la colonne de synthèse avant l’étape 4.4.
- Utilisez une spatule métallique sèche pour peser 6 à 12 mg de SalPCl dans un tube de microcentrifugation sec de 1,5 mL et dissolvez-la dans 100 μL de dioxane en faisant doucement griller le solvant de haut en bas dans le tube de microcentrifugation.
- Ajouter le SalPCl dissous à l’antichambre et le charger sur la colonne de synthèse, en suivant l’étape 3.3. Laissez-le réagir pendant 15 min, en agitant la solution toutes les 5 min. Retirez et jetez la solution SalPCl conformément à l’étape 3.4.
REMARQUE: SalPCl est ajouté en grand excès et récupérera toute l’eau absorbée pendant la préparation et le chargement dans la chambre de réaction. Cependant, l’introduction d’humidité au cours des étapes 4.5 et 4.6 compromettra le rendement final en oligonucléotide 5′-triphosphate. - Ajouter 250 μL de solution TBAP à l’antichambre et charger sur la colonne de synthèse, en suivant l’étape 3.3. Laissez-le réagir pendant 20 min, en agitant toutes les 5 min. Retirez et jetez la solution TBAP conformément à l’étape 3.4.
- Laver la colonne avec 0,5 mL de DMF, puis 0,5 mL d’ACN, en retirant le solvant après chaque addition selon les étapes 3.3 et 3.4.
- Ajouter 250 μL de solution comburante (0,1 M d’iode dans du tétrahydrofurane (THF)/pyridine/eau, 88:10:2) à l’antichambre et la charger sur la colonne de synthèse, en suivant l’étape 3.3. Laissez-le réagir pendant 30 min, en agitant toutes les 10 min. Retirer et jeter la solution comburante conformément à l’étape 3.4.
- Lavez la colonne avec 0,5 mL d’ACN et retirez-la, conformément aux étapes 3.3 et 3.4.
- Démontez l’appareil de réaction. Lavez la colonne de synthèse avec 5 mL d’ACN et séchez la colonne.
5. Décolleté du support solide, de la déprotection et de la purification
- Retirez la résine de support solide séchée de la colonne de synthèse et transférez-la dans un tube scellable à bouchon à vis en polypropylène de 1,5 mL avec un joint torique en silicone.
- Suspendre la résine dans 1 mL d’un mélange 1:1 d’ammoniac aqueux à 28% à 30% et de méthylamine aqueuse (AMA) à 40% et sceller hermétiquement le tube. Incuber à 65 °C pendant 10 min avec mélange intermittent par inversion douce39. Utiliser un traitement plus doux à température ambiante pendant 2 h pour les oligonucléotides de plus de 40 nt.
ATTENTION: Le chauffage de la solution AMA mettra le tube sous haute pression. Si le tube n’est pas hermétiquement scellé ou n’utilise pas de joint torique compatible avec l’ammoniac (silicone), le tube peut évacuer le gaz ou le solvant de fuite, ce qui peut compromettre la sécurité ou le rendement du produit final. N’ouvrez jamais le tube lorsqu’il est au-dessus de la température ambiante, car la solution AMA chaude peut produire violemment du gaz. - Refroidir le tube sur de la glace et le centrifuger brièvement (6 000 à 12 000 × g pendant 10 s). Retirez le surnageant de la résine, filtrez à travers une seringue équipée d’un filtre de 0,2 μm et transférez-le dans un nouveau tube stérile en polypropylène. Évaporer la solution à sec à l’aide d’une centrifugeuse à vide munie d’un piège chimique neutralisant l’ammoniac.
REMARQUE: Si l’oligonucléotide synthétique ne contient pas de nucléotides d’ARN avec des groupes protecteurs 2′-silyl, passez à l’étape 5.8. - Éliminer les groupes protecteurs du silyle en dissolvant la matière séchée dans 1 mL de 1 M de fluorure de tétrabutylammonium (TBAF) dans le THF, en chauffant à 55 °C, en agitant si nécessaire pour dissoudre complètement l’oligonucléotide et en incubant à température ambiante pendant 16-24 h40,41.
- Trempez la solution TBAF avec 1 mL de tampon Tris de 1 M, pH 7,5, et retirez le THF à l’aide d’une centrifugeuse à vide.
- Retirez les sels TBAF à l’aide d’une colonne d’exclusion de taille jetable, en suivant les instructions du fabricant. Pour les oligonucléotides de moins de 15 nt, confirmer l’élution du produit en collectant l’éluat en fractions, et en identifiant les principales fractions du produit par absorbance à 260 nm sur un spectrophotomètre UV-Vis.
- Concentrer l’oligonucléotide d’ARN déprotégé par lyophilisation ou centrifugeuse sous vide si elle est inférieure à 15 nt, ou par précipitation d’éthanol si supérieure à 15 nt.
- Préparez une échelle préparative 10%-20% de polyacrylamide / 8 M d’urée / 1x gel TBE en utilisant du bouillon d’acrylamide mono:bis 19: 1, selon les protocoles appropriés pour la taille de l’oligonucléotide, la plaque de gel et le support. Assemblez la plaque de gel dans le support de gel avec 1x TBE dans des réservoirs et pré-exécutez à 35 W (ou selon le cas pour le format de plaque de gel) pendant au moins 30 min.
- Dissoudre l’oligonucléotide solide dans un tampon de chargement de gel d’urée (8 M d’urée, 10 % de saccharose, 50 mM de Tris, pH 8, 1 mM d’EDTA avec des colorants courants de bromophénol et de xylène cyanol) et chauffer à 80 °C. Chargez sur le gel de polyacrylamide et courez pendant 1-2 h à 25-35 W (ou selon le cas pour le format de la plaque de gel et la taille de l’oligonucléotide).
REMARQUE: Le bleu de bromophénol ou le cyanol de xylène doivent être exclus du tampon de chargement du gel pour les oligonucléotides de moins de 15 nt si l’un ou l’autre co-migre avec le produit, car il est difficile d’éliminer ces colorants de l’oligonucléotide élué sans précipitation d’éthanol. - Lorsque l’électrophorèse sur gel est terminée, retirez la plaque de gel du support de gel, démontez la plaque de gel et enveloppez le gel dans un film de polychlorure de vinyle. Identifiez les bandes de produit en ombrage arrière avec une lumière UV de 254 nm et éliminez la bande de produit principale à l’aide d’une lame de rasoir.
- Extraire l’oligonucléotide du gel excisé par la méthode d’écrasement et de trempage42.
- Écrasez le gel excisé en l’extrudant à travers une seringue en plastique ou mécaniquement.
- Pour les oligonucléotides de plus de 15 nt:
- Éluer dans 3x volumes de tampon d’écrasement et de trempage (300 mM NaCl; 10 mM Tris, pH 8; 1 mM EDTA) pendant au moins 12 h avec agitation ou agitation.
- Retirer les morceaux de gel solides en faisant passer la solution à travers une seringue munie d’un filtre de 0,2 μm et concentrer l’oligonucléotide par précipitation d’éthanol.
- Pour les oligonucléotides inférieurs à 15 nt:
- Éluez dans 3x volumes d’eau sans nucléase pendant au moins 12 h avec agitation ou agitation.
- Retirer les morceaux de gel solides en faisant passer la solution à travers une seringue munie d’un filtre de 0,2 μm et concentrer par lyophilisation.
- Enlever les sels résiduels et les solutés à l’aide d’une colonne d’exclusion de taille jetable comme à l’étape 5.6. Concentré par lyophilisation.
- Conserver les oligonucléotides triphosphorylés purifiés à -20 °C dans un tampon TE (10 mM Tris, pH 8 ; 1 mM EDTA) ou dans un tampon de stockage d’oligonucléotides similaire.
- Déterminer la concentration de l’oligonucléotide en mesurant l’absorbance à 260 nm à l’aide d’un spectrophotomètre UV-Vis.
NOTE: Le coefficient d’extinction estimé de l’oligonucléotide ne doit pas être affecté par son état de phosphorylation 5′ et peut être calculé à partir de sa séquence à l’aide d’un calculateur de coefficient d’extinction d’oligonucléotide. - Vérifier la triphosphorylation par spectrométrie de masse. Recherchez une masse attendue de +239,94 Da par rapport à celle de l’oligonucléotide 5′-hydroxyle.
REMARQUE: La désorption/ionisation laser assistée par matrice ou la spectrométrie de masse par ionisation par électropulvérisation (MALDI-MS ou ESI-MS, respectivement) conviennent à l’identification de l’état de triphosphorylation de l’oligonucléotide lors de l’utilisation de protocoles optimisés pour les acides nucléiques. ESI-MS fournit toutefois des résultats plus cohérents, en raison de taux plus faibles de fragmentation ionique; un service commercial représentatif est fourni dans la Table des matériaux.
- Déterminer la concentration de l’oligonucléotide en mesurant l’absorbance à 260 nm à l’aide d’un spectrophotomètre UV-Vis.
6. Oligonucléotides triphosphorylés comme substrats pour l’auto-réplication du ribozyme
ATTENTION : Le 32P est un isotope radioactif et les étapes suivantes doivent être effectuées à l’aide de protocoles de sûreté normalisés pour travailler avec des matières radioactives en laboratoire et par un chercheur certifié pour l’utilisation de matières radioactives par les services de santé et de sécurité environnementales concernés. Comme alternative, le substrat de ribozyme A auto-réplicant peut être préparé synthétiquement avec une étiquette 5′-fluorescéine14 et imagé par fluorescence, comme à l’étape 7.9.
- Préparer la solution A sous forme de mélange de ribozyme E auto-réplicateur et de substrat d’ARN A14marqué 5′-32 P à des concentrations de 0,30 μM et 30 μM, respectivement. Préparer des solutions B-transcrites et B-synthétiques avec 15 μM de substrat d’ARN triphosphorylé B, préparées par transcription in vitro 14 ou triphosphorylation chimique comme ci-dessus, respectivement, dans un tampon EPPS de 75 mM, pH 8,5 et 37,5 MgCl2. Porter les deux solutions à 42 °C.
REMARQUE: Tous les composants de l’ARN sont répertoriés dans le tableau des matériaux. - Pour initier l’auto-réplication, mélanger rapidement 5 μL de solution A avec 10 μL de solution B-transcrite ou B-synthétique à une concentration finale de 0,1 μM E, 10 μM A, 10 μM B, 25 mM MgCl2 et 50 mM de tampon EPPS, pH 8,5. Incuber le mélange réactionnel à 42 °C.
- À intervalles réguliers, prendre des aliquotes de 0,5 μL et tremper dans 9,5 μL de tampon de chargement de gel de formamide (95 % de formamide ; 10 mM d’EDTA, pH 8).
- Préparer un gel analytique de polyacrylamide/8 M d’urée/1x TBE, selon les protocoles appropriés pour la plaque de gel et le support. Assemblez le gel coulé et la plaque dans un support de gel, remplissez les réservoirs avec 1x TBE et pré-exécutez à 40 W (ou selon le cas pour différentes plaques et supports de gel) pendant 30 min.
- Chauffer les échantillons de réaction trempés à 80 °C, charger 5 μL de l’échantillon dans chaque puits et faire fonctionner le gel à 40 W pendant environ 40 min (ou selon le cas pour différentes plaques et supports de gel).
- Retirez la plaque de gel du support de gel, démontez et enveloppez le gel dans un film de polychlorure de vinyle. Couvrir le gel avec un écran au phosphore et exposer pendant 1 h (ou selon le cas pour 32P cpm); scannez l’écran à l’aide d’un scanner de gel fluorescent/phosphorescent.
- Quantifiez le rendement de la réaction à l’aide d’un logiciel de quantification d’images en gel.
- Ouvrez l’image du gel à l’aide de la boîte à outils d’analyse, choisissez Analyse | Définition de la forme, sélectionnez Zones | la forme rectangulaire et dessinez des rectangles de la même taille autour des bandes correspondant à A et au produit E non réagis pour chaque fois et les deux réactions.
- Choisissez analysis | Soustraction d’arrière-plan, sélectionnez Zones | la forme rectangulaire et dessinez un rectangle de la même taille dans une partie vide de l’image gel. Remplacez la méthode de soustraction d’arrière-plan pour tous les rectangles par Rectangle d’image.
- Choisissez Fenêtre | 2 Fenêtre de zone, puis Modifier | Exportez vers Excel pour exporter les volumes de pixels de bande quantifiés vers un fichier de feuille de calcul.
- Tracez la concentration du produit E par rapport au temps et adaptez les données à la croissance logistique Eq (1) à l’aide d’un logiciel d’ajustement des données statistiques :
[E] =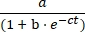 (1)
(1)
Où a est l’étendue maximale de la réaction, b est le degré de sigmoidicité et c est le taux de croissance exponentiel.- Dans la feuille de calcul exportée, divisez le volume du produit E par la somme des volumes du substrat A et du produit E pour déterminer le rendement fractionnaire du produit pour chaque fois et les deux réactions. Multipliez par la concentration initiale du substrat A (10 μM) pour déterminer le rendement du produit E en fonction du temps.
- Dans le logiciel d’ajustement des données statistiques, choisissez Fichier | Nouvelle | Nouveau fichier de projet, sélectionnez XY sous Nouveau tableau et graphique, puis cliquez sur Créer. Entrez les temps de réaction et les rendements du produit E pour les deux réactions dans les colonnes adjacentes, et étiquetez les colonnes en conséquence (par exemple, « temps », « B transcrit », « B synthétique »).
- Choisissez Insérer | Nouvelle analyse, certaines analyses XY | Régression non linéaire (ajustement de la courbe), puis cliquez sur OK. Choisissez courbes de croissance | Croissance logistique et cliquez sur OK; n’ajustez aucun autre paramètre. Observez les paramètres d’ajustement et les intervalles de confiance sous Résultats et le tracé des points de données et des courbes ajustées sous Graphiques.
7. Copie chirale croisée de l’ARN-L
- Préparer 10 μL de solution d’ARN contenant 20 μM d’ARN-D 27,3 t de polymérase chirale croisée, 2 μM d’amorce D-ARN marquée à la fluorescéine, 4 μM de gabarit de tête de marteau biotinylé L-ARN et 20 μM chacun d’ARN-L pppCUG, pppAUG, pppAGG et pppCGC, dans 10 μL de 50 mM Tris, pH 8,3. Recuit l’ARN en le chauffant à 90 °C pendant 1 min et en le refroidissant à 23 °C à 0,2 °C/s dans un thermocycleur PCR. Consultez le tableau des matériaux pour plus de détails sur la polymérase, l’apprêt et le modèle.
- Incuber la solution d’ARN à 17 °C et commencer la réaction en ajoutant 10 μL de tampon de démarrage 2x (400 mM MgCl2, 500 mM NaCl et 50 mM Tris, pH 8,3). S’assurer que les concentrations finales de tous les composants de la réaction sont de 10 μM de polymérase, 1 amorce de μM, 2 μM de gabarit, 10 μM de chaque trinucléotide 5′-triphosphate, 200 mM deMgCl2, 250 mM de NaCl et 50 mM de Tris, pH 8,3.
- Au fur et à mesure que la réaction progresse, prendre des aliquotes de 10 μL et tremper avec 5 μL de 0,5 M EDTA, pH 8.
- À chaque échantillon de réaction trempé, ajouter 0,1 mg de billes magnétiques recouvertes de streptavidine (capacité de liaison biotine-oligonucléotide de 20 pmol) en suspension dans 10 μL de 1 M De NaCl dans un tampon TE avec un détergent neutre à 0,05 % pour capturer les amorces prolongées par le trimère via le gabarit biotinylé et incuber pendant 30 min à température ambiante en agitant.
- Capturer les billes sur un aimant de capture de perles, retirer et jeter le liquide, et ajouter 50 μL de solution de lavage (250 mM de NaCl dans TE avec 0,05% de détergent neutre). Mélangez les perles, capturez-les à nouveau et retirez la solution de lavage. Répétez une fois de plus.
- Pour éluer les apprêts étendus des billes, ajoutez 50 μL de solution d’élution (25 mM de NaOH avec 0,05% de détergent neutre) et mélangez les perles. Recapturez les billes, retirez la solution d’élution, trempez avec 100 mM de Tris (pH 7,5) et précipitez avec de l’éthanol.
- Préparez un gel analytique de polyacrylamide/8 M d’urée/1x TBE, selon les protocoles appropriés pour la plaque de gel et le support. Assemblez le gel coulé et la plaque dans un support de gel, remplissez les réservoirs avec 1x TBE et pré-exécutez à 40 W (ou selon le cas pour différentes plaques et supports de gel) pendant 30 min.
- Dissoudre l’ARN précipité dans 10 μL de tampon de charge de gel de formamide et préparer un apprêt marqué à l’extrémité non réagi à 0,5 μM dans un tampon de chargement de gel de formamide. Chauffer les échantillons à 80 °C, charger 5 μL de l’échantillon dans chaque puits et faire fonctionner le gel à 40 W pendant environ 40 min (ou selon le cas pour différentes plaques et supports de gel).
- Retirez la plaque de gel du support et scannez à l’aide d’un scanner de gel fluorescent / phosphorescent pour visualiser les produits d’extension de l’ARN L chiral croisé.
Résultats
Les oligonucléotides doivent être synthétisés à l’aide de protocoles standard appropriés aux phosphoramidites et au synthétiseur automatisé ADN/ARN, en laissant l’oligonucléotide produit détaché du support solide dans la colonne de synthèse plastique d’origine, avec le groupe diméthoxytrityle 5ʹ-terminal retiré pour donner le 5ʹ-hydroxyle libre (section 1). Tous les oligonucléotides utilisés dans cette démonstration ont été préparés à l’aide de résine de verre à pores contrôlés (CPG) de 1 000 Å comme support solide et conduits à l’échelle de 0,2 ou 1 μmole. Des exemples représentatifs de colonnes de synthétiseur, de résines, de réactifs et de phosphoramidites sont fournis dans la Table des matériaux. Pour les réactions à plus grande échelle, les volumes et les temps utilisés dans les étapes suivantes peuvent devoir être ajustés.
La réaction de triphosphorylation est menée sur colonne dans une chambre de réaction construite sur mesure (figure 2, section 3) à l’aide de composants standard disponibles dans le commerce énumérés dans la table des matériaux et suit le schéma illustré à la figure 1 (section 4) 28. Il est essentiel que les conditions soient maintenues strictement anhydres pendant la triphosphorylation et que tous les solvants et réactifs soient préparés à l’avance sur des tamis moléculaires et laissés sécher complètement avant utilisation (rubrique 2). La triphosphorylation prend généralement 2 heures pour se produire, et par la suite, la colonne lavée et séchée peut être traitée selon les procédures standard de déprotection et de purification des oligonucléotides (rubrique 5).
Après déprotection, les oligonucléotides triphosphates sont purifiés par électrophorèse sur gel de polyacrylamide (PAGE) dénaturante, montrant une seule bande de produit majeure par ombrage UV qui peut être excisé et élué du gel. Le produit 5′-triphosphate est facilement séparé des produits secondaires de réaction pour les oligonucléotides courts, comme le montrent les trinucléotides d’ADN 5ʹ-triphosphates, pppAAA et pppCCC, et le trinucléotide D-ARN L 5ʹ-triphosphate pppGAA dans la figure 3A,B. Les produits 5′-hydroxyle et 5′-triphosphate pour les trimères d’ADN AAA et CCC ont été excisés et identifiés par spectrométrie de masse et marqués en conséquence à la figure 3A. Les bandes supplémentaires, telles que visibles pour le trimère d’ADN AAA, ne contiennent généralement pas assez de matière pour récupérer et identifier. La présence de ces bandes, cependant, est en corrélation avec des masses de produits supplémentaires dans les produits de réaction non purifiés (Figure 3C), représentant généralement des produits secondaires de 5′-diphosphate, de monophosphate et de H-phosphonate, comme indiqué ci-dessous.
Après la purification de PAGE, des oligonucléotides plus gros peuvent être élués à l’aide de la méthode de broyage et de trempage42 et de la précipitation ultérieure de l’éthanol. Toutefois, les oligonucléotides de moins de 15 nt ne peuvent pas être précipités efficacement à l’éthanol et nécessitent donc une procédure modifiée pour l’élution du gel (étape 5.11.3). La colonne d’exclusion de la taille jetable indiquée dans le tableau des matériaux n’est évaluée que pour une utilisation avec des oligonucléotides de plus de 10 nt. Cependant, nous avons constaté que des oligonucléotides aussi courts que des trimères peuvent être effectivement dessalés en utilisant le protocole recommandé par le fabricant. Néanmoins, lors du dessalement d’oligonucléotides courts (comme aux étapes 5.6 et 5.11.3), l’éluat de colonne soit collecté en fractions et que les fractions de produit soient identifiées par absorbance à 260 nm à l’aide d’un spectrophotomètre UV-Vis. Une colonne d’exclusion de taille optimisée pour les oligonucléotides plus courts est fournie dans le tableau des matériaux comme choix alternatif. Le rendement final de la synthèse d’oligonucléotides à l’échelle de 1 μmole après purification est de 50 à 300 nmol.
La triphosphorylation peut être confirmée par spectrométrie de masse, où le produit triphosphorylé a une masse +239,94 Da supérieure à l’oligonucléotide 5′-hydroxyle, bien que la présence de matériaux correspondant au 5′-di- et au monophosphate (+159,96 et +79,98 Da, respectivement) soit souvent observée. Un produit secondaire de 5′-H-phosphonate avec une masse de +63,98 Da de la masse de 5′-OH peut également être observé, et des niveaux élevés de ce produit indiquent que les conditions pendant la triphosphorylation n’étaient pas suffisamment anhydres. Avant la purification, les oligonucléotides déprotégés montreront généralement tous ces produits (Figure 3C), tandis que le matériau purifié montrera un pic correspondant au produit 5′-triphosphate avec les 5′-di- et les monophosphates (Figure 3D,E).
La spectrométrie de masse seule ne donnera généralement pas une mesure rigoureuse de la pureté du 5′-triphosphate en raison des taux différentiels d’ionisation et de fragmentation du triphosphate pendant l’ionisation. Pour mesurer la pureté du produit final, la chromatographie liquide en phase inverse et le tandem ESI-MS (RP-LC/ESI-MS) sont recommandés, en particulier pour les oligonucléotides plus longs. L’analyse des ARN-D 5ʹ-triphosphates pppACGAGG et pppGAGACCGCAACUUA par RP-LC/ESI-MS (Figure 4A,B, respectivement) montre une pureté typique du produit final, contenant 20% de 5ʹ-diphosphate car ces deux espèces sont difficiles à séparer lorsqu’elles sont présentes sur des oligonucléotides plus longs.
Les oligonucléotides synthétiques de 5′-triphosphate fonctionnent généralement aussi bien ou mieux que les matériaux préparés enzymatiquement dans les études biochimiques. Dans la section 6, à titre d’exemple, des substrats d’ARN 5′-triphosphate 14 nt préparés synthétiquement ou par transcription in vitro ont été comparés dans une réaction d’auto-réplication catalysée par l’ARN 14,15,43,44,45. Le ribozyme E catalyse l’assemblage des substrats A et B pour produire une nouvelle copie de E dans une réaction autocatalytique capable de croissance exponentielle (Figure 5A). Les composants E et 32composants A marqués P ont été préparés par transcription in vitro, et le substrat B triphosphorylé a été préparé soit synthétiquement, comme décrit ci-dessus, soit par transcription in vitro 14. La progression de la réaction d’auto-réplication a été surveillée en prélevant des échantillons périodiques qui ont été analysés par dénaturation de PAGE et quantifiés à l’aide d’un scanner de gel fluorescent / phosphorescent. Les données résultantes, adaptées à une fonction de croissance logistique, ont révélé que le substrat B transcrit ou synthétique supporte une croissance exponentielle, mais que le B synthétique donne une quantité légèrement supérieure de produit (Figure 5B). Ce résultat peut refléter l’hétérogénéité de composition à l’extrémité 5′ de l’ARN préparé par transcription in vitro 23,24.
La triphosphorylation chimique permet également la synthèse d’oligonucléotides triphosphates qui ne peuvent être préparés biologiquement, que ce soit in vitro ou dans les cellules. Dans la section 7, des oligonucléotides triphosphates non biologiques composés d’ARN-L, l’énantiomère de l’ARN-D naturel, préparés comme dans les sections 1 à 5, ont été utilisés comme substrats pour le ribozyme 27.3t de polymérase « chirale croisée » de l’ARN D (Figure 6A), qui catalyse la polymérisation dirigée par gabarit d’un produit d’ARN L plus long à partir d’oligonucléotides d’ARN L court 5′-triphosphates de manière générale. À titre d’exemple, le ribozyme peut synthétiser une version L-ARN du motif d’auto-clivage de la tête de marteau (Figure 6B)18. Les trinucléotides triphosphates d’ARN L purifiés ont été combinés avec un amorce d’ARN L marqué à la fluorescéine et un gabarit d’ARN-L (figure 6C) et ont réagi avec la ligase chirale croisée. Les échantillons au cours de la réaction ont été analysés par PAGE et imagés à l’aide d’un scanner de gel fluorescent/phosphorescent pour démontrer la synthèse d’une version L-ARN du ribozyme à tête de marteau codée par le gabarit (Figure 6D).
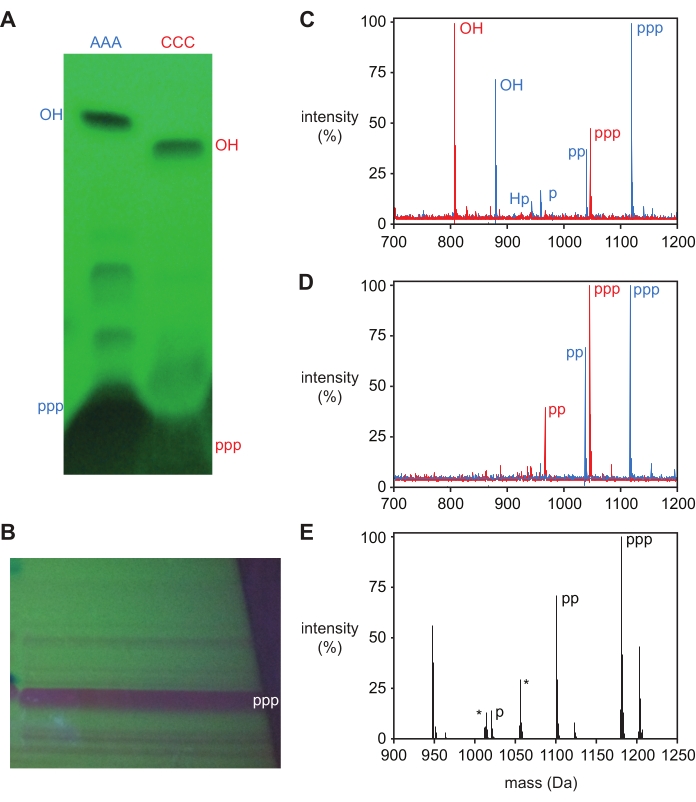
Figure 3 : Purification des trinucléotides 5ʹ-triphosphates. (A) Analyse PAGE (visualisée par ombrage UV) de la triphosphorylation des trinucléotides d’ADN tri-désoxyadénosine (AAA, bleu) et tri-désoxycytidine (CCC, rouge), intentionnellement surchargés pour visualiser des produits secondaires mineurs. Le produit 5ʹ-triphosphate (ppp) et la matière première du 5ʹ-hydroxyle (OH) ont été excisés et identifiés par MALDI-MS. (B) PAGE préparative de triphosphorylation du trinucléotide GAA de l’ARN-L, avec la bande de produits majeure excisée et identifiée comme le 5ʹ-triphosphate (ppp) par ESI-MS. (C) MALDI-MS de produits de réaction bruts après déprotection et (D) produits purifiés de (A). 5ʹ-triphosphate (ppp; pppAAA attendu 1 119 Da, observé 1 118 Da; pppCCC attendu 1 047 Da, observé 1 046); Le 5ʹ-diphosphate (pp), le 5ʹ-monophosphate (p), le 5ʹ-hydroxyle (OH) et le 5ʹ-H-phosphonate (Hp) sont étiquetés. (E) Spectre de masse déconvolu provenant de l’injection directe d’ESI-MS de produit isolé à base de 5ʹ-triphosphate de (B), avec des pics identifiés marqués (attendu 1 181,6 Da, observé 1 181,0 Da). Des produits à base de 5ʹ-diphosphate (pp) sont également observés, de même que des pics d’ions sodium pour les produits tri- et di-phosphate (+22 Da). Les pics de contaminants courants sont marqués d’un astérisque. Pour faciliter la comparaison, les spectres de masse ont été normalisés à l’intensité la plus élevée mesurée dans chaque spectre et sont rapportés en pourcentage par rapport à cette valeur. Abréviations : PAGE = électrophorèse sur gel de polyacrylamide; MALDI-MS = désorption/ionisation laser assistée par matrice; ESI-MS = spectrométrie de masse par ionisation par électropulvérisation. Veuillez cliquer ici pour voir une version agrandie de cette figure.
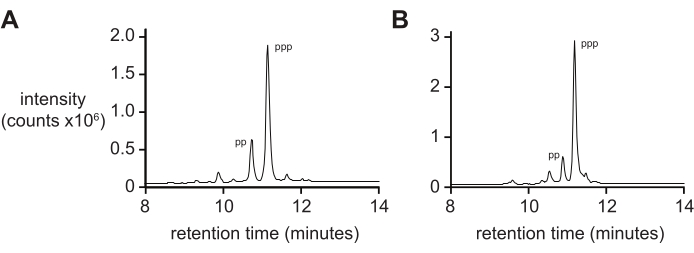
Figure 4 : RP-LC analytique de 6 nt et 14 nt D-ARN oligonucléotides triphosphates. (A) 5ʹ-pppACGAGG-3ʹ et (B) 5ʹ-pppGAGACCGCAACUUA-3ʹ. Tandem ESI-MS a identifié le pic majeur des deux (~70%) comme le 5ʹ-triphosphate (ppp), avec des quantités moindres de 5ʹ-diphosphate (pp). Abréviations : RP-LC = chromatographie liquide en phase inverse ; nt = nucléotides; ESI-MS = spectrométrie de masse par ionisation par électropulvérisation. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5 : Comparaison des substrats oligonucléotidiques 5ʹ-triphosphate préparés par synthèse chimique ou transcription in vitro. (A) Le ribozyme E auto-réplicant ligate l’ARN A et l’ARN B 5′-triphosphorylé. (B) Comparaison des réactions d’auto-réplication utilisant 10 μM A et 10 μM B, synthétiques (cercles ouverts) ou transcrits in vitro (cercles remplis). (B) Les données étaient adaptées à l’équation de croissance logistique: [E] = a / (1 + b e-ct), où a est le rendement final, b est le degré de sigmoïdité et c est le taux de croissance exponentiel. Les taux de croissance pour les deux réactions étaient identiques, à 1,14 h-1, tandis que l’étendue finale était 10% plus élevée pour les réactions avec B synthétique. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 6 : Polymérisation croisée de l’ARN-L chiral avec un ribozyme. (A) Le ribozyme de la polymérase D-ARN 27,3 t, qui catalyse la ligature de l’ARN-L dépendante du modèle. (B) Le produit L-ARN synthétisé par 27,3 t fait partie d’un motif d’endonucléase à tête de marteau. (C) Polymérisation de l’ARN-L catalysée par 27,3 t à l’aide d’un gabarit d’ARN L biotinylé (brun), d’un amorce d’ARN-L marqué en fin de compte (magenta) et de quatre trinucléotides trinucléotidiques d’ARN-L (cyan), préparés synthétiquement. (D) Analyse PAGE des produits d’extension de (B) à 4 h et 24 h, montrant chaque incorporation de trinucléotides jusqu’au produit pleine longueur (point noir). L’amorce d’ARN-L non réagie est incluse comme marqueur de référence. Abréviations : PAGE = électrophorèse sur gel de polyacrylamide; M = marqueur de référence. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
La procédure de triphosphorylation décrite ici est largement compatible avec la synthèse d’oligonucléotides en utilisant la chimie standard du phosphoramidite. Les phosphoramidites nucléosidiques devraient avoir des groupes protecteurs base-labile compatibles avec une déprotection rapide dans AMA39, y compris les groupes standard β-cyanoéthyle sur le phosphite, et isobutyryl, diméthylformamidyle, acétyle, phénoxyacétyle ou 4-isopropylphénoxyacétyle sur les amines exocycliques des nucléobases. Les groupes ribose 2'-hydroxyle doivent être protégés par des groupes protecteurs du silyle, soit le t-butyldiméthylsilyle (TBDMS), soit le tri-iso-propylsilyloxyméthyl (TOM)40,41. Le groupe pivaloyloxyméthyle base-labile (PivOM) a également été signalé comme étant compatible avec la triphosphorylation chimique30.
Plusieurs méthodes ont été décrites pour la triphosphorylation chimique des oligonucléotidessynthétiques 28,29,30,31,32,33,34,35. Nous avons constaté que la triphosphorylation utilisant le réactif Ludwig-Eckstein37 était l’une des plus accessibles, ne nécessitant aucune synthèse spécialisée de réactifs et aucun équipement spécialisé. Les oligonucléotides 5′-triphosphates préparés par cette méthode ont été couramment utilisés comme substrats pour les ribozymes d’ARN ligase, y compris l’utilisation de triphosphates d’oligonucléotides d’ARN-L enzymatiquement inaccessibles pour obtenir une synthèse et une réplication dépendantes du modèle de cet acide nucléique « image miroir » 14,16,17,18 . La méthode convient également à la préparation de petits ARN de la boucle de tige 5′-triphosphorylés qui sont de puissants activateurs de la réponse immunitaire innée chez les vertébrés 6,7.
Le réactif ludwig-Eckstein, la salicylphosphoochloridite37, est très réactif à l’eau et récupère efficacement toute eau introduite lors de la dissolution du réactif avant le chargement sur la colonne oligonucléotidique. Après ce point, cependant, l’oligonucléotide 5′-phosphitylé réagira préférentiellement avec toute eau introduite dans le système par rapport au pyrophosphate, formant un produit secondaire 5′-H-phosphonate après 28,37,38. Une préparation minutieuse des réactifs de triphosphorylation et de la chambre de réaction de triphosphorylation garantit que ce produit secondaire n’est pas formé. Pour le séchage au solvant, les tamis moléculaires de type 4 Å sont vendus préemballés dans des sacs en téflon compatibles avec la plupart des solvants organiques sous diverses marques par la plupart des sociétés de réactifs de synthèse d’oligonucléotides. Des précautions supplémentaires, telles que la triphosphorylation dans une boîte à gants sous atmosphère anhydre, ne sont généralement pas nécessaires.
La réaction de l’oligonucléotide 5′-phosphitylé avec le TBAP forme un intermédiaire cyclique 5′-trimétaphosphite, qui est ensuite oxydé en 5′-trimétaphosphate cyclique à l’aide d’une solution oxydante de synthèse d’oligonucléotides (iode dans l’eau/pyridine/THF). Il convient de noter que les solutions comburantes commerciales utilisent des quantités variables d’iode et qu’il est essentiel d’utiliser la concentration élevée d’iode de 0,1 M pour assurer une oxydation complète du triphosphate. Le produit cyclique est hydrolysé jusqu’au 5′-triphosphate linéaire final dans la même solution37, et des solutions oxydantes anhydres alternatives doivent être utilisées si une linéarisation avec des nucléophiles autres que l’eau est souhaitée (voir ci-dessous pour les applications)33. Cependant, tout trimétaphosphate cyclique résiduel sera linéarisé lors de la déprotection alcaline ultérieure de l’oligonucléotide. L’hydrolyse du 5′-trimétaphosphate cyclique ne donne que le triphosphate37,46 linéaire plutôt que ramifié.
La déprotection des oligonucléotides n’a généralement pas besoin d’être modifiée pour s’adapter à la triphosphorylation 5′, mais quelques précautions doivent être prises. Le triphosphate est relativement stable à une brève exposition à des conditions alcalines, mais il faut veiller à ne pas exposer le triphosphate à l’AMA plus longtemps que nécessaire. Il faut éviter de protéger les groupes qui nécessitent un traitement plus prolongé dans l’ammoniac ou l’AMA pendant plus de 10 minutes à 65 °C. Des traitements plus doux, tels que 2 h dans l’ammoniac à température ambiante, sont acceptables lorsqu’ils sont compatibles avec d’autres groupes protecteurs au phosphoramidite. Une méthode courante et rapide de déprotection des oligonucléotides d’ARN synthétiques protégés par le silyl utilise du trifluorure de triéthylamine et une température élevée47; cependant, cela doit être évité lors de la préparation de l’ARN 5′-triphosphates, car les conditions acides prolongées accélèrent l’hydrolyse du triphosphate31,32.
Preparative PAGE s’est avéré être la méthode la plus simple et la plus fiable pour la purification post-déprotection des oligonucléotides 5′-triphosphorylés (Figure 3 et Figure 4). Cependant, la CLHP préparative en phase inverse peut également être utilisée pour purifier les produits triphosphorylés. La présence de produits 5′-diphosphate et, dans une moindre mesure, de 5′-monophosphate est systématiquement observée lors de la vérification de la triphosphorylation par spectrométrie de masse. Nous avons observé une fragmentation du 5′-triphosphate au cours de la spectrométrie de masse à partir d’un matériau très pur préparé par synthèse chimique ou transcription, en particulier si l’instrument n’est pas optimisé pour l’analyse des oligonucléotides. Néanmoins, l’analyse RP-LC montre souvent que 10 % à 20 % du produit secondaire 5′-diphosphate est présent dans des oligonucléotides 5′-triphosphorylés plus longs (Figure 4). Les préparations commerciales de pyrophosphate de tributylammonium peuvent être contaminées par jusqu’à 20% de monophosphate, ce qui donnera du 5′-diphosphate comme produit secondaire pendant la triphosphorylation30,31. Une préparation minutieuse de ce réactif en interne peut donner des stocks TBAP beaucoup plus purs31. Cependant, nous avons constaté que les oligonucléotides triphosphorylés à l’aide de sources commerciales de TBAP présentent toujours une réactivité comparable ou supérieure lorsqu’ils sont utilisés comme substrats dans des réactions enzymatiques (Figure 5B), par rapport au matériel préparé par transcription in vitro.
Une autre utilisation notable de la triphosphorylation oligonucléotidique avec le réactif ludwig-Eckstein tire parti du trimétaphosphite cyclique intermédiaire33. Si l’étape d’oxydation suivante est effectuée avec du peroxyde de t-butyle de 1 M dans les hexanes, qui est souvent utilisé pour l’oxydation des oligonucléotides dans des conditions anhydres, l’oxydation du phosphite se produit sans hydrolyse d’ouverture de cycle, donnant le trimétaphosphate cyclique. Cet intermédiaire peut ensuite être réagi avec des nucléophiles primaires d’amine ou d’alcool pour donner des 5′-triphosphates avec des modifications au γ-phosphate. Ces modifications comprennent l’ajout d’une étiquette lipophile liée par une liaison phosphoramidate, qui facilite la purification rapide spécifique du triphosphate par RP-LC, suivie d’une hydrolyse acide de la balise du triphosphate33. Des modifications fluorescentes à la position γ-phosphate peuvent également être introduites pour être utilisées comme rapporteurs fluorescents en temps réel pour les réactions de ligature catalysées par le ribozyme15,33.
Déclarations de divulgation
Les auteurs déclarent qu’ils n’ont pas d’intérêts financiers concurrents.
Remerciements
Les auteurs sont reconnaissants à Greg Springsteen, Natasha Paul, Charles Olea, Jr., Jonathan Sczepanski et Katrina Tjhung pour leurs discussions utiles sur les meilleures pratiques en matière de réactions chimiques de triphosphorylation et à Gerald Joyce pour leurs commentaires utiles. Ce travail a été soutenu par une subvention MCB 2114588 de la National Science Foundation.
matériels
| Name | Company | Catalog Number | Comments |
| 0.22 µm polyethersulfone syringe filter | MilliporeSigma | SLMP025SS | Syringe filter for removing crushed polyacrylamide gel particles (Section 5) |
| 0.22 µm PTFE syringe filter | MilliporeSigma | SLLG013SL | Syringe filter for removing CPG resin (Section 5) |
| 1 mL plastic syringes | ThermoFisher Scientific | 14-823-434 (BD 309659) | Components of triphosphorylation apparatus (sections 2–4) |
| 1,4-Dioxane, anhydrous | MilliporeSigma | 296309 | Triphosphorylation solvent (sections 2–4) |
| 2-Chloro-4H-1,3,2-benzodioxaphosphorin-4-one, Salicyl Phosphorochloridite (SalPCl) | MilliporeSigma | 324124 | Triphosphorylation reagent (sections 2–4) |
| 30 mL glass bottles | MilliporeSigma | 23232 | Bottles for preparing triphosphorylation solvents and TBAP solution (section 2) |
| 3-way Stopcock, polycarbonate/polypropylene | Bio-Rad Laboratories | 7328103 | Component of triphosphorylation apparatus (sections 2–4) |
| 40% acrylamide/bis-acrylamide solution, 19:1 | Bio-Rad Laboratories | 1610144 | For PAGE (sections 5–7) |
| Acetonitrile, anhydrous, 100 mL | Glen Research | 40-4050-50 | Triphosphorylation solvent (sections 2–4) |
| Ammonia-neutralizing Trap | ThermoFisher Scientific | ANT100 and ANS121 | For use with Speedvac DNA130 (section 5) |
| Ammonium persulfate (APS) | Bio-Rad Laboratories | 1610700 | For PAGE, catalyst for acrylamide polymerization (sections 5–7) |
| Aqueous ammonia, 28% | MilliporeSigma | 338818 | For preparing AMA deprotection reagent (section 5) |
| Aqueous methylamine, 40% | TCI America | TCI-M0137 | For preparing AMA deprotection reagent (section 5) |
| Automated DNA/RNA oligonucleotide synthesizer | PerSeptive Biosystems | Expedite 8909 DNA/RNA Synthesizer | any column-based synthesizer is acceptable (section 1) |
| Bead-capture magnet | ThermoFisher Scientific | 12320D | For streptavidin bead capture (section 7) |
| Bromophenol blue | Bio-Rad Laboratories | 1610404 | For PAGE urea loading buffer (section 5) |
| Deep vacuum oil pump | ThermoFisher Scientific | VLP200-115 | For use with lyophilizer (section 5) |
| Drierite dessicant, 10-20 mesh | MilliporeSigma | 737828 | Desiccant for storing triphosphorylation chemicals and equipment (sections 1–2) |
| D-RNA 27.3t cross-chiral polymerase | prepared in house18 | 5′-GGUGGUGGAC GUGAUCAUUA CGGAUCACUA ACUCGUCAGU GCAUUGAGAA GGAGAAUAAA AUGCACAUAG GUCGAAAGAC CUUAUACAAG AACUGUAUCA CCGGAGGGCG AGCACCACC-3′ | For cross-chiral ribozyme reactions (section 7) |
| D-RNA CPG solid supports, 1,000Å, prepackaged 1 µmole synthesis columns | Glen Research | 20-3404-41E, 20-3415-41E, 20-3424-41E, 20-3430-41E | representative, for D-RNA oligonucleotide synthesis (section 1) |
| D-RNA TOM-protected phosphoramidites | ChemGenes | ANP-3201, 3202, 3203, 3205 | representative, for D-RNA oligonucleotide synthesis (section 1) |
| Empty Expedite Synthesis Columns, 1µm | Glen Research | 20-0021-01 | Synthesis columns for use with Expedite DNA/RNA synthesizer (section 1) |
| EPPS, N-(2-Hydroxyethyl)piperazine-N′-(3-propanesulfonic acid), solid | MilliporeSigma | E1894 | Ribozyme reaction buffer component (section 6) |
| Ethylenediaminetetraacetic acid (EDTA), solid | MilliporeSigma | EDS | Divalent metal ion chelator for use in various buffers (sections 5–7) |
| Filters for Expedite synthesis columns | Glen Research | 20-0021-0F | Expedite-style synthesis column filters, for use with empty synthesis columns (section 1) |
| Fluorescent/phosphorescent gel scanner | Cytiva | Amersham Typhoon RGB, 29187193 | For visualizing analytical PAGE (sections 6–7) |
| Formamide, deionized | VWR Life Science | 97062 | For PAGE formamide gel loading buffer (sections 6–7) |
| Gel image quantitation software | Cytiva | ImageQuant TL | For quantifying scanned gel images (section 6) |
| Glass desiccator | MilliporeSigma | CLS3121150 | Triphosphorylation solvent storage (section 2) |
| L-RNA CPG solid supports, 1,000Å, bulk | ChemGenes | N-4691-10, N-4692-10, N-4693-10, N-4694-10 | L-RNA oligonucleotide synthesis (section 1) |
| L-RNA hammerhead template | prepared in house18 | 5′-GCGCCUCAUC AGUCGAGCC-3′ | For cross-chiral ribozyme reactions (section 7) |
| L-RNA primer | prepared in house18 | 5′-fluorescein-GGCUCGA-3′ | For cross-chiral ribozyme reactions (section 7) |
| L-RNA TOM-protected phosphoramidites | ChemGenes | OP ANP-5201, 5202, 5203, 5205 | L-RNA oligonucleotide synthesis (section 1) |
| Lyophilizer/Freeze Dryer | VirTis | Benchtop K | For concentrating oligonucleotides (section 5) |
| Magnesium Chloride Hexahydrate, solid | MilliporeSigma | M2670 | For ribozyme reactions (sections 6–7) |
| N,N-Dimethylformamide, anhydrous | MilliporeSigma | 227056 | Triphosphorylation solvent (section 2) |
| NAP-25 Desalting column (Sephadex G-25 resin) | ThermoFisher Scientific | 45000150 | Disposable gravity-flow size exclusion chromatography columns containing Sephadex G-25 resin (section 5) |
| Non-coring stainless steel needle, 20 G | ThermoFisher Scientific | 14-815-410 | Needles for piercing rubber septa (sections 2–4) |
| Oligonucleotide extinction coefficient calculator | Integrated DNA Technologies | OligoAnalyzer Tool | Nearest-Neighbor Model Short Oligonucleotide 260nm extinction coefficient calculator (section 5) |
| Oxidizer solution, 0.1 M Iodine in THF/pyridine/water | ChemGenes | RN-1456 | Triphosphorylation reagent (section 4) |
| PAGE plates | Timberrock/CBS | NGP-250-BO and NO | For PAGE (sections 5–7) |
| PAGE power supply | Bio-Rad Laboratories | PowerPac HV 1645056 | For PAGE (sections 5–7) |
| PAGE spacers and combs (analytical) | Timberrock/CBS | VGS-0725 and VGC-0714 | For PAGE (sections 6–7) |
| PAGE spacers and combs (preparative) | Timberrock/CBS | VGS-3025R and VGC-3001 | For PAGE (section 5) |
| PAGE stand | Timberrock/CBS | ASG-250 | For PAGE (sections 5–7) |
| Parafilm M | ThermoFisher Scientific | 13-374-12 (Bemis PM999) | Wax sealing film for triphosphorylation apparatus (sections 2–4) |
| PCR thermocycler | Bio-Rad Laboratories | C1000 Touch Thermalcycler | For cross-chiral ribozyme reactions (section 7) |
| PD 10 Desalting column (Sephadex G-10 resin) | MilliporeSigma | GE17-0010-01 | Disposable gravity-flow size exclusion chromatography columns containing Sephadex G-10 resin, for oligonucleotides < 15 nt (section 5) |
| Phosphor screens | Cytiva | 28956480 | For visualizing 32P-labeled RNA (section 6) |
| Phosphoramidite synthesis reagents | Glen Research | 30-3142-52, 40-4050-53, 40-4012-52, 40-4122-52, 40-4132-52, 40-4060-62 | representative, for standard RNA/DNA synthesis (section 1) |
| Polypropylene screw-cap sealable tube | MilliporeSigma | BR780752 | 1.5 mL microcentrifuge tubes with screw-cap and silicone O-ring, for safe AMA deprotection (section 5) |
| Pyridine, anhydrous | MilliporeSigma | 270970 | Triphosphorylation solvent (section 2) |
| Reverse-phase liquid chromatography/electrospray ionization mass spectrometry (RP-LC/ESI-MS) | Novatia | n/a | Commercial service for LC/MS specializing in oligonucleotides (section 5) |
| Rubber Septa (ID x OD 7.9 mm x 14 mm), white | MilliporeSigma | Z564702 | Septa for preparing triphosphorylation solvents and TBAP (section 2) |
| Self-replicator ribozyme E | prepared in house14 | 5′-GGAAGUUGUG CUCGAUUGUU ACGUAAGUAA CAGUUUGAAU GGUUGAAGUA UGAGACCGCA ACUUA-3′ | For self-replicator ribozyme reactions (section 6) |
| Self-replicator substrate A | prepared in house14 | 5′-32P-GGAAGUUGUG CUCGAUUGUU ACGUAAGUAA CAGUUUGAAU GGUUGAAGUA U-3′-OH | For self-replicator ribozyme reactions (section 6) |
| Self-replicator substrate B, transcribed | prepared in house14 | 5′-pppGAGACCGCAA CUUA-3′ | For self-replicator ribozyme reactions (section 6) |
| Small Drying Traps, 4 Å molecular sieves | ChemGenes | DMT-1975 | Drying traps for DNA/RNA synthesizer phosphoramidites and triphosphorylation reagents (sections 1–2) |
| Sodium Chloride (NaCl), solid | MilliporeSigma | S7653 | Salt for use in various buffers (sections 5–7) |
| Sodium Hydroxide (NaOH), solid | MilliporeSigma | S8045 | Salt for use in various buffers (sections 5–7) |
| Statistical data-fitting software | GraphPad | Prism | For fitting data from analytical PAGE to kinetic models (section 6) |
| Streptavidin-coated magnetic beads | ThermoFisher Scientific | 65002 | For capturing biotin-labeled RNA in cross-chiral ribozyme reactions (section 7) |
| Sucrose | MilliporeSigma | 84097 | For PAGE urea loading buffer (section 5) |
| TBE running buffer, 10x | ThermoFisher Scientific | AAJ62788K3 | For PAGE (sections 5–7) |
| Tetrabutylammonium Fluoride, 1.0 M solution in Tetrahydrofuran | Aldrich | 216143 | For removing 2′-silyl protecting groups (section 5) |
| Tetramethylethylenediamine (TEMED) | Bio-Rad Laboratories | 1610801 | For polymerizing acrylamide for PAGE (sections 5–7) |
| Tributylamine | MilliporeSigma | 90781 | Triphosphorylation reagent (section 2) |
| Tributylammonium pyrophosphate (TBAP) | MilliporeSigma | P8533 | Triphosphorylation reagent (section 2) |
| Tris base | MilliporeSigma | T6666 | Buffering agent for use in various buffers (sections 5–7) |
| TWEEN20 polysorbate detergent | MilliporeSigma | P7949 | Neutral detergent for use with magnetic beads (Section 7) |
| Urea | MilliporeSigma | U5378 | For PAGE and gel loading buffer (sections 5–7) |
| UV-Vis spectrophotometer | ThermoFisher Scientific | NanoDrop 2000, ND2000 | For measuring oligonucleotide concentrations (section 5) |
| Vacuum centrifuge | ThermoFisher Scientific | Savant Speedvac DNA130-115 Vacuum Concentrator | For removing AMA and THF (section 5) |
| Xylene cyanol | Bio-Rad Laboratories | 1610423 | For PAGE urea loading buffer (section 5) |
Références
- Shuman, S. What messenger RNA capping tells us about eukaryotic evolution. Nature Reviews. Molecular Cell Biology. 3 (8), 619-625 (2002).
- Pichlmair, A., et al. RIG-I-mediated antiviral responses single-stranded RNA bearing 5'-phosphates. Science. 314 (5801), 997-1001 (2006).
- Hornung, V., et al. 5'-Triphosphate RNA is the ligand for RIG-I. Science. 314 (5801), 994-997 (2006).
- Myong, S., et al. Cytosolic viral sensor RIG-I is a 5'-triphosphate-dependent translocase on double-stranded RNA. Science. 323 (5917), 1070-1074 (2009).
- Takeuchi, O., Akira, S. Pattern recognition receptors and inflammation. Cell. 140, 805-820 (2010).
- Wang, Y., et al. Structural and functional insights into 5'-ppp RNA pattern recognition by the innate immune receptor RIG-I. Nature Structural & Molecular Biology. 17 (7), 781-787 (2010).
- Goubau, D., et al. Antiviral immunity via RIG-I-mediated recognition of RNA bearing 5'-diphosphates. Nature. 514 (7522), 372-375 (2014).
- Joyce, G. F. Forty years of in vitro evolution. Angewandte Chemie. 46 (34), 6420-6436 (2007).
- Robertson, M. P., Ellington, A. D. In vitro selection of an allosteric ribozyme that transduces analytes to amplicons. Nature Biotechnology. 17 (1), 62-66 (1999).
- Robertson, M. P., Hesselberth, J. R., Ellington, A. D. Optimization and optimality of a short ribozyme ligase that joins non-Watson-Crick base pairings. RNA. 7 (4), 513-523 (2001).
- Hesselberth, J. R., Robertson, M. P., Knudsen, S. M., Ellington, A. D. Simultaneous detection of diverse analytes with an aptazyme ligase array. Analytical Biochemistry. 312 (2), 106-112 (2003).
- Lam, B. J., Joyce, G. F. Autocatalytic aptazymes enable ligand-dependent exponential amplification of RNA. Nature Biotechnology. 27 (3), 288-292 (2009).
- Lam, B. J., Joyce, G. F. An isothermal system that couples ligand-dependent catalysis to ligand-independent exponential amplification. Journal of the American Chemical Society. 133 (9), 3191-3197 (2011).
- Olea, C., Horning, D. P., Joyce, G. F. Ligand-dependent exponential amplification of a self-replicating L-RNA enzyme. Journal of the American Chemical Society. 134 (19), 8050-8053 (2012).
- Olea, C., Joyce, G. F. Real-Time Detection of a Self-Replicating RNA Enzyme. Molecules. 21 (10), (2016).
- Sczepanski, J. T., Joyce, G. F. A cross-chiral RNA polymerase ribozyme. Nature. 515 (7527), 440-442 (2014).
- Tjhung, K. F., Sczepanski, J. T., Murtfeldt, E. R., Joyce, G. F. RNA-catalyzed cross-chiral polymerization of RNA. Journal of the American Chemical Society. 142 (36), 15331-15339 (2020).
- Bare, G. A. L., Joyce, G. F. Cross-chiral, RNA-catalyzed exponential amplification of RNA. Journal of the American Chemical Society. 143 (45), 19160-19166 (2021).
- Milligan, J. F., Groebe, D. R., Witherell, G. W., Uhlenbeck, O. C. Oligoribonucleotide synthesis using T7 RNA polymerase and synthetic DNA templates. Nucleic Acids Research. 15 (21), 8783-8798 (1987).
- Chelliserrykattil, J., Ellington, A. D. Evolution of a T7 RNA polymerase variant that transcribes 2'-O-methyl RNA. Nature Biotechnology. 22 (9), 1155-1160 (2004).
- Ibach, J., et al. Identification of a T7 RNA polymerase variant that permits the enzymatic synthesis of fully 2′-O-methyl-modified RNA. Journal of Biotechnology. 167 (3), 287-295 (2013).
- Esvelt, K. M., Carlson, J. C., Liu, D. R. A system for the continuous directed evolution of biomolecules. Nature. 472 (7344), 499-503 (2011).
- Pleiss, J. A., Derrick, M. L., Uhlenbeck, O. C. T7 RNA polymerase produces 5' end heterogeneity during in vitro transcription from certain templates. RNA. 4 (10), 1313-1317 (1998).
- Schenborn, E. T., Mierendorf, R. C. A novel transcription property of SP6 and T7 RNA polymerases: dependence on template structure. Nucleic Acids Research. 13 (17), 6223-6236 (1985).
- Martin, C. T., Muller, D. K., Coleman, J. E. Processivity in early stages of transcription by T7 RNA polymerase. Biochemistry. 27 (11), 3966-3974 (1988).
- Gholamalipour, Y., Karunanayake Mudiyanselage, A., Martin, C. T. 3' end additions by T7 RNA polymerase are RNA self-templated, distributive and diverse in character-RNA-Seq analyses. Nucleic Acids Research. 46 (18), 9253-9263 (2018).
- Vasilyev, N., Serganov, A. Preparation of short 5′-triphosphorylated oligoribonucleotides for crystallographic and biochemical studies. Nucleic Acid Crystallography: Methods and Protocols. , 11-20 (2016).
- Lebedev, A. V., Koukhareva, I. I., Beck, T., Vaghefi, M. M. Preparation of oligodeoxynucleotide 5'-triphosphates using solid support approach. Nucleosides, Nucleotides & Nucleic Acids. 20 (4-7), 1403-1409 (2001).
- Paul, N., Springsteen, G., Joyce, G. F. Conversion of a ribozyme to a deoxyribozyme through in vitro evolution. Chemistry & Biology. 13 (3), 329-338 (2006).
- Zlatev, I., et al. Efficient solid-phase chemical synthesis of 5'-triphosphates of DNA, RNA, and their analogues. Organic Letters. 12 (10), 2190-2193 (2010).
- Zlatev, I., Manoharan, M., Vasseur, J. -. J., Morvan, F. Solid-phase chemical synthesis of 5'-triphosphate DNA, RNA, and chemically modified oligonucleotides. Current Protocols in Nucleic Acid Chemistry. , (2012).
- Zlatev, I., et al. Automated parallel synthesis of 5'-triphosphate oligonucleotides and preparation of chemically modified 5'-triphosphate small interfering RNA. Bioorganic & Medicinal Chemistry. 21 (3), 722-732 (2013).
- Goldeck, M., Tuschl, T., Hartmann, G., Ludwig, J. Efficient solid-phase synthesis of pppRNA by using product-specific labeling. Angewandte Chemie. 53 (18), 4694-4698 (2014).
- Sarac, I., Meier, C. Efficient automated solid-phase synthesis of DNA and RNA 5′-triphosphates. Chemistry-A European Journal. 21 (46), 16421-16426 (2015).
- Sarac, I., Meier, C. Solid-phase synthesis of DNA and RNA 5'-O-triphosphates using cycloSal chemistry. Current Protocols in Nucleic Acid Chemistry. 64 (1), 4-67 (2016).
- Perez, J. T., et al. Influenza A virus-generated small RNAs regulate the switch from transcription to replication. Proceedings of the National Academy of Sciences of the United States of America. 107 (25), 11525-11530 (2010).
- Ludwig, J., Eckstein, F. Rapid and efficient synthesis of nucleoside 5'-0-(1-thiotriphosphates), 5'-triphosphates and 2',3'-cyclophosphorothioates using 2-chloro-4H-1,3,2-benzodioxaphosphorin-4-one. The Journal of Organic Chemistry. 54 (3), 631-635 (1989).
- Gaur, R. K., Sproat, B. S., Krupp, G. Novel solid phase synthesis of 2'-o-methylribonucleoside 5'-triphosphates and their α-thio analogues. Tetrahedron Letters. 33 (23), 3301-3304 (1992).
- Reddy, M. P., Hanna, N. B., Farooqui, F. Fast cleavage and deprotection of oligonucleotides. Tetrahedron Letters. 35 (25), 4311-4314 (1994).
- Hogrefe, R. I., McCaffrey, A. P., Borozdina, L. U., McCampbell, E. S., Vaghefi, M. M. Effect of excess water on the desilylation of oligoribonucleotides using tetrabutylammonium fluoride. Nucleic Acids Research. 21 (20), 4739-4741 (1993).
- Pitsch, S., Weiss, P. A., Jenny, L., Stutz, A., Wu, X. Reliable chemical synthesis of oligoribonucleotides (RNA) with 2′-O-[(Triisopropylsilyl)oxy]methyl(2′-O-tom)-protected phosphoramidites. Helvetica Chimica Acta. 84 (12), 3773-3795 (2001).
- Green, M. R., Sambrook, J. Isolation of DNA fragments from polyacrylamide gels by the crush and soak method. Cold Spring Harbor Protocols. 2019 (2), (2019).
- Paul, N., Joyce, G. F. A self-replicating ligase ribozyme. Proceedings of the National Academy of Sciences of the United States of America. 99 (20), 12733-12740 (2002).
- Lincoln, T. A., Joyce, G. F. Self-sustained replication of an RNA enzyme. Science. 323 (5918), 1229-1232 (2009).
- Robertson, M. P., Joyce, G. F. Highly efficient self-replicating RNA enzymes. Chemistry & Biology. 21 (2), 238-245 (2014).
- Singh, J., et al. Synthesis of modified nucleoside oligophosphates simplified: fast, pure, and protecting group free. Journal of the American Chemical Society. 141 (38), 15013-15017 (2019).
- Bellon, L. Oligoribonucleotides with 2'-O-(tert-butyldimethylsilyl) groups. Current Protocols in Nucleic Acid Chemistry. , (2001).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon