Method Article
Création de paires de modèles appariés in vivo/in vitro dérivés du patient d’organoïdes dérivés de PDX et de PDX pour la recherche en pharmacologie du cancer
Dans cet article
Résumé
Une méthode est décrite pour créer des organoïdes à l’aide de xénogreffes dérivées du patient (PDX) pour le dépistage in vitro, ce qui donne des paires appariées de modèles in vivo/in vitro. Des tumeurs de PDX ont été moissonnes/traitées en petits morceaux mécaniquement ou enzymatiquement, suivies du Clevers' méthode pour cultiver des organoids de tumeur qui ont été passaged, cryopreserved et caractérisés contre le PDX original.
Résumé
Les xénogreffes tumorales dérivées du patient (PDX) sont considérées comme les modèles précliniques les plus prédictifs, largement considérés comme pilotés par les cellules souches cancéreuses (CSC) pour l’évaluation conventionnelle des médicaments anticancéreux. Une grande bibliothèque de PDX reflète la diversité des populations de patients et permet ainsi des essais précliniques basés sur la population (« essais cliniques de souris de type II »); cependant, pdx ont des limites pratiques de faible débit, coûts élevés et longue durée. Les organoïdes tumoraux, étant également des modèles CSC-conduits patients-dérivés, peuvent être considérés comme l’équivalent in vitro de PDX, surmontant certaines limitations de PDX pour traiter de grandes bibliothèques d’organoïdes ou de composés. Cette étude décrit une méthode pour créer des organoïdes dérivés de PDX (PDXO), résultant ainsi en des modèles appariés pour la recherche en pharmacologie in vitro et in vivo. Des tumeurs PDX-CR2110 sous-cutanées-transplantées ont été rassemblées des souris de tumeur-roulement quand les tumeurs ont atteint 200-800millimètres 3,par une procédure approuvée d’autopsie, suivie du déplacement des tissus non-tumoraux adjacents et de la dissociation dans de petits fragments de tumeur. Les petits fragments tumoraux ont été lavés et passés à travers une passoire cellulaire de 100 μm pour enlever les débris. Des amas de cellules ont été rassemblés et suspendus dans la solution d’extrait de membrane de sous-sol (BME) et plaqués dans une plaque de 6 puits comme gouttelette solide avec des milieux liquides environnants pour la croissance dans un incubateur de CO2. La croissance d’Organoid a été surveillée deux fois par semaine sous la photomicroscopie et enregistrée par la photographie, suivie du changement liquide de milieu 2 ou 3 fois par semaine. Les organoïdes cultivés ont ensuite été croisés (7 jours plus tard) à un rapport de 1:2 en perturbant les organoïdes incorporés au BME à l’aide d’un cisaillement mécanique, aidés par l’ajout de trypsine et l’ajout de 10 μM d’Y-27632. Les organoïdes ont été cryoconservés dans des cryo-tubes pour le stockage à long terme, après libération de BME par centrifugation, et également échantillonnés (par exemple, l’ADN, l’ARN et le bloc FFPE) pour une caractérisation plus approfondie.
Introduction
Les cancers sont un ensemble de divers troubles génétiques et immunologiques. Le succès de la mise au point de traitements efficaces dépend fortement de modèles expérimentaux qui prédisent efficacement les résultats cliniques. De grandes bibliothèques de xénogreffes dérivées du patient (PDX) bien caractérisées ont longtemps été considérées comme le système translationnel in vivo de choix pour tester les thérapies chimio- et / ou ciblées en raison de leur capacité à récapituler les caractéristiques tumorales du patient, l’hétérogénéité et la réponse du médicament patient1,permettant ainsi aux essais cliniques de souris de type Phase II d’améliorer le succès clinique2,3. Les PDX sont généralement considérés comme des maladies des cellules souches cancéreuses, présentant une stabilité génétique, contrairement aux xénogreffes dérivées de lignées cellulaires2. Au cours des dernières décennies, de grandes collections de PDX ont été créées dans le monde entier, devenant le cheval de bataille du développement de médicaments anticancéreux aujourd’hui. Bien que largement utilisés et ayant une grande valeur translationnelle, ces modèles animaux sont intrinsèquement coûteux, longs et à faible débit, donc inadéquats pour le criblage à grande échelle. Les PDX sont également indésirables pour les tests d’immuno-oncologie (IO) en raison d’une nature immunodéprimée4. Il n’est donc pas pratique de tirer pleinement parti de la grande bibliothèque de PDX disponible.
Des découvertes récentes, initiées par le laboratoire5de Hans Clevers, ont conduit à l’établissement de cultures in vitro d’organoïdes générés à partir de cellules souches adultes dans la plupart des organes humains d’origine épithéliale5. Ces protocoles ont été affinés pour permettre la croissance d’organoïdes à partir de CSC supposés dans des carcinomes humains de diverses indications6,7. Ces organoïdes dérivés du patient (PDO) sont génomiquement stables8,9 et se sont avérés hautement prédictifs des résultats du traitement clinique10,11,12. En outre, la nature in vitro des PDO permet un criblage à haut débit (HTS)13,offrant ainsi potentiellement un avantage sur les modèles in vivo et tirant parti de grandes bibliothèques organoïdes comme substitut de la population de patients. Les PDO sont sur le point de devenir une importante plate-forme de découverte et de traduction, surmontant les nombreuses limitations des PDX décrites ci-dessus.
L’AOP et la PDX sont des modèles dérivés du patient et axés sur le SCC, qui ont la capacité d’évaluer les traitements dans le contexte d’un traitement personnalisé ou d’un format d’essai clinique. Les grandes bibliothèques existantes de PDX, comme la collection propriétaire de >3000 PDX14,15,16,17,conviennent donc à la génération rapide de bibliothèques d’organoïdes tumoraux (organoïdes dérivés de PDX, ou PDXO), résultant en une bibliothèque appariée de modèles PDX et PDXO appariés. Ce rapport décrit la procédure pour créer et caractériser le cancer côlorectal PDXO-CR2110 par rapport à son pdx-cr2110 parental modèle16.
Protocole
Tous les protocoles et modifications ou procédures concernant le soin et l’utilisation des animaux ont été examinés et approuvés par le Crown Bioscience Institutional Animal Care and Use Committee (IACUC) avant la réalisation des études. Le soin et l’utilisation des animaux ont été effectués conformément aux lignes directrices internationales de l’AAALAC (Association for Assessment and Accreditation of Laboratory Animal Care), telles qu’elles sont indiquées dans le Guide pour le soin et l’utilisation des animaux de laboratoire, Conseil national de recherches du Pays (2011). Toutes les procédures expérimentales sur les animaux ont été effectuées dans des conditions stériles dans des installations SPF (exempts d’agents pathogènes spécifiques) et menées en stricte conformité avec le Guide pour le soin et l’utilisation des animaux de laboratoire de différentes institutions gouvernementales (p. ex. les National Institutes of Health). Les protocoles ont été approuvés par le Comité d’éthique de l’expérimentation animale de l’établissement (p. ex. le Comité IACUC de l’établissement).
1. Préparation à la transplantation de tumeurs
-
Logement des animaux
- Souris nues Maison Balb/c (n = 5) dans des cages ventilées individuelles, à 20-26 ° C, 30-70% d’humidité, et un cycle d’éclairage de 12 h de lumière / 12 h foncé, avec une litière en épis de maïs changée chaque semaine, et l’irradiation stérilisée des aliments secs en granulés et de l’eau potable stérile ad libitum.
-
Préparation de fragments tumoraux donneurs
- Surveillez de près les souris donneuses porteuses de tumeurs pour le poids corporel (BW, via la balance de pesée) et le volume tumoral (TV, par mesure de l’étrier).
- Lorsque la télévision atteint 800-1000 mm3, euthanasier les animaux porteurs de tumeurs dans une hotte de biorisque.
- Placez les souris dans une chambre d’euthanasie avec un couvercle qui fournit du GAZ CO2 dans la chambre. Décharger du gaz dans la chambre à un débit qui produit une perte de conscience rapide avec un minimum de détresse pour l’animal. Le débit optimal pour le CO2 est d’environ 2-2,5 Lpm.
- Assurer l’euthanasie en observant que les animaux ne reprennent pas conscience.
- Après la mort clinique apparente, maintenir le débit de gaz pendant >1 min pour réduire au minimum la possibilité qu’un animal puisse récupérer.
- Stériliser la peau autour de la tumeur à l’aide d’écouvillons iodophores. Recueillir les tumeurs en enlevant les tissus non tumoraux adjacents et en les plaçant dans une boîte de Petri contenant 20 mL de PBS, pré-réfrigéré (4 °C) avant l’euthanasie.
- Lavez les tumeurs avec du PBS dans une autre boîte de Petri pour enlever les composants sanguins, puis coupez en deux et en enlevant toute peau supplémentaire, les vaisseaux sanguins, la calcification et /ou la nécrose.
- Placez seulement les morceaux intacts de tumeur dans un tube stérile de 50 mL de centrifugeuse avec 20 mL de PBS, avant de transporter à une chambre d’animal séparée pour la transplantation.
2. Croissance tumorale sous-cutanée
-
Inoculation sous-cutanée d’une pièce tumorale chez des souris immunodéprimées
- Coupez la tumeur pdx en morceaux de 2 millimètres avec un scalpel, et placez chacun dans des trocarts pour l’implantation sous-cutanée (SC).
- Anesthésier les animaux receveurs avec 5% d’isoflurane (maintenu par un cône de nez à 1%). Les animaux se détendent, perdent leur réflexe de redressement et finissent par devenir immobiles, jusqu’à ce qu’ils ne répondent pas à la douleur. Fixer les souris anesthésiées sur un tableau d’expérience dans la bonne position latérale et stériliser avec des écouvillons iodophores, en particulier les zones entourant le site de l’inoculation tumorale.
- Sur la peau du flanc gauche juste crânienne à la hanche, faites une incision de 0,5 cm avec un scalpel et créez un tunnel sous la peau vers le membre antérieur, 2-3 cm, en utilisant une pince émoussée.
- Transférer aseptiquement un cube de fragment de tumeur par emplacement d’inoculation du milieu et placer profondément à l’intérieur du tunnel sous-cutané. Confirmer visuellement la position du fragment avant la fermeture de la plaie avec des clips de plaie.
- Surveiller les souris implantées dans la tumeur (5) jusqu’à ce qu’elles deviennent conscientes de maintenir une réamence sternale, puis ne les renvoyer dans leur cage qu’après leur rétablissement complet de l’anesthésie.
-
Surveillance de la santé des souris porteuses de tumeurs
- Vérifiez quotidiennement la consommation d’eau et d’aliments et enregistrez le poids corporel chaque semaine.
- Vérifiez les souris pendant la surveillance de routine. Consigner tous les effets sur la mobilité, la respiration, le toilettage et l’apparence générale, la consommation d’aliments et d’eau, le gain ou la perte d’armes biologiques, l’ascite, etc.
- Mesurer tv deux fois par semaine à l’aide d’étriers et express en mm3 par: TV = 0,5 a × b2, où a et b sont la longueur et la largeur de la tumeur, respectivement.
- Sacrifier des animaux pour prélever des échantillons lorsque l’un des signes suivants apparaît : perte d’armes biologiques >20 %; mobilité réduite (incapable de manger ou de boire); incapable de se déplacer normalement en raison d’une ascite importante ou d’une hypertrophie de l’abdomen; effort dans la respiration; mort.
3. Nécropsie et récolte tumorale
- Lorsque le volume tumoral atteint 200-800 mm3,euthanasier les souris selon les protocoles approuvés (voir étape 1.2.2).
- Recueillir les tissus tumoraux en enlevant les tissus non tumoraux adjacents, puis en plaçant le tissu dans un tube en plastique de 50 mL avec AD+++ (sur la glace) avant la dissociation.
4. Préparation pour la culture organoïde dérivée de PDX
REMARQUE: Toutes les étapes suivantes ont été effectuées à l’intérieur d’une armoire de biosécurité conformément aux lignes directrices de la norme de culture tissulaire. Conservez les stocks préchauffés de plaques de 96, 24 et 6 puits dans un incubateur à 37 °C avant utilisation.
- Préparations réactives
- Préparer une solution d’extrait de membrane basale (BME) (facteur de croissance réduit, sans rouge de phénol). Conservez 10 mL de BME dans un réfrigérateur à 4 °C pendant la nuit. Une fois décongelée, faire tourbillonner la bouteille de BME pour assurer la dispersion.
- Préparez le support de base/tampon de lavage AD+++. Ajouter 5 mL de L-glutamine de 200 mM, de 1 M hepes et de stylo/streptocoque aux supports avancés DMEM/F12 par pipetage avec une pipette de 5 mL.
- Préparer le milieu organoïde du côlon tel que décrit par Sato et al.18 en complétant le milieu de base avec du N-Ac (1 mM), de l’A83-01 (500 nM), du B27 (1x), de l’EGF (50 ng/mL), du Noggin (100 ng/mL), du Nicotinamide (10 mM), du SD202190 (10 nM), de la R-spondine (500 ng/mL), de la L-glutamine (2 mM), de l’HEPES (10 μM), de la pénicilline-streptomycine (1x) et de l’Y-27623 (10 μM)19.
- Préparer le milieu de digestion AD+++: 10 mL de 1x milieu de culture organoïde avec 500 μL de collagénase de type II (20 mg/mL) et 10 μL de RhoKI Y-27632 (10 mM).
- Dissociationtumorale 20
- Transférer la tumeur dans un tube en plastique de 50 ml dans une boîte de Petri de 10 cm. Prenez une photographie macroscopique à côté d’une règle et notez une description de ses conditions (c.-à-d. taille, tissus adipeux, vascularisation, nécrose, etc.).
- Retirez l’excès d’AD+++ par aspiration, et coupez le tissu tumoral en petits morceaux par des ciseaux suivis d’un transfert de 1-2 morceaux dans un microtube de 2 mL pour casser le gel sur de la glace carbonique. Conserver à -80 °C pour le profilage génomique. Hachez les morceaux restants en morceaux plus fins par des ciseaux avant de les transférer dans un tube en plastique de 50 mL à l’aide d’AD +++.
- Laver les tissus hachés 2 à 3 fois par 35 mL de MA+++, suivi de l’ajout de 10 mL de milieu de digestion (voir étape 4.1.4) et de leur placement sur un agitateur orbital à 4 x g pendant 1 h à 37 °C.
- Homogénéiser le tissu digéré en pipetant de haut en bas à l’aide d’une pipette en plastique stérile de 5 mL, suivie de l’ajout de 20 mL d’AD+++ et de la filtration par une passoire cellulaire de 100 μm.
- Laver le pass-through deux fois avec AD+++ (tourner à 450 x g pendant 5 min), suivi de la remise en suspension dans le BME (ajouter 4x volume de BME à la pastille pour la suspension) et garder sur la glace19.
- Préparation de la culture organoïde
- Ajouter la suspension cellulaire BME dans une plaque de 6 puits en plusieurs gouttes pour un total de 200 μL par puits.
- Transférer la plaque dans un incubateur à 37 °C. Après 30 min, les gouttes de gel se solidifient.
- Ajouter 2 mL de milieux organoïdes à chaque puits, avec les gouttes représentatives enregistrées par photographie microscopique avant de les transférer dans un incubateur (37 °C et 5 % deCO2).
- Maintenir les cultures organoïdes avec un changement moyen tous les 3-4 jours, et passer à un rapport de 1:2 tous les 7 jours ou en fonction de leur croissance et de leur densité.
5. Analyse histopathologique et de séquençage de nouvelle génération (NGS)
- histopathologie
- Recueillir les organoïdes du puits avec le milieu existant à l’aide d’une pipette P1000, suivie d’une centrifugation (rotation à 450 x g pendant 5 min), d’un lavage au PBS et d’une fixation dans du formol à 10% pendant 1 h.
- Placer les organoïdes fixes dans de la gélatine à 100 % au fond du tube conique de 50 mL, suivi d’un traitement tissulaire et d’un encastrement de routine.
- Effectuer une coloration hématoxyline-éosine (H&E) à l’aide de protocoles standard sur des sections de paraffine de 4 mm.
- RNAseq et séquençage de l’exome entier
- Recueillir les organoïdes du puits avec le milieu de culture existant à l’aide d’une pipette P1000, suivie d’une centrifugation à l’aide d’une microcentrifuge à vitesse maximale (12 000 x g)pendant 5 min à 4 °C.
- Recueillir la pastille en enlevant le surnageant moyen pour s’assurer qu’il n’y avait pas de BME visible, puis en le faisant geler dans un microtube (glace carbonique), puis en la transférant dans un congélateur à -80 °C.
- Extraire l’ARN ou l’ADN en utilisant la procédure standard des fabricants et effectuer une analyse NGS pour rnAseq et le séquençage de l’exome entier (WES).
6. IC50 essai20
- Ensemencement organoïde dans une plaque de 384 puits pour le dosage IC50
- Dissocier les organoïdes dans les gouttes de BME de chaque puits (BME digérant) en ajoutant 20 μL de solution 100x dispase à chaque puits (plaque de 6 puits), qui contenait 2 mL de milieu organoïde, suivi de 10 min d’incubation à 37 °C.
- Pipette a digéré les organoïdes de tous les puits à travers un filtre de 70 μm dans un tube en plastique de 50 mL pour recueillir les organoïdes.
- Compter les organoïdes sous microscopie pour la détermination de la concentration d’organoïdes. Suspendre les organoïdes à l’aide du milieu de culture, avant d’ajouter du BME pour atteindre une concentration finale de 5 % (v/v) sur la glace.
- Ajouter 50 μL de la suspension organoïde dans chaque plaque de 384 puits avec le distributeur de liquide, selon la carte de la plaque avec une densité d’ensemencement de 200 CR2110 PDXO par puits dans le milieu de culture organoïde correspondant.
- Traitement au cisplatine et à l’irinotécan
- Utilisez du cisplatine (avec la concentration la plus élevée de 100 μM) et de l’irinotécan (avec la concentration la plus élevée de 10 μM). Ajouter le SN-38 (un métabolite de l’irinotécan, par opposition à l’irinotécan qui est habituellement utilisé pour les études in vivo) à chaque puits selon le schéma de dilution du médicament pendant 9 doses, en dilution en série par dispener numérique.
- Créez la carte à plaques à l’aide de l’outil logiciel distributeur numérique. Inclure un véhicule témoin négatif avec une viabilité de 100 % et un contrôle postif de la starurosporine de 5 μM, qui a montré une viabilité de 0 %.
- Replacez les plaques de 384 puits traitées par médicaments dans un incubateur à 37 °C.
- Détermination de la viabilité des cellules organoïdes après un traitement médicamenteux
- À la fin des 5 jours de traitement médicamenteux, déterminer la viabilité des cellules organoïdes à l’aide de réactifs de viabilité cellulaire luminescente conformément à la procédure recommandée par le fabricant. Ajouter un réactif luminescent dans chaque puits avec le dispener liquide et mélanger pendant 5 min sur un agitateur à plaques, suivi d’une incubation de 30 min à température ambiante dans l’obscurité.
- Enregistrez le signal luminescent sur un lecteur de plaques multi-puits à luminescence.
- Calculer les capacités normalisées de chaque puits à l’aide des lectures brutes du lecteur de plaque et créer une courbe dose-réponse et des valeurs IC50 par ajustement de courbe non linéaire.
Résultats
Morphologie des PDXO, typique des organoïdes sous microcopie légère, et compatible avec pdx parental par coloration H&E
En microscopie optique, PDXO-CR2110 démontre une morphologie kystique typique(figure 1A),comme décrit précédemment pour les organoïdes dérivés du patient (AOP), preuves soutenant la similitude entre PDXO et AOP dans les mêmes conditions de culture.
L’examen histopathologique par coloration H&E révèle que les structures tissulaires et les types de cellules de PDXO-CR2110 (figure 1B) reflètent le PDX-CR2110 original(figure 1C),ce qui soutient que le PDX et le PDXO ont été développés à partir de la même origine : CR2110. Cette observation fournit des preuves histopathologiques pour soutenir la similitude de PDXO à son PDX parental.
L’expression du transcriptome et le séquençage de l’exome entier démontrent une corrélation élevée entre PDXO-CR2110 et le PDX-CR2110 parent
Des tumeurs PDX-CR2110 ont été précédemment genomically-profiled utilisant le séquençage de transcriptome (RNAseq, ADN messagère)16 et l’exome entier (WES, ADN) séquencé. Nous avons maintenant profilé de la même manière le PDXO-CR2110 correspondant. Les comparaisons de profils génomiques des PDX et PDXO appariés correspondants (Figure 2) démontrent une corrélation élevée de 94,92 % dans l’expression du transcriptome (ARNm) (épigénétique) et une concordance élevée de 97,67 % des mutations de l’ADN (WES) (génétiques), ce qui suggère une similitude génomique globale entre cette paire de modèles.
Similitudes observées pour les propriétés pharmacologiques entre pdxo-CR2110 in vitro et pdx-cr2110 in vivo
Des essais de sensibilité aux médicaments ont été effectués sur PDXO-CR2110 dans des plaques de 384 puits, avec les résultats illustrés à la figure 3A. PDXO-CR2110 était sensible à l’irinotécan et résistant au cisplatine, ce qui correspond aux résultats du traitement PDX (figure 3B) où l’IGC (inhibition de la croissance tumorale) à des doses de 100 mg/kg, i.p., Q3Dx3 pour l’irinotécan et 5 mg/kg, i.p., Q4Dx4 pour le cisplatine est de 84,63 % et 6,61 % respectivement. Cette cohérence pharmacologique observée soutient l’équivalence potentiellement biologique des deux modèles et peut être utilisée pour des études pharmacologiques complémentaires in vitro et in vivo.
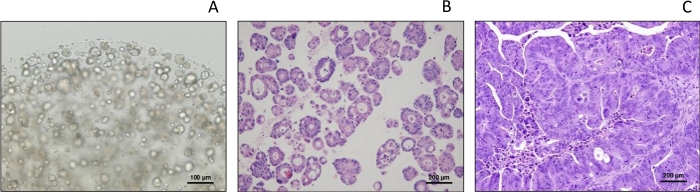
Figure 1 : Morphologie du PDXO-CR2110. (A) Morphologie de sous microscopie optique (type kystique). (B,C) Histopathologie de PDXO-CR2110 et PDX-CR2110, respectivement. Veuillez cliquer ici pour voir une version plus grande de cette figure.

Figure 2 : Profils génomiques de PDXO-CR2110 par rapport à PDX-CR2110, WES et RNAseq. Panel supérieur : corrélation globale d’expression de l’ARNm entre PDX-CR2110 et PDXO-CR2110 par RNAseq. Panel inférieur : tableau des corrélations d’expression de l’ARNm par RNAseq et concordance des mutations de l’ADN par WES : PDX- vs PDXO-CR2110. Veuillez cliquer ici pour voir une version plus grande de cette figure.
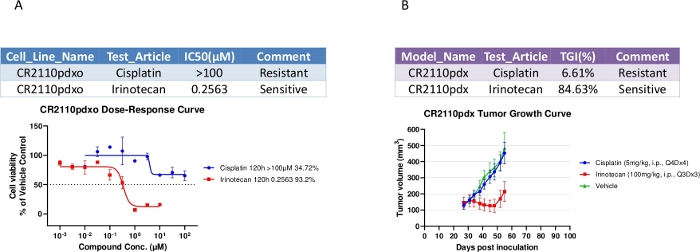
Figure 3 : Propriétés pharmacologiques du PDXO-CR2110 in vitro et du SC PDX-CR2110 in vivo. (A) PDXO-CR2110 in vitro dose-réponse aux composés d’essai. (B) Inhibition de la croissance tumorale induite par les mêmes composés d’essai sur CR2110 in vivo. Veuillez cliquer ici pour voir une version plus grande de cette figure.
Discussion
Les données préliminaires pour PDX-/PDXO-CR2110 dans ce rapport soutiennent l’équivalence biologique entre PDX et son dérivé, PDXO, en ce qui concerne la génomique, l’histopathologie et la pharmacologie, puisque les deux modèles représentent les formes de maladie dérivées du CSC original du patient. Les deux modèles sont des modèles de maladies dérivées du patient, potentiellement prédictifs de la réponse clinique des patients10,11,12,21. La paire appariée de modèles in vitro et in vivo peut se compléter pour le dépistage et la validation in vitro in vivo, améliorant ainsi le taux de réussite de la découverte de médicaments et réduisant potentiellement les taux d’attrition dans le développement clinique. Il convient de noter que PDXO peut maintenant permettre à HTS de tirer parti des grandes bibliothèques organoïdes dérivées du patient disponibles, où les PDX échouent en raison de coûts in vivo élevés et de délais plus longs. Il va sans dire qu’une bibliothèque PDX-PDXO appariée deviendrait probablement la plate-forme de choix pour soutenir la découverte de médicaments et la recherche translationnelle dans un avenir proche.
La conversion de la bibliothèque existante de modèles PDX annotés pourrait être une approche rapide et productive pour construire une bibliothèque d’organoïdes pratique en utilisant un processus industriel. Ce rapport a converti un PDX en PDXO pour explorer la faisabilité d’un tel processus, et la méthode utilisée pourrait être une base pour soutenir le processus à grande échelle de construction d’une vaste bibliothèque PDXO. Pratiquement, les méthodes de création de PDXO sont généralement similaires à la méthode largement décrite pour la génération de PDO18, à l’exception de la source du tissu patient étant des souris.
Il y a des étapes critiques pour s’assurer que pdxos sont créés avec succès : 1) les tumeurs fraîches de PDX sont fragmentées en petits morceaux ; 2) les conditions de culture décrites pour la culture organoïde telles que décrites par Clevers et ses collègues sont fidèlement mises en œuvre18,22,mais peuvent être ajustées pour différents organoïdes ; 3) différents organoïdes ont des taux de croissance différents, ce qui a un impact sur la durée de la culture et de la santé des organoïdes, ainsi que sur la durée du traitement médicamenteux; 4) un essai efficace pour déterminer la teneur en souris par rapport à la teneur en humains est absolument essentiel pour s’assurer que les cultures sont en grande partie des organoïdes humains, car certaines cultures peuvent inévitablement avoir une contamination persistante des tissus ou cellules de souris (dans le cas rapporté ici, il y a une contamination minimale des tissus de souris, données non présentées). La contamination par la souris pourrait être l’une des limites importantes de la création de biobanques de PDXO si des méthodes efficaces de détection et d’élimination ne sont pas utilisées.
Déclarations de divulgation
Tous les auteurs sont les employés à temps plein actuels de Crown Bioscience, Inc.
Remerciements
Les auteurs tiennent à remercier Jody Barbeau, Federica Parisi et Rajendra Kumari pour la lecture critique et l’édition du manuscrit. Les auteurs tiennent également à remercier l’équipe in vitro et in vivo de Crown Bioscience Oncology pour ses grands efforts techniques.
matériels
| Name | Company | Catalog Number | Comments |
| Advanced DMEM/F12 | Life Technologies | 12634028 | Base medium |
| DMEM | Hyclone | SH30243.01 | Washing medium |
| Collagenese type II | Invitrogen | 17101015 | Digest tumor |
| Matrigel | Corning | 356231 | Organoid culture matrix (Basement Membrane Extract, growth factor reduced) |
| N-Ac | Sigma | A9165 | Organoid culture medium |
| A83-01 | Tocris | 2939 | Organoid culture medium |
| B27 | Life Technologies | 17504044 | Organoid culture medium |
| EGF | Peprotech | AF-100-15 | Organoid culture medium |
| Noggin | Peprotech | 120-10C | Organoid culture medium |
| Nicotinamide | Sigma | N0636 | Organoid culture medium |
| SB202190 | Sigma | S7076 | Organoid culture medium |
| Gastrin | Sigma | G9145 | Organoid culture medium |
| Rspondin | Peprotech | 120-38-1000 | Organoid culture medium |
| L-glutamine | Life Technologies | 35050038 | Organoid culture medium |
| Hepes | Life Technologies | 15630056 | Organoid culture medium |
| penicillin-streptomycin | Life Technologies | 15140122 | Organoid culture medium |
| Y-27632 | Abmole | M1817 | Organoid culture medium |
| Dispase | Life Technologies | 17105041 | Screening assay |
| CellTiter-Glo 3D | Promega | G9683 | Screening assay (luminescent ATP indicator) |
| Multidrop dispenser | Thermo Fisher | Multidrop combi | Plating organoids/CellTiter-Glo 3D addition |
| Digital dispener | Tecan | D300e | Compound addition |
| Envision Plate reader | Perkin Elmer | 2104 | Luminescence reading |
| Balb/c nude mice | Beijing HFK Bio-Technology Co | ||
| RNAeasy Mini kit | Qiagen | 74104 | tRNA purification kit |
| DNAeasy Blood & Tissue Kit | Qiagen | 69506 | DNA purification kit |
| Histogel | Thermo Fisher | HG-4000-012 | Organoid embedding |
Références
- Tentler, J. J., et al. Patient-derived tumour xenografts as models for oncology drug development. Nature Reviews Clinical Oncology. 9 (6), 338-350 (2012).
- Gao, H., et al. High-throughput screening using patient-derived tumor xenografts to predict clinical trial drug response. Nature Medicine. 21 (11), 1318-1325 (2015).
- Yang, M., et al. Overcoming erlotinib resistance with tailored treatment regimen in patient-derived xenografts from naive Asian NSCLC patients. International Journal of Cancer. 132 (2), 74-84 (2013).
- Li, Q. X., Feuer, G., Ouyang, X., An, X. Experimental animal modeling for immuno-oncology. Pharmacology & Therapeutics. 173, 34-46 (2017).
- Sato, T., et al. Single Lgr5 stem cells build crypt-villus structures in vitro without a mesenchymal niche. Nature. 459 (7244), 262-265 (2009).
- Drost, J., Clevers, H. Organoids in Cancer Researchearch. Nature Reviews Cancer. 18 (7), 407-418 (2018).
- Muthuswamy, S. K. Organoid Models of Cancer Explode with Possibilities. Cell Stem Cell. 22 (3), 290-291 (2018).
- Sachs, N., et al. A Living Biobank of Breast Cancer Organoids Captures Disease Heterogeneity. Cell. 172 (1-2), 373-386 (2018).
- Weeber, F., et al. Preserved genetic diversity in organoids cultured from biopsies of human colorectal cancer metastases. Proceedings of the National Academy of Sciences of the United States of America. 112 (43), 13308-13311 (2015).
- Vlachogiannis, G., et al. Patient-derived organoids model treatment response of metastatic gastrointestinal cancers. Science. 359 (6378), 920-926 (2018).
- Yao, Y., et al. Patient-Derived Organoids Predict Chemoradiation Responses of Locally Advanced Rectal Cancer. Cell Stem Cell. 26 (1), 17-26 (2020).
- Ganesh, K., et al. A rectal cancer organoid platform to study individual responses to chemoradiation. Nature Medicine. 25 (10), 1607-1614 (2019).
- van de Wetering, M., et al. Prospective derivation of a living organoid biobank of colorectal cancer patients. Cell. 161 (4), 933-945 (2015).
- Yang, J. P., et al. A novel RNAi library based on partially randomized consensus sequences of nuclear receptors: identifying the receptors involved in amyloid beta degradation. Genomics. 88 (3), 282-292 (2006).
- Zhang, L., et al. A subset of gastric cancers with EGFR amplification and overexpression respond to cetuximab therapy. Scientific Reports. 3, 2992 (2013).
- Chen, D., et al. A set of defined oncogenic mutation alleles seems to better predict the response to cetuximab in CRC patient-derived xenograft than KRAS 12/13 mutations. Oncotarget. 6 (38), 40815-40821 (2015).
- Guo, S., et al. Molecular Pathology of Patient Tumors, Patient-Derived Xenografts, and Cancer Cell Lines. Cancer Research. 76 (16), 4619-4626 (2016).
- Sato, T., et al. Long-term expansion of epithelial organoids from human colon, adenoma, adenocarcinoma, and Barrett's epithelium. Gastroenterology. 141 (5), 1762-1772 (2011).
- Tiriac, H., French, R., Lowy, A. M. Isolation and Characterization of Patient-derived Pancreatic Ductal Adenocarcinoma Organoid Models. Journal of Visualized Experiments. (155), (2020).
- Kopper, O., et al. An organoid platform for ovarian cancer captures intra- and interpatient heterogeneity. Nature Medicine. 25 (5), 838-849 (2019).
- Corcoran, R. B., et al. Combined BRAF and MEK Inhibition With Dabrafenib and Trametinib in BRAF V600-Mutant Colorectal Cancer. Journal of Clinical Oncology. , (2015).
- Huch, M., et al. Long-term culture of genome-stable bipotent stem cells from adult human liver. Cell. 160 (1-2), 299-312 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon