Method Article
Un virus de la grippe Luciferase-fluorescent Reporter pour l'imagerie en direct et la quantification de l'infection virale
Dans cet article
Résumé
Les virus de la grippe A (IvIV) sont des agents pathogènes respiratoires contagieux qui causent des épidémies annuelles et des pandémies occasionnelles. Ici, nous décrivons un protocole pour suivre des infections virales in vivo utilisant une luciferase recombinante nouvelle et fluorescence-exprimant bi-reporter IAV (BIRFLU). Cette approche fournit aux chercheurs un excellent outil pour étudier l'IAV in vivo.
Résumé
Les virus de la grippe A (IvIV) causent des maladies respiratoires humaines qui sont associées à des conséquences économiques et sanitaires importantes. Comme pour d'autres virus, l'étude de la LUTTE anti-I nécessite l'utilisation d'approches secondaires laborieuses pour détecter la présence du virus dans les cellules infectées et/ou dans les modèles animaux d'infection. Cette limitation a été récemment contournée avec la génération de IAV recombinants exprimant facilement traçablefluorescent ou bioluminescent (luciferase) protéines reporter. Cependant, les chercheurs ont été forcés de sélectionner des gènes de reporter fluorescents ou de luciferase en raison de la capacité restreinte du génome de l'IAV pour inclure des séquences étrangères. Pour surmonter cette limitation, nous avons généré une réplication recombinante-compétente bi-reporter IAV (BIRFLU) exprimant de façon stable à la fois un gène fluorescent et un journaliste luciferase pour suivre facilement les infections par le VI in vitro et in vivo. À cette fin, les segments viraux non structuraux (NS) et hémagglutinine (HA) de la grippe A/Puerto Rico/8/34 H1N1 (PR8) ont été modifiés pour coder la Vénus fluorescente et les protéines bioluminescentes de luciférase Nanoluc, respectivement. Ici, nous décrivons l'utilisation de BIRFLU dans un modèle de souris de l'infection par le VI et la détection des deux gènes reporter à l'aide d'un système d'imagerie in vivo. Notamment, nous avons observé une bonne corrélation entre les expressions des journalistes et la réplication virale. La combinaison de techniques de pointe en biologie moléculaire, en recherche animale et en technologie d'imagerie offre aux chercheurs l'occasion unique d'utiliser cet outil pour la recherche sur la grippe, y compris l'étude des interactions virus-hôtes et la dynamique des infections virales. Fait important, la faisabilité de modifier génétiquement le génome viral pour exprimer deux gènes étrangers provenant de différents segments viraux ouvre la possibilité d'utiliser cette approche pour : (i) le développement de nouveaux vaccins anti-IAV, (ii) la génération de IAV recombinants qui peuvent être utilisés comme vecteurs vaccinaux pour le traitement d'autres infections pathogènes humaines.
Introduction
Le virus de la grippe A (IAV) est un virus d'ARN segmenté à sens négatif à un seul brin de la famille des Orthomyxoviridae 1,2,3. L'Organisation mondiale de la Santé (OMS) estime 3 à 5 millions de cas de grippe annuels et plus de 250 000 décès dus à la grippe dans le monde4,5,6. Les groupes particulièrement vulnérables à la grippe sont les personnes âgées, les personnes immunodéprimées et les enfants7,8,9,10,11. Bien que les vaccins soient disponibles et représentent l'intervention la plus courante et la plus efficace contre l'infection virale, le IAV est capable d'évoluer rapidement et d'échapper à l'immunité préexistante3,12,13, 14 (en) , 15. La réapparition d'une souche pandémique H1N1 en 2009 et l'émergence d'un IAV pathogène réitèrent la menace constante pour la santé publique humaine dans le monde4,16.
Lors d'une épidémie ou d'une pandémie, il est crucial de déterminer rapidement la pathogénicité et la transmissibilité des virus nouvellement isolés. Les techniques actuellement disponibles pour détecter le virus prennent beaucoup de temps et nécessitent parfois l'utilisation d'approches laborieuses, ce qui peut retarder l'achèvement de ces analyses17,18,19,20. En outre, les tests viraux actuels sont difficiles à intensifier, ce qui pourrait être nécessaire en cas d'éclosion. Enfin, l'utilisation de modèles animaux validés d'infection, tels que les souris, les cobayes et les furets, est couramment utilisée et est essentielle à l'étude des infections grippales, des réponses immunitaires et de l'efficacité de nouveaux vaccins et/ou antiviraux. Cependant, ces modèles sont restrictifs en raison de l'incapacité d'observer la dynamique virale en temps réel; cela limite les études à l'imagerie statique des infections virales21,22,23,24,25. Les animaux utilisés dans ces essais sont également euthanasiés afin de déterminer la charge virale, augmentant ainsi le nombre d'animaux nécessaires pour compléter ces études26. Pour contourner toutes ces limitations, de nombreux chercheurs s'appuient sur l'utilisation de recombinants réplication-compétents, journaliste-exprimant IAVs, qui sont capables d'accélérer les essais virologiques et de détecter la charge virale et la diffusion in vivo en temps réel26 ,27,28,29,30,31,32,33,34,35 ,36,37,38,39,40,41. Fait important, ces AIV exprimant des journalistes sont capables de se répliquer de la même façon que les AIV de type sauvage (WT) dans la culture cellulaire et dans les modèles animaux d'infection33,42.
Les protéines fluorescentes et bioluminescentes sont deux systèmes de reporter couramment utilisés par les chercheurs en raison de leur sensibilité, de leur stabilité et de leur facilité d'utilisation. En outre, il ya un soutien énorme et l'avancement dans les technologies de détection de protéines fluorescentes et bioluminescentes43,44,45,46,47,48 . Les protéines fluorescentes et la luciferase ont différentes propriétés qui leur permettent de briller, en particulier différentes dans la façon dont les états excités sont générés et comment l'émetteur est détecté43,44,45, 46,47,48. Les protéines fluorescentes sont d'abord excitées par l'absorption de l'énergie, qui est ensuite libérée sous forme de lumière à une longueur d'onde différente à mesure que les molécules diminuent à un état d'énergie inférieur43. D'autre part, la bioluminescence est dérivée d'une réaction chimique exothermic qui implique un substrat, l'oxygène, et parfois l'ATP afin de produire la lumière45. En raison des propriétés variables de ces deux types de protéines reporter, l'un peut-être plus avantageux que l'autre en fonction de l'étude de l'intérêt. Alors que les protéines fluorescentes sont largement utilisées pour observer la localisation cellulaire28,41, leurs signaux in vivo ont une intensité insuffisante et sont souvent obscurcis par l'autofluorescence dans les tissus vivants49. Par conséquent, les chercheurs s'appuient sur les luciferases pour évaluer la dynamique virale chez les organismes vivants, bien que les protéines fluorescentes peuvent être préférées pour les études ex vivo50,51,52,53. Contrairement aux protéines fluorescentes, les luciferases sont plus pratiques pour les études in vivo et plus applicables dans les approches non invasives26,27,28,29,30 , 31 Ans, états-unis ( , 32 Ans, états-unis ( , 33 Ans, états-unis ( , 34 Ans, états-unis ( , 35 Annonces , 36 Annonces , 37 Ans, états-unis ( , 38 Annonces , 39 Ans et plus qu'ils , 40 ans, états-unis ( , 41 Ans, états-unis ( , 54. En fin de compte, en fonction du type d'étude, les chercheurs doivent choisir entre l'utilisation d'une protéine fluorescente ou d'une protéine de journaliste de luciferase comme lecture, qui soumet leur étude à un compromis de fonctionnalités et de sensibilités, et sévèrement limite l'utilité des virus des journalistes recombinants. En outre, on s'inquiète de la corrélation entre l'expression de différents gènes de reporter utilisant des systèmes de fluorescence ou de luciferase et de réplication ou de diffusion virale, ce qui pourrait compromettre l'interprétation des données obtenues avec les VSI exprimant des journalistes.
Nous avons surmonté cette limitation en générant une réplication recombinante-compétente bi-reporter IAV (BIRFLU) qui code à la fois pour une protéine fluorescente et une luciférase dans le même génome viral55 (Figure 1). Ici, NanoLuc luciferase (Nluc), une petite protéine bioluminescentebrillante 48, a été insérée en amont de la séquence d'hémagglutinine (HA) dans le segment viral HA de la grippe A/Puerto Rico/08/1934 H1N1 (PR8)24,33, 40,55,56,57. En outre, Vénus, une protéine fluorescente monomeric fréquemment utilisée, a été insérée dans le segment viral non structurel (NS)32,33,36,41,55. Puisque BIRFLU code pour les gènes de journaliste fluorescents et de luciferase, le signal de protéine de journaliste peut être employé comme readout pour déterminer la réplication et la diffusion viraleines in vitro ou in vivo55. Des informations supplémentaires concernant la génération et la caractérisation in vitro ou in vivo de BIRFLU peuvent être trouvées dans notre récente publication55. BIRFLU peut être utilisé pour tester l'efficacité des médicaments antiviraux ou neutraliser les anticorps par le biais d'un nouvel essai de microneutralisation fluorescente et bioluminescente55. En outre, BIRFLU peut également être utilisé pour évaluer la dynamique virale dans un modèle de souris de l'infection55. Dans ce manuscrit, nous décrivons les procédures pour caractériser BIRFLU55 in vitro et comment étudier l'infection DE BIRFLU chez les souris utilisant des systèmes in vivo d'imagerie de luminescence pour la détection de Nluc in vivo ou de Vénus ex vivo.
La combinaison de techniques de pointe en biologie moléculaire, en recherche animale et en technologie d'imagerie offre aux chercheurs l'occasion unique d'utiliser BIRFLU pour la recherche sur les IAV, y compris l'étude des interactions virus-hôtes, la dynamique de l'infection virale; l'élaboration de nouvelles approches vaccinales pour le traitement thérapeutique des infections à VIH ou l'utilisation potentielle du VI comme vecteur vaccinal pour le traitement d'autres infections pathogènes.
Protocole
Tous les protocoles impliquant des souris ont été approuvés par le Comité institutionnel de soins et d'utilisation des animaux (IACUC) et le Comité de biosécurité institutionnelle (IBC) de l'Université de Rochester, École de médecine et de dentisterie. Toutes les expériences réalisées sur des animaux suivent les recommandations du Guide pour le soin et l'utilisation des animaux de laboratoire du Conseil national de recherches58. Le Vivarium and Division of Laboratory Animal Medicine facilities de l'École de médecine et de dentisterie de l'Université de Rochester est accrédité par l'Association for the Assessment and Accreditation of Laboratory Animal Care (AALAC) International et aux lois fédérales et des États et à la politique des National Institutes of Health (NIH). Un équipement de protection personnelle (EPI) approprié est nécessaire lorsque vous travaillez avec des souris. Des politiques et des exigences similaires devraient être mises en œuvre lors de l'exécution d'expériences décrites dans ce manuscrit dans chaque institution.
1. Utilisation de petits animaux vertébrés
- Achetez des souris BALB/c femelles de cinq à sept semaines et entretenez-les dans l'établissement de soins aux animaux dans des conditions spécifiques exemptes d'agents pathogènes. À l'arrivée des souris dans les installations déterminées, prévoyez une période de repos de 4 à 5 jours pour permettre aux animaux de s'acclimater à leur nouvel environnement.
- Suivant les protocoles de l'IACUC, placez un maximum de 5 souris par cage.
REMARQUE: À la fin de l'expérience, afin de s'assurer que l'animal est mort, les souris ont été euthanasiées à l'aide de deux procédures approuvées, en tenant compte du fait que la seconde doit être une méthode physique. Dans cette étude, après l'infection de souris avec BIRFLU et l'imagerie in vivo, les animaux sont euthanasiés avec une dose mortelle de 2,2,2-tribromoethanol (TBE), et en coupant la veine hépatique comme méthode secondaire physique comme nous l'avons précédemment montré23.
2. Biosécurité
REMARQUE: Dans ce manuscrit, BIRFLU a été généré dans l'épine dorsale de la grippe A/Puerto Rico/08/34 H1N1 (PR8), qui est une souche de laboratoire adaptée à la souris iAV23,32,33,56. Le virus a été généré à l'aide d'approches de génétique inverse à base de plasmides précédemment décrites et d'une description complète de la génération, et la caractérisation in vitro et in vivo de BIRFLU peut être trouvée dans notre récente publication55. Toutes les procédures impliquant des infections par le VI (in vitro ou in vivo) ont été effectuées dans un coffret de sécurité biologique dans des conditions de biosécurité (BSL)-2.
MISE EN GARDE: Un niveau de biosécurité approprié devrait être déterminé conformément à une évaluation des risques pour la biosécurité. Des renseignements supplémentaires sur l'exécution d'évaluations des risques liés à la biosécurité et l'établissement d'un confinement efficace de la biosécurité devraient être consultés auprès de l'établissement où les expériences seront effectuées.
- Nettoyez les armoires de biosécurité avec 70 % d'éthanol ou de désinfectant au dioxyde de chlore avant et après avoir effectué toutes les procédures expérimentales. Pour le travail des souris, stériliser tous les matériaux de dissection (ciseaux, forceps disséquants, etc.) et l'homogénéisateur Dounce avant et après leur utilisation.
- Jetez tout le matériel biologique produit au cours des procédures en vertu des directives appropriées du BAC et de l'IACUC.
3. Caractérisation in vitro de BIRFLU (figure 2)
REMARQUE: Consultez le tableau 1 pour toutes les compositions de mémoire tampon et de médias.
- Analyser l'expression des protéines par fluorescence (figure 2A) et l'immunofluorescence indirecte (figure 2B)
- Un jour avant l'infection, les plaques de semence24 puits avec madin-Darby Canine Kidney (MDCK) cellules épithéliales (1 x 105 cellules / puits, triplicates) dans les médias de culture tissulaire et de maintenir les plaques dans un incubateur de 37 oC avec 5% CO2. Préparez suffisamment de puits pour évaluer tous les anticorps choisis dans les cellules infectées par des simulacres et les cellules birFLU.
REMARQUE: Nous recommandons de visualiser les cellules sous un microscope léger pour confirmer une monocouche avant de commencer l'infection virale. Une monocouche de confluence de 90% des cellules de MDCK au moment de l'infection est recommandée. - Préparer des dilutions de WT ou BIRFLU IAVs dans les médias d'infection et infecter les cellules ensedutes MDCK avec une multiplicité d'infection (MOI) de 0,1 plaque-formation des unités (PFU) par cellule dans un volume final de 0,25 ml/puits de médias d'infection.
- Retirez le milieu de culture tissulaire de l'étape 3.1.1 et lavez les cellules MDCK deux fois avec 1x salin tamponné par phosphate (PBS). Ajouter les dilutions virales de 3.1.2 aux cellules MDCK et placer les plaques sur une plate-forme de bascule pendant 1 h à température ambiante pour permettre l'adsorption virale.
REMARQUE: Les cellules simulées ne sont incubées qu'avec des milieuis d'infection en l'absence de virus. - Après l'adsorption virale (étape 3.1.3), retirez l'inoculum viral par aspiration et ajoutez 1 ml de support post-infection contenant 1 g/mL de trypsine TPCK-traitée à chaque puits. Incuber les cellules pendant 18 h dans un incubateur humidifié co2 à 33 oC.
- Après 18 h d'infection, retirez le supernatant de culture tissulaire des plaques de 24 puits (3.1.4). Fixer et perméabiliser les cellules avec 0,25 ml/puits de solution de fixation/perméabilisation pendant 20 min à température ambiante.
REMARQUE: Préparer la solution de fixation/perméabilisation dans un capuchon de fumée pour éviter l'exposition au formaldéhyde. - Retirez la solution de fixation/perméabilisation de l'étape 3.1.5, lavez les cellules deux fois avec 1x PBS, et incubez les cellules avec 0,25 ml/puits de solution de blocage pendant 1 h à température ambiante.
REMARQUE: Passez à l'étape suivante ou entreposez les cellules dans une solution de blocage à 4 oC pendant la nuit. - Retirez la solution de blocage (étape 3.1.6) et ajoutez 0,25 ml/puits (1 g/mL) d'un anticorps monoclonal de souris (MAb) contre la nucléoprotéine IAV, NP (HB-65) ou une dilution de 1:1 000 d'un anticorps polyclonal de lapin (pAb) contre NLuc (voir la Table des Matériaux), tous deux dilués dans la solution de dilution d'anticorps (1x PBS, 2,5% BSA), et incubent les cellules pendant 1 h à 37 oC.
- Retirez l'anticorps primaire de l'étape 3.1.7, lavez les cellules trois fois avec 1x PBS et incubez-les avec 0,25 ml/puits d'un anti-souris ou d'anticorps secondaires anti-lapins du Texas (voir le Tableau des matériaux) dilué 1:200 dans l'anticorps solution de dilution.
REMARQUE: Les noyaux cellulaires peuvent être tachés en même temps en ajoutant 0,5 g/mL de 4',6'-diamidino-2-phenylindole (DAPI) à la même solution d'anticorps secondaire. Incuber les cellules pendant 1 h à 37 oC dans l'obscurité. D'autres anticorps secondaires conjugués à d'autres fluorophores peuvent être utilisés. - Retirez l'anticorps secondaire et Le DAPI de l'étape 3.1.8, lavez les cellules trois fois avec 1x PBS. Après le lavage, laisser les cellules dans 0,25 ml/puits de 1x PBS.
- Placez la plaque sur le stade d'un microscope à fluorescence pour détecter l'expression des journalistes de Vénus et Nluc, et NP à partir de cellules infectées à l'aide des filtres fluorescents appropriés. Capturez des images à l'aide d'un microscope à fluorescence (grossissement 20x) et fusionnez-les à l'aide d'un logiciel d'édition d'images (Figure 2A, B).
- Un jour avant l'infection, les plaques de semence24 puits avec madin-Darby Canine Kidney (MDCK) cellules épithéliales (1 x 105 cellules / puits, triplicates) dans les médias de culture tissulaire et de maintenir les plaques dans un incubateur de 37 oC avec 5% CO2. Préparez suffisamment de puits pour évaluer tous les anticorps choisis dans les cellules infectées par des simulacres et les cellules birFLU.
- Analyser l'activité du Nluc (Figure 2C) et la réplication virale (Figure 2D).
- Un jour avant l'infection, les plaques de semences de 12 puits avec des cellules MDCK (2 x 105cellules/bien, tripliciates) utilisant des supports de culture tissulaire dans un incubateur de 37 oC avec 5 % de CO2 pour atteindre environ 90 % de confluency au moment de l'infection.
REMARQUE: Avant l'infection, vérifiez les cellules au microscope pour vérifier une monocouche de cellules MDCK. - Le jour de l'infection prépare des dilutions des virus WT et BIRFLU dans les médias d'infection pour infecter les cellules MDCK (étape 3.2.1.) dans le triplement avec un MOI de 0.001 PFU dans 0.5 mL/puits. Enlever le milieu de culture tissulaire et laver les cellules MDCK deux fois avec 1x PBS.
- Ajouter les dilutions virales aux monocouches MDCK et permettre l'adsorption virale à température ambiante pendant 1 h sur une plate-forme à bascule. Après l'adsorption virale, retirez l'inoculum du virus et ajoutez 1,5 ml de support post-infection contenant 1 g/mL de trypsine traitée par TPCK, à chaque puits. Incuber les cellules infectées dans un incubateur humidifié au CO2 de 5 % à 33 oC et recueillir des supernatants aux points indiqués à l'étape 3.2.4.
- À 24, 48, 72 et 96 h après l'infection (p.i.) recueillir 150 L de supernatant de culture tissulaire de chaque puits et stocker les échantillons dans un tube de microcentrifuge à -80 oC pour effectuer les essais Nluc et les titres viraux.
- Pour effectuer l'exemple d'activité Nluc, suivez les indications du fabricant. Tout d'abord, décongeler le supernatant de culture tissulaire stocké à -80 oC (étape 3.2.4) sur la glace.
REMARQUE: Reportez-vous aux recommandations du fabricant et optimisez l'analyse si nécessaire. - Préparer la solution d'analyse de luciferase (voir la Table des Matériaux) en diluant le substrat Nluc 1:50 avec le tampon de dilution. Dans une microplaque blanche à fond plat de 96 puits pour les essais de luciferase, mélangez 25 ll de solution d'analyse de luciferase avec 10 à 25 l des échantillons de supernatant de culture de tissu recueillis. Incuber le mélange pendant 2 à 3 minutes avant de mesurer la luminescence à l'aide d'un luminomètre (figure 2C).
REMARQUE: La présence de particules virales infectieuses dans la culture tissulaire des supernatants est déterminée par l'analyse immunitaire-focus de fluorescence (Vénus) ou l'immunofluorescence indirecte (coloration virale d'antigène) comme précédemment décrit23,32, 33,56. - Un jour avant les titrations virales, les cellules de Semence MDCK dans une plaque de 96 puits (2 x 104 cellules/puits, tripliques) avec des supports de culture tissulaire et permettent aux cellules d'atteindre 90% de confluence au moment de l'infection dans un incubateur réglé à 37 oC avec 5% de CO2.
- Utilisez les échantillons de supernatant de culture tissulaire qui ont été décongelés sur la glace (étape 3.2.4.). Ajouter 90 lde de support d'infection à chacun des puits dans une nouvelle plaque de 96 puits, puis ajouter 10 l de la culture tissulaire décongelée supernatant (étape 3.2.4) au premier puits (rangée A). Utiliser une tuyauterie multicanal pour mélanger et transférer 10 L de la rangée A à la rangée B. Changez les pointes entre les dilutions et mélangez bien.
REMARQUE: Cette procédure doit être répétée jusqu'à la dernière rangée (H). Nous recommandons d'effectuer des titrations virales dans des tripliques pour une mesure précise des titres viraux. - Retirez le milieu de culture tissulaire des cellules MDCK dans les plaques de 96 puits (étape 3.2.7) et lavez deux fois avec 1x PBS. Ajouter 50 L des dilutions supernatant de la plaque de 96 puits contenant les dilutions virales (étape 3.2.8) à chaque puits dans la plaque de 96 puits contenant des cellules MDCK.
- Incuber la plaque de 96 puits sur une plate-forme à bascule pendant 1 h à température ambiante pour permettre l'adsorption virale. Après l'adsorption virale, retirez l'inoculum et ajoutez 100 L/puits de support post-infection contenant 1 g/mL de trypsine traitée par TPCK. Incuber les cellules infectées pendant 12 h dans unincubateur de 33 oC avec 5 % de CO 2.
- Depuis BIRFLU exprime Vénus, le nombre de cellules infectées peut être directement compté à l'aide d'un microscope à fluorescence. Alternativement, les titreleurs viraux peuvent être déterminés par immunofluorescence indirecte comme précédemment décrit utilisant un anticorps contre une protéine virale23,56,57,59. Pour ce dernier, procéder à la correction /perméabilize des cellules et la tache en utilisant l'anti-NP mAb HB-65 tel que décrit dans la section 4.4.
- Déterminer les titreleurs viraux en comptant le nombre d'unités fluorescentes (FFU)/mL à l'aide de la formule : ((nombre de FFU) x 20 x 1/dilution (Figure 2D).
- Un jour avant l'infection, les plaques de semences de 12 puits avec des cellules MDCK (2 x 105cellules/bien, tripliciates) utilisant des supports de culture tissulaire dans un incubateur de 37 oC avec 5 % de CO2 pour atteindre environ 90 % de confluency au moment de l'infection.
4. Caractérisation in Vivo de BIRFLU (Figure 3 et Figure 4)
- Infection de souris
REMARQUE : L'infection intranasale des souris a été faite comme précédemment décrit23. Pour un protocole plus détaillé de l'infection par le VI in vivo en utilisant le modèle de souris de l'infection, nous vous recommandons de visionner la vidéo associée à la publication précédente23. Cette section résume seulement les étapes requises pour l'infection de souris avec BIRFLU.- Inspecter les souris pour évaluer leur santé et leur apparence physique globale. Préparer la dilution de BIRFLU en 1x PBS pour inoculer les souris avec 1 x 106 PFU de BIRFLU dans un volume total de 30 L/souris. Maintenir le virus sur la glace jusqu'à l'inoculation de la souris.
REMARQUE: Des souris simulées (1x PBS) seront nécessaires comme contrôle interne pour l'imagerie. Placez des souris simulées dans une cage différente de celle des animaux infectés par le BIRFLU. - Anesthésiez des souris BALB/c femelles de cinq à sept semaines par voie intrapéritone avec 240 à 250 mg/kg de tribromoéthanol (TBE) en insérant l'aiguille dans le caudal 2/3 du côté droit de l'abdomen. Ensuite, retournez la souris dans la cage et attendez environ 5 min. Vérifiez que la souris est anesthésié avant l'inoculation du virus par l'absence du réflexe de pincement des orteils.
REMARQUE: Les sédatifs injectables sont recommandés sur les sédatifs inhalés pour les infections grippales in vivo, car ces derniers peuvent modifier le comportement et l'utilisation du virus par l'épithélium des voies respiratoires. En outre, ils peuvent également affecter les réponses immunitaires locales du poumon et interférer avec l'imagerie in vivo des souris infectées. Enfin, les sédatifs inhalés ont réduit la durée d'effet, ce qui constituerait un problème pour l'imagerie in vivo des animaux infectés. - Inoculer les souris intranasally avec les 30 L de la dilution BIRFLU préparée. Vérifiez que les souris respirent correctement avant de les retourner à la cage.
- Inspecter les souris pour évaluer leur santé et leur apparence physique globale. Préparer la dilution de BIRFLU en 1x PBS pour inoculer les souris avec 1 x 106 PFU de BIRFLU dans un volume total de 30 L/souris. Maintenir le virus sur la glace jusqu'à l'inoculation de la souris.
- Surveillance de la bioluminescence des souris infectées par BIRFLU (Figure 4A)
REMARQUE: Dans ce manuscrit, l'expression de Nluc ou de Vénus et la réplication virale dans les poumons de souris infectées par BIRFLU sont déterminées à 3 jours après l'infection. Cependant, la nature de l'imagerie par bioluminescence permet une surveillance répétée de l'expression de Nluc chez des animaux infectés par le BIRFLU sans qu'il soit nécessaire de les euthanasier pour chaque période expérimentale. Un système de formation image in vivo avec un collecteur d'anesthésie d'isoflurane est exigé pour exécuter les procédures expérimentales décrites. Consultez le Tableau des matériaux pour plus de détails concernant l'instrument d'imagerie et le logiciel d'image utilisés pour l'acquisition d'images et l'analyse des données.- Raser la poitrine des souris pour améliorer le signal de bioluminescence. Ouvrez le logiciel d'imagerie et appuyez sur Initialize. Ensuite, définiz les paramètres qui seront utilisés, y compris la mise en mode d'imagerie à la bioluminescence, l'économie automatique, le temps d'exposition à l'automobile, filtre ouvert, etc.
- Une fois que la machine est entièrement paraphée, allumez le système d'anesthésie isoflurane. Placer les animaux dans la chambre d'anesthésie. Les souris sont simultanément et légèrement anesthésiées avec un mélange de gaz d'oxygène et d'isoflurane vaporisé de 1 à 2 %.
- Une fois que les souris sont anesthésiées, administrer le substrat Nluc (voir le Tableau des matériaux)dilué 1:10 en 1x PBS (volume final 100 l/souris) par voie rétro-orbitale à l'aide d'une seringue avec une aiguille de 22 G.
- Immédiatement après l'administration du réactif Nluc, placez les animaux dans l'instrument d'imagerie avec leurs poitrines orientées vers le haut et le museau à l'intérieur du cône multiple pour garder les animaux anesthésiés pendant l'imagerie. Immédiatement après la fermeture de la porte de l'imageur, cliquez sur Acquérir dans le logiciel (Figure 4A, en haut).
- Après l'imagerie, renvoyez les souris dans leurs cages en les surveillant jusqu'à ce qu'elles aient complètement récupéré et éteignez le vaporisateur d'isoflurane. Ensuite, procédez à l'imagerie ex vivo des poumons de souris pour évaluer l'expression du gène de journaliste de Vénus (section 4.3).
- Utilisez les outils logiciels d'imagerie pour analyser les données de bioluminescence acquises. Utiliser l'outil ROI (région d'intérêt) pour désigner le signal spécifique et effectuer des mesures de flux dans la région d'intérêt (généralement autour de la poitrine) (Figure 4A, en bas). Bien que la forme du retour sur investissement ne soit pas pertinente, les iA plus importants sont généralement préférables pour capturer toute la zone de diffusion du signal.
- Cliquez sur Mesure. Évaluer la bioluminescence dans les photons puisqu'elle fournit une mesure absolue des émissions de photons qui est comparable aux mesures de sortie fournies par différents paramètres ou instruments d'imagerie.
- Analyse de fluorescence chez les souris infectées par BIRFLU (Figure 3 et Figure 4B)
- Recueillir les poumons de la souris après l'imagerie in vivo, comme précédemment décrit23.
- En bref, euthanasiez les souris avec une dose mortelle de TBE (500 mg/kg). Désinfecter le site d'incision avec 70% d'éthanol. À l'aide d'un scalpel, faire une incision du sternum à la base de l'abdomen, puis couper du bas de l'incision sur les côtés avec des ciseaux. Ensuite, couper la veine hépatique (deuxième méthode d'euthanasie physique) pour saigner l'animal.
REMARQUE: Pour éviter le signal de fond élevé pendant l'imagerie, il est important de réduire au minimum la quantité de sang dans les poumons (voir ci-dessous).
- En bref, euthanasiez les souris avec une dose mortelle de TBE (500 mg/kg). Désinfecter le site d'incision avec 70% d'éthanol. À l'aide d'un scalpel, faire une incision du sternum à la base de l'abdomen, puis couper du bas de l'incision sur les côtés avec des ciseaux. Ensuite, couper la veine hépatique (deuxième méthode d'euthanasie physique) pour saigner l'animal.
- Placez la souris dans la charge dorsale, et utilisez les ciseaux pour couper la pleure et ouvrir la cage thoracique. Par la suite, retirer les poumons en arrachant l'extrémité de la trachée avec des ciseaux tout en tenant doucement les poumons avec des forceps.
- Placer les poumons dans une plaque de six puits avec 2 ml de 1x PBS et laver les poumons trois fois avec 1x PBS.
REMARQUE: Pour éviter la contamination entre les échantillons, nettoyez et désinfectez les outils de dissection entre chaque animal.
- Placer les poumons dans une plaque de six puits avec 2 ml de 1x PBS et laver les poumons trois fois avec 1x PBS.
- Démarrez le logiciel d'acquisition d'images en cliquant sur initialiser et définir les paramètres pour l'imagerie, y compris le réglage du mode d'imagerie à la fluorescence, l'économie automatique, le temps d'exposition à l'automobile, l'excitation (500 nm) et les filtres d'émission (540 nm).
- Lorsque la machine est paraphée, placez les poumons dans un plateau de fond noir, en veillant à ce que les tissus sont séparés les uns des autres, introduisez le plateau dans l'imageur, et cliquez sur Acquérir sur le programme du système d'imagerie après la fermeture de la porte de l'imageur ( Figure 4B, en haut).
- Après l'imagerie, retirer immédiatement les tissus et les conserver sur de la glace (4 oC) si les échantillons sont traités le jour même; ou sur un tube et de la glace sèche pour les congeler rapidement, avant de les stocker à -80 oC, si les échantillons seront traités plus tard un autre jour.
- Pour le traitement de l'image, sélectionnez l'outil de retour sur investissement et dessinez des ROIs autour de chacun des poumons individuels. Cliquez sur Mesure. Ensuite, à l'aide des mesures moyennes d'efficacité radiante qui en résultent, soustrayez les valeurs des souris infectées par une simulacre (figure4B,en bas).
REMARQUE: Avec BIRFLU, il ya une bonne corrélation des niveaux d'expression De Nluc et Vénus, et la distribution du signal. Par conséquent, il est important d'analyser l'expression de Nluc (souris entière) et De vénus (poumons excisés) du même animal, et de maintenir la même orientation.
- Recueillir les poumons de la souris après l'imagerie in vivo, comme précédemment décrit23.
- Évaluation de la réplication DU BIRFLU dans les poumons des souris (figure3 et figure 4C)
- Homogénéiser les poumons des souris pour la titration virale par l'assay de plaque comme précédemment décrit23.
- Placer les poumons dans un homogénéisateur Dounce avec 1 ml de support d'infection réfrigérée. Homogériser les poumons avec le pilon pendant 1 min à température ambiante jusqu'à ce qu'il se soit complètement désintégré et stocker les échantillons dans un tube stérile à 4 oC.
- Centrifuger les échantillons à 300 x g pendant 10 min et recueillir le supernatant dans un tube stérile. Entreposez les échantillons sur la glace s'ils seront utilisés le même jour ou congelez-les à -80 oC pour évaluer les titreleurs viraux à une date ultérieure.
REMARQUE: Un nouvel homogénéisateur Dounce doit être utilisé pour chaque échantillon pulmonaire. - Pour effectuer l'analyse de plaque, la veille de l'exécution de la titration virale, les plaques de semence de six puits avec des cellules MDCK (5 x 105 cellules/ bien) dans les médias de culture tissulaire. Incuber les cellules pendant la nuit à 37 oC avec 5 % de CO2 pour atteindre 90 % de confluence le lendemain. Avant l'infection, confirmer que les cellules forment une monocouche sous un microscope léger.
- Préparer 1:10 dilutions sérielles du supernatant à partir des échantillons homogénéisés (étape 4.4.1) dans les médias d'infection. Préparer les tubes de microcentrifugeavec 540 l de supports d'infection, ajouter 60 L de l'échantillon de poumons homogénéisés au premier tube, mélanger en pipetting de haut en bas, puis en utilisant une nouvelle pointe, transférer 60 'L au tube suivant. Répétez ce processus de dilution en série jusqu'à la dernière dilution.
REMARQUE: Dans notre expérience 7 dilutions sont suffisantes pour déterminer les titreleurs viraux par l'assay de plaque des poumons des souris infectées. - Laver les cellules MDCK (étape 4.4.3) deux fois avec 1x PBS et transférer 500 L des échantillons dilués en série à chaque puits dans la plaque de six puits. Placer les plaques sur une plate-forme de bascule et permettre l'absorption virale de se produire pendant 1 h à température ambiante.
- Après 1 h d'absorption virale, retirez l'inoculum viral, ajoutez 2 mL/puits de milieu de superpose contenant 1 g/mL de trypsine traitée par TPCK, puis incubez les cellules à 33 oC sous 5 % de CO2 pendant 3 jours.
- Fixer les cellules infectées (étape 4.4.6) avec 1 ml/puits de 4% de formaldéhyde dilué dans 1x PBS à température ambiante pendant 2 h, puis retirer soigneusement le milieu de superposition et ajouter 1 ml de 1x PBS à chaque puits. Pour la visualisation de Vénus, imagez les plaques de six puits avec un système d'imagerie pour la détection de la fluorescence.
REMARQUE: Les plaques virales peuvent être colorées à l'aide d'un logiciel d'édition d'images (voir le Tableau des matériaux). - Pour évaluer l'expression de Nluc, visualisez les plaques virales en immunostaining utilisant un pAb anti-Nluc. Pour cela, retirer les 1x PBS, les cellules perméabilize en utilisant 0,5 ml/puits de solution de perméabilisation pendant 15 min à température ambiante.
- Retirez la solution de perméabilisation, lavez les cellules trois fois avec 1x PBS et bloquez avec 0,5 ml/puits de solution de blocage pendant 1 h à température ambiante.
- Retirez la solution de blocage de l'étape 4.4.9 et incubez les cellules avec 0,5 ml/puits de l'anti-Nluc pAb dilué 1:1,000 dans la solution de dilution pendant 1 h à 37 oC.
- Utilisez le kit ABC Alkaline Phosphatase et le Kit substrat Peroxidase DAB suivant la recommandation du fabricant pour la visualisation des plaques exprimant Nluc.
- En bref, laver les cellules à partir de 4,4,10 trois fois avec 1x PBS et les incuber avec 0,5 ml/puits d'anticorps secondaires anti-lapin biotinylated pendant 1 h à 37 oC.
- Retirez l'anticorps secondaire, lavez les cellules trois fois avec 1x PBS, et incubez avec la solution ABC pendant 1 h à 37 oC. Laver les cellules trois fois avec 1x PBS et visualiser les plaques virales avec le kit de substrat DAB HRP. Scanner les plaques immunostained à l'aide d'un scanner conventionnel.
REMARQUE: D'autres kits similaires pour l'immunostaining pourraient être utilisés.
- Tachez les plaques virales avec une solution violette cristalline pendant 1 h à température ambiante. Jeter la violette de cristal, laver les assiettes trois fois avec de l'eau, laisser sécher les assiettes et scanner à nouveau les assiettes.
- Pour déterminer les titreneurs viraux, comptez les plaques révélées après la coloration de la violette cristalline. Scanner les plaques à l'aide d'un scanner conventionnel. Calculer le titre de virus sous forme d'unités de formation de plaque (PFU) par mL (PFU/mL).
- Pour évaluer la stabilité birFLU in vivo, calculez le pourcentage de virus exprimant des journalistes en comptant le nombre de plaques teintées de violette cristalline (nombre de virus infectieux, étape 4.4.12) et comparez avec le nombre de plaques exprimant Vénus et Nluc ( l'étape 4.4.7 et l'étape 4.4.11, respectivement).
- Centrifuger les échantillons à 300 x g pendant 10 min et recueillir le supernatant dans un tube stérile. Entreposez les échantillons sur la glace s'ils seront utilisés le même jour ou congelez-les à -80 oC pour évaluer les titreleurs viraux à une date ultérieure.
Résultats
Génération et caractérisation de BIRFLU in vitro (figure 1 et figure 2)
Un IAV recombinant réplication-compétent exprimant deux gènes différents de journaliste (BIRFLU) a été construit utilisant la biologie moléculaire de l'état de l'art et les techniques de génétique inverse plasmides-basées (figure1). Ici, nous avons choisi d'utiliser Nluc en raison de plusieurs avantages par rapport à d'autres luciferases, y compris sa petite taille, ATP-indépendance, une plus grande intensité, et le substrat optimisé48,60. Nluc a été cloné dans le segment HA de l'IAV PR8, suivi du site de clivage porcin teschovirus (PTV) 2A (2A) devant le cadre de lecture ouvert (ORF) de HA (Figure 1). L'ORF de HA comprenait des mutations silencieuses pour enlever les signaux d'emballage d'origine et éviter toute recombinaison possible. Le signal complet d'emballage HA a été ajouté devant Nluc pour permettre l'incorporation appropriée du segment HA modifié dans la virion et l'expression Nluc et HA du même segment d'ARN viral (Figure 1). En outre, la protéine fluorescente Vénus a été clonée dans un segment modifié IAV PR8 NS qui code les deux protéines virales NS1 et NEP à partir d'une transcription unique32,36,41,54, 57. À cette fin, Vénus a été fusionnée au terminal C de NS1 et l'ensemble du NEP ORF a été cloné en aval du site de clivage PTV 2A qui a été placé entre les séquences NS1-Venus et NEP (Figure 1). En fin de compte, ces deux constructions modifiées de plasmide viral HA et NS ont été utilisées en combinaison avec le reste des plasmides de génétique inverse iAV PR8 pour générer BIRFLU (Figure 1). La caractérisation in vitro et in vivo de BIRFLU a été décrite précédemment55.
Dans la figure 2, nous avons caractérisé le BIRFLU in vitro en déterminant les niveaux d'expression de Vénus, de Nluc et de NP en utilisant des approches de fluorescence et d'immunofluorescence indirecte (figure2A,B). Les monocouches de confluents des cellules MDCK ont été soit simulées ou infectées (MOI 0.1) avec des virus WT ou BIRFLU PR8 et, à 18 h après l'infection, l'expression de Vénus a été directement évaluée à l'aide de la microscopie à fluorescence (figure 2A,B). L'expression Nluc (Figure 2A) et NP (Figure 2B) ont été visualisées par immunofluorescence indirecte à l'aide d'anticorps spécifiques pour chaque protéine. Comme prévu, l'expression de Vénus et de Nluc n'a été détectée que dans les cellules infectées par BIRFLU et non dans les cellules infectées par le virus WT PR8. En outre, la microscopie indirecte d'immunofluorescence a indiqué l'expression de NP dans les cellules WT et BIRFLU PR8-infectées. Aucune expression de Vénus, De Nluc ou de NP n'a été détectée, comme prévu, dans des cellules simulées infectées (figureA,B).
Pour évaluer les niveaux d'expression de Nluc in vitro, les cellules MDCK ont été infectées (MOI 0.001) avec des virus WT ou BIRFLU PR8 et l'activité de Nluc dans les supernatants de culture tissulaire a été évaluée à 24, 48, 72 et 96 h après l'infection (figure 2C). Seule l'activité de Nluc a été détectée chez les supernatants de culture tissulaire des cellules MDCK infectées par BIRFLU (figure 2C). L'activité de Nluc dans les supernatants de culture de tissu a été détectée dès 24 h après l'infection avec des niveaux d'expression plus élevés à 96 h après l'infection, probablement parce que l'effet cytopathique (CPE) induit pendant la libération virale d'infection la protéine de Nluc a conservé dans la cellule. Pour évaluer l'aptitude du BIRFLU dans les cellules cultivées, la cinétique de croissance des virus WT et BIRFLU PR8 a également été évaluée (Figure 2D) et la présence de virus infectieux dans les supernatants de culture tissulaire a été déterminée par l'analyse du foyer immunitaire (figure2D ). Notamment, la cinétique de réplication de BIRFLU était comparable à ceux du virus de PR8 de WT, bien que la réplication de BIRFLU ait été légèrement retardée et n'ait pas atteint les mêmes titers viraux que WT PR8. Cependant, BIRFLU a été en mesure d'atteindre des titreleurs de 5 x 107 PFU/mL (Figure 2D), ce qui indique que l'expression de deux gènes reporteurs dans le génome viral n'interfère pas de façon significative avec la réplication du BIRFLU dans les cellules MDCK.
Suivi de l'infection À BIRFLU chez la souris(figure3 et figure 4)
La figure 3 est un diagramme de flux schématique pour l'évaluation de la dynamique BIRFLU dans un modèle de souris de l'infection par le VI. Des souris femelles de CINQ à sept semaines de BALB/C ont été simulées-infectées avec 1x PBS ou infectées avec 1 x 106 PFU de BIRFLU intranasally. À 3 jours après l'infection, des souris ont été anesthésiées avec l'isoflurane et puis injectées avec le substrat de Nluc rétro-orbitalement. Toutes les souris ont été immédiatement placées dans l'instrument IVIS et le signal de Nluc a été évalué in vivo à l'aide de l'IVIS. Ensuite, les souris ont été euthanasiées et les poumons ont été récoltés. Les poumons excisés ont ensuite été analysés ex vivo à l'aide de l'imageur in vivo pour déterminer l'intensité de fluorescence par l'expression de Vénus. Enfin, les poumons de souris ont été homogénéisés, et les titres et la stabilité virales ont été déterminées par l'analyse de plaque. Les plaques ont été évaluées par la fluorescence directe de Vénus, par immunostaining utilisant des anticorps spécifiques pour Nluc et par la coloration de violette cristalline.
Les IAV exprimant des iAVs qui expriment des informations de réplication précédemment, qui expriment des iAV, expriment un seul gène de journaliste, le plus souvent une protéine fluorescente ou bioluminescente, comme substitut de l'infection virale et de la réplication. Cependant, BIRFLU, est capable d'exprimer les deux types de gènes reporter sur l'infection virale. Pour évaluer la corrélation entre la bioluminescence (imagerie in vivo) et la fluorescence (imagerie ex vivo) après l'infection BIRFLU, des souris balb/c femelles de cinq à sept semaines ont été simulées infectées par 1 x PBS ou inoculées par BIRFLU (106 PFU) intranasalement . L'activité de Nluc (figure 4A) a été évaluée par l'administration du substrat de Nluc injecté rétro-orbitalement à 3 jours après l'infection utilisant un instrument d'imagerie in vivo. Nous avons choisi d'évaluer la bioluminescence au jour 3 parce que les études précédentes indiquaient que la réplication du IAv, y compris le PR8, culmine entre les jours 2 et 4 après l'infection24,54. La bioluminescence a été surveillée (figure4A, en haut) et utilisée pour calculer le flux total moyen (Flux (log10 p/s) (Figure 4A, en bas). Comme prévu, les souris inoculées avec BIRFLU ont montré une activité de bioluminescence élevée, mais aucun signal n'a été détecté chez les souris infectées par un simulacre. Par la suite, les poumons des souris infectées ont été récoltés et l'expression de Vénus a été évaluée à l'aide de l'imagerie ex vivo (figure4B, en haut). De plus, l'efficacité radiante moyenne de fluorescence a été calculée (Figure 4B, en bas). Les poumons de souris excisées ont également été homogénéisés pour déterminer les titreleurs viraux et la stabilité génétique de BIRFLU in vivo (Figure 4C, D). La stabilité génétique de BIRFLU a été analysée par l'analyse de plaque utilisant les virus isolés des poumons de souris et la microscopie fluorescente (Vénus, dessus), l'immunostaining (Nluc, milieu) et la coloration de violette cristalline (en bas). BIRFLU récupéré à partir de souris poumons ont été en mesure de former des plaques et exprime de façon stable les deux gènes journaliste (Figure 4C). Notamment, nous avons observé une bonne corrélation entre les signaux de bioluminescence et de fluorescence avec la réplication virale.
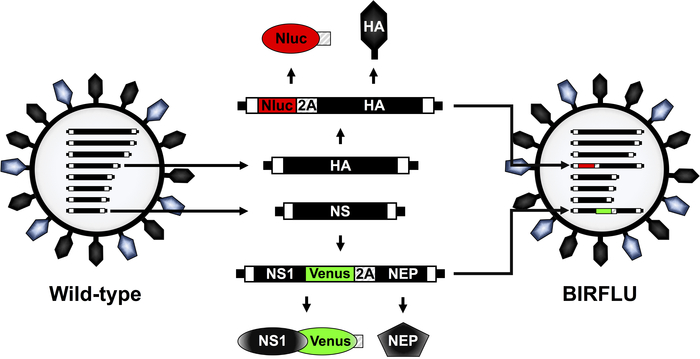
Figure 1 : Représentation schématique de la structure de virion IAV PR8 et BIRFLU et des segments génomiques. Les IAV sont entourés d'une couche lipidique contenant les deux principales glycoprotéines virales hémagglutinine (HA; noir) et neuraminidase (NA; bleu). Le IAV contient huit segments d'ARN à simple brin, à sens négatif (PB2, PB1, PA, HA, NP, NA, M et NS). Chaque segment viral contient des régions non codantes (NCR) aux extrémités 3' et 5' (boîtes noires). En outre, à la fin 3' et 5' des viraux (v)RNAs sont les signaux d'emballage, responsables de l'encapsidation efficace des vRNAs en virions naissantes (boîtes blanches). Les segments et produits viraux IAV PR8 HA et NS sont indiqués en noir. Les séquences de Nluc, Devénus et PTV 2A sont indiquées dans des boîtes rouges, vertes et rayées, respectivement. La représentation schématique des segments HA et NS modifiés exprimant Nluc et Vénus, respectivement, dans BIRFLU sont également indiquées. Ce chiffre a été adapté de Nogales et coll.55. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 2 : Caractérisation in vitro de BIRFLU. (A, B) Analyse de l'expression des protéines par fluorescence directe et immunofluorescence. Les cellules MDCK ont été simulées ou infectées (MOI 0.1) par des virus PR8 WT ou BIRFLU. Les cellules infectées ont été fixées à 18 h après l'infection pour visualiser directement l'expression de Vénus par microscopie fluorescente directe et pour visualiser l'expression Nluc (A) et virale NP (B) utilisant des anticorps spécifiques et l'immunofluorescence indirecte. Les noyaux ont été tachés avec DAPI. Des images représentatives (20x grossissement) sont affichées. Barres d'échelle de 100 m. (C, D) Cinétique de croissance de PR8 WT et BIRFLU. L'activité de Nluc (C) et les titres viraux (D) dans les supernatants de culture de tissu des cellules de MDCK infectées (MOI 0.001) avec wT et BIRFLU PR8 des virus ont été évalués aux heures indiquées après l'infection. Les données représentent les moyens de SD des tripliques. Les titrages viraux ont été déterminés par l'analyse immunisée-focus (FFU/mL). La ligne pointillée indique la limite de détection (200 FFU/ml). Ce chiffre a été adapté de Nogales et coll.55. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 3 : Représentation schématique pour l'étude du BIRFLU chez la souris. L'expression des gènes de journaliste de Nluc et de Vénus a été évaluée chez des souris infectées par 1 x 106 PFU de BIRFLU à l'aide d'imagerie in vivo ou ex vivo. En bref, le jour 1, 5 à 7 souris femelles de BALB/c de semaine ont été simulées-infectées (1x PBS) ou inoculées avec 1 x 106 PFU de BIRFLU intranasally. Au jour 3 après l'infection, des souris ont été légèrement anesthésiées utilisant l'isoflurane et le substrat de Nluc a été injecté rétro-orbitalement. Le signal de Nluc a été directement évalué utilisant l'imagerie in vivo. Immédiatement après la formation image, des souris ont été euthanasiées et l'expression de Vénus dans les poumons excisés entiers a été analysée utilisant la formation image ex vivo. Les poumons récupérés de souris ont été homogénéisés pour évaluer la réplication et la stabilité virales par l'analyse de plaque. Les flèches indiquent une corrélation entre la fluorescence (Vénus), l'immunostaining (Nluc) et la coloration de la violette cristalline. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
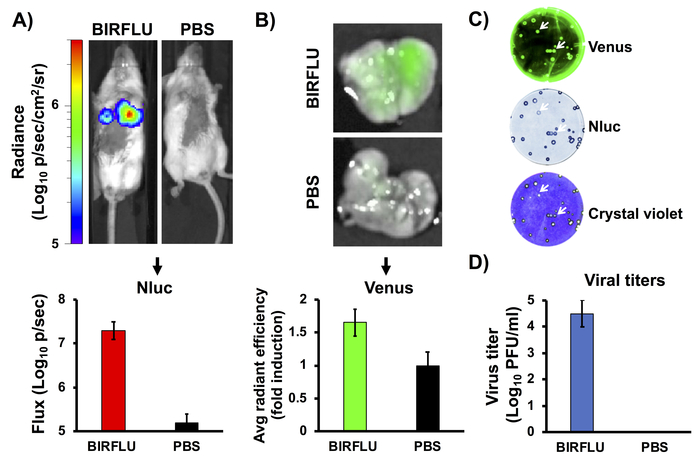
Figure 4 : Expression de bioluminescence et de fluorescence in vivo. Les souris femelles de CINQ à sept semaines de BALB/c ont été simulées-infectées (1x PBS) ou inoculées avec 1 x 106 PFU de BIRFLU intranasally. Au jour 3 post-infection, l'activité de Nluc (A) dans la souris entière a été déterminée. Images représentatives d'une souris unique montrant l'échelle d'éclat (p/sec/cm2/sr). Les valeurs d'éclat de bioluminescence ont été quanties et le flux total moyen est indiqué (Flux (Log10 p/s). Après l'imagerie De Nluc, les poumons ont été récoltés pour l'imagerie ex vivo (B). Des images représentatives de poumons entiers sont montrées. Pour quantifier l'expression de Vénus, les valeurs moyennes des régions d'intérêt (ROIs) ont été normalisées à l'autofluorescence pulmonaire des souris simulées-infectées et des changements de pli ont été calculés. Pour analyser la stabilité génétique de BIRFLU in vivo, les virus récupérés dans les poumons des souris ont été analysés par test de plaque à l'aide d'une microscopie fluorescente (Vénus, en haut), d'immunostaining (Nluc, milieu) et de coloration violette cristalline (en bas) (C). Des images représentatives d'une souris sont affichées. Pour évaluer la réplication du virus, des poumons entiers ont été homogénéisés après l'imagerie et utilisés pour infecter les cellules MDCK et déterminer les titreleurs viraux par test de plaque (PFU/mL) (D). Les flèches indiquent une corrélation entre la fluorescence (Vénus), l'immunostaining (Nluc) et la coloration de la violette cristalline. Les barres représentent la moyenne des titres de virus pulmonaires. Ce chiffre a été adapté de Logales et coll.55. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
| Médias et solutions de culture tissulaire | rédaction | espace de stockage | utiliser |
| Médias de culture tissulaire : Dulbecco modifié Eagle's medium (DMEM), 10 % Fetal Bovine Serum (FBS), 1% Penicillin-Streptomycin-L-glutamine (PSG) (DMEM 10 % FBS 1% PSG). | 445 ml d'AMEM, 50 ml de FBS et 5 ml de 100x PSG. | 4 oC | Entretien des cellules MDCK |
| Médias post-infection: DMEM 0,3% Bovine Albumin (BA), 1% PSG (DMEM 0.3 % BA 1% PSG). | 491 ml DMEM, 4,2 ml de 35 % BA et 5 ml de 100x PSG | 4 oC | Maintien des cellules MDCK après une infection virale |
| 10x Phosphate tamponné saline (PBS) | 80 g de NaCl, 2 g de KCl, 11,5 g de Na2HPO4.7H2O, 2 g de KH2PO4. Ajouter ddH2O jusqu'à 1 L. Ajuster le pH à 7,3 | température | Pour préparer 1x PBS |
| 1x PBS | Diluer 10x PBS avec ddH2O | température | Laver les cellules |
| Médias d'infection : 1x PBS, 0.3% BA, 1% Penicillin-Streptomycin (PS) (PBS/BA/PS). | 487 mL 1x PBS stérile, 4,2 mL de 35% BA et 5 ml de 100x 1% PS(100 U/mL) | 4 oC | Infections virales |
| Solution de fixation/perméabilisation : 4 % de formaldéhyde, 0,5 % de triton X-100 dilué en 1x PBS. | 400 mL de formaline tamponneutre neutre 10 %, 5 ml de Triton X-100 et 595 ml de 1x PBS | température | Correction et perméabilisation des cellules MDCK. |
| Solution de blocage : 2,5% d'albumine bovine (BSA) en 1x PBS. | 2,5 g de BSA en 97,5 ml de 1x PBS | 4 oC | Solution de blocage pour l'immunofluorescence et les essais de plaque. |
| Solution de dilution d'anticorps (1% BSA en 1x PBS) | 1 g de BSA en 99 ml de 1x PBS | 4 oC | Dilution des anticorps primaires et secondaires. |
| Solution de violette cristalline de 0,1 % | 1 g de violette cristalline dans 400 ml de méthanol. Ajouter 600 ml de ddH2O | température | Coloration des cellules MDCK dans les essais de plaque. |
| Tosylsulfonyl phenylalanyl chloromethyl cétone (TPCK)-traité trypsin | Préparer une solution de stock 1000x à 1 mg/mL en ddH2O | -20 oC | Pour les infections virales. |
Tableau 1 : Médias et solutions de culture tissulaire.
Discussion
Les chercheurs se sont appuyés sur les virus recombinants exprimant des journalistes comme outils moléculaires vitaux pour comprendre et développer la compréhension actuelle de la réplication virale et de la pathogénie26,27,28, 29 Ans et plus , 30 Ans, états-unis ( , 31 Ans, états-unis ( , 32 Ans, états-unis ( , 33 Ans, états-unis ( , 34 Ans, états-unis ( , 35 Annonces , 36 Annonces , 37 Ans, états-unis ( , 38 Annonces , 39 Ans et plus qu'ils , 40 ans, états-unis ( , 41 Ans, états-unis ( , 54. Les gènes de reporter les plus couramment favorisés sont les luciferases et les protéines fluorescentes, principalement en raison des progrès technologiques dans leur identification, le développement de variantes améliorées, et la détection par les technologies d'imagerie43 , 44 Ans, en est à qui , 45 Annonces , 46 Annonces , 47 Annonces , 48. Les virus reporterres sont souvent utilisés pour accélérer les essais virologiques, étudier la dynamique des virus in vitro et in vivo, et tester l'efficacité des approches vaccinales et thérapeutiques actuellement approuvées ou nouvelles26, 27,28,29,30,31,32,33,34,35, 36,37,38,39,40,41,54. Malheureusement, dans le cas de l'IAV, les études antérieures se limitaient à l'expression d'un gène reporter unique, ce qui entrave le type d'étude qui pourrait être menée 26,27,28,29 , 30 Ans, états-unis ( , 31 Ans, états-unis ( , 32 Ans, états-unis ( , 33 Ans, états-unis ( , 34 Ans, états-unis ( , 35 Annonces , 36 Annonces , 37 Ans, états-unis ( , 38 Annonces , 39 Ans et plus qu'ils , 40 ans, états-unis ( , 41 Ans, états-unis ( , 54. Pour éviter cette limitation, nous avons généré un IAV bi-reporter réplication-compétent qui exprime une luciferase de Nluc et une protéine fluorescente de Vénus (BIRFLU).
Dans ce rapport, nous décrivons la caractérisation in vitro de BIRFLU et les approches expérimentales pour employer BIRFLU pour suivre l'infection virale in vivo utilisant un modèle de souris de l'infection d'IAV. BirFLU Nluc et l'expression de Vénus se sont corrélées avec des titers viraux. En outre, BIRFLU est resté stable et a continué à exprimer les deux gènes de journaliste après avoir été récupéré des poumons des souris infectées. Cette approche offre aux chercheurs une excellente occasion d'étudier les iAV dans les cellules cultivées et dans les modèles animaux, y compris l'identification et le développement de nouvelles alternatives thérapeutiques pour le traitement des infections par le VIH.
Bien que BIRFLU ait été généré à l'aide de l'épine dorsale de PR8, d'autres IAV recombinants utilisant différents types, sous-types ou épines dorsales de souche virale pourraient être générés en utilisant la même approche expérimentale. De même, dans ce rapport, nous avons décrit les procédures expérimentales pour l'utilisation de BIRFLU dans un modèle de souris de la IAV. Cependant, BIRFLU pourrait être une technologie précieuse pour évaluer l'infection par le VI chez d'autres modèles animaux.
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
La recherche sur le virus de l'influenza dans le laboratoire LM-S est partiellement financée par le New York Influenza Center of Excellence (NYICE) (NIH 272201400005C), membre du contrat NO des Centres d'excellence pour la recherche et la surveillance de la grippe (CEIRS) du NIAID. HHSN272201400005C (NYICE) et par le Department of Defense (DoD) Peer Reviewed Medical Research Program (PRMRP) subvention W81XWH-18-1-0460.
matériels
| Name | Company | Catalog Number | Comments |
| 12-well Cell Culture Plate | Greiner Bio-one | 665102 | |
| 24-well Cell Culture Plate | Greiner Bio-one | 662160 | |
| 6-well Cell Culture Plate | Greiner Bio-one | 657160 | |
| 96-well Cell Culture Plate | Greiner Bio-one | 655-180 | |
| Adobe Photoshop CS4 | Adobe | This software is used in 3.1.10 and 4.4.7 | |
| Bovin Albumin solution (BA) | Sigma-Aldrich | A7409 | Store at 4 ° C |
| Bovin Serum Albumin (BSA) | Sigma-Aldrich | A9647 | Store at 4 °C |
| Cell Culture dishes 100mm | Greiner Bio-one | 664-160 | |
| ChemiDoc MP Imaging System | BioRad | This instrument is used in 4.4.7 | |
| Crystal Violet | Thermo Fisher Scientific | C581-100 | Store at Room temperature |
| Dounce Tissue Grinders | Thomas Scientific | 7722-7 | |
| Dulbecco’s modified Eagle’s medium (DMEM) | Corning Cellgro | 15-013-CV | Store at 4 °C |
| Fetal Bovine Serum (FBS) | Seradigm | 1500-050 | Store at -20 °C |
| 5 to 7 week-old female BALB/c mice | National Cancer Institute (NCI) | 555 | |
| Isoflurane | Baxter | 1001936040 | Store at Room temperature |
| IVIS Spectrum | PerkinElmer | 124262 | This instrument is used for in vivo imaging (4.2 and 4.3) |
| IX81 Motorized Inverted Microscope | Olympus | Olympus IX81 | |
| Living Image 4.7.2 software | PerkinElmer | This instrument is used for in vivo imaging (4.2 and 4.3) | |
| Lumicount | Packard | This instrument is used for quantifying luciferase activity (3.2.6) | |
| Madin-Darby Canine Kidney (MDCK) epithelial cells | ATCC | CCL-34 | |
| Monoclonal Antibody anti-NP Influenza A Virus HB-65 | ATCC | H16-L10-4R5 | Store at -20 °C |
| Nano-Glo Luciferase Assay Reagent | Promega | N1110 | This reagent is used to measure Nluc activity. Store at -20 °C |
| Neutral Buffered Formalin 10% | EMD | 65346-85 | Store at RT |
| Nunc MicroWell 96-Well Microplates | Thermo Fisher Scientific | 269620 | |
| Penicillin/Streptomycin (PS) 100x | Corning | 30-00-CI | Store at -20 °C |
| Penicillin/Streptomycin/L-Glutamine (PSG) 100x | Corning | 30-009-CI | Store at -20 °C |
| Retiga 20000R Fast1394 Camera | Qimaging | Retiga 2000R | |
| Scanner | HP | ||
| Texas Red-conjugated anti-mouse -rabbit secondary antibodies | Jackson | 715-075-150 | Store at -20 °C |
| Tosylsulfonyl phenylalanyl chloromethyl ketone (TPCK)-treated trypsin | Sigma-Aldrich | T8802 | Store at -20 °C |
| Triton X-100 | J.T.Baker | X198-07 | Store at RT |
| Vmax Kinetic plate reader | Molecular Devices |
Références
- Palese, P., Shaw, M. L., Knipe, D. M., Howley, P. M., Griffin, D. E., Lamb, R. A., Martin, M. A. Orthomyxoviridae: The Viruses and Their Replication. Fields Virology. , (2007).
- Martinez-Sobrido, L., Peersen, O., Nogales, A. Temperature Sensitive Mutations in Influenza A Viral Ribonucleoprotein Complex Responsible for the Attenuation of the Live Attenuated Influenza Vaccine. Viruses. 10 (10), (2018).
- Nogales, A., Martinez-Sobrido, L. Reverse Genetics Approaches for the Development of Influenza Vaccines. International Journal of Molecular Sciences. 18 (1), (2016).
- Neumann, G., Noda, T., Kawaoka, Y. Emergence and pandemic potential of swine-origin H1N1 influenza virus. Nature. 459 (7249), 931-939 (2009).
- Louie, J. K., et al. A review of adult mortality due to 2009 pandemic (H1N1) influenza A in California. PLoS One. 6 (4), e18221 (2011).
- Barr, I. G., et al. Epidemiological, antigenic and genetic characteristics of seasonal influenza A(H1N1), A(H3N2) and B influenza viruses: basis for the WHO recommendation on the composition of influenza vaccines for use in the 2009-2010 Northern Hemisphere season. Vaccine. 28 (5), 1156-1167 (2010).
- Simonsen, L., et al. Impact of influenza vaccination on seasonal mortality in the US elderly population. A.M.A. archives of internal medicine. 165 (3), 265-272 (2005).
- McLean, H. Q., Peterson, S. H., King, J. P., Meece, J. K., Belongia, E. A. School absenteeism among school-aged children with medically attended acute viral respiratory illness during three influenza seasons, 2012-2013 through 2014-2015. Influenza and Other Respiratory Viruses. 11 (3), 220-229 (2017).
- Principi, N., Esposito, S. Protection of children against influenza: Emerging problems. Human Vaccines and Immunotherapeutics. , 1-8 (2017).
- Falsey, A. R., Treanor, J. J., Tornieporth, N., Capellan, J., Randomized Gorse, G. J. double-blind controlled phase 3 trial comparing the immunogenicity of high-dose and standard-dose influenza vaccine in adults 65 years of age and older. Journal of Infectious Diseases. 200 (2), 172-180 (2009).
- Fuller, J. D., et al. Influenza vaccination of human immunodeficiency virus (HIV)-infected adults: impact on plasma levels of HIV type 1 RNA and determinants of antibody response. Clinical Infectious Diseases. 28 (3), 541-547 (1999).
- Carrat, F., Flahault, A. Influenza vaccine: the challenge of antigenic drift. Vaccine. 25 (39-40), 6852-6862 (2007).
- Doherty, P. C., Kelso, A. Toward a broadly protective influenza vaccine. Journal of Clinical Investigation. 118 (10), 3273-3275 (2008).
- Fiore, A. E., et al. Prevention and control of influenza with vaccines: recommendations of the Advisory Committee on Immunization Practices (ACIP), 2010. MMWR Recommendations and Reports. 59 (RR-8), 1-62 (2010).
- Pica, N., Palese, P. Toward a universal influenza virus vaccine: prospects and challenges. Annual Review of Medicine. 64, 189-202 (2013).
- To, K. K., Chan, J. F., Chen, H., Li, L., Yuen, K. Y. The emergence of influenza A H7N9 in human beings 16 years after influenza A H5N1: a tale of two cities. Lancet Infectious Diseases. 13 (9), 809-821 (2013).
- Baker, S. F., Nogales, A., Santiago, F. W., Topham, D. J., Martinez-Sobrido, L. Competitive detection of influenza neutralizing antibodies using a novel bivalent fluorescence-based microneutralization assay (BiFMA). Vaccine. 33 (30), 3562-3570 (2015).
- He, W., Mullarkey, C. E., Miller, M. S. Measuring the neutralization potency of influenza A virus hemagglutinin stalk/stem-binding antibodies in polyclonal preparations by microneutralization assay. Methods. , (2015).
- Kayali, G., et al. Testing human sera for antibodies against avian influenza viruses: horse RBC hemagglutination inhibition vs. microneutralization assays. Journal of Clinical Virology. 43 (1), 73-78 (2008).
- Stephenson, I., et al. Reproducibility of serologic assays for influenza virus A (H5N1). Emerging Infectious Diseases. 15 (8), 1252-1259 (2009).
- Maher, J. A., DeStefano, J. The ferret: an animal model to study influenza virus. Lab Animal (NY). 33 (9), 50-53 (2004).
- Webster, R. G., Cox, N., Stoehr, K. . WHO/CDS/CSR/NCS/2002.5 Rev. 1. , (2002).
- Rodriguez, L., Nogales, A., Martínez-Sobrido, L. Influenza A Virus Studies in a Mouse Model of Infection. Journal of Visualized Experiments. (127), (2017).
- Cox, A., Baker, S. F., Nogales, A., Martínez-Sobrido, L., Dewhurst, S. Development of a mouse-adapted live attenuated influenza virus that permits in vivo analysis of enhancements to the safety of live attenuated influenza virus vaccine. Journal of Virology. 89 (6), 3421-3426 (2015).
- Steel, J., Lowen, A. C., Mubareka, S., Palese, P. Transmission of influenza virus in a mammalian host is increased by PB2 amino acids 627K or 627E/701N. PLoS Pathogens. 5 (1), e1000252 (2009).
- Tran, V., Moser, L. A., Poole, D. S., Mehle, A. Highly sensitive real-time in vivo imaging of an influenza reporter virus reveals dynamics of replication and spread. Journal of Virology. 87 (24), 13321-13329 (2013).
- Fukuyama, S., et al. Multi-spectral fluorescent reporter influenza viruses (Color-flu) as powerful tools for in vivo studies. Nature Communications. 6, 6600 (2015).
- Manicassamy, B., et al. Analysis of in vivo dynamics of influenza virus infection in mice using a GFP reporter virus. Proceedings of the National Academy of Sciences U S A. 107 (25), 11531-11536 (2010).
- Perez, J. T., Garcia-Sastre, A., Manicassamy, B. Insertion of a GFP reporter gene in influenza virus. Current Protocols in Microbiology. , (2013).
- Reuther, P., et al. Generation of a variety of stable Influenza A reporter viruses by genetic engineering of the NS gene segment. Scientific Reports. 5, 11346 (2015).
- Tran, V., et al. Multi-Modal Imaging with a Toolbox of Influenza A Reporter Viruses. Viruses. 7 (10), 5319-5327 (2015).
- Breen, M., Nogales, A., Baker, S. F., Perez, D. R., Martinez-Sobrido, L. Replication-Competent Influenza A and B Viruses Expressing a Fluorescent Dynamic Timer Protein for In Vitro and In Vivo Studies. PLoS One. 11 (1), e0147723 (2016).
- Nogales, A., Baker, S. F., Martinez-Sobrido, L. Replication-competent influenza A viruses expressing a red fluorescent protein. Virology. , 206-216 (2014).
- Nogales, A., et al. Replication-competent fluorescent-expressing influenza B virus. Virus Research. 213, 69-81 (2015).
- Avilov, S. V., et al. Replication-competent influenza A virus that encodes a split-green fluorescent protein-tagged PB2 polymerase subunit allows live-cell imaging of the virus life cycle. Journal of Virology. 86 (3), 1433-1448 (2012).
- Breen, M., Nogales, A., Baker, S. F., Martínez-Sobrido, L. Replication-Competent Influenza A Viruses Expressing Reporter Genes. Viruses. 8 (7), (2016).
- Eckert, N., et al. Influenza A virus encoding secreted Gaussia luciferase as useful tool to analyze viral replication and its inhibition by antiviral compounds and cellular proteins. PLoS One. 9 (5), e97695 (2014).
- Karlsson, E. A., et al. Visualizing real-time influenza virus infection, transmission and protection in ferrets. Nature Communications. 6, 6378 (2015).
- Czako, R., et al. In Vivo Imaging of Influenza Virus Infection in Immunized Mice. MBio. 8 (3), (2017).
- Harding, A. T., Heaton, B. E., Dumm, R. E., Heaton, N. S. Rationally Designed Influenza Virus Vaccines That Are Antigenically Stable during Growth in Eggs. MBio. 8 (3), (2017).
- DiPiazza, A., et al. Pandemic 2009 H1N1 Influenza Venus reporter virus reveals broad diversity of MHC class II-positive antigen-bearing cells following infection in vivo. Scientific Reports. 7 (1), 10857 (2017).
- Yan, D., et al. Replication-Competent Influenza Virus and Respiratory Syncytial Virus Luciferase Reporter Strains Engineered for Co-Infections Identify Antiviral Compounds in Combination Screens. Biochemistry. 54 (36), 5589-5604 (2015).
- Shaner, N. C., Patterson, G. H., Davidson, M. W. Advances in fluorescent protein technology. Journal of Cell Science. 120 (Pt 24), 4247-4260 (2007).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nature Methods. 2 (12), 905-909 (2005).
- Kelkar, M., De, A. Bioluminescence based in vivo screening technologies. Current Opinion in Pharmacology. 12 (5), 592-600 (2012).
- Welsh, D. K., Noguchi, T. Cellular bioluminescence imaging. Cold Spring Harbor Protocols. 2012 (8), (2012).
- Zhao, H., et al. Emission spectra of bioluminescent reporters and interaction with mammalian tissue determine the sensitivity of detection in vivo. Journal of Biomedical Optics. 10 (4), 41210 (2005).
- Stacer, A. C., et al. NanoLuc reporter for dual luciferase imaging in living animals. Molecular Imaging. 12 (7), 1-13 (2013).
- Vintersten, K., et al. Mouse in red: red fluorescent protein expression in mouse ES cells, embryos, and adult animals. Genesis. 40 (4), 241-246 (2004).
- Stacer, A. C., et al. NanoLuc reporter for dual luciferase imaging in living animals. Mol Imaging. 12 (7), 1-13 (2013).
- Schoggins, J. W., et al. Dengue reporter viruses reveal viral dynamics in interferon receptor-deficient mice and sensitivity to interferon effectors in vitro. Proc Natl Acad Sci U S A. 109 (36), 14610-14615 (2012).
- Pan, W., et al. Visualizing influenza virus infection in living mice. Nat Commun. 4, 2369 (2013).
- Heaton, N. S., et al. In vivo bioluminescent imaging of influenza a virus infection and characterization of novel cross-protective monoclonal antibodies. J Virol. 87 (15), 8272-8281 (2013).
- Nogales, A., Baker, S. F., Martínez-Sobrido, L. Replication-competent influenza A viruses expressing a red fluorescent protein. Virology. 476, 206-216 (2015).
- Nogales, A., et al. A novel fluorescent and bioluminescent Bi-Reporter influenza A virus (BIRFLU) to evaluate viral infections. Journal of Virology. , (2019).
- Nogales, A., et al. Influenza A Virus Attenuation by Codon Deoptimization of the NS Gene for Vaccine Development. Journal of Virology. 88 (18), 10525-10540 (2014).
- Nogales, A., DeDiego, M. L., Topham, D. J., Martinez-Sobrido, L. Rearrangement of Influenza Virus Spliced Segments for the Development of Live-Attenuated Vaccines. Journal of Virology. 90 (14), 6291-6302 (2016).
- National Research Council (U.S.). Committee for the Update of the Guide for the Care and Use of Laboratory Animals., Institute for Laboratory Animal Research (U.S.) & National Academies Press (U.S.). . Guide for the care and use of laboratory animals. , (2011).
- Nogales, A., Martinez-Sobrido, L., Chiem, K., Topham, D. J., DeDiego, M. L. Functional Evolution of the 2009 Pandemic H1N1 Influenza Virus NS1 and PA in Humans. Journal of Virology. 92 (19), (2018).
- Hall, M. P., et al. Engineered luciferase reporter from a deep sea shrimp utilizing a novel imidazopyrazinone substrate. ACS Chemical Biology. 7 (11), 1848-1857 (2012).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon