Method Article
Un virus de la gripe Reportero fluorescente de Luciferase para imágenes en vivo y cuantificación de la infección viral
En este artículo
Resumen
Los virus de la gripe A son patógenos respiratorios contagiosos que causan epidemias anuales y pandemias ocasionales. Aquí, describimos un protocolo para rastrear infecciones virales in vivo usando una nueva luciferasa recombinante y bi-reportero expreso de fluorescencia IAV (BIRFLU). Este enfoque proporciona a los investigadores una excelente herramienta para estudiar IAV in vivo.
Resumen
Los virus de la gripe A causan enfermedades respiratorias humanas que se asocian con consecuencias económicas y sanitarias significativas. Al igual que con otros virus, el estudio de la AIF requiere el uso de enfoques secundarios laboriosos para detectar la presencia del virus en células infectadas y/o en modelos animales de infección. Esta limitación se ha eludido recientemente con la generación de IAVs recombinantes que expresan proteínas de reportero fluorescentes o bioluminiscentes (luciferasa) fácilmente trazables. Sin embargo, los investigadores se han visto obligados a seleccionar genes de reportero fluorescentes o luciferasa debido a la capacidad restringida del genoma de IAV para incluir secuencias extranjeras. Para superar esta limitación, hemos generado un IAV bi-reportero (BIRFLU) de replicación recombinante que expresa fácilmente un gen fluorescente y un gen de reportero de luciferasa para rastrear fácilmente las infecciones por IAV in vitro e in vivo. Con este fin, se modificaron los segmentos virales no estructurales (NS) y hemaglutininas (HA) de la gripe A/Puerto Rico/8/34 H1N1 (PR8) para codificar la Venus fluorescente y las proteínas bioluminiscentes Nanoluc luciferasa, respectivamente. Aquí, describimos el uso de BIRFLU en un modelo de ratón de infección por IAV y la detección de ambos genes reporteros utilizando un sistema de imágenes in vivo. En particular, hemos observado una buena correlación entre las expresiones de los reporteros y la replicación viral. La combinación de técnicas de vanguardia en biología molecular, investigación animal y tecnologías de imagen, proporciona a los investigadores la oportunidad única de utilizar esta herramienta para la investigación de la gripe, incluyendo el estudio de las interacciones virus-huésped y la dinámica de infecciones virales. Es importante destacar que la viabilidad de alterar genéticamente el genoma viral para expresar dos genes extraños de diferentes segmentos virales abre oportunidades para utilizar este enfoque para: (i) el desarrollo de nuevas vacunas IAV, (ii) la generación de IAVs recombinantes que pueden utilizarse como vectores de vacunas para el tratamiento de otras infecciones por patógenos humanos.
Introducción
El virus de la gripe A (IAV) es un virus de ARN segmentado de sentido negativo de una sola cadena envuelto de la familia Orthomyxoviridae 1,2,3. La Organización Mundial de la Salud (OMS) estima entre 3 y 5 millones de casos anuales de gripe y más de 250.000 muertes por gripe en todo el mundo4,5,6. Los grupos que son particularmente vulnerables a la gripe incluyen ancianos, personas inmunodeprimidas y niñosde 7,8,9,10,11. Aunque las vacunas están disponibles y representan la intervención más común y eficaz contra la infección viral, iAV es capaz de evolucionar rápidamente y escapar de la inmunidad preexistente3,12,13, 14 , 15. La reaparición de una cepa pandémica H1N1 en 2009 y la aparición de IAV patógena reiteran la amenaza constante para la salud pública humana en todo el mundo4,16.
Durante una epidemia o pandemia, es crucial determinar rápidamente la patogenicidad y la transmisibilidad de los virus recién aislados. Las técnicas actualmente disponibles para detectar el virus consumen mucho tiempo y a veces requieren el uso de enfoques laboriosos, lo que puede retrasar la realización de estos análisis17,18,19,20. Además, los ensayos virales actuales son difíciles de ampliar, lo que podría ser necesario durante el caso de un brote. Por último, el uso de modelos animales validados de infección, como ratones, conejillos de indias y hurones, se utiliza de forma rutinaria y son vitales para estudiar las infecciones por gripe, las respuestas inmunitarias y la eficacia de nuevas vacunas y/o antivirales. Sin embargo, estos modelos son restrictivos debido a la incapacidad de observar la dinámica viral en tiempo real; esto limita los estudios a la toma de imágenes estáticas de infecciones virales21,22,23,24,25. Los animales utilizados en estos ensayos también son eutanasiados con el fin de determinar la carga viral, aumentando así el número de animales necesarios para completar estos estudios26. Para eludir todas estas limitaciones, muchos investigadores confían en el uso de IAV sordosas y con opiniones que expresan periodistas, que son capaces de acelerar los ensayos virológicos y detectar la carga viral y la diseminación in vivo en tiempo real26 ,27,28,29,30,31,32,33,34,35 ,36,37,38,39,40,41. Es importante destacar que estos IAV que expresan a los periodistas son capaces de replicarse de forma similar a los IAV de tipo salvaje (WT) en el cultivo celular y en modelos animales de infección33,42.
Las proteínas fluorescentes y bioluminiscentes son dos sistemas de reportero comúnmente utilizados por los investigadores debido a su sensibilidad, estabilidad y facilidad de uso. Además, hay un enorme apoyo y avance en las tecnologías de detección de proteínas fluorescentes y bioluminiscentes43,44,45,46,47,48 . Las proteínas fluorescentes y la luciferasa tienen diferentes propiedades que les permiten brillar, diferenciando específicamente en cómo se generan los estados excitados y cómo se detecta la emisión43,44,45, 46,47,48. Las proteínas fluorescentes se excitan primero absorbiendo la energía, que posteriormente se libera como luz en una longitud de onda diferente a medida que las moléculas disminuyen a un estado de energía más bajo43. Por otro lado, la bioluminiscencia se deriva de una reacción exotérmica química que implica un sustrato, oxígeno, y a veces ATP con el fin de producir la luz45. Debido a las diversas propiedades de estos dos tipos de proteínas de reportero, uno tal vez más ventajoso que el otro dependiendo del estudio de interés. Mientras que las proteínas fluorescentes se utilizan ampliamente para observar la localización celular28,41, sus señales in vivo tienen una intensidad inadecuada y a menudo se oscurecen por la autofluorescencia en los tejidos vivos49. Por lo tanto, los investigadores confían en las luciferasas para evaluar la dinámica viral en organismos vivos, aunque las proteínas fluorescentes pueden ser preferidas para estudios ex vivo50,51,52,53. A diferencia de las proteínas fluorescentes, las luciferases son más convenientes para estudios in vivo y más aplicables en enfoques no invasivos26,27,28,29,30 , 31 , 32 , 33 , 34 , 35 , 36 , 37 , 38 , 39 , 40 , 41 , 54. En última instancia, en función del tipo de estudio, los investigadores deben elegir entre el uso de una proteína fluorescente o una proteína de reportero de luciferasa como su lectura, que somete su estudio a un equilibrio de funcionalidades y sensibilidades, y severamente restringe la utilidad de los virus de reportero recombinante. Además, existen preocupaciones con respecto a la correlación entre la expresión de diferentes genes reporteros utilizando sistemas de fluorescencia o luciferasa y replicación o diseminación viral, lo que podría poner en peligro la interpretación de los datos obtenidos con iAVs que expresan a un reportero.
Hemos superado esta limitación generando un IAV bi-reportero competente para replicación recombinante (BIRFLU) que codifica tanto para un fluorescente como para una proteína luciferasa en el mismo genoma viral55 (Figura1). Aquí, NanoLuc luciferase (Nluc), una pequeña y brillante proteína bioluminiscente48,se insertó aguas arriba de la secuencia de hemaglutinina (HA) en el segmento viral HA de la gripe A/Puerto Rico/08/1934 H1N1 (PR8)24,33, 40,55,56,57. Además, Venus, una proteína fluorescente monomérica de uso frecuente, se insertó en el segmento viral no estructural (NS)32,33,36,41,55. Dado que BIRFLU codifica los genes de reportero fluorescentes y luciferasa, la señal de proteína del reportero se puede utilizar como lectura para determinar la replicación viral y la diseminación in vitro o in vivo55. Puede encontrar información adicional sobre la generación y la caracterización in vitro o in vivo de BIRFLU en nuestra reciente publicación55. BIRFLU se puede utilizar para probar la eficacia de los medicamentos antivirales o anticuerpos neutralizantes a través de un nuevo ensayo de microneutralización basado en fluorescentes y bioluminiscentes55. Por otra parte, BIRFLU también se puede utilizar para evaluar la dinámica viral en un modelo de ratón de infección55. En este manuscrito, describimos los procedimientos para caracterizar BIRFLU55 in vitro y cómo estudiar la infección BIRFLU en ratones utilizando sistemas de imágenes de luminiscencia in vivo para la detección de Nluc in vivo o de Venus ex vivo.
La combinación de técnicas de vanguardia en biología molecular, investigación animal y tecnologías de imagen, ofrece a los investigadores la oportunidad única de utilizar BIRFLU para la investigación de IAV, incluyendo el estudio de interacciones virus-huésped, dinámica de la infección viral; el desarrollo de nuevos enfoques vacunales para el tratamiento terapéutico de las infecciones por IAV o el posible uso de la IAV como vector de vacunapara el tratamiento de otras infecciones patógenas.
Protocolo
Todos los protocolos relacionados con ratones han sido aprobados por el Comité Institucional de Cuidado y Uso de Animales (IACUC) y el Comité Institucional de Bioseguridad (IBC) de la Universidad de Rochester, Facultad de Medicina y Odontología. Todos los experimentos realizados en animales siguen las recomendaciones de la Guía para el Cuidado y Uso de Animales de Laboratorio del Consejo Nacional de Investigación58. El Vivarium y la División de Instalaciones de Medicina Animal de Laboratorio en la Facultad de Medicina y Odontología de la Universidad de Rochester está acreditado por la Asociación para la Evaluación y Acreditación del Cuidado Animal de Laboratorio (AALAC) Internacional y cumplir con las leyes federales y estatales y la política de los Institutos Nacionales de Salud (NIH). Se requiere un equipo de protección personal (EPP) adecuado cuando se trabaja con ratones. Se deben implementar políticas y requisitos similares al realizar experimentos descritos en este manuscrito en cada institución.
1. Uso de pequeños animales vertebrados
- Comprar ratones BALB/c hembra de cinco a siete semanas de edad y mantenerlos en el centro de cuidado animal en condiciones específicas libres de patógenos. A la llegada de los ratones a las instalaciones determinadas, permita un período de descanso de 4 a 5 días para permitir que los animales se aclimaten a su nuevo entorno.
- Siguiendo los protocolos IACUC, coloque un máximo de 5 ratones por jaula.
NOTA: Al concluir el experimento, con el fin de garantizar que el animal está muerto, los ratones fueron eutanasiados utilizando dos procedimientos aprobados, teniendo en cuenta que el segundo debe ser un método físico. En este estudio, después de la infección por ratones con BIRFLU y la toma de imágenes in vivo, los animales se eutanasian con una dosis letal de 2,2,2-tribromoetanol (TBE), y cortando la vena hepática como método secundario físico como hemos demostrado previamente23.
2. Bioseguridad
NOTA: En este manuscrito, BIRFLU se generó en la columna vertebral de la gripe A/Puerto Rico/08/34 H1N1 (PR8), que es una cepa IAV de laboratorio común adaptada al ratón23,32,33,56. El virus se generó utilizando enfoques de genética inversa descritos anteriormente basados en plásmidos y una descripción completa de la generación, y la caracterización in vitro e in vivo de BIRFLU se puede encontrar en nuestra reciente publicación55. Todos los procedimientos que involucran infecciones por ASI (in vitro o in vivo) se realizaron en un gabinete de seguridad biológica en condiciones de nivel de bioseguridad (BSL)-2.
PRECAUCION: Debe determinarse un nivel adecuado de bioseguridad de conformidad con una evaluación del riesgo de bioseguridad. Deberá consultarse con la institución en la que se realizarán los experimentos información adicional sobre la realización de evaluaciones del riesgo de seguridad de la bioseguridad y el establecimiento de una contención eficaz de la seguridad de la biotecnología.
- Limpie los gabinetes de bioseguridad con un 70% de desinfectante de etanol o dióxido de cloro antes y después de realizar todos los procedimientos experimentales. Para el trabajo en ratones, esterilizar todo el material de disección (tijeras, fórceps de disección, etc.) y el homogeneizador Dounce antes y después de su uso.
- Deseche todo el material biológico producido durante los procedimientos bajo las directrices apropiadas de IBC e IACUC.
3. Caracterización in vitro de BIRFLU (Figura 2)
NOTA: Refiera al cuadro 1 para todas las composiciones del buffer y de los medios.
- Analizar la expresión proteica por fluorescencia (Figura2A)e inmunofluorescencia indirecta (Figura 2B)
- Un día antes de la infección, sesemilla placas de 24 pocillos con células epiteliales madin-darby canino riñón (MDCK) (1 x 105 células/pozo, triplicados) en medios de cultivo de tejido y mantener las placas en una incubadora de 37 oC con 5% de CO2. Preparar suficientes pozos para evaluar todos los anticuerpos elegidos en células infectadas por simulacros y infectadas por BIRFLU.
NOTA: Recomendamos visualizar las células bajo un microscopio ligero para confirmar una monocapa antes de iniciar la infección viral. Se recomienda una monocapa de confluencia del 90% de células MDCK en el momento de la infección. - Preparar diluciones de IAV de WT o BIRFLU en medios de infección e infectar las células MDCK sembradas con una multiplicidad de infección (MOI) de 0,1 unidades formadoras de placa (PFU) por célula en un volumen final de 0,25 ml/pozo de medios de infección.
- Retire el medio de cultivo de tejido del paso 3.1.1 y lave las células MDCK dos veces con 1 solución salina tamponada con fosfato (PBS). Agregue las diluciones del virus de 3.1.2 a las células MDCK y coloque las placas en una plataforma de balanceo durante 1 h a temperatura ambiente para permitir la adsorción viral.
NOTA: Las células infectadas por la burla se incuban solo con medios de infección en ausencia de virus. - Después de la adsorción viral (paso 3.1.3), retire el inóculo viral por aspiración y agregue 1 ml de medios post-infección que contengan 1 g/ml de trippsina tratada con TPCK a cada poc. Incubar células durante 18 h en una incubadora humidificada de CO2 al 5% a 33oC.
- Después de 18 h de infección, retire el sobrenadante de cultivo de tejido de las placas de 24 pocillos (3.1.4). Fijar y permeabilizar las células con 0,25 ml/pozo de solución de fijación/permeabilización durante 20 minutos a temperatura ambiente.
NOTA: Prepare la solución de fijación/permeabilización en una campana de humo sin humos para evitar la exposición al formaldehído. - Retire la solución de fijación/permeabilización del paso 3.1.5, lave las células dos veces con 1pbS e incubar las células con 0,25 ml/bien de solución de bloqueo durante 1 h a temperatura ambiente.
NOTA: Continúe con el siguiente paso o almacene las celdas en solución de bloqueo a 4 oC durante la noche. - Retire la solución de bloqueo (paso 3.1.6) y agregue 0,25 ml/pozo (1 g/ml) de un anticuerpo monoclonal de ratón (MAb) contra la nucleoproteína IAV, NP (HB-65) o una dilución de 1:1.000 de un anticuerpo policlonal de conejo (pAb) contra NLuc (ver la Tabla de Materiales), ambos diluidos en solución de dilución de anticuerpos (1x PBS, 2,5% BSA), e incubar las células durante 1 h a 37 oC.
- Retire el anticuerpo primario del paso 3.1.7, lave las células tres veces con 1x PBS e incubarlas con 0,25 ml/bien de un antiratón conjugado en rojo de Texas o anticuerpos secundarios anticonejo (ver la Tabla de Materiales)diluido 1:200 en anticuerpos solución de dilución.
NOTA: Los núcleos celulares se pueden teñir al mismo tiempo añadiendo 0,5 g/ml de 4',6'-diamidino-2-phenylindole (DAPI) a la misma solución secundaria de anticuerpos. Incubar las células durante 1 h a 37 oC en la oscuridad. Se pueden utilizar otros anticuerpos secundarios conjugados con otros fluoróforos. - Retire el anticuerpo secundario y el DAPI del paso 3.1.8, lave las células tres veces con 1 x PBS. Después del lavado, deje las células en 0,25 ml/pozo de 1pbS.
- Coloque la placa en el escenario de un microscopio de fluorescencia para detectar la expresión del reportero de Venus y Nluc, y NP de células infectadas utilizando los filtros fluorescentes adecuados. Capture imágenes utilizando un microscopio de fluorescencia (aumento de 20x) y combínelas utilizando un software de edición de imágenes (Figura2A,B).
- Un día antes de la infección, sesemilla placas de 24 pocillos con células epiteliales madin-darby canino riñón (MDCK) (1 x 105 células/pozo, triplicados) en medios de cultivo de tejido y mantener las placas en una incubadora de 37 oC con 5% de CO2. Preparar suficientes pozos para evaluar todos los anticuerpos elegidos en células infectadas por simulacros y infectadas por BIRFLU.
- Analizar la actividad Nluc (Figura2C)y la replicación viral (Figura2D).
- Un día antes de la infección, las placas de 12 pocillos de semilla con células MDCK (2 x 105células/pozo, triplicados) utilizan los medios de cultivo de tejido en una incubadora de 37 oC con 5% de CO2 para alcanzar aproximadamente el 90% de confluencia en el momento de la infección.
NOTA: Antes de la infección, revise las células bajo un microscopio para verificar una monocapa de células MDCK. - El día de la infección preparar diluciones de los virus WT y BIRFLU en los medios de infección para infectar las células MDCK (paso 3.2.1.) en triplicado con un MOI de 0.001 PFU en 0.5 mL/pozo. Retire el medio de cultivo de tejido y lave las células MDCK dos veces con 1 pbS.
- Añada las diluciones del virus a las monocapas MDCK y permita la adsorción viral a temperatura ambiente durante 1 h en una plataforma de balanceo. Después de la adsorción viral, eliminar el inóculo del virus y añadir 1,5 ml de medios post-infección que contengan 1 g/ml de trippsina tratada con TPCK, a cada poca. Incubar las células infectadas en una incubadora humidificada de CO2 al 5% a 33 oC y recoger los sobrenadores en los puntos de tiempo indicados en el paso 3.2.4.
- A las 24, 48, 72 y 96 h después de la infección (p.i.) recoger 150 l de sobrenadante de cultivo tisular de cada pocal y almacenar las muestras en un tubo de microcentrífuga a -80 oC para realizar los ensayos de Nluc y las valoraciones virales.
- Para realizar el ensayo de actividad Nluc, siga las indicaciones del fabricante. En primer lugar, descongelar el sobrenadante de cultivo de tejido almacenado a -80 oC (paso 3.2.4) sobre hielo.
NOTA: Consulte las recomendaciones del fabricante y optimice el ensayo si es necesario. - Preparar la solución de ensayo de luciferasa (ver la Tabla de Materiales)diluyendo el sustrato nluc 1:50 con el tampón de dilución. En una microplaca blanca de fondo plano de 96 pocillos para ensayos de luciferasa, mezcle 25 ml de solución de ensayo de luciferasa con 10 a 25 l de las muestras de sobrenadantes de cultivo de tejido recogido. Incubar la mezcla durante 2-3 minutos antes de medir la luminiscencia utilizando un luminómetro (Figura2C).
NOTA: La presencia de partículas virales infecciosas en los sobrenatantes del cultivo de tejidos se determina mediante ensayo de enfoque inmune por fluorescencia (Venus) o inmunofluorescencia indirecta (tinción de antígeno sórmicos virales) como se describió anteriormente23,32, 33,56. - Un día antes de las valoraciones virales, las células MDCK de semillas en una placa de 96 pocillos (2 x 104 células/pozo, triplicados) con medios de cultivo de tejido y permiten que las células alcancen una confluencia del 90% en el momento de la infección en una incubadora establecida a 37 oC con 5% de CO2.
- Utilice las muestras de sobrenadantes de cultivo de tejidos que se descongelaron sobre hielo (paso 3.2.4.). Añadir 90 l de medios de infección a cada uno de los pozos en una nueva placa de 96 pocillos, luego añadir 10 l del sobrenadante de cultivo de tejido descongelado (paso 3.2.4) al primer pozo (fila A). Utilice una tubería multicanal para mezclar y transferir 10 l de la fila A a la fila B. Cambie las puntas entre las diluciones y mezcle bien.
NOTA: Este procedimiento debe repetirse hasta la última fila (H). Recomendamos realizar valoraciones virales en triplicados para una medición precisa de los valoradores virales. - Retire el medio de cultivo de tejido de las células MDCK en las placas de 96 pocillos (paso 3.2.7) y lávelo dos veces con 1 pbS. Añadir 50 l de las diluciones de sobrenadantes de la réplica placa de 96 pocillos que contiene las diluciones del virus (paso 3.2.8) a cada pocto en la placa de 96 pocillos que contiene células MDCK.
- Incubar la placa de 96 pocillos en una plataforma de balanceo durante 1 h a temperatura ambiente para permitir la adsorción viral. Después de la adsorción viral, retire el inóculo y agregue 100 l/pozo de medios post-infección que contengan 1 g/ml de trippsina tratada con TPCK. Incubar células infectadas durante 12 h en una incubadorade 33 oC con 5% de CO2.
- Dado que BIRFLU expresa Venus, el número de células infectadas se puede contar directamente mediante un microscopio de fluorescencia. Alternativamente, los títulos virales pueden determinarse mediante inmunofluorescencia indirecta como se describió anteriormente utilizando un anticuerpo contra una proteína viral23,56,57,59. Para este último, proceda a fijar/permeabilizar las células y la mancha utilizando el anti-NP mAb HB-65 como se describe en la sección 4.4.
- Determinar los lanzadores virales contando el número de unidades formadoras de fluorescentes (FFU)/ml utilizando la fórmula: ((número de FFU) x 20 x 1/dilución (Figura 2D).
- Un día antes de la infección, las placas de 12 pocillos de semilla con células MDCK (2 x 105células/pozo, triplicados) utilizan los medios de cultivo de tejido en una incubadora de 37 oC con 5% de CO2 para alcanzar aproximadamente el 90% de confluencia en el momento de la infección.
4. En Vivo Caracterización de BIRFLU (Figura3 y Figura 4)
- Infección del ratón
NOTA: La infección intranasal de ratones se realizó como se describió anteriormente23. Para un protocolo más detallado de la infección por IAV in vivo utilizando el modelo de ratón de infección, recomendamos ver el vídeo asociado con la publicación anterior23. Esta sección sólo resume los pasos necesarios para la infección del ratón con BIRFLU.- Inspeccione a los ratones para evaluar su salud y su aspecto físico general. Preparar la dilución de BIRFLU en 1x PBS para inocular ratones con 1 x 106 PFU de BIRFLU en un volumen total de 30 l/ratón. Mantenga el virus sobre hielo hasta la inoculación del ratón.
NOTA: Se necesitarán ratones infectados por la burla (1x PBS) como control interno para la toma de imágenes. Coloque ratones infectados por simulacros en una jaula diferente a la de los animales infectados por BIRFLU. - Anestetizar ratones BALB/c hembra de cinco a siete semanas de edad por vía intraperitoneal con 240–250 mg/kg de tribromoetanol (TBE) insertando la aguja en el caudal 2/3 del lado derecho del abdomen. A continuación, devolver el ratón a la jaula y esperar alrededor de 5 min. Compruebe que el ratón está anestesiado antes de la inoculación del virus por la ausencia del reflejo del dedo del pie-pinch.
NOTA: Se recomiendan sedantes inyectables sobre sedantes inhalados para infecciones gripales in vivo, ya que estos últimos pueden modificar el comportamiento y la toma del virus por el epitelio de las vías respiratorias. Además, también pueden afectar las respuestas inmunitarias locales del pulmón e interferir con la imagen in vivo de ratones infectados. Por último, los sedantes inhalados han reducido la duración del efecto, lo que sería un problema para la toma de imágenes in vivo de los animales infectados. - Inocular los ratones por vía intranasal con los 30 ml de la dilución BIRFLU preparada. Compruebe que los ratones respiran correctamente antes de devolverlos a la jaula.
- Inspeccione a los ratones para evaluar su salud y su aspecto físico general. Preparar la dilución de BIRFLU en 1x PBS para inocular ratones con 1 x 106 PFU de BIRFLU en un volumen total de 30 l/ratón. Mantenga el virus sobre hielo hasta la inoculación del ratón.
- Monitorización de la bioluminiscencia de ratones infectados con BIRFLU (Figura 4A)
NOTA: En este manuscrito se determinan la expresión de reportero de Nluc o Venus y la replicación viral en los pulmones de ratones infectados con BIRFLU a los 3 días posteriores a la infección. Sin embargo, la naturaleza de las imágenes de bioluminiscencia permite un seguimiento repetido de la expresión de Nluc en animales individuales infectados por BIRFLU sin necesidad de eutanasiarlos para cada punto de tiempo experimental. Se requiere un sistema de diagnóstico por imágenes in vivo con un colector de anestesia isoflurano para realizar los procedimientos experimentales descritos. Consulte la Tabla de materiales para obtener más información sobre el instrumento de imagen y el software de imagen utilizados para la adquisición de imágenes y el análisis de datos.- Afeitar el pecho de los ratones para mejorar la señal de bioluminiscencia. Abra el software de imágenes y pulse Inicializar. A continuación, establezca los parámetros que se utilizarán, incluyendo el ajuste del modo de imagen a bioluminiscencia, ahorro automático, tiempo de exposición a automático, filtro abierto, etc.
- Una vez que la máquina esté completamente inicializada, encienda el sistema de anestesia de isoflurano. Coloque los animales en la cámara de anestesia. Los ratones son anestesiados simultáneamente y ligeramente con una mezcla de gas de oxígeno y vaporizados 1–2% de isoflurano.
- Una vez anestesiados los ratones, administre el sustrato de Nluc (ver la Tabla de Materiales)diluido 1:10 en 1x PBS (volumen final 100 l/ratón) por vía retroorbital utilizando una jeringa con una aguja de 22 G.
- Inmediatamente después de la administración del reactivo Nluc, coloque los animales en el instrumento de imágenes con los pechos hacia arriba y hacia fuera dentro del cono del colector para mantener al animal anestesiado durante la toma de imágenes. Inmediatamente después de cerrar la puerta del imager, haga clic en Adquirir en el programa de software (Figura4A,superior).
- Después de la toma de imágenes, devuelva a los ratones a sus jaulas monitorizándolos hasta que se hayan recuperado por completo y apague el vaporizador de isoflurano. A continuación, proceda con la toma de imágenes ex vivo de los pulmones de ratones para evaluar la expresión génica del reportero de Venus (sección 4.3).
- Utilice las herramientas de software de imágenes para analizar los datos de bioluminiscencia adquiridos. Utilice el ROI de la herramienta (región de interés) para designar la señal específica y realizar mediciones de flujo en la región de interés (normalmente alrededor del pecho) (Figura4A,inferior). Aunque la forma del ROI es irrelevante, generalmente se prefieren los ROI más grandes para capturar toda el área de difusión de la señal.
- Haga clic en Medir. Evaluar la bioluminiscencia en fotones ya que proporciona una medición absoluta de emisión de fotones que es comparable a las mediciones de salida proporcionadas por diferentes parámetros o instrumentos de imagen.
- Análisis de fluorescencia en ratones infectados con BIRFLU (Figura3 y Figura 4B)
- Recoger los pulmones del ratón después de la toma de imágenes in vivo, como se describió anteriormente23.
- Brevemente, eutanasia ratones con una dosis letal de TBE (500 mg/kg). Desinfectar el lugar de la incisión con 70% de etanol. Con un bisturí, haga una incisión desde el esternón hasta la base del abdomen y luego corte desde la parte inferior de la incisión a los lados con tijeras. A continuación, corte la vena hepática (segundo método de eutanasia física) para sangrar al animal.
NOTA: Para evitar una señal de fondo alta durante la toma de imágenes, es importante minimizar la cantidad de sangre en los pulmones (ver más abajo).
- Brevemente, eutanasia ratones con una dosis letal de TBE (500 mg/kg). Desinfectar el lugar de la incisión con 70% de etanol. Con un bisturí, haga una incisión desde el esternón hasta la base del abdomen y luego corte desde la parte inferior de la incisión a los lados con tijeras. A continuación, corte la vena hepática (segundo método de eutanasia física) para sangrar al animal.
- Coloque el ratón en la recumbencia dorsal y use las tijeras para cortar la pleura y abrir la caja torácica. Posteriormente, retire los pulmones cortando el extremo de la tráquea con tijeras mientras sostiene suavemente los pulmones con fórceps.
- Coloque los pulmones en una placa de seis pocillos con 2 ml de 1pbS y lave los pulmones tres veces con 1 x PBS.
NOTA: Para evitar la contaminación entre muestras, limpie y desinfecte las herramientas de diselación entre cada animal.
- Coloque los pulmones en una placa de seis pocillos con 2 ml de 1pbS y lave los pulmones tres veces con 1 x PBS.
- Inicie el software de adquisición de imágenes haciendo clic en inicializar y establecer los parámetros para la imagen, incluyendo la configuración del modo de imagen a fluorescencia, ahorro automático, tiempo de exposición a los filtros automáticos, excitación (500 nm) y emisión (540 nm).
- Cuando se inicialice la máquina, coloque los pulmones en una bandeja de fondo negro, asegurándose de que los tejidos estén separados entre sí, introduzca la bandeja en el imager y haga clic en Adquirir en el programa del sistema de imágenes después de cerrar la puerta del imager ( Figura 4B,superior).
- Después de la toma de imágenes, retire los tejidos inmediatamente y guárdelos en hielo (4 oC) si las muestras se procesan el mismo día; o en un tubo y hielo seco para congelarlos rápidamente, antes de almacenarlos a -80 oC, si las muestras se procesarán más tarde en un día diferente.
- Para el procesamiento de imágenes, seleccione la herramienta ROI y dibuje ROI alrededor de cada uno de los pulmones individuales. Haga clic en Medir. A continuación, utilizando las mediciones de eficiencia radiante media resultantes, reste los valores de los ratones infectados por el simulacro (Figura4B,inferior).
NOTA: Con BIRFLU, hay una buena correlación de los niveles de expresión de Nluc y Venus, y la distribución de la señal. Por lo tanto, es importante analizar la expresión Nluc (ratón entero) y Venus (pulmones extirpados) del mismo animal, y mantener la misma orientación.
- Recoger los pulmones del ratón después de la toma de imágenes in vivo, como se describió anteriormente23.
- Evaluación de la replicación de BIRFLU en pulmones de ratones (Figura 3 y Figura 4C)
- Homogeneizar los pulmones de ratones para la valoración viral por ensayo de placa como se describió anteriormente23.
- Coloque los pulmones en un homogeneizador Dounce con 1 ml de medios de infección refrigerados. Homogeneizar los pulmones con el pestillo durante 1 min a temperatura ambiente hasta que se haya desintegrado por completo y almacenar las muestras en un tubo estéril a 4oC.
- Centrifugar las muestras a 300 x g durante 10 min y recoger el sobrenadante en un tubo estéril. Almacene las muestras en hielo si se utilizarán el mismo día o congele a -80 oC para evaluar los lanzadores virales más adelante.
NOTA: Se debe utilizar un nuevo homogeneizador Dounce para cada muestra de pulmón. - Para realizar el ensayo de placa, el día antes de realizar la valoración viral, las placas de seis pocillos de semilla con células MDCK (5 x 105 células/pozo) en los medios de cultivo de tejido. Incubar las células durante la noche a 37 oC con un 5% de CO2 para alcanzar el 90% de confluencia al día siguiente. Antes de la infección, confirme que las células forman una monocapa bajo un microscopio de luz.
- Preparar las diluciones en serie 1:10 del sobrenadante a partir de las muestras homogeneizadas (paso 4.4.1) en los medios de infección. Preparar tubos de microcentrífuga con 540 ml de medios de infección, añadir 60 ml de la muestra de pulmones homogeneizados al primer tubo, mezclar pipeteando hacia arriba y hacia abajo, y luego usando una nueva punta, transferir 60 s al siguiente tubo. Repita este proceso de dilución en serie hasta la última dilución.
NOTA: En nuestra experiencia 7 diluciones son suficientes para determinar los tituladores virales por ensayo de placa de los pulmones de ratones infectados. - Lave las células MDCK (paso 4.4.3) dos veces con 1pbS y transfiera 500 l de las muestras diluidas en serie a cada pocal de la placa de seis pocillos. Coloque las placas sobre una plataforma de balanceo y permita que se produzca una absorción viral durante 1 h a temperatura ambiente.
- Después de 1 h de absorción viral, eliminar el inóculo viral, añadir 2 ml/pozo de medio de superposición que contenga 1 g/ml de trippsina tratada con TPCK y, a continuación, incubar las células a 33 oC por debajo del 5% de CO2 durante 3 días.
- Fijar las células infectadas (paso 4.4.6) con 1 mL/pozo de 4% de formaldehído diluido en 1x PBS a temperatura ambiente durante 2 h, luego quitar cuidadosamente el medio de superposición y añadir 1 mL de 1x PBS a cada pocto. Para la visualización de Venus, ilumine las placas de seis pocillos con un sistema de imágenes para la detección de fluorescencia.
NOTA: Las placas virales se pueden colorear utilizando un software de edición de imágenes (consulte la Tabla de materiales). - Para evaluar la expresión de Nluc, visualice las placas virales mediante inmunomancha utilizando un pAb anti-Nluc. Para ello, retire el 1x PBS, permeabilize las células usando 0.5 mL/pozo de solución de permeabilización durante 15 minutos a temperatura ambiente.
- Retire la solución de permeabilización, lave las células tres veces con 1pbS y bloquee con 0,5 ml/pozo de solución de bloqueo durante 1 h a temperatura ambiente.
- Retire la solución de bloqueo del paso 4.4.9 e incubar las células con 0,5 ml/pozo del pAb anti-Nluc diluido 1:1.000 en solución de dilución durante 1 h a 37 oC.
- Utilice el kit de fosfatasa alcalina ABC y el kit de sustrato de peroxidasa DAB siguiendo las recomendaciones del fabricante para la visualización de placas que expresan Nluc.
- Brevemente, lave las células de 4.4.10 tres veces con 1x PBS e incubarlas con 0,5 ml/pozo de anticuerpo secundario anticonejo biotinilado durante 1 h a 37 oC.
- Retire el anticuerpo secundario, lave las células tres veces con 1 x PBS e incubar con la solución ABC durante 1 h a 37 oC. Lave las células tres veces con 1x PBS y visualice las placas virales con el kit de sustrato DAB HRP. Escanee las placas inmunomanchadas utilizando un escáner convencional.
NOTA: Se podrían utilizar otros kits similares para la inmunomancha.
- Mancha las placas virales con solución violeta cristalina durante 1 h a temperatura ambiente. Deseche el cristal violeta, lave las placas tres veces con agua, deje que las placas se sequen y escanee las placas de nuevo.
- Para determinar los lanzadores virales, cuente las placas reveladas después de la tinción violeta cristalina. Escanee las placas con un escáner convencional. Calcular el títer de virus como unidades formadoras de placa (PFU) por ml (PFU/ml).
- Para evaluar la estabilidad de BIRFLU in vivo, calcule el porcentaje de virus que expresan a los periodistas contando el número de placas teñidas de cristal violeta (número de virus infecciosos, paso 4.4.12) y compártalo con el número de placas que expresan Venus y Nluc ( 4.4.7 y paso 4.4.11, respectivamente).
- Centrifugar las muestras a 300 x g durante 10 min y recoger el sobrenadante en un tubo estéril. Almacene las muestras en hielo si se utilizarán el mismo día o congele a -80 oC para evaluar los lanzadores virales más adelante.
Resultados
Generación y caracterización de BIRFLU in vitro (Figura 1 y Figura 2)
Se construyó un IAV de replicación recombinante que expresaba dos genes de reportero diferentes (BIRFLU) utilizando técnicas de biología molecular y genética inversa basadas en plásmidos de última generación (Figura1). Aquí, elegimos utilizar Nluc debido a varias ventajas sobre otras luciferasas, incluyendo su pequeño tamaño, independencia ATP, mayor intensidad, y sustrato optimizado48,60. Nluc fue clonado en el segmento HA de IAV PR8 seguido del teschovirus porcino (PTV) 2A sitio de escisión (2A) delante del marco de lectura abierto (ORF) de HA (Figura 1). El ORF de HA incluía mutaciones silenciosas para eliminar las señales de embalaje originales y evitar cualquier posible recombinación. La señal de embalaje HA completa se añadió delante de Nluc para permitir la incorporación adecuada del segmento HA modificado en la expresión virión y Nluc y HA del mismo segmento de ARN viral (Figura1). Además, la proteína fluorescente Venus fue clonada en un segmento modificado IAV PR8 NS que codifica las dos proteínas virales NS1 y NEP de una sola transcripción32,36,41,54, 57. Con ese fin, Venus se fusionó con el Terminal C de NS1 y todo el ORF NEP fue clonado aguas abajo del sitio deescisión PTV 2A que se colocó entre las secuencias NS1-Venus y NEP (Figura 1). En última instancia, estas dos construcciones modificadas de plásmido viral HA y NS se utilizaron en combinación con el resto de los plásmidos de genética inversa IAV PR8 para generar BIRFLU (Figura1). La caracterización in vitro e in vivo de BIRFLU se ha descrito previamente55.
En la Figura2, caracterizamos birFLU in vitro determinando los niveles de expresión de Venus, Nluc y NP utilizando enfoques de fluorescencia e inmunofluorescencia indirecta (Figura 2A,B). Las monocapas confluentes de células MDCK fueron infectadas o infectadas por simulacros (MOI 0.1) con virus WT o BIRFLU PR8 y, a las 18 h después de la infección, la expresión de Venus se evaluó directamente mediante microscopía de fluorescencia (Figura2A,B). La expresión Nluc (Figura2A)y NP (Figura2B)se visualizó mediante inmunofluorescencia indirecta utilizando anticuerpos específicos para cada proteína. Como se preveía, la expresión de Venus y Nluc se detectó sólo en células infectadas por BIRFLU y no en células infectadas por el virus WT PR8. Además, la microscopía de inmunofluorescencia indirecta reveló la expresión NP en células infectadas por WT y BIRFLU PR8. No se detectó ninguna expresión de Venus, Nluc o NP, como se esperaba, en células infectadas simuladas (FiguraA,B).
Para evaluar los niveles de expresión de Nluc in vitro, las células MDCK se infectaron (MOI 0.001) con virus WT o BIRFLU PR8 y se evaluó la actividad de Nluc en los sobrenatos de cultivo tisular a 24, 48, 72 y 96 h después de la infección (Figura2C). Sólo se detectó actividad nluc en supernatantes de cultivo de tejido de células MDCK infectadas con BIRFLU (Figura2C). La actividad de Nluc en los sobrenatantes del cultivo tisular se detectó tan pronto como 24 h después de la infección con niveles más altos de expresión a 96 h después de la infección, probablemente porque el efecto citopático (CPE) inducido durante la infección viral libera la proteína Nluc retenida en la celda. Para evaluar la idoneidad de BIRFLU en células cultivadas, también se evaluó la cinética de crecimiento de los virus WT y BIRFLU PR8 (Figura 2D) y la presencia de virus infecciosos en los sobrenatos de cultivo de tejidos se determinó mediante un ensayo de enfoque inmune (Figura2D) ). En particular, la cinética de replicación de BIRFLU fue comparable a la del virus WT PR8, aunque la replicación de BIRFLU se retrasó ligeramente y no alcanzó los mismos lanzadores virales que EL WT PR8. Sin embargo, BIRFLU fue capaz de alcanzar lanzadores de 5 x 107 PFU/ml (Figura2D),lo que indica que la expresión de dos genes reporteros en el genoma viral no interfiere significativamente con la replicación de BIRFLU en células MDCK.
Seguimiento de la infección por BIRFLU en ratones(Figura3 y Figura 4)
La Figura 3 es un diagrama de flujo esquemático para la evaluación de la dinámica BIRFLU en un modelo de ratón de infección por IAV. Los ratones BALB/C de cinco a siete semanas de edad se infectaron simulados con 1 pbS o se infectaron con 1 x 106 PFU de BIRFLU por vía intranasal. A los 3 días después de la infección, los ratones fueron anestesiados con isoflurano y luego inyectados con sustrato de Nluc retro-orbitalmente. Todos los ratones se colocaron inmediatamente en el instrumento IVIS y la señal Nluc se evaluó in vivo utilizando el IVIS. A continuación, los ratones fueron eutanasiados y los pulmones fueron cosechados. Los pulmones extirpados fueron analizados ex vivo utilizando el imager in vivo para determinar la intensidad de la fluorescencia a través de la expresión de Venus. Por último, los pulmones de los ratones fueron homogeneizados, y los lanzadores virales y la estabilidad se determinaron mediante el ensayo de placa. Las placas fueron evaluadas por la fluorescencia directa de Venus, mediante inmunomancha utilizando anticuerpos específicos para Nluc y por tinción violeta cristalina.
Las iAV de expresión de reporteros competentes para la replicación anteriormente expresan un solo gen de reportero, con mayor frecuencia ya sea una proteína fluorescente o bioluminiscente, como sustituto de la infección viral y la replicación. Sin embargo, BIRFLU, es capaz de expresar ambos tipos de genes de reportero sobre la infección viral. Para evaluar la correlación entre la bioluminiscencia (imágenes in vivo) y la fluorescencia (imágenes ex vivo) después de la infección por BIRFLU, los ratones BALB/c hembras de cinco a siete semanas de edad se infectaron simuladas con 1 x PBS o se inocularon con BIRFLU (106 PFU) intranasalmente . La actividad de Nluc (Figura4A)se evaluó mediante la administración del sustrato de Nluc inyectado retroorbitalmente a los 3 días posteriores a la infección utilizando un instrumento de imagen in vivo. Elegimos evaluar la bioluminiscencia en el día 3 porque estudios anteriores indicaron que la replicación de IAV, incluyendo PR8, alcanza los picos entre los días 2 y 4 después de la infección24,54. Se monitorizó la bioluminiscencia (Figura4A,parte superior) y se utilizó para calcular el flujo total promedio (Flujo (log10 p/s) (Figura4A,inferior). Como se predijo, los ratones inoculados con BIRFLU mostraron una alta actividad de bioluminiscencia, pero no se detectó ninguna señal en ratones infectados por simulacros. A partir de entonces, se cosecharon los pulmones de los ratones infectados y se evaluó la expresión de Venus mediante imágenes ex vivo (Figura4B,superior). Además, se calculó la eficiencia radiante media de fluorescencia (Figura4B,inferior). Los pulmones de los ratones extirpados también se homogeneizaron paradeterminar los lanzadores virales y la estabilidad genética de BIRFLU in vivo (Figura 4C,D). La estabilidad genética de BIRFLU se analizó mediante el ensayo de placa utilizando los virus aislados de los pulmones de ratones y la microscopía fluorescente (Venus, superior), la inmunomancha (Nluc, medio) y la tinción violeta cristalina (abajo). BIRFLU recuperado de ratones pulmones fueron capaces de formar placas y expresa establemente ambos genes reportero (Figura4C). En particular, observamos una buena correlación entre la bioluminiscencia y las señales de fluorescencia con la replicación viral.
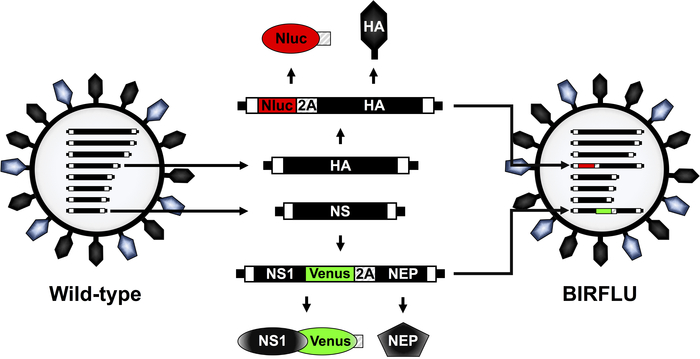
Figura 1: Representación esquemática de la estructura y los segmentos del genoma de la estructura y el genoma del IAV PR8 WT y BIRFLU. El IAV está rodeado por una bicapa lipídica que contiene las dos principales glicoproteínas virales hemagglutinina (HA; negro) y neuraminidasa (NA; azul). IAV contiene ocho segmentos de ARN de una sola cadena, sentido negativo(PB2, PB1, PA, HA, NP, NA, M y NS). Cada segmento viral contiene regiones no codificante (NCR) en los extremos de 3' y 5' (cajas negras). Además, en el extremo de 3' y 5' de los virales (v)RNA se encuentran las señales de embalaje, responsables de la encapidción eficiente de los ARNN en viriones nacientes (cajas blancas). Los segmentos y productos virales IAV PR8 HA y NS están indicados en negro. Las secuencias de Nluc, Venus y PTV 2A se indican en cajas rojas, verdes y rayadas, respectivamente. También se indica la representación esquemática de los segmentos HA y NS modificados que expresan Nluc y Venus, respectivamente, en BIRFLU. Esta figura ha sido adaptada de Nogales et al.55. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Caracterización in vitro de BIRFLU. (A, B) Análisis de la expresión proteica por fluorescencia directa e inmunofluorescencia. Las células MDCK se infectaron o infectaron (MOI 0.1) con virus PR8 WT o BIRFLU. Las células infectadas se fijaron a 18 h después de la infección para visualizar directamente la expresión de Venus mediante microscopía fluorescente directa y para visualizar la expresión Nluc (A) y VIRAL NP (B) utilizando anticuerpos específicos e inmunofluorescencia indirecta. Los núcleos se mancharon con DAPI. Se muestran imágenes representativas (aumento de 20x). Barras de escala a 100 m. (C, D) Cinética de crecimiento de PR8 WT y BIRFLU. La actividad Nluc (C) y los valoradores virales (D) en los sobrenatantes de cultivo de tejido de células MDCK infectadas (MOI 0.001) con los virus WT y BIRFLU PR8 se evaluaron en los momentos indicados después de la infección. Los datos representan los medios : SD de los triplicados. Los lanzadores virales fueron determinados por el ensayo de enfoque inmune (FFU/ml). La línea de puntos indica el límite de detección (200 FFU/ml). Esta figura ha sido adaptada de Nogales et al.55. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Representación esquemática para el estudio de BIRFLU en ratones. La expresión de los genes reporteros de Nluc y Venus se evaluó en ratones infectados con 1 x 106 PFU de BIRFLU utilizando imágenes in vivo o ex vivo. Brevemente, el día 1, 5 a 7 semanas de edad, los ratones hembra según las ratones BALB/c fueron infectados por simulacros (1x PBS) o inoculados con 1 x 106 PFU de BIRFLU por vía intranasal. En el día 3 después de la infección, los ratones fueron anestesiados levemente usando isoflurano y el sustrato de Nluc se inyectó retro-orbitalmente. La señal Nluc se evaluó directamente utilizando imágenes in vivo. Inmediatamente después de la toma de imágenes, los ratones fueron eutanasiados y la expresión de Venus en pulmones enteros extirpado fue analizada utilizando imágenes ex vivo. Los pulmones de ratones recuperados fueron homogeneizados para evaluar la replicación viral y la estabilidad mediante el ensayo de placa. Las flechas indican correlación entre fluorescencia (Venus), inmunomancha (Nluc) y tinción violeta cristalina. Haga clic aquí para ver una versión más grande de esta figura.
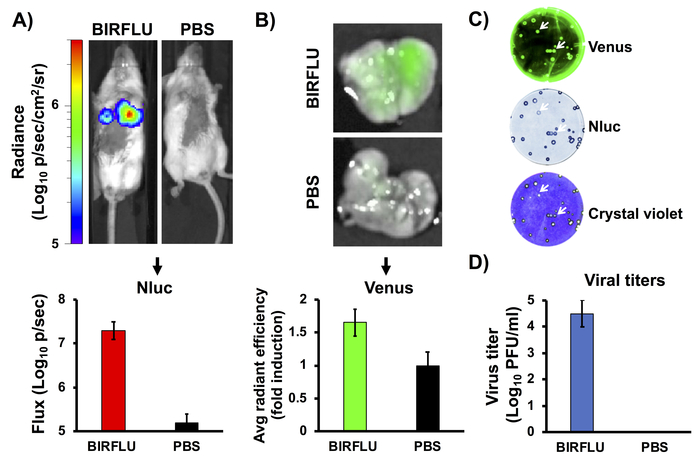
Figura 4: Expresión de bioluminiscencia y fluorescencia in vivo. Los peratones hembras de BALB/c de cinco a siete semanas de edad fueron ininfectados (1x PBS) o inoculados con 1 x 106 PFU de BIRFLU por vía intranasal. En el día 3 después de la infección, se determinó la actividad de Nluc (A) en todo el ratón. Imágenes representativas de un solo ratón que muestra la escala de resplandor (p/seg/cm2/sr). Se cuantificaron los valores de resplandor de bioluminiscencia y se muestra el flujo total medio (Flujo (Log10 p/s). Después de la toma de imágenes de Nluc, se cosecharon los pulmones para obtener imágenes ex vivo (B). Se muestran imágenes representativas de pulmones enteros. Para cuantificar la expresión de Venus, se normalizaron los valores medios de las regiones de interés (ROI) a la autofluorescencia pulmonar de ratones infectados por burlas y se calcularon los cambios de pliegue. Para analizar la estabilidad genética de BIRFLU in vivo, los virus recuperados de los pulmones de ratones fueron analizados mediante ensayo de placa utilizando microscopía fluorescente (Venus, superior), inmunomanchado (Nluc, medio) y tinción violeta cristalina (abajo) (C). Se muestran imágenes representativas de un ratón. Para evaluar la replicación del virus, los pulmones enteros se homogeneizaron después de la toma de imágenes y se utilizaron para infectar las células MDCK y determinar los valoradores virales mediante el ensayo de placa (PFU/ml) (D). Las flechas indican correlación entre fluorescencia (Venus), inmunomancha (Nluc) y tinción violeta cristalina. Las barras representan la media de las tetas de los virus pulmonares. Esta figura ha sido adaptada de Logales et al.55. Haga clic aquí para ver una versión más grande de esta figura.
| Medios y soluciones de cultivo de tejidos | Composición | Almacenamiento | Uso |
| Medios de cultivo de tejidos: El medio de eagle modificado de Dulbecco (DMEM), el suero de la vid fetal (FBS) del 10 % y el 1% de la penicilina-estreptomicina-L-glutamina (PSG)(DMEM 10 % FBS 1% PSG). | 445 ml DMEM, 50 mL de FBS y 5 mL de 100x PSG. | 4 oC | Mantenimiento de células MDCK |
| Medios de posinfección: DMEM 0,3% albúmina bovina (BA), 1% PSG (DMEM 0,3 % BA 1% PSG). | 491 ml DMEM, 4,2 ml de 35 % BA y 5 ml de 100x PSG | 4 oC | Mantenimiento de las células MDCK después de una infección viral |
| 10x Solución salina tamponada de fosfato (PBS) | 80 g de NaCl, 2 g de KCl, 11,5 g de Na2HPO4.7H2O, 2 g de KH2PO4. Añadir ddH2O hasta 1 L. Ajustar el pH a 7.3 | Temperatura ambiente | Para preparar 1x PBS |
| 1x PBS | Diluir 10pbS con ddH2O | Temperatura ambiente | Lavar las células |
| Medios de infección: 1x PBS, 0.3% BA, 1% Penicilina-Streptomicina (PS) (PBS/BA/PS). | 487 mL 1x PBS estéril, 4,2 ml de 35% BA y 5 ml de 100x 1% PS (100 U/mL) | 4 oC | Infecciones virales |
| Solución de fijación/permeabilización: 4% formaldehído, 0,5% tritón X-100 diluido en 1pbS. | 400 ml de formalina tamponada neutra 10%, 5 ml de Triton X-100 y 595 mL de 1x PBS | Temperatura ambiente | Corrección y permeabilización de células MDCK. |
| Solución de bloqueo: 2.5% albúmina de suero bovino (BSA) en 1x PBS. | 2,5 g de BSA en 97,5 ml de 1pbS | 4 oC | Solución de bloqueo para inmunofluorescencia y ensayos de placa. |
| Solución de dilución de anticuerpos (1% BSA en 1x PBS) | 1 g de BSA en 99 mL de 1x PBS | 4 oC | Dilución de anticuerpos primarios y secundarios. |
| 0.1% solución violeta cristalina | 1 g de cristal violeta en 400 ml de metanol. Añadir 600 ml de ddH2O | Temperatura ambiente | Manchado de células MDCK en ensayos de placa. |
| Tosylsulfonyl phenylalanyl chloromethyl ketone (TPCK) -trató de trippsina | Preparar una solución de stock de 1.000x a 1 mg/ml en ddH2O | -20 oC | Para infecciones virales. |
Tabla 1: Medios y soluciones de cultivo de tejidos.
Discusión
Los investigadores han confiado en los virus recombinantes que expresan a los periodistas como herramientas moleculares vitales para comprender y ampliar la comprensión actual de la replicación viral y la patogénesis26,27,28, 29 , 30 , 31 , 32 , 33 , 34 , 35 , 36 , 37 , 38 , 39 , 40 , 41 , 54. Los genes de reportero más comúnmente favorecidos son las luciferas y las proteínas fluorescentes, principalmente debido a los avances tecnológicos en su identificación, desarrollo de variantes mejoradas y detección por tecnologías de imagen43 , 44 , 45 , 46 , 47 , 48. Los virus de reporteros recombinantes se utilizan a menudo para acelerar los ensayos virológicos, estudiar la dinámica de los virus in vitro e in vivo, y para probar la eficacia de las vacunas y enfoques terapéuticos actualmente aprobados o nuevos26, 27,28,29,30,31,32,33,34,35, 36,37,38,39,40,41,54. Desafortunadamente, en el caso de IAV, estudios anteriores se limitaron a la expresión de un gen de un solo reportero, lo que dificulta el tipo de estudio que se podía llevar a cabo 26,27,28,29 , 30 , 31 , 32 , 33 , 34 , 35 , 36 , 37 , 38 , 39 , 40 , 41 , 54. Para evitar esta limitación, hemos generado un IAV bi-reportero competente para la replicación que expresa una Nluc luciferasa y una proteína fluorescente Venus (BIRFLU).
En este informe, describimos la caracterización in vitro de BIRFLU y los enfoques experimentales para utilizar BIRFLU para realizar un seguimiento de la infección viral in vivo utilizando un modelo de ratón de infección por IAV. Expresión BIRFLU Nluc y Venus correlacionada con tituladores virales. Además, BIRFLU se mantuvo estable y continuó expresando ambos genes de reportero después de ser recuperados de los pulmones de ratones infectados. Este enfoque ofrece a los investigadores una excelente oportunidad para estudiar el IAV en células cultivadas y en modelos animales, incluyendo la identificación y desarrollo de nuevas alternativas terapéuticas para el tratamiento de infecciones por IAV.
Aunque BIRFLU se ha generado utilizando la columna vertebral de PR8, se podrían generar otros IAV recombinantes utilizando diferentes tipos, subtipos o cepas de tensión viral utilizando el mismo enfoque experimental. Asimismo, en este informe describimos los procedimientos experimentales para el uso de BIRFLU en un modelo de ratón de IAV. Sin embargo, BIRFLU podría ser una tecnología valiosa para evaluar la infección por IAV en otros modelos animales.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
La investigación sobre el virus de la gripe en el laboratorio LM-S está parcialmente financiada por el Centro de Excelencia en La Gripe de Nueva York (NYICE) (NIH 272201400005C), miembro del contrato No de los Centros de Excelencia para la Investigación y Vigilancia de la Gripe (CEIRS) de NIAID. HHSN272201400005C (NYICE) y por el Programa de Investigación Médica Revisada por Pares (PRMRP) del Departamento de Defensa (DoD) otorgan W81XWH-18-1-0460.
Materiales
| Name | Company | Catalog Number | Comments |
| 12-well Cell Culture Plate | Greiner Bio-one | 665102 | |
| 24-well Cell Culture Plate | Greiner Bio-one | 662160 | |
| 6-well Cell Culture Plate | Greiner Bio-one | 657160 | |
| 96-well Cell Culture Plate | Greiner Bio-one | 655-180 | |
| Adobe Photoshop CS4 | Adobe | This software is used in 3.1.10 and 4.4.7 | |
| Bovin Albumin solution (BA) | Sigma-Aldrich | A7409 | Store at 4 ° C |
| Bovin Serum Albumin (BSA) | Sigma-Aldrich | A9647 | Store at 4 °C |
| Cell Culture dishes 100mm | Greiner Bio-one | 664-160 | |
| ChemiDoc MP Imaging System | BioRad | This instrument is used in 4.4.7 | |
| Crystal Violet | Thermo Fisher Scientific | C581-100 | Store at Room temperature |
| Dounce Tissue Grinders | Thomas Scientific | 7722-7 | |
| Dulbecco’s modified Eagle’s medium (DMEM) | Corning Cellgro | 15-013-CV | Store at 4 °C |
| Fetal Bovine Serum (FBS) | Seradigm | 1500-050 | Store at -20 °C |
| 5 to 7 week-old female BALB/c mice | National Cancer Institute (NCI) | 555 | |
| Isoflurane | Baxter | 1001936040 | Store at Room temperature |
| IVIS Spectrum | PerkinElmer | 124262 | This instrument is used for in vivo imaging (4.2 and 4.3) |
| IX81 Motorized Inverted Microscope | Olympus | Olympus IX81 | |
| Living Image 4.7.2 software | PerkinElmer | This instrument is used for in vivo imaging (4.2 and 4.3) | |
| Lumicount | Packard | This instrument is used for quantifying luciferase activity (3.2.6) | |
| Madin-Darby Canine Kidney (MDCK) epithelial cells | ATCC | CCL-34 | |
| Monoclonal Antibody anti-NP Influenza A Virus HB-65 | ATCC | H16-L10-4R5 | Store at -20 °C |
| Nano-Glo Luciferase Assay Reagent | Promega | N1110 | This reagent is used to measure Nluc activity. Store at -20 °C |
| Neutral Buffered Formalin 10% | EMD | 65346-85 | Store at RT |
| Nunc MicroWell 96-Well Microplates | Thermo Fisher Scientific | 269620 | |
| Penicillin/Streptomycin (PS) 100x | Corning | 30-00-CI | Store at -20 °C |
| Penicillin/Streptomycin/L-Glutamine (PSG) 100x | Corning | 30-009-CI | Store at -20 °C |
| Retiga 20000R Fast1394 Camera | Qimaging | Retiga 2000R | |
| Scanner | HP | ||
| Texas Red-conjugated anti-mouse -rabbit secondary antibodies | Jackson | 715-075-150 | Store at -20 °C |
| Tosylsulfonyl phenylalanyl chloromethyl ketone (TPCK)-treated trypsin | Sigma-Aldrich | T8802 | Store at -20 °C |
| Triton X-100 | J.T.Baker | X198-07 | Store at RT |
| Vmax Kinetic plate reader | Molecular Devices |
Referencias
- Palese, P., Shaw, M. L., Knipe, D. M., Howley, P. M., Griffin, D. E., Lamb, R. A., Martin, M. A. Orthomyxoviridae: The Viruses and Their Replication. Fields Virology. , (2007).
- Martinez-Sobrido, L., Peersen, O., Nogales, A. Temperature Sensitive Mutations in Influenza A Viral Ribonucleoprotein Complex Responsible for the Attenuation of the Live Attenuated Influenza Vaccine. Viruses. 10 (10), (2018).
- Nogales, A., Martinez-Sobrido, L. Reverse Genetics Approaches for the Development of Influenza Vaccines. International Journal of Molecular Sciences. 18 (1), (2016).
- Neumann, G., Noda, T., Kawaoka, Y. Emergence and pandemic potential of swine-origin H1N1 influenza virus. Nature. 459 (7249), 931-939 (2009).
- Louie, J. K., et al. A review of adult mortality due to 2009 pandemic (H1N1) influenza A in California. PLoS One. 6 (4), e18221 (2011).
- Barr, I. G., et al. Epidemiological, antigenic and genetic characteristics of seasonal influenza A(H1N1), A(H3N2) and B influenza viruses: basis for the WHO recommendation on the composition of influenza vaccines for use in the 2009-2010 Northern Hemisphere season. Vaccine. 28 (5), 1156-1167 (2010).
- Simonsen, L., et al. Impact of influenza vaccination on seasonal mortality in the US elderly population. A.M.A. archives of internal medicine. 165 (3), 265-272 (2005).
- McLean, H. Q., Peterson, S. H., King, J. P., Meece, J. K., Belongia, E. A. School absenteeism among school-aged children with medically attended acute viral respiratory illness during three influenza seasons, 2012-2013 through 2014-2015. Influenza and Other Respiratory Viruses. 11 (3), 220-229 (2017).
- Principi, N., Esposito, S. Protection of children against influenza: Emerging problems. Human Vaccines and Immunotherapeutics. , 1-8 (2017).
- Falsey, A. R., Treanor, J. J., Tornieporth, N., Capellan, J., Randomized Gorse, G. J. double-blind controlled phase 3 trial comparing the immunogenicity of high-dose and standard-dose influenza vaccine in adults 65 years of age and older. Journal of Infectious Diseases. 200 (2), 172-180 (2009).
- Fuller, J. D., et al. Influenza vaccination of human immunodeficiency virus (HIV)-infected adults: impact on plasma levels of HIV type 1 RNA and determinants of antibody response. Clinical Infectious Diseases. 28 (3), 541-547 (1999).
- Carrat, F., Flahault, A. Influenza vaccine: the challenge of antigenic drift. Vaccine. 25 (39-40), 6852-6862 (2007).
- Doherty, P. C., Kelso, A. Toward a broadly protective influenza vaccine. Journal of Clinical Investigation. 118 (10), 3273-3275 (2008).
- Fiore, A. E., et al. Prevention and control of influenza with vaccines: recommendations of the Advisory Committee on Immunization Practices (ACIP), 2010. MMWR Recommendations and Reports. 59 (RR-8), 1-62 (2010).
- Pica, N., Palese, P. Toward a universal influenza virus vaccine: prospects and challenges. Annual Review of Medicine. 64, 189-202 (2013).
- To, K. K., Chan, J. F., Chen, H., Li, L., Yuen, K. Y. The emergence of influenza A H7N9 in human beings 16 years after influenza A H5N1: a tale of two cities. Lancet Infectious Diseases. 13 (9), 809-821 (2013).
- Baker, S. F., Nogales, A., Santiago, F. W., Topham, D. J., Martinez-Sobrido, L. Competitive detection of influenza neutralizing antibodies using a novel bivalent fluorescence-based microneutralization assay (BiFMA). Vaccine. 33 (30), 3562-3570 (2015).
- He, W., Mullarkey, C. E., Miller, M. S. Measuring the neutralization potency of influenza A virus hemagglutinin stalk/stem-binding antibodies in polyclonal preparations by microneutralization assay. Methods. , (2015).
- Kayali, G., et al. Testing human sera for antibodies against avian influenza viruses: horse RBC hemagglutination inhibition vs. microneutralization assays. Journal of Clinical Virology. 43 (1), 73-78 (2008).
- Stephenson, I., et al. Reproducibility of serologic assays for influenza virus A (H5N1). Emerging Infectious Diseases. 15 (8), 1252-1259 (2009).
- Maher, J. A., DeStefano, J. The ferret: an animal model to study influenza virus. Lab Animal (NY). 33 (9), 50-53 (2004).
- Webster, R. G., Cox, N., Stoehr, K. . WHO/CDS/CSR/NCS/2002.5 Rev. 1. , (2002).
- Rodriguez, L., Nogales, A., Martínez-Sobrido, L. Influenza A Virus Studies in a Mouse Model of Infection. Journal of Visualized Experiments. (127), (2017).
- Cox, A., Baker, S. F., Nogales, A., Martínez-Sobrido, L., Dewhurst, S. Development of a mouse-adapted live attenuated influenza virus that permits in vivo analysis of enhancements to the safety of live attenuated influenza virus vaccine. Journal of Virology. 89 (6), 3421-3426 (2015).
- Steel, J., Lowen, A. C., Mubareka, S., Palese, P. Transmission of influenza virus in a mammalian host is increased by PB2 amino acids 627K or 627E/701N. PLoS Pathogens. 5 (1), e1000252 (2009).
- Tran, V., Moser, L. A., Poole, D. S., Mehle, A. Highly sensitive real-time in vivo imaging of an influenza reporter virus reveals dynamics of replication and spread. Journal of Virology. 87 (24), 13321-13329 (2013).
- Fukuyama, S., et al. Multi-spectral fluorescent reporter influenza viruses (Color-flu) as powerful tools for in vivo studies. Nature Communications. 6, 6600 (2015).
- Manicassamy, B., et al. Analysis of in vivo dynamics of influenza virus infection in mice using a GFP reporter virus. Proceedings of the National Academy of Sciences U S A. 107 (25), 11531-11536 (2010).
- Perez, J. T., Garcia-Sastre, A., Manicassamy, B. Insertion of a GFP reporter gene in influenza virus. Current Protocols in Microbiology. , (2013).
- Reuther, P., et al. Generation of a variety of stable Influenza A reporter viruses by genetic engineering of the NS gene segment. Scientific Reports. 5, 11346 (2015).
- Tran, V., et al. Multi-Modal Imaging with a Toolbox of Influenza A Reporter Viruses. Viruses. 7 (10), 5319-5327 (2015).
- Breen, M., Nogales, A., Baker, S. F., Perez, D. R., Martinez-Sobrido, L. Replication-Competent Influenza A and B Viruses Expressing a Fluorescent Dynamic Timer Protein for In Vitro and In Vivo Studies. PLoS One. 11 (1), e0147723 (2016).
- Nogales, A., Baker, S. F., Martinez-Sobrido, L. Replication-competent influenza A viruses expressing a red fluorescent protein. Virology. , 206-216 (2014).
- Nogales, A., et al. Replication-competent fluorescent-expressing influenza B virus. Virus Research. 213, 69-81 (2015).
- Avilov, S. V., et al. Replication-competent influenza A virus that encodes a split-green fluorescent protein-tagged PB2 polymerase subunit allows live-cell imaging of the virus life cycle. Journal of Virology. 86 (3), 1433-1448 (2012).
- Breen, M., Nogales, A., Baker, S. F., Martínez-Sobrido, L. Replication-Competent Influenza A Viruses Expressing Reporter Genes. Viruses. 8 (7), (2016).
- Eckert, N., et al. Influenza A virus encoding secreted Gaussia luciferase as useful tool to analyze viral replication and its inhibition by antiviral compounds and cellular proteins. PLoS One. 9 (5), e97695 (2014).
- Karlsson, E. A., et al. Visualizing real-time influenza virus infection, transmission and protection in ferrets. Nature Communications. 6, 6378 (2015).
- Czako, R., et al. In Vivo Imaging of Influenza Virus Infection in Immunized Mice. MBio. 8 (3), (2017).
- Harding, A. T., Heaton, B. E., Dumm, R. E., Heaton, N. S. Rationally Designed Influenza Virus Vaccines That Are Antigenically Stable during Growth in Eggs. MBio. 8 (3), (2017).
- DiPiazza, A., et al. Pandemic 2009 H1N1 Influenza Venus reporter virus reveals broad diversity of MHC class II-positive antigen-bearing cells following infection in vivo. Scientific Reports. 7 (1), 10857 (2017).
- Yan, D., et al. Replication-Competent Influenza Virus and Respiratory Syncytial Virus Luciferase Reporter Strains Engineered for Co-Infections Identify Antiviral Compounds in Combination Screens. Biochemistry. 54 (36), 5589-5604 (2015).
- Shaner, N. C., Patterson, G. H., Davidson, M. W. Advances in fluorescent protein technology. Journal of Cell Science. 120 (Pt 24), 4247-4260 (2007).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nature Methods. 2 (12), 905-909 (2005).
- Kelkar, M., De, A. Bioluminescence based in vivo screening technologies. Current Opinion in Pharmacology. 12 (5), 592-600 (2012).
- Welsh, D. K., Noguchi, T. Cellular bioluminescence imaging. Cold Spring Harbor Protocols. 2012 (8), (2012).
- Zhao, H., et al. Emission spectra of bioluminescent reporters and interaction with mammalian tissue determine the sensitivity of detection in vivo. Journal of Biomedical Optics. 10 (4), 41210 (2005).
- Stacer, A. C., et al. NanoLuc reporter for dual luciferase imaging in living animals. Molecular Imaging. 12 (7), 1-13 (2013).
- Vintersten, K., et al. Mouse in red: red fluorescent protein expression in mouse ES cells, embryos, and adult animals. Genesis. 40 (4), 241-246 (2004).
- Stacer, A. C., et al. NanoLuc reporter for dual luciferase imaging in living animals. Mol Imaging. 12 (7), 1-13 (2013).
- Schoggins, J. W., et al. Dengue reporter viruses reveal viral dynamics in interferon receptor-deficient mice and sensitivity to interferon effectors in vitro. Proc Natl Acad Sci U S A. 109 (36), 14610-14615 (2012).
- Pan, W., et al. Visualizing influenza virus infection in living mice. Nat Commun. 4, 2369 (2013).
- Heaton, N. S., et al. In vivo bioluminescent imaging of influenza a virus infection and characterization of novel cross-protective monoclonal antibodies. J Virol. 87 (15), 8272-8281 (2013).
- Nogales, A., Baker, S. F., Martínez-Sobrido, L. Replication-competent influenza A viruses expressing a red fluorescent protein. Virology. 476, 206-216 (2015).
- Nogales, A., et al. A novel fluorescent and bioluminescent Bi-Reporter influenza A virus (BIRFLU) to evaluate viral infections. Journal of Virology. , (2019).
- Nogales, A., et al. Influenza A Virus Attenuation by Codon Deoptimization of the NS Gene for Vaccine Development. Journal of Virology. 88 (18), 10525-10540 (2014).
- Nogales, A., DeDiego, M. L., Topham, D. J., Martinez-Sobrido, L. Rearrangement of Influenza Virus Spliced Segments for the Development of Live-Attenuated Vaccines. Journal of Virology. 90 (14), 6291-6302 (2016).
- National Research Council (U.S.). Committee for the Update of the Guide for the Care and Use of Laboratory Animals., Institute for Laboratory Animal Research (U.S.) & National Academies Press (U.S.). . Guide for the care and use of laboratory animals. , (2011).
- Nogales, A., Martinez-Sobrido, L., Chiem, K., Topham, D. J., DeDiego, M. L. Functional Evolution of the 2009 Pandemic H1N1 Influenza Virus NS1 and PA in Humans. Journal of Virology. 92 (19), (2018).
- Hall, M. P., et al. Engineered luciferase reporter from a deep sea shrimp utilizing a novel imidazopyrazinone substrate. ACS Chemical Biology. 7 (11), 1848-1857 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados