Method Article
À l’aide de Sniper-Cas9 pour minimiser les effets hors cible de CRISPR-Cas9 sans la perte d’activité sur la cible Via évolution dirigée
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Nous présentons ici un protocole pour optimiser CRISPR-Cas9 pour parvenir à une plus grande spécificité sans la perte d’activité sur la cible. Nous utilisons une approche d’évolution dirigée appelée Sniper-écran pour trouver un mutant Cas9 avec les caractéristiques souhaitées. Sniper-Cas9 est compatible avec tronqué ARN simple-guide et livraison dans un format ribonucléoprotéines, célèbres stratégies pour atteindre les spécificités plus élevées.
Résumé
Le développement de cluster régulièrement dois‑je répétitions courtes palindromiques (CRISPR)-protéine associée 9 (Cas9) dans les modalités thérapeutiques nécessite d’éviter les effets potentiellement préjudiciables hors cible. Plusieurs méthodes ont été élaborées pour réduire ces effets. Nous présentons ici un Escherichia coli-évolution dirigée selon méthode appelée Sniper-écran pour obtenir une variante Cas9 avec spécificité optimisée et conservé l’activité sur la cible, appelée Sniper-Cas9. À l’aide de Sniper-écran, sélection positif et négatif peut être effectuée simultanément. L’écran peut également être répété avec autres séquences d’ARN (sgRNA) single-guide pour enrichir les vrai succès positive. En utilisant le promoteur double CMV-PltetO1 pour exprimer les variantes Cas9, la performance de la bibliothèque de mise en commun peut être rapidement vérifiée dans les cellules de mammifères. On décrit également les méthodes pour augmenter la spécificité du tireur d’élite-Cas9. Tout d’abord, l’utilisation de sgRNAs tronqué antérieurement s’est avérée qu’en augmenter la spécificité Cas9. Contrairement à d’autres Cas9s ingénierie, tireur d’élite-Cas9 conserve un niveau de (WT) de type sauvage de l’activité sur la cible lorsqu’il est combiné avec sgRNAs tronqué. Deuxièmement, l’exécution du tireur d’élite-Cas9 dans un format de ribonucléoprotéines (RNP) non pas un plasmide est possible sans affecter son activité sur la cible.
Introduction
Dans cet article, notre objectif est d’améliorer la spécificité de Cas9 en combinant différentes stratégies. Diverses méthodes d’éviter les hors-cible des effets de CRISPR-Cas9 ont été développées. Par exemple, sgRNAs tronqué permet d’atteindre plus de spécificité1. En outre, la méthode de livraison Cas9 peut être modifiée d’un format de plasmide dans un format de RNP pour obtenir plus de spécificité2. Résidus d’acides aminés spécifiques de la Cas9 Streptococcus pyogenes protéine (SpCas9) ont été modifiés selon le rationnel conception décrite précédemment3,4,5. Alternativement, les résidus d’acides aminés ont été modifiés de façon aléatoire et les variantes de Cas9 avec la spécificité plus élevée ont été identifiés à l’aide d’une levure6 ou7, e.coli8 système de dépistage.
Cependant, de nombreux groupes ont rapporté que Cas9 variantes conçues à l’aide de la conception à affaiblir les interactions non spécifiques entre Cas9 et le substrat pièce faible activité sur la cible7,8,9, 10 , 11 , 12. nous avons développé un e. coli-système d’évolution dirigée, Sniper-écran, à l’écran au hasard mutagénisé Cas9 variantes. Un e. coli , système de contrôle a des avantages par rapport à un système de levure en raison de l’efficacité de transformation de temps et plus plus vite doublement d’Escherichia coli.
Sélection tant négative que positive, basée sur trois différents plasmides et un gène d’intérêt (GOI) intégré dans le génome d’e. coli , sont utilisés en Sniper-écran. Variantes de Cas9 sont exprimées selon le système de double-promoteur de CMV-PltetO1 d’un plasmide numéro de faibles copies afin que les candidats identifiés chez e. coli peuvent être testés dans des cellules de mammifères sans nécessiter de subcloning. Le gouvernement indonésien est introduit dans le génome de Escherichia coli en utilisant le système de transposon Tn7. Le plasmide sgRNA, qui contient une origine sensibles à la température de réplication, exprime une sgRNA ciblant le GOI; Toutefois, les sgRNA et les séquences GOI parfaitement ne correspondent pas. Un site de cible parfaitement appariées sgRNA existe sur un troisième plasmide contenant le gène de la BCDC , qui encode un produit mortel qui empoisonne la gyrase. Dans ce système, les cellules exprimant Cas9 variantes avec une activité élevée hors cible sont supprimés parce que cassures double-brin (CDB) sont introduits dans le site ne correspondent pas, situé dans l’ADN génomique. En revanche, les cellules exprimant Cas9 variantes avec faible activité sur la cible sont également supprimés à cause de l’expression des gènes létaux BCDC . Le niveau d’expression des variantes Cas9 peut être modifié en changeant la concentration de l’anhydrotétracycline (ATC), qui ajuste la force de la sélection.
Nous avons pensé que la localisation du site de cible ne correspondent pas sgRNA dans l’ADN génomique, plutôt que sur un plasmide augmenterait la sensibilité du système. L’avantage de cette approche est qu’il y a qu’un seul site génomique, alors qu’il y aurait plusieurs plasmides, chacun contenant un site cible, au sein d’une même cellule d’e. coli .
Grâce à ce système, nous avons identifié une variante Cas9, tireur d’élite-Cas9, qui montre les activités au niveau des WT sur la cible et réduit les activités hors-cible par rapport au WT Cas9. Sniper-Cas9 peut atteindre des taux de spécificité encore plus élevé en utilisant sgRNAs tronqué ou prestation RNP plutôt que prestation axée sur le plasmide.
Protocole
1. intégration d’un humain IgE dans la souche e. coli de la BW25141
-
Clonage de l' IgE
- Réaction en chaîne par polymérase (PCR) amplifient une longueur de 500 bp d’un humain GOI contenant divers sites cible de candidats grâce à des méthodes PCR standards avec des amorces contenant des sites de restriction enzyme NotI et XhoI.
Remarque : Dans cette expérience, le gouvernement indonésien a été le gène humain de l’EMX1 . - Digérer le produit de la PCR et le vecteur de13 pgrg36 avec des enzymes de restriction NotI et XhoI.
- Gel de purifier les fragments désirés (500 bp et 12ko).
- Ligaturer les fragments ensemble à l’aide de T4 ligase. Pour ce faire, mélanger 50 ng de pgrg36 digérées et 6 ng de l’insert de la PCR dans un volume réactionnel de 20 μL contenant 1 tampon de ligase x et enzyme de ligase 0,5 U. Incuber une nuit à température ambiante (RT).
- Transformer le plasmide ligaturé en cellules compétentes DH5-α. Poussent les transformants qui en résulte sur les boîtes de gélose de bouillon (LB) de Luria contenant l’ampicilline (100 μg/mL) à 32 ° C et incuber pendant la nuit. Chercher une colonie et grandir dans un milieu LB contenant ampicilline durant la nuit. Isoler l’ADN de l' e. coli, en utilisant un miniprep commercialement disponible Kit de plasmide. Confirmer l’insertion du fragment GOI par Sanger sequencing le plasmide obtenu à partir du mini-plasmide préparation (mini-préparent)13.
- Réaction en chaîne par polymérase (PCR) amplifient une longueur de 500 bp d’un humain GOI contenant divers sites cible de candidats grâce à des méthodes PCR standards avec des amorces contenant des sites de restriction enzyme NotI et XhoI.
-
Préparation de la BW25141 -GOI
- Transformer le plasmide obtenu pgrg36 -GOI la souche e. coli de la BW25141. Il est essentiel d’utiliser une souche de BW25141 afin de minimiser le nombre de fausses positifs colonies.
- Cultiver les cellules transformées dans le tampon LB à 32 ° C durant la nuit. GDI est insérée dans l’ADN génomique de la souche BW25141 (BW25141 -GOI). Retirez le plasmide pgrg36 -GOI de la souche BW25141 -GOI utilisant le protocole standard pgrg3613. Brièvement, diluer une colonie (environ 107-fold) et il se développent sur un plat de livre à 42 ° C pendant la nuit. Ensemencer les colonies sur la plaque LB et cultivez-les à 42 ° C pendant la nuit.
- Confirmer l’insertion correcte de GOI par colonie PCR, utilisant les amorces a proposé dans le protocole de pgrg36 : 5'-GATGCTGGTGGCGAAGCTGT-3 'et 5'-GATGACGGTTTGTCACATGGA-3'. L’apprêt amplifie le site d’insertion de l’ADN génomique, et la taille des produits PCR obtenus sera 904 bp plus la taille de l’insert (500 bp dans ce cas).
- Préparer electrocompetent cellules BW25141 -GOI (un protocole détaillé est décrit dans les étapes 3.2.6–3.2.10).
2. préparation de la bibliothèque de variante Cas9
-
Préparation de la bibliothèque
- Transformer le vecteur de Cas97 dans un commercial e. coli mutateur souche (Table des matières) et suivez les instructions du fabricant pour obtenir une variante bibliothèque (le mutateur).
- Effectuer des erreurs PCR sur la séquence entière de Cas9 WT dans le vecteur de Cas9, utilisant un kit PCR sujette (Table des matières).
Remarque : Le protocole du taux d’erreur faible a été adopté dans l’affaire Sniper-Cas9 pour ne pas perturber la fonction d’origine de la protéine. - Digérer le vecteur Cas9 avec des enzymes de restriction appropriées. Gel de purifier le produit PCR (de l’étape 2.1.2) et la colonne vertébrale digérée.
Remarque : La taille du gène SpCas9 est d’environ 4,3 kb. XhoI et KpnI ont été choisis pour digérer le vecteur pBLC-SpCas9 qui a été utilisé dans le cas de Sniper-Cas9. - Assembler le fragment d’épine dorsale (de l’étape 2.1.3) et l’insert amplifié par PCR erreurs (de l’étape 2.1.3) par isotherme in vitro de recombinaison.
NOTE : plus de 500 ng de la colonne vertébrale était nécessaire pour obtenir une forte concentration de bibliothèque (erreurs PCR [EP]). Deux erreurs différents kits de PCR ont été utilisées pour préparer des bibliothèques EP dans l’affaire Sniper-Cas9 (bibliothèque de l’EP I et II). - Purifier les produits de l’Assemblée (de l’étape 2.1.4) à l’aide d’un kit de purification de l’ADN qui permet à faible volume d’élution (Table des matières). Éluer avec 6 μL d’eau exempte de nucléase (NFW) et mesurer la concentration de l’ADN.
- Transformer plus de 500 ng du vecteur de bibliothèque Cas9 (pour chacun des trois bibliothèques) dans 50 μl de cellules d’Escherichia coli electrocompetent (Table des matières). Voir le protocole d’électroporation dans étapes 3.2.1-3.2.4. Pour la préparation de cette bibliothèque, utiliser 1 mL de milieu SOC au lieu de 250 μL / 50 μL de cellules compétentes.
- Rendre au 1/100, 1:1, 000 et des dilutions de 1/10 000 du mélange contenant les cellules récupérées avec le médium de la SOC. Plaque des cellules dilués sur plaques de gélose 100 mm LB additionnés de chloramphénicol (12,5 μg/mL). Plaque les cellules restantes sur une plaque de2 245 mm. Incuber à 37 ° C pendant la nuit.
-
Calcul de la complexité de la bibliothèque
- Photographier les plaques de dilution via un système de documentation de gel ou d’une caméra numérique ordinaire. Exécutez OpenCFU logiciel14 et télécharger les photographies des plaques dilution. Définissez la zone de comptage à l’intérieur d’une plaque et supprimer les colonies fausses.
- Manuellement, multipliez le nombre de colonies par le facteur de dilution pour obtenir le nombre initial de transformants. Convertir ces numéros en forme logarithmique (base 10). Calculer la moyenne afin de déterminer la complexité de la bibliothèque.
- Lorsque la valeur de complexité souhaitée est obtenue, rassembler toutes les colonies sur la plaque carrée de 245 mm (de l’étape 2.1.7) à l’aide d’un épandeur et 20 mL de LB additionné de chloramphénicol. Ne pas pousser les colonies se sont réunis et purifier la bibliothèque de plasmide à l’aide d’un kit de midiprep commerciale.
Remarque : Plus la complexité de la bibliothèque, le mieux c’est. Lorsque le tireur d’élite-Cas9 a été identifié, une diversité de 3 x 106 a été obtenue pour chaque bibliothèque.
3. positifs et négatifs de dépistage pour faire évoluer Cas9
-
Sélection de la cible et de la construction plasmidique
- Sélectionnez une séquence cible de l’espaceur sgRNA dans gouvernement de l’Inde. Remplacer par un ou deux résidus dans le nucléotide aléatoire pour produire une séquence ne correspondent pas.
NOTE : Site de cible humaine EMX1 3 (GAGTCCGAGCAGAAGAAGAA avec GGG PAM) a été utilisé dans le cas de Sniper-Cas9. Voici les séquences non utilisées : GAGTCCGAGCAGAAagAGAA, GAacCCGAGCAGAAGAAGAA, GAGTCCGAGCAGAgGAAGAA et GAGcCCGAGCAGAAGAAGAA. - Insérez la séquence ne correspondent pas (voir l’étape 3.1.1) dans le plasmide sgRNA utilisant oligonucléotide standard (oligo)7de la procédure de clonage.
- Insérez la séquence incompatible avec un PAM à l’extrémité 3' p11-dentelle-wtx1 (Table des matières) pour construire le plasmide BCDC à l’aide de procédures clonage standard oligo15.
- Sélectionnez une séquence cible de l’espaceur sgRNA dans gouvernement de l’Inde. Remplacer par un ou deux résidus dans le nucléotide aléatoire pour produire une séquence ne correspondent pas.
-
Préparation du tireur d’élite-dépistage e. coli cellules compétentes
- Décongelez les cellules electrocompetent BW25141 -GOI sur la glace.
- Ajouter 1 ng de chacun de la BCDC plasmide et le plasmide sgRNA avec doubles non-correspondances dans 50 μl de cellules BW25141 -GOI décongelées. Doucement les cellules de la composition de pipetage et déplacez-les dans une cupule d’électroporation prérefroidies 0,1 cm.
- Transformer les e. coli avec les deux plasmides par électroporation. Ajouter 250 μL de milieu SOC immédiatement après l’électroporation. Pipetez doucement la solution pour mélanger les cellules et le milieu. Transvaser le mélange dans un tube de microtubes de 1,5 mL.
Remarque : Pour une efficacité maximale, réglez la tension 1.80 kV et que le runtime doivent être comprise entre ms 4,8 et 5,0 ms. - Récupérer les cellules transformées et les incuber à 32 ° C pendant 1 h avec agitant doucement.
- Plaque de 125 μL des cellules récupérées sur l’ampicilline (50 μg/mL) / plaque de gélose LB kanamycine (25 μg/mL) (condition de culture). Plaque les autres cellules sur un ampicilline/kanamycine/arabinose (1,5 mg/mL) de gélose LB (BCDC-exprimant la condition). Incuber à 32 ° C durant la nuit.
- Vérifier l’absence de survivants des colonies sur la base de données-exprimant la plaque de l’état. Recueillir des colonies de la plaque de condition de culture à l’aide d’un écarteur et la culture dans 250 mL de bouillon super optimale (SOB) additionné de 50 μg/mL d’ampicilline et 25 μg/mL de kanamycine à 32 ° C, avec douce agitation.
- Quand la densité optique à 600 nm (OD600) atteint 0,4, faites refroidir le ballon sur glace. Préparer de l’eau désionisée prérefroidie et une solution de glycérol à 10 % prérefroidies (stériliser avant usage).
- Centrifuger (à 4 000 x g pendant 5 min à 4 ° C) les cellules et éliminer le surnageant. Ajouter 200 mL d’eau désionisée prérefroidie. Remettre en suspension les cellules à l’aide d’une pipette sérologique de 10 mL. Répétez cette opération 3 fois.
- Laver les cellules avec 50 mL de solution de glycérol prérefroidies 10 %. Centrifuger à eux comme avant (à 4 000 x g pendant 5 min à 4 ° C).
- Jeter le surnageant et resuspendre le culot dans 300 μL d’une solution de glycérol de 10 %. Faire 50 parties aliquotes μL et congelez-les dans l’azote liquide. Stocker les cellules (cellules de dépistage Sniper) à-80 ° C.
-
Sniper-dépistage
- Transformer les Sniper-dépistage des cellules (de l’étape 3.2.10) avec 100 ng des plasmides Cas9 variant de chaque bibliothèque (à partir de mesures de 2.2.3.See étape 2.1.1 et 2.1.4). Suivez les étapes d’électroporation décrites dans les étapes 3.2.1–3.2.3.
- Transférer 250 μL des cellules dans un tube de microcentrifuge fraîches 1,5 mL. Ajouter 250 pg d’ATC pour faire une concentration finale de 10 ng/mL. Récupérer des cellules fois contenant des ATC et sans ATC (voir l’étape 3.2.4 pour l’étape de récupération).
- Plaque de 25 μl de cellules de sans ATC récupérés sur une gélose au chloramphénicol/kanamycine LB (condition non sélectif). Ajouter ATC aux cellules contenant de l’ATC récupérés pour faire une concentration finale de 100 ng/mL sur un plat de livre de 245 mm. La plaque immédiatement les cellules sur une gélose LB chloramphénicol/kanamycine/arabinose (condition sélective). Incuber une nuit à 32 ° C.
Remarque : La taille et le numéro de la plaque LB sont déterminés par la taille de la diversité de la couverture de dépistage. Dans le cas d’une boîte de pétri avec 20 mL de LB de 100 mm, ajouter 2 μg d’ATC. - Photographier les plaques. Compter le nombre de colonies viables à l’aide de logiciels de OpenCFU14. (Voir étape 2.2.1) S’assurer que le nombre de colonies sur la plaque non sélectif est au moins 10 x plus grande que la diversité de la bibliothèque pour couvrir toutes les variantes.
- Calculer la fréquence de survie comme suit.
Fréquence de survie = le nombre de colonies sur une gélose sélective / (le nombre de colonies sur une plaque non sélectif x 10) - Mettre en commun les colonies qui ont survécu sur les géloses sélectives de tous les trois bibliothèques. Incuber les colonies survivantes dans 250 mL de LB additionné avec du chloramphenicol 12,5 μg/mL à 42 ° C pendant la nuit. Isoler la bibliothèque grillagée de Cas9 ADN à l’aide d’un kit de midiprep.
Remarque : Cette étape efface le plasmide sgRNA. - Répétez le processus de présélection des étapes 3.3.1–3.3.6 jusqu'à ce que la fréquence de survie atteint un plateau. Utilisation 10 ng de plasmide Cas9 sélectionné pour transformation et 10 ng/mL d’ATV pendant la récupération. Maintenir la concentration de l’ATC à 100 ng/mL pour la condition sélective.
-
Mixer et la seconde projection
- Mélanger les variantes regroupées sélectionnées en utilisant le protocole suivant ADN-brassage. PCR amplification l’insert Cas9 dans le plasmide de Cas9 en utilisant des amorces de flanquement, 150 nucléotides de l’insertion des limites. Digest 2 μg de produit PCR amplifié avec la DNase I pour 1 min à 37 ° C.
- Purifier bp 70 – 200 des fragments de longueur à l’aide de 2 % gel d’agarose. PCR amplification des fragments purifiés. Utiliser le produit comme modèle RP amplifier l’insert Cas9 avec des amorces appropriées flanquant Cas9. Utilisez le produit final de la PCR pour construire une bibliothèque Cas9 comme indiqué au point 2.1.4.
- Préparer les nouvelles cellules de Sniper-dépistage (voir la section 3.2) avec un autre sgRNA dépareillées plasmide (voir étape 3.1.1). Refaire le processus de présélection (sections 3.2, 3.3) jusqu'à ce que la survie taux atteint un plateau. Utilisation 10 ng de plasmide Cas9 sélectionné pour transformation et 10 ng/mL ATC pendant la récupération. Maintenir la concentration de l’ATC à 10 ng/mL pour la condition sélective.
-
Sélection d’évoluée Cas9 mutants plasmides
- Après la dernière étape de la présélection, au hasard de prélever des colonies d’une centaine de la gélose sélective et de leur culture dans un milieu LB contenant du chloramphénicol à 42 ° C pendant la nuit.
- Isoler les plasmides en utilisant une procédure de miniprep et Sanger-séquence les inserts utilisant les amorces de séquençage dans Cas9.
- Choisir les variantes plus fréquentes trois albums pour les tester dans des lignées cellulaires humaines.
4. livraison de Cas9 comme un RNP avec sgRNA tronqué
-
Sélection de gènes de cible à l’aide de Cas-OFFinder
- Choisir des sites cibles avec Cas-OFFinder (http://www.rgenome.net/cas-offinder/). Sélectionnez le type de PAM approprié pour le type spécifique de Cas9 et le génome de cibles (homme, souris, poisson zèbre, etc..). Remplir dans l’onglet requête souches , choisissez le nombre de non-correspondanceet cliquez sur le bouton soumettre .
- Après que quelques secondes, le sur la cible (avec un nombre d’incompatibilité de '0') et la volonté de sites hors cible apparaissent. En général, choisissez les sites hors cible avec des décalages d’un à trois.
-
Préparation du modèle sgRNA
- Commander des oligos crRNA et tracrRNA avec les séquences suivantes de modèle, à savoir crRNA séquence : 5'-TAATACGACTCACTATAGGNNNNNNNNNNNNNNNNNNNNGTTTTAGAGCTAGAA-3'; séquence tracrRNA : 5'-AGCACCGACTCGGTGCCACTTTTTCAAGTTGATAACGGACTAGCCTTATTTTAACTTGCTATTTCTAGCTCTAAAAC-3'. Compléter la séquence de crRNA avec la séquence cible obtenue à l’étape 4.1.2 N20. Pour concevoir sgRNAs tronqué, supprimer des bases de l’extrémité 5' pour obtenir des séquences sgRNA N19, N18 ou N17.
- Préparer le mélange PCR pour l’amplification de la séquence d’encodage sgRNA comme suit : mélanger 10 μL de 5 x tampon, 0,5 μL de crRNA oligo (100 pmol/μL), 0,5 μL de tracrRNA oligo (100 pmol/μL), 2,5 μL des dNTPs (10 mM), 0,5 μL de l’ADN polymérase et 36 μL de NFW (Table des matières).
- Amplifier le modèle en utilisant les conditions suivantes, à savoir, pour la dénaturation initiale : 1 min à 98 ° C ; pour la dénaturation, recuit et extension : 10 s à 98 ° C, 15 s à 54 ° C, 20 s à 72 ° C, pendant 25 cycles ; pour l’extension finale : 5 min à 72 ° C.
- Analysons 2 μL du modèle amplifié ADN (de l’étape 4.2.3) sur un gel d’agarose de 2 %. Purifier le modèle à l’aide d’un kit de purification de PCR.
Remarque : La taille du modèle ADN est 125 bp.
-
Synthèse de sgRNA
- Préparer le mélange réactionnel pour la synthèse de sgRNA comme suit : combiner 8,5 μL du modèle ADN (de l’étape 4.2.3), 1 μL de UTP (25 mM), 1 μL de CTP (25 mM), 1 μL de GTP (25 mM), 1 μL d’ATP (25 mM), 4,2 μL de MgCl2 (100 mM) , 4,5 μL d’ARN polymérase T7 (50 U/μl), 3 μL de tampon de l’ARN polymérase x T7 10, 1,2 μL de la pyrophosphatase (0,5 U/μl), 0.75 μL d’inhibiteur de RNase (40 U/μl) et 4,2 μL de NFW.
- Incuber le mélange réactionnel à 37 ° C pendant la nuit (au moins pendant 10 h). Ajouter 0,5 μL de DNase (2 U/μl) au mélange réactionnel et incuber à 37 ° C pendant 15 à 30 min. purifier le sgRNA à l’aide d’un kit de purification d’ARN.
- Pour réduire la toxicité causée par une réponse immunitaire innée, déclenchée par la 5'-triphosphate le sgRNA16, retirez la 5'-triphosphate le guide ARN à la phosphatase alcaline intestinale de veau (CIP) comme suit : traiter 10 µg de in vitro- ARN transcrit avec 250 U de l’ICU pendant 3 h à 37 ° C en présence d’inhibiteur de U de RNase 100. Purifier le sgRNA CIP-traités à l’aide d’un kit de purification d’ARN.
5. Cas9 WT - et Sniper expression de la protéine et de purification
-
Expression de la protéine chez e. coli
- Transformer les plasmides se peloter18 encodage Cas9 WT et tireur d’élite, His-tag dans le BL21 (DE3) e. coli de souche.
- Ensemencer à 50 mL de milieu LB contenant 50 kanamycine μg/mL avec une colonie frais nourrissait l’animal-Cas9 expression plasmide et la secousse il nuit (200 tr/min) à 37 ° C (préculture).
- Transférer 10 mL de la culture au jour le jour à 500 mL de milieu LB frais contenant 50 kanamycine μg/mL. Incuber la culture avec la secousse (200 tr/min) à 37 ° C pendant 2 h.
- Surveiller l' OD600 jusqu'à ce que la culture atteint la phase logarithmique de croissance (OD600 ≈ 0,6 à 0,7).
- Induire l’expression de la protéine WT - ou Sniper-Cas9 avec isopropyl β-D-1-thiogalactopyranoside (IPTG, à une concentration finale de 0,25 nM). Incubez la culture à 18 ° C durant la nuit.
-
Purification de protéine
- Récolter les cellules par centrifugation à 5 000 x g pendant 10 min à 4 ° C.
- Resuspendre le culot dans le tampon de lyse (50 mM NaH2PO4, 300 mM NaCl, imidazole de 10 mM, 4 mM dithiothréitol [TNT], 5 mM benzamidine, fluorure de phénylméthylsulfonyle 100 mM [PMSF], pH 8) à 20 mL par gramme de poids frais.
- Ajouter PMSF, DTT et le lysozyme à une concentration finale de 1 mg/mL chacun et incuber le mélange sur la glace pendant 30 min.
- Laisser agir les cellules sur la glace. Pouls à plusieurs reprises pendant 10 s à 200 – 300 W avec un 10 s délai entre chaque impulsion, pour une durée totale de 20 min de refroidissement.
- Centrifuger le lysat à 6 000 x g pendant 30 min à 4 ° C.
- Retirez le surnageant dans un nouveau tube. Ajouter 1 mL de résine d’agarose His-Bind à 5 mL d’effacé lysat et agiter doucement pendant 1 h à 4 ° C.
- Charger le mélange de résine de Agarose lysat/His-Bind sur une colonne avec une sortie vers le bas plafonnés.
- Retirer le bouchon de fond et récupérer la colonne intermédiaire.
- Laver la colonne 2 x avec tampon de lavage (50 mM NaH2PO4, 300 mM NaCl, imidazole de 20 mM, pH 8) ; recueillir la fraction de lavage pour l’analyse par électrophorèse sur gel sodium dodecyl sulfate polyacrylamide (SDS-PAGE).
- Éluer la protéine 10 x 1 ml d’élution buffer (50 mM NaH2PO4, 300 mM NaCl, imidazole de 250 mM, pH 8), échantillons collecteurs pour SDS-PAGE.
- Concentrer la protéine éluée WT - ou Sniper-Cas9 en utilisant un filtre de colonne 100 kDa. Stocker les échantillons dans une solution de 10 mM Tris-HCl, NaCl 150 mM et 50 % de glycérol à-80 ° C.
6. livraison de RNP
-
Transfection et préparation des cellules pour la livraison de RNP
- Maintenir les cellules HEK293T dans de Dulbecco aigle modifié (DMEM) additionné de 10 % sérum fœtal (SVF) et 1 % des antibiotiques à 37 ° C, avec 5 % de CO2.
- Mélanger protéine WT - ou Sniper-Cas9 (2 μg) avec sgRNA (2 μg) et incuber pendant 10 min à ta faire complexes RNP.
- Trypsinize et compter les cellules non vides. Préparer 2 x 104 cellules par une réaction. Laver les cellules avec une solution saline tamponnée au phosphate (PBS) et centrifuger. Aspirer le surnageant et resuspendre le culot avec le tampon d’électroporation.
- Complexes d’oligonucléotides RNP dans les cellules en utilisant les paramètres suivants, à savoir 1 300 V, 30 ms et une impulsion. Plaque de cellules sur une plaque de 48 puits remplis de 500 μL de DMEM supplémenté avec FBS et antibiotiques (comme indiqué au point 6.1.1) juste après l’électroporation. Incuber à 37 ° C, avec 5 % de CO2.
- Isoler l’ADN génomique avec un kit de préparation Adng, 48 h après la transfection.
7. la transfection des plasmides codant pour Sniper-Cas9 et sgRNA
-
Construction d’un plasmide sgRNA
- Commander avant et marche arrière oligos avec les séquences suivantes du modèle, à savoir avant : 5'-CACCGNNNNNNNNNNNNNNNNNNNN-3'; inverse : 5'-AAACNNNNNNNNNNNNNNNNNNNNC-3'. Remplacer N20 avec la séquence cible obtenue à l’étape 4.1.2. Raccourcir la séquence cible à une longueur de N19, N18, N17 ou à synthétiser sgRNA tronqué.
- Recuire les deux oligos dans 1 x tampon de T4 DNA ligase.
- Digérer le vecteur pRG2 avec BsaI enzyme de restriction.
- Gel de purifier le vecteur digéré (3 300 bp) à l’aide d’un gel d’agarose 0,8 %.
- Ligaturer les oligo recuit et le fragment purifié à l’aide de ligase T4 à 37 ° c : mélange 50 ng de pRG2 digérées et 1 ng de recuit oligo dans un volume réactionnel de 20 μL. Incuber à RT pendant 15 min.
- Transformer le mélange de la ligature de la souche de DH5 alpha et croissance transformants sur plaques de gélose LB contenant l’ampicilline (100 μg/mL) à 37 ° C. Confirmer l’insertion de l’oligo du vecteur par séquençage standard.
-
Transfection des plasmides codant pour Sniper-Cas9 et sgRNA
- Maintenir les cellules HEK293T DMEM supplémenté avec des antibiotiques de SVF et 1 % 10 % à 37 ° C, avec 5 % de CO2. La veille de la transfection, trypsinize et compter les cellules non vides. Lorsque vous travaillez à l’échelle 48 puits, plaque 1 x 105 cellules / puits dans 250 μL de milieu de culture complet. Les cellules doivent être anastomosé de 50 à 80 % le jour de la transfection.
- À l’échelle de 48 puits, préparer 250 ng de plasmide p3s-Cas9 et 250 ng de plasmide de sgRNA pour la transfection, utilisant un réactif à base de lipides transfection. Mélanger les plasmides dans 25 μL de sérum gratuit-MEM.
- Diluer 1 μL de réactif de transfection avec 25 μL de sérum gratuit-MEM. Incuber le mélange à ta pendant 5 min. combiner les deux mélanges et incuber la solution obtenue à ta pendant 20 min pour former des complexes plasmide-lipofectamine.
- Après 20 min d’incubation, ajouter 50 μL de la solution contenant les plasmide-transfection réactif complexes directement à chaque puits contenant des cellules et mélangent doucement en balançant la plaque avant et en arrière. Incuber les cellules à 37 ° C dans un incubateur à CO2 pour la transfection de poteau de 48 à 72 h avant l’analyse de l’expression du transgène.
8. calcul des fréquences indel pour déterminer les activités sur la cible et hors cible
-
Ciblées de séquençage en profondeur pour l’analyse des sites hors cible potentielles sur la cible
- Isoler l’ADN génomique de l’étape 6.1.5 ou 7.2.4 avec un kit de préparation Adng. Générer des bibliothèques de séquençage en profondeur par amplification par PCR de l’Adng avec des amorces ciblant sur la cible et hors cible.
- Utiliser les amorces indice d’étiqueter chaque échantillon. Sous réserve des bibliothèques regroupées au séquençage de bout couplé à l’aide d’une machine de nouvelle génération séquençage.
-
Analyse de séquençage en profondeur à l’aide de Cas-Analyzer
- Analyser les données de séquençage en profondeur à l’aide de l’analyseur de Cas-évaluation outil17.
- Choisir les fichiers de Fastq sous les onglets lecture 1 et 2 de lecture (lecture 1 = XX_SXX_L001_R1_001.fastq, lire 2 = XX_SXX_L001_R2_001.fastq).
- Compléter l’onglet Informations de base et l’onglet des Paramètres d’analyse , cliquez sur le bouton soumettre .
Résultats
Après que Sniper-écran est effectuée, le pourcentage des colonies de survie peut être calculé en divisant le nombre de colonies sur le plat de livre contenant du chloramphénicol, kanamycine, arabinose et ATC (CKAA) par le nombre de colonies dans la plaque LB contenant chloramphénicol et kanamycine seulement (CK). Ce pourcentage était généralement très faible lorsque le tireur d’élite à l’écran a été réalisée avec les bibliothèques de SpCas9. Hits de vrai positif peuvent être enrichis en répétant l’écran avec la piscine survivante. Dans cet écran Sniper représentant, par exemple, un taux de survie de 100 % a été obtenu après le troisième écran (Figure 1). Transfections utilisant RNP ou plasmidique codée Sniper-Cas9 peuvent être faites pour différentes cibles et la résultante sur la cible et activités hors cible mesurées par ciblées de séquençage de l’amplicon (Figure 2). Au plus objectifs, tireur d’élite-Cas9 montre le même niveau d’activités sur la cible et des ratios plus élevés de spécificité par rapport à la sgRNAs en poids tronqué permet également d’améliorer encore la spécificité (Figure 3). Toutefois, leur utilisation est limitée à seulement quelques cibles parce qu’elles résultent de faible activité sur la cible par rapport à sgRNAs pleine longueur, dans la plupart des cas. Par conséquent, sgRNAs avec différentes longueurs (de 17 à 20 mers) doivent être testées et activités sur la cible tant hors cible doivent être mesurées pour optimiser la spécificité.

Figure 1 : représentant Sniper-écrans effectué les différentes bibliothèques de variantes aléatoires de Cas9. Mutateur bibliothèque, EP I et bibliothèque EP II indiquer bibliothèques apportées à l’aide de différents kits commerciaux. Premier, deuxième et final indiquent le nombre de fois que l’écran de l’enrichissement était effectué7. Ce chiffre a été modifié par Lee et al.,7. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
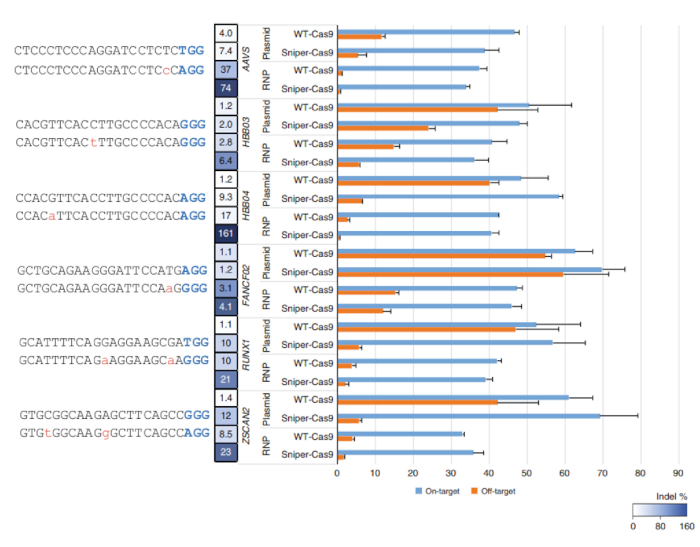
Figure 2 : sur la cible et hors cible activités de WT-Cas9 ou Sniper-Cas9 jumelé avec une séquence de guide 20-mer livrée par plasmide ou RNP. Rapports de spécificité ont été déterminés en divisant l’activité sur la cible de l’activité hors-cible. Barres d’erreur indiquent SEM (n = 3)7. Ce chiffre a été modifié par Lee et al.,7. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
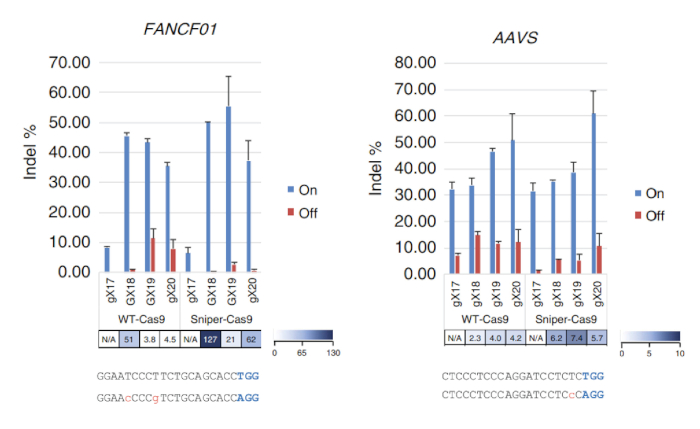
Figure 3 : sur la cible et les activités de Sniper-Cas9 par rapport au WT-Cas9 obtenues à l’aide de sgRNAs avec des longueurs variables ciblant les sites FANCF01 et AAVS hors cible. Rapports de spécificité ont été déterminés en divisant indel fréquences sur les sites sur la cible par ceux sur les sites respectifs de hors-cible. sgRNAs avec une guanine correspondante à l’extrémité 5' (GX18 ou GX19) et ceux avec une guanine dépareillée (gX17, gX18, gX19 ou gX20) sont indiqués. Rapports de spécificité n’ont été pas calculés lorsque les activités sur la cible normalisées étaient < 70 %7. Ce chiffre a été modifié par Lee et al.,7. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Discussion
Pour ceux qui veulent éviter de lourdes épreuves de présélection pour obtenir Sniper-Cas9, la protéine Sniper-Cas9 et le plasmide codant sont disponibles. À l’aide de ces matériaux, la longueur optimale de la sgRNA, qui fournit le plus haut ratio de spécificité, doit être déterminée. En outre, livraison de Sniper-Cas9 et sgRNA dans un format de RNP est recommandée car elle se traduit généralement par un ratio plus élevé de spécificité que la prestation sous forme de plasmide. Contrairement à Sniper-Cas9, les autres variantes de Cas9 machinés ne sont pas compatibles avec tronqué sgRNAs6,7 ou livraison au RNP formulaire8 (à l’exception de HiFi-Cas9).
Pour tireur d’élite à l’écran, la sélection de la séquence dépareillée est l’étape la plus importante. Sélection d’un sgRNA ne correspondent pas associé à une activité faible clivage envers la séquence GOI doit être évitée. Si ce n’est pas le cas, Cas9 variantes avec un niveau WT de spécificité ne seront pas cliver l’ADN génomique d’e. coli avec le site cible ne correspondent pas. Cet effet se traduira par un grand nombre de colonies de fond, mettre en péril toute procédure de dépistage.
Parce qu’e. coli a un jeûne doublement de temps et efficacité de transformation élevé par rapport à la levure, tireur d’élite-écran est avantageux par rapport aux méthodes de dépistage basé sur la levure. En outre, tireur d’élite-écran doit être plus sensible que les autres e. coli-issu des systèmes où le site ne correspondent pas a lieu un plasmide : il y a une copie de l’ADN génomique et ainsi qu’une copie du site ne correspondent pas à notre système, mais un grand nombre de plasmides dans une unique cellule d’e. coli .
Les spécificités des autres endonucléases ADN qui induisent la BDB, tels que SaCas9 ou Cpf1s, pourraient également être améliorées à l’aide de tireur d’élite à l’écran. Malheureusement, l’écran Sniper ne peut servir à augmenter la spécificité des éditeurs de base directement, parce que les éditeurs de base n’induisent pas de BDB dans l’ADN génomique d’e. coli. Comme base éditeurs utilisent la version morte Cas9 dans le coeur de leur système ou les nickase, les spécificités des éditeurs de base pourraient être augmentées en utilisant les succès obtenus à partir de tireur d’élite à l’écran.
Déclarations de divulgation
ToolGen a déposé une demande de brevet (PCT/KR2017/006212) couvrant l’écran Sniper (statut : en attente, inventeur : Jungjoon K. Lee). Jungjoon K. Lee, Joonsun Lee, Minhee Jung et Euihwan Jeong sont des employés de ToolGen, Inc.
Remerciements
Cette recherche a été financée par des subventions du ministère de la Science et TIC de Corée (2017M3A9B4061406) et la National Research Foundation de Corée (NRF) financé par le gouvernement coréen (MSIT) (subvention numéros 2017M3A9B4061404 et 2018M3A9H3020844) pour Jungjoon K. Lee. Le plasmide codant pGRG36 était un cadeau de Nancy Craig (Addgene plasmide #16666) et p11-LacY-wtx1 était un cadeau de Huimin Zhao.
matériels
| Name | Company | Catalog Number | Comments |
| Alkaline Phosphatase, Calf Intestinal (CIP) | NEB | M0290L | |
| Ampicillin Sodium Salt | Fisher Chemical | BP1760-25 | |
| Anhydrotetracycline hydrochloride | Sigma Aldrich | 37919 or 94664 | |
| Antibiotic Antimycotic solution | Welgene | LS203-01 | Media component |
| BamHI | Enzynomics | R003L | |
| Chloramphenicol | Sigma Aldrich | R4408 | |
| Diversify PCR Random Mutagenesis | Clontech | 630703 | Error-prone PCR kit |
| DNA-Shuffling Kit | Jena Bioscience | PP-103 | |
| Dulbecco's Modified Eagle's Medium (DMEM) | Welgene | LM001-17 | Culture media |
| Endura Electrocompetent Cells (DUO) | Lucigen | 60242-2 | E. coli electrocompetent cells for library preparation |
| Fetal Bovine Serum (FBS) | Welgene | S101-01 | Media component |
| Gene Pulser II | Bio-Rad | E. coli electroporation equipment | |
| GeneMorph II Random Mutagenesis Kit | Agilent | 200550 | Error-prone PCR kit |
| genomic DNA prep kit | GenAll | 106-152 | gDNA-prep kit |
| Glycerol | BioShop | GLY001.1 | |
| GP/MP Cuvette, 0.1cm | Bio-Rad | BR165-2089 | E. coli electroporation equipment |
| HEK293T cell | ATCC | CRL-11268 | Human cell line |
| Kanamycin sulfate | Acros Organics | 450810500 | |
| L-(+)-Arabinose | Sigma Aldrich | A3256-25G | |
| LaboPass PCR Purification Kit | Cosmogenetech | CMR0112 | |
| LaboPass Plasmid Mini | Cosmogenetech | CMP0112 | Mini-prep kit |
| LB Agar Miller | Formedium | LMM0202 | |
| LB Broth Miller | Formedium | LMM0102 | |
| Lipofectamine | Invitrogen | 11668019 | Transfection reagent |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | Gibson assembly (isothermal in vitro recombination) mixture |
| Neon Transfection System | Thermo | MPK5000 | RNP Electroporation equipment |
| Neon Transfection System 10 µL Kit | Thermo | MPK1096 | RNP Electroporation equipment |
| NucleoBond Xtra Midi EF | Macherey-Nagel | 740420.50 | Midi-prep kit |
| Oligo Clean & Concentrator | Zymo Research | D4061 | DNA purification kit that enables low-volume elution |
| Opti-MEM | Gibco | LS31985070 | Transfection media |
| p11-lacY-wtx1 | Addgene | #123945 | |
| pgrg36 | Addgene | #16666 | E. coli mutator strain |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530L | |
| Pyrophophatase | NEB | M2403L | |
| Ribonucleotide Solution Set | NEB | N0450L | |
| RNase inhibitor | NEB | M0314L | |
| T7 RNA polymerase | NEB | M0251L | |
| Turbo Dnase | Ambion | AM2238 | |
| XbaI | Enzynomics | R013L | |
| XL1-Red Competent Cells | Agilent | 200129 |
Références
- Fu, Y., Sander, J. D., Reyon, D., Cascio, V. M., Joung, J. K. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nature Biotechnology. 32 (3), 279-284 (2014).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Research. 24 (6), 1012-1019 (2014).
- Chen, J. S., et al. Enhanced proofreading governs CRISPR-Cas9 targeting accuracy. Nature. 550 (7676), 407-410 (2017).
- Kleinstiver, B. P., et al. High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects. Nature. 529 (7587), 490-495 (2016).
- Slaymaker, I. M., et al. Rationally engineered Cas9 nucleases with improved specificity. Science. 351 (6268), 84-88 (2016).
- Casini, A., et al. A highly specific SpCas9 variant is identified by in vivo screening in yeast. Nature Biotechnology. 36 (3), 265-271 (2018).
- Lee, J. K., et al. Directed evolution of CRISPR-Cas9 to increase its specificity. Nature Communications. 9 (1), 3048(2018).
- Vakulskas, C. A., et al. A high-fidelity Cas9 mutant delivered as a ribonucleoprotein complex enables efficient gene editing in human hematopoietic stem and progenitor cells. Nature Medicine. 24 (8), 1216-1224 (2018).
- Anderson, K. R., et al. CRISPR off-target analysis in genetically engineered rats and mice. Nature Methods. 15, 512-514 (2018).
- Kim, S., Bae, T., Hwang, J., Kim, J. S. Rescue of high-specificity Cas9 variants using sgRNAs with matched 5' nucleotides. Genome Biology. 18 (1), 218(2017).
- Kulcsar, P. I., et al. Crossing enhanced and high fidelity SpCas9 nucleases to optimize specificity and cleavage. Genome Biology. 18 (1), 190(2017).
- Zhang, D., et al. Perfectly matched 20-nucleotide guide RNA sequences enable robust genome editing using high-fidelity SpCas9 nucleases. Genome Biology. 18 (1), 191(2017).
- McKenzie, G. J., Craig, N. L. Fast, easy and efficient: site-specific insertion of transgenes into enterobacterial chromosomes using Tn7 without need for selection of the insertion event. BMC Microbiology. 6, 39(2006).
- Geissmann, Q. OpenCFU, a new free and open-source software to count cell colonies and other circular objects. PLoS One. 8 (2), e54072(2013).
- Chen, Z., Zhao, H. A highly sensitive selection method for directed evolution of homing endonucleases. Nucleic Acids Research. 33 (18), e154(2005).
- Kim, S., et al. CRISPR RNAs trigger innate immune responses in human cells. Genome Research. 28 (3), 367-373 (2018).
- Park, J., et al. Cas-Analyzer: an online tool for assessing genome editing results using NGS data. Bioinformatics. 33, 286-288 (2017).
- Studier, F. W., et al. Use of T7 RNA Polymerase to Direct Expression of Cloned Genes. Methods in Enzymology. 185, 60-89 (1990).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon