Method Article
Verwendung von Sniper-Cas9, Off-Target Auswirkungen von CRISPR-Cas9 ohne den Verlust der Zielaktivität durch gerichtete Evolution
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier präsentieren wir Ihnen ein Protokoll zur Optimierung der CRISPR-Cas9 um eine höhere Spezifität verlustfrei auf-Aktivität zu erreichen. Wir verfolgen einen gerichteten Evolution genannt Sniper-Bildschirm, um einen Mutant Cas9 mit den gewünschten Eigenschaften zu finden. Sniper-Cas9 ist kompatibel mit gekürzten Single-Guide RNAs und Lieferung in einem Ribonucleoprotein Format, bekannte Strategien zur Erreichung höherer Besonderheiten.
Zusammenfassung
Die Entwicklung der gruppierten regelmäßig dazwischen kurze palindromische Wiederholungen (CRISPR)-assoziierten Protein 9 (Cas9) in therapeutischen Modalitäten erfordert die Vermeidung ihrer potenziell schädlichen Wirkungen Ziel. Mehrere Methoden sind entwickelt worden, um solche Effekte zu reduzieren. Hier präsentieren wir eine Escherichia coli-basierte gerichtete Evolution Methode namens Sniper-Bildschirm, um eine Cas9 Variante mit optimierten Spezifität zu erhalten und zielgerichtet Aktivität, genannt Sniper-Cas9 beibehalten. Sniper-Bildschirm verwenden, kann positive und negative Auswahl gleichzeitig durchgeführt werden. Der Bildschirm kann auch mit anderen Single-Guide-(SgRNA) RNA-Sequenzen für die wahre positive Treffer zu bereichern wiederholt werden. Durch die Verwendung der CMV-PltetO1 dual Promoter Cas9 Varianten zum Ausdruck zu bringen, kann die Leistung der gepoolten Bibliothek schnell in Säugetierzellen überprüft werden. Methoden zur Steigerung der Spezifität der Sniper-Cas9 werden ebenfalls beschrieben. Zuerst nachweislich die Verwendung des abgeschnittenen SgRNAs zuvor Cas9 Spezifität zu erhöhen. Im Gegensatz zu anderen technischen Cas9s bewahrt Sniper-Cas9 ein Wildtyp (WT) auf Ziel-Aktivität in Kombination mit abgeschnittenen SgRNAs. Zweitens ist die Lieferung von Sniper-Cas9 in einem Ribonucleoprotein (RNP) Format anstelle von einem Plasmid-Format ohne Auswirkungen auf die Zielaktivität möglich.
Einleitung
In diesem Beitrag wollen wir die Besonderheit des Cas9 durch Kombination verschiedener Strategien zu verbessern. Verschiedene Methoden zur Vermeidung der Ziel-Auswirkungen der CRISPR-Cas9 entstanden sind. Z. B. kann abgeschnittene SgRNAs verwendet werden, um höhere Spezifität1zu erreichen. Darüber hinaus kann die Art der Cas9 Lieferung aus einem Plasmid-Format RNP Format zu höheren Spezifität2geändert werden. Spezifischen Aminosäureresten des Streptococcus Pyogenes Cas9 (SpCas9) Protein nach dem rationalen geändert wurden zuvor beschriebenen Design3,4,5. Alternativ Aminosäurereste in zufälliger Weise verändert wurden und die Cas9-Varianten mit der höchsten Spezifität identifiziert mit einem Hefe-6 oder eine E. Coli7,8 screening-System.
Jedoch haben viele Gruppen berichtet, dass Cas9 Varianten entwickelt, mit dem Design um zu schwächen die unspezifische Interaktion zwischen Cas9 und dem Substrat Ausstellung niedrigen zielgerichtet Aktivitäten7,8,9, 10 , 11 , 12. wir entwickelt ein E. Coli-basiertes gerichtete Evolution System, Sniper-Bildschirm, nach dem Zufallsprinzip mutagenisierter Cas9 Varianten auf den Bildschirm. Ein E. Coli screening-System hat Vorteile gegenüber einem Hefe-System wegen der schneller Verdoppelung Zeit und höhere Transformation Wirkungsgrade von E. Coli.
Sowohl positive als auch negative Auswahl, basierend auf drei verschiedenen Plasmiden und ein Gen des Interesses (GOI) in das E. Coli -Genom integriert werden in Sniper-Bildschirm verwendet. Cas9 Varianten sind unter dem CMV-PltetO1 Dual-Promotor System von einem niedrig-Kopie Nummer Plasmid ausgedrückt, so dass Kandidaten identifiziert in E. Coli in Säugetierzellen ohne subcloning getestet werden können. Die indische Regierung wird in der E. Coli -Genom mit dem Tn7 Transposon-System eingeführt. Das Plasmid SgRNA, das enthält einen temperaturempfindliche Ursprung der Replikation, drückt eine SgRNA Ausrichtung der GOI; Allerdings sind die SgRNA und GOI Sequenzen nicht perfekt aufeinander abgestimmt. Ein perfekt aufeinander abgestimmtes SgRNA Ziel-Site existiert auf einem dritten Plasmid enthält das CcdB -gen, das welches eine tödliche Produkt kodiert, die Gyrase vergiftet. In diesem System werden Zellen mit dem Ausdruck Cas9 Varianten mit Aktivitäten mit hohem Ziel entfernt, weil Doppel-Strangbrüchen (DSB) in die nicht übereinstimmende Standort in der genomischen DNA eingeschleust werden. Auf der anderen Seite werden Zellen mit dem Ausdruck Cas9 Varianten mit niedrigen zielgerichtet Aktivitäten auch wegen tödlichen CcdB Genexpression entfernt. Die Expression der Cas9 Varianten kann geändert werden, durch Veränderung der Konzentration von Anhydrotetracycline (ATC), welches die Auswahl Kraft einstellt.
Wir begründete, dass Ortung der Zielwebsite nicht übereinstimmende SgRNA in der genomischen DNA statt auf einem Plasmid die Empfindlichkeit des Systems erhöhen würde. Der Vorteil dieses Ansatzes ist, dass es nur eine genomische Site, während gäbe es viele Plasmide, jeweils eine Zielsite innerhalb einer einzigen E. Coli Zelle.
Mit diesem System haben wir eine Cas9 Variante, Sniper-Cas9, zeigt WT-Ebene zielgerichtet Aktivitäten und reduzierte Ziel-Aktivitäten im Vergleich zu WT Cas9 identifiziert. Sniper-Cas9 kann sogar höhere Spezifität Verhältnisse mit abgeschnittenen SgRNAs oder RNP-basierte Lieferung als Plasmid-basierte Lieferung erreichen.
Protokoll
(1) Integration von einem menschlichen GOI BW25141 E. Coli -Stamm
-
Klonen von GOI
- Polymerase-Kettenreaktion (PCR) verstärken 500 bp Länge eines menschlichen GOI mit verschiedenen Kandidaten-Ziel-Sites über standard-PCR-Methoden mit Primer mit NotI und XhoI Restriktionsenzym Standorten.
Hinweis: In diesem Experiment war die indische Regierung das menschliche EMX1 gen. - Das PCR-Produkt und der pgrg3613 Vektor mit Restriktionsenzymen NotI und XhoI zu verdauen.
- Gel reinigen die gewünschte Fragmente (500 bp und 12 kb).
- Verbinden Sie die Fragmente zusammen mit T4-Ligase. Dazu mischen Sie 50 ng der verdauten pgrg36 und 6 ng PCR-Einsatzes in einem Reaktionsvolumen von 20 μl, 1 X Ligase Puffer und 0,5 U-Ligase-Enzym enthält. Inkubation bei Raumtemperatur (RT) über Nacht.
- Umwandeln Sie die aufgespaltenen Plasmid in DH5-α zuständigen Zellen. Die daraus resultierende Transformanten auf Luria Brühe (LB) Agarplatten mit Ampicillin (100 μg/mL) bei 32 ° C zu wachsen und über Nacht inkubieren. Nehmen Sie eine Kolonie und in LB-Medium mit Ampicillin über Nacht wachsen. Das Plasmid-DNA aus den E. Coli, mit einem im Handel erhältlichen Miniprep Kit zu isolieren. Bestätigen Sie das Einfügen des Fragments GOI von Sanger-Sequenzierung das Plasmid Mini-Plasmid Vorbereitung (Mini-Prep)13entnommen.
- Polymerase-Kettenreaktion (PCR) verstärken 500 bp Länge eines menschlichen GOI mit verschiedenen Kandidaten-Ziel-Sites über standard-PCR-Methoden mit Primer mit NotI und XhoI Restriktionsenzym Standorten.
-
Vorbereitung der BW25141 -GOI
- Verwandeln Sie das erhaltenen pgrg36 -GOI -Plasmid in BW25141 E. Coli -Stamm. Es ist wichtig, um eine BW25141 Sorte verwenden, um die Anzahl der falsch positiven Kolonien zu minimieren.
- Wachsen Sie die transformierten Zellen in der LB-Puffer bei 32 ° C über Nacht. GOI ist in der genomischen DNA des BW25141-Stammes (BW25141 -GOI) eingefügt. Entfernen Sie das pgrg36 -GOI -Plasmid aus dem BW25141 -GOI -Stamm mit den standard pgrg36 Protokoll Nr.13. Kurz gesagt, eine Kolonie zu verdünnen (ca. 107-Falten) und über Nacht auf eine LB-Platte bei 42 ° C wachsen. Streifen der Kolonien auf der LB-Platte und wachsen sie über Nacht bei 42 ° C.
- Bestätigen Sie das korrekte GOI Einfügen von Colony PCR unter Verwendung der Zündkapseln in das pgrg36-Protokoll vorgeschlagen: 5'-GATGCTGGTGGCGAAGCTGT-3 'und 5'-GATGACGGTTTGTCACATGGA-3'. Der Primer verstärkt die genomische DNA-Einstichstelle, und die Größe der entstehenden PCR-Produkts werden 904 bp plus die Größe des Einsatzes (500 bp in diesem Fall).
- Bereiten Sie Electrocompetent BW25141 -GOI Zellen (ein detailliertes Protokoll wird in 3.2.6–3.2.10 Schritten beschrieben).
2. Vorbereitung der Variante Cas9-Bibliothek
-
Bibliothek-Vorbereitung
- Cas9 Vektor7 zu verwandeln in eine kommerzielle e.coli Mutator strain (Table of Materials) und folgen Sie den Anweisungen des Herstellers, eine Variante Bibliothek (die Mutator) zu erhalten.
- Durchführen Sie fehleranfällige PCR auf die gesamte Sequenz der WT-Cas9 im Cas9 Vektor, mit eine fehleranfällige PCR Kit (Table of Materials).
Hinweis: Die niedrige Fehlerquote-Protokoll wurde im Sniper-Cas9 Fall nicht zu stören die ursprüngliche Funktion des Proteins angenommen. - Den Cas9-Vektor mit geeigneten Restriktionsenzymen zu verdauen. Gel reinigen das PCR-Produkt (aus Schritt 2.1.2) und das verdaute Rückgrat.
Hinweis: Die Größe des SpCas9-Gens ist etwa 4,3 kb. XhoI und KpnI wurden ausgewählt, um pBLC SpCas9 Vektor zu verdauen, die in der Sniper-Cas9 Fall verwendet wurde. - Montieren Sie das Rückgrat-Fragment (aus Schritt 2.1.3) und die Einlage verstärkt mittels fehleranfällige PCR (aus Schritt 2.1.3) über isothermen in-vitro- Rekombination.
Hinweis: mehr als 500 ng des Rückgrats ist erforderlich, um eine hohe Konzentration von fehleranfällige PCR [EP] (Bibliothek). Zwei verschiedene fehleranfällige PCR-Kits wurden verwendet, um EP Bibliotheken im Sniper-Cas9 Fall vorbereiten (EP-Bibliothek I und II). - Reinigen Sie die Produkte aus der Assembly (aus Schritt 2.1.4) mithilfe eines DNA-Reinigung-Kits, die leise Elution (Table of Materials) ermöglicht. Eluieren mit 6 μL der Nuklease-freies Wasser (NFW) und messen die Konzentration von DNA.
- Transformieren von mehr als 500 ng der Cas9 Bibliothek Vektor (für jede der drei Bibliotheken) in 50 μL Electrocompetent E. Coli Zellen (Table of Materials). Siehe die Elektroporation Protocol in Schritten 3.2.1-3.2.4. Verwenden Sie für diese Bibliothek Vorbereitung 1 mL der SOC-Medium anstelle von 250 μl pro 50 μl kompetente Zellen.
- Machen 1: 100, 1:1 000 und 1: 10.000 Verdünnungen der Mischung mit der wiederhergestellten Zellen mit SOC-Medium. Platte die verdünnten Zellen auf 100 mm LB-Agar-Platten mit Chloramphenicol (12,5 μg/mL) ergänzt. Die verbleibenden Zellen auf einem 245 mm2 Teller Platte. Über Nacht bei 37 ° C inkubieren.
-
Berechnung der Bibliothek Komplexität
- Die Verdünnung Platten über eine Gel-Dokumentations-System oder eine gewöhnliche Digitalkamera zu fotografieren. Führen Sie OpenCFU Software14 und laden Sie die Fotos der Verdünnung Platten. Setzen Sie die Zählung im Inneren einen Teller und entfernen Sie falsche Kolonien.
- Multiplizieren Sie die Anzahl der Kolonien manuell mit der Verdünnungsfaktor die ursprüngliche Anzahl der Transformanten zu erhalten. Wandeln Sie diese Zahlen in logarithmischer Form (Basis 10). Berechnen Sie den Mittelwert um die Komplexität der Bibliothek zu bestimmen.
- Wenn die gewünschte Multiplikator gewonnen wird, entnehme die Kolonien auf den 245 mm quadratischen Platte (Schritt 2.1.7) mit einem Streuer und 20 mL LB mit Chloramphenicol ergänzt. Nicht die gesammelten Kolonien wachsen und Reinigen der Plasmid-Bibliothek mit einem kommerziellen Midiprep-Kit.
Hinweis: Je höher die Komplexität der Bibliothek, desto besser. Als Sniper-Cas9 identifiziert wurde, wurde eine Vielfalt von 3 x 106 für jede Bibliothek erreicht.
(3) positive und negative Screening für neue Cas9
-
Zielauswahl und Plasmid-Bau
- Wählen Sie eine Zielsequenz SgRNA Abstandhalter im GOI. Ersetzen Sie ein oder zwei Rückstände in der zufälligen Nukleotid, eine nicht übereinstimmende Sequenz zu produzieren.
Hinweis: Menschliche EMX1 Zielsite 3 (GAGTCCGAGCAGAAGAAGAA mit GGG PAM) wurde in der Sniper-Cas9 Fall verwendet. Sind die nicht übereinstimmenden Sequenzen verwendet: GAGTCCGAGCAGAAagAGAA, GAacCCGAGCAGAAGAAGAA, GAGTCCGAGCAGAgGAAGAA und GAGcCCGAGCAGAAGAAGAA. - Die nicht übereinstimmende Sequenz einfügen (siehe Punkt 3.1.1) in der SgRNA-Plasmid mit standard Oligonukleotid (Oligo) Klonen Verfahren7.
- P11-Spitzen-wtx1 (Tabelle der Materialien), die CcdB Plasmid mit standard Oligo Klonen Verfahren15zu konstruieren stecken Sie die nicht übereinstimmende Sequenz mit einem PAM am 3'-Ende.
- Wählen Sie eine Zielsequenz SgRNA Abstandhalter im GOI. Ersetzen Sie ein oder zwei Rückstände in der zufälligen Nukleotid, eine nicht übereinstimmende Sequenz zu produzieren.
-
Vorbereitung der Sniper-Screening E. Coli kompetente Zellen
- BW25141 -GOI Electrocompetent Zellen auf Eis auftauen.
- Addiere 1 ng jeder CcdB Plasmid und SgRNA Plasmid mit doppelten Fehlanpassungen in 50 μL der aufgetauten BW25141 -GOI -Zellen. Vorsichtig mischen Sie die Zellen durch pipettieren und verschieben Sie sie in eine prechilled 0,1 cm Elektroporation Küvette.
- E. Coli mit zwei Plasmide über Elektroporation zu verwandeln. Fügen Sie 250 μl SOC-Medium sofort nach Elektroporation. Pipette vorsichtig die Lösung um die Zellen und das Medium zu mischen. Übertragen Sie die Mischung zu einem 1,5 mL Microcentrifuge Schlauch.
Hinweis: Für maximale Effizienz, stellen Sie die Spannung bei 1,80 kV und die Laufzeit sollte zwischen 4,8 ms und 5,0 ms. - Erholen Sie die transformierten Zellen und inkubieren sie bei 32 ° C für 1 h mit sanft schütteln.
- Platte 125 μL der wiederhergestellten Zellen auf einem Ampicillin (50 μg/mL) / Kanamycin (25 μg/mL) LB-Agar-Platte (Kultur-Bedingung). Platte der verbleibenden Zellen auf einem Ampicillin/Kanamycin/Arabinose (1,5 mg/mL) LB-Agar-Platte (CcdB-Zustand auszudrücken). Über Nacht bei 32 ° C inkubieren.
- Überprüfen Sie, ob das Fehlen von Überlebenden Kolonien auf den CcdB-Zustand Platte zum Ausdruck zu bringen. Sammeln Sie Kolonien aus der Kultur-Zustand-Platte mit einem Streuer und Kultur, die sie in 250 mL super optimale Brühe (SOB) Medium ergänzt mit 50 μg/mL von Ampicillin und 25 μg/mL Kanamycin bei 32 ° C, mit schütteln sanft.
- Wenn die Extinktion bei 600 nm (OD600) erreicht 0.4, chill den Kolben auf Eis. Bereiten prechilled deionisiertes Wasser und eine prechilled 10 % Glycerin-Lösung (vor Gebrauch sterilisieren).
- Zentrifugieren Sie (bei 4.000 X g für 5 min bei 4 ° C) der Zellen und verwerfen Sie den überstand. 200 mL prechilled deionisiertes Wasser hinzugeben. Aufzuwirbeln Sie die Zellen mit einer serologischen 10 mL-Pipette. Wiederholen Sie diesen Schritt 3 X.
- Waschen Sie die Zellen mit 50 mL prechilled 10 % Glycerin-Lösung. Zentrifugieren sie wie zuvor (bei 4.000 X g für 5 min bei 4 ° C).
- Den Überstand verwerfen und erneut das Pellet in 300 μl 10 % Glycerin-Lösung. 50 μL Aliquote machen und Einfrieren in flüssigem Stickstoff. Speichern der Zellen (Sniper-Screening) bei-80 ° C.
-
Scharfschütze-screening
- Verwandeln die Sniper-Screening-Zellen (aus Schritt 3.2.10) mit 100 ng des Cas9 variant Plasmide aus jeder Bibliothek (ab Schritt 2.2.3.See Schritte 2.1.1 und 2.1.4). Die Elektroporation Schritte, beschriebenen Schritte 3.2.1–3.2.3.
- Übertragen Sie 250 μL der Zellen zu einem frischen 1,5 mL Microcentrifuge Schlauch. Fügen Sie 250 Pg von ATC, eine Endkonzentration von 10 ng/mL zu machen. ATC-haltigen sowohl ATC-freie Zellen (siehe Schritt 3.2.4 für die Wiederherstellungsschritt) zu erholen.
- Platte 25 μL der wiederhergestellten ATC-freie Zellen auf einer Chloramphenicol/Kanamycin LB-Agar-Platte (obstructions Zustand). ATC hinzu kommt die wiederhergestellten ATC-haltigen Zellen, eine Endkonzentration von 100 ng/mL auf eine 245 mm LB-Platte zu machen. Sofort Platte der Zellen auf eine Chloramphenicol/Kanamycin/Arabinose LB Nährbodenplatte (selektive Zustand). Inkubation über Nacht bei 32 ° C.
Hinweis: Die Größe und die Anzahl der LB-Platte werden durch die Größe der Vielfalt der Screening-Abdeckungen bestimmt. Fügen Sie bei einem 100 mm Petrischale mit 20 mL LB 2 μg des ATC. - Fotografieren Sie die Platten. Die Anzahl der lebensfähigen Kolonien mit OpenCFU Software14. (Siehe Schritt 2.2.1) Stellen Sie sicher, dass die Anzahl der Kolonien auf der obstructions Platte mindestens 10 x größer als die Vielfalt der Bibliothek um alle Varianten zu decken ist.
- Berechnen Sie die Überlebens-Frequenz wie folgt.
Überleben-Frequenz = Anzahl der Kolonien auf einem selektiven Teller / (die Anzahl der Kolonien auf einem obstructions Teller X 10) - Der Pool der Kolonien, die auf den selektiven Platten aus allen drei Bibliotheken überlebt. Inkubieren Sie die Überlebenden Kolonien in 250 mL LB-Medium ergänzt mit 12,5 μg/mL Chloramphenicol bei 42 ° C über Nacht. Die abgeschirmte Cas9 Bibliothek DNA mit einem Midiprep-Kit zu isolieren.
Hinweis: Dieser Schritt löscht das Plasmid SgRNA. - Wiederholen Sie das Screening-Verfahren von Schritte 3.3.1–3.3.6 bis die Überlebens-Frequenz ein Plateau erreicht. Einsatz 10 ng des ausgewählten Cas9 Plasmids für Transformation und 10 ng/mL von ATC während der Wiederherstellung. Die ATC-Konzentration bei 100 ng/mL für den selektiven Zustand zu erhalten.
-
Mischen und das zweite screening
- Mischen Sie die ausgewählten gepoolten Varianten unter Verwendung des folgenden DNA Schlurfen Protokolls. PCR verstärken den Cas9 Einsatz in der Cas9-Plasmid mit flankierenden Primern, 150 Nukleotide von den Grenzen einfügen. Digest 2 μg amplifizierten PCR-Produktes mit DNase I für 1 min bei 37 ° C.
- Reinigen Sie Fragmente 70 – 200 bp lang mit 2 % Agarose-Gelelektrophorese. PCR verstärken die gereinigten Fragmenten. Verwendung das Produkt als Vorlage, um PCR verstärken die Cas9 Einlage mit geeigneten Primern flankierende Cas9. Verwenden Sie das Endprodukt PCR, um eine Cas9-Bibliothek zu bauen, wie in Schritt 2.1.4 beschrieben.
- Neue Sniper-Screening Zellen (siehe Abschnitt 3.2) mit einem anderen nicht übereinstimmende SgRNA Plasmid vorzubereiten (siehe Punkt 3.1.1). Wiederholen Sie das Screening-Verfahren (Abschnitte 3.2, 3.3), bis die überleben Rate erreicht eine Hochebene. Einsatz 10 ng des ausgewählten Cas9 Plasmids für Transformation und 10 ng/mL ATC während der Wiederherstellung. Die ATC-Konzentration bei 10 ng/mL für den selektiven Zustand zu erhalten.
-
Auswahl des weiterentwickelten Cas9 mutierte Plasmide
- Nach dem letzten Screening Schritt nach dem Zufallsprinzip wählen Sie 100 Kolonien von der selektiven Platte und Kultur sie in Chloramphenicol-haltigen LB-Medium bei 42 ° C über Nacht.
- Isolieren Sie die Plasmide mit ein Miniprep-Verfahren und Sanger-Sequenz mit Sequenzierung Primer innerhalb Cas9 Einsätzen.
- Wählen Sie die oberen drei häufigsten Varianten in humanen Zelllinien zu testen.
4. Lieferung von Cas9 als ein RNP mit abgeschnittenen sgRNA
-
Gen der Zielauswahl mit Cas-OFFinder
- Wählen Sie Ziel-Sites mit Cas-OFFinder (http://www.rgenome.net/cas-offinder/). Wählen Sie den PAM-Typ für den bestimmten Typ des Cas9 und des Ziel-Genoms (Mensch, Maus, Zebrafisch, etc..). Füllen Sie die Registerkarte " Abfrage-Sequenzen ", wählen Sie die Mismatch-Anzahl, und klicken Sie auf die Schaltfläche " senden ".
- Nach ein paar Sekunden, die auf Ziel (mit einer Fehlanpassung Anzahl von '0') und Ziel-Websites wird angezeigt. In der Regel wählen Sie die Ziel-Websites mit ein bis drei Fehlanpassungen.
-
Vorbereitung der SgRNA Vorlage
- Bestellen Sie CrRNA und TracrRNA Oligos mit der folgenden Vorlage Sequenzen, nämlich CrRNA Sequenz: 5'-TAATACGACTCACTATAGGNNNNNNNNNNNNNNNNNNNNGTTTTAGAGCTAGAA-3 "; TracrRNA-Sequenz: 5'-AGCACCGACTCGGTGCCACTTTTTCAAGTTGATAACGGACTAGCCTTATTTTAACTTGCTATTTCTAGCTCTAAAAC-3 ". Füllen Sie die CrRNA-Sequenz mit der Zielsequenz in Schritt 4.1.2 erhaltenen N20. Abgeschnittene SgRNAs entwerfen, entfernen Sie Basen aus 5'-Ende, N19, N18 oder N17 SgRNA Sequenzen zu erhalten.
- Bereiten Sie die PCR Mischung für die Verstärkung der SgRNA-Codierung Sequenz wie folgt: 10 μl 5 x Puffer, 0,5 μL CrRNA Oligo (100 Pmol/μL), 0,5 μL TracrRNA Oligo (100 Pmol/μL), 2,5 μL von dNTPs (10 mM), 0,5 μL der DNA-Polymerase zu kombinieren , und 36 μL NFW (Tabelle der Materialien).
- Verstärken Sie die Vorlage mit den folgenden Bedingungen, nämlich für die erste Denaturierung: 1 min bei 98 ° C; für die Denaturierung, Glühen und Erweiterung: 10 s bei 98 ° C, 15 s bei 54 ° C, 20 s bei 72 ° C für 25 Zyklen; für die letzte Erweiterung: 5 min bei 72 ° C.
- Analysieren Sie 2 μL der verstärkten Vorlage DNA (aus Schritt 4.2.3) auf ein 2 % Agarose-Gel. Reinigen Sie die Vorlage mit einem PCR-Reinigung-Kit.
Hinweis: Die Größe der Schablone DNA ist 125 bp.
-
Synthese von sgRNA
- Wie folgt SgRNA Synthese das Reaktionsgemisch vorbereiten: kombinieren Sie 8,5 μL der Schablone DNA (aus Schritt 4.2.3), 1 μL der UTP (25 mM), 1 μL der CTP-Version (25 mM), 1 μL GTP (25 mM), 1 μL des ATP (25 mM), 4.2 μL von MgCl2 (100 mM) , 4,5 μL der T7 RNA-Polymerase (50 U/μl), 3 μL der 10 x T7-RNA-Polymerase-Puffer, 1,2 μL der Pyrophosphatase (0,5 U/μl), 0,75 μl RNase-Inhibitor (40 U/μl) und 4.2 μL NFW.
- Über Nacht die Reaktionsmischung bei 37 ° C inkubieren (mindestens 10 h). Fügen Sie 0,5 μL der DNase (2 U/μl) zum Reaktionsgemisch und Inkubation bei 37 ° C für 15-30 min. Reinigen der SgRNA mit einer RNA-Reinigung-Kit.
- Verringerung die Toxizität verursacht durch eine angeborene Immunantwort ausgelöst durch die 5'-Triphosphat am SgRNA16, entfernen Sie die 5'-Triphosphat aus dem Guide RNAs mit Kalb Darm alkalische Phosphatase (CIP) wie folgt: 10 µg von in Vitro- behandeln transkribierten RNA mit 250 U CIP 3 h bei 37 ° C in Anwesenheit von 100 U RNase-Inhibitor. Reinigen Sie die CIP-behandelte SgRNA mit einer RNA-Reinigung-Kit.
5. WT und Sniper-Cas9-Protein-Expression und Reinigung
-
Protein-Expression in E. coli
- Verwandeln pET Plasmide18 codieren sein-tagged WT - und Sniper-Cas9 in der BL21 (DE3) E. Coli -Stamm.
- 50 mL LB-Medium mit 50 μg/mL Kanamycin mit einer frischen Kolonie beherbergen die pET-Cas9-Expressionsplasmid und schütteln Sie es über Nacht (200 u/min) bei 37 ° C (Vorkultur) zu impfen.
- Übertragen Sie 10 mL über Nacht Kultur auf 500 mL frisches LB-Medium mit 50 μg/mL Kanamycin. Inkubieren Sie die Kultur mit schütteln (200 u/min) bei 37 ° C für 2 h.
- Überwachen Sie die OD600 , bis die Kultur die Mid Log-Phase des Wachstums (OD600 ≈ 0,6 – 0,7) erreicht.
- Induzieren die Expression des Proteins WT oder Sniper-Cas9 mit Isopropyl-β-D-1-Thiogalactopyranoside (IPTG in einer Endkonzentration von 0,25 nM). Über Nacht inkubieren Sie die Kultur bei 18 ° C.
-
Proteinreinigung
- Ernte der Zellen durch Zentrifugation bei 5.000 X g für 10 min bei 4 ° C.
- Das Pellet in der Lyse-Puffer (50 mM NaH2PO4, 300 mM NaCl, 10 mM Imidazol, 4 mM Dithiothreitol [DVB], 5 mM Benzamidine, 100 mM Phenylmethylsulfonyl Fluorid [PMSF], pH 8) bei 20 mL pro Gramm Frischgewicht aufzuwirbeln.
- Eine Endkonzentration von 1 mg/mL PMSF, DVB-t und Lysozym hinzu und inkubieren Sie die Mischung für 30 min auf Eis.
- Beschallen Sie die Zellen auf dem Eis. Puls immer wieder für 10 s bei 200 – 300 W mit 10 s, die Zeit zwischen jedem Impuls für eine Gesamtzeit von 20 Minuten kühlen.
- Zentrifugieren der lysate bei 6.000 X g für 30 min bei 4 ° C.
- Entfernen Sie den Überstand zu einem frischen Schlauch. Fügen Sie 1 mL der His-Bind Agarose Harz 5 mL lysate gelöscht und schütteln Sie sanft für 1 h bei 4 ° C.
- Laden der lysate/His-Bind Agarose Harzmischung auf eine Spalte mit einem capped Grundablass.
- Entfernen Sie die untere Kappe und sammeln Sie die Spalte durchströmten.
- Waschen Sie die Spalte 2 X mit Waschpuffer (50 mM NaH2PO4, 300 mM NaCl, 20 mM Imidazol, pH 8); sammeln Sie waschen Bruchteil für die Analyse von Sodium Dodecyl Sulfat-Polyacrylamid-Gelelektrophorese (SDS-PAGE).
- Eluieren Protein 10 X mit 1 mL der Elution buffer (50 mM NaH2PO4, 300 mM NaCl, 250 mM Imidazol, pH 8), sammeln von Proben für die SDS-PAGE.
- Konzentration der eluierten WT oder Sniper-Cas9 Protein mit 100 kDa Spaltenfilter. Speichern Sie die Proben in einer Lösung aus 10 mM Tris-HCl, 150 mM NaCl und 50 % Glycerin bei-80 ° C.
(6) RNP Lieferung
-
Transfektion und Vorbereitung der Zellen für die RNP Lieferung
- Pflegen Sie HEK293T Zellen in Dulbecco des Adlers Medium (DMEM) ergänzt mit 10 % fetalen bovine Serum (FBS) und 1 % Antibiotika bei 37 ° C mit 5 % CO2geändert.
- WT oder Sniper-Cas9 Protein (2 μg) mit SgRNA (2 μg) mischen und 10 min bei RT RNP-komplexe zu inkubieren.
- Trypsinize und die Zellen zu zählen. 2 x 104 Zellen pro eine Reaktion vorzubereiten. Waschen Sie die Zellen mit Phosphat-gepufferte Kochsalzlösung (PBS) und Zentrifuge. Aspirieren Sie überstand und Aufschwemmen Sie das Pellet mit Elektroporation Puffer.
- Electroporate RNP-komplexe in die Zellen mit den folgenden Einstellungen, Puls nämlich 1.300 V, 30 ms und ein. Platte die Zellen auf einem 48-Well-Platte mit 500 μL der DMEM ergänzt mit FBS und Antibiotika (wie in Schritt 6.1.1 beschrieben) direkt nach der Elektroporation gefüllt. Inkubation bei 37 ° C mit 5 % CO2.
- Die genomische DNA mit einer gDNA Vorbereitungssatz 48 h nach der Transfektion zu isolieren.
7. die Transfektion von Plasmiden Codierung Sniper-Cas9 und sgRNA
-
Bau einer SgRNA-plasmid
- Bestellen vorwärts und rückwärts Oligos mit der folgenden Vorlage Sequenzen, nämlich nach vorn: 5'-CACCGNNNNNNNNNNNNNNNNNNNN-3 "; Rückseite: 5'-AAACNNNNNNNNNNNNNNNNNNNNC-3 ". Ersetzen Sie N20 durch die Zielsequenz in Schritt 4.1.2 erhaltenen. Verkürzen Sie die Zielsequenz auf eine Länge von N19, N18 oder N17, abgeschnittene SgRNA zu synthetisieren.
- Tempern Sie beide Oligos in 1 x T4 DNA-Ligase Puffer.
- Verdauen Sie den pRG2-Vektor mit BsaI Restriktionsenzym.
- Gel zu reinigen den verdauten Vektor (3.300 bp) mit einem 0,8 % Agarose-Gel.
- Verbinden der geglühten Oligo und das gereinigte Fragment mit T4-Ligase bei 37 ° C: Mischung 50 ng der verdauten pRG2 und 1 ng des geglühten Oligo in einem Reaktionsvolumen von 20 μl. 15 min bei RT inkubieren.
- Verwandeln Sie die Ligatur-Mischung in die DH5 alpha Belastung und wachsen Sie Transformanten auf LB-Agar-Platten mit Ampicillin (100 μg/mL) bei 37 ° C. Bestätigen Sie das Einfügen von Oligo im Vektor durch standard Sequenzierung.
-
Transfektion von Plasmiden Codierung Sniper-Cas9 und sgRNA
- Pflegen Sie HEK293T Zellen in DMEM mit 10 % FBS und 1 % Antibiotika bei 37 ° C mit 5 % CO2ergänzt. Am Vortag Transfektion, trypsinize und die Zellen zu zählen. Beim Arbeiten in einer Skala 48-Well Platte 1 x 105 Zellen pro Bohrloch in 250 μL des vollständigen Wachstumsmedium. Die Zellen sollten am Tag der Transfektion Zusammenfluss von 50 – 80 % sein.
- Auf der Skala 48-gut vorzubereiten 250 ng Plasmid p3s-Cas9 und 250 ng SgRNA Plasmid zur Transfektion mit einem Lipid-basierte Transfection Reagens. Mischen Sie die Plasmide in 25 μL Serum frei-MEM.
- Verdünnen Sie 1 μL Transfection Reagens mit 25 μL Serum frei-MEM. Brüten Sie der Mischung bei RT 5 min kombinieren die beiden Mischungen und inkubieren Sie die resultierende Lösung bei RT für 20 min in Form Plasmid-Lipofectamine komplexe.
- Fügen Sie nach 20 min Inkubation 50 μL der Lösung mit der Plasmid-Transfection Reagens komplexe direkt zu jedem gut enthaltende Zellen, und mischen Sie leicht von der Platte hin und her schaukeln. Inkubation der Zellen bei 37 ° C in einem CO-2 -Inkubator für 48-72 h Post Transfektion vor Prüfung für Transgene Ausdruck.
8. Berechnung der Indel Frequenzen, Ziel- und Ziel-Tätigkeiten zu bestimmen
-
Gezielte Tiefe Sequenzierung zur Analyse von zielgerichtet und mögliche Ziel-Websites
- Genomischen DNA aus Schritt 6.1.5 oder 7.2.4 mit einem gDNA Vorbereitungssatz zu isolieren. Generieren Sie Tiefe Sequenzierung Bibliotheken durch PCR-Amplifikation von der gDNA mit Primern, die Ausrichtung am Ziel und Ziel.
- Verwenden Sie Index-Primer, um jede Probe zu kennzeichnen. Vorbehaltlich gepoolte Bibliotheken gepaart-End Sequenzierung mit einem Next Generation Sequencing-Maschine.
-
Tiefe Sequenzierung Analyse mit Cas-Analyzer
- Tiefe Sequenzierung Datenanalyse mit der Cas-Analyzer Assessment Tool17.
- Wählen Sie die Fastq-Dateien unter den Tabs Lesen 1 und 2 lesen (Read 1 = XX_SXX_L001_R1_001.fastq, lesen Sie 2 = XX_SXX_L001_R2_001.fastq).
- Füllen Sie die Registerkarte " Basisinformationen " und die Registerkarte " Parameter " klicken Sie auf die Schaltfläche " senden ".
Ergebnisse
Nachdem Sniper-Bildschirm durchgeführt wurde, kann der Anteil der überleben Kolonien berechnet werden, indem die Anzahl der Kolonien auf der LB-Platte mit Chloramphenicol, Kanamycin, Arabinose und ATC (CKAA) durch die Anzahl der Kolonien in der LB-Platte mit Chloramphenicol und Kanamycin nur (CK). Dieser Anteil war in der Regel sehr niedrig, als Sniper-Bildschirm mit den Bibliotheken des SpCas9 durchgeführt wurde. Wahr-Positive Treffer können durch die Wiederholung des Bildschirms mit den Überlebenden Pool angereichert werden. In diesem Vertreter Sniper-Bildschirm erhielt beispielsweise eine Überlebensrate von 100 % nach dem dritten Bildschirm (Abbildung 1). Transfektionen mit RNPs oder Plasmid-kodierte Sniper-Cas9 können für verschiedene Ziele und die daraus resultierende zielgerichtet erfolgen und Ziel-Aktivitäten gemessen durch gezielte Amplikons Sequenzierung (Abbildung 2). Bei den meisten Zielen, Sniper-Cas9 zeigt das gleiche Maß an Aktivitäten zielgerichtet und höhere Spezifität Verhältnisse im Vergleich zu den WT gestauchtes SgRNAs können auch zur Spezifität (Abbildung 3) weiter zu verbessern. Ihr Einsatz ist jedoch beschränkt sich auf nur wenige Ziele, weil sie niedrig auf Ziel-Aktivitäten im Vergleich zu Full-length SgRNAs, in den meisten Fällen führen. Daher SgRNAs mit unterschiedlichen Längen (von 17 - bis 20-Mers) müssen getestet werden und am Ziel und Ziel-Aktivitäten müssen gemessen werden, um die Spezifität zu optimieren.

Abbildung 1: Vertreter Sniper-Bildschirme mit verschiedenen Bibliotheken von zufälligen Cas9 Varianten durchgeführt. Mutator Bibliothek, EP I und II EP-Bibliothek Bibliotheken gemacht, mit verschiedenen kommerziellen Kits zeigen. Erste, zweite und Finale zeigen, dass die Anzahl der Zeiten, die der Anreicherung Bildschirm war7durchgeführt. Diese Zahl wurde von Lee Et Al.7geändert. Bitte klicken Sie hier für eine größere Version dieser Figur.
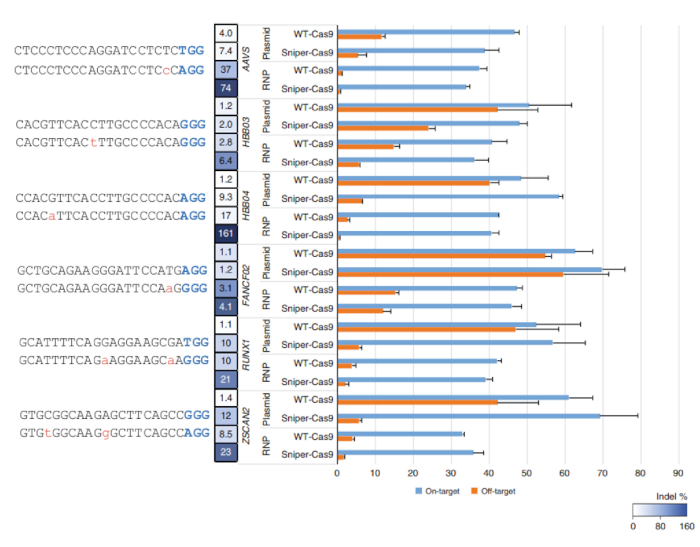
Abbildung 2: am Ziel und Ziel-Aktivitäten der WT-Cas9 oder Sniper-Cas9 gepaart mit einer 20-Mer Führer Sequenz per Plasmid oder RNP. Spezifität Verhältnisse waren bestimmt, indem die Zielaktivität durch die Ziel-Aktivität. Fehlerbalken zeigen SEM (n = 3)7. Diese Zahl wurde von Lee Et Al.7geändert. Bitte klicken Sie hier für eine größere Version dieser Figur.
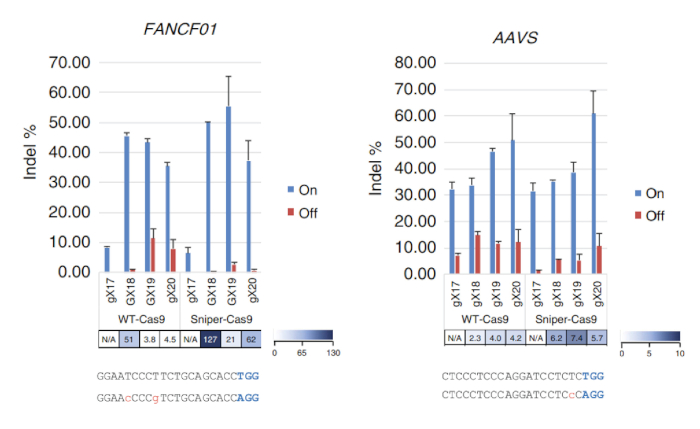
Abbildung 3: am Ziel und Ziel Aktivitäten der Sniper-Cas9 im Vergleich zum WT-Cas9 unter Verwendung von SgRNAs mit variabler Länge, die an den Standorten FANCF01 und Flugabwehrpanzer. Spezifität Verhältnisse waren bestimmt, indem Indel Frequenzen auf Ziel-Sites durch die an den jeweiligen Standorten Ziel. SgRNAs mit einem übereinstimmenden Guanin am 5' Terminus (GX18 oder GX19) und solche mit einer nicht übereinstimmenden Guanin (gX17, gX18, gX19 oder gX20) werden angezeigt. Spezifität Verhältnisse wurden nicht berechnet, wenn die normalisierten zielgerichtet Aktivitäten waren < 70 %7. Diese Zahl wurde von Lee Et Al.7geändert. Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
Für diejenigen, die umständlich Screening-Verfahren zu Sniper-Cas9 vermeiden wollen, gibt es das Sniper-Cas9 Protein und die Codierung Plasmid. Unter Verwendung dieser Materialien die optimale Länge der SgRNA, das bietet die höchste Spezifität Verhältnis ermittelt werden soll. Lieferung von Sniper-Cas9 und SgRNA in einem RNP-Format wird darüber hinaus empfohlen, da es in der Regel ergibt sich eine höhere Spezifität-Verhältnis als Lieferung in einem Plasmid-Format. Im Gegensatz zu Sniper-Cas9 sind andere technischen Cas9-Varianten nicht kompatibel mit abgeschnittenen SgRNAs6,7 oder Lieferung im RNP Form8 (mit Ausnahme von HiFi-Cas9).
Für Sniper-Bildschirm ist die Auswahl der nicht übereinstimmenden Sequenz der wichtigste Schritt. Auswahl an eine nicht übereinstimmende SgRNA, die mit niedrigen Spaltung Aktivität in Richtung der GOI -Sequenz ist, sollte vermieden werden. Wenn dies nicht der Fall ist, Cas9 Varianten mit einem WT Spezifität werden die E. Coli genomischen DNA mit den nicht übereinstimmenden Zielstandort nicht Spalten. Dieser Effekt führt eine große Anzahl von Hintergrund-Kolonien, gefährden die gesamte screening-Verfahren.
Da E. Coli eine Fast hat Verdoppelung der Zeit und hohe Transformationseffizienz verglichen mit Hefe, Sniper-Bildschirm vorteilhaft Hefe-basierten Screening-Methoden gegenüber. Sniper-Bildschirm sollte sein empfindlicher als andere E. Coli-basierten Systemen, die nicht übereinstimmende Website erfolgt, auf einem Plasmid: gibt es eine Kopie der genomischen DNA und damit nur eine Kopie der nicht übereinstimmenden Site in unserem System, sondern eine große Anzahl von Plasmide innerhalb einer einzigen E. Coli Zelle.
Die Besonderheiten des anderen DNA-Endonucleases, die DSB, z. B. SaCas9 oder Cpf1s, induzieren könnte auch verbessert werden, mithilfe von Sniper-Bildschirm. Leider kann nicht Sniper-Bildschirm direkt, erhöhen die Spezifität des Basis-Editoren verwendet werden, da base Editoren nicht dazu, DSB in der genomischen DNA von E. Coli veranlassen. Als Basis Editoren die Nickase oder tot Cas9 in den Kern ihres Systems verwenden, könnte die Besonderheiten von Basis-Editoren erhöht werden, mit den Hits von Sniper-Bildschirm erhalten.
Offenlegungen
ToolGen hat eine Patentanmeldung (PCT/KR2017/006212) für Sniper-Bildschirm eingereicht (Status: pending, Erfinder: Jungjoon K. Lee). Jungjoon K. Lee, Joonsun Lee, Minhee Jung und Euihwan Jeong sind Mitarbeiter von ToolGen, Inc.
Danksagungen
Diese Forschung wurde unterstützt durch Zuschüsse aus dem Ministerium für Wissenschaft und IKT-Korea (2017M3A9B4061406) und die National Research Foundation von Korea (NRF) finanziert von der koreanischen Regierung (MSIT) (Zuschuss Zahlen 2017M3A9B4061404 und 2018M3A9H3020844) Jungjoon K. Lee. Das Plasmid pGRG36 Codierung war ein Geschenk von Nancy Craig (Addgene Plasmid #16666) und p11-LacY-wtx1 war ein Geschenk von Huimin Zhao.
Materialien
| Name | Company | Catalog Number | Comments |
| Alkaline Phosphatase, Calf Intestinal (CIP) | NEB | M0290L | |
| Ampicillin Sodium Salt | Fisher Chemical | BP1760-25 | |
| Anhydrotetracycline hydrochloride | Sigma Aldrich | 37919 or 94664 | |
| Antibiotic Antimycotic solution | Welgene | LS203-01 | Media component |
| BamHI | Enzynomics | R003L | |
| Chloramphenicol | Sigma Aldrich | R4408 | |
| Diversify PCR Random Mutagenesis | Clontech | 630703 | Error-prone PCR kit |
| DNA-Shuffling Kit | Jena Bioscience | PP-103 | |
| Dulbecco's Modified Eagle's Medium (DMEM) | Welgene | LM001-17 | Culture media |
| Endura Electrocompetent Cells (DUO) | Lucigen | 60242-2 | E. coli electrocompetent cells for library preparation |
| Fetal Bovine Serum (FBS) | Welgene | S101-01 | Media component |
| Gene Pulser II | Bio-Rad | E. coli electroporation equipment | |
| GeneMorph II Random Mutagenesis Kit | Agilent | 200550 | Error-prone PCR kit |
| genomic DNA prep kit | GenAll | 106-152 | gDNA-prep kit |
| Glycerol | BioShop | GLY001.1 | |
| GP/MP Cuvette, 0.1cm | Bio-Rad | BR165-2089 | E. coli electroporation equipment |
| HEK293T cell | ATCC | CRL-11268 | Human cell line |
| Kanamycin sulfate | Acros Organics | 450810500 | |
| L-(+)-Arabinose | Sigma Aldrich | A3256-25G | |
| LaboPass PCR Purification Kit | Cosmogenetech | CMR0112 | |
| LaboPass Plasmid Mini | Cosmogenetech | CMP0112 | Mini-prep kit |
| LB Agar Miller | Formedium | LMM0202 | |
| LB Broth Miller | Formedium | LMM0102 | |
| Lipofectamine | Invitrogen | 11668019 | Transfection reagent |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | Gibson assembly (isothermal in vitro recombination) mixture |
| Neon Transfection System | Thermo | MPK5000 | RNP Electroporation equipment |
| Neon Transfection System 10 µL Kit | Thermo | MPK1096 | RNP Electroporation equipment |
| NucleoBond Xtra Midi EF | Macherey-Nagel | 740420.50 | Midi-prep kit |
| Oligo Clean & Concentrator | Zymo Research | D4061 | DNA purification kit that enables low-volume elution |
| Opti-MEM | Gibco | LS31985070 | Transfection media |
| p11-lacY-wtx1 | Addgene | #123945 | |
| pgrg36 | Addgene | #16666 | E. coli mutator strain |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530L | |
| Pyrophophatase | NEB | M2403L | |
| Ribonucleotide Solution Set | NEB | N0450L | |
| RNase inhibitor | NEB | M0314L | |
| T7 RNA polymerase | NEB | M0251L | |
| Turbo Dnase | Ambion | AM2238 | |
| XbaI | Enzynomics | R013L | |
| XL1-Red Competent Cells | Agilent | 200129 |
Referenzen
- Fu, Y., Sander, J. D., Reyon, D., Cascio, V. M., Joung, J. K. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nature Biotechnology. 32 (3), 279-284 (2014).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Research. 24 (6), 1012-1019 (2014).
- Chen, J. S., et al. Enhanced proofreading governs CRISPR-Cas9 targeting accuracy. Nature. 550 (7676), 407-410 (2017).
- Kleinstiver, B. P., et al. High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects. Nature. 529 (7587), 490-495 (2016).
- Slaymaker, I. M., et al. Rationally engineered Cas9 nucleases with improved specificity. Science. 351 (6268), 84-88 (2016).
- Casini, A., et al. A highly specific SpCas9 variant is identified by in vivo screening in yeast. Nature Biotechnology. 36 (3), 265-271 (2018).
- Lee, J. K., et al. Directed evolution of CRISPR-Cas9 to increase its specificity. Nature Communications. 9 (1), 3048(2018).
- Vakulskas, C. A., et al. A high-fidelity Cas9 mutant delivered as a ribonucleoprotein complex enables efficient gene editing in human hematopoietic stem and progenitor cells. Nature Medicine. 24 (8), 1216-1224 (2018).
- Anderson, K. R., et al. CRISPR off-target analysis in genetically engineered rats and mice. Nature Methods. 15, 512-514 (2018).
- Kim, S., Bae, T., Hwang, J., Kim, J. S. Rescue of high-specificity Cas9 variants using sgRNAs with matched 5' nucleotides. Genome Biology. 18 (1), 218(2017).
- Kulcsar, P. I., et al. Crossing enhanced and high fidelity SpCas9 nucleases to optimize specificity and cleavage. Genome Biology. 18 (1), 190(2017).
- Zhang, D., et al. Perfectly matched 20-nucleotide guide RNA sequences enable robust genome editing using high-fidelity SpCas9 nucleases. Genome Biology. 18 (1), 191(2017).
- McKenzie, G. J., Craig, N. L. Fast, easy and efficient: site-specific insertion of transgenes into enterobacterial chromosomes using Tn7 without need for selection of the insertion event. BMC Microbiology. 6, 39(2006).
- Geissmann, Q. OpenCFU, a new free and open-source software to count cell colonies and other circular objects. PLoS One. 8 (2), e54072(2013).
- Chen, Z., Zhao, H. A highly sensitive selection method for directed evolution of homing endonucleases. Nucleic Acids Research. 33 (18), e154(2005).
- Kim, S., et al. CRISPR RNAs trigger innate immune responses in human cells. Genome Research. 28 (3), 367-373 (2018).
- Park, J., et al. Cas-Analyzer: an online tool for assessing genome editing results using NGS data. Bioinformatics. 33, 286-288 (2017).
- Studier, F. W., et al. Use of T7 RNA Polymerase to Direct Expression of Cloned Genes. Methods in Enzymology. 185, 60-89 (1990).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten