Method Article
Rab10 détection de Phosphorylation par LRRK2 l’activité à l’aide de SDS-PAGE avec une balise de Phosphate-binding
Dans cet article
Résumé
La présente étude décrit une méthode simple de détecter des taux endogènes de la phosphorylation de Rab10 de leucine-rich repeat kinase 2.
Résumé
Mutations de leucine-rich repeat kinase 2 (LRRK2) ont été démontrées d’être liée à la maladie de Parkinson familiale (FPD). Étant donné que l’activation anormale de l’activité de la kinase de LRRK2 a été impliquée dans la pathogénie de PD, il est essentiel d’établir une méthode permettant d’évaluer les niveaux physiologiques de l’activité de la kinase de LRRK2. Des études récentes ont révélé que LRRK2 phosphoryle des membres de la famille Rab GTPase, y compris Rab10, dans des conditions physiologiques. Bien que la phosphorylation de Rab10 endogène par LRRK2 dans des cellules cultivées puisse être détectée par spectrométrie de masse, il a été difficile de le détecter par immunotransfert en raison de la sensibilité médiocre d’anticorps de phosphorylation spécifiques actuellement disponibles pour Rab10. Nous décrivons ici une simple méthode de détecter les niveaux endogènes de la phosphorylation de Rab10 par LRRK2 basée sur immunoblotting utilisant sur gel de polyacrylamide sodium dodecyl sulfate (SDS-PAGE) combinée avec une étiquette de liaison phosphate (P-tag), qui N-(5-(2-aminoethylcarbamoyl)pyridin-2-ylmetyl) -N,N',N'- tris (pyridin-2-yl-méthyl) - 1,3 - diaminopropan-2-ol. Le présent protocole non seulement fournit un exemple de la méthode utilisant la balise P mais permet également l’évaluation de comment les mutations comme inhibiteur traitement/administration ou tout autre facteur modifie la signalisation en aval de LRRK2 dans les cellules et tissus .
Introduction
PD est l’un des maladies neurodégénératives plus répandues, affectant principalement les neurones dopaminergiques dans le midbrain, ce qui entraîne un dysfonctionnement des systèmes moteurs dans personnes âgées1. Alors que la plupart des patients développent des PD de manière sporadique, il y a des familles qui héritent de la maladie. Mutations dans plusieurs gènes ont été retrouvées à être assemblé avec le FPD2. Un des gènes responsables de FPD est LRRK2, dans lequel huit mutations faux-sens (N1437H, R1441C/G/H/S, Y1699C, G2019S et I2020T), liées à un FPD principalement héréditaire appelé PARK8 ont été jusqu'à présent signalé3,4,5. Plusieurs association pangénomique (GWAS) des patients atteints de la MP sporadiques ont également recensée variations génomiques du locus LRRK2 comme un facteur de risque pour PD, suggérant que l’anomalie dans le fonctionnement du LRRK2 est une cause fréquente de neurodégénérescence dans deux sporadiques et PARK8 FPD6,7,8.
LRRK2 est une grosse protéine (2 527 acides aminés), consistant en un domaine riche en leucine, un liant le GTP de Ras du domaine des protéines complexes (ROC), un C-terminal du domaine, un domaine kinase de sérine/thréonine protéine et un domaine de WD409ROC (COR). Les mutations de FPD huit localiser dans ces domaines fonctionnels ; N1437H et R1441C/G/H/S dans le domaine ROC, Y1699C dans le domaine de la CDR, G2019S et I2020T dans le domaine kinase. Car la mutation G2019S, que l'on trouve le plus souvent de mutation du PD patients10,11,12, augmente l’activité de la kinase de LRRK2 par 2-3 fois in vitro13, on fait l’hypothèse que l’augmentation anormale du phosphorylation de fonction LRRK2 est toxique pour les neurones. Cependant, il a été impossible d’étudier si la phosphorylation des substrats de LRRK2 physiologiquement pertinents est altérée dans les patients Parkinsoniens familial/sporadiques en raison du manque de méthodes évaluant dans les échantillons de patients dérivées.
La phosphorylation des protéines est généralement détectée par immunotransfert ou dosage immuno-enzymatique (ELISA) utilisant des anticorps reconnaissant spécifiquement l’État phosphorylé de protéines ou par spectrométrie de masse. Toutefois, la stratégie de l’ancienne parfois impossible d’appliquer à cause des difficultés dans la création d’anticorps spécifiques à la phosphorylation. Un marquage métabolique des cellules avec phosphate radioactif est une autre option à examiner les niveaux physiologiques de la phosphorylation quand la phosphorylation propres anticorps ne sont pas facilement disponibles. Toutefois, elle nécessite une grande quantité de matières radioactives et implique donc un équipement spécialisé pour la radioprotection14. L’analyse par spectrométrie de masse est plus sensible par rapport à ces méthodes immunochimiques et est devenu populaire dans l’analyse de la phosphorylation des protéines. Cependant, la préparation de l’échantillon est fastidieux et coûteux instruments sont requis pour l’analyse.
Un sous-ensemble de la famille Rab GTPase, y compris Rab10 et Rab8 a été récemment rapporté que des substrats physiologiques directs pour LRRK2 basée sur le résultat d’une analyse de phosphoproteomic à grande échelle15. Ensuite, nous avons démontré que la phosphorylation de Rab10 a été augmentée de mutations de FPD dans les fibroblastes embryonnaires de souris et dans les poumons des souris16de knockin. Dans ce rapport, nous avons choisi d’employer une électrophorèse de gel de polyacrylamide sodium dodécyl sulfate (SDS-PAGE)-méthode dans laquelle une molécule de P-tag est co polymérisée en gel SDS-PAGE (balise P SDS-PAGE) pour détecter les niveaux endogènes de la phosphorylation de Rab10, fondée sur parce qu’un anticorps très sensible spécifique pour Rab10 phosphorylés manquait encore. Nous n’avons pas réussi à détecter la phosphorylation de Rab8 endogène en raison de la faible sélectivité des anticorps actuellement disponibles pour Rab8 total. Par conséquent, nous avons décidé de mettre l’accent sur la phosphorylation de Rab10. LRRK2 phosphoryle Rab10 à Thr73 localiser au milieu de la région hautement conservée « switch II ». Haute conservation des sites de phosphorylation des protéines Rab est peut-être une des raisons pour lesquelles phosphospecific anticorps reconnaissant les protéines Rab distinctes sont difficiles à faire.
La phosphorylation de Rab8A par LRRK2 inhibe la liaison du Rabin8, un facteur d’échange de nucléotide de guanine (FEM) qui active la Rab8A en échangeant le PIB lié avec GTP15. La phosphorylation de Rab10 et Rab8A par LRRK2 inhibe aussi la liaison des inhibiteurs de la dissociation du PIB (GDIs), qui sont essentiels à l’activation des protéines Rab en extrayant Rab PIB lié aux protéines des membranes15. Collectivement, c’est l’hypothèse que la phosphorylation des protéines Rab par LRRK2 les empêche d’activation bien que le mécanisme moléculaire précis et les conséquences physiologiques de la phosphorylation demeurent flous.
P-tag SDS-PAGE a été inventé par Kinoshita et al. , en 2006 : dans cette méthode, acrylamide était par covalence associée à P-tag, une molécule capturant des phosphates avec une haute affinité, qui copolymérisés en SDS-PAGE gélifie17. Parce que les molécules de P-tag dans un gel SDS-PAGE retardent sélectivement la mobilité électrophorétique des protéines phosphorylées, P-tag SDS-PAGE peut séparer des protéines phosphorylées de ceux non-phosphorylés (Figure 1). Si la protéine d’intérêt est phosphorylé sur les résidus de multiples, on observera une échelle des bandes correspondant aux formes phosphorylées différemment. Dans le cas de Rab10, nous n'observons qu’une bande décalée, indiquant que Rab10 est phosphorylé uniquement à Thr73. L’avantage majeur de P-tag SDS-PAGE immunoblotting avec des anticorps spécifiques à la phosphorylation est que Rab10 phosphorylés peuvent être détectées par immunotransfert avec des anticorps non spécifiques à la phosphorylation (c.-à-d., reconnaissance Rab10 total) Après avoir été transféré sur la membrane, qui est généralement plus spécifiques, sensibles et disponible de sources commerciales et universitaires. Un autre avantage d’utiliser la balise P SDS-PAGE, c’est qu’on peut obtenir une estimation approximative de la stoechiométrie de la phosphorylation, ce qui est impossible par immunotransfert avec des anticorps spécifiques à la phosphorylation ou par marquage métabolique des cellules avec radioactif phosphates.
Hormis l’utilisation peu coûteuse balise P acrylamide et quelques modifications mineures liées à celui-ci, la présente méthode pour la détection de la phosphorylation de Rab10 par LRRK2 suit un protocole général d’immunoblotting.Par conséquent, il devrait être simple et facilement exécutable dans les laboratoires où immunoblotting est une pratique habituelle, avec tous les types d’échantillons, y compris des homogénats tissulaires, lysats cellulaires et protéines purifiées.
Protocole
1. préparation pour la balise P SDS-PAGE
- Enlever et jeter les médias de la vaisselle de 10 cm dans laquelle les cellules croissent en utilisant une aspiration et laver les cellules avec tampon phosphate salin de Dulbecco 5 mL (SPD) de première SPD ajoutant au côté des plats pour ne pas perturber la couche cellulaire rock manuellement les plats arrière et plusieurs fois de suite.
- Retirer et jeter le SPD à l’aide d’une aspiration et ajouter 2 mL de trypsine de 0,25 % (p/v) diluée dans du SPD et balancer doucement les plats pour couvrir la couche de cellules. Mettre les plats dans un incubateur à CO2 (37 ° C, l’air humidifié, 5 % de CO2) pendant 5 min.
- Après avoir effectué de haut en bas en utilisant une pipette jetable pour briser les cellules individuelles, recueillir la suspension de cellules dans un tube de 15 mL et mesurer la densité des cellules à l’aide d’un hematocytometer sous un microscope18.
- Diluer les cellules à 2,5 × 105 cellules/mL avec le milieu Eagle modifié de Dulbecco (DMEM) additionnés de 10 % (v/v) sérum fœtal (SVF), 100 U/mL de pénicilline et 100 µg/mL de streptomycine. Ajouter 2 mL (5 × 105 cellules) de la suspension cellulaire dilué dans chaque puits de plaques 6 puits.
- Cultiver les cellules pendant la nuit dans un incubateur à CO2 (37 ° C, l’air humidifié, 5 % de CO2).
- Transfection dans les cellules HEK293
Remarque : Si la phosphorylation de Rab10 endogène doit être examinée, passer au point 1.7. On trouvera des plasmides utilisés dans le présent protocole auprès des auteurs sur demande. Voir Table des matières pour brèves informations et Figure S1 pour leur DNA sequences.- Aliquote à 200 µL de DMEM dans des tubes de 1,5 mL.
- Dans chaque tube, ajouter les deux 0,266 µg (concentration finale des 1,33 µg/mL) d’un plasmide codant HA-Rab10 et 1,066 µg (concentration finale de 5,33 µg/mL) d’un plasmide codant 3 × drapeau-LRRK2 provenant des stocks de plasmide 500 µg/mL. Ajouter ensuite 4 µL de solution de 1 mg/mL de POLYÉTHYLÈNEIMINE (dissous dans 20 mM HEPES-NaOH, pH 7,0) et mélanger immédiatement la solution au Vortex pendant 5 s.
- Laisser les tubes reposer à température ambiante pendant 10 min et ajouter le contenu d’un tube goutte à goutte d’un bien en utilisant une micropipette. Doucement et manuellement de roche les plaques de culture plusieurs fois en arrière pour permettre le mélange de transfection diffuse uniformément tout au long du puits.
- Laisser les cellules à se développer pendant encore 24 à 36 h dans un incubateur à CO2 (37 ° C, l’air humidifié, 5 % de CO2).
- Si la phosphorylation de Rab10 endogène est à examiner, traiter des cellules avec ou sans inhibiteurs LRRK2 pendant 1 h avant la lyse des cellules.
- Préparer les solutions d’inhibiteurs LRRK2 en dissolvant les inhibiteurs dans le diméthylsulfoxyde (DMSO) à 10 mM. Nous vous recommandons d’utiliser IML-2 et GSK2578215A, qui sont très spécifiques et puissants inhibiteurs LRRK2. Stocker les solutions mères à-80 ° C.
- Préparer des stocks de travail des inhibiteurs en diluant les solutions mères avec le DMSO : pour IML-2, préparer 10 µM et 30 µM endogène et surexprimée LRRK2, respectivement. Pour GSK2578215A, préparer 1 mM et 3 mM pour endogène et surexprimée LRRK2, respectivement.
- Ajouter 2 µL des stocks travail d’IML-2 ou GSK2578215A au milieu d’un bien à l’aide d’une micropipette, faisant doucement tourner la plaque manuellement en arrière à plusieurs reprises de laisser les inhibiteurs diffuse uniformément tout au long du puits.
- Ajouter 2 µL de DMSO à un puits différent comme témoin négatif d’une manière similaire à l’inhibiteur dans l’étape 1.8.3.
- Remettre les plaques dans l’incubateur et la culture des cellules pendant 1 h.
- Lyse des cellules.
- Mettre les plaques de culture sur la glace. Retirez et jetez les médias. Laver les cellules premier ajout 2 ml SPD au côté des plats pour ne pas perturber la cellule couche et de roche manuellement les plats en arrière à plusieurs reprises.
- Retirer et jeter le SPD et ajoute les cellules 100 µL de la mémoire tampon de lyse (50 mM Tris-HCl de pH 7.5, 1 % (v/v) polyoxyethylene(10) octylphényle éther, 1 mM EGTA (éthylène glycol-bis(2-aminoethylether)-N, N, N', N'-tétraacétique), orthovanadate de sodium 1 mM, 50 mM le fluorure de sodium, 10 mM β-glycérophosphate, pyrophosphate de sodium 5 mM, 0,1 µg/mL microcystine-LR, saccharose 270 mM, inhibiteur de la protéase cocktail).
- Inclinez les plaques sur la glace et raclez les cellules en utilisant un grattoir de cellules pour recueillir autant cellule lysat que possible. Recueillir les lysats à l’aide d’une micropipette dans des tubes de 1,5 mL (préalablement refroidis sur la glace).
ATTENTION : Microcystine-LR peut être fatal si avalé ou contact avec la peau.
- Laisser les tubes sur la glace pendant 10 min pour la lyse complète. Clarifier les lysats cellulaires par centrifugation (20 000 × g, 10 min à 4 ° C) et transférer les surnageants dans de nouveaux tubes de 1,5 mL préalablement refroidis sur la glace.
- Mesurer la concentration de protéines (µg/µL) des lysats défrichées par analyse de Bradford.
- Établir l’albumine sérique bovine (BSA) des normes propres (0,2, 0,4, 0,6, 0,8 et 1 mg/mL) en diluant la solution-mère avec de l’eau distillée. Diluer les lysats de cellules défrichées par 20 fois avec de l’eau distillée.
- Mettre 5 µL/puits des normes de BSA, blank (eau distillée) et chaque cellule dilué lysat dans une plaque à 96 puits en triple exemplaire.
- Ajouter 150 µL/puits de réactif d’analyse de la Bradford à l’aide d’une micropipette 12 canaux et laisser la plaque à température ambiante pendant 5 min.
- Mesurer l’absorbance à 595 nm sur un lecteur de plaques et de la comparer aux normes de la BSA.
- Préparer 100 µL d’échantillons SDS-PAGE. La concentration de protéine des échantillons est de 1 µg/µL pour surexprimée HA-Rab10 et 2 µg/µL pour Rab10 endogène.
- En utilisant les concentrations quantifiées de l’étape 1.11.4, calculer le volume (µL) des lysats cellulaires équivalant à 100 µg (surexprimée HA-Rab10) ou 200 µg (Rab10 endogènes) en divisant la quantité de protéine (100 µg ou 200 µg) par la concentration de protéines de lysats (µ g/µL).
- Ajouter 25 µL de tampon de Laemmli 4 × SDS-PAGE (62,5 mM Tris-HCl, pH 6,8, 8 % (p/v) SDS, 40 % (v/v) de glycérol, bleu de bromophénol 0,02 % (p/v), 4 % (v/v) de β-mercaptoéthanol) dans de nouveaux tubes de 1,5 mL conservés à température ambiante.
- Ajouter de la valeur de la cellule lysats à chaque tube et mélangent au Vortex pendant 5 s à température ambiante.
- Porter le volume total à 100 µL avec le tampon de lyse et mélanger au Vortex pendant 5 s à température ambiante.
- Supplément de 10 mM MnCl2 pour étancher l’agent chélatant. Ajouter 1 µL de 500 mM MnCl2. Mix au Vortex pendant 5 s à température ambiante.
Remarque : Les échantillons contenant MnCl2 peuvent ne pas convenir pour SDS-PAGE normale.
2. moulage Gels pour P-tag SDS-PAGE
Remarque : Les Gels doivent être effectuées sur le même jour que les gels en cours d’exécution. Gels en vertu des conditions de luminosité ambiantes.
- Préparer 5 mM P-tag acrylamide solution première dissoudre 10 mg de poudre/solide P-tag acrylamide complètement avec 100 µL de méthanol et ensuite porter à 3,3 mL en ajoutant de l’eau bidistillée.
Remarque : L’acrylamide P-tag est sensible à la lumière. La solution ainsi préparée doit être stockée dans l’obscurité à 4 ° C jusqu'à l’utilisation. - Plaques propre clair et entaillées par pulvérisation de l’éthanol à 70 % et essuyer avec une serviette de papier. Assembler les plaques de gel. Les dimensions des plaques gel utilisés dans ce protocole particulier sont 80 ou 100 mm de long par 100 mm de large. Entretoises de silicium pur sont mis entre plaine et plaques entaillées et l’assemblé sont fixés avec des clips de liant.
Remarque : N’importe quel type de plaques de gel qui fonctionnent pour SDS-PAGE ordinaire peut être utilisé. - Mettre un peigne pour être utilisé (peigne en plastique 17 puits) dans la marque sur la plaque de gel la position du fond des puits et des plaques de gel assemblé avec un marqueur permanent.
- Préparer 10 mL de mélange de gel d’acrylamide 10 % (10 % (p/v) d’acrylamide (acrylamide:bis-acrylamide = 29 : 1), 375 mM Tris-HCl (pH 8,8), 0,1 % (p/v) SDS) dans un tube de 15 mL.
Remarque : La concentration optimale d’acrylamide peut varier selon les réactifs utilisés. Tétraméthyléthylènediamine (TEMED) et ammonium persulfate (APS) ne doit pas être ajouté à ce stade. - Ajouter 100 µL d’acrylamide P-balise de 5 mM et 10 µL de 1 M MnCl2 solutions à la concentration finale de 50 µM et 100 µM, respectivement.
Remarque : Les concentrations optimales de l’acrylamide P-tag et le MnCl2 peuvent également varier selon les réactifs utilisés. - Ajouter 15 µL de TEMED dans le mélange de gel à une concentration finale de 0,15 % (v/v) et ensuite 50 µL de 10 % (p/v) : APS à une concentration finale de 0,05 % (p/v). Bien mélanger en agitant doucement le tube pendant 5 s et verser dans les boîtes assemblées immédiatement jusqu'à une hauteur de 2 mm ci-dessous la position indiquée à l’étape 2.3.
- Doucement la couche 2-propanol dans la solution de gel pour aplatir le dessus de séparer les gels.
- Les gels, laissez reposer pendant 30 min à température ambiante. Il n’est pas nécessaire de protéger les gels de la lumière.
NOTE : Il pourrait prendre plus de temps pour les gels en vertu de la température de la pièce froide. Le mélange de gel de dégazage avant d’ajouter le TEMED et APS permet d’accélérer cette étape. À cette fin, le mélange de gel peut être préparé dans un erlenmeyer de 100-200 mL relié à une aspiration. Dégazer la solution pendant 10 min. - Enlever le couches 2-propanol en absorbant avec du papier absorbant.
- Laver l’espace supérieur des gels en remplissant l’espace avec de l’eau distillée d’un flacon laveur et jeter l’eau de décanter dans le bassin. Répéter 3 fois de lavage.
- Enlever l’eau résiduelle restant dans l’espace supérieur des gels en absorbant avec du papier absorbant.
- Préparer 3 mL de mélange de gel d’acrylamide 4 % (4 % (p/v) d’acrylamide (a: crylamide:bis-acrylamide = 29 : 1), 125 mM Tris-HCl (pH 6,8), 0,1 % (p/v) SDS) dans un tube de 15 mL.
Remarque : N’ajoutez pas de balise P acrylamide ou MnCl2 solution pour le mélange de gel de gerbage. - Ajouter 7,5 µL de TEMED et 24 µL de 10 % (p/v) : APS à la concentration finale de 0,25 % (v/v) et 0,08 % (p/v), respectivement. Bien mélanger en agitant doucement le tube pendant 5 s et versez le mélange sur le dessus du gel de séparation. Mettre immédiatement les peignes appropriés (les peignes en plastique 17-bien accueillir jusqu'à 25 µL d’échantillons, par exemple).
- Les gels, laissez reposer pendant 30 min à température ambiante. Il n’est pas nécessaire de protéger les gels de la lumière. Après que les gels empilement défini, exécutez-les sans être stockées.
3. SDS-PAGE et immunoblot
- Retirez les peignes de gels. Supprimez les entretoises de silicium et puis clips de gels.
- Mettre les gels coulées dans des réservoirs de gel et fixer le gel au réservoir en pinçant avec les clips de liant.
- Verser le tampon en cours d’exécution (25 mM Tris, 192 mM, Glycine, 0,1 % (p/v) SDS) en bas et haut des gels. Puits propres en rinçant le tampon en cours d’exécution à l’aide d’une seringue de 5 mL et une aiguille de 21G pour enlever les morceaux de gel.
- Enlever les bulles d’air de l’espace en bas des gels à l’aide d’une aiguille tordue, attachée à une seringue. Pour rendre une aiguille tordue, courber manuellement une aiguille 21G au milieu de l’aiguille pour que l’angle entre la pointe et la base de l’aiguille devient 30-45 °.
- Tournez en bas précipités causées par l’ajout de MnCl2 à 20 000 × g pendant 1 min à température ambiante pour prélever des échantillons de claires.
- Charger 10 µg de protéines pour la détection de la phosphorylation de Rab10 surexprimée et 30 µg de protéines pour Rab10 endogène.
Remarque : Il est essentiel de charger un volume égal d’échantillons dans tous les puits. Les ruelles vides doivent être chargés avec tampon de Laemmli de × 1 SDS-PAGE. Si les échantillons contiennent MnCl2, ajouter la même concentration de MnCl2 aux échantillons factices. - Le bien chargé avec un marqueur de poids moléculaire (MWM) devrait également être complété avec tampon de Laemmli 1 × SDS-PAGE donc le volume d’échantillons chargés est la même dans tous les puits.
Remarque : Encore une fois, si les échantillons contiennent MnCl2, s’ajoute la même concentration de MnCl2 le MWM. Par ailleurs, un MWM sans EDTA peut être utilisé. - Exécuter les gels à 50 V pour le gerbage (environ 30 min) jusqu'à ce que le front de colorant traverse dans le gel de séparation.
- Après que les échantillons de la pile, changer la tension de 120 V pour séparation jusqu'à ce que le front de colorant atteigne le fond des gels (environ 50 et 80 min pour 80 et 100 mm long gels, respectivement).
Remarque : Les habitudes migratoires de la MWM devrait être différente de celle sur les gels de SDS-PAGE normales. Il ne doit pas être utilisé pour estimer le poids moléculaire des protéines sur gel SDS-PAGE de P-tag, mais peut être utilisé pour vérifier la reproductibilité des gels P-tag. Pour plus d’informations, voir analyse . - Laver les gels pour retirer les gels de MnCl2 .
- Versez le transfert tampon (48 mM Tris, 39 mM, Glycine, méthanol 20 % (v/v)) contenant 10 mM EDTA et 0,05 % (p/v) : SDS dans un récipient (par exemple, gros bateaux de pesage).
Remarque : Utiliser la mémoire tampon fraîche pour chaque lavage.
- Placer un filtre en papier (10 x 7 cm) sur un plateau en mousse pour le transfert. Placer le gel sur le papier filtre. Assurez-vous qu’il n’y a aucune bulle d’air entre le gel et le papier filtre.
- Mettre une membrane (10 x 7 cm) sur le gel et assurez-vous qu’il n’y a aucune bulle d’air entre le gel et la membrane.
- Mettre un autre filtre en papier (10 x 7 cm) sur la membrane et, encore une fois, assurez-vous qu’il n’y a aucune bulle d’air entre la membrane et le papier filtre.
- Mettre un autre plateau en mousse sur le papier filtre. Placez la pile de papiers/filtre à membrane dans une cassette pour le transfert.
- Mettre la cassette dans un réservoir de transfert, en veillant à ce que la membrane se situe entre le gel et l’anode chargé positivement.
- Connectez le vase sur un bloc d’alimentation et mettre le réservoir dans une boîte de mousse de styrène remplie d’eau glacée. Démarrer le transfert à 100 V à 180 min.
NOTE : Déplacement prolongé est nécessaire étant donné que le transfert des protéines à partir de gels P-tag SDS-PAGE ne sont pas aussi efficace que celui de normales gels SDS-PAGE. Un refroidissement efficace est essentiel pour éviter la fonte des gels pendant le transfert.
- Retirer les membranes les gels à l’aide de la pince à épiler et tremper les membranes dans une solution de Ponceau S (0,1 % (p/v) Ponceau S, 5 % (v/v) d’acide acétique) pour colorer les protéines transférées sur les membranes dans un récipient en plastique. Le volume de la solution devrait être suffisant pour couvrir une membrane.
- Incuber les membranes pendant 1 min à température ambiante en poussant manuellement.
Remarque : Une échelle des bandes devienne visible dans chaque ruelle. - Ramasser la membrane par la solution de coloration avec des pincettes et voir si l’échelle des bandes a schéma uniforme et l’intensité dans chaque ruelle où les échantillons ont été chargés.
- Après avoir vérifié la coloration, retirer la solution colorante et ajouter un tampon TBST (20 mM Tris-HCl pH 7,4, 150 mM NaCl, 0,1 % (v/v) polyoxyethylenesorbitan monolaurate). Le volume de la mémoire tampon doit être suffisant pour couvrir une membrane.
Remarque : La solution de Ponceau S peut être collectée et réutilisé plusieurs fois. - Balancer les membranes dans TBST sur un agitateur basculant (~ 60 tr/min) à température ambiante jusqu'à ce qu’aucune bande visible sur les membranes.
- Répétez l’étape de lavage avec un mélange TBST frais pendant 5 min.
NOTE : Temps d’exposition peut varier selon la solution ECL et le système utilisé pour la détection de chimiluminescence.
- Allumez un imageur équipé d’une caméra à dispositif à couplage de charge (CCD) et un ordinateur connecté à l’imageur. Démarrez le logiciel de contrôle pour l’imageur. Attendez que la température de la caméra CCD a atteint-25 ° C.
- Mettre 1 mL de la solution ECL pour une des membranes sur une pellicule de plastique sur un banc.
- Mettre un membrane gel-côté-vers le haut sur la solution ECL et puis rapidement Retournez-le afin que les deux côtés de la membrane sont enduites avec la solution.
- Ramasser la membrane et égoutter en laissant un côté de la touche de membrane papier absorbant pendant 5 s.
- Mettre la membrane entre clair films (par exemple, papier clair poches).
NOTE : Le film plastique n’est pas recommandé pour envelopper les membranes. Les films clairs utilisés pour l’emballage de la membrane doivent être aussi plate que possible sans rides visibles afin d’éviter le fond inégal. - Placer la membrane sur un plateau noir. Placez le plateau dans l’imageur et fermer la porte.
- Cliquez sur le bouton « Mise au point » dans la fenêtre du logiciel de contrôle. Vérifier que la membrane est correctement positionnée. Cliquez sur le bouton « Retour ».
- Sélectionnez « Précision » pour « Type d’exposition ». Sélectionnez « Manuel » pour « Temps d’exposition » et définissez la durée d’exposition à 1 s.
- Sélectionnez « Élevé » pour « Résolution/sensibilité ». « Ajouter des images de numérisation » laissez désactivé. Cliquez sur le bouton « Start » pour prendre la photo.
- Enregistrez l’image qui apparaît sur l’écran de l’ordinateur au format TIFF.
- Répéter de prendre des photos avec la durée d’exposition de 1, 3, 10, 30, 60, 90, 120, 150 s et jusqu'à 180 s. Lorsque vous prenez la dernière image, cochez « Ajouter des images de numérisation » afin de l’image numérique, pas de la chimiluminescence, de la membrane peut être pris simultanément.
- Sélectionnez la meilleure image avec la plus grande gamme dynamique et sans n’importe quel pixel saturante (indiqué en rouge) dans les bandes d’intérêt.
NOTE : Films radiographiques classiques peuvent également servir pour détection19.
Résultats
Surexpression système : Phosphorylation des HA-Rab10 de 3 × drapeau-LRRK2 dans HEK293 cellules :
Les cellules HEK293 ont été transfectées avec 0,266 µg d’HA-Rab10 sauvage et 1,066 µg de 3 × drapeau-LRRK2 (mutant sauvage, kinase-inactive (K1906M), ou des mutants FPD). La phosphorylation de Rab10 a été étudiée par P-tag SDS-PAGE, suivi par immunotransfert à l’aide d’un anti-HA anticorps (Figure 2). 10 µg de protéines ont été exécutés sur un gel de 10 % (80 x 100 x 1 mm), contenant 50 µM P-tag acrylamide et 100µm MnCl2. Le temps d’exposition pour le gel P-tag (panneau supérieur) était de 10 s à l’aide d’une solution titrée de ECL. Co-surexpression de LRRK2 avec Rab10 causé Maj de bande sur le gel P-tag (marqué avec un cercle ouvert dans le panneau supérieur ; comparer les pistes 2 et 4). En revanche, la co-expression avec un mutant kinase inactive LRRK2 (K1906M) Impossible de changer la mobilité des Rab10 (cf. voies 4 et 5), ce qui indique que le passage de la bande est due à l’activité de la kinase LRRK2. Le passage de la bande a été augmenté de toutes les mutations de FPD (comparer les voies 4 et 6-13) en accord avec le précédent rapport15.
Système endogène : La Phosphorylation de Rab10 par LRRK2 dans des cellules cultivées :
Cellules de fibroblastes embryonnaires de souris 3 t 3-Swiss albinos ont été traités avec ou sans un LRRK2 inhibiteur (GSK2578215A)20 à une concentration finale de 1 µM pendant 1 h (Figure 3 a). Cellules A549 le carcinome du poumon humain ont été traités avec ou sans un autre LRRK2 inhibiteur (IML-2)21 à une concentration finale de 10 nM pendant 1 h (Figure 3 b). Endogènes Rab10 phosphorylation par LRRK2 endogène a été examinée par la balise P SDS-PAGE, suivi par immunotransfert avec un anticorps anti-Rab10. 30 µg de protéines ont été exécutés sur un gel de 10 % (100 x 100 x 1 mm pour les cellules 3 t 3-Swiss albinos) et 80 mm pour les cellules A549 contenant 50 µM P-tag acrylamide et 100µm MnCl2. L’exposition pour le gel de la P-tag (panneau supérieur) dans les figures 3 a et 3 b Figure étaient 3 min et 90 s, respectivement. Le déplacement de bande correspondant à phosphorylés Rab10 endogène a été observé sur le gel de P-tag (marqué avec un cercle ouvert dans le panneau supérieur ; Comparez voies 1-2 et de 3-4) qui ont disparu après traitement des cellules avec GSK2578215A ou MLi-2. L’efficacité des inhibiteurs LRRK2 a été validée par immunotransfert utilisant les anticorps de LRRK2 anti-pSer935 (le troisième panneau du bas), qui est une lecture bien établie d’inhibition LRRK2 dans cellules22.

Figure 1. Représentation schématique de la balise P SDS-PAGE. Quand un mélange de phosphorylée (cercles rouges) et de protéines non-phosphorylés (cercles bleus) (p. ex., Rab10), qui traverse un gel où le balise P acrylamide est co polymérisé, la mobilité des protéines phosphorylées seuls retarde en raison de la forte interaction des protéines phosphorylées avec les molécules de P-tag (broken arrows). Étant donné que Rab10 est phosphorylé par LRRK2 à un seul résidu Thr73, Rab10 s’exécute sous forme de deux bandes : la bande supérieure représente Rab10 phosphorylés et la bande de fond Rab10 non-phosphorylés. Lorsque les cellules sont traitées avec des inhibiteurs de LRRK2, la bande supérieure devrait disparaître. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
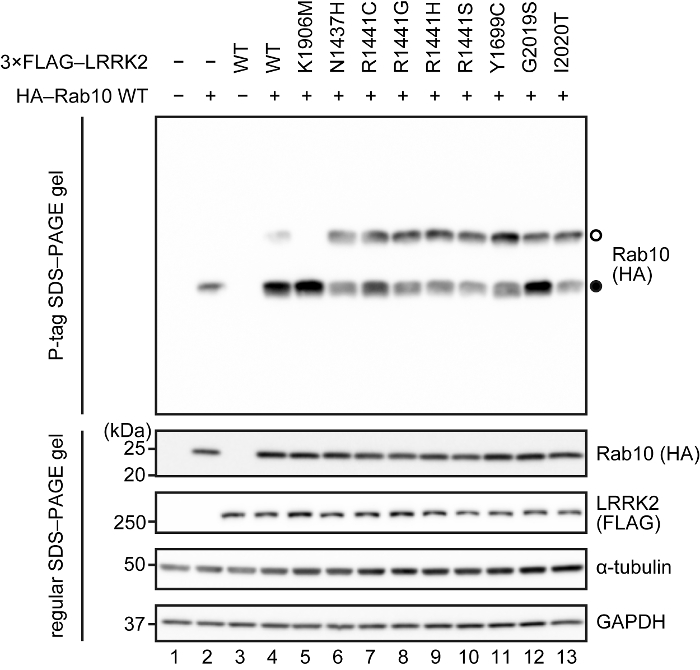
Figure 2. Un résultat représentatif de la tache de P-tag : système de surexpression. Le panneau supérieur montre la tache de P-tag à l’aide d’un anti-HA anticorps, où Rab10 phosphorylées et non-phosphorylés sont marqués avec ouvrir (○) et fermé des cercles (●), respectivement. Les panneaux ci-dessous la tache P-tag sont immuno-Blot sur des gels SDS-PAGE normales à l’aide d’un anti-HA, Anti-Flag, anti-α-tubuline et anticorps anti-GAPDH. La tache HA et tache de drapeau sont indiqués pour assurer la surexpression de HA-Rab10 et 3xFLAG-LRRK2, respectivement. Le α-tubuline et GAPDH taches apparaissent pour assurer le chargement égal. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 3. Un résultat représentatif de la tache de P-tag : système endogène. Deux lignées de cellules, à savoir les cellules 3 t 3-Swiss albinos (A) et les cellules A549 (B), ont été utilisés. Dans les deux chiffres, les panneaux supérieurs montrent que la balise P-taches à l’aide d’un anticorps anti-Rab10, où Rab10 endogène phosphorylées et non-phosphorylés sont marqués avec open (○) et fermé des cercles (●), respectivement. Les panneaux ci-dessous les taches P-tag sont immuno-Blot sur des gels SDS-PAGE normales à l’aide d’un anti-Rab10 anti-pSer935 LRRK2, anti-LRRK2 et anticorps anti-α-tubuline. Le Rab10 et la LRRK2 sont fournis pour assurer l’expression similaire d’endogènes Rab10 et LRRK2 entre les échantillons, respectivement. La tache de LRRK2 pSer935 sont indiqués pour veiller à ce que GSK2578215A (A) et MLi-2 (B) a fonctionné comme prévu. La tache de tubuline α sont indiquées pour assurer le chargement égal. Les images des taches P-tag superposées avec le marqueur de poids moléculaire sont indiquées en Figure S4. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Figure S1. Des séquences d’ADN des plasmides. Les séquences d’ADN du plasmide codant HA-Rab10/pcDNA5 FRT à et celle de 3 × × drapeau-LRRK2/p3 drapeau-CMV-10. Tous les plasmides utilisés dans le présent protocole sont disponibles auprès des auteurs sur demande. S’il vous plaît cliquez ici pour télécharger ce fichier.
Figure S2. Un exemple de l’optimisation de la balise P SDS-PAGE. Le même ensemble d’échantillons que celle utilisée dans la Figure 3 a a été utilisé pour l’optimisation de la balise P SDS-PAGE. Quatre conditions différentes ont été testées comme indiqué sur la figure. Les bandes correspondant à Rab10 phosphorylées et non-phosphorylés sont mis en évidence des rectangles de ligne solide et en pointillés, respectivement.Après cette expérience, la condition avec 10 % d’acrylamide, acrylamide de balise P 50 µM et 100 µM MnCl2 a donné la meilleure séparation des deux bandes, les deux localisation au milieu du gel. S’il vous plaît cliquez ici pour télécharger ce fichier.
Figure S3. Comparaison entre le modèle de migration d’un marqueur de poids moléculaire. Le marqueur de poids moléculaire (MWM) a été exécuté sur un gel de SDS-PAGE régulier (panneau de gauche) ou sur un gel SDS-PAGE de P-tag (panneau central), et les gels ont été colorées par coloration au bleu de Coomassie. Le panneau de droite montre un immunoblot des échantillons utilisés dans la Figure 2 (voies 4 et 5) sur le même gel P-tag SDS-PAGE, comme le panneau central pour montrer les positions des Rab10 phosphorylées et non-phosphorylés. La pointe de la flèche sur le côté droit de l’immunoblot indique la position de l’un du MWM sur gel SDS-PAGE de P-tag. S’il vous plaît cliquez ici pour télécharger ce fichier.
Figure S4. La position d’un marqueur de poids moléculaire sur des gels de P-tag SDS-PAGE. Les images numérisées des membranes utilisées pour les figures 3 a et 3 b de la Figure sont affichés sous forme de (A) et(B), respectivement. L’immuno-Blot, illustré à la Figure 3 est superposées pour afficher la position relative du marqueur de poids moléculaire (MWM). Le MWM n’a cédé bien aux membranes mais le marqueur (pointe de flèche) dans la Figure S3 a été faiblement mais régulièrement observé. La position de la MWM est indiquée en pointillés. Notez que les voies sans rapport avec les chiffres entre le MWM et les voies d’intérêt sont barrés. S’il vous plaît cliquez ici pour télécharger ce fichier.
Discussion
Nous décrivons ici une méthode facile et robuste de détecter Rab10 phosphorylation par LRRK2 niveaux endogènes basé sur la méthodologie P-tag. Parce que l’anticorps actuellement disponibles contre phosphorylés Rab10 fonctionne uniquement avec des protéines surexprimée15, la présente méthode utilisant la balise P SDS-PAGE est la seule façon d’évaluer les concentrations endogènes de la phosphorylation de Rab10. En outre, la présente méthode permet l’estimation de la stoechiométrie de la phosphorylation de Rab10 dans les cellules. Parce que la méthodologie P-tag est génériquement applicable aux phospho-protéines, le présent protocole peut être un « prototype » pour l’établissement des méthodes similaires pour les autres phospho-protéines.
Des étapes cruciales dans le protocole sont des gels de coulée et préparation des échantillons. P-balise l’acrylamide est un réactif photo relativement labiles et la capacité de retarder la mobilité électrophorétique des protéines phosphorylées est parfois perdue après un stockage prolongé à 4 ° C. Les chercheurs devraient prendre grand soin pour éviter d’exposer l’acrylamide P-tag à la lumière. En outre, la molécule de P-tag a besoin former un complexe avec les ions Mn2 + pour capturer des phosphates. Ainsi, les échantillons ne doivent pas contenir des agents chélateurs comme l’EDTA, qui sont des composants habituels des tampons d’échantillons SDS-PAGE disponibles dans le commerce. Nous recommandons d’utiliser le tampon de Laemmli le classique pour préparer les échantillons pour P-tag SDS-PAGE.
Un autre point critique est d’optimiser soigneusement les concentrations d’acrylamide, P-tag acrylamide et MnCl2 dans le mélange de gel de séparation (Figure S2). Les distances de migration des bandes phosphorylées et non-phosphorylés variera selon les réactifs utilisés (lot, pureté, etc.), et l’optimisation des concentrations en testant plusieurs différentes concentrations en combinaison appropriées est obligatoire (par exemple, P-tag acrylamide (25, 50, 75 µM), MnCl2 (1:2 ou 1 / 3 rapport molaire à l’acrylamide P-tag) et l’acrylamide (7.5, 10, 12,5 %)). Les bandes phosphorylées et non-phosphorylés doivent apparaître au milieu les gels et bien séparent les uns des autres. Pour les processus d’optimisation, il est recommandé d’utiliser Rab10 surexprimée parce que la bande décalée est facilement détectable. Les auteurs peuvent fournir des échantillons de contrôle pour ce protocole.
Un de la plus grande confusion que ce protocole peut provoquer sera due à la différence des schémas migratoires de la MWM entre ordinaire et gels de P-tag SDS-PAGE. Comme illustré à la Figure S3, les schémas de migration de la MWM régulière et gels (10 %) de la balise P SDS-PAGE diffèrent grandement, et on ne peut pas utiliser le MWM pour estimer le poids moléculaire des protéines sur gel SDS-PAGE de P-tag. Toutefois, sur les gels de P-tag SDS-PAGE, d'entre le MWM se démarque en migrant vers le milieu du gel (pointe de flèche en Figure S3), qui est régulièrement observé parmi les gels (voir Figure S4). Cette MWM toujours migre entre les bandes de Rab10 phosphorylées et non-phosphorylés. Par conséquent, ce marqueur est utile non seulement pour s’assurer que la balise P SDS-PAGE fonctionne avant le transfert, mais aussi comme un point de repère pour avoir un sens approximatif d’où les bandes de Rab10 phosphorylées et non-phosphorylés verra.
Pour la détection des bandes par immunoblot, nous avons utilisé un imageur équipé d’une caméra CCD refroidie (voir Table des matières) ainsi comme une radiographie conventionnelle film système en développement et constaté que les deux systèmes fonctionnent bien. Pour la détection des niveaux endogènes de la phosphorylation de Rab10 dans les cellules cultivées, il est nécessaire d’utiliser des lignées de cellules qui ont des niveaux d’expression élevée de LRRK2 endogènes, tels que les fibroblastes embryonnaires de souris (soit primaire cultivés ou immortalisé cellule (lignes Albino 3 t 3-suisses, etc.)) et les cellules A549 dérivé de carcinome du poumon humain. Pour la détection des niveaux endogènes de la phosphorylation de Rab10 dans les tissus de souris, il est recommandé d’utiliser du poumon16.
La quantification de la phosphorylation de Rab10 n’est pas supérieure à celle d’immunotransfert habituel. Si la stoechiométrie de la phosphorylation de Rab10 est très faible (comme illustré à la Figure 3), l’intensité de la bande non-phosphorylés (marquée d’un cercle fermé) tend à être loin au-dessus du niveau de saturation, rendant la détermination précise de la stoechiométrie impossible. Néanmoins, estimation qualitative est encore possible dans tous les cas, qui n’est pas possible sans l’aide de la méthode actuelle. Étant donné que la détection de la phosphorylation de Rab10 endogène exige une exposition prolongée, il tend à être certaines bandes non spécifiques figurant sur l’étiquette P blot même si les anticorps anti-Rab10 utilisé dans le présent protocole donne des immunoblots assez propres pour une utilisation normale . Pour distinguer les protéines phosphorylées de bandes non spécifiques, il est essentiel d’utiliser un contrôle inhibiteur-traitement, où bandes phosphorylées, mais pas des bandes, devraient disparaître. LRRK2 phosphoryle Rab8 outre Rab1015, et nous avons appliqué avec succès le présent protocole à Rab8A ainsi (données non présentées). Le présent protocole est potentiellement utilisable pour étudier la phosphorylation des protéines, bien qu’il pourrait y avoir des difficultés techniques si la protéine est grande (> 100 kDa) ou multiplier phosphorylés. Parce que Rab10 est une petite protéine (25 kDa) et individuellement phosphorylés à Thr73 par LRRK2, le résultat de la balise P SDS-PAGE est simple lorsqu’on observe qu’une seule bande décalée.
Dans un proche avenir, il sera essentiel d’établir la méthode pour détecter quantitativement l’activité kinasique de LRRK2 dans des échantillons de patient dérivé d’une manière de haut débit afin d’évaluer l’altération de l’activité de la kinase de LRRK2 dans les patients Parkinsoniens, ainsi que d’évaluer l’effet des drogues sur LRRK2 dans les essais cliniques. Depuis le sang périphérique humain cellules mononucléaires (PBMC) expriment des niveaux relativement élevés d’endogène LRRK223, PBMC ou encore isolé des globules seront d’une valeur de test pour la phosphorylation de Rab10 endogène. La présente méthode va non seulement être utile dans l’étude de la biologie fondamentale de la LRRK2, voie de signalisation, mais également de l’aide à obtenir une base de validation pour décider quel échantillon dans les échantillons de patient dérivé doit être analysé dans ces études à grande échelle.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions le Dr Takeshi Iwatsuboa (Université de Tokyo, Japon) aimablement les plasmides codant pour 3xFLAG-LRRK2 WT et mutants. Nous remercions également m. Dario Alessi (Université de Dundee, Royaume-Uni) aimablement IML-2 et le plasmide codant HA-Rab10. Ce travail a été soutenu par la société japonaise pour la Promotion of Science (JSPS) KAKENHI Grant nombre JP17K08265 (G.I.).
matériels
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Dulbecco's phosphate-buffered saline (DPBS) | homemade | 150 mM NaCl, 8 mM Na2HPO4-12H2O, 2.7 mM KCl, 1.5 mM KH2PO4 in MilliQ water and sterilized by autoclaving | |
| Sodium chloride | Nacalai Tesque | 31320-34 | |
| Sodium Disodium Hydrogenphosphate 12-Water | Wako | 196-02835 | |
| Potassium chloride | Wako | 163-03545 | |
| Potassium Dihydrogen Phosphate | Wako | 169-04245 | |
| 2.5% Trypsin (10X) | Sigma-Aldrich | T4549 | Dilute 10-fold with sterile DPBS for preparing working solution |
| Dulbecco's modified Eagle medium (DMEM) | Wako | 044-29765 | |
| Fetal bovine serum | BioWest | S1560 | Heat-inactivated at 56 °C for 30 min |
| Penicillin-Streptomycin (100X) | Wako | 168-23191 | |
| HEPES | Wako | 342-01375 | |
| Sodium hydroxide | Wako | 198-13765 | |
| Polyethylenimine HCl MAX, Linear, Mw 40,000 (PEI MAX 40000) | PolySciences, Inc. | 24765-1 | Stock solution was prepared in 20 mM HEPES-NaOH pH 7.0 at 1 mg/mL and the pH was then adjusted to 7.0 with NaOH |
| Dimethyl sulfoxide | Wako | 045-28335 | |
| Tris | STAR | RSP-THA500G | |
| Hydrochloric acid | Wako | 080-01066 | |
| Polyoxyethylene(10) Octylphenyl Ether | Wako | 160-24751 | Equivalent to Triton X-100 |
| Ethylene glycol-bis(2-aminoethylether)-N,N,N’,N’-tetraacetic acid (EGTA) | Wako | 346-01312 | |
| Sodium orthovanadate(V) | Wako | 198-09752 | |
| Sodium fluoride | Kanto Chemical | 37174-20 | |
| β-Glycerophosphoric Acid Disodium Salt Pentahydrate | Nacalai Tesque | 17103-82 | |
| Sodium pyrophosphate decahydrate | Kokusan Chemical | 2113899 | |
| Microcystin-LR | Wako | 136-12241 | |
| Sucrose | Wako | 196-00015 | |
| Complete EDTA-free protease inhibitor cocktail | Roche | 11873580001 | Dissolve one tablet in 1 mL water, which can be stored at -20 °C for a month. Use it at 1:50 dilution for cell lysis |
| Pierce Coomassie (Bradford) Protein Assay Kit | Thermo Fisher Scientific | 23200 | |
| Sodium dodecyl sulfate | Nacalai Tesque | 31607-65 | |
| Glycerol | Wako | 075-00616 | |
| Bromophenol blue | Wako | 021-02911 | |
| β-mercaptoethanol | Kanto Chemical | 25099-00 | |
| Ethanol | Wako | 056-06967 | |
| Methanol | Wako | 136-01837 | |
| Phosphate-binding tag acrylamide | Wako | AAL-107 | P-tag acrylamide |
| 40% (w/v) acrylamide solution | Nacalai Tesque | 06119-45 | Acrylamide:Bis = 29:1 |
| Tetramethylethylenediamine (TEMED) | Nacalai Tesque | 33401-72 | |
| Ammonium persulfate (APS) | Wako | 016-08021 | 10% (w/v) solution was prepared by dissolving the powder of ammonium persulfate in MilliQ water |
| 2-propanol | Wako | 166-04831 | |
| Manganese chloride tetrahydrate | Sigma-Aldrich | M3634 | |
| Precision Plus Protein Prestained Standard | Bio-Rad | 1610374, 1610373, 1610377 | Molecular weight marker used in the protocol |
| WIDE-VIEW Prestained Protein Size Marker III | Wako | 230-02461 | |
| Glycine | Nacalai Tesque | 17109-64 | |
| Amersham Protran NC 0.45 | GE Healthcare | 10600007 | Nitrocellulose membrane |
| Durapore Membrane Filter | EMD Millipore | GVHP00010 | PVDF membrane |
| Filter Papers No.1 | Advantec | 00013600 | |
| Ponceau S | Nacalai Tesque | 28322-72 | |
| Acetic acid | Wako | 017-00251 | |
| Tween-20 | Sigma-Aldrich | P1379 | polyoxyethylenesorbitan monolaurate |
| Ethylenediaminetetraacetic acid (EDTA) | Wako | 345-01865 | |
| Skim milk powder | Difco Laboratories | 232100 | |
| Immunostar | Wako | 291-55203 | ECL solution (Normal sensitivity) |
| Immunostar LD | Wako | 290-69904 | ECL solution (High sensitivity) |
| CBB staining solution | homemade | 1 g CBB R-250, 50% (v/v) methanol, 10% (v/v) acetic acid in 1 L of MilliQ water | |
| CBB R-250 | Wako | 031-17922 | |
| CBB destaining solution | homemade | 12% (v/v) methanol, 7% (v/v) acetic acid in 1 L MilliQ water | |
| Name | Company | Catalog Number | Comments |
| Antibodies | |||
| anti-HA antibody | Sigma-Aldrich | 11583816001 | Used at 0.2 μg/mL for immunoblotting. |
| anti-Rab10 antibody | Cell Signaling Technology | #8127 | Used at 1:1000 for immunoblotting. Specificity was confirmed by CRISPR KO in Ito et al., Biochem J, 2016. |
| anti-pSer935 antibody | Abcam | ab133450 | Used at 1 μg/mL for immunoblotting. |
| anti-LRRK2 antibody | Abcam | ab133518 | Used at 1 μg/mL for immunoblotting. |
| anti-α-tubulin antibody | Sigma-Aldrich | T9026 | Used at 1 μg/mL for immunoblotting. |
| anti-GAPDH antibody | Santa-Cruz | sc-32233 | Used at 0.02 μg/mL for immunoblotting. |
| Peroxidase AffiniPure Sheep Anti-Mouse IgG (H+L) | Jackson ImmunoResearch | 515-035-003 | Used at 0.16 μg/mL for immunoblotting. |
| Peroxidase AffiniPure Goat Anti-Rabbit IgG (H+L) | Jackson ImmunoResearch | 111-035-003 | Used at 0.16 μg/mL for immunoblotting. |
| Name | Company | Catalog Number | Comments |
| Inhibitors | |||
| GSK2578215A | MedChem Express | HY-13237 | Stock solution was prepared in DMSO at 10 mM and stored at -80 °C |
| MLi-2 | Provided by Dr Dario Alessi (University of Dundee) | Stock solution was prepared in DMSO at 10 mM and stored at -80 °C | |
| Name | Company | Catalog Number | Comments |
| Plasmids | |||
| Rab10/pcDNA5 FRT TO HA | Provided by Dr Dario Alessi (University of Dundee) | This plasmid expresses amino-terminally HA-tagged human Rab10. | |
| LRRK2 WT/p3xFLAG-CMV-10 | Provided by Dr Takeshi Iwatsubo (University of Tokyo) | Ito et al., Biochemistry, 46: 1380–1388 (2007). This plasmid expresses amino-terminally 3xFLAG-tagged wild-type human LRRK2. | |
| LRRK2 K1906M/p3xFLAG-CMV-10 | Provided by Dr Takeshi Iwatsubo (University of Tokyo) | Ito et al., Biochemistry, 46: 1380–1388 (2007). This plasmid expresses amino-terminally 3xFLAG-tagged K1906M kinase-inactive mutant of human LRRK2. | |
| LRRK2 N1437H/p3xFLAG-CMV-10 | This paper. This plasmid expresses amino-terminally 3xFLAG-tagged N1437H FPD mutant of human LRRK2. | ||
| LRRK2 R1441C/p3xFLAG-CMV-10 | Provided by Dr Takeshi Iwatsubo (University of Tokyo) | Kamikawaji et al., Biochemistry, 48: 10963–10975 (2013). This plasmid expresses amino-terminally 3xFLAG-tagged R1441C FPD mutant of human LRRK2. | |
| LRRK2 R1441G/p3xFLAG-CMV-10 | Provided by Dr Takeshi Iwatsubo (University of Tokyo) | Kamikawaji et al., Biochemistry, 48: 10963–10975 (2013). This plasmid expresses amino-terminally 3xFLAG-tagged R1441G FPD mutant of human LRRK2. | |
| LRRK2 R1441H/p3xFLAG-CMV-10 | Provided by Dr Takeshi Iwatsubo (University of Tokyo) | Kamikawaji et al., Biochemistry, 48: 10963–10975 (2013). This plasmid expresses amino-terminally 3xFLAG-tagged R1441H FPD mutant of human LRRK2. | |
| LRRK2 R1441S/p3xFLAG-CMV-10 | This paper. This plasmid expresses amino-terminally 3xFLAG-tagged R1441S FPD mutant of human LRRK2. | ||
| LRRK2 Y1699C/p3xFLAG-CMV-10 | Provided by Dr Takeshi Iwatsubo (University of Tokyo) | Kamikawaji et al., Biochemistry, 48: 10963–10975 (2013). This plasmid expresses amino-terminally 3xFLAG-tagged Y1699C FPD mutant of human LRRK2. | |
| LRRK2 G2019S/p3xFLAG-CMV-10 | Provided by Dr Takeshi Iwatsubo (University of Tokyo) | Kamikawaji et al., Biochemistry, 48: 10963–10975 (2013). This plasmid expresses amino-terminally 3xFLAG-tagged G2019S FPD mutant of human LRRK2. | |
| LRRK2 I2020T/p3xFLAG-CMV-10 | Provided by Dr Takeshi Iwatsubo (University of Tokyo) | Kamikawaji et al., Biochemistry, 48: 10963–10975 (2013). This plasmid expresses amino-terminally 3xFLAG-tagged I2020T FPD mutant of human LRRK2. | |
| Name | Company | Catalog Number | Comments |
| Equipments | |||
| CO2 incubator | Thermo Fisher Scientific | Forma Series II 3110 Water-Jacketed | |
| Auto Pipette | Drummond | Pipet-Aid PA-400 | |
| Micropipette P10 | Nichiryo | 00-NPX2-10 | 0.5–10 μL |
| Micropipette P200 | Nichiryo | 00-NPX2-200 | 20–200 μL |
| Micropipette P1000 | Nichiryo | 00-NPX2-1000 | 100–1000 μL |
| Tips for micropipette P10 | STAR | RST-481LCRST | Sterile |
| Tips for micropipette P200 | FUKAEKASEI | 1201-705YS | Sterile |
| Tips for micropipette P1000 | STAR | RST-4810BRST | Sterile |
| 5 mL disporsable pipette | Greiner | 606180 | Sterile |
| 10 mL disporsable pipette | Greiner | 607180 | Sterile |
| 25 mL disporsable pipette | Falcon | 357535 | Sterile |
| Hematocytometer | Sunlead Glass | A126 | Improved Neubeuer |
| Microscope | Olympus | CKX53 | |
| 10 cm dishes | Falcon | 353003 | For tissue culture |
| 6-well plates | AGC Techno Glass | 3810-006 | For tissue culture |
| Vortex mixer | Scientific Industries | Vortex-Genie 2 | |
| Cell scrapers | Sumitomo Bakelite | MS-93100 | |
| 1.5 mL tubes | STAR | RSV-MTT1.5 | |
| 15 mL tubes | AGC Techno Glass | 2323-015 | |
| 50 mL tubes | AGC Techno Glass | 2343-050 | |
| Centrifuges | TOMY | MX-307 | |
| 96-well plates | Greiner | 655061 | Not for tissue culture |
| Plate reader | Molecular Devices | SpectraMax M2e | |
| SDS–PAGE tanks | Nihon Eido | NA-1010 | |
| Transfer tanks | Nihon Eido | NA-1510B | |
| Gel plates (notched) | Nihon Eido | NA-1000-1 | |
| Gel plates (plain) | Nihon Eido | NA-1000-2 | |
| Silicon spacers | Nihon Eido | NA-1000-16 | |
| 17-well combs | Nihon Eido | Custom made | |
| Binder clips | Nihon Eido | NA-1000-15 | |
| 5 mL syringe | Terumo | SS-05SZ | |
| 21G | Terumo | NN-2138R | |
| Power Station 1000 VC | ATTO | AE-8450 | Power supply for SDS–PAGE and transfer |
| Large weighing boats | Ina Optika | AS-DL | |
| Plastic containers | AS ONE | PS CASE No.4 | 10 x 80 x 50 mm |
| Rocking shaker | Titech | NR-10 | |
| Styrene foam box | generic | The internal dimensions should fit one transfer tank (200 x 250 x 250 mm). | |
| ImageQuant LAS-4000 | GE Healthcare | An imager equipped with a cooled CCD camera for detection of ECL |
Références
- Sveinbjornsdottir, S. The clinical symptoms of Parkinson's disease. J. Neurochem. 139 (Suppl. 1), 318-324 (2016).
- Hernandez, D. G., Reed, X., Singleton, A. B. Genetics in Parkinson disease: Mendelian versus non-Mendelian inheritance. J. Neurochem. 139 (Suppl. 1), 59-74 (2016).
- Paisán-Ruíz, C., et al. Cloning of the gene containing mutations that cause PARK8-linked Parkinson's disease. Neuron. 44 (4), 595-600 (2004).
- Zimprich, A., et al. Mutations in LRRK2 cause autosomal-dominant parkinsonism with pleomorphic pathology. Neuron. 44 (4), 601-607 (2004).
- Gilks, W. P., et al. A common LRRK2 mutation in idiopathic Parkinson's disease. Lancet. 365 (9457), 415-416 (2005).
- Satake, W., et al. Genome-wide association study identifies common variants at four loci as genetic risk factors for Parkinson's disease. Nat. Genet. 41 (12), 1303-1307 (2009).
- Simón-Sánchez, J., et al. Genome-wide association study reveals genetic risk underlying Parkinson's disease. Nat. Genet. 41 (12), 1308-1312 (2009).
- Klein, C., Ziegler, A. Imputation of sequence variants for identification of genetic risks for Parkinson's disease: a meta-analysis of genome-wide association studies. Lancet. 377 (9766), 641-649 (2011).
- Cookson, M. R. The role of leucine-rich repeat kinase 2 (LRRK2) in Parkinson's disease. Nat. Rev. Neurosci. 11 (12), 791-797 (2010).
- Ozelius, L. J., et al. LRRK2 G2019S as a Cause of Parkinson’s Disease in Ashkenazi Jews. N. Engl. J. Med. 354 (4), 424-425 (2006).
- Lesage, S., et al. LRRK2 G2019S as a Cause of Parkinson’s Disease in North African Arabs. N. Engl. J. Med. 354 (4), 422-423 (2006).
- Bouhouche, A., et al. LRRK2 G2019S Mutation: Prevalence and Clinical Features in Moroccans with Parkinson's Disease. Parkinsons. Dis. , 1-7 (2017).
- West, A. B., et al. Parkinson's disease-associated mutations in leucine-rich repeat kinase 2 augment kinase activity. Proc. Natl. Acad. Sci. U. S. A. 102 (46), 16842-16847 (2005).
- Ito, G., et al. GTP binding is essential to the protein kinase activity of LRRK2, a causative gene product for familial Parkinson's disease. Biochemistry. 46 (5), 1380-1388 (2007).
- Steger, M., et al. Phosphoproteomics reveals that Parkinson's disease kinase LRRK2 regulates a subset of Rab GTPases. Elife. 5, (2016).
- Ito, G., et al. Phos-tag analysis of Rab10 phosphorylation by LRRK2: a powerful assay for assessing kinase function and inhibitors. Biochem. J. 473, 2671-2685 (2016).
- Kinoshita, E., Kinoshita-Kikuta, E., Takiyama, K., Koike, T. Phosphate-binding tag, a new tool to visualize phosphorylated proteins. Mol. Cell. Proteomics. 5 (4), 749-757 (2006).
- Database, J. S. E. Using a Hemacytometer to Count Cells. J. Vis. Exp. , Available from: https://www-jove-com.remotexs.ntu.edu.sg/science-education/5048/using-a-hemacytometer-to-count-cells (2017).
- Ni, D., Xu, P., Gallagher, S. Immunoblotting and Immunodetection. Curr. Protoc. Mol. Biol. (114), 10.8.1-10.8.37 (2016).
- Reith, A. D., et al. GSK2578215A; a potent and highly selective 2-arylmethyloxy-5-substitutent-N-arylbenzamide LRRK2 kinase inhibitor. Bioorg. Med. Chem. Lett. 22 (17), 5625-5629 (2012).
- Fell, M. J., et al. MLi-2, a potent, selective and centrally active compound for exploring the therapeutic potential and safety of LRRK2 kinase inhibition. J. Pharmacol. Exp. Ther. 355, 397-409 (2015).
- Dzamko, N., et al. Inhibition of LRRK2 kinase activity leads to dephosphorylation of Ser(910)/Ser(935), disruption of 14-3-3 binding and altered cytoplasmic localization. Biochem. J. 430 (3), 405-413 (2010).
- Thévenet, J., Pescini Gobert, R., Hooft van Huijsduijnen , R., Wiessner, C., Sagot, Y. J. Regulation of LRRK2 expression points to a functional role in human monocyte maturation. PLoS One. 6 (6), e21519(2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon