Method Article
Rab10 Phosphorylierung Erkennung von LRRK2 Aktivität mit SDS-PAGE mit einem Phosphat-Bindung
In diesem Artikel
Zusammenfassung
Die vorliegende Studie beschreibt eine einfache Methode zur Erkennung von endogenen Ebenen der Rab10 Phosphorylierung von Leucin-reiche wiederholen Kinase 2.
Zusammenfassung
Mutationen im Leucin-reiche wiederholen Kinase 2 (LRRK2) haben gezeigt, mit familiären Parkinson-Erkrankung (FPD) verknüpft werden. Da abnormale Aktivierung der Kinase-Aktivität von LRRK2 in der Pathogenese von Morbus Parkinson in Verbindung gebracht hat, ist es wichtig, eine Methode zur Evaluierung der physiologischen Ebenen die Kinase-Aktivität von LRRK2 zu etablieren. Neuere Studien gezeigt, dass LRRK2 Rab GTPase Familienmitglieder, einschließlich Rab10, unter physiologischen Bedingungen phosphorylates. Obwohl die Phosphorylierung des endogenen Rab10 von LRRK2 in kultivierten Zellen durch Massenspektrometrie nachgewiesen werden konnte, ist es schwierig zu erkennen durch Immunoblotting aufgrund der schlechten Empfindlichkeit der derzeit verfügbaren Phosphorylierung-spezifische Antikörper für gewesen. Rab10. Hier beschreiben wir eine einfache Methode zum Nachweis der endogenen Ebenen der Rab10 Phosphorylierung von LRRK2 basierend auf Immunoblotting Verwendung von Sodium Dodecyl Sulfat Polyacrylamid Gelelektrophorese (SDS-PAGE) kombiniert mit einem Phosphat-Bindung-Tag (P-Tag), die ist N-(5-(2-aminoethylcarbamoyl)pyridin-2-ylmetyl) -N,N',N'- Tris (Pyridin-2-Yl-Methyl) - 1,3 - Diaminopropan-2-Ol. Dieses Protokoll nicht nur liefert ein Beispiel für die Methode, die Nutzung im P-Tags, sondern ermöglicht auch die Bewertung von Mutationen sowie Inhibitor Behandlung/Verwaltung oder andere Faktoren, wie alter der nachgeschalteten Signalisierung von LRRK2 in Zellen und Geweben .
Einleitung
PD gehört zu den häufigsten neurodegenerativen Erkrankungen, überwiegend dopaminergen Neuronen im Mittelhirn, beeinflussen, was zu Funktionsstörungen des motorischen Systems in Senioren1. Während die meisten Patienten PD auf sporadische Weise entwickeln, gibt es Familien, die Krankheit zu erben. Mutationen in verschiedenen Genen wurden gefunden, mit FPD2verknüpft werden. Die verursachenden Gene für FPD gehört LRRK2, in denen acht Missense-Mutationen (N1437H, R1441C/G/H/S, Y1699C, G2019S und I2020T) verbunden mit einer dominant vererbten FPD genannt PARK8 bisher gemeldeten3,4,5gewesen sein. Mehreren genomweiten Assoziationsstudien (GWAS) sporadische PD-Patienten haben auch genomische Variationen bei den LRRK2-Locus als Risikofaktor für PD, darauf hindeutet, dass die Anomalie in der Funktion von LRRK2 ist eine häufige Ursache für Neurodegeneration in beiden sporadisch identifiziert und PARK8 FPD6,7,8.
LRRK2 ist ein großes Protein (2.527 Aminosäuren) bestehend aus einer Leucin-reiche wiederholen Sie Domäne, ein GTP-Bindung Ras komplexe Proteine (ROC) Domäne, eine C-terminale ROC (ADR) Domäne, eine Serin/Threonin Protein Kinase-Domäne und einer WD40 wiederholen Domäne9. Die acht FPD-Mutationen finden in diesen funktionellen Domänen; N1437H und R1441C/G/H/S in der ROC-Domäne, Y1699C im Bereich ADR, G2019S und I2020T in der Kinase-Domäne. Da G2019S Mutation, die am häufigsten in PD Patienten10,11,12Mutation gefunden wird, die Kinase-Aktivität von LRRK2 2-3 Fach in-vitro-13erhöht, wird es vermutet. dass die abnorme Zunahme der Phosphorylierung von LRRK2 Substrate giftig für Neuronen. Es war jedoch unmöglich zu prüfen, ob die Phosphorylierung von physiologisch relevanten LRRK2 Substraten in familiären/sporadische PD-Patienten aufgrund der fehlenden Methoden in abgeleiteten Patientenstichproben Bewertung geändert wird.
Protein-Phosphorylierung wird in der Regel durch Immunoblotting oder Enzym-linked Immunosorbentprobe Assay (ELISA) mit Antikörpern, die spezifisch erkennen des phosphorylierten Zustands Proteine oder durch massenspektrometrische Analyse erkannt. Die ehemalige Strategie kann jedoch manchmal wegen der Schwierigkeiten bei der Schaffung von Phosphorylierung-spezifische Antikörper angewendet werden. Metabolische Kennzeichnung von Zellen mit radioaktivem Phosphat ist eine weitere Option, physiologische Ebenen der Phosphorylierung zu untersuchen, wenn Phosphorylierung-spezifischen Antikörper nicht leicht verfügbar sind. Aber es erfordert eine große Menge radioaktiver Stoffe und daher beinhaltet einige spezielle Ausrüstung für Strahlenschutz14. Massenspektrometrische Analyse ist empfindlicher gegenüber diesen immunchemische Methoden und populär bei der Analyse von Protein-Phosphorylierung. Jedoch die Probenvorbereitung ist zeitaufwändig und teuer Instrumente sind für die Analyse erforderlich.
Eine Teilmenge der Rab GTPase Familie einschließlich Rab10 und Rab8 wurde kürzlich berichtet, wie das Ergebnis eines umfangreichen Phosphoproteomic Analyse15direkte physiologische Substrate für LRRK2 zugrunde. Wir zeigten dann, dass Rab10 Phosphorylierung von FPD Mutationen im embryonalen Fibroblasten der Maus und in den Lungen von Profi Mäuse16erhöht wurde. In diesem Bericht, wir entschieden uns für eine Sodium Dodecyl Sulfat Polyacrylamid Gelelektrophorese (SDS-PAGE) beschäftigen-basierte Methode, bei der ein P-Tag-Molekül Co polymerisierten in SDS-PAGE Gelen (P-Tag SDS-PAGE) zur Erfassung der endogenen Ebenen der Rab10 Phosphorylierung denn ein hochempfindlicher Antikörper spezifisch für phosphorylierten Rab10 noch fehlte. Wir haben versäumt, die Phosphorylierung des endogenen Rab8 aufgrund der schlechten Selektivität der derzeit verfügbaren Antikörper für insgesamt Rab8 erkennen. Daher entschieden wir uns konzentrieren auf die Rab10 Phosphorylierung. LRRK2 phosphorylates Rab10 bei Thr73 Ortung in der Mitte der Region hoch konservierte "Schalter II". Hohe Erhaltung der Phosphorylierung Websites unter Rab-Proteine möglicherweise einer der Gründe, warum Phosphospecific Antikörper erkennen unterschiedliche Rab-Proteine schwer sind zu machen.
Die Phosphorylierung des Rab8A von LRRK2 hemmt die Bindung von Rabin8, ein Guanin-Nukleotid Austausch Faktor (GEF) die Rab8A aktiviert, durch den Austausch der gebundenen BIP mit GTP15. Phosphorylierung von Rab10 und Rab8A von LRRK2 hemmt auch die Bindung von BIP-Dissoziation-Inhibitoren (GDIs), die wesentlich für die Aktivierung des Rab-Proteine durch die Gewinnung von BIP-gebundenen Rab-Proteine von Membranen15. Gemeinsam wird es vermutet, dass die Phosphorylierung des Rab-Proteine von LRRK2 sie Aktivierung daran hindert, obwohl die genauen molekularen Mechanismus und physiologischen Folgen der Phosphorylierung unklar bleiben.
P-Tag SDS-PAGE wurde erfunden von Kinoshita Et Al. 2006: bei dieser Methode wurde Acrylamid kovalent verbunden mit P-Tag, ein Molekül Erfassung Phosphate mit hoher Affinität, die in SDS-PAGE copolymerisiert Gele17. Da die P-Tag-Moleküle in einem SDS-PAGE Gel selektiv elektrophoretische Mobilität der phosphorylierten Proteine verzögern, kann P-Tag SDS-PAGE phosphorylierten Proteine von nicht phosphoryliert zu trennen (Abbildung 1). Wenn das Protein des Interesses auf mehrere Reste phosphoryliert wird, wird eine Leiter Bands differentiell phosphorylierten Formen entsprechend beobachtet werden. Im Falle von Rab10 beobachten wir nur eine verschobene Band darauf hinweist, dass Rab10 nur bei Thr73 phosphoryliert wird. Der große Vorteil der P-Tag SDS-PAGE über Immunoblotting mit Phosphorylierung-spezifischen Antikörpern ist, dass phosphorylierten Rab10 durch Immunoblotting mit Phosphorylierung-unspezifische Antikörper (d.h.erkennen total Rab10) nachgewiesen werden können nach auf Membranen übertragen wird, ist die in der Regel bestimmten, sensiblen und verfügbar aus kommerziellen/akademische Quellen. Ein weiterer Vorteil der Verwendung von P-Tag SDS-PAGE ist eine ungefähre Schätzung der Stöchiometrie der Phosphorylierung, erhalten kann, das ist unmöglich, durch Immunoblotting mit Phosphorylierung-spezifische Antikörper oder durch metabolische Kennzeichnung von Zellen mit radioaktiven Phosphate.
Abgesehen von der Verwendung von preiswerten P-Tag Acrylamid und einige geringfügigen Änderungen mit sich bringen folgt das vorliegende Verfahren zur Erkennung von Rab10 Phosphorylierung von LRRK2 ein allgemeines Protokolls Immunoblotting.Daher sollte es unkompliziert und leicht ausführbare in jedem Labor sein, wo Immunoblotting eine übliche Praxis, mit allen Arten von Proben ist, einschließlich der gereinigten Proteine, Zelle Lysates und Gewebe Homogenates.
Protokoll
(1) Probenvorbereitung für die P-Tag SDS-PAGE
- Entfernen Sie und entsorgen Sie die Medien aus 10 cm Gerichte, in denen Zellen angebaut werden mit einer Absaugung, waschen Sie Zellen mit 5 mL Dulbecco Phosphat-gepufferte Kochsalzlösung (DPBS) durch die erste Zugabe DPBS auf die Seite der Gerichte um nicht zu stören die Zellschicht und manuell rocken Sie die Gerichte zurück und her mehrere Male.
- Entfernen Sie und entsorgen Sie des DPBS mit einer Absaugung fügen Sie 2 mL von 0,25 % (w/V) Trypsin in DPBS verdünnt hinzu und schütteln Sie die Gerichte zur Deckung der Zellschicht. Genommen Sie die Gerichte in eine CO2 -Inkubator (37 ° C, feuchte Luft, 5 % CO2) für 5 min.
- Nach Pipettierung nach oben und unten mit einer Einweg-Pipette freistehende Zellen aufzubrechen, sammeln Sie die Zellsuspension in ein 15 mL Röhrchen und Messen Sie die Zelldichte mit einem Hematocytometer unter einem Mikroskop18.
- Verdünnen Sie die Zellen bis 2,5 × 105 Zellen/mL mit Dulbeccos modifizierten Eagle Medium (DMEM) mit 10 % (V/V) fetalen bovine Serum (FBS), 100 U/mL Penicillin und 100 µg/mL Streptomycin ergänzt. Geben Sie 2 mL (5 × 105 Zellen) von verdünnter Zellsuspension in jedem 6-Well Platten.
- Wachsen Sie die Zellen über Nacht in eine CO2 -Inkubator (37 ° C, feuchte Luft, 5 % CO2).
- Transfektion in HEK293 Zellen
Hinweis: Wenn Phosphorylierung des endogenen Rab10 werden untersucht soll, fahren Sie mit Schritt 1,7. In diesem Protokoll verwendeten Plasmide erhalten Sie von den Autoren auf Anfrage. Siehe Tabelle der Materialien für kurze Informationen und Abbildung S1 für ihre DNA-Sequenzen.- Aliquoten 200 µL DMEM in 1,5 mL Röhrchen.
- Jedes Rohr fügen Sie beide 0,266 µg (Endkonzentration von 1,33 µg/mL) von einem Plasmid Codierung HA-Rab10 und 1.066 µg (Endkonzentration von 5,33 µg/mL) von einem Plasmid Codierung 3 × Flagge LRRK2 von 500 µg/mL Plasmid Aktien hinzu. Dann fügen Sie 4 µL 1 mg/mL Lösung von Polyethyleneimine (gelöst in 20 mM HEPES-NaOH, pH 7,0) und mischen Sie sofort die Lösung durch Vortexen für 5 s.
- Lassen Sie die Rohre für 10 min bei Raumtemperatur stehen und fügen Sie den Inhalt der eine Röhre tropfenweise zu einem gut mit einer Mikropipette. Rocken Sie sanft und manuell die Kultur Platten mehrere Male hin und her, um die Transfektion Mischung gleichmäßig in den Brunnen diffundieren zu lassen.
- Lassen Sie die Zellen, die für ein weiteres 24-36 h in einem CO-2 -Inkubator (37 ° C, feuchte Luft, 5 % CO2) wachsen.
- Wenn Phosphorylierung des endogenen Rab10 werden untersucht soll, behandeln Sie Zellen mit und ohne LRRK2-Inhibitoren für 1 h vor der Lyse der Zellen.
- Bereiten Sie Stammlösungen von LRRK2-Inhibitoren durch Auflösen der Inhibitoren in Dimethyl Sulfoxid (DMSO) bei 10 mM. Wir empfehlen die Verwendung von MLi-2 und GSK2578215A, die sehr spezifisch und potente LRRK2-Inhibitoren sind. Speichern Sie auf lagerlösungen bei-80 ° C.
- Arbeiten Bestände der Inhibitoren durch Verdünnung der Stammlösungen mit DMSO vorzubereiten: für MLi-2, 10 µM und 30 µM für endogene und überexprimieren LRRK2 bzw. vorzubereiten. GSK2578215A Vorbereitung 1 mM und 3 mM für endogene und überexprimieren LRRK2, beziehungsweise.
- Fügen Sie 2 µL der Arbeiten Bestände von MLi-2 oder GSK2578215A in die Mitte eines nun mit einer Mikropipette und schütteln Sie die Platte manuell hin und her mehrere Male, um die Inhibitoren gleichmäßig im ganzen gut diffundieren zu lassen.
- Geben Sie 2 µL von DMSO in ein anders als Negativkontrolle in einer ähnlichen Weise zu den Inhibitor im Schritt 1.8.3.
- Setzen Sie die Platten wieder in den Inkubator und Kultur die Zellen 1 h.
- Lyse der Zellen.
- Die Kultur-Platten auf Eis gelegt. Entfernen und entsorgen der Medien. Waschen Sie die Zellen mit ersten Hinzufügen von 2 mL DPBS auf die Seite der Gerichte zu vermeiden, stören die Zelle Schicht und manuell die Gerichte mehrere Male hin und her schaukeln.
- Entfernen und entsorgen des DPBS und fügen Sie die Zellen 100 µL des Puffers lyse (50 mM Tris-HCl pH 7,5, 1 % (V/V) polyoxyethylene(10) Octylphenyl Äther, 1 mM EGTA (Ethylenglykol-bis(2-aminoethylether)-N, N, N', N'-Tetraacetic Säure), 1 mM Natrium Orthovanadate, 50 mM Natriumfluorid, β-Glycerophosphat 10 mM, 5 mM Natrium Pyrophosphat, 0,1 µg/mL Microcystin-LR, 270 mM Saccharose, Protease-Inhibitor cocktail).
- Kippen Sie die Platten auf dem Eis und kratzen Sie die Zellen mit einem Zelle Schaber, um soviel Zelle lysate wie möglich zu sammeln. Sammeln Sie die Lysates mit einer Mikropipette in 1,5 mL Röhrchen (vorab gekühlt auf Eis).
Achtung: Microcystin-LR kann tödlich wenn verschlucken oder Berührung mit der Haut sein.
- Lassen Sie die Rohre auf Eis für 10 min für komplette Lyse stehen. Klären Sie der Zelle Lysates durch Zentrifugieren (20.000 × g, 10 min bei 4 ° C) und übertragen Sie die Überstände auf neue 1,5 mL Röhrchen vorab auf Eis gekühlt.
- Proteinkonzentration (µg/µL) von den gerodeten Lysates von Bradford-Test zu messen.
- Bereiten Sie Rinderserumalbumin (BSA) Normen (0,2, 0,4, 0,6, 0,8 und 1 mg/mL) durch eine Verdünnung der Stammlösung mit destilliertem Wasser. 20-fache der geräumten Zelle Lysates durch Verdünnen mit destilliertem Wasser.
- 5 µL/Well der BSA Standards setzen, verdünnt Blank (destilliertes Wasser), und jede Zelle lysate in eine 96-Well-Platte in dreifacher Ausfertigung.
- 150 µL/Well des Bradford-Assay Reagenz mit 12-Kanal Mikropipette und lassen Sie die Platte für 5 min bei Raumtemperatur stehen.
- Messung der Extinktion bei 595 nm auf einer Platte Leser und im Vergleich zu BSA Standards.
- SDS-PAGE 100 µL der Proben vorbereiten. Die Konzentration des Proteins der Proben ist 1 µg/µL für überexprimieren HA-Rab10 und 2 µg/µL für endogene Rab10.
- Mit der quantifizierten Konzentrationen von Schritt 1.11.4, berechnen Sie das Volumen (µL) der Zelle Lysates entspricht 100 µg (überexprimieren HA-Rab10) oder 200 µg (endogene Rab10) durch Division der Protein-Menge (100 µg oder 200 µg) durch die Proteinkonzentration von Lysaten (µ g/µL).
- Fügen Sie 25 µL 4 × Laemmli SDS-PAGE Probenpuffer (62,5 mM Tris-HCl, pH 6,8, 8 % (w/V) SDS, 40 % (V/V) Glycerin, 0,02 % (w/V) Bromophenol Blue, 4 % (V/V) β-Mercaptoethanol hinzu), neue 1,5 mL Röhrchen bei Raumtemperatur aufbewahrt.
- Fügen Sie das berechnete Volumen der Zelle Lysates zu jedem Rohr und Mischen von vortexen für 5 s bei Raumtemperatur.
- Das Gesamtvolumen zu 100 µL mit Lyse Puffer und Mischung zu bringen, durch Vortexen für 5 s bei Raumtemperatur.
- Ergänzen Sie mit 10 mM MnCl2 der Chelatbildner zu stillen. Fügen Sie 1 µL von 500 mM MnCl2. Mix von vortexen für 5 s bei Raumtemperatur.
Hinweis: Proben mit MnCl2 sind möglicherweise nicht für normale SDS-PAGE geeignet.
2. Casting Gele für P-Tag SDS-PAGE
Hinweis: Gele sollten am selben Tag wie die Ausführung der Gele erfolgen. Gele können unter Lichtverhältnissen gemacht werden.
- 5 mM P-Tag Acrylamid-Stammlösung durch erste auflösen die 10 mg Pulver/Solid P-Tag Acrylamid komplett mit 100 µL des Methanols vorbereiten und dann bis 3,3 mL indem doppelt destilliertes Wasser bringen.
Hinweis: P-Tag Acrylamid ist lichtempfindlich. Die fertige Lösung ist bis zur Verwendung im Dunkeln bei 4 ° C aufzubewahren. - Saubere schlicht und gekerbte Platten durch 70 % igem Ethanol Spritzen und mit einem Papiertuch abwischen. Montieren Sie die Gel-Platten. Die Abmessungen der in diesem bestimmten Protokoll verwendeten Gel-Platten sind 80 oder 100 mm lang 100 mm breit. Sauberen Silizium Abstandhalter werden zwischen Ebene und gekerbte und montierten Platten mit Binder Clips eingespannt sind.
Hinweis: Jede Art von Gel-Platten, die Arbeiten für den normalen SDS-PAGE verwendet werden kann. - Setzen Sie einen Kamm früher (17-Brunnen Binderücken) in die montierten Gel-Platten und Mark auf die Gelplatte die Position des unteren Rands der Brunnen mit einem wasserfesten Filzstift.
- 10 mL 10 % Acrylamid Gel Mischung vorbereiten (10 % (w/V) Acrylamid (Acrylamide:bis-Acrylamid = 29,1), 375 mM Tris-HCl (pH 8,8), 0,1 % (w/V) SDS) in der 15 mL Tube.
Hinweis: Die optimale Konzentration des Acrylamids variieren je nach verwendeten Reagenzien. Tetramethylethylenediamine (TEMED) und Ammonium Bleichen (APS) sollten nicht an dieser Stelle hinzugefügt werden. - Fügen Sie 100 µL 5 mM P-Tag Acrylamid und 10 µL 1 M MnCl2 Lösungen bei Endkonzentrationen von 50 µM und 100 µM bzw..
Hinweis: Die optimalen Konzentrationen der P-Tag Acrylamid und MnCl2 variieren auch je nach verwendeten Reagenzien. - Fügen Sie 15 µL TEMED, die Gel-Mischung auf eine Endkonzentration von 0,15 % (V/V) und dann 50 µL 10 % (w/V) APS auf eine Endkonzentration von 0,05 % (w/V). Mischen Sie gut durch sanft schütteln das Rohr für 5 s und gießen in den montierten Platten sofort bis zu einer Höhe d. h. 2 mm unterhalb der Position im Schritt 2.3 markiert.
- Sanft layer-2-Propanol auf die Gel-Lösung zu der Spitze der Trennung Gele zu glätten.
- Lassen Sie die Gele für 30 min bei Raumtemperatur stehen. Es ist nicht notwendig, die Gele vor Licht zu schützen.
Hinweis: Es möglicherweise für Gele, legen Sie unter Zellentemperatur länger dauern. Die Gel-Mischung vor dem Hinzufügen von TEMED und APS Entgasung hilft, diesen Schritt zu beschleunigen. Zu diesem Zweck kann die Gel-Mischung in ein 100-200 mL-Erlenmeyerkolben an eine Absaugung angeschlossen vorbereitet werden. Entgasen Sie die Lösung für 10 min. - Entfernen Sie die geschichteten 2-Propanol durch die Aufnahme mit einem Papiertuch.
- Waschen Sie den obersten Raum der Gele durch den Raum auffüllen mit destilliertem Wasser aus einer Waschflasche und entsorgen Sie das Wasser durch das Gießen ab ins Becken. Wiederholen Sie 3-Mal waschen.
- Entfernen Sie Restwasser verbleiben im oberen Bereich der Gele durch die Aufnahme mit einem Papiertuch.
- 3 mL 4 % Acrylamid Gel Mischung vorbereiten (4 % (w/V) Acrylamid (a: Crylamide:bis-Acrylamid = 29,1), 125 mM Tris-HCl (pH 6,8), 0,1 % (w/V) SDS) in der 15 mL Tube.
Hinweis: Keine hinzu P-Tag Acrylamid oder MnCl2 Lösung Stapeln Gel-Mischung. - Fügen Sie 7,5 µL TEMED und 24 µL 10 % (w/V) APS bei endgültigen Konzentrationen von 0,25 % (V/V) und 0,08 % (w/V), beziehungsweise. Mischen Sie gut durch sanft schütteln das Rohr für 5 s und gießen Sie das Gemisch auf die Trennung Gel. Sofort setzen Sie geeignete Kämme (17-Brunnen Plastikbinderücken die bis zu 25 µL der Proben, z. B. beherbergen).
- Lassen Sie die Gele für 30 min bei Raumtemperatur stehen. Es ist nicht notwendig, die Gele vor Licht zu schützen. Nachdem die Stapeln Gele eingerichtet haben, führen Sie sie ohne weitere Lagerung.
(3) SDS-PAGE und Immunoblotting
- Entfernen Sie die Kämme aus der Gele. Dann entfernen Sie das Silizium Abstandhalter und dann Clips die Gele.
- Die gegossenen Gele in Gel-Tanks und befestigen Sie das Gel in den Tank durch Klemmung mit Bindemittel-Clips.
- Gießen Sie den laufenden Puffer (25 mM Tris, 192 mM Glycin, 0,1 % (w/V) SDS) an der Unterseite und Oberseite der Gele. Sauberen Brunnen durch Spülen des laufenden Puffers mit einer 5 mL Spritze und Nadel 21G um Gel Stücke zu entfernen.
- Entfernen Sie Luftblasen aus dem unteren Raum der Gele mit einer gebogenen Nadel befestigt, eine Spritze. Um eine verbogene Nadel machen, manuell beugen einer 21G-Nadel in der Mitte der Nadel, so dass der Winkel zwischen der Spitze und der Basis der Nadel 30-45 wird °.
- Spin-down Ausscheidungen verursacht durch die Zugabe von MnCl2 bei 20.000 × g für 1 min bei Raumtemperatur klare Proben erhalten.
- Laden Sie 10 µg Proteine für die Erkennung der Phosphorylierung von überexprimieren Rab10 und 30 µg Proteinen für endogene Rab10.
Hinweis: Es ist wichtig, gleiches Volumen der Proben in alle Wells zu laden. Leere Gassen sollten mit 1 × Laemmli SDS-PAGE Probenpuffer eingelegt werden. Den Dummy-Proben Proben MnCl2enthalten, die gleiche Konzentration an MnCl2 hinzugefügt. - Die gut geladen mit einem Molekulargewicht Marker (MWM) sollte auch mit 1 × Laemmli SDS-PAGE Probenpuffer ergänzt werden so dass das Volumen der Proben geladen in alle Wells ist.
Hinweis: Auch wenn die Proben MnCl2enthalten, fügen Sie die gleiche Konzentration an MnCl2 der MWM hinzu. Alternativ kann eine EDTA-freie MWM verwendet werden. - Führen Sie Gele bei 50 V zum Stapeln (ca. 30 min.), bis die Farbstoff-Front in der Trennung Gel kreuzt.
- Nachdem die Proben zu stapeln, ändern die Spannung auf 120 V für die Trennung bis Farbstoff vorne den Boden der Gele erreicht (ca. 50 bis 80 min für 80-100 mm lange Gele, beziehungsweise).
Hinweis: Die Migration Muster von der MWM wird voraussichtlich auf normalen SDS-PAGE Gelen anders sein. Es sollte nicht verwendet werden, für die Schätzung des Molekulargewichts der Proteine auf P-Tag SDS-PAGE Gelen aber zur Überprüfung der Reproduzierbarkeit der P-Tag Gele eingesetzt werden. Informationen finden Sie unter Diskussion . - Waschen Sie die Gele um MnCl2 aus der Gele zu entfernen.
- Gießen Sie die Übertragung mit 10 mM EDTA Puffer (48 mM Tris, 39 mM Glycin, 20 % (V/V) Methanol) und 0,05 % (w/V) SDS in einen Behälter (z.B.große Boote wiegen).
Hinweis: Verwenden Sie frische Puffer für jede Wäsche.
- Legen Sie ein Filterpapier (10 x 7 cm) auf einem Schwamm-Pad für die Übertragung. Platzieren Sie das Gel auf dem Filterpapier. Stellen Sie sicher, es gibt keine Luftblasen zwischen dem Filterpapier und das Gel.
- Eine Membran (10 x 7 cm) auf das Gel und stellen sicher, dass keine Luftblasen zwischen dem Gel und der Membran.
- Legen Sie ein weiteres Filterpapier (10 x 7 cm) auf der Membran und wieder, stellen Sie sicher, es gibt keine Luftblasen zwischen der Membran und das Filterpapier.
- Setzen Sie ein anderes Schwamm-Pad auf dem Filterpapier. Legen Sie den Papierstapel Membranfilter/in eine Kassette für den Transfer.
- Legen Sie die Kassette in einen Transfer-Tank, um sicherzustellen, dass die Membran zwischen dem Gel und der positiv geladenen Anode befindet.
- Schließen Sie den Tank an ein Netzteil an und den Tank in ein Styrol Schaum Box mit eiskaltem Wasser gefüllt. Starten Sie Transfer bei 100 V für 180 min.
Hinweis: Längere Transfer ist notwendig, da der Transfer von Proteinen aus P-Tag SDS-PAGE Gelen sind nicht so effizient wie die von normalen SDS-PAGE Gelen. Effiziente Kühlung ist unbedingt zu vermeiden, schmelzen Gele während der Übertragung.
- Entfernen Sie die Membranen aus die Gele mit einer Pinzette und genießen Sie die Membranen in einer Ponceau S-Lösung (0,1 % (w/V) Ponceau S, 5 % (V/V) Essigsäure), übertragenen Proteine auf den Membranen in einem Plastikbehälter zu beflecken. Das Volumen der Lösung sollte ausreichen, um eine Membran zu decken.
- Inkubieren Sie die Membranen für 1 min bei Raumtemperatur durch Schaukeln manuell.
Hinweis: Eine Leiter der Bands sollten in jeder Bahn sichtbar. - Holen Sie die Membran aus der Färbelösung mit Pinzette und sehen Sie, ob die Leiter der Bands einheitliche Muster und Intensität in jeder Gasse hat wo die Samples geladen wurden.
- Entfernen Sie nach der Überprüfung der Färbung die Färbelösung zu, und fügen Sie TBST Puffer (20 mM Tris-HCl pH 7.4, 150 mM NaCl, 0,1 % (V/V) Polyoxyethylenesorbitan Monolaurate). Das Volumen des Puffers sollte ausreichen, um eine Membran zu decken.
Hinweis: Die Ponceau S-Lösung kann gesammelt werden und mehrmals wiederverwendet. - Rocken Sie die Membranen in TBST auf einen rockigen Shaker (~ 60 u/min) bei Raumtemperatur, bis keine sichtbaren Bänder auf den Membranen bleiben.
- Wiederholen Sie die Waschschritt mit frischen TBST für 5 min.
Hinweis: Die Belichtungszeit variieren je nach der ECL-Lösung und das System zur Erkennung von Chemolumineszenz verwendet.
- Schalten Sie ein Imager ausgerüstet mit einer – Coupled Ladegerät (CCD) Kamera und einem Computer an die Kamera angeschlossen. Starten Sie die Steuerungs-Software für den Imager. Warten Sie, bis die Temperatur der CCD Kamera-25 ° c erreicht hat
- Setzen Sie 1 mL der ECL-Lösung für eine Membran auf einer Plastikfolie ausgebreitet auf einer Bank.
- Die ECL-Lösung setzen Sie eine Membran Gel-Seite nach oben auf und dann umdrehen Sie schnell, dass beiden Seiten der Membran mit der Lösung beschichtet sind.
- Die Membran holen und abtropfen lassen indem man eine Seite der Membran Touch ein Papiertuch für 5 s.
- Setzen Sie die Membran zwischen klaren Folien (z.B. klare Papier Taschen).
Hinweis: Plastikfolie wird nicht empfohlen für die Umwicklung der Membranen. Klaren zum Einwickeln der Membran verwendeten Folien müssen so flach wie möglich ohne sichtbare Falten, unebenem Hintergrund zu vermeiden. - Legen Sie die Membran auf einem schwarzen Tablett. Stellte das Tablett in den Imager und schließen Sie die Tür.
- Klicken Sie auf "Focusing", in das Fenster der Steuerungssoftware. Überprüfen Sie, dass die Membran richtig positioniert ist. Klicken Sie auf die Schaltfläche "Zurück".
- Wählen Sie "Präzision" für"Belichtung". Wählen Sie "Manuell" für "Belichtungszeit" und legen Sie die Belichtungszeit auf 1 s.
- Wählen Sie "Hoch" für "Empfindlichkeit/Auflösung". Lassen Sie "Digitalisierung Bild hinzufügen" deaktiviert. Klicken Sie auf "Start", um das Bild aufzunehmen.
- Speichern Sie das Bild, das erschien auf dem Display des Computers als TIFF-Datei.
- Wiederholen Sie die Aufnahmen mit der Belichtungszeit von 1, 3, 10, 30, 60, 90, 120, 150 s und bis zu 180 s. Bei der Einnahme von des letzten Bildes überprüfen Sie "Digitalisierung Bild hinzufügen", damit das digitale Bild, nicht die Chemilumineszenz der Membran gleichzeitig entnommen werden kann.
- Wählen Sie das beste Bild mit der größten Dynamik und ohne jede sättigbaren Pixel (rot dargestellt) in den Bands von Interesse.
Hinweis: Konventionelle Röntgenaufnahmen können auch für Erkennung19verwendet werden.
Ergebnisse
Überexpression System: Phosphorylierung des HA-Rab10 von 3 Zellen × Fahne-LRRK2 in HEK293:
HEK293 Zellen wurden mit 0,266 µg von HA-Rab10 Wildtyp und 1.066 µg von 3 × Flagge LRRK2 (Wildtyp, Kinase-inaktive Mutant (K1906M) oder FPD Mutanten) transfiziert. Rab10 Phosphorylierung wurde geprüft durch P-Tag SDS-PAGE gefolgt von Immunoblotting mit einem Anti-HA Antikörper (Abbildung 2). 10 µg Proteine wurden auf einem 10 % Gel (80 x 100 x 1 mm) mit 50 µM P-Tag Acrylamid und 100 µM MnCl2ausgeführt. Die Belichtungszeit für das P-Tag-Gel (Oberseite) war 10 s mit einer Standardlösung ECL. Co-Überexpression von LRRK2 mit Rab10 verursacht Band Verschiebung auf dem P-Tag-Gel (markiert mit einem offenen Kreis in der oberen Leiste; Vergleiche die Bahnen 2 und 4). Im Gegensatz dazu Co Ausdruck mit einer Kinase-inaktive Mutant (K1906M) LRRK2 konnte nicht die Mobilität der Rab10 geändert werden (Vergleiche die Bahnen 4 und 5), mitteilt, daß die Band Verschiebung aufgrund der LRRK2-Kinase-Aktivität. Die Band-Verschiebung wurde durch alle FPD Mutationen (Vergleiche die Bahnen 4 und 6-13) im Einvernehmen mit den vorherigen Bericht15erhöht.
Endogene System: Phosphorylierung des Rab10 von LRRK2 in kultivierten Zellen:
3 t 3-Swiss Albino embryonalen Fibroblasten Mauszellen behandelt wurden, mit oder ohne ein LRRK2-Inhibitor (GSK2578215A)20 auf eine Endkonzentration von 1 µM für 1 h (Abb. 3A). Menschlichen Lunge Karzinom A549 Zellen wurden behandelt, mit oder ohne ein weiteres LRRK2-Inhibitor (MLi-2)21 um eine Endkonzentration von 10 nM für 1 h (Abb. 3 b). Endogene Rab10 Phosphorylierung von endogenen LRRK2 wurde von P-Tag SDS-PAGE gefolgt von Immunoblotting mit einem Anti-Rab10-Antikörper untersucht. 30 µg Proteine wurden auf einer 10 % Gel (100 x 100 x 1 mm für 3 t 3-Swiss Albino-Zellen) und 80 mm für A549 Zellen mit 50 µM P-Tag Acrylamid und 100 µM MnCl2ausgeführt. Die Belichtung für das P-Tag-Gel (Oberseite) in Abbildung 3A und 3 b Abbildung wurden 3 min und 90 s, beziehungsweise. Die Band-Verschiebung entspricht phosphorylierten endogenen Rab10 beobachtet auf dem P-Tag-Gel (markiert mit einem offenen Kreis Deckplatte; Vergleiche Bahnen 1-2 und 3-4) die bei der Behandlung von Zellen mit GSK2578215A oder MLi-2 verschwunden. Die Wirksamkeit von LRRK2-Hemmern wurde durch Immunoblotting mit der Anti-pSer935 LRRK2 Antikörper (das dritte Panel von unten), ist eine gut etablierte Auslesen von LRRK2 Hemmung in Zellen22validiert.

Abbildung 1: Schematische Darstellung der P-Tag SDS-PAGE. Wenn eine Mischung aus phosphorylierten (rote Kreise) und nicht phosphoryliert (blaue Kreise) Proteine (z.B.Rab10) durch ein Gel wo ist P-Tag Acrylamid Co polymerisierten läuft, verzögert die Mobilität der einzige phosphorylierten Proteine aufgrund der starken Zusammenspiel der phosphorylierten Proteine mit dem P-Tag-Moleküle (gestrichelte Pfeile). Da Rab10 von LRRK2 bei einem einzigen Rückstand Thr73 phosphoryliert wird, läuft Rab10 als zwei Bands: die Top-Band phosphorylierten Rab10 und die unteren Band darstellt nicht phosphoryliert Rab10. Wenn Zellen mit LRRK2-Hemmern behandelt werden, sollte die Top-Band verschwinden. Bitte klicken Sie hier für eine größere Version dieser Figur.
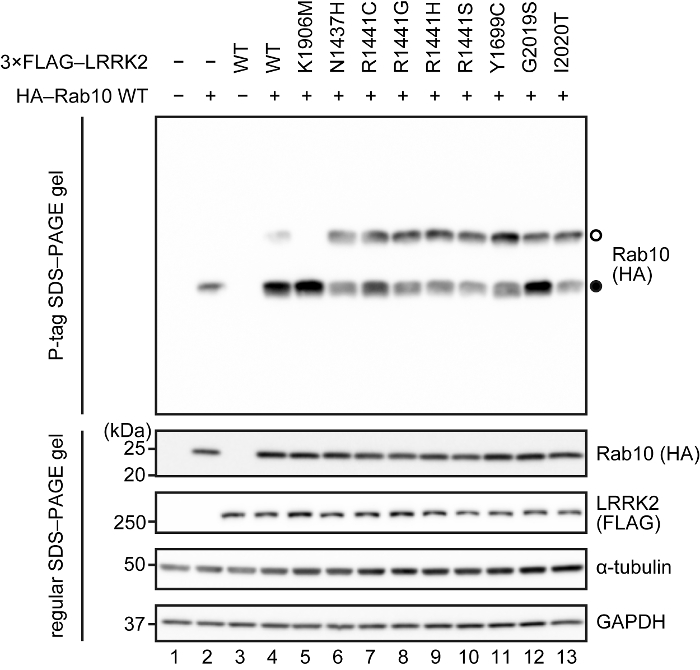
Abbildung 2: Repräsentatives Ergebnis der P-Tag-Blot: Überexpression System. Der obere Bereich zeigt den P-Tag-Fleck mit einem Anti-HA Antikörper, wo phosphorylierten und nicht phosphoryliert Rab10 mit gekennzeichnet sind (○) geöffnet und geschlossen (●) Kreisen, beziehungsweise. Die Paneele der P-Tag Fleck unten sind Immunoblots auf normalen SDS-PAGE Gelen mit einer Anti-HA, Anti-FLAG, Anti-α-Tubulin und Anti-GAPDH Antikörper. Die HA-Blot und Flagge Blot werden dafür die Überexpression von HA-Rab10 und 3xFLAG-LRRK2 bzw. angezeigt. Die α-Tubulin und GAPDH Flecken sind dafür die gleiche Belastung gezeigt. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 3. Repräsentatives Ergebnis der P-Tag-Blot: endogene System. Zwei Zellinien, nämlich (A) 3 t 3-Swiss Albino und (B) A549 Zellen, wurden verwendet. In beiden Abbildungen zeigen die Paneele im P-Tag blots mit Antikörpers Anti-Rab10, wo phosphorylierten und nicht phosphoryliert endogenen Rab10 sind gekennzeichnet mit offenen (○) und geschlossen (●) Kreise bzw.. Die Platten unten P-Tag Flecken sind Immunoblots auf normalen SDS-PAGE Gelen mit einer Anti-Rab10, Anti-pSer935 LRRK2, Anti-LRRK2 und Anti-α-Tubulin Antikörper. Die Rab10 und LRRK2 ergeben sich für die Gewährleistung des ähnlichen Ausdrucks der endogenen Rab10 und LRRK2 zwischen den Proben bzw.. Der pSer935 LRRK2 Blot sind dafür gezeigt, dass GSK2578215A (A) und MLi-2 (B) wie erwartet funktionierte. Der α-Tubulin-Blot werden dafür die gleiche Beladung angezeigt. Die Bilder von den P-Tag Flecken überlagert mit dem Molekulargewicht Marker sind in Abbildung S4gezeigt. Bitte klicken Sie hier für eine größere Version dieser Figur.
Abbildung S1. DNA-Sequenzen von den Plasmiden. Die DNA-Sequenzen des Plasmids HA-Rab10/pcDNA5 FRT, und dass 3 × × Fahne-LRRK2/p3 FLAG-CMV-10-Codierung. Alle Plasmide verwendet in diesem Protokoll sind von den Autoren auf Anfrage erhältlich. Bitte klicken Sie hier, um diese Datei herunterladen.
Abbildung S2. Ein Beispiel der P-Tag SDS-PAGE-Optimierung. Der gleiche Satz von Proben wie in Abbildung 3A wurde zur Optimierung der P-Tag SDS-PAGE verwendet. Vier verschiedene Bedingungen getestet wurden, wie in der Abbildung gezeigt. Die Bänder entsprechend phosphorylierten und nicht phosphoryliert Rab10 werden durch solide und gestrichelte Linie Rechtecke bzw. hervorgehoben.Basierend auf diesem Experiment, gab die Bedingung mit 10 % Acrylamid, 50 µM P-Tag Acrylamid und 100 µM MnCl2 die beste Trennung der beiden Bands, beide suchen in der Mitte des Gels. Bitte klicken Sie hier, um diese Datei herunterladen.
Abbildung S3. Vergleich der Migration Muster eines Markers Molekulargewicht. Der Molekulargewicht Marker (MWM) ausgeführt wurde, auf einem normalen SDS-PAGE-Gel (linken) oder auf einem P-Tag SDS-PAGE-Gel (mittlere Panel) und die Gele waren voller Flecken von Coomassie-Färbung. Im Rechte Bereich zeigt ein Immunoblot der in Abbildung 2 (Bahn 4 und 5) verwendeten Proben auf dem gleichen P-Tag SDS-PAGE-Gel als mittleren Feld, um die Positionen der phosphorylierten und nicht phosphoryliert Rab10 zeigen. Die Pfeilspitze an der rechten Seite der Immunoblot gibt die Position eines der MWM auf dem P-Tag SDS-PAGE-Gel. Bitte klicken Sie hier, um diese Datei herunterladen.
Abbildung S4. Die Position eines Markers Molekulargewicht auf P-Tag SDS-PAGE Gelen. Digitalisierte Bilder der Membranen zur Abbildung 3A und 3 b Abbildung erscheinen als (A) und(B), beziehungsweise. Der Immunoblots in Abbildung 3 dargestellt werden überlagert, um die relative Position des Markers Molekulargewicht (MWM) zeigen. Die MWM nicht gut auf die Membranen zu übertragen, sondern der Marker (Pfeilspitze) in Abbildung S3 war schwach, aber konsequent beobachtet. Die Position der MWM ist mit gepunkteten Linien gekennzeichnet. Beachten Sie, dass die Bahnen irrelevant für die Zahlen zwischen den MWM und die Gassen von Interesse durchgestrichen sind. Bitte klicken Sie hier, um diese Datei herunterladen.
Diskussion
Hier beschreiben wir eine einfache und robuste Methode zum Nachweis Rab10 Phosphorylierung von LRRK2 auf endogene Ebenen basierend auf der P-Tag-Methode. Da die derzeit verfügbaren Antikörper gegen phosphorylierten Rab10 nur mit überexprimieren Proteine15arbeitet, ist das vorliegende Verfahren unter Verwendung von P-Tag SDS-PAGE die einzige Möglichkeit, endogene Ebenen der Rab10 Phosphorylierung zu beurteilen. Darüber hinaus erlaubt das vorliegende Verfahren die Schätzung der Stöchiometrie der Rab10 Phosphorylierung in den Zellen. Da die P-Tag-Methodik auf Phospho-Proteine allgemein anwendbar ist, kann dieses Protokolls ein "Prototyp" für ähnliche Methoden für andere Phospho-Proteine.
Wichtige Schritte im Protokoll sind Gießen Gele und Vorbereitung der Proben. P-Tag Acrylamid ist ein relativ Foto-labile Reagenz und die Fähigkeit, die elektrophoretische Mobilität der phosphorylierten Proteine zu verzögern ist manchmal verloren nach längerer Lagerung bei 4 ° C. Forscher sollten alle Sorgfalt zu vermeiden, dass P-Tag Acrylamid ans Licht. Darüber hinaus muss der P-Tag-Molekül zu einem Komplex mit Mn2 + -Ionen, Phosphate zu erfassen. Daher sollten Proben nicht Komplexbildnern wie EDTA, enthalten die üblichen Komponenten der handelsüblichen SDS-PAGE-Probe-Puffer. Wir empfehlen die klassische Laemmli Probenpuffer zur Vorbereitung von Proben für P-Tag SDS-PAGE.
Ein weiterer kritischer Punkt ist die Konzentration von Acrylamid, P-Tag Acrylamid und MnCl2 in der Trennung Gel Mischung (Abbildung S2) gründlich zu optimieren. Die Migration Abstände der phosphorylierten und nicht phosphoryliert Bands variiert je nach verwendeten Reagenzien (Menge, Reinheit usw.)und Optimierung der entsprechenden Konzentrationen durch das Testen von mehreren unterschiedlicher Konzentrationen in Kombination ist obligatorisch (z. B. P-Tag Acrylamid (25, 50, 75 µM), MnCl2 (1:2 oder 1:3 molare Verhältnis zu Acrylamid P-Tag), und Acrylamid (7,5, 10, 12,5 %)). Die phosphorylierten und nicht phosphoryliert Bands sollten erscheinen in der Mitte die Gele und gut voneinander getrennt. Für Prozess-Optimierung empfiehlt es sich, überexprimieren Rab10 zu verwenden, da die verschobene Band leicht nachweisbar ist. Die Autoren bieten Kontrollproben für dieses Protokoll.
Die größte Verwirrung, die dieses Protokoll verursachen können werden aufgrund der Differenz der Migrationsmuster der MWM zwischen normalen und P-Tag SDS-PAGE Gelen. Wie in Abbildung S3gezeigt, die Migrationsmuster der MWM auf regelmäßige und P-Tag SDS-PAGE Gelen (10 %) sind sehr unterschiedlich und man kann nicht mit der MWM für die Schätzung des Molekulargewichts der Proteine auf P-Tag SDS-PAGE Gelen. Jedoch auf P-Tag SDS-PAGE Gelen zeichnet eines der MWM sich durch die Migration in der Mitte des Gels (Pfeilspitze in Abbildung S3), die konsequent unter Gele beobachtet wird (siehe Abbildung S4). Diese MWM wandert immer zwischen den Bands von phosphorylierten und nicht phosphoryliert Rab10. Daher eignet sich dieser Marker nicht nur dafür, dass P-Tag SDS-PAGE funktioniert vor der Übertragung, sondern auch als ein Wahrzeichen für einen groben Sinn für wo die Bands der phosphorylierten und nicht phosphoryliert Rab10 gesehen werden.
Für die Erkennung von Bands auf Immunoblots, haben wir ein Imager ausgerüstet mit einer gekühlten CCD Kamera verwendet (siehe Tabelle der Materialien) sowie als einer herkömmlichen Röntgenaufnahme film entwickelndes System, und festgestellt, dass beide Systeme gut funktionieren. Für die Erkennung von endogenen Ebenen der Rab10 Phosphorylierung in kultivierten Zellen ist es notwendig, die Zell-Linien nutzen die hohe Expression von endogenen LRRK2 wie Maus embryonalen Fibroblasten haben (entweder primär kultiviert oder Zelle Linien (verewigt 3 t 3-Swiss Albino, etc.)) und menschlichen Lunge Karzinom abgeleitet A549 Zellen. Für die Erkennung von endogenen Ebenen der Rab10 Phosphorylierung in Maus-Geweben empfiehlt es sich, Lunge16verwenden.
Die Quantifizierung der Rab10 Phosphorylierung ist nicht jenseits der üblichen Immunoblotting. Wenn die Stöchiometrie der Rab10 Phosphorylierung ist sehr gering (wie in Abbildung 3dargestellt), tendenziell die Intensität der nicht phosphoryliert Band (mit einem geschlossenen Kreis markiert) weit über die Sättigungsniveau, so dass die exakte Bestimmung der Stöchiometrie unmöglich. Dennoch ist qualitative Schätzung noch möglich auf jeden Fall ohne mit dem vorliegenden Verfahren nicht erhältlich ist. Da die Erkennung der Phosphorylierung des endogenen Rab10 über längere Zeit erfordert, es neigt dazu, dass einige unspezifische Bands, die auf dem P-Tag beflecken, obwohl der Anti-Rab10-Antikörper, die in diesem Protokoll verwendeten ziemlich sauber Immunoblots für den normalen Gebrauch gibt . Um phosphorylierten Proteine aus unspezifische Banden zu unterscheiden, ist es wichtig, ein Inhibitor-Behandlung-Steuerelement verwenden, in denen phosphorylierten Bands, aber keine unspezifische Bänder verschwinden sollte. LRRK2 phosphorylates Rab8 neben Rab1015haben, und wir erfolgreich dieses Protokolls, Rab8A sowie (Daten nicht gezeigt). Dieses Protokolls kann potenziell zu prüfen, die Phosphorylierung des Proteine verwendet werden, obwohl es möglicherweise technische Schwierigkeiten, wenn das Protein groß ist (> 100 kDa) oder multiplizieren phosphorylierten. Denn Rab10 ein kleines Protein (25 kDa ist) und einzeln auf Thr73 von LRRK2 phosphoryliert, das Ergebnis der P-Tag SDS-PAGE einfach ist nur eine verschobene Band festgestellt.
In naher Zukunft wird es entscheidend für die Methode um quantitativ die Kinase-Aktivität von LRRK2 bei Patienten stammenden Proben in gewissem Sinne Hochdurchsatz-zur Veränderung der die Kinase-Aktivität von LRRK2 bei PD-Patienten sowie Bewertung Bewertung erkennen zu etablieren die Wirkung von Medikamenten auf LRRK2 in klinischen Studien. Seit Menschenblut peripheren Mononukleäre Zellen (PBMCs) express relativ hohen endogenen LRRK223, PBMCs oder weiter isolierten Blutzellen werden mal testen für endogene Rab10 Phosphorylierung. Das vorliegende Verfahren wird nicht nur bei der Untersuchung der Grundlagenbiologie die LRRK2 Signalweg nützlich sein, sondern auch Hilfe bei der Erlangung einer grundlegenden Proof of Concept für die Entscheidung, welche Probe in Patienten stammenden Proben in solchen breit angelegten Studien analysiert werden.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Wir danken Dr. Takeshi Iwatsubo (Universität von Tokyo, Japan) freundlicherweise dafür die Plasmiden Codierung 3xFLAG LRRK2 WT und Mutanten. Wir danken auch Dr. Dario Alessi (Universität von Dundee, UK) freundlicherweise zur MLi-2 und das Plasmid Codierung HA-Rab10. Diese Arbeit wurde von der Japan Society für Promotion of Science (JSPS) KAKENHI Grant Anzahl JP17K08265 (G.I) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Dulbecco's phosphate-buffered saline (DPBS) | homemade | 150 mM NaCl, 8 mM Na2HPO4-12H2O, 2.7 mM KCl, 1.5 mM KH2PO4 in MilliQ water and sterilized by autoclaving | |
| Sodium chloride | Nacalai Tesque | 31320-34 | |
| Sodium Disodium Hydrogenphosphate 12-Water | Wako | 196-02835 | |
| Potassium chloride | Wako | 163-03545 | |
| Potassium Dihydrogen Phosphate | Wako | 169-04245 | |
| 2.5% Trypsin (10X) | Sigma-Aldrich | T4549 | Dilute 10-fold with sterile DPBS for preparing working solution |
| Dulbecco's modified Eagle medium (DMEM) | Wako | 044-29765 | |
| Fetal bovine serum | BioWest | S1560 | Heat-inactivated at 56 °C for 30 min |
| Penicillin-Streptomycin (100X) | Wako | 168-23191 | |
| HEPES | Wako | 342-01375 | |
| Sodium hydroxide | Wako | 198-13765 | |
| Polyethylenimine HCl MAX, Linear, Mw 40,000 (PEI MAX 40000) | PolySciences, Inc. | 24765-1 | Stock solution was prepared in 20 mM HEPES-NaOH pH 7.0 at 1 mg/mL and the pH was then adjusted to 7.0 with NaOH |
| Dimethyl sulfoxide | Wako | 045-28335 | |
| Tris | STAR | RSP-THA500G | |
| Hydrochloric acid | Wako | 080-01066 | |
| Polyoxyethylene(10) Octylphenyl Ether | Wako | 160-24751 | Equivalent to Triton X-100 |
| Ethylene glycol-bis(2-aminoethylether)-N,N,N’,N’-tetraacetic acid (EGTA) | Wako | 346-01312 | |
| Sodium orthovanadate(V) | Wako | 198-09752 | |
| Sodium fluoride | Kanto Chemical | 37174-20 | |
| β-Glycerophosphoric Acid Disodium Salt Pentahydrate | Nacalai Tesque | 17103-82 | |
| Sodium pyrophosphate decahydrate | Kokusan Chemical | 2113899 | |
| Microcystin-LR | Wako | 136-12241 | |
| Sucrose | Wako | 196-00015 | |
| Complete EDTA-free protease inhibitor cocktail | Roche | 11873580001 | Dissolve one tablet in 1 mL water, which can be stored at -20 °C for a month. Use it at 1:50 dilution for cell lysis |
| Pierce Coomassie (Bradford) Protein Assay Kit | Thermo Fisher Scientific | 23200 | |
| Sodium dodecyl sulfate | Nacalai Tesque | 31607-65 | |
| Glycerol | Wako | 075-00616 | |
| Bromophenol blue | Wako | 021-02911 | |
| β-mercaptoethanol | Kanto Chemical | 25099-00 | |
| Ethanol | Wako | 056-06967 | |
| Methanol | Wako | 136-01837 | |
| Phosphate-binding tag acrylamide | Wako | AAL-107 | P-tag acrylamide |
| 40% (w/v) acrylamide solution | Nacalai Tesque | 06119-45 | Acrylamide:Bis = 29:1 |
| Tetramethylethylenediamine (TEMED) | Nacalai Tesque | 33401-72 | |
| Ammonium persulfate (APS) | Wako | 016-08021 | 10% (w/v) solution was prepared by dissolving the powder of ammonium persulfate in MilliQ water |
| 2-propanol | Wako | 166-04831 | |
| Manganese chloride tetrahydrate | Sigma-Aldrich | M3634 | |
| Precision Plus Protein Prestained Standard | Bio-Rad | 1610374, 1610373, 1610377 | Molecular weight marker used in the protocol |
| WIDE-VIEW Prestained Protein Size Marker III | Wako | 230-02461 | |
| Glycine | Nacalai Tesque | 17109-64 | |
| Amersham Protran NC 0.45 | GE Healthcare | 10600007 | Nitrocellulose membrane |
| Durapore Membrane Filter | EMD Millipore | GVHP00010 | PVDF membrane |
| Filter Papers No.1 | Advantec | 00013600 | |
| Ponceau S | Nacalai Tesque | 28322-72 | |
| Acetic acid | Wako | 017-00251 | |
| Tween-20 | Sigma-Aldrich | P1379 | polyoxyethylenesorbitan monolaurate |
| Ethylenediaminetetraacetic acid (EDTA) | Wako | 345-01865 | |
| Skim milk powder | Difco Laboratories | 232100 | |
| Immunostar | Wako | 291-55203 | ECL solution (Normal sensitivity) |
| Immunostar LD | Wako | 290-69904 | ECL solution (High sensitivity) |
| CBB staining solution | homemade | 1 g CBB R-250, 50% (v/v) methanol, 10% (v/v) acetic acid in 1 L of MilliQ water | |
| CBB R-250 | Wako | 031-17922 | |
| CBB destaining solution | homemade | 12% (v/v) methanol, 7% (v/v) acetic acid in 1 L MilliQ water | |
| Name | Company | Catalog Number | Comments |
| Antibodies | |||
| anti-HA antibody | Sigma-Aldrich | 11583816001 | Used at 0.2 μg/mL for immunoblotting. |
| anti-Rab10 antibody | Cell Signaling Technology | #8127 | Used at 1:1000 for immunoblotting. Specificity was confirmed by CRISPR KO in Ito et al., Biochem J, 2016. |
| anti-pSer935 antibody | Abcam | ab133450 | Used at 1 μg/mL for immunoblotting. |
| anti-LRRK2 antibody | Abcam | ab133518 | Used at 1 μg/mL for immunoblotting. |
| anti-α-tubulin antibody | Sigma-Aldrich | T9026 | Used at 1 μg/mL for immunoblotting. |
| anti-GAPDH antibody | Santa-Cruz | sc-32233 | Used at 0.02 μg/mL for immunoblotting. |
| Peroxidase AffiniPure Sheep Anti-Mouse IgG (H+L) | Jackson ImmunoResearch | 515-035-003 | Used at 0.16 μg/mL for immunoblotting. |
| Peroxidase AffiniPure Goat Anti-Rabbit IgG (H+L) | Jackson ImmunoResearch | 111-035-003 | Used at 0.16 μg/mL for immunoblotting. |
| Name | Company | Catalog Number | Comments |
| Inhibitors | |||
| GSK2578215A | MedChem Express | HY-13237 | Stock solution was prepared in DMSO at 10 mM and stored at -80 °C |
| MLi-2 | Provided by Dr Dario Alessi (University of Dundee) | Stock solution was prepared in DMSO at 10 mM and stored at -80 °C | |
| Name | Company | Catalog Number | Comments |
| Plasmids | |||
| Rab10/pcDNA5 FRT TO HA | Provided by Dr Dario Alessi (University of Dundee) | This plasmid expresses amino-terminally HA-tagged human Rab10. | |
| LRRK2 WT/p3xFLAG-CMV-10 | Provided by Dr Takeshi Iwatsubo (University of Tokyo) | Ito et al., Biochemistry, 46: 1380–1388 (2007). This plasmid expresses amino-terminally 3xFLAG-tagged wild-type human LRRK2. | |
| LRRK2 K1906M/p3xFLAG-CMV-10 | Provided by Dr Takeshi Iwatsubo (University of Tokyo) | Ito et al., Biochemistry, 46: 1380–1388 (2007). This plasmid expresses amino-terminally 3xFLAG-tagged K1906M kinase-inactive mutant of human LRRK2. | |
| LRRK2 N1437H/p3xFLAG-CMV-10 | This paper. This plasmid expresses amino-terminally 3xFLAG-tagged N1437H FPD mutant of human LRRK2. | ||
| LRRK2 R1441C/p3xFLAG-CMV-10 | Provided by Dr Takeshi Iwatsubo (University of Tokyo) | Kamikawaji et al., Biochemistry, 48: 10963–10975 (2013). This plasmid expresses amino-terminally 3xFLAG-tagged R1441C FPD mutant of human LRRK2. | |
| LRRK2 R1441G/p3xFLAG-CMV-10 | Provided by Dr Takeshi Iwatsubo (University of Tokyo) | Kamikawaji et al., Biochemistry, 48: 10963–10975 (2013). This plasmid expresses amino-terminally 3xFLAG-tagged R1441G FPD mutant of human LRRK2. | |
| LRRK2 R1441H/p3xFLAG-CMV-10 | Provided by Dr Takeshi Iwatsubo (University of Tokyo) | Kamikawaji et al., Biochemistry, 48: 10963–10975 (2013). This plasmid expresses amino-terminally 3xFLAG-tagged R1441H FPD mutant of human LRRK2. | |
| LRRK2 R1441S/p3xFLAG-CMV-10 | This paper. This plasmid expresses amino-terminally 3xFLAG-tagged R1441S FPD mutant of human LRRK2. | ||
| LRRK2 Y1699C/p3xFLAG-CMV-10 | Provided by Dr Takeshi Iwatsubo (University of Tokyo) | Kamikawaji et al., Biochemistry, 48: 10963–10975 (2013). This plasmid expresses amino-terminally 3xFLAG-tagged Y1699C FPD mutant of human LRRK2. | |
| LRRK2 G2019S/p3xFLAG-CMV-10 | Provided by Dr Takeshi Iwatsubo (University of Tokyo) | Kamikawaji et al., Biochemistry, 48: 10963–10975 (2013). This plasmid expresses amino-terminally 3xFLAG-tagged G2019S FPD mutant of human LRRK2. | |
| LRRK2 I2020T/p3xFLAG-CMV-10 | Provided by Dr Takeshi Iwatsubo (University of Tokyo) | Kamikawaji et al., Biochemistry, 48: 10963–10975 (2013). This plasmid expresses amino-terminally 3xFLAG-tagged I2020T FPD mutant of human LRRK2. | |
| Name | Company | Catalog Number | Comments |
| Equipments | |||
| CO2 incubator | Thermo Fisher Scientific | Forma Series II 3110 Water-Jacketed | |
| Auto Pipette | Drummond | Pipet-Aid PA-400 | |
| Micropipette P10 | Nichiryo | 00-NPX2-10 | 0.5–10 μL |
| Micropipette P200 | Nichiryo | 00-NPX2-200 | 20–200 μL |
| Micropipette P1000 | Nichiryo | 00-NPX2-1000 | 100–1000 μL |
| Tips for micropipette P10 | STAR | RST-481LCRST | Sterile |
| Tips for micropipette P200 | FUKAEKASEI | 1201-705YS | Sterile |
| Tips for micropipette P1000 | STAR | RST-4810BRST | Sterile |
| 5 mL disporsable pipette | Greiner | 606180 | Sterile |
| 10 mL disporsable pipette | Greiner | 607180 | Sterile |
| 25 mL disporsable pipette | Falcon | 357535 | Sterile |
| Hematocytometer | Sunlead Glass | A126 | Improved Neubeuer |
| Microscope | Olympus | CKX53 | |
| 10 cm dishes | Falcon | 353003 | For tissue culture |
| 6-well plates | AGC Techno Glass | 3810-006 | For tissue culture |
| Vortex mixer | Scientific Industries | Vortex-Genie 2 | |
| Cell scrapers | Sumitomo Bakelite | MS-93100 | |
| 1.5 mL tubes | STAR | RSV-MTT1.5 | |
| 15 mL tubes | AGC Techno Glass | 2323-015 | |
| 50 mL tubes | AGC Techno Glass | 2343-050 | |
| Centrifuges | TOMY | MX-307 | |
| 96-well plates | Greiner | 655061 | Not for tissue culture |
| Plate reader | Molecular Devices | SpectraMax M2e | |
| SDS–PAGE tanks | Nihon Eido | NA-1010 | |
| Transfer tanks | Nihon Eido | NA-1510B | |
| Gel plates (notched) | Nihon Eido | NA-1000-1 | |
| Gel plates (plain) | Nihon Eido | NA-1000-2 | |
| Silicon spacers | Nihon Eido | NA-1000-16 | |
| 17-well combs | Nihon Eido | Custom made | |
| Binder clips | Nihon Eido | NA-1000-15 | |
| 5 mL syringe | Terumo | SS-05SZ | |
| 21G | Terumo | NN-2138R | |
| Power Station 1000 VC | ATTO | AE-8450 | Power supply for SDS–PAGE and transfer |
| Large weighing boats | Ina Optika | AS-DL | |
| Plastic containers | AS ONE | PS CASE No.4 | 10 x 80 x 50 mm |
| Rocking shaker | Titech | NR-10 | |
| Styrene foam box | generic | The internal dimensions should fit one transfer tank (200 x 250 x 250 mm). | |
| ImageQuant LAS-4000 | GE Healthcare | An imager equipped with a cooled CCD camera for detection of ECL |
Referenzen
- Sveinbjornsdottir, S. The clinical symptoms of Parkinson's disease. J. Neurochem. 139 (Suppl. 1), 318-324 (2016).
- Hernandez, D. G., Reed, X., Singleton, A. B. Genetics in Parkinson disease: Mendelian versus non-Mendelian inheritance. J. Neurochem. 139 (Suppl. 1), 59-74 (2016).
- Paisán-Ruíz, C., et al. Cloning of the gene containing mutations that cause PARK8-linked Parkinson's disease. Neuron. 44 (4), 595-600 (2004).
- Zimprich, A., et al. Mutations in LRRK2 cause autosomal-dominant parkinsonism with pleomorphic pathology. Neuron. 44 (4), 601-607 (2004).
- Gilks, W. P., et al. A common LRRK2 mutation in idiopathic Parkinson's disease. Lancet. 365 (9457), 415-416 (2005).
- Satake, W., et al. Genome-wide association study identifies common variants at four loci as genetic risk factors for Parkinson's disease. Nat. Genet. 41 (12), 1303-1307 (2009).
- Simón-Sánchez, J., et al. Genome-wide association study reveals genetic risk underlying Parkinson's disease. Nat. Genet. 41 (12), 1308-1312 (2009).
- Klein, C., Ziegler, A. Imputation of sequence variants for identification of genetic risks for Parkinson's disease: a meta-analysis of genome-wide association studies. Lancet. 377 (9766), 641-649 (2011).
- Cookson, M. R. The role of leucine-rich repeat kinase 2 (LRRK2) in Parkinson's disease. Nat. Rev. Neurosci. 11 (12), 791-797 (2010).
- Ozelius, L. J., et al. LRRK2 G2019S as a Cause of Parkinson’s Disease in Ashkenazi Jews. N. Engl. J. Med. 354 (4), 424-425 (2006).
- Lesage, S., et al. LRRK2 G2019S as a Cause of Parkinson’s Disease in North African Arabs. N. Engl. J. Med. 354 (4), 422-423 (2006).
- Bouhouche, A., et al. LRRK2 G2019S Mutation: Prevalence and Clinical Features in Moroccans with Parkinson's Disease. Parkinsons. Dis. , 1-7 (2017).
- West, A. B., et al. Parkinson's disease-associated mutations in leucine-rich repeat kinase 2 augment kinase activity. Proc. Natl. Acad. Sci. U. S. A. 102 (46), 16842-16847 (2005).
- Ito, G., et al. GTP binding is essential to the protein kinase activity of LRRK2, a causative gene product for familial Parkinson's disease. Biochemistry. 46 (5), 1380-1388 (2007).
- Steger, M., et al. Phosphoproteomics reveals that Parkinson's disease kinase LRRK2 regulates a subset of Rab GTPases. Elife. 5, (2016).
- Ito, G., et al. Phos-tag analysis of Rab10 phosphorylation by LRRK2: a powerful assay for assessing kinase function and inhibitors. Biochem. J. 473, 2671-2685 (2016).
- Kinoshita, E., Kinoshita-Kikuta, E., Takiyama, K., Koike, T. Phosphate-binding tag, a new tool to visualize phosphorylated proteins. Mol. Cell. Proteomics. 5 (4), 749-757 (2006).
- Using a Hemacytometer to Count Cells. J. Vis. Exp Available from: https://www-jove-com.remotexs.ntu.edu.sg/science-education/5048/using-a-hemacytometer-to-count-cells (2017)
- Ni, D., Xu, P., Gallagher, S. Immunoblotting and Immunodetection. Curr. Protoc. Mol. Biol. (114), 10.8.1-10.8.37 (2016).
- Reith, A. D., et al. GSK2578215A; a potent and highly selective 2-arylmethyloxy-5-substitutent-N-arylbenzamide LRRK2 kinase inhibitor. Bioorg. Med. Chem. Lett. 22 (17), 5625-5629 (2012).
- Fell, M. J., et al. MLi-2, a potent, selective and centrally active compound for exploring the therapeutic potential and safety of LRRK2 kinase inhibition. J. Pharmacol. Exp. Ther. 355, 397-409 (2015).
- Dzamko, N., et al. Inhibition of LRRK2 kinase activity leads to dephosphorylation of Ser(910)/Ser(935), disruption of 14-3-3 binding and altered cytoplasmic localization. Biochem. J. 430 (3), 405-413 (2010).
- Thévenet, J., Pescini Gobert, R., Hooft van Huijsduijnen , R., Wiessner, C., Sagot, Y. J. Regulation of LRRK2 expression points to a functional role in human monocyte maturation. PLoS One. 6 (6), e21519 (2011).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten