Method Article
Rab10 detección de fosforilación por la actividad de LRRK2 mediante SDS-PAGE con una etiqueta de enlace de fosfato
En este artículo
Resumen
El presente estudio describe un método sencillo de detectar niveles endógenos de la fosforilación Rab10 quinasa repetición rica en leucina 2.
Resumen
Mutaciones de la cinasa repetición rica en leucina 2 (LRRK2) han demostrado estar relacionados con la enfermedad de Parkinson familiar (FPD). Puesto que la activación anormal de la actividad quinasa de LRRK2 se ha implicado en la patogénesis de la EP, es esencial establecer un método para evaluar los niveles fisiológicos de la actividad quinasa de LRRK2. Estudios recientes revelaron que LRRK2 fosforila a miembros de la familia de Rab GTPase, incluyendo Rab10, bajo condiciones fisiológicas. Aunque la fosforilación de Rab10 endógeno de LRRK2 en células cultivadas podría detectarse por espectrometría de masas, ha sido difícil de detectar por immunoblotting debido a la pobre sensibilidad de anticuerpos específicos de fosforilación actualmente disponibles Rab10. Aquí, describimos un sencillo método para detectar los niveles endógenos de Rab10 fosforilación de LRRK2 basado en immunoblotting utilizando sodio electroforesis dodecyl del sulfato gel de poliacrilamida (SDS-PAGE) combinado con una enlace de fosfato etiqueta (P-), que es N-(5-(2-aminoethylcarbamoyl)pyridin-2-ylmetyl) -N,N',N'- tris (pyridin-2-yl-metil) - 1,3 - diaminopropan-2-ol. El presente Protocolo no sólo proporciona un ejemplo de la metodología que utiliza la etiqueta P pero también permite la evaluación de cómo mutaciones así como tratamiento y administración de inhibidor u otros factores alteran la señalización corriente abajo de LRRK2 en células y tejidos .
Introducción
PD es una de las enfermedades neurodegenerativas más comunes, que afecta predominante a las neuronas dopaminérgicas en el cerebro medio, dando por resultado la disfunción de los sistemas de motor en personas de edad avanzada1. Mientras que la mayoría de los pacientes desarrollará PD de manera esporádica, hay familias que heredar la enfermedad. Se han encontrado mutaciones en varios genes vinculados con FPD2. Uno de los genes causales de la FPD es LRRK2, en el que ocho mutaciones sin sentido (N1437H, R1441C/G/H/S, Y1699C, G2019S y I2020T) vinculadas a un dominante heredado FPD llamada PARK8 hasta ahora han sido reportados3,4,5. Varios estudios de Asociación de genoma completo (GWAS) de pacientes con EP esporádicos también han identificado variaciones genómicas en el locus del LRRK2 como factor de riesgo para PD, lo que sugiere que la anormalidad en la función de LRRK2 es una causa común de la neurodegeneración en los esporádicos y PARK8 FPD6,7,8.
LRRK2 es una proteína grande (2.527 aminoácidos) que consiste en un dominio repetición rica en leucina, un Ras de unión a GTP dominio de proteínas complejas (ROC), un C-terminal de dominio, un dominio de la cinasa de serina/treonina proteína y WD40 dominio repetición9ROC (CDR). Las ocho mutaciones FPD localizar en estos dominios funcionales; N1437H y R1441C/G/H/S en el dominio ROC, Y1699C en el dominio de la COR, G2019S y I2020T en el dominio de la cinasa. Puesto que la mutación G2019S, que es lo más frecuentemente encontrado mutación en PD pacientes10,11,12, aumenta la actividad de la quinasa de LRRK2 en 2-3 veces en vitro13, se presume que el aumento anormal de fosforilación de los substratos de LRRK2 es tóxico para las neuronas. Sin embargo, ha sido imposible estudiar si se altera la fosforilación de sustratos fisiológicamente relevantes de LRRK2 en pacientes con EP familiar/esporádico debido a la falta de métodos de evaluación en muestras derivadas.
Fosforilación de la proteína es generalmente detectada por immunoblotting o análisis enzima-ligado del inmunosorbente (ELISA) usando los anticuerpos específicamente reconociendo el estado fosforilado de proteínas o por análisis de espectrometría de masa. Sin embargo, la estrategia anterior a veces no se puede aplicar debido a las dificultades en la creación de anticuerpos específicos de fosforilación. Etiquetado metabólico de las células con fosfato radiactivo es otra opción para examinar niveles fisiológicos de la fosforilación de cuando los anticuerpos específicos de fosforilación no están disponibles. Sin embargo, requiere una gran cantidad de materiales radiactivos y por lo tanto implica algunos equipos especializados para protección radiológica14. El análisis de espectrometría de masa es más sensible frente a estos métodos inmunoquímicos y llegó a ser popular en el análisis de la fosforilación de la proteína. Sin embargo, la preparación de la muestra es desperdiciador de tiempo y costosos instrumentos son necesarios para el análisis.
Un subconjunto de la familia de Rab GTPase como Rab10 y Rab8 fue divulgado recientemente como sustratos fisiológicos directos para LRRK2 basan en el resultado de un análisis a gran escala phosphoproteomic del15. Entonces hemos demostrado que la fosforilación Rab10 fue aumentada por mutaciones de la FPD en fibroblastos embrionarios de ratón y en los pulmones de knockin ratones16. En este informe, optamos por emplear una electroforesis en gel de poliacrilamida sodio dodecil sulfato (SDS-PAGE)-método en el cual una molécula de P-tag es co polimerizada en geles de SDS-PAGE (etiqueta P SDS-PAGE) para la detección de los niveles endógenos de la fosforilación Rab10, basado en porque todavía le faltaba un anticuerpo altamente sensible específico para Rab10 fosforilada. Nosotros hemos podido detectar la fosforilación de Rab8 endógeno debido a la pobre selectividad de anticuerpos disponibles para Rab8 total. Por lo tanto, decidimos enfocarnos en la fosforilación Rab10. LRRK2 fosforila Rab10 en Thr73 ubicación en el centro de la región altamente conservada "interruptor II". Conservación de los sitios de fosforilación entre proteínas Rab podría ser una de las razones por qué anticuerpos phosphospecific reconociendo distintas proteínas Rab son difíciles de hacer.
La fosforilación de Rab8A por LRRK2 inhibe la Unión de Rabin8, un factor de intercambio del nucleótido guanina (GEF) que activa Rab8A intercambiando el PIB consolidado con GTP15. Fosforilación de Rab10 y Rab8A por LRRK2 también inhibe la Unión de los inhibidores de la disociación de PIB (GDIs), que son esenciales para la activación de las proteínas Rab extrayendo proteínas Rab PIB-limite de membranas15. Colectivamente, se presume que la fosforilación de proteínas Rab por LRRK2 les impide la activación aunque el mecanismo molecular exacto y consecuencias fisiológicas de la fosforilación no quedan claros.
P-etiqueta SDS-PAGE fue inventado por Kinoshita et al. en 2006: en este método, acrilamida covalente fue juntada con la etiqueta P, una molécula captura de fosfatos con alta afinidad, que copolymerized en SDS-PAGE gel17. Porque las moléculas de P etiqueta en un gel de SDS-PAGE selectivamente retardan la movilidad electroforética de las proteínas fosforiladas, etiqueta P SDS-PAGE puede separar proteínas fosforiladas de los no-phosphorylated (figura 1). Si el proteína de interés es fosforilado en residuos múltiples, se observará una escalera de bandas correspondientes a las formas fosforiladas diferencialmente. En el caso de Rab10, observamos sólo una banda cambiada de puesto, lo que indica que Rab10 es fosforilado en Thr73. La gran ventaja de la etiqueta P SDS-PAGE en immunoblotting con anticuerpos específicos de fosforilación es que Rab10 fosforilada puede detectarse por immunoblotting con fosforilación-anticuerpos inespecíficos (es decir, reconocimiento total Rab10) Tras ser trasladado en las membranas, que suele ser más específicos, sensibles y disponibles de fuentes comerciales y académico. Otra ventaja de usar la etiqueta P SDS-PAGE es que se puede obtener una estimación aproximada de la estequiometría de la fosforilación, que es imposible por immunoblotting con anticuerpos específicos de fosforilación o por el etiquetado metabólico de las células con radiactivo fosfatos.
Aparte del uso de acrilamida etiqueta P barata y algunas modificaciones menores relacionados con ella, el método actual de detección de Rab10 fosforilación de LRRK2 sigue un protocolo general de immunoblotting.Por lo tanto, debe ser sencillo y fácilmente ejecutable en cualquier laboratorios donde immunoblotting es una práctica habitual, con cualquier tipo de muestras incluyendo proteínas purificadas, lysates de la célula y homogenados de tejido.
Protocolo
1. preparación para la SDS-PAGE de etiqueta P
- Quitar y desechar el medio de los platos de 10 cm en la que las células se cultivan utilizando una aspiración y lavar las células con tamponada con fosfato solución salina de 5 mL Dulbecco (DPBS) por primer DPBS agregando al lado de los platos para evitar perturbar la capa de células y manualmente los platos detrás de la roca y varias veces.
- Retire y deseche la DPBS mediante una succión y añadir 2 mL de tripsina 0.25% (p/v) diluido en DPBS y con cuidado los platos para cubrir la capa de células. Poner los platos en un incubador de CO2 (37 ° C, aire humidificado, 5% CO2) durante 5 minutos.
- Después de su uso y con una pipeta desechable para romper las células separadas, recoger la suspensión de células en un tubo de 15 mL y medir la densidad de células usando un hematocitómetro bajo un microscopio18.
- Diluir las células 2,5 × 105 células/mL con medio de Eagle modificado de Dulbecco (DMEM) suplidas con 10% (v/v) de suero bovino fetal (FBS), 100 U/mL de penicilina y 100 de μg/mL estreptomicina. Añadir 2 mL (5 × 105 células) de la suspensión celular diluido en cada pocillo de las placas de 6 pozos.
- Crecen las células durante la noche en un incubador de CO2 (37 ° C, aire humidificado, 5% CO2).
- Transfección en células HEK293
Nota: Si la fosforilación de Rab10 endógeno debe ser examinado, proceda al paso 1.7. Plásmidos utilizados en este protocolo se pueden obtener de los autores a petición. Véase Tabla de materiales para obtener información breve y Figura S1 para su DNA secuencias.- Alícuota a 200 μL de DMEM en tubos de 1,5 mL.
- A cada tubo, agregue ambos 0.266 μg (concentración final de 1.33 μg/mL) de un plásmido de codificación HA-Rab10 y 1.066 μg (concentración final de 5.33 μg/mL) de un plásmido codificación 3 × LRRK2 bandera de stocks de plásmido de 500 μg/mL. Luego añadir 4 μL de solución 1 mg/mL de polietilamina (disuelto en 20 mM HEPES-NaOH, pH 7.0) y mezclar inmediatamente la solución con un vórtex durante 5 s.
- Deje que los tubos reposar a temperatura ambiente durante 10 min y añadir el contenido de un tubo de gota a gota a un bien con una micropipeta. Manualmente y suavemente rock las placas de cultivo y hacia atrás varias veces para dejar la mezcla de transfección difusa uniformemente a lo largo del pozo.
- Que las células crecen para otro 24-36 h en un incubador de CO2 (37 ° C, aire humidificado, 5% CO2).
- Si la fosforilación de Rab10 endógeno debe ser examinado, tratar células con y sin inhibidores de LRRK2 para 1 h antes de la lisis de las células.
- Preparar las soluciones madre de LRRK2 inhibidores disolviendo los inhibidores en dimetil sulfóxido (DMSO) al 10 mM. Se recomienda utilizar MLi-2 y GSK2578215A, que son altamente específicos y potentes inhibidores de LRRK2. Almacene las soluciones stock a-80 ° C.
- Preparar las acciones de trabajo de los inhibidores diluyendo las soluciones stock con DMSO: por MLi-2, preparar 10 μm y 30 LRRK2 endógeno y sobreexpresado, respectivamente. Para GSK2578215A, prepare 1 mM y 3 mM para LRRK2 endógeno y sobreexpresado, respectivamente.
- Añadir 2 μl de las acciones de trabajo de MLi-2 o GSK2578215A a la mitad de uno con una micropipeta y con cuidado la placa manualmente hacia adelante y hacia atrás varias veces para dejar los inhibidores difusa uniformemente a lo largo del pozo.
- Añadir 2 μl de DMSO a un pozo diferente como un control negativo en una manera similar a los del inhibidor en paso 1.8.3.
- Poner las placas a la incubadora y las células de 1 h de la cultura.
- Lisar las células.
- Poner las placas de hielo. Retire y deseche los medios de comunicación. Lavar las células por primera agregando 2 mL DPBS al lado de los platos para evitar molestar a la célula de la capa y roca manualmente los platos hacia adelante y hacia atrás varias veces.
- Quite y deseche la DPBS y agregue a la células 100 μl del buffer de lisis (50 mM Tris-HCl de pH 7,5, 1% (v/v) polyoxyethylene(10) Octylpheny éter, 1 mM EGTA (glicol de etileno-bis(2-aminoethylether)-N, N, N', N'-ácido etilendiaminotetraacético), ortovanadato de sodio 1 mM, 50 mM fluoruro de sodio, glicerofosfato-β de 10 mM, pirofosfato de sodio 5 mM, 0,1 μg/mL microcistina-LR, 270 mM sacarosa, cóctel del inhibidor de la proteasa).
- Inclinación de las placas de hielo y raspe las células usando un raspador celular reunir tanto celular lysate como sea posible. Recoger los lisados utilizando una micropipeta en tubos de 1,5 mL (previamente enfriados en hielo).
PRECAUCIÓN: Microcistina-LR puede ser fatal si se traga o contacto con la piel.
- Dejar los tubos reposar en hielo durante 10 min para lisis completa. Aclarar los lysates de la célula por centrifugación (20.000 × g, 10 min a 4 ° C) y transferir el sobrenadante a nuevos tubos de 1.5 mL previamente enfriados en hielo.
- Medir la concentración de proteína (μg/μl) de los lisados despejados por el análisis de Bradford.
- Preparar albúmina de suero bovino (BSA) normas (0.2, 0.4, 0.6, 0.8 y 1 mg/mL) diluyendo la solución con agua destilada. Diluir los lysates de la célula autorizado por 20-fold con agua destilada.
- Poner 5 μL/pocillo de las normas de la BSA, blanco (agua destilada) y cada célula diluido lisado en una placa de 96 pocillos por triplicado.
- Añadir 150 μL/pocillo del reactivo de ensayo de Bradford, utilizando una micropipeta de 12 canales y dejar la placa reposar a temperatura ambiente durante 5 minutos.
- Medir la absorbancia a 595 nm en un lector de placas y comparar con estándares de BSA.
- 100 μl de las muestras se preparan para SDS-PAGE. La concentración de proteína de las muestras es de 1 μg/μl para HA-Rab10 sobreexpresado y 2 μg/μl de Rab10 endógena.
- Las concentraciones cuantificadas de paso 1.11.4, calcular el volumen (μL) de los lysates de la célula equivalente a 100 μg (sobreexpresada HA-Rab10) o 200 μg (Rab10 endógena) dividiendo la cantidad de proteína (μg 100 o 200 μg) por la concentración de proteína de lisados (μ g/μL).
- Añada 25 μl del tampón de muestra de SDS-PAGE de Laemmli 4 × (62,5 mM Tris-HCl, pH 6.8, 8% (p/v) de SDS, glicerol 40% (v/v), azul de bromofenol 0,02% (w/v), 4% (v/v) β-mercaptoetanol) a nuevos tubos de 1,5 mL almacenados temperatura ambiente.
- Agregar el volumen calculado de la célula lisados a cada tubo y mezclan con un vórtex durante 5 s a temperatura ambiente.
- Llevar el volumen total a 100 μl con el tampón de lisis y mezclar con un vórtex durante 5 s a temperatura ambiente.
- Suplemento con 10 mM MnCl2 para calmar al agente quelante. Añadir 1 μl de 500 mM MnCl2. Mezclar con un vórtex durante 5 s a temperatura ambiente.
Nota: Las muestras que contienen MnCl2 pueden no ser convenientes para el normal de SDS-PAGE.
2. fundición geles de SDS-PAGE de etiqueta P
Nota: Geles deben hacerse el mismo día como correr los geles. Los geles pueden realizarse bajo condiciones de luz ambiente.
- Preparar solución stock de acrilamida 5 mM etiqueta P disolviendo primero la 10 mg de polvo sólido etiqueta P acrilamida completamente con 100 μl de metanol y luego traer a 3.3 mL añadiendo agua bidestilada.
Nota: La acrilamida de la etiqueta P es sensible a la luz. La solución preparada debe almacenarse en la oscuridad a 4 ° C hasta su uso. - Placas limpio sencillo y entalladas rociar etanol al 70% y limpiándola con una toalla de papel. Montar las placas de gel. Las dimensiones de las placas de gel usadas en este protocolo particular son 80 o 100 mm de largo por 100 mm de ancho. Separadores de silicona limpia se ponen entre el llano y con muescas de la placas y las placas ensambladas se sujetan con clips binder.
Nota: Puede utilizarse cualquier tipo de placas de gel de SDS-PAGE ordinario. - Poner un peine que utiliza (17-pozo canutillo) en las placas de gel montado y marca en la placa la posición de la parte inferior de los pocillos del gel con un marcador permanente.
- Preparar 10 mL de mezcla de gel de acrilamida de 10% (10% (p/v) acrilamida (acrylamide:bis-acrilamida = 29: 1), 375 mM Tris-HCl (pH 8.8), 0.1% (p/v) de SDS) en un tubo de 15 mL.
Nota: La óptima concentración de acrilamida puede variar dependiendo de los reactivos usados. Tetramethylethylenediamine (TEMED) y amonio persulfato (APS) no se debe agregar en este punto. - Añadir 100 μl de acrilamida de etiqueta P de 5 mM y 10 μl de 1 M MnCl2 soluciones en concentraciones finales de 50 μm y 100 μm, respectivamente.
Nota: Las concentraciones óptimas de la etiqueta P acrilamida y MnCl2 también pueden variar dependiendo de los reactivos usados. - Añadir 15 μl de TEMED a la mezcla del gel a una concentración final de 0,15% (v/v) y luego 50 μl de 10% (p/v) APS a una concentración final de 0.05% (w/v). Mezclar bien agitando suavemente el tubo para 5 s y verter en las placas montadas inmediatamente hasta una altura que es 2 mm más abajo que la posición marcada en el paso 2.3.
- Suavemente de la capa 2-propanol en la solución de gel para aplanar la parte superior del gel de separación.
- Dejar los geles reposar 30 min a temperatura ambiente. No es necesario proteger a los geles de la luz.
Nota: Puede tardar más tiempo para los geles fijar bajo temperatura ambiente frío. Desgasificación de la mezcla de gel antes de agregar el TEMED y el APS ayuda a acelerar este paso. Para ello, la mezcla de gel se puede preparar en un matraz Erlenmeyer de 100-200 mL conectado a una succión. Desgasificar la solución durante 10 minutos. - Retire la capas 2-propanol absorbiendo con una toalla de papel.
- Lavado del espacio superior de los geles por llenar el espacio con agua destilada de una botella de lavado y descarte el agua vertiendo apagado en Cuenca. Repita el lavado 3 veces.
- Eliminar el agua residual que queda en el espacio superior de los geles absorbiendo con una toalla de papel.
- Preparar 3 mL de mezcla de gel de acrilamida de 4% (4% (w/v) acrilamida (a: crylamide:bis-acrilamida = 29: 1), 125 mM Tris-HCl (pH 6.8), 0.1% (p/v) de SDS) en un tubo de 15 mL.
Nota: No agregue etiqueta P acrilamida o MnCl2 solución a la mezcla de gel de apilamiento. - Agregar 7.5 μl de TEMED y 24 μl de 10% (p/v) APS en concentraciones finales de 0.25% (v/v) y 0.08% (w/v), respectivamente. Mezclar bien agitando suavemente el tubo para 5 s y vierta la mezcla en la parte superior del gel de separación. Inmediatamente poner peines apropiados (canutillos de plástico de 17 pozos que acomodar hasta 25 μl de las muestras, por ejemplo).
- Dejar los geles reposar 30 min a temperatura ambiente. No es necesario proteger a los geles de la luz. Después el gel de apilamiento, ejecutarlos sin almacenaje adicional.
3. SDS-PAGE e Immunoblotting
- Quitar los peines de los geles. Luego retire los espaciadores de silicio y luego los clips de los geles.
- Poner los geles fundidos en gel tanques y arreglar el gel al tanque de sujeción con los clips de la carpeta.
- Vierta el buffer corriente (25 mM Tris, 192 mM glicina, 0.1% (p/v) de SDS) en la parte inferior y superior de los geles. Limpieza pozos por descarga el buffer corriente utilizando una jeringa de 5 mL y una aguja de 21G para eliminar piezas de gel.
- Eliminar burbujas de aire desde el espacio de la parte inferior de los geles con una aguja doblada unida a una jeringa. Para hacer una aguja doblada, doble una aguja de 21G en medio de la aguja manualmente para que el ángulo entre la punta y la base de la aguja se convierte en 30-45 °.
- Desactivación de precipitados causados por la adición de MnCl2 a 20.000 x g durante 1 min a temperatura ambiente para obtener muestras de claras.
- Carga de 10 μg proteínas para la detección de la fosforilación de Rab10 sobreexpresado y 30 μg proteínas de Rab10 endógena.
Nota: Es crítico para cargar un volumen igual de muestras en todos los pocillos. Calles vacías deben colocarse con el tampón de muestra de 1 × Laemmli SDS-PAGE. Si las muestras contienen MnCl2, añadir la misma concentración de MnCl2 para las muestras simuladas. - Bien cargado con un marcador de peso molecular (MWM) debería también complementarse con tampón de SDS-PAGE de Laemmli 1 × así que el volumen de muestras cargados es igual en todos los pocillos.
Nota: Una vez más, si las muestras contienen MnCl2, añadir la misma concentración de MnCl2 al MWM. Como alternativa, puede usarse un MWM libre de EDTA. - Correr los geles a 50 V para apilar (aproximadamente 30 minutos) hasta que el frente de tinte se cruza en el gel de separación.
- Después de las muestras de la pila, cambiar el voltaje a 120 V para la separación hasta que la parte delantera del tinte llega a la parte inferior de los geles (aproximadamente 50 y 80 min de 80 y 100 mm de longitud y geles, respectivamente).
Nota: Se espera que el patrón de migración de la MWM sea distinto en geles de SDS-PAGE normales. No debe utilizarse para estimar el peso molecular de proteínas en geles de SDS-PAGE de etiqueta P pero puede utilizarse para comprobar la reproducibilidad de los geles de la etiqueta P. Consulte la discusión para más detalles. - Lave los geles para quitar MnCl2 de los geles.
- Vierta a la transferencia búfer (48 mM Tris, 39 mM glicina, 20% (v/v) de metanol) que contiene 10 mM EDTA y 0.05% (w/v) SDS en un recipiente (por ejemplo, grandes barcos de pesaje).
Nota: Utilice buffer fresca para cada lavado.
- Coloque un papel de filtro (10 x 7 cm) sobre una almohadilla de esponja para la transferencia. Coloque el gel en el papel de filtro. Asegúrese de que no hay burbujas de aire entre el papel de filtro y el gel.
- Poner una membrana (10 x 7 cm) sobre el gel y asegúrese de que no hay burbujas de aire entre el gel y la membrana.
- Poner otro papel de filtro (10 x 7 cm) en la membrana y, una vez más, asegúrese de que no hay burbujas de aire entre la membrana y el papel de filtro.
- Coloque otra almohadilla de esponja en el papel de filtro. Poner la pila de papeles de filtro de membrana en un cassette para la transferencia.
- Poner el cassette en un tanque de la transferencia, asegúrese de que la membrana se encuentra entre el gel y el ánodo cargado positivamente.
- Conecte el tanque a una fuente de alimentación y poner el depósito en una caja de espuma de estireno llenada con agua helada. Iniciar transferencia a 100 V durante 180 minutos.
Nota: Traslado prolongado es necesario puesto que la transferencia de proteínas de geles de SDS-PAGE de etiqueta P no son tan eficientes que de normales geles de SDS-PAGE. Enfriamiento eficiente es esencial para evitar que se derritan los geles durante la transferencia.
- Quite las membranas de los geles con unas pinzas y remoje las membranas en una solución de Ponceau S (0.1% (peso/volumen) Ponceau S, ácido acético al 5% (v/v)) para teñir proteínas transferidas en las membranas en un recipiente de plástico. El volumen de la solución debe ser suficiente para cubrir una membrana.
- Incubar las membranas durante 1 min a temperatura ambiente, meciendo manualmente.
Nota: Una escalera de bandas debe ser visible en cada carril. - Recoger la membrana de la solución de tinción con pinzas y ver si la escalera de bandas tiene patrón uniforme y la intensidad en cada carril donde las muestras hayan sido cargadas.
- Después de comprobar la coloración, retirar la solución de tinción y añadir tampón TBST (20 mM Tris-HCl pH 7.4, 150 mM NaCl, 0,1% (v/v) polyoxyethylenesorbitan monolaurato). El volumen del buffer debe ser suficiente para cubrir una membrana.
Nota: La solución de Ponceau S puede recogerse y reutilizarse varias veces. - Roca de las membranas en TBST en un agitador oscilante (~ 60 rpm) a temperatura ambiente hasta que no queden bandas visibles en las membranas.
- Repetir el lavado con TBST fresco durante 5 minutos.
Nota: Tiempo de exposición puede variar dependiendo de la solución de ECL y el sistema utilizado para la detección de la quimioluminescencia.
- Activar un sensor equipado con un dispositivo de carga acoplada (CCD) de cámara y un ordenador conectado al reproductor de imágenes. Inicie el software de control para el reproductor de imágenes. Espere hasta que alcanza la temperatura de la cámara CCD-25 ° C.
- Poner 1 mL de la solución de ECL para una membrana en una envoltura de plástico extendido en un banco.
- Poner una membrana gel hacia arriba en la solución de ECL y entonces rápidamente Dale la vuelta para que ambos lados de la membrana están cubiertos con la solución.
- Recoger la membrana y escurrir dejando a un lado del toque de la membrana una toalla de papel para 5 s.
- Poner la membrana entre películas claro (por ejemplo, bolsas de papel claro).
Nota: No se recomienda papel plástico para envolver las membranas. Las películas claro utilizadas para envolver la membrana deben ser tan plano como sea posible sin arrugas visibles para evitar el fondo irregular. - Coloque la membrana sobre una bandeja negra. Poner la bandeja en el reproductor de imágenes y cierre la puerta.
- Haga clic en el botón de "Enfoque" en la ventana del software de control. Comprobar que la membrana esté correctamente posicionada. Haga clic en el botón "Volver".
- Seleccione "Precisión" para "Tipo de exposición". Seleccione "Manual" para "Tiempo de exposición" y establezca el tiempo de exposición en 1 s.
- Seleccione "Alto" para la "Resolución de sensibilidad". Dejar "Añadir la digitalización imagen" desactivada. Haga clic en el botón "Start" para tomar una imagen.
- Guardar la imagen que apareció en la pantalla de la computadora como un archivo TIFF.
- Repetir la toma de imágenes con el tiempo de exposición de 1, 3, 10, 30, 60, 90, 120, 150 s y 180 s. Cuando se toma la última imagen, compruebe "Añadir la digitalización imagen" por lo que la imagen digital, no la quimioluminescencia, de la membrana puede tomarse simultáneamente.
- Seleccionar la mejor imagen con mayor rango dinámico y sin ninguna saturación píxeles (mostrados en rojo) en los grupos de interés.
Nota: Las películas de radiografía convencional también pueden utilizarse para detección19.
Resultados
Sistema de sobreexpresión: Fosforilación de la HA-Rab10 por 3 × bandera-LRRK2 en HEK293 células:
Las células HEK293 fueron transfectadas con 0.266 μg de HA-Rab10 tipo de salvaje y 1.066 μg 3 × bandera LRRK2 (mutante de tipo salvaje, quinasa inactiva (K1906M), o mutantes FPD). Fosforilación de Rab10 fue examinada por etiqueta P SDS-PAGE seguido immunoblotting usando un anti-HA anticuerpo (figura 2). 10 μg de proteínas se corrieron en un gel 10% (80 x 100 x 1 mm) que contiene 50 acrilamida de etiqueta P μm y 100 μm MnCl2. El tiempo de exposición para el gel de la etiqueta P (panel superior) fue 10 s con una solución estándar de ECL. Co-sobreexpresión de LRRK2 con Rab10 causado cambio de banda en el gel de la etiqueta P (marcado con un círculo abierto en el panel superior; comparar carriles 2 y 4). En cambio, coexpresión con un mutante quinasa inactiva LRRK2 (K1906M) no se pudo cambiar la movilidad de los Rab10 (Comparar con carriles 4 y 5), lo que indica que el cambio de banda es debido a la actividad de la cinasa de LRRK2. El cambio de la banda fue aumentado en todas las mutaciones de FPD (Comparar con calles 4 y 6-13) acuerdo con el informe anterior15.
Sistema endógeno: Fosforilación de la Rab10 de LRRK2 en células cultivadas:
Células de fibroblastos embrionarios de ratón 3T3-Swiss albino fueron tratadas con o sin un inhibidor (GSK2578215A) de LRRK220 a una concentración final de 1 μm para 1 h (Figura 3A). Células pulmonares humanas A549 fueron tratadas con o sin otro LRRK2 inhibidor (MLi-2)21 a una concentración final de 10 nM para 1 h (figura 3B). Endógeno Rab10 fosforilación de LRRK2 endógena fue examinada por etiqueta P SDS-PAGE seguido immunoblotting con un anticuerpo anti-Rab10. 30 μg de proteínas se corrieron en un gel 10% (100 x 100 x 1 mm para las células 3T3-Swiss albino) y 80 mm para las células A549 con 50 etiqueta P acrilamida μm y 100 μm MnCl2. La exposición para el gel de la etiqueta P (panel superior) en la Figura 3A y 3B figura fueron 3 min y 90 s, respectivamente. Se observó el cambio de la banda correspondiente a Rab10 endógeno fosforilada en el gel de la etiqueta P (marcado con un círculo abierto en el panel superior; comparar carriles 1-2 y 3-4) que desapareció sobre el tratamiento de células con GSK2578215A o MLi-2. La eficacia de los inhibidores de LRRK2 fue validada por inmunotransferencia utilizando el anticuerpo anti-pSer935 del LRRK2 (el tercer panel de la parte inferior), que es una lectura bien establecida de la inhibición de LRRK2 en células22.

Figura 1. Representación esquemática de la etiqueta P SDS – PAGE. Cuando una mezcla de fosforilada (círculos rojos) y las proteínas no-phosphorylated (círculos azules) (e.g., Rab10) corre a través de un gel de acrilamida etiqueta P se co polimerizada, la movilidad de las proteínas fosforiladas sólo retarda debido a la fuerte interacción de las proteínas fosforiladas con las moléculas de la etiqueta P (flechas quebradas). Ya que Rab10 es fosforilado por LRRK2 en un solo residuo de Thr73, Rab10 funciona como dos bandas: la banda superior representa Rab10 fosforilada y la banda inferior representa Rab10 no-phosphorylated. Cuando las células son tratadas con inhibidores de LRRK2, la banda superior debe desaparecer. Haga clic aquí para ver una versión más grande de esta figura.
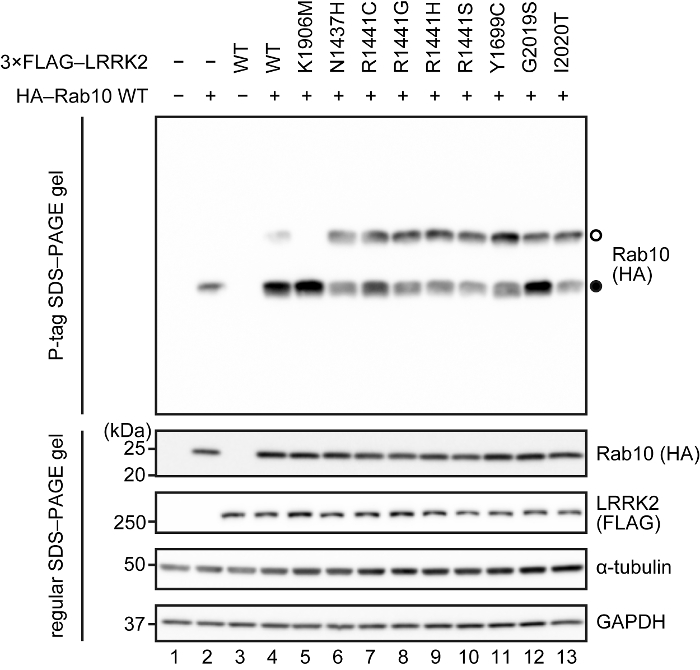
Figura 2. Resultado representativo de la mancha etiqueta P: sistema sobreexpresión. El panel superior muestra la mancha blanca /negra de la etiqueta P usando un anti-HA anticuerpo, donde Rab10 fosforilados y no fosforilados son marcados con abra (○) y cerrado círculos (●), respectivamente. Los paneles se muestra a continuación la mancha etiqueta P son immunoblots en geles de SDS-PAGE normales usando un anti-HA, anti-FLAG, anti-α-tubulina y anticuerpos anti-GAPDH. La mancha HA y bandera blot se muestran para asegurar la sobreexpresión de Rab10 HA y 3xFLAG-LRRK2, respectivamente. La α-tubulina y GAPDH lacras aparecen para asegurar la carga igual. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3. Resultado representativo de la mancha etiqueta P: sistema endógeno. Dos células, a saber: (A) albino Swiss 3T3 células y células A549 (B), fueron utilizados. En ambas figuras, los paneles superiores muestran que la etiqueta P blots utilizando un anticuerpo anti-Rab10, donde Rab10 endógeno fosforilados y no fosforilados se marcan con el abrir (○) y cerrado círculos (●), respectivamente. Los paneles que se muestra a continuación la bienvenida de etiqueta P son immunoblots en geles de SDS-PAGE normales usando un anti-Rab10 anti-pSer935 LRRK2, anti-LRRK2 y anticuerpos anti-α-tubulina. La Rab10 y LRRK2 aparecen para garantizar la expresión similar de endógeno Rab10 y LRRK2 entre las muestras, respectivamente. La pSer935 LRRK2 blot se muestran para asegurar que GSK2578215A (A) y MLi-2 (B) trabajó como se esperaba. La mancha de la α-tubulina se muestran para asegurar la carga igual. Las imágenes de las manchas blancas /negras de la etiqueta P superpuestas con el marcador de peso molecular se muestran en la Figura S4. Haga clic aquí para ver una versión más grande de esta figura.
Figura S1. Secuencias de ADN de la plásmidos. Las secuencias de ADN del plásmido codificación FRT a HA-Rab10/pcDNA5 y el de 3 × × p3-LRRK2/bandera bandera-CMV-10. Todos los plásmidos utilizados en este protocolo están disponibles de los autores a petición. Haga clic aquí para descargar este archivo.
Figura S2. Un ejemplo de la optimización de la etiqueta P SDS-PAGE. El mismo conjunto de muestras que se utiliza en la Figura 3A se utilizó para la optimización de la etiqueta P SDS-PAGE. Se analizaron cuatro diferentes condiciones como se muestra en la figura. Las bandas correspondientes a Rab10 fosforilados y no fosforilados se destacan con rectángulos de línea sólida y punteada, respectivamente.En base a este experimento, la condición con 10% de acrilamida, acrilamida de etiqueta P de 50 μm y 100 μm MnCl2 dio la mejor separación de las dos bandas, tanto localizar en medio del gel. Haga clic aquí para descargar este archivo.
Figura S3. Comparación del patrón de migración de un marcador de peso molecular de. El marcador de peso molecular (MWM) era correr en un gel de SDS-PAGE regular (panel de la izquierda) o en un gel de SDS-PAGE de la etiqueta P (panel central), y los geles fueron teñidos mediante tinción de Coomassie. El panel de la derecha muestra un immunoblot de las muestras utilizadas en la figura 2 (carril 4 y 5) en el mismo gel de SDS-PAGE de etiqueta P como el panel central para mostrar las posiciones de Rab10 fosforilados y no fosforilados. La punta de flecha en el lado derecho del immunoblot indica la posición de uno de lo MWM en el gel SDS-PAGE de la etiqueta P. Haga clic aquí para descargar este archivo.
Figura S4. La posición de un marcador de peso molecular en geles de SDS-PAGE de la etiqueta P. Imágenes digitalizadas de las membranas que se utiliza para figuras 3A y 3B de la figura se muestran como (A) y(B), respectivamente. Los immunoblots que se muestra en la figura 3 se superponen para mostrar la posición relativa del marcador de peso molecular (MWM). El MWM no transferencia bien a las membranas pero el marcador (flecha) Figura S3 fue débilmente pero consistentemente observado. La posición de la MWM está marcada con líneas punteadas. Tenga en cuenta que carriles irrelevantes las cifras entre lo MWM y los carriles de interés han sido tachados. Haga clic aquí para descargar este archivo.
Discusión
Aquí, describimos un método robusto y fácil de detectar Rab10 fosforilación de LRRK2 nivel endógeno basado en la metodología de la etiqueta P. Porque el anticuerpo actualmente disponible contra Rab10 fosforilada sólo funciona con las proteínas sobreexpresadas15, el presente método utiliza la etiqueta P SDS-PAGE es la única manera de evaluar los niveles endógenos de la fosforilación Rab10. Por otra parte, el presente método permite la estimación de la estequiometría de la fosforilación en las células Rab10. Porque la metodología de la etiqueta P es genéricamente aplicable a fosfo-proteínas, el presente Protocolo puede ser un "prototipo" para el establecimiento de métodos similares para otros fosfo-proteínas.
Pasos críticos en el protocolo son geles de casting y preparación de muestras. Acrilamida de la etiqueta P es un reactivo relativamente lábil foto y la capacidad de retardar la movilidad electroforética de las proteínas fosforiladas a veces se pierde después de almacenamiento a largo plazo a 4 ° C. Los investigadores deben tomar cada cuidado para evitar la exposición a acrilamida etiqueta P a la luz. Además, la molécula de P-etiqueta necesita formar un complejo con iones2 + Mn para capturar los fosfatos. Por lo tanto, las muestras no deben contener a agentes quelantes como el EDTA, que son generalmente componentes comercialmente disponibles buffer de muestra de SDS-PAGE. Se recomienda utilizar el tampón de muestra de Laemmli clásica para preparar muestras para etiqueta P SDS-PAGE.
Otro punto crítico es bien optimizar las concentraciones de acrilamida, etiqueta P acrilamida y MnCl2 en la mezcla del gel de separación (Figura S2). Las distancias de migración de bandas fosforiladas y no fosforiladas variará dependiendo de los reactivos utilizados (lote, pureza, etc.), y la optimización de las concentraciones adecuadas probando varias concentraciones diferentes en combinación es obligatorio (por ejemplo, etiqueta P acrilamida (25, 50, 75 μm), MnCl2 (1:2 o 1:3 relación molar a acrilamida etiqueta P) y acrilamida (7,5, 10, 12,5%)). Las bandas fosforiladas y no fosforiladas aparecen en medio de los geles y bien separaron entre sí. Para el proceso de optimización, se recomienda utilizar Rab10 sobreexpresado porque la banda cambió de puesto es fácilmente detectable. Los autores pueden proporcionar muestras de control para este protocolo.
Uno de la confusión más grande que puede causar este Protocolo será debido a la diferencia de los patrones de migración de la MWM entre regular y geles de SDS-PAGE de la etiqueta P. Como se muestra en la Figura S3, los patrones de migración de MWM en regular y geles de SDS-PAGE de la etiqueta P (ambas 10%) son muy diferentes y uno no puede usar el MWM para estimar el peso molecular de proteínas en geles de SDS-PAGE de la etiqueta P. Sin embargo, en geles de SDS-PAGE de la etiqueta P, uno de lo MWM destaca por migrar a la mitad del gel (flecha en Figura S3), que constantemente se observa entre geles (véase la Figura S4). Este MWM siempre migra entre las bandas de Rab10 fosforilados y no fosforilados. Por lo tanto, este marcador es útil no sólo para garantizar que la etiqueta P SDS-PAGE funciona antes de la transferencia, sino también como un punto de referencia para tener una sensación áspera de donde se verá las bandas de Rab10 fosforilados y no fosforilados.
Para la detección de bandas immunoblots, hemos utilizado un sensor equipado con una cámara CCD refrigerada (véase Tabla de materiales) así como una radiografía convencional película sistema en desarrollo y encontró que ambos sistemas trabajan bien. Para la detección de niveles endógenos de la fosforilación Rab10 en cultivos de células, es necesario utilizar líneas celulares que tienen niveles de expresión alta de LRRK2 endógenos, tales como fibroblastos embrionarios de ratón (ya sea primaria cultivadas o inmortalizado (líneas) de la célula Albino Swiss 3T3, etc.)) y las células de pulmón humano derivadas de carcinoma A549. Para la detección de niveles endógenos de la fosforilación Rab10 en tejidos de ratón, se recomienda utilizar el pulmón16.
La cuantificación de la fosforilación Rab10 no es más allá de la habitual immunoblotting. Si la estequiometría de la fosforilación Rab10 es muy baja (como se muestra en la figura 3), la intensidad de la banda no-phosphorylated (marcado con un círculo cerrado) tiende a ser muy por encima del nivel de saturación, haciendo la determinación precisa de la estequiometría imposible. Sin embargo, estimación cualitativa es posible en cualquier caso, que no es obtenible sin utilizar el método actual. Puesto que la detección de la fosforilación de Rab10 endógeno requiere una exposición prolongada, tiende a haber algunas bandas no específicos que aparecen en la etiqueta P blot aunque el anticuerpo anti-Rab10 usado en este protocolo da immunoblots bastante limpios para el uso normal . Para distinguir las proteínas phosphorylated de bandas inespecíficas, es fundamental para utilizar un control de tratamiento del inhibidor, en el cual bandas fosforiladas, pero bandas no inespecíficos, deben desaparecer. LRRK2 fosforila Rab8 además de Rab1015, y nos hemos aplicado con éxito el presente Protocolo a Rab8A así (datos no mostrados). El presente Protocolo potencialmente puede utilizarse para examinar la fosforilación de alguna proteínas, aunque puede haber dificultades técnicas si la proteína es grande (> kDa 100) o multiplicar fosforilada. Porque Rab10 es una proteína pequeña (25 kDa) y fosforilada por separado en Thr73 de LRRK2, el resultado de la etiqueta P SDS-PAGE es simple donde se observa una única banda cambiada de puesto.
En un futuro próximo, será fundamental para establecer el método para detectar cuantitativamente la actividad quinasa de LRRK2 en muestras de paciente deriva de una forma de alto rendimiento para evaluar la alteración de la actividad quinasa de LRRK2 en pacientes con EP, así como evaluar el efecto de las drogas en LRRK2 en ensayos clínicos. Desde sangre periférica células mononucleares (PBMCs) expresan niveles relativamente altos de endógeno LRRK223, PBMCs o más células aisladas de sangre será prueba para fosforilación endógena de Rab10. El presente método no sólo ser útil en la investigación de la biología básica de la LRRK2 vía de señalización, sino que también ayuda en la obtención de un prueba de concepto básico para decidir que muestra en muestras derivadas de paciente debe ser analizado en estos estudios a gran escala.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos a Dr. Takeshi Iwatsubo (Universidad de Tokio, Japón) por proporcionar amablemente la plásmidos codifican 3xFLAG LRRK2 WT y mutantes. También agradecemos a Dr. Dario Alessi (Universidad de Dundee, Reino Unido) por proporcionar amablemente MLi-2 y el plásmido codificación HA-Rab10. Este trabajo fue apoyado por la sociedad japonesa para la promoción de la ciencia (JSPS) KAKENHI concesión número JP17K08265 (G.I.).
Materiales
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Dulbecco's phosphate-buffered saline (DPBS) | homemade | 150 mM NaCl, 8 mM Na2HPO4-12H2O, 2.7 mM KCl, 1.5 mM KH2PO4 in MilliQ water and sterilized by autoclaving | |
| Sodium chloride | Nacalai Tesque | 31320-34 | |
| Sodium Disodium Hydrogenphosphate 12-Water | Wako | 196-02835 | |
| Potassium chloride | Wako | 163-03545 | |
| Potassium Dihydrogen Phosphate | Wako | 169-04245 | |
| 2.5% Trypsin (10X) | Sigma-Aldrich | T4549 | Dilute 10-fold with sterile DPBS for preparing working solution |
| Dulbecco's modified Eagle medium (DMEM) | Wako | 044-29765 | |
| Fetal bovine serum | BioWest | S1560 | Heat-inactivated at 56 °C for 30 min |
| Penicillin-Streptomycin (100X) | Wako | 168-23191 | |
| HEPES | Wako | 342-01375 | |
| Sodium hydroxide | Wako | 198-13765 | |
| Polyethylenimine HCl MAX, Linear, Mw 40,000 (PEI MAX 40000) | PolySciences, Inc. | 24765-1 | Stock solution was prepared in 20 mM HEPES-NaOH pH 7.0 at 1 mg/mL and the pH was then adjusted to 7.0 with NaOH |
| Dimethyl sulfoxide | Wako | 045-28335 | |
| Tris | STAR | RSP-THA500G | |
| Hydrochloric acid | Wako | 080-01066 | |
| Polyoxyethylene(10) Octylphenyl Ether | Wako | 160-24751 | Equivalent to Triton X-100 |
| Ethylene glycol-bis(2-aminoethylether)-N,N,N’,N’-tetraacetic acid (EGTA) | Wako | 346-01312 | |
| Sodium orthovanadate(V) | Wako | 198-09752 | |
| Sodium fluoride | Kanto Chemical | 37174-20 | |
| β-Glycerophosphoric Acid Disodium Salt Pentahydrate | Nacalai Tesque | 17103-82 | |
| Sodium pyrophosphate decahydrate | Kokusan Chemical | 2113899 | |
| Microcystin-LR | Wako | 136-12241 | |
| Sucrose | Wako | 196-00015 | |
| Complete EDTA-free protease inhibitor cocktail | Roche | 11873580001 | Dissolve one tablet in 1 mL water, which can be stored at -20 °C for a month. Use it at 1:50 dilution for cell lysis |
| Pierce Coomassie (Bradford) Protein Assay Kit | Thermo Fisher Scientific | 23200 | |
| Sodium dodecyl sulfate | Nacalai Tesque | 31607-65 | |
| Glycerol | Wako | 075-00616 | |
| Bromophenol blue | Wako | 021-02911 | |
| β-mercaptoethanol | Kanto Chemical | 25099-00 | |
| Ethanol | Wako | 056-06967 | |
| Methanol | Wako | 136-01837 | |
| Phosphate-binding tag acrylamide | Wako | AAL-107 | P-tag acrylamide |
| 40% (w/v) acrylamide solution | Nacalai Tesque | 06119-45 | Acrylamide:Bis = 29:1 |
| Tetramethylethylenediamine (TEMED) | Nacalai Tesque | 33401-72 | |
| Ammonium persulfate (APS) | Wako | 016-08021 | 10% (w/v) solution was prepared by dissolving the powder of ammonium persulfate in MilliQ water |
| 2-propanol | Wako | 166-04831 | |
| Manganese chloride tetrahydrate | Sigma-Aldrich | M3634 | |
| Precision Plus Protein Prestained Standard | Bio-Rad | 1610374, 1610373, 1610377 | Molecular weight marker used in the protocol |
| WIDE-VIEW Prestained Protein Size Marker III | Wako | 230-02461 | |
| Glycine | Nacalai Tesque | 17109-64 | |
| Amersham Protran NC 0.45 | GE Healthcare | 10600007 | Nitrocellulose membrane |
| Durapore Membrane Filter | EMD Millipore | GVHP00010 | PVDF membrane |
| Filter Papers No.1 | Advantec | 00013600 | |
| Ponceau S | Nacalai Tesque | 28322-72 | |
| Acetic acid | Wako | 017-00251 | |
| Tween-20 | Sigma-Aldrich | P1379 | polyoxyethylenesorbitan monolaurate |
| Ethylenediaminetetraacetic acid (EDTA) | Wako | 345-01865 | |
| Skim milk powder | Difco Laboratories | 232100 | |
| Immunostar | Wako | 291-55203 | ECL solution (Normal sensitivity) |
| Immunostar LD | Wako | 290-69904 | ECL solution (High sensitivity) |
| CBB staining solution | homemade | 1 g CBB R-250, 50% (v/v) methanol, 10% (v/v) acetic acid in 1 L of MilliQ water | |
| CBB R-250 | Wako | 031-17922 | |
| CBB destaining solution | homemade | 12% (v/v) methanol, 7% (v/v) acetic acid in 1 L MilliQ water | |
| Name | Company | Catalog Number | Comments |
| Antibodies | |||
| anti-HA antibody | Sigma-Aldrich | 11583816001 | Used at 0.2 μg/mL for immunoblotting. |
| anti-Rab10 antibody | Cell Signaling Technology | #8127 | Used at 1:1000 for immunoblotting. Specificity was confirmed by CRISPR KO in Ito et al., Biochem J, 2016. |
| anti-pSer935 antibody | Abcam | ab133450 | Used at 1 μg/mL for immunoblotting. |
| anti-LRRK2 antibody | Abcam | ab133518 | Used at 1 μg/mL for immunoblotting. |
| anti-α-tubulin antibody | Sigma-Aldrich | T9026 | Used at 1 μg/mL for immunoblotting. |
| anti-GAPDH antibody | Santa-Cruz | sc-32233 | Used at 0.02 μg/mL for immunoblotting. |
| Peroxidase AffiniPure Sheep Anti-Mouse IgG (H+L) | Jackson ImmunoResearch | 515-035-003 | Used at 0.16 μg/mL for immunoblotting. |
| Peroxidase AffiniPure Goat Anti-Rabbit IgG (H+L) | Jackson ImmunoResearch | 111-035-003 | Used at 0.16 μg/mL for immunoblotting. |
| Name | Company | Catalog Number | Comments |
| Inhibitors | |||
| GSK2578215A | MedChem Express | HY-13237 | Stock solution was prepared in DMSO at 10 mM and stored at -80 °C |
| MLi-2 | Provided by Dr Dario Alessi (University of Dundee) | Stock solution was prepared in DMSO at 10 mM and stored at -80 °C | |
| Name | Company | Catalog Number | Comments |
| Plasmids | |||
| Rab10/pcDNA5 FRT TO HA | Provided by Dr Dario Alessi (University of Dundee) | This plasmid expresses amino-terminally HA-tagged human Rab10. | |
| LRRK2 WT/p3xFLAG-CMV-10 | Provided by Dr Takeshi Iwatsubo (University of Tokyo) | Ito et al., Biochemistry, 46: 1380–1388 (2007). This plasmid expresses amino-terminally 3xFLAG-tagged wild-type human LRRK2. | |
| LRRK2 K1906M/p3xFLAG-CMV-10 | Provided by Dr Takeshi Iwatsubo (University of Tokyo) | Ito et al., Biochemistry, 46: 1380–1388 (2007). This plasmid expresses amino-terminally 3xFLAG-tagged K1906M kinase-inactive mutant of human LRRK2. | |
| LRRK2 N1437H/p3xFLAG-CMV-10 | This paper. This plasmid expresses amino-terminally 3xFLAG-tagged N1437H FPD mutant of human LRRK2. | ||
| LRRK2 R1441C/p3xFLAG-CMV-10 | Provided by Dr Takeshi Iwatsubo (University of Tokyo) | Kamikawaji et al., Biochemistry, 48: 10963–10975 (2013). This plasmid expresses amino-terminally 3xFLAG-tagged R1441C FPD mutant of human LRRK2. | |
| LRRK2 R1441G/p3xFLAG-CMV-10 | Provided by Dr Takeshi Iwatsubo (University of Tokyo) | Kamikawaji et al., Biochemistry, 48: 10963–10975 (2013). This plasmid expresses amino-terminally 3xFLAG-tagged R1441G FPD mutant of human LRRK2. | |
| LRRK2 R1441H/p3xFLAG-CMV-10 | Provided by Dr Takeshi Iwatsubo (University of Tokyo) | Kamikawaji et al., Biochemistry, 48: 10963–10975 (2013). This plasmid expresses amino-terminally 3xFLAG-tagged R1441H FPD mutant of human LRRK2. | |
| LRRK2 R1441S/p3xFLAG-CMV-10 | This paper. This plasmid expresses amino-terminally 3xFLAG-tagged R1441S FPD mutant of human LRRK2. | ||
| LRRK2 Y1699C/p3xFLAG-CMV-10 | Provided by Dr Takeshi Iwatsubo (University of Tokyo) | Kamikawaji et al., Biochemistry, 48: 10963–10975 (2013). This plasmid expresses amino-terminally 3xFLAG-tagged Y1699C FPD mutant of human LRRK2. | |
| LRRK2 G2019S/p3xFLAG-CMV-10 | Provided by Dr Takeshi Iwatsubo (University of Tokyo) | Kamikawaji et al., Biochemistry, 48: 10963–10975 (2013). This plasmid expresses amino-terminally 3xFLAG-tagged G2019S FPD mutant of human LRRK2. | |
| LRRK2 I2020T/p3xFLAG-CMV-10 | Provided by Dr Takeshi Iwatsubo (University of Tokyo) | Kamikawaji et al., Biochemistry, 48: 10963–10975 (2013). This plasmid expresses amino-terminally 3xFLAG-tagged I2020T FPD mutant of human LRRK2. | |
| Name | Company | Catalog Number | Comments |
| Equipments | |||
| CO2 incubator | Thermo Fisher Scientific | Forma Series II 3110 Water-Jacketed | |
| Auto Pipette | Drummond | Pipet-Aid PA-400 | |
| Micropipette P10 | Nichiryo | 00-NPX2-10 | 0.5–10 μL |
| Micropipette P200 | Nichiryo | 00-NPX2-200 | 20–200 μL |
| Micropipette P1000 | Nichiryo | 00-NPX2-1000 | 100–1000 μL |
| Tips for micropipette P10 | STAR | RST-481LCRST | Sterile |
| Tips for micropipette P200 | FUKAEKASEI | 1201-705YS | Sterile |
| Tips for micropipette P1000 | STAR | RST-4810BRST | Sterile |
| 5 mL disporsable pipette | Greiner | 606180 | Sterile |
| 10 mL disporsable pipette | Greiner | 607180 | Sterile |
| 25 mL disporsable pipette | Falcon | 357535 | Sterile |
| Hematocytometer | Sunlead Glass | A126 | Improved Neubeuer |
| Microscope | Olympus | CKX53 | |
| 10 cm dishes | Falcon | 353003 | For tissue culture |
| 6-well plates | AGC Techno Glass | 3810-006 | For tissue culture |
| Vortex mixer | Scientific Industries | Vortex-Genie 2 | |
| Cell scrapers | Sumitomo Bakelite | MS-93100 | |
| 1.5 mL tubes | STAR | RSV-MTT1.5 | |
| 15 mL tubes | AGC Techno Glass | 2323-015 | |
| 50 mL tubes | AGC Techno Glass | 2343-050 | |
| Centrifuges | TOMY | MX-307 | |
| 96-well plates | Greiner | 655061 | Not for tissue culture |
| Plate reader | Molecular Devices | SpectraMax M2e | |
| SDS–PAGE tanks | Nihon Eido | NA-1010 | |
| Transfer tanks | Nihon Eido | NA-1510B | |
| Gel plates (notched) | Nihon Eido | NA-1000-1 | |
| Gel plates (plain) | Nihon Eido | NA-1000-2 | |
| Silicon spacers | Nihon Eido | NA-1000-16 | |
| 17-well combs | Nihon Eido | Custom made | |
| Binder clips | Nihon Eido | NA-1000-15 | |
| 5 mL syringe | Terumo | SS-05SZ | |
| 21G | Terumo | NN-2138R | |
| Power Station 1000 VC | ATTO | AE-8450 | Power supply for SDS–PAGE and transfer |
| Large weighing boats | Ina Optika | AS-DL | |
| Plastic containers | AS ONE | PS CASE No.4 | 10 x 80 x 50 mm |
| Rocking shaker | Titech | NR-10 | |
| Styrene foam box | generic | The internal dimensions should fit one transfer tank (200 x 250 x 250 mm). | |
| ImageQuant LAS-4000 | GE Healthcare | An imager equipped with a cooled CCD camera for detection of ECL |
Referencias
- Sveinbjornsdottir, S. The clinical symptoms of Parkinson's disease. J. Neurochem. 139 (Suppl. 1), 318-324 (2016).
- Hernandez, D. G., Reed, X., Singleton, A. B. Genetics in Parkinson disease: Mendelian versus non-Mendelian inheritance. J. Neurochem. 139 (Suppl. 1), 59-74 (2016).
- Paisán-Ruíz, C., et al. Cloning of the gene containing mutations that cause PARK8-linked Parkinson's disease. Neuron. 44 (4), 595-600 (2004).
- Zimprich, A., et al. Mutations in LRRK2 cause autosomal-dominant parkinsonism with pleomorphic pathology. Neuron. 44 (4), 601-607 (2004).
- Gilks, W. P., et al. A common LRRK2 mutation in idiopathic Parkinson's disease. Lancet. 365 (9457), 415-416 (2005).
- Satake, W., et al. Genome-wide association study identifies common variants at four loci as genetic risk factors for Parkinson's disease. Nat. Genet. 41 (12), 1303-1307 (2009).
- Simón-Sánchez, J., et al. Genome-wide association study reveals genetic risk underlying Parkinson's disease. Nat. Genet. 41 (12), 1308-1312 (2009).
- Klein, C., Ziegler, A. Imputation of sequence variants for identification of genetic risks for Parkinson's disease: a meta-analysis of genome-wide association studies. Lancet. 377 (9766), 641-649 (2011).
- Cookson, M. R. The role of leucine-rich repeat kinase 2 (LRRK2) in Parkinson's disease. Nat. Rev. Neurosci. 11 (12), 791-797 (2010).
- Ozelius, L. J., et al. LRRK2 G2019S as a Cause of Parkinson’s Disease in Ashkenazi Jews. N. Engl. J. Med. 354 (4), 424-425 (2006).
- Lesage, S., et al. LRRK2 G2019S as a Cause of Parkinson’s Disease in North African Arabs. N. Engl. J. Med. 354 (4), 422-423 (2006).
- Bouhouche, A., et al. LRRK2 G2019S Mutation: Prevalence and Clinical Features in Moroccans with Parkinson's Disease. Parkinsons. Dis. , 1-7 (2017).
- West, A. B., et al. Parkinson's disease-associated mutations in leucine-rich repeat kinase 2 augment kinase activity. Proc. Natl. Acad. Sci. U. S. A. 102 (46), 16842-16847 (2005).
- Ito, G., et al. GTP binding is essential to the protein kinase activity of LRRK2, a causative gene product for familial Parkinson's disease. Biochemistry. 46 (5), 1380-1388 (2007).
- Steger, M., et al. Phosphoproteomics reveals that Parkinson's disease kinase LRRK2 regulates a subset of Rab GTPases. Elife. 5, (2016).
- Ito, G., et al. Phos-tag analysis of Rab10 phosphorylation by LRRK2: a powerful assay for assessing kinase function and inhibitors. Biochem. J. 473, 2671-2685 (2016).
- Kinoshita, E., Kinoshita-Kikuta, E., Takiyama, K., Koike, T. Phosphate-binding tag, a new tool to visualize phosphorylated proteins. Mol. Cell. Proteomics. 5 (4), 749-757 (2006).
- Database, J. S. E. Using a Hemacytometer to Count Cells. J. Vis. Exp. , Available from: https://www-jove-com.remotexs.ntu.edu.sg/science-education/5048/using-a-hemacytometer-to-count-cells (2017).
- Ni, D., Xu, P., Gallagher, S. Immunoblotting and Immunodetection. Curr. Protoc. Mol. Biol. (114), 10.8.1-10.8.37 (2016).
- Reith, A. D., et al. GSK2578215A; a potent and highly selective 2-arylmethyloxy-5-substitutent-N-arylbenzamide LRRK2 kinase inhibitor. Bioorg. Med. Chem. Lett. 22 (17), 5625-5629 (2012).
- Fell, M. J., et al. MLi-2, a potent, selective and centrally active compound for exploring the therapeutic potential and safety of LRRK2 kinase inhibition. J. Pharmacol. Exp. Ther. 355, 397-409 (2015).
- Dzamko, N., et al. Inhibition of LRRK2 kinase activity leads to dephosphorylation of Ser(910)/Ser(935), disruption of 14-3-3 binding and altered cytoplasmic localization. Biochem. J. 430 (3), 405-413 (2010).
- Thévenet, J., Pescini Gobert, R., Hooft van Huijsduijnen , R., Wiessner, C., Sagot, Y. J. Regulation of LRRK2 expression points to a functional role in human monocyte maturation. PLoS One. 6 (6), e21519(2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados