Method Article
Protein Isolation du développement embryonnaire Souris Coeur Région Valve
Dans cet article
Résumé
The analysis of protein expression in young embryonic mouse valves has been hampered by the limited tissue available. This manuscript provides a protocol for preparing protein from developing embryonic mouse valve regions for western blot analysis.
Résumé
Western blot analysis is a commonly employed technique for detecting and quantifying protein levels. However, for small tissue samples, this analysis method may not be sufficiently sensitive to detect a protein of interest. To overcome these difficulties, we examined protocols for obtaining protein from adult human cardiac valves and modified these protocols for the developing early embryonic mouse counterparts. In brief, the mouse embryonic aortic valve regions, including the aortic valve and surrounding aortic wall, are collected in the minimal possible volume of a Tris-based lysis buffer with protease inhibitors. If required based on the breeding strategy, embryos are genotyped prior to pooling four embryonic aortic valve regions for homogenization. After homogenization, an SDS-based sample buffer is used to denature the sample for running on an SDS-PAGE gel and subsequent western blot analysis. Although the protein concentration remains too low to quantify using spectrophotometric protein quantification assays and have sample remaining for subsequent analyses, this technique can be used to successfully detect and semi-quantify phosphorylated proteins via western blot from pooled samples of four embryonic day 13.5 mouse aortic valve regions, each of which yields approximately 1 μg of protein. This technique will be of benefit for studying cell signaling pathway activation and protein expression levels during early embryonic mouse valve development.
Introduction
Etre capable d'identifier et de quantifier les niveaux d'expression de protéine est une technique standard pour animale et des expériences basées sur des cellules. Cependant, malgré un intérêt de longue date dans le développement de valve cardiaque embryonnaire précoce, l'évaluation de l'expression des protéines dans ce tissu spécifique au cours du développement est actuellement limité à l'immunohistochimie à la fois le poussin et 1,2 de la souris. Une partie de la difficulté à quantifier l'expression des protéines dans les soupapes de développement de la plupart des organismes modèles (par exemple, poulet et de souris) est la petite taille des vannes, ce qui limite la quantité de protéine qui peut être obtenue. Ainsi, pour des analyses quantitatives, les chercheurs s'appuient généralement sur l'extraction de l'ARN et l'amplification de la PCR quantitative ultérieure ou l'analyse des microréseaux 2-5. Cependant, les niveaux d'expression d'ARN et de protéines ne sont pas entièrement corrélative 6, donc en mettant l'accent sur l'expression de l'ARN ne peuvent pas fournir un compte rendu rigoureux des nombreux changements qui se produisent dans toute signalisation donnévoie à différents moments de l'évolution. Sur la base de cette limite dans la méthodologie actuellement disponibles, l'objectif de cette procédure était de développer un protocole pour obtenir de manière fiable des quantités suffisantes de protéine à partir de la souris embryonnaire régions de valve cardiaque en développement pour l'analyse quantitative des changements qui se produisent dans différentes voies de signalisation qui sont importantes dans la maturation de ce tissu.
Vannes embryonnaires sont déjà couramment disséqués de souris pour l'isolement de l'ARN et l'analyse ultérieure gène d'expression 2-5. Toutefois, ces études ont été limitées à l'utilisation de l'expression des gènes en tant que sortie de lecture de signalisation activation de la voie, ce qui ne permet pas la détection de détecter des modifications post-traductionnelles des protéines qui sont susceptibles d'affecter la signalisation en aval. En utilisant les techniques d'isolation de l'ARN comme un point de départ, nous avons commencé par disséquer les régions d'intérêt. Parce que notre intérêt détectait protéines phosphorylées qui sont des indices de pat signalisationHway activité pendant une période spécifique du développement de la valve aortique (E13.5-14.5), on a effectué toutes les dissections dans du tampon Tris-phosphate libre et recueilli les soupapes dans un tampon de lyse à base de Tris avec des inhibiteurs de phosphatase et protéase. Dans notre cas particulier, que les régions de la valve aortique ont été recueillies, mais la région de la valve pulmonaire peuvent facilement être obtenues en même temps. Les régions de soupapes sont ensuite homogénéisé et combiné avec un tampon d'échantillon qui est actuellement utilisé pour étudier l'expression des protéines dans les valves cardiaques adultes 7. En utilisant des volumes d'échantillon de petite taille (par exemple, 2 pi) et la mise en commun des régions de soupape à partir d'embryons avec le même génotype, nous avons pu détecter des protéines phosphorylées et non phosphorylés à jour embryonnaire 13,5 8. Étant donné que les régions de la vanne peuvent être congelés et stockés dans un tampon de lyse, les embryons peuvent être génotypés si nécessaire avant la mise en commun.
Cette technique élargit l'ensemble des outils qui sont disponibles pour l'évaluation signalin cellulaireg voies au cours du développement et fournit un compliment quantitative de l'immunohistochimie, en particulier pour les valves cardiaques en développement. Cette technique devrait être bénéfique non seulement pour les cardiologues de développement, mais aussi à tous les biologistes du développement qui travaillent avec des embryons de stade précoce sont intéressés dans les régions qui contiennent des tissus limitée.
Protocole
NOTE: Toutes les expériences ont été approuvées par le soin et l'utilisation des animaux Commission institutionnelle à l'Université de Caroline du Nord à Chapel Hill.
1. accise de la valve aortique
- En utilisant une technique d'euthanasie approuvé pour le stade embryonnaire d'intérêt, euthanasier une souris enceinte de chiots à la journée embryonnaire souhaitée (E) de développement.
- Utiliser 70% d'éthanol pour stériliser l'abdomen de la femme enceinte. Soulevez la peau et les muscles du bas-ventre et à l'écart des organes internes, et de disséquer cavité abdominale inférieure ouverte la femme de permettre l'accès à la corne utérine.
- Pour récupérer la corne utérine, maintenez le col avec une pince et caudale soigneusement découpées à la pince. Soulevez la corne utérine, la coupe de tout le tissu conjonctif qui maintient la corne en place, et terminer la suppression en coupant la jonction de la corne de l'utérus avec l'oviducte.
- Placez la corne utérine disséqué dans un col boîte de Pétri contenantd 0,1 M de tampon Tris (pH 7,6) et rincer au besoin. Ensuite, couper la corne utérine ouverte sens de la longueur pour exposer les sacs embryonnaires.
- Pour exposer l'embryon, couper ouvrir un sac embryonnaire à la jonction du placenta et de l'embryon et couper les vaisseaux ombilicaux pour libérer l'embryon. Placez l'embryon disséqué dans une deuxième boîte de Pétri avec un tampon froid Tris 0,1 M. Retour de la première boîte de Pétri avec les embryons restants undissected à glace jusqu'au moment de la prochaine embryon.
- Pour améliorer l'accès de la paroi thoracique dans l'embryon, décapiter l'embryon. Si le génotypage est nécessaire, enlever et sauver un morceau de tissu supplémentaire dans un tube Eppendorf placé sur la glace.
- Placer l'embryon sur le dos, et couper la paroi thoracique à la verticale le long du côté de la cage thoracique, à proximité d'un membre antérieur, et horizontalement au-dessus du diaphragme pour visualiser le coeur. Ensuite, ouvrez la paroi thoracique à exposer le cœur.
- En utilisant une pince pour tenir haut le long des gros vaisseaux, soulever le cœur à l'aide de pinces et couper les navires de moins. Lan, couper au-dessus de la pince pour libérer le cœur. Si les vaisseaux pulmonaires restent non coupés, les poumons peuvent être enlevées avec le cœur et doivent être retirés avant de poursuivre.
- Couper et enlever l'artère pulmonaire au-dessus du niveau de la vanne; aux stades embryonnaires décrits ici, l'artère pulmonaire est légèrement opaque, ce qui facilite sa discrimination de la région de la soupape. Coupez juste en dessous de la valve pulmonaire, en évitant le myocarde ventriculaire trabéculaire et de recueillir comme décrit à l'étape 1.10 si cette région est également intéressant. Ici et dans l'étape suivante, utilisez l'action capillaire pour attirer tampon Tris excès loin de la zone d'intérêt avant de recueillir dans un tampon de lyse.
- Retirer soigneusement la aorte distale de la valvule aortique, juste au-dessus du niveau de la vanne; comme l'artère pulmonaire, l'aorte reste légèrement opaque à ces stades embryonnaires, ce qui facilite son extraction. Coupez juste en dessous de la valve aortique, en évitant encore le myocarde ventriculaire trabéculaire, et transférer levanne à l'aide de pinces dans un tube Eppendorf avec du tampon de lyse 2 ul (décrit dans le tableau des matériaux) contenant des inhibiteurs de protease et de phosphatase sur de la glace.
- Répétez les étapes 1.6 à 1.10 jusqu'à ce que les vannes ont été excisés de tous les embryons, la collecte de chaque valve dans un tube Eppendorf séparé. Si tous les embryons ont le même génotype, toutes les vannes peuvent être collectées dans un seul tube Eppendorf.
- Si génotypage doit être effectuée afin de déterminer les vannes à mettre en commun, mettre immédiatement les échantillons dans un tampon de lyse à -80 ° C jusqu'à utilisation ultérieure. Les échantillons peuvent ensuite être regroupées après génotypage (étape 2.1.1).
Extraction de la protéine 2
- Décongeler les tissus prélevés sur la glace et centrifuger à 13 100 g pendant 1 min pour recueillir les tissus dans un tampon de lyse au fond du tube.
- Si les embryons ont été génotypés, utiliser une pipette pour recueillir doucement et le tampon de lyse piscine contenant les régions de soupape à partir d'embryons avec des génotypes similaires. Pour assurer une forte bet, la mise en commun des régions 3-4 de soupape à partir d'embryons E13.5 par échantillon est recommandé.
- Si tous les embryons sont du même génotype et toutes les régions de la vanne ont été rassemblées à l'étape 1.10, lyser l'ensemble de l'échantillon, comme décrit dans l'étape 2.2.
- Vérifiez le volume de l'échantillon global résultant et ajouter un tampon de lyse à un volume total de 40 ul; alors, perturber et homogénéiser les échantillons à l'aide de 5 mm billes d'acier inoxydable dans des tubes Eppendorf. Actionnez le lyser pour 2-4 min à 50 Hz, selon les instructions du fabricant. tubes à centrifuger brièvement, (~ 13 100 x g pendant 1 min), et de transfert des échantillons dans de nouveaux tubes, laissant derrière les débris insolubles.
- Préparer des échantillons distincts pour SDS-PAGE et transfert ultérieur pour analyse western blot, à la suite d'un protocole tel que Eslami et al 9. Buvard pour une protéine de contrôle du chargement est essentiel pour la quantification des protéines.
Résultats
Grâce à cette technique de préparation, nous avons pu détecter phosphorylée Smad 1,5,8 (pSmad) dans les régions de la valve aortique unique à partir de E13.5 embryons. Comme le montre la Figure 1A, la protéine isolée à partir d'une même région de la soupape est suffisant pour détecter une bande de pSmad faible. L'intensité du signal augmente proportionnellement au nombre de régions de soupapes qui sont mises en commun. Fait important, la pSmad rapport a / β-actine reste à peu près constant à travers les différentes tailles de l'échantillon (figure 1c). En raison des faibles taux de protéines disponibles, le même transfert ne peut être dépouillé et sondé pour un total Smad. Cependant, une tache distincte montrant totale Smad 1 et β-actine montre le même profil d'expression (figure 1B).
Ces régions de soupapes sont presque entièrement dépourvu de contamination myocarde ventriculaire. Grâce à cette technique de préparation en combinaison avec des échantillons du ventricule droit, nous n'avons pas pu détecter ventricparti- ANP marqueur du myocarde dans les régions de la valve aortique, alors que ce marqueur a été facilement détectée dans le ventricule (Figure 2). En outre, l'infarctus du marqueur chaîne légère de myosine ventriculaire 2V est également facilement détectée dans l'échantillon ventriculaire mais est à peine détectable dans les régions de la valve aortique. Ces résultats confirment la précision de la dissection.
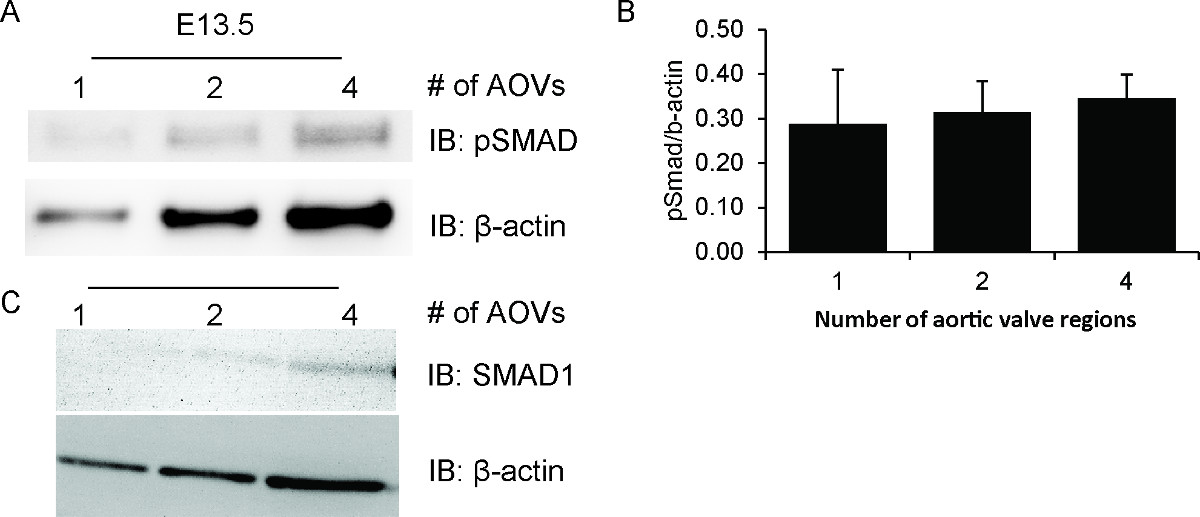
Figure 1 Smad phosphorylation dans les régions de la valve aortique embryonnaires. Échantillons ont été préparés avec un nombre croissant de régions de valve aortique de souris embryonnaire regroupées, allant de 1 à 4 régions de soupape par échantillon. Chaque échantillon comprend toutes les protéines isolées à partir de la région de l'ensemble de valve (s) dans un volume total de 25 à 28 pl. Préparé tissus ont ensuite été soumis à une analyse Western blot pour phosphorylée Smad1,5,8 (pSmad) (A), Smad1 totale (C), et β-actine (A, C). La protéine a été détectée en utilisant Lumigen ECL Ultra et imagé avec un logiciel UVM VisionWorks. L'intensité des bandes de pSmad et β-actine est proportionnelle à la quantité de matière de départ, telle que quantifiée en (B) à l'aide du logiciel ImageJ. Les données présentées sont la moyenne et l'écart type de deux taches distinctes. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
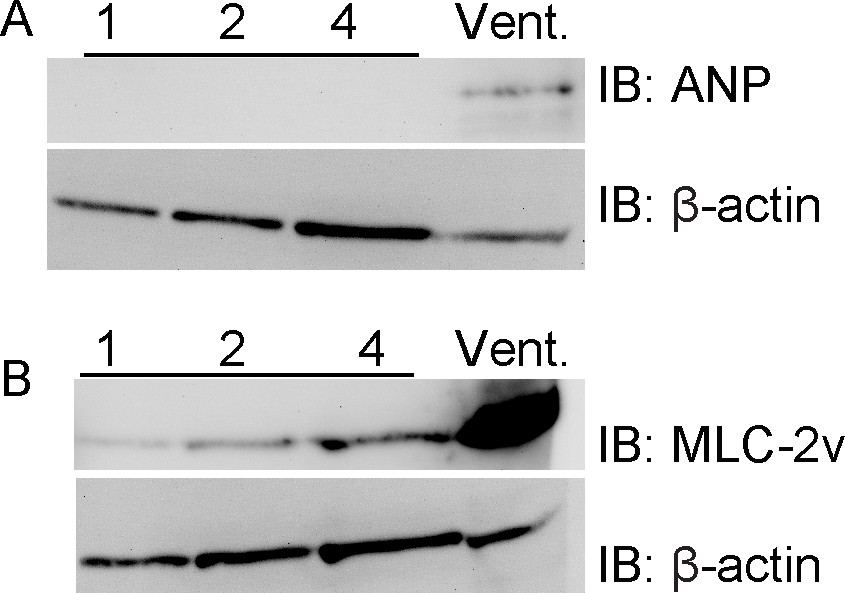
Figure 2: les régions de la valve aortique n'expriment pas les marqueurs de myocarde ventriculaire. Échantillons ont été préparés avec un nombre croissant de régions de valve aortique de souris embryonnaire regroupées, allant de 1 à 4 régions de vannes par exemple, ou avec unExtrait du ventricule droit. Les échantillons ont été traités comme décrit à la figure 1 et transférés pour le myocarde ventriculaire marqueurs ANP (A) et de la chaîne légère de myosine (MLC) -2V (B). ANP est complètement défendu à l'échantillon ventriculaire, et MLC-2v est exprimée à des niveaux extrêmement bas par rapport à l'échantillon ventriculaire. β-actine est disponible en tant que témoin de chargement.
Discussion
La capacité à quantifier les niveaux de protéines au début embryon de souris et poussin régions cardiaques de soupape constitue un outil supplémentaire pour comprendre les événements de signalisation cellulaires critiques pour le développement de la vanne. Notre protocole décrit ici ne diffère pas beaucoup de procédures d'isolement de protéines standard. Cependant, en modifiant quelques étapes clés, nous avons réussi à obtenir des protéines phosphorylées de tailles très petits échantillons. Pour atteindre ce résultat, les étapes suivantes sont d'une importance particulière. Afin de s'assurer que la protéine de qualité est obtenu, il est essentiel de garder les échantillons réfrigérés, si de la glace ou à l'aide de tampons glacées, en présence de la protéase et, le cas échéant, des inhibiteurs de phosphatase. En outre, l'homogénéisation d'un dispositif qui est conçu pour traiter les petits volumes d'échantillons est essentielle pour perturber de manière adéquate les membranes cellulaires. Même avec ces précautions, le rendement obtenu de la protéine peut être trop faible pour détecter par l'intermédiaire du dosage de Bradford avec un spectrophotomètreou un kit de quantification des protéines et ont encore l'échantillon disponible pour des analyses ultérieures. Nous avons pu déterminer que chaque région de la valve aortique contient environ 1 ug de protéine par mise en commun des régions 7 de la valve aortique et l'utilisation de la totalité de l'échantillon pour quantification. Malgré cette limitation, suffisamment de protéines est obtenue pour détecter les protéines phosphorylées et non phosphorylées, si l'absence de quantification préalable fait un contrôle de chargement essentiel. Nous avons spécifiquement utilisés β-actine, car il est une protéine de haute abondance, qui nous a permis de détecter même après décapage des membranes à de nombreuses reprises; son poids moléculaire différent de nos protéines d'intérêt; et il est exprimé de façon ubiquitaire. Un essai western blot avec différents nombres d'échantillons mis en commun est recommandé de déterminer le nombre de tissus qui doit l'être pour les régions soit des valves cardiaques embryonnaires à d'autres stades de développement ou d'autres petits tissus d'intérêt. En outre, nous recommandons la mise en commun au moins trois téchantillons d'émission de chaque échantillon de protéine pour rendre compte de la variabilité de la taille des tissus disséqués ainsi que dans la durée de chaque embryon assis sur de la glace avant la dissection. Si la protéine d'intérêt ne peut pas être détectée par western blot, même après la mise en commun des échantillons, nous vous recommandons de réduire le volume de l'échantillon et l'augmentation du temps d'homogénéisation afin de s'assurer que toutes les protéines potentiel est disponible pour la détection. En outre, les inhibiteurs de protéase peuvent également être ajoutés à un tampon de Tris refroidi à la glace pendant dissections de protéines instables ou facilement dégradés. Si la protéine d'intérêt est exprimée à de faibles niveaux, dépannage pendant la partie western blot de l'expérience, comme le réglage de l'incubation de l'anticorps primaire ou de la méthode de détection de substrat, peut aider à la visualisation.
Si les éléments (par exemple, l'immunohistochimie ou l'hybridation in situ) suggère qu'une protéine d'intérêt est exprimée à la fois dans les valves cardiaques et la la tranquiliténding myocarde, le myocarde de l'artère aortique ou pulmonaire peut être taquiné dehors doucement avec des aiguilles de tungstène, même des vannes de E12.5 embryons 5. Parce que cette étape supplémentaire augmente le temps de dissection et ajouter difficulté technique importante, nous vous proposons employant au besoin seulement. Dans notre étude initiale, nous avons commencé par une analyse immunohistochimique qui a montré que la phosphorylation de Smad a seulement changé dans le mésenchyme de soupape; donc, nous étions convaincus que les différences observées par western blot étaient dus à des changements dans l'expression dans le mésenchyme de soupape 8. En outre, parce que les vannes des sorties des voies de développement sont assis à la jonction des grandes artères et les ventricules, effectuer un transfert de contrôle de la contamination ventriculaire en utilisant un anticorps spécifique de myocarde ventriculaire telle que l'ANP ou MLC-2v est recommandée.
Parce que les soupapes subissent une réorganisation spectaculaire au cours de leur développement, nous offrons la r spécifique au stade suivantes recommandations pour les coussins / vannes et leur tissu environnant. Pour les premiers coussins (E9.5-11.5), les deux voies de sortie et coussins auriculo-ventriculaires sont facilement visualisées et disséqués en raison de la translucidité du myocarde 10; Toutefois, ces embryons sont de petite taille, et les aiguilles de dissection sont recommandés au lieu de forceps et microciseaux. Pour la différenciation à mi-étape (E12.5-14.5) 10, les valves semi-lunaires (c.-à-aortiques et les vannes de l'artère pulmonaire) seront plus facilement accessibles que les valves auriculo-ventriculaires (c'est à dire, la valvules mitrale et tricuspide). Cependant, les valvules semi-lunaires doivent être soigneusement disséqués en raison de l'aorte et l'artère pulmonaire sont septating et tournant pendant ce laps de temps 11,12. Une attention particulière doit être donnée à la rotation de la base de l'aorte et l'artère pulmonaire, et le point médian entre ces artères doit être constamment identifié. Lors des étapes ultérieures de maturation de la vanne et de la condensation (E15.5 et plus tard) 10, le valvules mitrale et tricuspide peut être taquiné loin à l'intérieur des ventricules; Toutefois, les valves semi-lunaires devraient être plus facilement accessible.
Western blot est une technique de longue date pour quantifier les niveaux d'expression de protéines dans les valves cardiaques adultes 13, où la taille de la biopsie est grande. Au contraire, les analyses complémentaires embryonnaires ont été axées sur l'immunohistochimie ou l'autre non quantitative pour détecter l'expression de la protéine ou la transcription inverse PCR, où la petite quantité de départ de l'ARNm isolé peut être amplifié avant analyse. Ces techniques actuellement employées fournissent des informations pertinentes concernant les changements dans la signalisation et l'expression des gènes, mais n'ont pas la capacité d'évaluer quantitativement les niveaux de protéines. Avec ce protocole, nous pouvons maintenant ajouter des données quantitatives d'expression de protéines à corréler avec les données d'expression de l'ARNm quantitative et analyses immunohistochimiques.
Ce protocole compliments techniques établies pour isoler l'ARNm à partir des valves cardiaques embryonnaires et expression de la protéine de formation d'image. A ce titre, la combinaison de ces techniques permet des analyses plus complètes de l'amont et la régulation des voies de signalisation, à partir de l'ARNm de la modification post-traductionnelle de protéine. En outre, cette technique permet la quantification de l'expression de la protéine, ce qui permettra d'améliorer les techniques d'immunohistochimie actuellement utilisés. Ensemble, nous pensons que cette technique sera d'intérêt pour tout chercheur intéressé par les valves cardiaques ou la préparation d'autres explants de tissus en particulier les petits pour l'analyse western blot quantitative.
Déclarations de divulgation
The authors have nothing to disclose.
Remerciements
We would like to thank Andrea Portbury and Davin Townley-Tilson for critical reading of the manuscript and the NIH (grant # R01HL061656) for funding support.
matériels
| Name | Company | Catalog Number | Comments |
| Timed-pregnant mouse | To be dissected at the embryonic stage of interest | ||
| Stereoscopic microscope | Nikon | SMZ645 | |
| 0.1 M Tris, pH 7.6 | |||
| Microscissors | Fine Science Tools | 15003-08 | |
| Fine forceps, #5 | Fine Science Tools | 11251-30 | |
| Dissecting needle holders | Ted Pella Inc. | 13560 | |
| Dissecting needles | Ted Pella Inc. | 13561-10 | |
| Micropipette, 20 μl, with tips | |||
| Lysis buffer | 50 mM Tris-HCl, pH 7.5, 150 mM NaCl, 5 mM EDTA, 1% Triton | ||
| PhosSTOP | Roche | 4906845001 | Add 1 tablet to 10 ml lysis buffer |
| TissueLyser LT | Qiagen | 85600 | |
| Stainless steel beads | Qiagen | 69989 | |
| Microcentrifuge |
Références
- Wirrig, E. E., Hinton, R. B., Yutzey, K. E. Differential expression of cartilage and bone-related proteins in pediatric and adult diseased aortic valves. J Mol Cell Cardiol. 50, 561-569 (2011).
- Chakraborty, S., et al. Twist1 promotes heart valve cell proliferation and extracellular matrix gene expression during development in vivo and is expressed in human diseased aortic valves. Dev Biol. 347, 167-179 (2010).
- Lee, M. P., Yutzey, K. E. Twist1 directly regulates genes that promote cell proliferation and migration in developing heart valves. PLoS One. 6 (e29758), (2011).
- Peacock, J. D., Huk, D. J., Ediriweera, H. N., Lincoln, J. Sox9 transcriptionally represses Spp1 to prevent matrix mineralization in maturing heart valves and chondrocytes. PLoS One. 6 (e26769), (2011).
- Chakraborty, S., Cheek, J., Sakthivel, B., Aronow, B. J., Yutzey, K. E. Shared gene expression profiles in developing heart valves and osteoblast progenitor cells. Physiol Genomics. 35, 75-85 (2008).
- Vogel, C., Marcotte, E. M. Insights into the regulation of protein abundance from proteomic and transcriptomic analyses. Nat Rev Genet. 13, 227-232 (2012).
- Meng, X., et al. Expression of functional Toll-like receptors 2 and 4 in human aortic valve interstitial cells: potential roles in aortic valve inflammation and stenosis. Am J Physiol Cell Physiol. 294, 29-35 (2008).
- Dyer, L. A., Wu, Y., Moser, M., Patterson, C. BMPER-induced BMP signaling promotes coronary artery remodeling. Developmental Biology. 386, 385-394 (2014).
- Eslami, A., Lujan, J. Western blotting: sample preparation to detection. J Vis Exp. (44), (2010).
- Garside, V. C., Chang, A. C., Karsan, A., Hoodless, P. A. Co-ordinating Notch, BMP, and TGF-beta signaling during heart valve development. Cell Mol Life Sci. 70, 2899-2917 (2013).
- Jiang, X., Rowitch, D. H., Soriano, P., McMahon, A. P., Sucov, H. M. Fate of the mammalian cardiac neural. Development. 127, 1607-1616 (2000).
- Scherptong, R. W., et al. Morphogenesis of outflow tract rotation during cardiac development: the pulmonary push concept. Dev Dyn. 241, 1413-1422 (2012).
- Mohler, E. R., Adam 3rd, L. P., McClelland, P., Graham, L., Hathaway, D. R. Detection of osteopontin in calcified human aortic valves. Arterioscler Thromb Vasc Biol. 17, 547-552 (1997).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon