Method Article
Imagen longitudinal intravital de la dinámica vascular en la médula ósea del calvario
En este artículo
Resumen
La microscopía intravital permite el estudio de procesos biológicos dinámicos como la regeneración de tejidos y el desarrollo de tumores. La médula ósea del calvario, un tejido altamente dinámico, ofrece información sobre la hematopoyesis y la función vascular. El uso de un implante de fijación de cabeza biocompatible impreso en 3D permite obtener imágenes longitudinales repetitivas, lo que mejora nuestra comprensión de la dinámica de los tejidos y el microambiente tumoral.
Resumen
La microscopía de fluorescencia longitudinal intravital se ha convertido en una técnica crucial para estudiar los procesos biológicos dinámicos, especialmente en el contexto de la regeneración de tejidos, el desarrollo tumoral y las respuestas terapéuticas. En particular, la médula ósea del calvario es un tejido altamente dinámico, donde el destino hematopoyético está interconectado con el microambiente circundante, con vasos especializados que responden a la hematopoyesis normal y patológica. Las imágenes tradicionales de los tejidos fijos ofrecen información estática, lo que a menudo limita una comprensión completa de estos procesos.
La integración de animales transgénicos que expresan marcadores celulares específicos, trazadores de células vivas, avances en equipos de imagen y el uso de cámaras especializadas han elevado la microscopía intravital a una herramienta fundamental para obtener información sobre los fenómenos biológicos dinámicos. Una aplicación de las imágenes intravitales es la investigación del comportamiento de los vasos tumorales y sus efectos terapéuticos. Un implante de fijación de cabeza de titanio impreso en 3D de nuevo diseño se puede conectar de forma estable al cráneo del ratón y es adecuado para la obtención de imágenes longitudinales durante varias sesiones.
El protocolo propuesto permite el examen espacial y temporal de la dinámica vascular en la médula ósea del calvario, incluyendo la visualización y cuantificación de la heterogeneidad vascular, la interacción con las células estromales y hematopoyéticas, y la medición de los parámetros funcionales vasculares. Además, la técnica permite la visualización de los lechos vasculares establecidos y el seguimiento de los efectos terapéuticos, la movilización de células madre y la localización de compuestos quimioterapéuticos a lo largo del tiempo mediante microscopía de dos fotones. En general, este protocolo de imágenes longitudinales intravitales proporciona una plataforma completa para investigar tanto el comportamiento de los vasos tumorales como la dinámica de las células hematopoyéticas, ofreciendo información valiosa sobre los intrincados procesos que gobiernan estos fenómenos biológicos.
Introducción
La microscopía intravital de la médula ósea (MO) de la calvaria sirve como una técnica poderosa e indispensable para investigar los procesos dinámicos de la hematopoyesis, la regulación del microambiente tisular y la dinámica vascular dentro del nicho de la MO. El objetivo principal de este enfoque metodológico es permitir la visualización y el análisis en tiempo real de los comportamientos celulares, las interacciones y la organización espacial dentro del microambiente BM in vivo. Mediante la observación directa de la médula ósea calvaria utilizando técnicas de imagen avanzadas junto con el marcaje fluorescente, los investigadores pueden dilucidar la compleja interacción entre las células madre hematopoyéticas (HSC), las células estromales y la vasculatura circundante, proporcionando así información crucial sobre la regulación de la hematopoyesis y las respuestas inmunitarias.
El desarrollo y la utilización de células madre de microscopía intravital parten de las limitaciones de los métodos tradicionales de imagen histológica y ex vivo, que a menudo no logran capturar la naturaleza dinámica de los comportamientos celulares y las interacciones de un tejido. A diferencia de las técnicas de imagen estática, la microscopía intravital permite a los investigadores observar la dinámica celular en tiempo real, lo que permite el estudio de procesos longitudinales como la migración, proliferación y diferenciación celular dentro de su nicho nativo, sin sacrificar a los animales de experimentación. Además, la microscopía intravital proporciona la ventaja única de estudiar los comportamientos funcionales in vivo, como la funcionalidad vascular (por ejemplo, perfusión, permeabilidad, hipoxia), preservando así la relevancia fisiológica y evitando artefactos asociados con la fijación y el procesamiento de tejidos. Estudios pioneros en el campo han demostrado las enormes ventajas de este enfoque 1,2, y sus hallazgos han sido corroborados y ampliados por enfoques más recientemente refinados 3,4,5 que utilizaron microscopía intravital para rastrear la localización endógena de HSC, la migración y las interacciones con la vasculatura dentro del nicho de BM. Además, la microscopía intravital ha sido fundamental para dilucidar los mecanismos subyacentes a los trastornos hematopoyéticos, como la leucemia y los síndromes de fracaso de la MO, ofreciendo nuevos conocimientos sobre la motilidad de las células leucémicas 6,7, las implicaciones vasculares asociadas a la enfermedad8 y la respuesta a los fármacos9.
Existen varias ventajas de la microscopía intravital, la imagen de la médula ósea de la pantorrilla cubre sitios óseos alternativos. En primer lugar, el BM contenido en el hueso del cráneo proporciona un fácil acceso para la obtención de imágenes intravitales en comparación con los huesos más profundos, como el fémur o la tibia. Esta accesibilidad facilita la observación directa del microambiente tisular, incluido el propio hueso, a través de la visualización de la generación de segundos armónicos (SHG)10, sin necesidad de procedimientos quirúrgicos invasivos. En segundo lugar, el cráneo es relativamente delgado y translúcido, lo que permite una mejor visualización de la dinámica celular dentro del nicho BM. Esta transparencia facilita la obtención de imágenes de alta resolución con microscopía de dos fotones, microscopía confocal y microscopía de lámina de luz, sin necesidad de técnicas de aclaramiento o aclaramiento óseo. El hueso de la pantorrilla ofrece una plataforma estable, plana y rígida para los experimentos de imágenes intravitales, lo que minimiza los artefactos de movimiento del tejido y garantiza condiciones de imagen consistentes durante períodos de observación prolongados. Esta estabilidad es particularmente ventajosa para los estudios longitudinales que rastrean los comportamientos y respuestas celulares a lo largo del tiempo. La reproducibilidad es otra gran ventaja, dada la estructura relativamente pequeña y espacialmente definida del hueso del calvario en los animales de experimentación. Esta uniformidad facilita la comparación entre diferentes grupos experimentales y permite un análisis estadístico sólido de los datos de imagen.
Aquí describimos un método para obtener imágenes de la BM de la calvaria del ratón mediante microscopía intravital de dos fotones mediante la introducción de un implante de fijación de la cabeza11 recientemente desarrollado, impreso en 3D utilizando una aleación de titanio de grado 23 biocompatible (Ti6Al4V), que cuenta con una cubierta dura dedicada y fácil de colocar hecha de la misma aleación de titanio, lo que permite un cierre seguro de la herida para evitar infecciones o daños en el sitio de la cirugía. El implante fija la cabeza del ratón de forma firme y estable a la platina del microscopio a través de un soporte de implante, lo que minimiza los artefactos respiratorios y permite obtener imágenes longitudinales de las mismas áreas a lo largo del tiempo. Se proporcionan algunos ejemplos de imágenes multicolor que representan células y estructuras del microambiente BM (superficie ósea SHG+; células mesenquimales nestina-GFP+; células endoteliales cdh5-DSRED+, cdh5-GFP+ o pdgfb-GFP+) y el compartimento hematopoyético maligno (células de LMA tdTOMATO+), así como agentes de contraste fluorescentes que representan la luz de los vasos (dextrano-TRITC). Los parámetros vasculares medidos durante horas o días, incluida la longitud, la rectitud y el diámetro de los vasos, así como la permeabilidad en diferentes regiones vasculares, pueden proporcionar información importante sobre el comportamiento y la salud de los tejidos.
Protocolo
Todos los experimentos con animales se realizaron bajo el acuerdo ético APAFIS#27215-2020041513522374 v6, aprobado por el "Ministère de l'enseignement supérieur, de la recherche et de l'innovation" francés.
1. Diseño de un implante de fijación de cabeza de titanio biocompatible para impresión 3D
NOTA: Diseñamos un implante de fijación de cabeza biocompatible utilizando un software de diseño asistido por computadora (CAD) de modelado paramétrico con capacidades integradas de análisis de elementos finitos (FEA) (consulte la tabla de materiales). Los insumos principales para el proceso de diseño incluyen un modelo anatómico de alta resolución del cráneo del ratón, un modelo del objetivo del microscopio y un modelo del sistema de fijación, que en conjunto informan las dimensiones y la configuración del implante. El resultado final es un archivo de malla independiente de la plataforma, normalmente en formato STL o STEP, compatible con la mayoría de los programas de impresión 3D. Este formato de archivo garantiza una transferencia perfecta a la impresora 3D para una fabricación precisa del implante.
- Preparación del modelo 3D del ratón
- Coloque el cráneo del ratón, preparado o in vivo, en el escáner de micro-CT.
- Configure los parámetros de escaneo para una resolución óptima de detalles anatómicos finos: abra el Panel de control y, en la sección Condiciones de escaneo , elija un campo de visión (FOV). Utilice LiveView para definir el volumen de análisis.
NOTA: A través de la resolución del detector, el campo de visión determinará el tamaño del vóxel en función de la resolución del detector. En las máquinas micro-CT más nuevas, un campo de visión de 5 mm suele corresponder a un tamaño de vóxel de 5 μm, un campo de visión de 10 mm a un tamaño de vóxel de 10 μm, un campo de visión de 25 mm a un tamaño de vóxel de 21 μm. El campo de visión limitado a esta resolución puede requerir múltiples escaneos para cubrir todo el cráneo. Varias exploraciones requieren superposición entre exploraciones. - Combine varios escaneos de alta resolución en un software de imágenes para crear un modelo único y completo del cráneo.
- Importe archivos DICOM micro-CT en software de segmentación (por ejemplo, 3D Slicer, Amira, Avizo o Mimics) para aislar el cráneo.
- Realice una segmentación basada en umbrales para separar el tejido óseo de las estructuras circundantes y realice ajustes manuales para refinar regiones complejas o ruidosas.
- En 3D Slicer, primero importe archivos DICOM y cargue el conjunto de datos y observe los segmentos CT en las vistas de corte axial, sagital y coronal .
- Abra la Edición de segmentos, haga clic en Agregar para crear una nueva segmentación | Agregue para crear un nuevo segmento, que se va a umbral.
- En el panel Editor de segmentos , haga clic en el efecto Umbral . Ajuste los reguladores de umbral inferior y superior (o escriba valores numéricos) para que la región que se va a segmentar se resalte en rojo en las vistas de sector. Un segmento 3D con umbral está listo.
- Para exportar el cráneo segmentado como un archivo STL, cambie al módulo de segmentación. Asegúrese de que el segmento con umbral esté seleccionado. En la sección Exportar/Importar modelos y mapas de etiquetas , establezca Tipo de exportación en Modelos. y Formato de archivo: STL. Haga clic en el botón Exportar .
NOTA: Los archivos STL utilizan un formato de malla triangular, que representa la superficie del cráneo con miles de triángulos, capturando la forma anatómica con gran detalle.- Si es necesario, realice la costura en MeshLab de la siguiente manera:
- Importar mallas haciendo clic en Archivo | Importe la malla y seleccione una como escaneo de referencia.
- Alinee otros escaneos con la herramienta Alinear (Filtros | Inscripción | Alinear) marcando los puntos correspondientes y utilizando Proceso.
- Combine los escaneos alineados haciendo clic en Filtros | Capa de malla | Aplanar capas visibles con la opción Fusionar solo capas visibles .
- Si es necesario, realice la costura en MeshLab de la siguiente manera:
- Limpie y repare la malla, eliminando los vértices y triángulos duplicados de la siguiente manera, para garantizar la eliminación de los triángulos inducidos por el ruido y el relleno de cualquier agujero involuntario, para obtener una superficie lisa y continua:
- Haga clic en Filtros | Limpieza y reparación | Eliminar vértices duplicados.
- Haga clic en Filtros | Limpieza y reparación | Eliminar caras duplicadas.
- Haga clic en Filtros | Limpieza y reparación | Retire las piezas aisladas (por número de cara). Especifique un umbral para el número mínimo de caras que se van a conservar (por ejemplo, establezca este umbral bajo para eliminar pequeños clústeres o ruido).
- Haga clic en Filtros | Remallado, simplificación y reconstrucción | Cierre los agujeros.
- Realice la simplificación de la malla a ~ 10,000-30,000 triángulos, ya que los archivos STL iniciales de micro-CT contienen recuentos de triángulos muy altos que no son compatibles con el software CAD. En las áreas de alto detalle, conserve triángulos más pequeños en la parte superior del cráneo para garantizar un ajuste preciso del implante. En regiones más gruesas, simplifique las regiones menos críticas con triángulos más grandes para reducir el tamaño del archivo y optimizar la eficiencia del procesamiento.
NOTA: La simplificación de la malla se realiza por áreas y se detalla en 1.1.8.1-1.1.8.3.- Utilice la herramienta de selección Seleccionar componentes conectados en una región para seleccionar áreas y simplificarlas.
- Utilice la diezmación de colapso de borde cuadrático en las áreas seleccionadas con parámetros. Haga clic en Filtros | Remallado, simplificación y reconstrucción | Simplificación: Diezmado de colapso de borde cuádrico. Establezca el umbral de calidad en 0,8, la opción Conservar el límite de la malla en SÍ, la conservación de la topología en SÍ, el número objetivo de caras: establezca un número objetivo correspondiente a la resolución necesaria.
- Itere según sea necesario hasta que el número total de triángulos se reduzca al nivel deseado. A continuación, guarde el modelo simplificado en un archivo STL.
- Preparación del modelo de cráneo en el modelo CAD
- Importe el modelo de cráneo simplificado en formato STL en el software CAD como una nueva pieza o modelo. Asegúrese de que el archivo importado se convierta correctamente en un cuerpo sólido o de superficie si es necesario, en función de las capacidades del software. Guarde el modelo en el formato de archivo CAD nativo para permitir modificaciones posteriores y garantizar la compatibilidad con los pasos de diseño posteriores.
- Coloque puntos de referencia en Bregma y Lambda y cree el eje Bregma-Lambda en este modelo.
- Cree el plano Mediano que incluya el eje Bregma-Lambda.
- en el plano Mediana, cree un eje tangencial a la calvaria y, a continuación, guarde el cráneo preparado (Figura suplementaria S1).
- Cree un nuevo ensamblaje e inserte el cráneo preparado de manera que Bregma coincida con el origen, el plano mediano con el plano derecho y el eje de la pantorrilla sea horizontal.
- El implante de fijación de la cabeza se diseñará directamente dentro del ensamblaje para garantizar un ajuste perfecto con el modelo de cráneo preparado. Para lograr esto, cree el implante como una nueva pieza dentro del ensamblaje. Comience navegando a Insertar | Componente | Nueva pieza en el software CAD. Cuando se le solicite, seleccione la superficie de la calvaria o el plano superior del ensamblaje como referencia para colocar y anclar la nueva pieza.
- Crea un conjunto de planos igualmente espaciados (espaciado de 2 mm) a lo largo del cráneo. En cada plano, cree un boceto, y en cada boceto, dibuje una curva proyectada con un desplazamiento de 0,2 mm en forma de una spline simétrica con nueve puntos, siguiendo la sección de la superficie superior del cráneo 0,2 mm por encima del cráneo (Figura suplementaria S2).
- Cree una superficie barrida utilizando estas ranuras, la superficie límite del cráneo, asegurando que esta superficie sea lisa y sin ningún defecto, para ser utilizada para la construcción de las características del implante.
- Definición de la superficie observable en el cráneo
NOTA: Con base en el modelo 3D, se identifica la región más plana del hueso de la calvaria ideal para la observación en 3D. Esta región se elige cuidadosamente para garantizar un acceso óptimo al objetivo del microscopio de dos fotones, teniendo en cuenta la integridad biológica y estructural del cráneo. La definición puede utilizar coordenadas estereotáxicas donde el punto Bregma sirve como origen del sistema de coordenadas.- Cree un boceto en el plano del calvario y cree una spline en forma de pera de AP +6,5 a -2,6 mm de ancho en AP 0,0. Este será el Contorno de la Superficie Observable (Figura Suplementaria S3).
NOTA: El objetivo del microscopio está colocado virtualmente en relación con la superficie observable previamente definida en el cráneo. Este paso implica determinar las limitaciones espaciales impuestas por el tamaño y la forma del objetivo, asegurando que el diseño del implante se adapte a los requisitos del microscopio. - Inserte el modelo de objetivo del microscopio en el conjunto verticalmente. Restrinja su punto de enfoque en un plano paralelo al plano de la pantorrilla en el hueso de la pantorrilla y defina el volumen delimitador del objetivo del microscopio.
- Cree un volumen barriendo el objetivo siguiendo el contorno de la superficie observable.
- Cree una ventana de observación alrededor de la superficie observable definida, asegurando un acceso sin obstrucciones para el objetivo del microscopio.
NOTA: La ventana de observación es una estructura en forma de anillo que tiene funciones secundarias como la retención de líquidos por inmersión y estructura sólida para el correcto cierre de la cubierta protectora. - Cree un croquis en el plano de la calvaria y copie el contorno de la superficie observable.
- Crea un contorno paralelo de 0,5 mm de grosor. Extruya este doble contorno hacia arriba 0,9 mm y hacia abajo hasta la superficie Skull Limit Surface con un ángulo de inclinación de 4°.
- Cree un boceto en el plano del calvario y cree una spline en forma de pera de AP +6,5 a -2,6 mm de ancho en AP 0,0. Este será el Contorno de la Superficie Observable (Figura Suplementaria S3).
- Definición de las estructuras de cementación, cola y cola de milano.
- Cree un boceto en el plano del calvario y dibuje una forma de C de 0,5 mm de grosor que se conecte a la ventana de observación y extruya 2 mm de altura y hacia abajo hasta la superficie de la superficie del límite del cráneo con un ángulo de inclinación de 4° (Figura suplementaria S4).
- Crea un boceto en el plano del calvario y rellena este espacio con rectángulos de 0,4 mm de grosor colocados radialmente con un espacio de 1 mm entre ellos.
- Extruir de forma similar a la ventana de observación.
- Crear la cola del implante apoyándose en el cráneo para la fijación respetando el volumen disponible.
- Crea un plano transversal tangencialmente a la estructura de cementación y otro de 12,5 mm hacia atrás.
- Dibuja rectángulos en estos planos. Barre un cuerpo piramidal entre ellos, estableciendo los ángulos aproximadamente 20° y 37° con respecto a la horizontal.
- Agregue un orificio roscado M1.6 en la cola cerca de la estructura de cementación.
- Crea un boceto en el extremo de la cola y dibuja el perfil de cola de milano.
- Extruya este perfil en 8 mm.
- Verificación de la no injerencia en el objetivo.
- Examine todo el diseño del implante, incluida la ventana de observación y la cola de milano, para asegurarse de que no haya interferencias mecánicas con el objetivo del microscopio. Compruebe si hay interferencias con el volumen creado en el paso 1.3.3.
NOTA: Este paso de verificación es fundamental para garantizar que el objetivo se pueda colocar y mover según sea necesario sin obstrucciones.
- Examine todo el diseño del implante, incluida la ventana de observación y la cola de milano, para asegurarse de que no haya interferencias mecánicas con el objetivo del microscopio. Compruebe si hay interferencias con el volumen creado en el paso 1.3.3.
- Retire el material innecesario de la cola del implante para minimizar el peso y reducir las posibles molestias para el animal.
- Cree un boceto en el plano mediano y dibuje recortes triangulares en él para formar una estructura de celosía cuando se extraiga por extrusión del cuerpo de cola.
- Repita en el plano perpendicular.
- Verifique el desplazamiento máximo mediante la simulación del método de elementos finitos (FEM) para analizar las propiedades mecánicas del implante en diversas condiciones.
NOTA: Esta simulación ayuda a predecir el desplazamiento máximo y las tensiones dentro del implante durante su uso, lo que garantiza su fiabilidad.- Habilite el complemento de simulación de software CAD.
- En la barra de herramientas Simulación, cree un nuevo estudio estático. Vaya a la pestaña Simulación (si está visible) o vaya a Simulación | Estudio. En el cuadro de diálogo Estudio , elija Estático como tipo de estudio. Haga clic en Aceptar para crear el nuevo estudio (Figura complementaria S5).
NOTA: Este tipo de estudio es adecuado para analizar la deformación y la tensión bajo cargas estáticas, como se espera en el uso del implante. - Elija un material para el implante que coincida con la composición real del implante (Ti6Al4V).
- Defina restricciones fijas para simular los puntos de fijación del implante donde entraría en contacto con la fijación de cola de milano. Defina la superficie superior de la cola de milano como fija.
- Defina la carga vertical en la superficie inferior del implante. Ajuste 1 N de fuerza vertical.
- Genere una malla. Refinar la malla en áreas donde se espera una alta tensión o deformación (Figura suplementaria S6). Para ello, haga clic con el botón derecho del ratón en el elemento de malla del árbol de estudio y seleccione Crear malla... En el cuadro de diálogo Malla , ajuste la Densidad de malla con el control deslizante o activando el cuadro Control de malla para establecer los tamaños de elemento máximo y mínimo de forma independiente. Haga clic en Aceptar para generar la malla.
NOTA: El software genera automáticamente una malla inicial para el estudio, pero para obtener resultados más precisos, la malla se puede refinar para seleccionar una malla más fina, particularmente en regiones que son críticas para la estabilidad del implante (como la ventana de observación). Un mallado más fino en estas regiones mejora la precisión de la simulación al capturar las concentraciones de tensión locales. - Inicie el análisis estático para calcular las distribuciones de desplazamiento, tensión y deformación a través del implante.
- Analiza los resultados. Examine la gráfica de desplazamiento para identificar las áreas de máxima deformación. Este gráfico muestra cuánto se deformaría el implante bajo las cargas aplicadas, resaltando cualquier área con riesgo de movimiento excesivo. Asegúrese de que la deformación dentro de la ventana de observación no supere las 3 micras.
- Por último, diseñe una cubierta protectora para proteger la ventana de observación, cuando no esté en uso. Esta tapa protege el hueso expuesto de los contaminantes ambientales y los daños físicos.
2. Tratamientos con ratones, anestesia e implantación quirúrgica del implante de cabeza
NOTA: Aquí, ya sea macho o hembra, se pueden usar ratones C57BL/6 o transgénicos de 7 a 12 semanas de edad, como se muestra. Para inducir la colonización leucémica del MO, las células leucémicas generadas, según lo descrito por Horton et al.12, se administran por vía intravenosa 2-3 semanas antes de la imagen. Para garantizar la salud de la herida, se deben utilizar técnicas estériles.
- Debajo del capó, ensamble las herramientas quirúrgicas estériles (tijeras y pinzas sin filo), la recortadora y los consumibles, incluidos gel para ojos, solución salina, desinfectante, cemento dental, hisopos absorbentes y pañuelos de limpieza. Encienda el esterilizador de perlas.
- Llene la cámara de inducción anestésica con isoflurano al 4% y oxígeno atmosférico y coloque el ratón en la cámara. Cuando el animal esté completamente anestesiado (verifique si hay una pérdida del reflejo de enderezamiento y un patrón de respiración más profundo y lento), cambie el flujo de isoflurano al cono de la nariz de la máscara anestésica y baje la concentración de isoflurano al 2%.
- Coloque el ratón sobre una almohadilla térmica a 37 °C y controle visualmente la frecuencia respiratoria. Opcional: Para ratones con un fenotipo patológico, agregue una manta de supervivencia para preservar la temperatura correcta durante toda la anestesia.
- Administrar analgésicos por vía subcutánea (Buprecare, 0,01 mg/kg) 30 min antes de la cirugía.
- Afeita la cabeza del ratón con una maquinilla de afeitar eléctrica.
- Agregue una gota de gel oftálmico en los ojos del ratón para evitar la sequedad mientras está bajo anestesia, asegurándose de que el gel no gotee sobre el cuero cabelludo.
- Limpie el ratón para eliminar los pelos residuales de la zona quirúrgica. Frote la parte superior del cuero cabelludo con desinfectante en un pañuelo de algodón. Asegúrese de eliminar todo el vello para evitar artefactos de imagen, así como el riesgo de infección de la herida.
- Con pinzas y tijeras estériles (después de 30 s en el esterilizador), haga una pequeña incisión en la porción central del cuero cabelludo para exponer la cicatriz ósea central. Sígalo para definir la longitud y el ancho correctos del área de imagen. Retire con cuidado el trozo del cuero cabelludo, levante la piel entre las orejas con pinzas y haga una pequeña incisión en la parte posterior de la cabeza. Sosteniendo la piel hacia arriba, pase las tijeras por debajo de la piel y corte con cuidado fuera del área de la imagen.
- Extirpar el tejido conectivo entre el cráneo y el cuero cabelludo. Limpie el hueso expuesto con PBS estéril en un hisopo de algodón. Proceda rápidamente a la fijación del implante de cabeza al cráneo del ratón para evitar la exposición prolongada del hueso del cráneo (<10 min).
- Haga una pasta con suficiente cemento dental en una placa de Petri siguiendo las instrucciones del fabricante y aplíquela rápidamente en la parte inferior del implante de fijación de la cabeza.
- Sin que entre cemento dental en el área de la imagen, coloque el soporte de la cabeza sobre el cráneo expuesto del ratón y espere a que se asiente.
- Una vez configurado (normalmente dentro de los 3-5 minutos), agregue unas gotas de PBS a 37 °C en la cámara de imágenes para mantener el cráneo hidratado.
3. Obtención de imágenes con un microscopio de dos fotones
- Antes de comenzar la cirugía, encienda el láser y deje que se caliente para estabilizarse. Encienda el microscopio e inicie el software de adquisición. Seleccione el objetivo apropiado (inmersión en agua 25x/0.95 para este experimento).
- Utilice un soporte de microscopio vertical de etapa fija que tenga suficiente espacio para colocar una placa de pruebas veterinaria con una almohadilla térmica, todo para moverlo integralmente con la platina xy. Utilice una placa de pruebas óptica de aluminio sólido como plataforma para sujetar el implante de cabeza y la máscara estereotáxica. Conecte las mangueras flexibles y sus conectores asociados con la inyección y extracción de isoflurano a la máscara en la jaula del microscopio.
- Configure los ajustes de adquisición para escanear rápido: formato 512 x 512 píxeles; velocidad 600 Hz; y factor de zoom 1.
- Ajuste la línea láser a 880 nm para poder detectar SHG, GFP y tdTOMATO con la misma longitud de onda de excitación.
- Apague la luz de la habitación y cierre correctamente la caja alrededor del soporte del microscopio antes de activar los detectores NDD y defina las puertas de detección espectral: PMT1, para detectar SHG [388 - 431 nm]; HyD2, para GFP [485 - 548 nm] y HyD3 para tdTOMATO [551 - 645 nm]. Deje el desplazamiento en 0.
- Cree un nuevo conjunto de datos de proyecto y cámbiele el nombre de acuerdo con el experimento específico.
- Una vez que el implante de fijación de la cabeza se adhiere al cráneo del ratón (confirmado mediante el endurecimiento del cemento), conecte el isoflurano hacia la máscara estereotáxica del microscopio.
- Acerque el ratón rápidamente al microscopio. Inserte con cuidado los dientes del ratón en la mascarilla para permitir la penetración del isoflurano a través de la nariz del ratón levantando su nariz con una mano.
- Mientras sostiene el mouse con una mano, deslice suavemente la cola de milano del implante de fijación de la cabeza en el soporte de fijación con la otra mano y asegúrelo con media vuelta de la perilla del tornillo.
- Introducir una sonda rectal preincrustada con un gel a base de agua para el control de la temperatura y añadir gotas oftálmicas en los ojos del ratón.
- Llene la cámara de imágenes de la cabeza con una gran cantidad de gel a base de agua o PBS y baje el objetivo de inmersión en agua para sumergirlo completamente para obtener una excitación óptima y la detección de las señales generadas por la excitación de dos fotones.
- Mueva la etapa x-y y la unidad z para enfocar el hueso central en la cicatriz del andamio.
NOTA: Es conveniente utilizar los oculares del microscopio y una lámpara de tipo halogenuros metálicos como fuente de luz, filtrada por un filtro de emisión de excitación de triple banda. La falta de corte óptico hace imposible obtener una imagen nítida; Pero con la práctica, el área central del cráneo es fácilmente reconocible. - Después de identificar el tejido, marcado por una superficie ósea y una vena central, apague la lámpara de halogenuros metálicos y cierre las ventanas de la jaula del microscopio para proteger los detectores HyD y permitir el paso del láser infrarrojo (IR).
- Ajuste la ganancia PMT/SHG a 850 V (su rango de funcionamiento lineal) y ajuste las ganancias HyD al 100%. Aumente la potencia del láser IR hasta obtener una imagen con un rango dinámico de 200 niveles de gris (con detectores dinámicos de 8 bits) en el más bajo de los tres canales. Reduzca las ganancias de los otros detectores si las diferencias de emisión son demasiado importantes o si hay saturación utilizando el modo de tabla de búsqueda (LUT) "inferior inferior".
NOTA: En nuestro caso, una potencia láser del 30% fue suficiente para obtener contraste en todos los canales. - En el software de adquisición, busque una región hecha de bolsas BM (GFP+, tdTOMATO+) encerradas dentro de las superficies óseas (SHG).
- Para encontrar diferentes ROI, active LAS NAVIGATOR y cree una visión general de toda la región utilizando el modo en espiral. Detenga la adquisición cuando la superficie adquirida sea lo suficientemente grande. Guarde la vista general, combínela y cámbiele el nombre: esta será la imagen de referencia en el caso de las imágenes longitudinales.
NOTA: Tenga cuidado de no generar una espiral demasiado grande, ya que esto podría hacer que el soporte del dispositivo entre en contacto con el objetivo. - Registre algunas posiciones de ROI haciendo clic en el icono de una sola imagen y cambie el nombre de cada posición en la lista de tareas. Realice una captura de pantalla para tener la posición relativa de los distintos ROI superpuesta a la vista general guardada anteriormente.
NOTA: Esta captura de pantalla será esencial para reemplazar los ROIs en la imagen longitudinal, ya que el software de adquisición no permite recuperar este grupo de posiciones en la versión utilizada para este experimento. - Para adquirir un volumen de pila z, seleccione el modo de pila z, defina el tamaño del paso a 3 μm y anule la selección del mismo tamaño de pila para todas las regiones . Seleccione la primera posición, defina las posiciones superior e inferior , verifique el tamaño del paso z y haga clic en redefinir la pila. Aparece un icono de cubo junto al nombre de la posición en la lista de posiciones. Repita este paso para todas las posiciones de los diferentes ROI.
- Presione adquirir y guarde las imágenes (tres canales z-stacks con ~50 planes) en la carpeta correspondiente. Este paso marca Tctrl del experimento; Compruebe que los parámetros de adquisición son correctos (obtenga el rango dinámico correcto en los tres canales, evite la saturación de la señal en la medida de lo posible y verifique el tiempo de adquisición durante un ciclo completo).
- Para medir una característica dinámica (permeabilidad vascular en este caso), adquiera un lapso de tiempo.
- En esta configuración, los tres detectores permiten la obtención simultánea de imágenes de tres canales; si se requiere un cuarto canal para el experimento (monitoreo de Dextran en este caso), active el modo secuencial y agregue una segunda secuencia de adquisición. Ajuste la línea láser IR a 820 nm para poder excitar el fluoróforo Dextran Cy5. Cambie el rango de detección de Hyd2 a [650 - 744 nm] y desactive los otros detectores. Seleccione entre el modo de adquisición de pilas .
NOTA: De forma predeterminada, la segunda secuencia de adquisición tiene los mismos parámetros que la secuencia 1. - Antes de iniciar el timelapse, inicie la adquisición de todas las posiciones para garantizar que el tiempo de grabación no supere los 3 min (teniendo en cuenta la adición del modo secuencial), el tiempo mínimo necesario para detectar la fuga de dextrano.
- Antes de la inyección de Dextran, cambie el modo de adquisición a xyzt. En el módulo t, ajuste el intervalo de tiempo a 3 min y la duración a 1 h.
- En esta configuración, los tres detectores permiten la obtención simultánea de imágenes de tres canales; si se requiere un cuarto canal para el experimento (monitoreo de Dextran en este caso), active el modo secuencial y agregue una segunda secuencia de adquisición. Ajuste la línea láser IR a 820 nm para poder excitar el fluoróforo Dextran Cy5. Cambie el rango de detección de Hyd2 a [650 - 744 nm] y desactive los otros detectores. Seleccione entre el modo de adquisición de pilas .
- Inyectar 100 μL de Dextran 70 KDa-TRITC (o 500 KDa-Cy5) a 3 mg/ratón por vía intravenosa e iniciar la adquisición.
- Después de la adquisición, guarde el conjunto de datos en la carpeta adecuada.
NOTA: Durante estos numerosos pasos, controle la frecuencia respiratoria y la temperatura del ratón con frecuencia y ajuste el flujo de isoflurano si es necesario. Idealmente, la temperatura, el ECG y la frecuencia respiratoria podrían registrarse e incluirse como metadatos del experimento.
4. Recuperación del ratón
- Una vez finalizada la sesión de imagen, desactive el detector HyDs dentro del software de adquisición y encienda la caja de calentamiento del ratón (37 °C). Abra la jaula del microscopio, levante el objetivo y retire la sonda rectal del ratón. A continuación, deslice suavemente la cola de milano del implante de fijación de la cabeza fuera del soporte de fijación y mueva el ratón de la máscara estereotáxica del microscopio a la máscara de cirugía en la almohadilla térmica.
- Apague la inyección de isoflurano bajo el microscopio y apague la almohadilla térmica.
- Retire suavemente el gel a base de agua o PBS del cráneo del ratón con un hisopo estéril.
- Agregue gel intrasitio para mantener la humedad del cráneo entre las sesiones de imagen, como se describió anteriormente13.
- Cierre el área de imagen del implante de cabeza con la cubierta específica y asegúrela cuidadosamente con el tornillo.
- Coloque el mouse en la caja de calentamiento a 37 ° C y espere hasta que se despierte. Lleve el ratón a la instalación de animales y colóquelo en una jaula limpia con hidrogel y enriquecimiento. Monitoree a los animales todos los días para detectar cualquier signo de dolor o infección, en cuyo caso administre Buprecare diariamente. Para preservar la humedad del pantalón, cambie el gel intrasite 2 veces por semana.
NOTA: Elija una jaula alta con refugios de cartón y material de anidación y asegúrese de poner a los ratones en jaulas individuales.
5. Adquisiciones longitudinales
NOTA: El ratón puede volver a ser fotografiado durante los días siguientes. Sin embargo, asegúrese de no repetir más de tres sesiones de imágenes por semana para evitar los efectos indeseables de la anestesia repetida, como la sequedad ocular o la fatiga excesiva, así como la dificultad respiratoria y la hipotermia.
- Para volver a crear una imagen de un ratón con un soporte para la cabeza ya instalado, siga el paso 2.2 y coloque una pequeña gota de gel oftálmico en los ojos del ratón para evitar la sequedad durante el procedimiento.
- Siga la sección 3 hasta el paso 3.16 para obtener la imagen general.
NOTA: El uso del implante de titanio de la cabeza sin tener que cambiar la altura o el ángulo de la posición del soporte hace que sea muy fácil encontrar el área general de interés. - Si es necesario, vuelva a alinear las imágenes anteriores y nuevas utilizando el módulo de abrir imagen y alinear.
- Abra la imagen de captura de pantalla del paso 3.17 para marcar las posiciones en su lugar original.
NOTA: El uso de contraste SHG es muy útil, especialmente cuando se espera una remodelación de los vasos sanguíneos y las células asociadas que expresan GFP. - Adquiera z-stacks y timelapses de la misma manera que se describe en los pasos 3.18-3.22.
6. Cuantificación de parámetros vasculares
- Parámetros vasculares
- Para abrir las imágenes generadas con el software (archivos .lif) en IMARIS, primero conviértalas a formato ".ims" con el Convertidor de archivos. Abra el archivo ".ims" con IMARIS, verifique los metadatos (escalas X, Y, Z) y seleccione el módulo Filamento . Seleccione omitir la creación automática, editar manualmente.
- Para una mejor visualización durante la generación de filamentos, vaya a Configuración (símbolo de hoja), seleccione Línea en estilo y deje el número de píxeles en el valor predeterminado (1). Marque las casillas Mostrar dendritas, incluidos el punto inicial, los puntos de ramificación y los puntos terminales. Desmarque Mostrar lomo.
- En dibujar (símbolo de pincel), seleccione Método AutoPath; 5 μm como diámetro, y la fuente de canal asociada con el vaso (TRITC-Dextrano en este caso). Por último, marque el centro automático y el diámetro automáticor.
- Comience a dibujar los filamentos usando shift + clic derecho del mouse para elegir un punto inicial de rama y shift + clic izquierdo para elegir un punto final de una rama.
NOTA: El punto de inicio está en azul claro, los puntos de ramificación están en rojo o azul oscuro y los puntos terminales están en verde. - Para establecer el diámetro correcto del filamento mientras dibuja, use el botón de desplazamiento del mouse.
- Para unir dos filamentos con puntos terminales distintos, cree un nuevo punto de partida entre ellos y conéctelo en cada punto final.
NOTA: Por lo tanto, los puntos terminales verdes aparecen en los puntos de bifurcación. - Una vez creado el árbol vascular, vuelva a la configuración y asegúrese de que los números de puntos iniciales, ramificados y terminales sean correctos. Seleccione el cono para visualizar los diámetros y verificar su apariencia.
- Si el diámetro (o la conexión) de un filamento parece incorrecto, vaya a Editar (símbolo de lápiz), seleccione el filamento que desea eliminar con un clic derecho y haga clic en Eliminar. Vuelva a dibujar el filamento ajustando el diámetro del filamento con el botón de desplazamiento del ratón.
- Para extraer todos los datos estadísticos generados y guardarlos, haga clic en el icono Estadísticas | exportar todas las estadísticas a un archivo (icono de varios disquetes en la parte inferior de la pantalla).
NOTA: Para evitar cualquier filamento potencialmente aberrante que no corresponda a los vasos, solo se incluyeron en el análisis estadístico los filamentos de más de 20 μm.
- Cuantificación de fugas
NOTA: Para abrir las imágenes generadas con el software (archivo .lif) en IMARIS, primero conviértalas a formato ".ims" con el Convertidor de archivos.- Para fusionar imágenes tomadas antes y después de la inyección de dextrano en la misma serie temporal, vaya al modo Surpass, haga clic en la primera imagen (Tctrl , antes de la inyección de dextran) y en la imagen de lapso de tiempo (tiempo 1, 60 min) para abrirlas, y combínelas haciendo clic en Editar y agregar puntos de tiempo.
NOTA: Para corregir la deriva 3D eventual, siga los pasos 6.2.2-6.2.7 utilizando la función de puente entre Fiji e IMARIS. Instale los siguientes complementos en Fiji: "IMARIS_Bridge96.jar" y "IMARISBridgeUtils.jar". El registro de deriva 3D se realiza mediante el uso de "StackReg Pluging" (ya disponible en Fiji). Si no es necesario, vaya al paso 6.2.8 - Para hacer la corrección de deriva en 3D, abra Fiji para puentear imágenes de IMARIS haciendo clic en Plugins | IMARIS | Imagen de IMARIS.
- En Fiyi, selecciona Plugins | Inscripción | Corrija la deriva 3D.
- En las nuevas ventanas que aparecen, elija el canal adecuado para el registro.
NOTA: En este caso era Nestin-GFP, pero podría ser cualquier canal que resalte objetos inmóviles y una alta relación señal-fondo. - Para mejorar la detección de pequeñas desviaciones, seleccione Cálculo de escala de tiempo múltiple, Corrección de desviación de subpíxeles e Imágenes de mejora de bordes, y deje los demás ajustes como predeterminados.
- Para conectar la imagen corregida a IMARIS, selecciónela y haga clic en Plugins | IMARIS | Imagen a IMARIS.
- Guárdelo en la carpeta correspondiente haciendo clic en Archivo | Guardar como.
- Para visualizar todos los canales adquiridos, haga clic en Editar | Mostrar ajuste de pantalla. Seleccione el canal de dextrano y el tiempo 1 (fotograma 2 en la parte inferior de la pantalla).
- Cree una nueva superficie que defina el lumen vascular en el punto de tiempo 1 marcando la casilla de verificación agregar nueva superficie (objeto azul)
- En Surface 1, vaya a Propiedades y desmarque todos los parámetros en Configuración del algoritmo. Haga clic en el siguiente paso (flecha azul)
- Seleccione el canal vascular (dextrano en este caso) en canal de origen y marque Resta suave y de fondo. Deje todos los demás parámetros en los valores predeterminados. Haga clic en el siguiente paso (flecha azul).
- Ajuste el valor del umbral de acuerdo con la apariencia de la superficie en términos de grosor y cobertura de todas las áreas positivas. Haga clic en el siguiente paso (flecha azul).
- Excluya superficies no específicas (en su mayoría puntos pequeños) utilizando el filtro de forma predeterminada Número de vóxeles. Haga clic en el paso final (flecha verde).
- Si es necesario, continúe ajustando la superficie manualmente haciendo clic en editar (icono de lápiz) en Configuración de parámetros. Para seleccionar un objeto para eliminarlo, haga clic con el botón izquierdo del ratón sobre él y elimínelo seleccionando eliminar. Si el objeto que se va a eliminar está intercalado con otro, desconecte las dos partes separadas de la superficie haciendo clic en Mayús + clic a la izquierda entre las dos y seleccionando Cortar superficie. Repita este paso tantas veces como sea necesario.
- Finalmente, combine todos los segmentos usando el modo de selección (icono de destino a la derecha) y presione control + desplazamiento para recuperar todos los objetos. Cuando todos estén seleccionados, haga clic con el botón izquierdo y haga clic en unificar.
- Para generar la superficie de referencia, copie esta superficie en los otros puntos de tiempo. Seleccione la superficie global y duplíquela haciendo clic en Duplicar y cámbiele el nombre a Intravascular.
- Seleccione la superficie Intravascular y haga clic en Duplicar en todos los puntos temporales.
- Divida la señal de dextrano para distinguir las áreas intra (IN) y extravasculares (OUT). Para crear un canal IN para todos los puntos temporales, haga clic en la superficie Intravascular | Editar (botón de lápiz) | Enmascarar todo.
- En Selección de canal, seleccione el canal apropiado y marque duplicar canal antes de aplicar la máscara. En Configuración de máscara, marque solo Constante interior/exterior con la superficie exterior de vóxeles establecida en 0. Por último, marque también Aplicar a todos los puntos de tiempo.
- En Ajuste de pantalla, se crea un nuevo canal de forma predeterminada con el nombre enmascarado. Cámbiele el nombre.
- Para crear un canal OUT para todos los puntos de tiempo, repita los pasos 6.2.18-6.2.20, pero seleccione Establecer vóxeles dentro de la superficie en 0. Cambie el nombre de este nuevo canal a OUT.
- Para extraer datos estadísticos, haga clic en el icono Estadística | Exportar todas las estadísticas a un archivo (icono de varios disquetes en la parte inferior de la pantalla)
- Para analizar la fuga, divida el canal de suma de intensidad exterior por el canal de suma de intensidad interior para cada punto de tiempo.
- Para fusionar imágenes tomadas antes y después de la inyección de dextrano en la misma serie temporal, vaya al modo Surpass, haga clic en la primera imagen (Tctrl , antes de la inyección de dextran) y en la imagen de lapso de tiempo (tiempo 1, 60 min) para abrirlas, y combínelas haciendo clic en Editar y agregar puntos de tiempo.
Resultados
En la Figura 1 y la Figura 2, se muestra el modelo CAD de un implante de fijación de cabeza de titanio colocado en un cráneo de ratón escaneado, que está diseñado para seguir la estructura anatómica del cráneo y proporcionar un dispositivo liviano y biocompatible capaz de sujetarse firmemente a la etapa del microscopio asegurando la estabilidad a nivel celular. Siguiendo este protocolo paso a paso, el implante se adhiere de forma estable al cráneo del ratón y puede fijarse firmemente al soporte del microscopio mediante su cola de milano, lo que permite una zona de imagen plana para la retención de líquidos y la observación intravital a lo largo del tiempo. Se puede cerrar con una cubierta para minimizar cualquier daño o infección de la herida, lo que permite la repetición de imágenes de la misma área de tejido durante semanas. Una vez despierto, el ratón con un implante en la cabeza puede caminar, alimentarse y tener una rutina regular libremente.
La Figura 3 muestra una vista de escaneo de la vasculatura BM del calvario hecha de capilares heterogéneos, que incluyen arteriolas, capilares de transición y sinusoides. Los vasos están incrustados en un microambiente de tejido complejo en estrecho contacto con la superficie ósea y las células mesenquimales perivasculares. Durante el desarrollo de la leucemia, se pueden detectar células leucémicas aisladas individuales dentro del microambiente BM en estrecha proximidad con los vasos, y su injerto aumenta con el tiempo, llenando la pantorrilla en las últimas etapas de la enfermedad.
En la Figura 4 se muestra cómo las imágenes obtenidas con este protocolo pueden proporcionar datos cuantitativos, que pueden ser analizados con métodos estadísticos. Mostramos cómo segmentar vasos con la herramienta de filamento IMARIS y medir la longitud y el diámetro de los fragmentos vasculares, así como su rectitud. También se puede evaluar la correlación de estos parámetros.
La figura 5 muestra la adquisición de imágenes longitudinales de dos posiciones diferentes de la MO de la pantorrilla durante la progresión de la LMA en los días 4, 7 y 10, y el día 10 se asocia con un injerto de ~50% de la MO con células leucémicas, medido mediante citometría de flujo (no se muestra). Podemos observar una importante remodelación del tamaño de los vasos preexistentes, así como la formación de nuevos vasos en zonas específicas asociadas a la pérdida ósea local.
Finalmente, en la Figura 6, mostramos cómo se puede medir la permeabilidad vascular como un parámetro dinámico con imágenes de lapso de tiempo que muestran la capacidad de diferentes barreras vasculares para retener un tinte fluorescente a lo largo del tiempo.
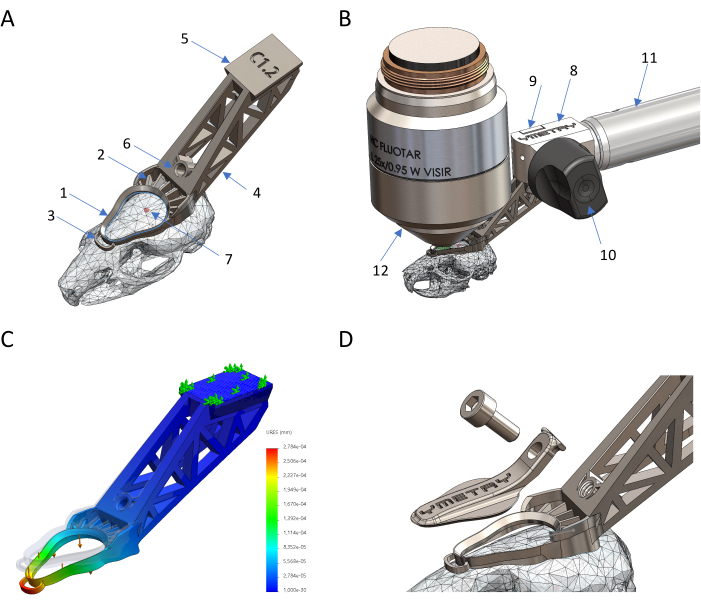
Figura 1: Diseño y producción de un soporte de cabezal biocompatible a base de titanio. (A) Partes del implante in situ: 1 anillo de observación, 2 características de cementación, 3 anclajes estabilizadores, 4 colas, 5 colas de milano, 6 orificios roscados, 7 Bregma. (B) Conexión del implante de la cabeza al soporte: 8 cuerpos de fijación, 9 abrazaderas, 10 palancas excéntricas, 11 estructuras, 12 objetivos de microscopio. (C) Deformación del implante contra carga mediante simulación FEM donde el desplazamiento máximo es de 0,23 μm contra una fuerza de 0,04 N. (D) Tapa protectora y su tornillo. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Preparación del ratón para la obtención de imágenes intravitales. (A) Vista del implante de la cabeza y el área de imagen expuesta quirúrgicamente antes de la obtención de imágenes. (B) Implante de cabeza firmemente unido al cráneo del ratón. (C) Ratón despierto en la jaula de recuperación con la cubierta cerrada en el implante de la cabeza. Haga clic aquí para ver una versión más grande de esta figura.
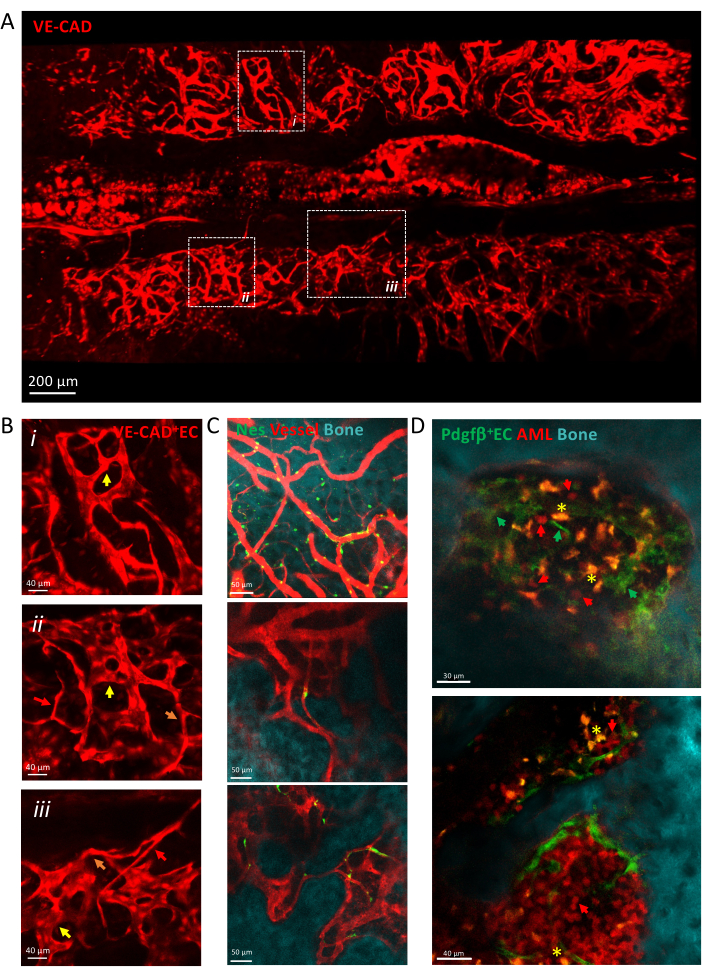
Figura 3: Imagen intravital de la vasculatura de la calvaria. (A) Proyección z de la vista de escaneo de mosaico de la vasculatura de la calvaria marcada por cdh5-DSRED. (B) Acérquese a las áreas representadas para describir diferentes tipos de vasos representados por flechas, arteriolas por flechas rojas, capilares de transición por flechas naranjas, sinusoides por flechas amarillas. (i) y (ii) proyecciones z de tejido X μm; (iii) rebanada única. (C) Cortes individuales de varios campos de visión de los vasos BM, que muestran la superficie ósea (SHG), las células perivasculares (nes-GFP+) y la luz vascular (dextran-TRITC). (D) Nicho vascular asociado con la progresión de la LMA. Segmentos representativos de puntos de tiempo tempranos (superiores) y tardíos (inferiores) del desarrollo de AML. La leucemia MLL-AF9 se marca con tdTOMATO (flechas rojas), mientras que los vasos se marcan con pdgfb-GFP (flechas verdes), la superficie ósea con SHG y los macrófagos con amarillo (autofluorescencia, asterisco amarillo). Barras de escala = 200 μm (A), 40 μm (B,D-panel inferior), 50 μm (C). Abreviaturas: BM = médula ósea; GFP = proteína verde fluorescente; LMA = leucemia mieloide aguda; EC= Células endoteliales. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Cuantificación de parámetros vasculares. (A) Medición de parámetros vasculares a través de la herramienta de filamento IMARIS en una proyección z representativa de vasos de médula ósea marcados con TRITC-dextrano. Se muestran representaciones de líneas y conos. (B) Cuantificación de los parámetros del recipiente en la imagen que se muestra en A. (C) Correlación entre parámetros vasculares que muestran una correlación opuesta entre la rectitud y la longitud del vaso (negativo, Spearman r = -3523; p < 0,0001; R2 = 0,2102) vs diámetro (positivo; Lancero r = 0,4110; p < 0,0001; R2 = 0,1299). Barras de escala = 100 μm. Haga clic aquí para ver una versión más grande de esta figura.
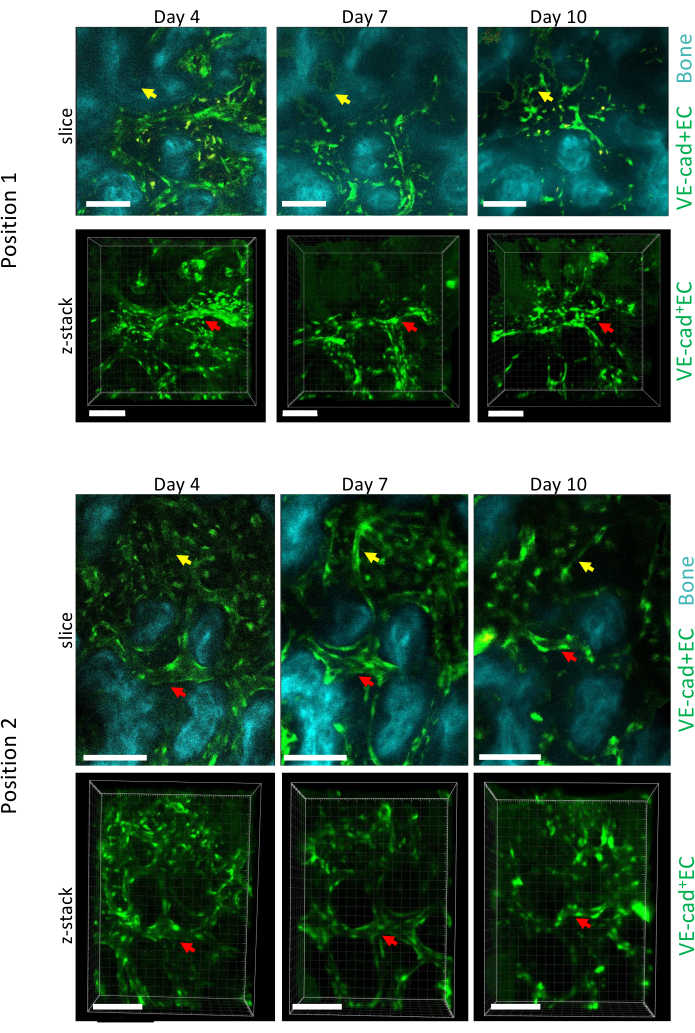
Figura 5: Imagen longitudinal de dos posiciones diferentes de la médula ósea de la calvaria sobre el desarrollo de la LMA. Las células endoteliales que recubren los vasos se marcan con cdh5-GFP, la superficie ósea con SHG y los macrófagos en amarillo (autofluorescencia). Se muestra la remodelación de vasos preexistentes (flechas rojas) y la formación de nuevos vasos (flechas amarillas). Barras de escala = 100 μm. Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: Permeabilidad vascular. (A) Esquemas de la medición de la permeabilidad vascular a través de la herramienta de superficie IMARIS. (B) Proyección Z de la misma área fotografiada longitudinalmente durante 1 h. (C) Cuantificación de la permeabilidad vascular dentro de las áreas como se muestra en A. Haga clic aquí para ver una versión más grande de esta figura.
Figura suplementaria S1: Cráneo preparado. En el plano mediano, crea un eje tangencial a la calvaria y, a continuación, guarda el cráneo preparado. Haga clic aquí para descargar este archivo.
Figura suplementaria S2: Planos espaciados 2 mm. Cómo crear un conjunto de planos igualmente espaciados (espaciado de 2 mm) a lo largo del cráneo. Haga clic aquí para descargar este archivo.
Figura suplementaria S3: Contorno de superficie observable. Cree un boceto en el plano del calvario y cree una spline en forma de pera de AP +6,5 a -2,6 mm de ancho en AP 0,0. Haga clic aquí para descargar este archivo.
Figura suplementaria S4: Vino de observación. Cree un boceto en el plano de la pantorrilla y dibuje una forma de C de 0,5 mm de grosor que se conecte a la ventana de observación . Haga clic aquí para descargar este archivo.
Figura complementaria S5: Haga clic en nuevo estudio. Vaya a la pestaña Simulación (si está visible) o vaya a Simulación | Estudio. En el cuadro de diálogo Estudio , elija Estático como tipo de estudio. Haga clic en Aceptar para crear el nuevo estudio. Haga clic aquí para descargar este archivo.
Figura complementaria S6: Haga clic con el botón derecho para crear la malla. Refinar la malla en las áreas donde se espera una alta tensión o deformación. Haga clic aquí para descargar este archivo.
Discusión
La principal fortaleza de la microscopía intravital es su capacidad para capturar procesos celulares dinámicos en tiempo real dentro de su entorno nativo, superando las limitaciones de los métodos tradicionales de imagen histológica y ex vivo. Mediante la observación directa de la MO de la pantorrilla utilizando las técnicas avanzadas de imagen junto con el marcaje fluorescente descrito en este protocolo, los investigadores pueden estudiar no solo los parámetros funcionales vasculares 8,14, sino también procesos longitudinales como el injerto y la migración de células leucémicas 6,15, la proliferación y la actividad metabólica16,17 preservando la relevancia fisiológica y evitando los artefactos asociados con la fijación y el procesamiento de tejidos.
Las ventajas de las imágenes intravitales BM calvaria sobre los sitios óseos alternativos incluyen la fácil accesibilidad, la transparencia del hueso del cráneo que permite una mejor visualización sin daño óseo, la estabilidad del hueso del cráneo que minimiza los artefactos de movimiento del tejido y la reproducibilidad en animales de experimentación que facilita la comparación entre diferentes grupos experimentales. La posibilidad de recuperar los animales y realizar imágenes longitudinales permite también reducir el número de animales utilizados en un estudio. Vale la pena mencionar que, debido a que se han observado diferencias específicas de la cepa en los patrones de crecimiento de los vasos del cráneo y la consecuente osteogénesis18, es importante tener esto en cuenta al diseñar el implante de imagen específico para la cepa de ratón deseada para permitir una compatibilidad anatómica perfecta y el acceso al sitio de imagen deseado.
Otro punto importante a mencionar son las posibles diferencias en las propiedades vasculares y hematopoyéticas dentro del BM del calvario en comparación con otros huesos, una cuestión poco explorada hasta los últimos años. Estudios novedosos sugieren funciones localizadas para diferentes huesos, con la MO de la pantorrilla diferenciándose de otros huesos en términos de hematopoyesis, estructura y función ósea y vascular 19,20,21,22,23, así como respuesta a patologías neurológicas24. Estas diferencias deben ser exploradas y tenidas en cuenta antes de generalizar los hallazgos específicos de la calvaria.
El diseño y la construcción de un implante de imagen es un paso clave para esta configuración experimental, empezando por la elección del material. Los implantes biocompatibles desempeñan un papel fundamental en la investigación biomédica, facilitando una amplia gama de aplicaciones que van desde la ingeniería de tejidos y la medicina regenerativa hasta los sistemas de administración de fármacos y los dispositivos de monitorización in vivo . La elección del material para un implante de cabeza adaptado para la obtención de imágenes intravitales es fundamental. Idealmente, el material debe exhibir una excelente biocompatibilidad, propiedades mecánicas adecuadas para garantizar una flexibilidad mínima y estabilidad general y, finalmente, la capacidad de integrarse dentro del cráneo sin inducir inflamación. El titanio es ideal por su buena tolerabilidad dentro del cuerpo animal, sin inducción de reacciones adversas en contacto con tejidos biológicos, así como por su resistencia a la corrosión, lo que garantiza la estabilidad a largo plazo incluso si se expone a fluidos corporales o soluciones experimentales. Además, su resistencia mecánica lo hace resistente a la deformación y a la fractura. A pesar de su resistencia, el titanio tiene una densidad relativamente baja, lo que da como resultado dispositivos de imagen intravital livianos que minimizan la carga para los animales de experimentación y los investigadores. Por último, su versatilidad en la fabricación permite la personalización de los dispositivos de imagen intravital para adaptarse a requisitos experimentales específicos, como el tamaño, la forma y la funcionalidad.
Aquí, diseñamos un implante de fijación de cabeza biocompatible utilizando software CAD de modelado paramétrico con capacidades integradas de análisis de elementos finitos (FEA), específicamente SolidWorks. Este enfoque permite ajustes precisos e iterativos a los requisitos estructurales y espaciales del implante, lo que garantiza tanto la compatibilidad anatómica como la resistencia mecánica. Las alternativas gratuitas, como FreeCAD, ofrecen funcionalidades similares de modelado y simulación. Los insumos principales para el proceso de diseño incluyen un modelo anatómico de alta resolución del cráneo del ratón, un modelo del objetivo del microscopio y un modelo del sistema de fijación, que en conjunto informan las dimensiones y la configuración del implante. El resultado final es un archivo de malla independiente de la plataforma, normalmente en formato STL o STEP, compatible con la mayoría de los programas de impresión 3D.
El paso inicial consiste en capturar características anatómicas detalladas de la cabeza del ratón utilizando técnicas de imagen de alta resolución, como la microtomografía computarizada in vivo . Este enfoque proporciona el mayor detalle anatómico y precisión, capturando las microestructuras del cráneo. El escaneo 3D de un cráneo desnudo preparado también podría ser una opción. Este método, que utiliza láser o escáneres de luz estructurada, se emplea comúnmente en un cráneo preparado y proporciona contornos de superficie precisos, aunque con menos detalles internos en comparación con la micro-TC. De lo contrario, los modelos de anatomía se pueden descargar de publicaciones y bases de datos de código abierto25 o DigiMorph {https://www.digimorph.org/specimens/Mus_musculus/}. Si bien son convenientes, estos modelos pueden carecer de detalles específicos del espécimen, por lo que a menudo se necesitan ajustes para el animal específico en el estudio, como el escalado a la distancia real de Bregma-Lambda. Los datos adquiridos se utilizan para crear un modelo 3D preciso del cráneo del ratón, que sirve como plantilla fundamental para el diseño del implante.
Para asegurar el implante en el cráneo, se diseña una estructura de cementación para cubrir la superficie restante del cráneo que no está ocupada por la ventana de observación. Esta estructura debe proporcionar puntos de fijación robustos al tiempo que evita las características anatómicas críticas. La estructura de cementación tiene múltiples aberturas para garantizar una polimerización óptima del cemento debajo del implante a través de la difusión. Además, las paredes de la estructura de cementación tienen un ángulo de tiro pequeño, lo que permite que el cemento se ancle de forma segura contra estas paredes en ángulo. Se diseña la cola del implante, que se extiende desde el cuerpo principal hasta el sistema de fijación. Este componente es crucial para alinear y estabilizar el implante durante la observación, y su diseño debe tener en cuenta el espacio disponible y las limitaciones anatómicas de la cabeza del ratón. Por último, se integra un mecanismo de cola de milano en el diseño del implante para facilitar la fijación y separación del implante del sistema de fijación. Esta característica mejora la practicidad y la facilidad de uso del implante durante las observaciones repetidas. El mecanismo de cola de milano proporciona una fijación repetible para una fácil recuperación de los tejidos observados para sesiones de imágenes repetidas.
Los lectores que estén considerando la adopción de la microscopía intravital de la médula ósea del calvario deben evaluar cuidadosamente sus objetivos de investigación y los requisitos experimentales para determinar si este método es apropiado para sus estudios. Si bien la microscopía intravital ofrece información sin precedentes sobre la hematopoyesis, la regulación del microambiente tisular y la dinámica vascular in vivo, también presenta ciertos desafíos y limitaciones técnicas. Los investigadores deben estar preparados para abordar estos desafíos a través de un diseño experimental cuidadoso, la optimización de los parámetros de imagen y la utilización de controles apropiados. Además, los investigadores deben considerar la disponibilidad de equipos de imagen especializados, la experiencia en técnicas de etiquetado fluorescente y los recursos computacionales para el análisis de imágenes. En general, las imágenes de microscopía intravital de la MO de la pantorrilla tienen un enorme potencial para avanzar en nuestra comprensión de la hematopoyesis y la biología vascular, ofreciendo una ventana única a los procesos dinámicos que ocurren dentro del microambiente de la MO.
Divulgaciones
Jozsua Fodor es el fundador de la empresa YMETRY (n.º de registro 888312352). Los demás autores no tienen conflictos de intereses.
Agradecimientos
Los autores quieren agradecer a todo el personal del IMAG'IC y de las instalaciones de animales del Institut Cochin por su apoyo con los experimentos de microscopía y el alojamiento de ratones. Los ratones Tg(Nes-EGFP)33Enik y Tg(Pdgfb-icre/ERT2)1Frut fueron un amable regalo del Dr. Bonnet (The Francis Crick Institute, Londres). Tg(Cdh5-cre/ERT2)1Rha y B6. Los ratones Cg-Gt(ROSA)26Sortm9(CAG-tdTomato)Hze/J fueron un amable regalo del Dr. Rafii (Weill Cornell Medicine, Nueva York). El trabajo descrito ha sido apoyado por el CNRS, el INSERM y la Université de Paris Cité, y ha sido becado por ATIP-AVENIR, Fondation ARC pour la recherche sur le cancer (R19084KS - RSE20008KSA), Ville de Paris "Emergence" (R20192KK - RPH20192KKA), Laurette Fugain (R23197KK), Cancéropôle IDF (RPH23177KKA), INCA PLBIO (RPH21162KKA), Fondation de France (RAF23152KKA), Ligue contre le cancer (282273/807251), Institut du cancer Paris Carpem, Asociación Europea de Hematología (RAK23130KKA) y Consejo Europeo de Investigación ERC-STG (EEA24092KKA). La instalación central de IMAG'IC cuenta con el apoyo de la Infraestructura Nacional France BioImaging (subvención ANR-10-INBS-04). El laboratorio Passaro está afiliado al "Institut Hors Murs des Sciences Cardiovasculaires" y al "Leukemia Institute Paris Saint-Louis.
Materiales
| Name | Company | Catalog Number | Comments |
| Amira | Thermo ScientificTM | ||
| Anesthesia | Isoflurane 2% to 3% | ||
| Anesthesic mask (animal detection) | Minerve | ||
| Anesthesic unit | Minerve | ||
| B6.Cg-Gt(ROSA)26Sortm9(CAG-tdTomato)Hze | Jackson laboratories | MGI:3809523 | |
| CalvariaVue | Ymetry | https://ymetry.com/site/head-fixation-implants/44-mouse-head-fixation-implant-for-calvaria.html | |
| Dental cement | Kemdent | SUN527 | |
| Dextran 500 kDa-Cy5 | Tebu-bio | DX500-S5-1 | 3 mg/mouse |
| Dextran 70 kDa-TRITC | Sigma | T1162 | 3 mg/mouse |
| Disinfectant | MP-Labo | Dermidine-60ml | |
| Electric razor | Aescular | Isis | |
| Eye gel | Ocry-gel | 10g | |
| Fiji | https://imagej.net/software/fiji/downloads | v 17 May 30 | |
| Fiji plugings to bridge with Imaris | https://imagej.net/software/fiji/downloads | Imaris_Bridge96.jar ; ImarisBridgeUtils.jar | |
| Heating box | Datesand | Thermacage | |
| Heating pad for surgery | Minerve | ||
| Imaging heating pad & rectal probe | F. Haer | ||
| Imaris v9.6.0 | Oxford instruments | ||
| Intrasite gel | Chinoxia | 2390766 | |
| LAS AF Software | Leica | LAS X 3.5.7.23225 | |
| Medication | Buprecare, 0.01 mg/kg | ||
| Objective HCX IRAPO L 25x/0.95 WATER | Leica | 506374 | |
| Saline buffer (PBS 1x) | Sigma | P4417 | Sterilize by autoclave |
| SP8 DIVE FALCON Multiphoton Microscope | Leica | ||
| Stereotoxic mask | Minerve | 1201261 | |
| Sterilizator beads | Sigma | Z742555 | |
| Surgery tools | Moria | 4877A; 2183 | |
| Survival blanket | SECURIMED | 11006 | |
| Swabs / Tissues | Sterilize by autoclave | ||
| Syringe 1 mL 26 G | BD Plastipak | 305501 | |
| Temperature controller | F. Haer | 40-90-5D-02 | |
| Tg(Nes-EGFP)33Enik mice | Jackson laboratories | MGI:5523870 | |
| Tg(Pdgfb-icre/ERT2)1Frut mice | Jackson laboratories | MGI:3793852 | |
| Tg(Cdh5-cre/ERT2)1Rha mice | Jackson laboratories | MGI:3848982 | |
| Ultrasound gel | Parker laboratories | Aquasonic 100 |
Referencias
- Lo Celso, C., et al. Live-animal tracking of individual haematopoietic stem/progenitor cells in their niche. Nature. 457 (7225), 92-96 (2009).
- Pittet, M. J., Weissleder, R. Intravital imaging. Cell. 147 (5), 983-991 (2011).
- Itkin, T., et al. Distinct bone marrow blood vessels differentially regulate haematopoiesis. Nature. 532 (7599), 323-328 (2016).
- Christodoulou, C., et al. Live-animal imaging of native haematopoietic stem and progenitor cells. Nature. 578 (7794), 278-283 (2020).
- Upadhaya, S., et al. Intravital imaging reveals motility of adult hematopoietic stem cells in the bone marrow niche. Cell Stem Cell. 27 (2), 336-345.e4 (2020).
- Hawkins, E. D., et al. T-cell acute leukaemia exhibits dynamic interactions with bone marrow microenvironments. Nature. 538 (7626), 518-522 (2016).
- Duarte, D., et al. Defining the in vivo characteristics of acute myeloid leukemia cells behavior by intravital imaging. Immunol Cell Biol. 97 (2), 229-235 (2019).
- Passaro, D., et al. Increased vascular permeability in the bone marrow microenvironment contributes to disease progression and drug response in acute myeloid leukemia. Cancer Cell. 32 (3), 324-341.e6 (2017).
- Jia, Y., et al. FLT3 inhibitors upregulate CXCR4 and E-selectin ligands via ERK suppression in AML cells and CXCR4/E-selectin inhibition enhances anti-leukemia efficacy of FLT3-targeted therapy in AML. Leukemia. 37 (6), 1379-1383 (2023).
- Pendleton, E. G., Tehrani, K. F., Barrow, R. P., Mortensen, L. J. Second harmonic generation characterization of collagen in whole bone. Biomed Opt Express. 11 (8), 4379-4396 (2020).
- Ventalon, C., Bourdieu, L., Leger, J. F., Fodor, J. Dispositif de liaison d'un animal de laboratoire à au moins un système d'expérimentation, et procédé de fixation d'un tel dispositif. France patent. , (2021).
- Horton, S. J., et al. Acute myeloid leukemia induced by MLL-ENL is cured by oncogene ablation despite acquisition of complex genetic abnormalities. Blood. 113 (20), 4922-4929 (2009).
- Scott, M. K., Akinduro, O., Lo Celso, C. In vivo 4-dimensional tracking of hematopoietic stem and progenitor cells in adult mouse calvarial bone marrow. J Vis Exp. (91), e51683 (2014).
- Jung, Y., et al. Intravital imaging of mouse bone marrow: Hemodynamics and vascular permeability. Methods Mol Biol. 1763, 11-22 (2018).
- Le, V. H., et al. In vivo longitudinal visualization of bone marrow engraftment process in mouse calvaria using two-photon microscopy. Sci Rep. 7, 44097 (2017).
- Spencer, J. A., et al. Direct measurement of local oxygen concentration in the bone marrow of live animals. Nature. 508 (7495), 269-273 (2014).
- Yang, M., Mahanty, A., Jin, C., Wong, A. N. N., Yoo, J. S. Label-free metabolic imaging for sensitive and robust monitoring of anti-CD47 immunotherapy response in triple-negative breast cancer. J Immunother Cancer. 10 (9), e005199 (2022).
- Li, W., et al. Tracking strain-specific morphogenesis and angiogenesis of murine calvaria with large-scale optoacoustic and Ultrasound Microscopy. J Bone Miner Res. 37 (5), 1032-1043 (2022).
- Lassailly, F., Foster, K., Lopez-Onieva, L., Currie, E., Bonnet, D. Multimodal imaging reveals structural and functional heterogeneity in different bone marrow compartments: functional implications on hematopoietic stem cells. Blood. 122 (10), 1730-1740 (2013).
- Rindone, A. N., et al. Quantitative 3D imaging of the cranial microvascular environment at single-cell resolution. Nat Commun. 12 (1), 6219 (2021).
- Mills, W. A., Coburn, M. A., Eyo, U. B. The emergence of the calvarial hematopoietic niche in health and disease. Immunol Rev. 311 (1), 26-38 (2022).
- Bixel, M. G., et al. Angiogenesis is uncoupled from osteogenesis during calvarial bone regeneration. Nat Commun. 15 (1), 4575 (2024).
- Koh, B. I., et al. Adult skull bone marrow is an expanding and resilient haematopoietic reservoir. Nature. 636 (8041), 172-181 (2024).
- Kolabas, Z. I., et al. Distinct molecular profiles of skull bone marrow in health and neurological disorders. Cell. 186 (17), 3706-3725.e9 (2023).
- Rosenhain, S., et al. A preclinical micro-computed tomography database including 3D whole body organ segmentations. Sci Data. 5, 180294 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados