Method Article
颅骨骨髓血管动力学的活体纵向成像
摘要
活体显微镜可以研究动态生物过程,例如组织再生和肿瘤发展。颅骨骨髓是一种高度动态的组织,有助于了解造血和血管功能。使用生物相容性 3D 打印的头部固定植入物可以进行重复纵向成像,从而增强我们对组织动力学和肿瘤微环境的理解。
摘要
活体纵向荧光显微镜成像已成为研究动态生物过程的关键技术,特别是在组织再生、肿瘤发展和治疗反应的背景下。特别是,颅骨骨髓是一种高度动态的组织,其中造血命运与周围的微环境相互连接,专门的血管对正常和病理造血做出反应。固定组织的传统成像提供静态信息,通常会限制对这些过程的全面理解。
表达细胞特异性标记物的转基因动物、活细胞示踪剂的整合、成像设备的进步以及专用腔室的使用已将活体显微镜提升为深入了解动态生物现象的关键工具。活体成像的一个应用是研究肿瘤血管行为和治疗效果。新设计的 3D 打印钛头固定植入物可以稳定地连接到小鼠颅骨,适用于多次治疗的纵向成像。
拟议的方案允许对颅骨骨髓中的血管动力学进行空间和时间检查,包括血管异质性的可视化和量化、与基质和造血细胞的相互作用以及血管功能参数的测量。此外,该技术能够使用双光子显微镜对已建立的血管床进行可视化,并监测治疗效果、干细胞动员和化疗化合物随时间推移的定位。总体而言,这种活体纵向成像方案为研究肿瘤血管行为和造血细胞动力学提供了一个全面的平台,为控制这些生物现象的复杂过程提供了有价值的见解。
引言
颅骨骨髓 (BM) 的活体显微镜成像是研究 BM 生态位内造血、组织微环境调节和血管动力学动态过程的一种强大且不可或缺的技术。这种方法的主要目的是能够实时可视化和分析 体内 BM 微环境中的细胞行为、相互作用和空间组织。通过使用先进的成像技术结合荧光标记直接观察 BM 颅骨,研究人员可以阐明造血干细胞 (HSC)、基质细胞和周围脉管系统之间的复杂相互作用,从而为造血和免疫反应的调节提供重要见解。
活体显微镜成像干细胞的开发和利用摆脱了传统组织学和离体成像方法的局限性,这些方法通常无法捕捉细胞行为和组织相互作用的动态性质。与静态成像技术不同,活体显微镜使研究人员能够实时观察细胞动力学,从而可以在不牺牲实验动物的情况下研究其天然生态位内的纵向过程,例如细胞迁移、增殖和分化。此外,活体显微镜提供了研究体内功能行为的独特优势,例如血管功能(例如灌注、通透性、缺氧),从而保持生理相关性并避免与组织固定和加工相关的伪影。该领域的开创性研究已经证明了这种方法的巨大优势 1,2,他们的发现已被最近改进的方法 3,4,5 证实和扩展,这些方法利用活体显微镜来跟踪内源性 HSC 定位、迁移以及与 BM 生态位内脉管系统的相互作用。此外,活体显微镜检查有助于阐明造血系统疾病的潜在机制,例如白血病和 BM 衰竭综合征,为白血病细胞运动 6,7、疾病相关血管影响8 和药物反应9 提供新的见解。
颅骨 BM 覆盖替代骨部位的活体显微镜成像有几个优点。首先,与股骨或胫骨等较深的骨骼相比,颅骨中包含的 BM 为活体成像提供了便利。这种可访问性有助于通过二次谐波产生 (SHG) 可视化10 直接观察组织微环境,包括骨骼本身,而无需侵入性外科手术。其次,颅骨相对较薄且半透明,可以更好地可视化 BM 生态位内的细胞动力学。这种透明度有助于使用双光子显微镜、共聚焦显微镜和光片显微镜进行高分辨率成像,而无需骨稀释或透明化技术。颅骨为活体成像实验提供了一个稳定、平坦和刚性的平台,最大限度地减少了组织运动伪影,并确保了长时间观察期间成像条件的一致性。这种稳定性特别有利于跟踪细胞行为和反应随时间变化的纵向研究。可重复性是另一个巨大的优势,因为实验动物的颅骨结构相对较小且空间定义。这种均匀性有助于不同实验组之间的比较,并能够对成像数据进行稳健的统计分析。
在这里,我们描述了一种通过活体双光子显微镜对小鼠颅骨 BM 进行成像的方法,方法是引入一种新开发的头部固定植入物11,该植入物使用生物相容性 23 级钛合金 (Ti6Al4V) 进行 3D 打印,它具有由相同钛合金制成的专用且易于定位的硬盖,可以安全闭合伤口,以避免感染或损伤手术部位。植入物通过植入物支架将小鼠头部牢固稳定地固定在显微镜载物台上,最大限度地减少呼吸伪影,并允许随着时间的推移对同一区域进行纵向成像。提供了一些多色图像的示例,这些图像描绘了来自 BM 微环境(SHG + 骨表面;巢蛋白 GFP + 间充质细胞;cdh5-DSRED+、cdh5-GFP+ 或 pdgfb-GFP+ 内皮细胞)和恶性造血区室(tdTOMATO+ AML 细胞),以及描绘血管腔的荧光造影剂(葡聚糖-TRITC)。在数小时或数天内测量的血管参数,包括血管长度、直线度和直径,以及不同血管区域的通透性,可以提供有关组织行为和健康状况的重要信息。
研究方案
所有动物实验均根据道德协议 APAFIS#27215-2020041513522374 v6 进行,该协议已获得法国“Ministère de l'enseignement supérieur, de la recherche et de l'innovation”的批准。
1. 用于 3D 打印的生物相容性钛头部固定植入物的设计
注意:我们使用具有内置有限元分析 (FEA) 功能的参数建模计算机辅助设计 (CAD) 软件设计了一种生物相容性头部固定植入物(参见 材料表)。设计过程的主要输入包括小鼠颅骨的高分辨率解剖模型、显微镜物镜模型和固定系统模型,它们共同决定了植入物的尺寸和配置。最终输出是一个独立于平台的网格文件,通常为 STL 或 STEP 格式,与大多数 3D 打印软件兼容。这种文件格式可确保无缝传输到 3D 打印机,以实现精确的植入物制造。
- 准备鼠标的 3D 模型
- 将准备好的或 体内的小鼠头骨置于显微 CT 扫描仪中。
- 设置扫描参数以获得最佳精细解剖细节的分辨率:打开 控制面板 ,然后在扫描 条件 部分选择视野 (FOV)。使用 LiveView 定义扫描卷。
注意:通过探测器分辨率,FOV 将根据探测器分辨率确定体素大小。在较新的显微 CT 机器中, 5 mm FOV 通常对应于 5 μm 体素大小,10 mm FOV 对应于 10 μm 体素大小,25 mm FOV 对应于 21 μm 体素大小。在此分辨率下的有限 FOV 可能需要多次扫描才能覆盖整个头骨。多次扫描需要在扫描之间重叠。 - 在成像软件中将多个高分辨率扫描拼接在一起,以创建单个全面的颅骨模型。
- 将显微 CT DICOM 文件导入分割软件(例如 3D Slicer、Amira、Avizo 或 Mimics)以隔离颅骨。
- 执行基于阈值的分割,将骨组织与周围结构分开,并进行手动调整以优化复杂或嘈杂的区域。
- 在 3D Slicer 中,首先导入 DICOM 文件并加载数据集,然后在 轴向、 矢状和 冠状 切片视图中观察 CT 切片。
- 打开 Segment Editor,单击 Add 以创建新的区段 |Add 以创建新区段,该区段将进行阈值处理。
- 在 Segment Editor 面板中,单击 Threshold 效果。调整 Lower threshold 和 Upper threshold 滑块(或键入数值),以便要分段的区域在切片视图中以红色突出显示。阈值 3D 段已准备就绪。
- 要将分割后的头骨导出为 STL 文件,请切换到 Segmentation Module(分割模块)。确保已选择阈值区段。在 Export/Import Models and Labelmaps 部分中,将 Export type 设置为 Models。和 文件格式:STL。点击 Export 按钮。
注意:STL 文件使用三角形网格格式,用数千个三角形表示头骨表面,以精细的细节捕捉解剖形状。- 如有必要,请在 MeshLab 中进行拼接,如下所示:
- 通过单击 File (文件) |Import Mesh 并选择一个作为参考扫描。
- 使用 “对齐”工具 (滤镜 |注册 |Align) 通过标记相应的点并使用 Process.
- 通过单击 过滤器 |Mesh Layer (网格层) | 使用 Merge Only Visible Layers 选项拼合可见图层。
- 如有必要,请在 MeshLab 中进行拼接,如下所示:
- 清理并修复网格,消除重复的顶点和三角形,如下所示,以确保去除噪声引起的三角形并填充任何意外的孔洞,以获得光滑连续的表面:
- 点击 过滤器 |清洁和维修 |Remove Duplicated Vertices (删除重复的顶点)。
- 点击 过滤器 |清洁和维修 |Remove Duplicated Faces(删除重复的面)。
- 点击 过滤器 |清洁和维修 |删除孤立的碎片(按面编号)。指定要保留 的最小面数 的阈值(例如,将此阈值设置为较低以删除小簇或噪波)。
- 点击 过滤器 |重新划分网格、简化和重建 |关闭孔。
- 将网格简化为 ~10,000-30,000 个三角形,因为来自 micro-CT 的初始 STL 文件包含非常高的三角形数量,与 CAD 软件不兼容。在高细节区域,在颅骨顶部保留较小的三角形,以确保植入物准确贴合。在较粗糙的区域中,使用较大的三角形简化不太关键的区域,以减小文件大小并优化处理效率。
注意:网格简化按区域进行,详见 1.1.8.1-1.1.8.3。- 使用 Region 选择工具中的 Select Connected Components 选择要简化的区域。
- 使用带参数的选定区域上的 Quadratic Edge Collapse Decimation。点击过滤器 |重新划分网格、简化和重建 |简化:二次边塌折叠抽取。将 Quality Threshold 设置为 0.8,将 Preserve Boundary of the mesh 设置为 YES,将 Preserve topology 设置为 YES,Target Number of faces:设置与所需分辨率对应的目标编号。
- 根据需要迭代,直到三角形的总数减少到所需的级别。然后,将简化的模型保存到 STL 文件。
- 在 CAD 模型中准备颅骨模型
- 将 STL 格式的简化颅骨模型作为新零件或模型导入 CAD 软件。如有必要,请确保将导入的文件正确转换为实体或曲面体,具体取决于软件的功能。将模型保存为原生 CAD 文件格式,以便进行进一步修改并确保与后续设计步骤兼容。
- 将参考点放在 Pregma 和 Lambda 上,并在此模型中创建 Bregma-Lambda 轴。
- 创建包括 Bregma-Lambda 轴的 Median 平面。
- 在正中平面中,创建一个与颅骨相切的轴,然后保存准备好的头骨(补充图 S1)。
- 创建一个新组件,并以 Pregma 与原点重合、中平面与正确平面重合且颅骨轴水平的方式插入准备好的头骨。
- 头部固定植入物将直接在组件内设计,以确保与准备好的颅骨模型完美匹配。为此,请在装配体中创建 implant 作为新零件。首先导航到 Insert |组件 |CAD 软件中的新零件 。出现提示时,选择颅骨曲面或装配体的顶部基准面作为定位和锚定新零件的参考。
- 在头骨上创建一组等距平面(间距为 2 mm)。在每个平面上,创建一个草图,然后在每个草图上,沿着头骨顶面的部分在头骨上方 0.2 毫米处绘制一条 0.2 毫米的偏移投影曲线,该曲线具有九个点的对称样条形式(补充图 S2)。
- 使用这些样条曲线( 颅骨极限曲面)创建一个扫描曲面,确保该表面光滑且没有任何缺陷,用于构建植入物特征。
- 颅骨上可观察表面的定义
注意:根据 3D 模型,确定了非常适合 3D 观察的颅骨最平坦的区域。该区域经过精心选择,以确保双光子显微镜物镜的最佳通道,同时考虑颅骨的生物和结构完整性。该定义可以使用立体定位坐标,其中 Pregma 点作为坐标系的原点。- 在颅骨平面上创建草图,并在 AP 0.0 处创建一个从 AP +6.5 到 -2、6 mm 宽的梨形样条曲线。这将是 可观察表面等值线 (补充图 S3)。
注意:显微镜物镜实际上是相对于颅骨上先前定义的可观察表面定位的。此步骤涉及确定物镜尺寸和形状施加的空间限制,确保植入物设计符合显微镜的要求。 - 将显微镜物镜模型垂直插入组件中。将其焦点约束在平行于颅骨的颅骨平面的平面上,并定义 显微镜物镜的边界体积。
- 通过沿着 Observable Surface Contour 扫描物镜来创建体积。
- 在定义的可观察表面周围创建一个观察窗口,确保显微镜物镜畅通无阻。
注意:观察窗是一个环状结构,具有辅助功能,例如浸液保留和固体结构,用于正确关闭保护盖。 - 在颅骨平面中创建草图并复制可观察的表面轮廓。
- 创建厚度为 0.5 mm 的平行轮廓。将此双轮廓向上拉伸 0.9 毫米并向下拉伸到表面 颅骨极限表面,拔模角度为 4°。
- 在颅骨平面上创建草图,并在 AP 0.0 处创建一个从 AP +6.5 到 -2、6 mm 宽的梨形样条曲线。这将是 可观察表面等值线 (补充图 S3)。
- 定义胶合、尾部和燕尾榫结构。
- 在颅骨平面上创建一个草图,并绘制一个 0.5 毫米厚的 C 形,连接到观察窗,并以 4° 拔模角向下拉伸 2 毫米高并向下拉伸到表面颅骨极限表面(补充图 S4)。
- 在颅骨平面上创建一个草图,并用 0.4 mm 厚的矩形填充此空间,这些矩形径向放置,它们之间有 1 mm 的间距。
- 挤出方式类似于 Observation Window。
- 通过尊重可用体积,依靠颅骨创建植入物的尾部以固定。
- 在胶合结构切线上创建一个横向平面,然后向后再创建一个 12.5 mm。
- 在这些基准面上绘制矩形。在两者之间扫掠金字塔形实体,将角度设置为与水平面的大约 20° 和 37°。
- 在靠近胶合结构的尾部添加一个螺纹 M1.6 孔。
- 在 Tail 的末端创建草图并绘制燕尾榫轮廓。
- 将此轮廓拉伸 8 mm。
- 验证不干扰目标。
- 检查整个植入物设计,包括观察窗和燕尾榫,以确保显微镜物镜没有机械干扰。检查是否干扰了在步骤 1.3.3 中创建的卷。
注意:此验证步骤对于确保物镜可以根据需要无阻碍地定位和移动至关重要。
- 检查整个植入物设计,包括观察窗和燕尾榫,以确保显微镜物镜没有机械干扰。检查是否干扰了在步骤 1.3.3 中创建的卷。
- 从植入物的尾部去除不必要的材料,以减轻重量并减少动物的潜在不适。
- 在中间平面上创建一个草图,并在其上绘制三角形切口,以便在从 Tail 主体中通过拉伸提取时形成桁架结构。
- 在垂直平面中重复。
- 通过有限元法 (FEM) 仿真验证最大位移,以分析植入物在各种条件下的机械性能。
注意:此模拟有助于预测使用过程中植入物内的最大位移和应力,确保其可靠性。- 启用 CAD 软件仿真插件。
- 在 Simulation 工具栏中,创建新的 静态算例。导航到 Simulation 选项卡(如果可见)或转到 Simulation |学习。在 Study (分析 ) 对话框中,选择 Static (静态) 作为 分析类型。单击 OK 创建新研究(补充图 S5)。
注意:这种研究类型适用于分析静载荷下的变形和应力,正如植入物的使用所预期的那样。 - 为植入物选择与实际植入物成分相匹配的材料 (Ti6Al4V)。
- 定义固定约束以模拟植入物与燕尾榫固定接触的附着点。将 燕尾榫的顶面 定义为 fixed。
- 定义种植体 底面上的垂直载荷 。设置 1 N 垂直力。
- 生成网格。在预期高应力或变形的区域细化网格(补充图 S6)。为此,请右键单击研究树的网格单元,然后选择 Create Mesh...在 Mesh 对话框中,使用滑块或通过激活 Mesh Control 框来调整 Mesh Density 以单独设置最大和最小单元大小。单击 OK 生成网格。
注意:软件会自动为研究生成初始网格,但为了获得更准确的结果,可以细化网格以选择更精细的网格,特别是在对植入物稳定性至关重要的区域(例如观察窗口)。在这些区域中进行更精细的网格划分可以通过捕获局部应力集中来提高仿真的准确性。 - 开始静态分析以计算整个植入物的位移、应力和应变分布。
- 分析结果。检查位移图解以确定最大变形的区域。该图显示了植入物在施加的载荷下会变形的程度,突出显示了任何有过度移动风险的区域。确保观察窗内的变形不超过 3 微米。
- 最后,设计一个保护罩,以便在不使用时保护观察窗。这个盖子可以保护裸露的骨骼免受环境污染和物理损伤。
2. 小鼠治疗、麻醉和头部植入物的手术植入
注意:这里可以使用雄性或雌性、7 至 12 周龄的 C57BL/6 或转基因小鼠,如图所示。为了诱导 BM 的白血病定植,产生的白血病细胞如 Horton 等人 12 所述,在成像前 2-3 周静脉内给药。为了保证伤口的健康,必须使用无菌技术。
- 在引擎盖下,组装无菌手术工具(钝剪刀和镊子)、修剪器和耗材,包括眼胶、生理盐水、消毒剂、牙科水泥、吸收棉签和清洁纸。打开珠子消毒器。
- 用 4% 异氟醚和大气氧气填充麻醉诱导室,然后将小鼠放入腔室中。当动物完全麻醉时(检查扶正反射是否丧失以及更深和更慢的呼吸模式),将异氟醚流量切换到麻醉面罩的鼻锥,并将异氟醚浓度降低到 2%。
- 将鼠标放在 37 °C 加热垫上,并目视监测呼吸频率。可选:对于具有病理表型的小鼠,添加生存毯以在整个麻醉过程中保持正确的温度。
- 手术前 30 分钟皮下注射止痛药 (Buprecare,0.01 mg/kg)。
- 用电动剃须刀剃掉鼠标头。
- 在小鼠的眼睛上滴一滴眼凝胶,以防止麻醉时干燥,确保凝胶不会滴到头皮上。
- 清洁鼠标以去除手术区域的任何残留毛发。用消毒剂在棉纸上擦拭头皮顶部。请务必去除所有毛发,以避免成像伪影和伤口感染的风险。
- 使用无菌镊子和剪刀(在消毒器中 30 秒后),在头皮的中央部分做一个小切口,露出中央骨疤痕。按照它定义正确的长度和宽度成像区域。小心地去除头皮,用镊子提起耳朵之间的皮肤,然后在后脑勺做一个小切口。抬起皮肤,将剪刀穿过皮肤下,小心地在成像区域外剪开。
- 去除颅骨和头皮之间的结缔组织。用棉签用无菌 PBS 擦拭裸露的骨头。快速将头部植入物连接到小鼠的头骨上,以避免颅骨长时间暴露(<10 分钟)。
- 按照制造商的说明在培养皿中用足够的牙科水泥制成糊状物,然后迅速将其涂抹在头部固定植入物的底部。
- 在成像区域内不让任何牙科粘固剂的情况下,将头部支架放在暴露的鼠标头骨上,然后等待其凝固。
- 凝固后(通常在 3-5 分钟内),在成像室中加入几滴 37 °C PBS 以保持颅骨水分。
3. 使用双光子显微镜进行成像
- 在开始手术之前,打开激光并让它预热以稳定下来。打开显微镜并启动采集软件。选择合适的物镜(本实验为 25x/0.95 WATER 浸泡)。
- 使用固定载物台立式显微镜支架,该支架有足够的空间放置带有加热垫的兽医面包板,所有这些都可以与 xy 载物台整体移动。使用实心铝制光学面包板作为平台来固定头部植入物和立体定位面罩。将与异氟醚注射和萃取相关的柔性软管及其连接器连接到显微镜笼中的面罩上。
- 配置采集设置以快速扫描: 格式 512 x 512 像素;速度 600 Hz; 和 缩放系数 1。
- 在 880 nm 处调整激光线,以便能够检测具有相同激发波长的 SHG、GFP 和 tdTOMATO。
- 在激活 NDD 检测器之前,请关闭房间内的灯并正确关闭显微镜支架周围的盒子,并定义光谱检测门:PMT1,用于检测 SHG [388 - 431 nm]; HyD2,用于 GFP [485 - 548 nm] 和 HyD3 ,用于 tdTOMATO [551 - 645 nm]。将 偏移 量保留为 0。
- 创建新的项目数据集,并根据特定实验对其进行重命名。
- 一旦头部固定植入物连接到小鼠头骨上(通过水泥硬化确认),将异氟醚朝向显微镜的立体定位面罩打开。
- 将鼠标快速带到显微镜前。小心地将小鼠牙齿插入面罩中,用一只手抬起小鼠的鼻子,让异氟醚穿过小鼠鼻子。
- 一只手握住鼠标,用另一只手轻轻地将头部固定植入物的燕尾榫滑入固定支架中,然后用螺丝旋钮半圈将其固定。
- 引入预埋有水基凝胶的直肠探针,用于温度监测,并在小鼠眼睛中添加滴眼液。
- 用大量水基凝胶或 PBS 填充头部成像室,并降低水浸物镜以使其完全浸没,以获得最佳激发和检测双光子激发产生的信号。
- 移动 x-y 舞台和 z 驱动器,将焦点放在支架疤痕中的中央骨骼上。
注意:使用显微镜的目镜和金属卤化物型灯作为光源,通过三波段激发发射滤光片过滤,非常方便。缺乏光学切片使得无法获得清晰的图像;但通过练习,颅骨的中央区域很容易识别。 - 识别出以骨表面和中央静脉为标志的组织后,关闭金属卤化物灯并关闭显微镜笼窗以保护 HyD 检测器并让红外 (IR) 激光通过。
- 将 PMT/SHG 增益 设置为 850 V (其线性工作范围),并将 HyD 增益 设置为 100%。增加 IR 激光功率,直到在三个通道中最低的通道上获得动态范围为 200 灰度级(使用 8 位检测器动态)的图像。如果发射差异太重要或存在饱和,则使用“低于”查找表 (LUT) 模式来降低其他检测器的增益。
注意:在我们的例子中,30% 的激光功率足以在所有通道中获得对比度。 - 在采集软件上,搜索由包裹在骨表面 (SHG) 内的 BM 口袋(GFP +、tdTOMATO +)组成的区域。
- 要找到不同的 ROI,请激活 LAS NAVIGATOR 并使用 螺旋模式创建整个区域的概览。当采集的表面足够大时停止采集。保存概览,合并并重命名:这将是纵向成像时的参考图像。
注意:小心不要产生太大的螺旋,因为这可能会导致设备支架与物镜接触。 - 通过单击 单个图像 图标记录一些 ROI 位置,并重命名 任务列表中的每个位置。进行屏幕截图,将各种 ROI 的相对位置叠加在之前保存的概览上。
注意:此屏幕截图对于替换纵向成像中的 ROI 至关重要,因为采集软件不允许在用于此实验的版本中调用这组位置。 - 要获取 z 堆栈卷,请选择 z 堆栈模式,定义 3 μm 的步长,然后取消选择所有区域的相同堆栈大小选项。选择第一个位置,定义顶部和底部位置,检查 z 步长,然后单击 redefine stack。在位置列表中,位置名称旁边会显示一个立方体图标。对不同 ROI 的所有位置重复此步骤。
- 按 acquire 并将图像(具有 ~50 个计划的三个通道 z 堆栈)保存在相应的文件夹中。此步骤标志着实验的 Tctrl ;检查采集参数是否正确(在三个通道中获得正确的动态范围,尽可能避免信号饱和,并检查整个周期的采集时间)。
- 要测量动态特征(在本例中为血管通透性),请获取延时摄影。
- 在这种设置中,三个探测器允许同时对三个通道进行成像;如果实验需要第四个通道(在本例中为 Dextran 监测),则激活 序列模式 并添加第二个采集序列。在 820 nm 处调整 IR 激光线,以便能够激发葡聚糖 Cy5 荧光团。将 Hyd2 检测范围更改为 [650 - 744 nm] 并停用其他检测器。 在堆栈 采集模式之间进行选择。
注意:默认情况下,第二个采集序列与序列 1 具有相同的参数。 - 在开始延时摄影之前,启动所有位置的采集,以确保记录时间不超过 3 分钟(考虑到添加顺序模式),这是检测葡聚糖泄漏所需的最短时间。
- 在葡聚糖进样之前,将 采集模式 更改为 xyzt。在 t 模块中,将 时间间隔 调整为 3 分钟 , 将持续时间 调整为 1 小时。
- 在这种设置中,三个探测器允许同时对三个通道进行成像;如果实验需要第四个通道(在本例中为 Dextran 监测),则激活 序列模式 并添加第二个采集序列。在 820 nm 处调整 IR 激光线,以便能够激发葡聚糖 Cy5 荧光团。将 Hyd2 检测范围更改为 [650 - 744 nm] 并停用其他检测器。 在堆栈 采集模式之间进行选择。
- 以 3 mg/小鼠的剂量静脉注射 100 μL 葡聚糖 70 KDa-TRITC(或 500 KDa-Cy5)并开始采集。
- 采集后,将数据集保存在相应的文件夹中。
注意:在这许多步骤中,请经常监测鼠标的呼吸频率和温度,并在必要时调整异氟醚流量。理想情况下,可以记录体温、心电图和呼吸频率并作为实验的元数据。
4. 鼠标恢复
- 成像会话完成后,在采集软件中停用 HyDs 检测器并打开鼠标加热箱 (37 °C)。打开显微镜笼,提起物镜,然后从鼠标中取出直肠探针。接下来,轻轻地将头部固定植入物的燕尾榫从固定支架中滑出,然后将鼠标从显微镜的立体定位面罩移动到加热垫上的手术面罩上。
- 在显微镜下关闭异氟醚注射并关闭加热垫。
- 用无菌棉签轻轻去除小鼠颅骨上的水基凝胶或 PBS。
- 如前所述,添加现场内凝胶以在成像之间保持颅骨水分13。
- 用特定的盖子封闭头部植入物的成像区域,并用螺钉小心地固定它。
- 将鼠标放入 37 °C 的加热箱中,等待它醒来。将小鼠带到动物设施,并将其安置在装有水凝胶和富集剂的干净笼子中。每天监测动物以发现任何疼痛或感染的迹象,在这种情况下,每天服用 Buprecare。为了保持颅骨湿度,每周更换 2 次现场内凝胶。
注意:选择带有纸板庇护所和筑巢材料的高笼,并确保将老鼠放入单独的笼子中。
5. 纵向收购
注意:在接下来的几天里,可以再次对鼠标进行成像。但是,请确保每周重复的成像治疗不要超过三次,以避免反复麻醉的不良影响,例如眼睛干涩或过度疲劳,以及呼吸窘迫和体温过低。
- 要对已安装头部支架的鼠标重新成像,请按照步骤 2.2 将一小滴眼用凝胶滴在鼠标的眼睛上,以防止手术过程中干燥。
- 按照第 3 节直到步骤 3.16 获取概览图像。
注意:使用头部钛植入物时,无需改变支架位置的高度或角度,可以很容易地找到整个感兴趣区域。 - 如果需要,请使用 open image and align 模块重新对齐以前的图像和新图像。
- 打开步骤 3.17 中的屏幕截图图像,以在其原始位置标记位置。
注意:使用 SHG 造影剂非常有用,尤其是在预期表达 GFP 的血管和相关细胞重塑时。 - 以步骤 3.18-3.22 中描述的相同方式获取 z 堆栈和延时摄影。
6. 血管参数量化
- 血管参数
- 要在 IMARIS 中打开使用软件生成的图像(.lif 文件),请首先使用文件转换器将它们转换为“.ims”格式。使用 IMARIS 打开“.ims”文件,检查元数据(X、Y、Z 刻度),然后选择 Filament 模块。选择 skip automatic creation, edit manually(跳过自动创建)、edit manually(手动编辑)。
- 为了在灯丝生成过程中更好地可视化,请导航到 设置 (叶子符号),选择 线条 in 样式,并将 像素数 保留为默认值 (1)。勾选 显示树突 框,包括 起点、分支点 和 终点 。取消勾选 Show Spine。
- 在 “绘制 (画笔符号)”下,选择 “方法自动路径”;直径为 5 μm,以及与血管关联的通道源(本例中为 TRITC-葡聚糖)。最后,勾选 automatic center 和 automatic diameter。
- 使用 shift + 鼠标右键单击 选择树枝的 起点 ,使用 shift + 左键单击 选择树枝的 终点 ,开始绘制细丝。
注意:起点为浅蓝色,分支点为红色或深蓝色,终点为绿色。 - 要在绘图时设置正确的细丝直径,请使用鼠标滚动按钮。
- 要连接具有不同终端节点的两个细丝,请在它们之间创建一个新的起点,并在每个终端节点处连接它。
注意:因此,绿色端点出现在分支点中。 - 创建维管树后,返回 设置 并确保起始点、分支点和终点的数量正确。选择 圆锥 体以可视化直径并验证其外观。
- 如果细丝直径(或连接)似乎不正确,请导航到 编辑 (铅笔符号),右键单击选择要删除的细丝,然后单击 删除。使用鼠标滚轮按钮调整灯丝上的直径,重新绘制灯丝。
- 要提取所有生成的统计数据并保存它们,请单击 统计 图标 |将所有统计数据导出到文件-(屏幕底部的多张软盘图标)。
注意:为避免任何与血管不对应的潜在异常细丝,统计分析中仅包括长度超过 20 μm 的细丝。
- 泄漏定量
注意:要在 IMARIS 中打开使用软件生成的图像(.lif 文件),请首先使用文件转换器将它们转换为“.ims”格式。- 要在同一时间序列中合并葡聚糖注射前后拍摄的图像,请转到 Surpass 模式,单击第一张图像(Tctrl ,葡聚糖注射前)和延时图像(时间 1,60 分钟)以打开它们,然后单击 编辑 并 添加时间点来组合它们。
注意:要纠正最终的 3D 漂移,请使用 Fijis 和 IMARIS 之间的桥接功能执行步骤 6.2.2-6.2.7。在斐济安装以下插件:“IMARIS_Bridge96.jar”和“IMARISBridgeUtils.jar”。3D 漂移注册是使用“StackReg Pluging”(已在斐济提供)执行的。如果不需要,请转到步骤 6.2.8 - 要进行 3D 漂移校正,请单击插件 |伊玛丽丝 |图片来自 IMARIS。
- 在斐济,选择 插件 |注册 |校正 3D 漂移。
- 在显示的新窗口中,选择适当的 注册渠道。
注意:在本例中是 Nestin-GFP,但它可以是任何突出显示固定物体和高信噪比的通道。 - 要增强对小漂移的检测,请选择 Multi time scale computation (多时间尺度计算)、Sub pixel drift correction (子像素漂移校正) 和 Edge enhance images(边缘增强图像),并将其他设置保留为默认值。
- 要将校正后的图像桥接到 IMARIS,请选择它并单击 插件 |伊玛丽丝 |图片来自 IMARIS。
- 通过单击 文件 |另存为。
- 要可视化所有获取的通道,请单击 Edit |显示显示调整。选择 葡聚糖通道 和 时间 1 (屏幕底部的第 2 帧 )。
- 通过勾选 Add new surface 复选框(蓝色对象),在时间点 1 创建一个新表面,定义血管腔
- 在 Surface 1 中,转到 Properties 并取消勾选 Algorithm Settings 中的所有参数。单击 Next step (下一步)(蓝色箭头)
- 在源通道下选择血管通道(在本例中为葡聚糖),然后勾选 平滑和背景减法.将所有其他参数保留为默认值。单击 Next step(下一步)(蓝色箭头)。
- 根据表面外观(所有正区域的厚度和覆盖率)调整 Threshold 值。单击 Next step(下一步)(蓝色箭头)。
- 默认情况下,使用过滤器排除非特定表面(主要是小点) 体 素数 (Number of Voxels)。单击 Final step (最后一步)(绿色箭头)。
- 如有必要,通过单击设置参数中的编辑(铅笔图标)继续手动调整表面。要选择要删除的对象,请左键单击该对象,然后选择 delete 进行删除。如果要删除的对象与另一个对象进行整理,请单击 Shift + 单击两者之间的左侧并选择 Cut Surface (切割曲面),以断开曲面的两个分离部分。根据需要多次重复此步骤。
- 最后,使用 选择模式 (右侧的目标图标)合并所有段,然后按 Ctrl + 滚动 以恢复所有对象。选择所有选项后,单击鼠标左键,然后单击 unify。
- 要生成参考表面,请将此表面复制到其他时间点。选择全局表面,然后单击 Duplicate (复制 ) 进行复制,然后将其重命名为 Intravascular(血管内)。
- 选择表面 Intravascular 并单击 Duplicate to all Timepoints。
- 分离葡聚糖信号以区分血管内 (IN) 和血管外 (OUT) 区域。要为所有时间点创建 IN channeI,请单击表面 Intravascular |编辑 (铅笔按钮) |遮罩全部。
- 在 Channel Selection 中,选择适当的通道并在 应用蒙版之前勾选 duplicate channel。在 遮罩设置(Mask Settings) 中,仅勾选 常量内部/外部(Constant inside/outside),并将体素外部表面设置为 0。最后,勾选 也适用于所有时间点。
- 在 Display Adjustment 中,默认情况下会创建一个名称被遮罩的新通道。将其重命名为 IN。
- 要为所有时间点创建通道 OUT,请重复步骤 6.2.18-6.2.20,但选择 将体素内部表面设置为 0。将此新通道重命名为 OUT。
- 要提取统计数据,请单击 Statistic 图标 |Export All Statistics to File(将所有统计信息导出到文件 )(屏幕底部的多个软盘图标)
- 要分析泄漏性,请将 Intensity sum channel outside (外部强度总和通道 ) 除以 每个时间点的 intensity sum channel inside (内部强度总和通道 )。
- 要在同一时间序列中合并葡聚糖注射前后拍摄的图像,请转到 Surpass 模式,单击第一张图像(Tctrl ,葡聚糖注射前)和延时图像(时间 1,60 分钟)以打开它们,然后单击 编辑 并 添加时间点来组合它们。
结果
在图 1 和图 2 中,CAD 模型显示了放置在扫描小鼠头骨上的钛头固定植入物,该植入物旨在遵循头骨的解剖结构,并提供一个轻便且生物相容性的装置,能够牢固地固定在显微镜载物台上,确保细胞水平的稳定性。通过遵循此分步方案,植入物可以稳定地附着在小鼠颅骨上,并可以通过燕尾榫牢固地固定在显微镜支架上,从而形成平坦的成像区域,以便随着时间的推移进行液体保留和活体观察。它可以用盖子闭合,以尽量减少伤口的任何损伤或感染,从而允许在数周内对同一组织区域进行重复成像。一旦醒来,佩戴头部植入物的老鼠就可以自由走动、进食和有规律的作息。
图 3 显示了由异质毛细血管(包括小动脉、过渡毛细血管和正弦管)制成的颅骨 BM 脉管系统的平铺扫描视图。血管嵌入到与骨表面和血管周围间充质细胞紧密接触的复杂组织微环境中。在白血病发展过程中,可以在靠近血管的 BM 微环境中检测到单个孤立的白血病细胞,并且它们的植入随着时间的推移而增加,在疾病的晚期填满颅骨。
图 4 显示了使用该协议获得的图像如何提供定量数据,这些数据可以使用统计方法进行分析。我们展示了如何使用 IMARIS 细丝工具分割血管,并测量血管碎片的长度和直径,以及它们的直线度。还可以评估这些参数的相关性。
图 5 显示了在第 4 天、第 7 天和第 10 天的 AML 进展过程中颅骨 BM 的两个不同位置的纵向成像采集,第 10 天与 ~50% 的 BM 白血病细胞植入有关,通过流式细胞术测量(未显示)。我们可以观察到先前存在的血管大小的重要重塑,以及与局部骨质流失相关的特定区域新血管的形成。
最后,在 图 6 中,我们展示了如何将血管通透性作为动态参数进行测量,延时成像显示了不同血管屏障随时间保留荧光染料的能力。
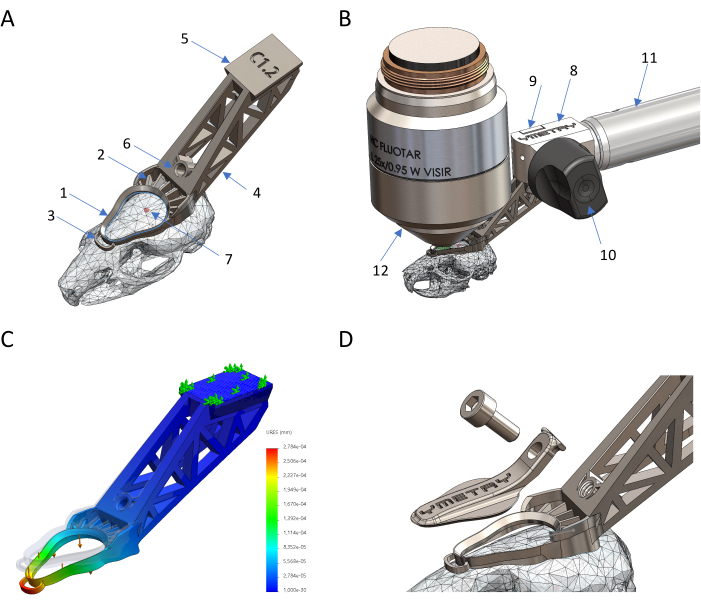
图 1:钛基生物相容性头架的设计和生产。 (A) 原位种植体的部件:1 个观察环、2 个粘接特征、3 个稳定锚、4 个尾部、5 个燕尾榫、6 个螺纹孔、7 个前囟。(B) 头部植入物与支架的连接:8 固定体、9 夹具、10 偏心杆、11 结构、12 显微镜物镜。(C) 通过 FEM 模拟测量的植入物在载荷作用下的变形,其中最大位移为 0.23 μm,作用力为 0.04 N。(D) 保护盖及其螺丝。 请单击此处查看此图的较大版本。

图 2:小鼠用于活体成像的准备。 (A) 成像前头部植入物和手术暴露的成像区域的视图。(B) 头部植入物牢固地附着在小鼠头骨上。(C) 小鼠在恢复笼中醒来,头部植入物上有封闭的盖子。 请单击此处查看此图的较大版本。
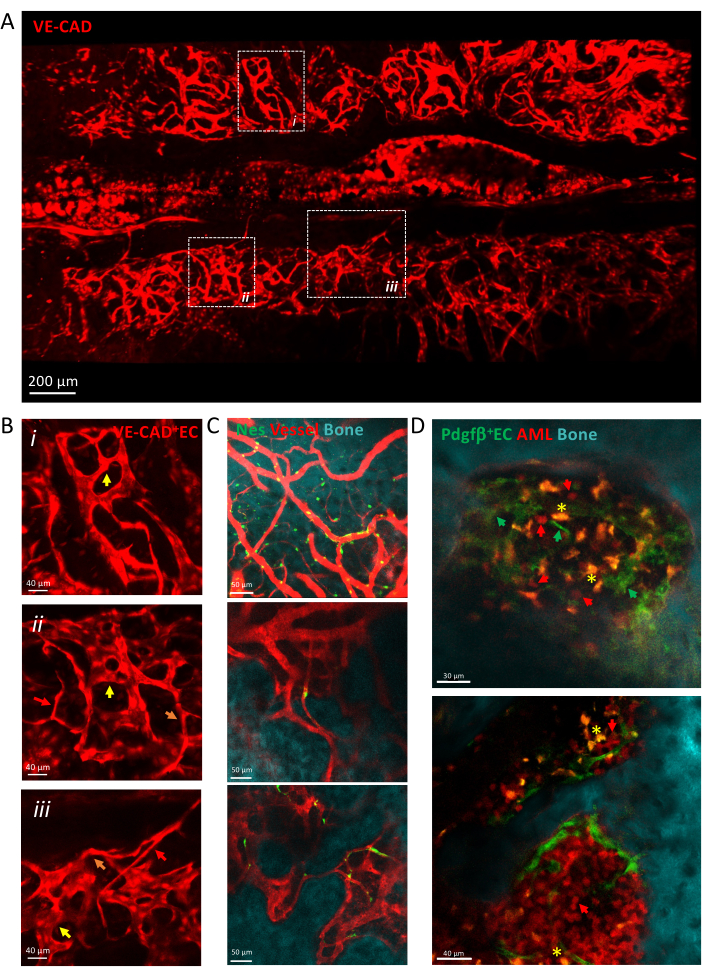
图 3:颅骨脉管系统的活体成像。(A) cdh5-DSRED 标记的颅骨脉管系统的平铺扫描视图的 z 投影。(B) 放大描绘区域以描述箭头描述不同类型的血管,用红色箭头描述小动脉,用橙色箭头描述过渡毛细血管,用黄色箭头描述正弦。(i) 和 (ii) X μm 组织的 z 突起;(iii) 单片。(C) BM 血管多个视野的单片,显示骨表面 (SHG)、血管周围细胞 (nes-GFP+) 和血管腔 (葡聚糖-TRITC)。(D) 与 AML 进展相关的血管生态位。AML 发展的早期(顶部)和晚期(底部)时间点的代表性切片。MLL-AF9 白血病用 tdTOMATO 标记(红色箭头),而血管用 pdgfb-GFP(绿色箭头)标记,骨表面用 SHG 标记,巨噬细胞用黄色标记(自发荧光,黄色星号)。比例尺 = 200 μm (A)、40 μm (B,D - 下面板)、50 μm (C)。缩写: BM = 骨髓;GFP = 绿色荧光蛋白;AML = 急性髓系白血病;EC= 内皮细胞。请单击此处查看此图的较大版本。

图 4:血管参数量化。 (A) 通过 IMARIS 细丝工具 在用 TRITC-葡聚糖标记的骨髓血管的代表性 z 投影中测量血管参数。将显示线和圆锥表示。(B) A 中所示图像中血管参数的 量化。(C) 血管参数之间的相关性显示血管直线度和长度之间存在相反的相关性(负,Spearman r = -3523;p < 0.0001;R2 = 0.2102) 与直径(正;斯皮尔曼 r = 0.4110;p < 0.0001;R2 = 0.1299)。比例尺 = 100 μm。 请点击此处查看此图的较大版本。
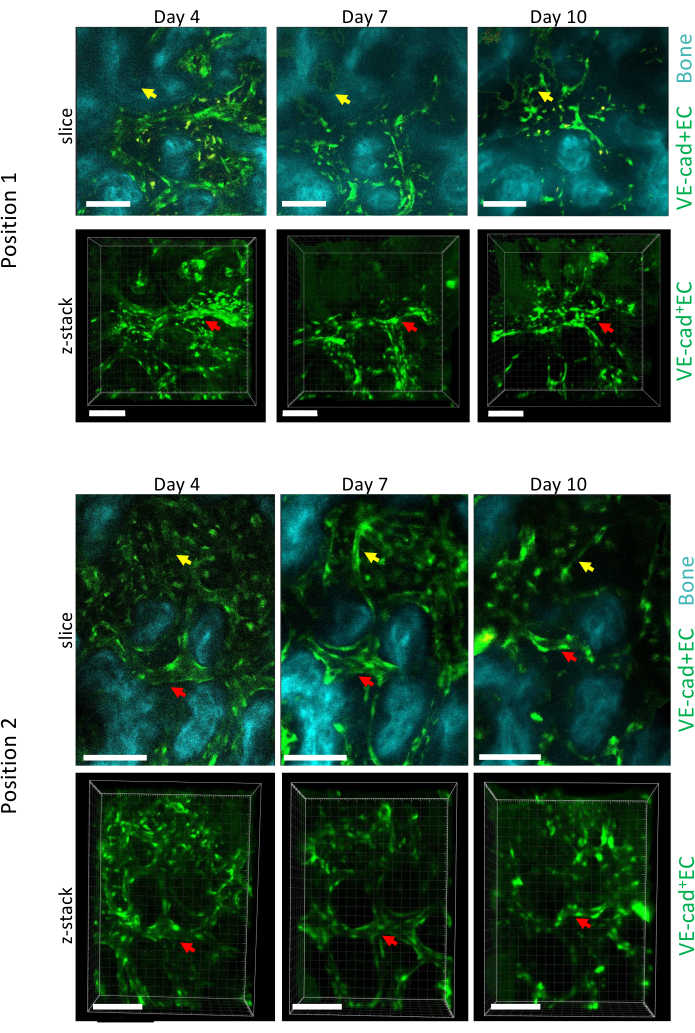
图 5:AML 发展中颅骨 BM 两个不同位置的纵向成像。 血管内皮细胞用 cdh5-GFP 标记,骨表面用 SHG 标记,巨噬细胞用黄色标记(自发荧光)。显示了现有血管的重塑(红色箭头)和新血管的形成(黄色箭头)。比例尺 = 100 μm。 请点击此处查看此图的较大版本。

图 6:血管通透性。 (A) 通过 IMARIS 表面工具 测量血管通透性的示意图。(B) 在 1 小时内纵向成像的同一区域的 Z 投影。(C) 如 A 所示区域内血管通透性的量化。 请单击此处查看此图的较大版本。
补充图 S1:准备好的头骨。 在正中平面中,创建一个与颅骨相切的轴,然后保存准备好的头骨。 请点击此处下载此文件。
补充图 S2:间距 2 毫米的计划。 如何在头骨上创建一组等距平面(间距 2 mm)。 请点击此处下载此文件。
补充图 S3:可观察的表面轮廓。 在颅骨平面上创建草图,并在 AP 0.0 处创建一个从 AP +6.5 到 -2、6 mm 宽的梨形样条曲线。 请点击此处下载此文件。
补充图 S4:观察 vindow。 在颅骨平面上创建草图,并绘制一个 0.5 mm 厚的 C 形,连接到 观察 窗口。 请点击此处下载此文件。
补充图 S5:单击 New study(新建研究)。 导航到 Simulation 选项卡(如果可见)或转到 Simulation |学习。在 Study (分析 ) 对话框中,选择 Static (静态) 作为 分析类型。单击 OK 创建新研究。 请点击此处下载此文件。
补充图 S6:右键单击以创建网格。 在预期高应力或变形的区域细化网格。 请点击此处下载此文件。
讨论
活体显微镜的主要优势在于它能够在其天然环境中实时捕获动态细胞过程,克服了传统组织学和离体成像方法的局限性。通过使用先进的成像技术直接观察颅骨 BM,结合本协议中描述的荧光标记,研究人员不仅可以研究血管功能参数 8,14,还可以研究白血病细胞植入和迁移 6,15、增殖和代谢活动等纵向过程16,17保持生理相关性并避免与组织固定和处理相关的伪影。
与其他骨骼部位相比,BM 颅骨活体成像的优势包括易于接近、颅骨透明性允许在没有骨损伤的情况下更好地可视化、颅骨的稳定性最大限度地减少组织运动伪影,以及跨实验动物的可重复性,便于不同实验组之间的比较。恢复动物和进行纵向成像的可能性也可以减少研究中使用的动物数量。值得一提的是,由于已经观察到颅管生长模式的菌株特异性差异和随之而来的成骨作用18,因此在为所需小鼠菌株设计特定成像植入物时,重要的是要考虑到这一点,以实现完美的解剖兼容性并进入所需的成像部位。
另一个值得一提的重要一点是,与其他骨骼相比,颅骨 BM 内的血管和造血特性存在潜在差异,直到最近几年,这个问题还很少被探索。新的研究表明,不同骨骼的局部功能不同,颅骨 BM 在造血、骨骼和血管结构和功能方面与其他骨骼不同 19,20,21,22,23,以及对神经系统病理的反应24。在概括特定的颅骨发现之前,需要进一步探索和考虑这些差异。
成像植入物的设计和构造是该实验设置的关键步骤,从材料的选择开始。生物相容性植入物在生物医学研究中发挥着关键作用,促进了从组织工程和再生医学到药物输送系统和 体内 监测设备的广泛应用。为适用于活体成像的头部植入物选择材料至关重要。理想情况下,材料应表现出优异的生物相容性、适合确保最小柔韧性和整体稳定性的机械性能,以及最后能够在颅骨内整合而不会引起炎症。钛在动物体内具有良好的耐受性,与生物组织接触时不会引起不良反应,并且具有耐腐蚀性,即使暴露于体液或实验溶液中也能确保长期稳定性,因此是理想的选择。此外,它的机械强度使其不易变形和断裂。尽管钛很坚固,但其密度相对较低,因此可以制造出轻便的活体成像设备,从而最大限度地减少实验动物和研究人员的负担。最后,它在制造方面的多功能性允许定制活体成像设备,以满足特定的实验要求,例如尺寸、形状和功能。
在这里,我们使用具有内置有限元分析 (FEA) 功能的参数建模 CAD 软件(特别是 SolidWorks)设计了一个生物相容性头部固定植入物。这种方法能够对植入物的结构和空间要求进行精确、迭代的调整,确保解剖兼容性和机械弹性。FreeCAD 等免费替代品提供类似的建模和仿真功能。设计过程的主要输入包括小鼠颅骨的高分辨率解剖模型、显微镜物镜模型和固定系统模型,它们共同决定了植入物的尺寸和配置。最终输出是一个独立于平台的网格文件,通常为 STL 或 STEP 格式,与大多数 3D 打印软件兼容。
第一步包括使用高分辨率成像技术(例如 体内 显微 CT 扫描)捕捉小鼠头部的详细解剖特征。这种方法提供了最高的解剖细节和准确性,可以捕捉头骨的微观结构。准备好的裸露头骨的 3D 扫描也是一种选择。这种方法使用激光或结构光扫描仪,通常用于准备好的头骨,并提供准确的表面轮廓,但与显微 CT 相比,内部细节较少。否则,可以从开源出版物和数据库25 或 DigiMorph {https://www.digimorph.org/specimens/Mus_musculus/} 下载解剖模型。虽然方便,但这些模型可能缺乏特定于标本的细节,因此通常需要对研究中的特定动物进行调整,例如缩放到实际的 Bregma-Lambda 距离。然后,使用获取的数据创建小鼠头骨的精确 3D 模型,作为植入物设计的基础模板。
为了将植入物固定在颅骨上,设计了粘合结构以覆盖未被观察窗占据的剩余颅骨表面。这种结构必须提供坚固的连接点,同时避免关键的解剖学特征。粘接结构具有多个开口,以确保通过扩散在植入物下粘接剂的最佳聚合。此外,胶结结构的墙体具有较小的拔模角,这使得胶结剂能够牢固地锚固在这些倾斜的墙体上。设计了从主体延伸到固定系统的植入物尾部。该组件对于在观察过程中对齐和稳定植入物至关重要,其设计必须考虑可用空间和鼠标头部的解剖学限制。最后,将燕尾榫机构集成到植入物设计中,以便于植入物与固定系统的连接和分离。此功能增强了植入物在重复观察期间的实用性和可用性。燕尾榫机构提供可重复的固定,便于检索观察到的组织以进行重复成像。
考虑采用颅骨 BM 活体显微镜成像的读者应仔细评估他们的研究目标和实验要求,以确定这种方法是否适合他们的研究。虽然活体显微镜为 体内造血、组织微环境调节和血管动力学提供了无与伦比的见解,但它也带来了一定的技术挑战和局限性。研究人员应准备好通过仔细的实验设计、优化成像参数和利用适当的对照措施来应对这些挑战。此外,研究人员应考虑专用成像设备的可用性、荧光标记技术的专业知识以及用于图像分析的计算资源。总体而言,颅骨 BM 的活体显微镜成像在促进我们对造血和血管生物学的理解方面具有巨大潜力,为了解 BM 微环境中发生的动态过程提供了一个独特的窗口。
披露声明
Jozsua Fodor 是 YMETRY 公司(注册号 888312352)的创始人。其他作者没有利益冲突。
致谢
作者要感谢 IMAG'IC 和科钦研究所动物设施的所有工作人员,感谢他们对显微镜实验和小鼠饲养的支持。Tg(Nes-EGFP)33Enik 和 Tg(Pdgfb-icre/ERT2)1Frut 小鼠是 Bonnet 博士(伦敦弗朗西斯克里克研究所)的一份善意礼物。Tg(Cdh5-cre/ERT2)1Rha 和 B6。Cg-Gt(ROSA)26Sortm9(CAG-tdTomato)Hze/J 小鼠是 Rafii 博士(纽约威尔康奈尔医学院)的一份礼物。所描述的工作得到了 CNRS、INSERM 和巴黎城市大学的支持,并得到了 ATIP-AVENIR、ARC pour la recherche sur le cancer (R19084KS - RSE20008KSA)、Ville de Paris “Emergence” (R20192KK - RPH20192KKA)、Laurette Fugain (R23197KK)、坎塞罗波尔 IDF (RPH23177KKA)、INCA PLBIO (RPH21162KKA)、法兰西基金会 (RAF23152KKA)、Ligue contre le cancer (282273/807251)、Institut du cancer Paris Carpem 的资助, 欧洲血液学协会 (RAK23130KKA) 和欧洲研究委员会 ERC-STG (EEA24092KKA)。IMAG'IC 核心设施由法国国家基础设施生物成像公司(授予 ANR-10-INBS-04)提供支持。Passaro 实验室隶属于“Institut Hors Murs des Sciences Cardiovasculaires”和“巴黎圣路易白血病研究所”。
材料
| Name | Company | Catalog Number | Comments |
| Amira | Thermo ScientificTM | ||
| Anesthesia | Isoflurane 2% to 3% | ||
| Anesthesic mask (animal detection) | Minerve | ||
| Anesthesic unit | Minerve | ||
| B6.Cg-Gt(ROSA)26Sortm9(CAG-tdTomato)Hze | Jackson laboratories | MGI:3809523 | |
| CalvariaVue | Ymetry | https://ymetry.com/site/head-fixation-implants/44-mouse-head-fixation-implant-for-calvaria.html | |
| Dental cement | Kemdent | SUN527 | |
| Dextran 500 kDa-Cy5 | Tebu-bio | DX500-S5-1 | 3 mg/mouse |
| Dextran 70 kDa-TRITC | Sigma | T1162 | 3 mg/mouse |
| Disinfectant | MP-Labo | Dermidine-60ml | |
| Electric razor | Aescular | Isis | |
| Eye gel | Ocry-gel | 10g | |
| Fiji | https://imagej.net/software/fiji/downloads | v 17 May 30 | |
| Fiji plugings to bridge with Imaris | https://imagej.net/software/fiji/downloads | Imaris_Bridge96.jar ; ImarisBridgeUtils.jar | |
| Heating box | Datesand | Thermacage | |
| Heating pad for surgery | Minerve | ||
| Imaging heating pad & rectal probe | F. Haer | ||
| Imaris v9.6.0 | Oxford instruments | ||
| Intrasite gel | Chinoxia | 2390766 | |
| LAS AF Software | Leica | LAS X 3.5.7.23225 | |
| Medication | Buprecare, 0.01 mg/kg | ||
| Objective HCX IRAPO L 25x/0.95 WATER | Leica | 506374 | |
| Saline buffer (PBS 1x) | Sigma | P4417 | Sterilize by autoclave |
| SP8 DIVE FALCON Multiphoton Microscope | Leica | ||
| Stereotoxic mask | Minerve | 1201261 | |
| Sterilizator beads | Sigma | Z742555 | |
| Surgery tools | Moria | 4877A; 2183 | |
| Survival blanket | SECURIMED | 11006 | |
| Swabs / Tissues | Sterilize by autoclave | ||
| Syringe 1 mL 26 G | BD Plastipak | 305501 | |
| Temperature controller | F. Haer | 40-90-5D-02 | |
| Tg(Nes-EGFP)33Enik mice | Jackson laboratories | MGI:5523870 | |
| Tg(Pdgfb-icre/ERT2)1Frut mice | Jackson laboratories | MGI:3793852 | |
| Tg(Cdh5-cre/ERT2)1Rha mice | Jackson laboratories | MGI:3848982 | |
| Ultrasound gel | Parker laboratories | Aquasonic 100 |
参考文献
- Lo Celso, C., et al. Live-animal tracking of individual haematopoietic stem/progenitor cells in their niche. Nature. 457 (7225), 92-96 (2009).
- Pittet, M. J., Weissleder, R. Intravital imaging. Cell. 147 (5), 983-991 (2011).
- Itkin, T., et al. Distinct bone marrow blood vessels differentially regulate haematopoiesis. Nature. 532 (7599), 323-328 (2016).
- Christodoulou, C., et al. Live-animal imaging of native haematopoietic stem and progenitor cells. Nature. 578 (7794), 278-283 (2020).
- Upadhaya, S., et al. Intravital imaging reveals motility of adult hematopoietic stem cells in the bone marrow niche. Cell Stem Cell. 27 (2), 336-345.e4 (2020).
- Hawkins, E. D., et al. T-cell acute leukaemia exhibits dynamic interactions with bone marrow microenvironments. Nature. 538 (7626), 518-522 (2016).
- Duarte, D., et al. Defining the in vivo characteristics of acute myeloid leukemia cells behavior by intravital imaging. Immunol Cell Biol. 97 (2), 229-235 (2019).
- Passaro, D., et al. Increased vascular permeability in the bone marrow microenvironment contributes to disease progression and drug response in acute myeloid leukemia. Cancer Cell. 32 (3), 324-341.e6 (2017).
- Jia, Y., et al. FLT3 inhibitors upregulate CXCR4 and E-selectin ligands via ERK suppression in AML cells and CXCR4/E-selectin inhibition enhances anti-leukemia efficacy of FLT3-targeted therapy in AML. Leukemia. 37 (6), 1379-1383 (2023).
- Pendleton, E. G., Tehrani, K. F., Barrow, R. P., Mortensen, L. J. Second harmonic generation characterization of collagen in whole bone. Biomed Opt Express. 11 (8), 4379-4396 (2020).
- Ventalon, C., Bourdieu, L., Leger, J. F., Fodor, J. Dispositif de liaison d'un animal de laboratoire à au moins un système d'expérimentation, et procédé de fixation d'un tel dispositif. France patent. , (2021).
- Horton, S. J., et al. Acute myeloid leukemia induced by MLL-ENL is cured by oncogene ablation despite acquisition of complex genetic abnormalities. Blood. 113 (20), 4922-4929 (2009).
- Scott, M. K., Akinduro, O., Lo Celso, C. In vivo 4-dimensional tracking of hematopoietic stem and progenitor cells in adult mouse calvarial bone marrow. J Vis Exp. (91), e51683 (2014).
- Jung, Y., et al. Intravital imaging of mouse bone marrow: Hemodynamics and vascular permeability. Methods Mol Biol. 1763, 11-22 (2018).
- Le, V. H., et al. In vivo longitudinal visualization of bone marrow engraftment process in mouse calvaria using two-photon microscopy. Sci Rep. 7, 44097 (2017).
- Spencer, J. A., et al. Direct measurement of local oxygen concentration in the bone marrow of live animals. Nature. 508 (7495), 269-273 (2014).
- Yang, M., Mahanty, A., Jin, C., Wong, A. N. N., Yoo, J. S. Label-free metabolic imaging for sensitive and robust monitoring of anti-CD47 immunotherapy response in triple-negative breast cancer. J Immunother Cancer. 10 (9), e005199 (2022).
- Li, W., et al. Tracking strain-specific morphogenesis and angiogenesis of murine calvaria with large-scale optoacoustic and Ultrasound Microscopy. J Bone Miner Res. 37 (5), 1032-1043 (2022).
- Lassailly, F., Foster, K., Lopez-Onieva, L., Currie, E., Bonnet, D. Multimodal imaging reveals structural and functional heterogeneity in different bone marrow compartments: functional implications on hematopoietic stem cells. Blood. 122 (10), 1730-1740 (2013).
- Rindone, A. N., et al. Quantitative 3D imaging of the cranial microvascular environment at single-cell resolution. Nat Commun. 12 (1), 6219 (2021).
- Mills, W. A., Coburn, M. A., Eyo, U. B. The emergence of the calvarial hematopoietic niche in health and disease. Immunol Rev. 311 (1), 26-38 (2022).
- Bixel, M. G., et al. Angiogenesis is uncoupled from osteogenesis during calvarial bone regeneration. Nat Commun. 15 (1), 4575 (2024).
- Koh, B. I., et al. Adult skull bone marrow is an expanding and resilient haematopoietic reservoir. Nature. 636 (8041), 172-181 (2024).
- Kolabas, Z. I., et al. Distinct molecular profiles of skull bone marrow in health and neurological disorders. Cell. 186 (17), 3706-3725.e9 (2023).
- Rosenhain, S., et al. A preclinical micro-computed tomography database including 3D whole body organ segmentations. Sci Data. 5, 180294 (2018).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。
我们使用 cookie 来增强您在我们网站上的体验。
继续使用我们的网站或单击“继续”,即表示您同意接受我们的 cookie。