Method Article
Caracterización de procesos de reparación del ADN en transitoria y duradera doble cadena DNA roturas por microscopia de la inmunofluorescencia
* Estos autores han contribuido por igual
En este artículo
Resumen
Reparación de roturas del DNA de doble cadena es un proceso dinámico, que requiere no sólo la formación de complejos de reparación en los descansos, pero también su resolución después de la lesión se trata. Aquí, utilizamos microscopia de la inmunofluorescencia para roturas de doble hebra transitorias y duradera como una herramienta para diseccionar este mecanismo de mantenimiento del genoma.
Resumen
La reparación de roturas de doble hebra (distritales) en el ADN es un proceso altamente coordinado, que requiere la formación y resolución de los complejos de multi-proteínas reparación. Este proceso está regulado por una gran variedad de proteínas que promueven la asociación y disociación de las proteínas a estas lesiones. Gracias en gran parte a la capacidad para realizar pantallas funcionales de una vasta biblioteca de proteínas, hay una mayor apreciación de los genes necesarios para la reparación de rotura de ADN de doble cadena. Pantallas a menudo geneticamente o químico inhibidor identifican las proteínas implicadas en procesos de reparación mediante aumento de la toxicidad como un marcador para una proteína que se requiere para la reparación DSB. Aunque útil para identificables nuevas proteínas celulares involucradas en mantener la fidelidad del genoma, análisis funcional requiere la determinación de si la proteína de interés promueve la localización, formación o resolución de los complejos de reparación.
La acumulación de proteínas de reparación puede ser fácilmente detectada como distintos focos nucleares por microscopia de la inmunofluorescencia. Por lo tanto, asociación y disociación de estas proteínas en los sitios de daño del ADN pueden accederse mediante la observación de estos focos nucleares representante a intervalos después de la inducción de roturas del DNA de doble cadena. Este enfoque también puede identificar proteínas de factor de reparación mal localizada, si reparar los defectos no ocurren simultáneamente con incompletos retrasos en la reparación. En este escenario, roturas de DNA de doble cadena larga duración pueden diseñarse expresando una endonucleasa de corte raro (p. ej., SceI) en las células donde el sitio de reconocimiento para la enzima dijo se ha integrado en el genoma celular. La lesión resultante es particularmente difícil de resolver como reparación fiel será reintroducir sitio de reconocimiento de la enzima, lo que provocó otra ronda de escote. Como resultado, se eliminan las diferencias en la cinética de reparación. Si no se forman complejos de reparación, localización ha sido impedida. Este protocolo describe la metodología necesaria para identificar cambios en la cinética de reparación así como localización de la proteína de reparación.
Introducción
Cada día, cada célula en el cuerpo humano es bombardeado con un estimado 10.000 lesiones de ADN1. Esta amenaza existencial nos pone en riesgo de mutaciones, oncogénesis, así como la célula muerte. Para proteger la fidelidad de genoma, células de mamíferos han evolucionado para responder a daños en el ADN con una compleja serie de asociaciones de proteínas y modificaciones. Esta respuesta está organizada en múltiples vías, conocidas colectivamente como la DNA daños respuesta (DDR)2,3. El DDR consiste en la acumulación de proteínas de reparación del ADN en las lesiones del ADN, coordinadas tanto temporal como espacialmente. DDR con frecuencia induce arresto del ciclo celular para evitar la propagación o la intensificación de los daños que pueden ocurrir durante la replicación del ADN dañado2,de4,5. A su vez, también es necesario para la viabilidad celular desactivar la detención del ciclo celular por desvincularlo complejos de reparación después de concluida la reparación.
Entre los distintos tipos de daños en el ADN, distritales son los más perjudiciales. Distritales de reparación, pueden fallar los cambios del cromosoma o eliminaciones a gran escala tales como la pérdida de los brazos del cromosoma entero. La reparación de los distritales se divide en dos vías6,7,8. Recombinación homóloga (HR) requiere una hermana cromosoma para utilizar como una plantilla de ADN y por lo tanto se limita a últimas fases S y G2/M del ciclo celular9,10. No homólogos se terminan uniendo (NHEJ) no tiene estas restricciones, pero puede causar pequeñas canceladuras al reparar distritales11,12.
Reparación de DSB específicamente y el DDR en general son áreas activas de investigación. A pesar de ser organizados en vías convenientemente separadas, hay una gran cantidad de redundancia. De hecho, muchas proteínas (BRCA1, BRCA2 y el EPR complejo por ejemplo) están implicadas en múltiples vías13,14,15,16. La reparación de una lesión por una vía, puede llevar a un daño intermedio que debe ser reparado por otro camino14. El entrelazamiento de estas vías, combinadas con su compleja tarea de reclutar las proteínas adecuadas para el lugar correcto para la cantidad exacta de tiempo necesario, requiere de un proceso regulador multi-con gradas.
Un informe reciente destaca las complejidades de DDR demostrando la formación de complejos de reparación, la resolución y localización de cada uno por separado que pueden deteriorada17. El objetivo general del siguiente protocolo es diseccionar definitivamente la capacidad de las células para reparación de DSB. Usando microscopia de la inmunofluorescencia, acumulación de proteínas de reparación en los sitios de daño puede ser visualizada en los puntos de tiempo representativo después de la inducción de los consejos escolares distritales.
Esta técnica tiene varias ventajas a los enfoques utilizados. Con frecuencia, es investigado en momentos solo e incapaz de representar el proceso dinámico de ensamblaje y disociación de complejos de reparación. Observar la gama completa de la reparación de la activación inicial de la resolución asegura que un retraso en la reparación no es identificado erróneamente como inhibición completa. Por el contrario, asegura que la inducción de una respuesta de reparación que no es capaz de inactivar dicha respuesta no identificado erróneamente como la normal o la activación excesiva.
Formación de complejos proteína retrasada y la localización errónea de proteínas de reparación, sin embargo, no se puedan claramente distinguir con este enfoque. Para determinar si las proteínas de reparación mal localizadas versus demora en su localización, se puede introducir un OSD de "larga duración" a través de la hendidura enzimática del ADN celular. La lesión resultante se corte de nuevo cada vez que se repara, resultando en un enfoque distinto reparación nuclear grande y eliminar la restricción temporal de contratación. Esto puede lograrse mediante la modificación de lo existente con el uso de una endonucleasa de corte raro (p. ej., SceI) para inducir un DSB de larga duración. La longevidad de los consejos escolares distritales permite la visualización de las proteínas de difícil reparación por microscopia de la inmunofluorescencia. La mayor abundancia también podría mejorar la visualización cuando la detección se ve obstaculizada por la limitación en la calidad de anticuerpos, una característica que puede ser útil cuando menos proteínas estudiadas se identifican como teniendo un impacto en la reparación del ADN.
En particular, ofrecemos instrucciones explícitas de una imagen libre procesamiento y análisis de software (por ejemplo, ImageJ). Esto elimina una barrera financiera importante en análisis de imagen, apertura de la interrogación de la reparación del daño de ADN a un público más amplio.
Protocolo
Nota: el presente Protocolo es escrito para U2OS celdas que contengan un-SceI reconocimiento sitio18. Las células no necesitan ser U2OS pero deben contener el sitio SceI. El protocolo puede necesitar ser ajustado (p. ej., número de células sembradas y tiempos de incubación) dependiendo del tipo de células utilizadas.
1. definir la cinética de formación de complejos de reparación DSB
- Cultivar células de U20S-DRGFP en una placa de cultivo de tejidos de 10 cm hasta 85-90% confluente.
- Retire los medios de comunicación e incubar a temperatura ambiente (RT) en 3 mL de EDTA por 2 min.
- Vuelva a colocar EDTA con 1 mL de tripsina e incubar a 37 ° C durante 5 minutos, asegúrese de que las células son separadas de la superficie de la placa por visualización al microscopio. Neutralizar la tripsina con 5 mL de Dulbecco modificado Eagle Medium (DMEM) suplementado con 10% suero bovino fetal (FBS) y 1% de penicilina/estreptomicina.
- Determinar la concentración de células en la suspensión usando un hemocitómetro y el método de exclusión azul de tripano.
- 6 x 103 células/pozo en 200 μL de una placa de 96 pocillos de fondo de cristal de la semilla. Por otra parte, semilla 4 x 106 células en 8 mL de 12 mm cubreobjetos colocan en una placa de 10 cm.
Nota: Cada bien o cubreobjetos representa un momento único, incluyendo el control. - Crecen las células durante la noche a 37 ° C y 5% CO2.
- Inducción de distritales
- Sustituir los medios de comunicación de la fuente de placa de 96 pocillos, 10 cm H2O2 diluida a una concentración de 25 μm con DMEM + 10% FBS e incubar a 37 ° C durante 1 h.
- Lavar las células 3 x 1 x PBS y reemplazar con DMEM fresco suplementados con 10% de SBF y el 1% de penicilina/estreptomicina. Incube las células a 37 ° C durante 0, 1, 4, 8, 24 y 48 h. Asegúrese que las diluciones frescas de H2O2 se utilizan para cada experimento.
Nota: Para evitar matar a las células de la concentración de H2O2 usado aquí, asegúrese de que la dosis de daños es baja bastante que la mayor parte de las células será evitar la apoptosis y la senescencia. Es fundamental para observar el proceso de recuperación. Esto se logra mejor mediante la medición de sensibilidad a una gama de dosis. Realizar un ensayo MTT u otro ensayo de viabilidad celular para determinar la concentración mínima de peróxido de hidrógeno que se puede utilizar.
- Fijación y permeabilizing células
- Lavar las células 3 veces con PBS 1 x. Retirar un cubreobjetos de la placa de 10 cm para cada momento y lugar en un único pozo de una placa de 24 pocillos para la fijación. Utilice la aguja y pinzas para extraer cuidadosamente el cubreobjetos y evitar rayar las células de la superficie del cubreobjetos.
- Fijar las células por incubar durante 15 min con paraformaldehído al 4% (PFA) en señalan momentos después de 1 h de H2O2 exposición y recambio de DMEM fresco (0, 1, 4, 8, 24 y 48 h).
- Lavar los cubreobjetos fija 3 veces con PBS. Permeabilizar las células con detergente no iónico 0,5% diluido en 1 x PBS. Incubar por 15 min a temperatura ambiente, antes de lavar 3 veces con PBS.
Nota: Después de permeabilización, almacenamiento de cubreobjetos es posible en PBS a 4 ° C durante por lo menos una semana.
- Reparación compleja visualización
- Bloque de la placa de 96 pocillos/cubreobjetos en una solución de 3% albúmina de suero bovino (BSA) y detergente no iónico 0.1% diluido en PBS 1 x (solución de BSA 3%) durante 1 h a TA.
- Incubar con anticuerpos primarios dirigidos a la proteína de reparación del interés, diluido según las especificaciones del fabricante en una solución de BSA 3% durante 1 h a temperatura ambiente, antes de lavar 3 veces con PBS 1 x.
- Incubar la placa de 96 pocillos/cubreobjetos con anticuerpo secundario diluido según protocolo del fabricante de la solución de BSA 3%. Confirman que los fluoróforos secundarias no tienen excitación superpuesta o espectros de emisión.
- Después de lavar las células con PBS 3 veces añadir DAPI, diluido según las especificaciones del fabricante de 1 x PBS e incubar a temperatura ambiente durante 5 minutos.
- Coloque el cubreobjetos sobre el portaobjetos con una gota de agente de montaje, asegurándose de que las caras laterales de la celda hacia abajo. Presione con cuidado el cubreobjetos y empaparse de cualquier agente de montaje exceso con una toallita.
- Selle el cubreobjetos con esmalte transparente de secado rápido y asegurar un sellado completo del cubreobjetos con la diapositiva.
- Tomar las imágenes de microscopio confocal, centrándose en el canal DAPI, antes de otros canales (con señales de gene de la reparación del ADN de interés) para evitar la introducción de sesgos en el experimento.
- Alternativamente, se pueden adquirir imágenes Z-secciones de la región deseada y la imagen de condensación en un solo plano para visualizar todos los focos detectables.
Nota: Si la resolución del OSD o la proteína de interés no se observa en los puntos de tiempo elegidos, determinar cuándo distritales resolución seleccionando inicialmente una amplia gama de puntos de tiempo (0-48 h post inducción OSD). Los puntos de tiempo de interés variará según el método de inducción de OSD y la proteína de reparación del interés.
- Uso libre de software ImageJ para definir los núcleos de las imágenes de microscopía de inmunofluorescencia.
Nota: Para abrir imágenes de microscopio, debe instalarse el plugin de Bio-formatos en ImageJ.- Abra las imágenes confocales (.czi o .tif) arrastrando el archivo de imagen en la ventana de ImageJ, o seleccionando "Bio-formatos importador" de la barra "Plugin" en ImageJ.
Nota: Aparecerá la ventana de "Opciones de importación de Bio-formatos". - Seleccione "Dividir canales" para dividir la imagen en DAPI constitutiva e imágenes fluorescentes dentro de la ventana de "Opciones de importación de Bio-formatos".
- Seleccione "Colorized" en "Opciones de Color" en el menú desplegable. Seleccione "OK" para abrir la DAPI e histona H2AX (H2AX) imágenes como separar de windows y escoge la imagen DAPI.
- Selecciona la barra de herramientas "Imagen" y "Umbral de ajuste" para seleccionar los núcleos.
Nota: Aparecerá una ventana de "Umbral". - Ajustar el umbral de la imagen deslizando la barra inferior de la ventana de "Umbral" completamente a la derecha. Ajuste la barra superior hasta que los núcleos aparecen completamente rojos con distintos esquemas y el negro de fondo. NO seleccione aplicar. Cierre la ventana "Ajustar umbral".
- Selecciona la barra de herramientas de "Analizar" y "Analizar partículas". Ver una ventana de "Analizar las partículas" que aparecen en la pantalla.
- De entrada el tamaño mínimo de un núcleo.
Nota: Las partículas por debajo del tamaño introducido no contará como núcleos. - En el menú desplegable de "Mostrar", seleccione "Contornos". Seleccione la opción "Agregar al administrador". Haga clic en "Aceptar" para abrir una ventana de "Administrador de ROI".
Nota: Esquemas de núcleos aparecerá en una ventana independiente. - Asignar a números a los núcleos que pueden seleccionarse desde la ventana de "Administrador de ROI".
- Abra las imágenes confocales (.czi o .tif) arrastrando el archivo de imagen en la ventana de ImageJ, o seleccionando "Bio-formatos importador" de la barra "Plugin" en ImageJ.
- Utilizar ImageJ para cuantificar H2AX focos en imágenes de microscopia de la inmunofluorescencia.
- Seleccione la ventana de focos "H2AX" (teñida con fluorescente conjugado anticuerpo secundario). Luego, seleccione la barra de herramientas de "Proceso" y elegir "Buscar Maxima" para encontrar los focos dentro de los núcleos.
Nota: Se abrirá una ventana de "Buscar la Maxima". - Elige la opción "Solo puntos" en el menú desplegable de "Tipo de salida" y seleccionar "Selección de puntos de vista previa" para visualizar los maxima/focos. De entrada un valor de "Tolerancia de ruido" dependiendo de la intensidad de la fluorescencia de la imagen (figura 1). Compruebe el aspecto de una ventana blanca con puntos negros pequeños.
Nota: Estos puntos representan la focos/maxima H2AX que han sido detectada. El valor de tolerancia de ruido debe mantenerse constante para todas las imágenes analizadas. Una tolerancia baja/ruido representan también muchos/pocos maxima (focos) dentro del núcleo y no será una representación exacta de los focos dentro del núcleo. - Seleccionar los núcleos para que H2AX focos se deben cuantificar para desde la ventana de "Administrador de ROI". Seleccione todos los núcleos marcando la opción "Mostrar todos".
- Seleccione "Medida" para medir el número de máximos/focos dentro de los núcleos seleccionados.
Nota: El número de focos de maxima aparece como múltiplos de 255, es decir, un máximo tiene una lectura de "RawIntDen" (densidad integrado) de 255. - Copiar los valores de "RawIntDen" y pegarlos en software para análisis de datos.
- Seleccione la ventana de focos "H2AX" (teñida con fluorescente conjugado anticuerpo secundario). Luego, seleccione la barra de herramientas de "Proceso" y elegir "Buscar Maxima" para encontrar los focos dentro de los núcleos.
2. Análisis de localización a una rotura de ADN de doble cadena de larga duración
- Células de semilla en 96 pocillos platos/cubreobjetos como se describe en las secciones 1.1 y 1.2.
-
Inducción de roturas del DNA de doble cadena
- Transfectar las células con el vector de expresión-SceI utilizando un reactivo de transfección basada en lípidos según las especificaciones del fabricante. Espere 24-72 h después de transfección para maximizar la expresión de la endonucleasa. Determinar si la transfección tuvo éxito localizando las células con un enfoque singular gran nuclear γ-H2AX.
-
Adquisición de imágenes
- Fijar, permeabilizar, bloquear y lave la placa de 96 pocillos/cubreobjetos, como se describe en la sección 1.8.
- Co incubar las células con un anticuerpo contra γ-H2AX y la proteína de reparación del interés (aquí, un anticuerpo primario contra RAD51 fue utilizado) diluido según las especificaciones del fabricante en la solución de BSA 3%.
- Lave la placa de 96 pocillos/cubreobjetos 3 veces con PBS 1 x. Incubar la placa de 96 pocillos/cubreobjetos con anticuerpo secundario diluido según protocolo del fabricante de la solución de BSA 3%. Confirman que los fluoróforos secundarias no tienen excitación superpuesta o espectros de emisión.
- Identificar las células que tienen un foco grande γ-H2AX nuclear. Sólo tomar fotografías de estas células para evitar el sesgo.
-
Análisis de larga duración-SceI inducida focos
- Analizar los focos de larga duración manualmente, contando el número de células que tienen los focos grandes de γ-H2AX y co localizados focos de la proteína de reparación del interés.
Resultados
La figura 1 muestra la selección de la discriminación de ruido correcto para la cuantificación de maxima/focos con ImageJ. Las imágenes combinadas de DAPI y la proteína de reparación del interés están en el panel izquierdo. Figura 1A muestra una discriminación de ruido de 90 y marca el número correcto de focos. Los núcleos en el borde (representado con una flecha de color rosa) y focos fuera de los núcleos (representados con una flecha amarilla) no cuentan en la cuantificación. Figura 1B representa una tolerancia de bajo nivel de ruido de 50. Numerosos máximos son identificados fuera del núcleo y dentro del núcleo. Figura 1 muestra una tolerancia de ruido alta de 220. Se detectó un solo foco (una flecha blanca). Para ofrecer al lector un sentido de "positivo" y resultados "negativos", también hemos recopilado resultados representativos en la figura 2. Específicamente, la figura 2A representa colocalización entre un foco de γ-H2AX (verde) y una proteína de reparación de interés (rojo). Asimismo, en la parte superior izquierda de este panel se muestra una célula untransfected y un celular con un enfoque no-nucleares (false) γ-H2AX se muestra en la parte superior derecha. Por favor nota que los micronúcleos asocian con maquinaria de daño del ADN son una fuente probable de estos focos no nuclear19. Figura 2B proporciona un aumento mayor de las dos células de la derecha de la figura 2A para proporcionar claridad en cómo se determinaron a ser interiores y exteriores al núcleo, respectivamente. En la figura 2se muestra un ejemplo de un enfoque de γ-H2AX nuclear sin la localización de una proteína de reparación. Un esquema de este protocolo se proporciona en la figura 3 que resume esta aproximación a la caracterización de reparación del ADN.

Figura 1 : Representante análisis de cinética de reparación de doble cadena DNA rotura. Imágenes representativas de γ-H2AX cuantificación de focos usando el software ImageJ (con formatos de Bio plugin). Los paneles de la izquierda son imágenes compuestas de núcleos (40 aumentos), teñidos azul DAPI y verde para γ-H2AX. Paneles central muestran referencia núcleos no analizados con γ-H2AX focos/maxima. (A) el panel de la derecha muestra la detección del maxima/focos con una tolerancia de ruido de 90 y es representante de la estimación real del número de focos dentro del núcleo. (B) el derecho panel muestra la cuantificación máxima con una tolerancia de poco ruido (50). Focos de maxima puede ser detectados fuera del núcleo (flecha amarilla), mientras que la mayoría de los máximos situados en el núcleo son las señales de fondo no específica. (C) el derecho panel muestra cuantificación maxima tolerancia de ruido alto (220). Sólo un máximo/foco (flecha blanca) se detecta dentro del núcleo y no es representativo de los focos dentro del núcleo. Haga clic aquí para ver una versión más grande de esta figura.
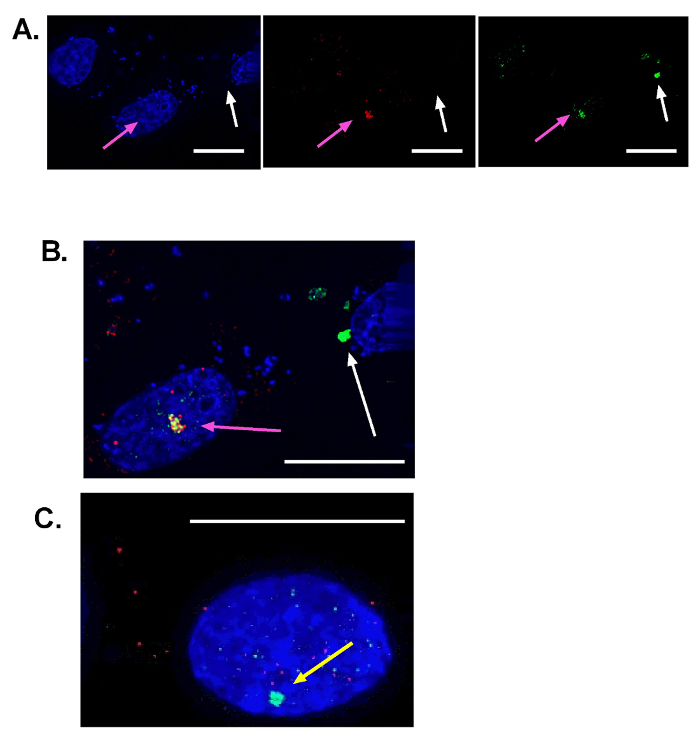
Figura 2 : Representante SceI inducida por roturas de DNA de doble cadena. Imágenes representativas de los núcleos de las células cultivadas en cubreobjetos (40 aumentos). Los núcleos se tiñen de azul. Una proteína de reparación (RAD51) se tiñe de rojo y γ-H2AX se tiñe de verde. (A) los tres paneles mostrar los mismo tres células. En la izquierda panel de mayoría, los núcleos son visibles. En el panel central, se muestra coloración para RAD51. En el panel derecho, γ-H2AX coloración aparece. El núcleo en la esquina superior derecha no muestra que ningún signo de SceI inducida distritales. El núcleo en el centro (flecha rosa) representa un verdadero evento positivo ya que tiene un foco nuclear grande γ-H2AX y un foco nuclear de RAD51 Co localizado. El núcleo en el lado derecho tiene un enfoque falso grande γ-H2AX como extranuclear. Focos fuera del núcleo no deben utilizarse para el análisis de colocalización. (B) se trata de un aumento mayor de la imagen fusionada en parte A (centro y derecha mayoría de las células). El foco amarillo nuclear grande que indica la superposición de señal RAD51 y H2AX es denotado por la flecha rosa. (C) A imagen representativa de un foco nuclear de γ-H2AX sin co la coloración de la proteína de reparación de ADN de interés. A diferencia del enfoque que se denota con una flecha blanca en (A) y (B), la flecha amarilla muestra resultados típicos cuando las proteínas de reparación se impiden localizar a los sitios de daño. Barra de escala = 15 μm. haga clic aquí para ver una versión más grande de esta figura.

Figura 3 : Representación visual del protocolo para el análisis de reparación de rotura de doble cadena ADN. Pasos (1) representación del protocolo 1.1 para la siembra de células sobre cubreobjetos vidrio inferior placas. (2) ilustración de la inducción de doble cadena se rompe por tratamiento con peróxido de hidrógeno (derecha) o inducción de larga duración double-strand breaks por transferencia 3 μg de SceI. (3) representación de los pasos del protocolo 1.4 a 1.5.3 para visualización de roturas de doble cadena. (4a) imagen de microscopía confocal de células teñidas con DAPI (azul) y γ-H2AX focos (verde). (4b) imagen de singular γ-H2AX grandes focos (verde) otra vez núcleos tinción DAPI (azul). (5a) representación del análisis de focos de γ-H2AX con ImageJ (1.10.1–1.11.5 pasos de protocolo). (5b) Descripción de la cuenta manual de grandes focos de γ-H2AX duradero (paso de protocolo 2.4). Haga clic aquí para ver una versión más grande de esta figura.
Discusión
El análisis de ADN reparación de daños en general y la reparación de roturas del DNA de doble hebra es específicamente un área activa de investigación porque sus consecuencias abarcan tumorigenesis biología básica6,20. Datos de este manuscrito un enfoque que disecciona con precisión la contribución de proteínas RAD51 y H2AX γ a la resolución de los consejos escolares distritales a través de horas esperando, este método puede utilizarse para aclarar funciones adicionales de proteínas de reparación a los consejos escolares distritales14. La comparación de la cinética de reparación y la contratación de maquinaria de reparación-SceI inducida distritales entre las células control y las células con la proteína de interés noqueado, definiría la contribución de esa proteína a la selección, localización, y resolución de los factores de reparación para distritales.
Visualizar reparación cinética tiene varias ventajas sobre los métodos utilizados. Con frecuencia, reparación es investigada en puntos del tiempo solo, que es incapaz de representar el proceso dinámico de ensamblaje y disociación de complejos de reparación. Observar toda la gama de reparación de activación inicial a máxima resolución asegura que un retraso en la reparación no es identificado erróneamente como inhibición completa. Por el contrario, asegura la inducción de una respuesta de reparación sin la capacidad de inactivar que respuesta no es identificado erróneamente como activación normal o excesiva. Estos son en gran parte cambios en la aplicación de un enfoque establecido, pero el uso de una endonucleasa de corte raro para inducir un DSB de larga duración es una novedosa herramienta. Permite al investigador a determinar claramente si la localización de una proteína de reparación se inhibe en lugar de su reclutamiento se retrasó un período corto de tiempo, más allá de la conclusión del experimento. SceI inducida puede OSD persiste durante días y teóricamente como la enzima se expresa (la célula permanezcan viable). También se pueden observar la cinética de reparación DSB vía proyección de imagen de células vivas. Sin embargo, aunque en proyección de imagen de célula permite la detección de los eventos de reparación en tiempo real, su aplicación se ve obstaculizada por el requisito de que las proteínas marcado con un fluoróforo como GFP. Tal manipulación genética tiene el potencial de alterar la función de la proteína y representa una barrera técnica adicional.
Análisis de la imagen mediante software ImageJ pueden resultar engorroso para maestro. Las instrucciones paso a paso de reparación foco reconocimiento y cuantificación utilizando este software se proporcionan con la esperanza de eliminar este obstáculo para otros grupos. Esto podría llevar a la mayor utilización de lo software de gran alcance y eliminar el costo de los programas más caros en un momento de contracción de grupos de apoyo de subsidio federal.
En principio, la duración de estos distritales debe permitir la visualización de las proteínas de difícil reparación por microscopia de la inmunofluorescencia. Este podría ser el caso para proteínas que son difíciles de ver porque no acumulan en un alto suficiente concentración para ser detectado, por ejemplo, la persistencia de los resultados de OSD-SceI inducida en una mayor que la típica acumulación de proteínas de reparación en la lesión. La mayor abundancia también podría mejorar la visualización cuando la detección se ve obstaculizada por una limitación en la calidad de anticuerpos; una característica que podría ser particularmente útil cuando menos proteínas estudiadas se identifican como teniendo un impacto en el ADN de la reparación. Hasta la fecha, este enfoque ha sido verificado por 4 proteínas, es decir, RAD51 RPA70, BRCA1 y BRCA2 de reparación del ADN (datos no mostrados, figura 2y referencia17)
Por último, hay modificaciones que podrían introducirse en el presente Protocolo para ampliar los tipos de daños en el ADN que puede ser interrogado así como los ambientes celulares. La modificación más evidente sería cambiar la fuente o el tipo de daño del ADN inducido. La prueba del principio para el uso de UVB, radiación ionizante o radiomimetics (phleomycin, bleomicina y etopósido) para estudiar la cinética de reparación del ADN ya ha sido publicado18,19,23,24 , 25. con ligera modificación, este enfoque puede dilucidar respuesta de proteínas de reparación por NHEJ a distritales. alternativamente, Britton et al. describen una técnica de extracción previa que podría ser utilizado26. Además, son homing endonucleasas (-HmuI o BasI) que tienen sitios de reconocimiento extenso, similares a-SceI27de mellar. Distinguiéndolos de SceI, estas enzimas causan rupturas monocatenarias de DNA (SSBs). Introduciendo el sitio de reconocimiento para estas enzimas en una línea celular permitiría el análisis de la reparación SSB que es paralelo al protocolo descrito en este manuscrito para interrogar duradero distritales. en particular, el proceso de integración el reconocimiento SceI sitio y verificación de la introducción del sitio pueden ser laboriosos. Afortunadamente los avances en tecnología (tales como sistemas de verduras/Cas9) de edición genómica han facilitado esta carga28. También hay un campo de investigación activo que ha tenido éxito en ingeniería endonucleases autoguiados hacia el blanco en una genoma ubicación deseada, que sugiere que en el futuro, estos avances de la ingeniería y el uso de CRISPR/Cas9 puede llevar esta clonación paso a ser prescindible28,29.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos a Joel Sanneman y Dr. Philine Wangemann de la base de la microscopia Confocal, financiado por el Kansas estado Universidad Universidad de medicina veterinaria, por su apoyo de los esfuerzos para desarrollar esta técnica. pCBASceI fue un regalo de Maria Jasin (plásmido Addgene # 26477) 30. Las células U2OS DR-GFP eran un regalo bueno Maria Jasin18.
Materiales
| Name | Company | Catalog Number | Comments |
| 12mm Coverslips | VWR | 89015-725 | |
| 16% Paraformaldehyde (PFA) | ThermoFisher Scientific | 28908 | |
| 24-well plate | VWR | 82050-892 | |
| 96-well glass bottom plate | Cellvis | P96-1.5H-N | |
| Anti-H2AX Alexa488 | EMD Millipore | 05-636-AF488 | |
| Anti-Rad51 (D4B10) | Cell Signaling Technology | 8875S | |
| Bio-formats plugin for ImageJ | National Institute of Health (NIH) | https://imagej.nih.gov/ij/plugins/index.html | |
| Bovine Serum Albumin (BSA) | VWR | 97061-416 | |
| DAPI | ThermoFisher Scientific | D1306 | |
| DMEM, High Glucose | ThermoFisher Scientific | 12100046 | |
| EDTA | Invitrogen | 15576-028 | |
| Fetal Bovine Serum (FBS) | VWR | 89510-194 | |
| Goat Anti-Rabbit IgG Alexa594 | ThermoFisher Scientific | A-11012 | |
| Hydrogen Peroxide | sigma-Aldrich | 216763-100ML | |
| ImageJ Software | National Institute of Health (NIH) | https://imagej.nih.gov/ij/ | |
| I-SceI Expression Vector | Addgene | 26477 | |
| Nail Polish- Insta Dri | Sally and Hansen | Clearly Quick (103) | |
| Phosphate Buffered Saline (PBS) | Bio Basic | PD8117 | |
| ProLong Gold Antifade Reagent | Life Technologies | P36930 | |
| Triton X-100 | Sigma-Aldrich | X100-100ML | |
| Trypsin-EDTA | Sigma-Aldrich | T4049-500ML | |
| TurboFect Transfection Reagent | ThermoFisher Scientific | R0531 | |
| Tween-20 | Fisher Scientific | BP337-500 |
Referencias
- Lindahl, T. Instability and decay of the primary structure of DNA. Nature. 362 (6422), 709-715 (1993).
- Branzei, D., Foiani, M. Regulation of DNA repair throughout the cell cycle. Nat Rev Mol Cell Biol. 9 (4), 297-308 (2008).
- Ciccia, A., Elledge, S. J. The DNA damage response: making it safe to play with knives. Mol Cell. 40 (2), 179-204 (2010).
- Auclair, Y., Rouget, R., Drobetsky, E. A. ATR kinase as master regulator of nucleotide excision repair during S phase of the cell cycle. Cell Cycle. 8 (12), 1865-1871 (2009).
- Awasthi, P., Foiani, M., Kumar, A. ATM and ATR signaling at a glance. J Cell Sci. 128 (23), 4255-4262 (2015).
- Ceccaldi, R., Rondinelli, B., D'Andrea, A. D. Repair Pathway Choices and Consequences at the Double-Strand Break. Trends Cell Biol. 26 (1), 52-64 (2016).
- Chapman, J. R., Taylor, M. R. G., Boulton, S. J. Playing the End Game: DNA Double-Strand Break Repair Pathway Choice. Mol Cell. 47 (4), 497-510 (2012).
- Daley, J. M., Sung, P. 53BP1, BRCA1, and the Choice between Recombination and End Joining at DNA Double-Strand Breaks. Mol Cell Biol. 34 (8), 1380-1388 (2014).
- Hartlerode, A., Odate, S., Shim, I., Brown, J., Scully, R. Cell Cycle-Dependent Induction of Homologous Recombination by a Tightly Regulated I-SceI Fusion Protein. PLOS ONE. 6 (3), e16501 (2011).
- Brandsma, I., Gent, D. C. Pathway choice in DNA double strand break repair: observations of a balancing act. Genome Integr. 3, 9 (2012).
- Davis, A. J., Chen, D. J. DNA double strand break repair via non-homologous end-joining. Transl Cancer Res. 2 (3), 130-143 (2013).
- Reid, D. A., et al. Organization and dynamics of the nonhomologous end-joining machinery during DNA double-strand break repair. Proc Natl Acad Sci. 112 (20), E2575-E2584 (2015).
- Chen, J. J., Silver, D., Cantor, S., Livingston, D. M., Scully, R. BRCA1, BRCA2, and Rad51 Operate in a Common DNA Damage Response Pathway. Cancer Res. 59 (7 Supplement), 1752s-1756s (1999).
- D'Andrea, A. D. BRCA1: A Missing Link in the Fanconi Anemia/BRCA Pathway. Cancer Discov. 3 (4), 376-378 (2013).
- Gudmundsdottir, K., Ashworth, A. The roles of BRCA1 and BRCA2 and associated proteins in the maintenance of genomic stability. Oncogene. 25 (43), 5864-5874 (2006).
- Liu, S., et al. Distinct roles for DNA-PK, ATM and ATR in RPA phosphorylation and checkpoint activation in response to replication stress. Nucleic Acids Res. 40 (21), 10780-10794 (2012).
- Wallace, N. A., Khanal, S., Robinson, K. L., Wendel, S. O., Messer, J. J., Galloway, D. A. High Risk Alpha Papillomavirus Oncogenes Impair the Homologous Recombination Pathway. J Virol. , (2017).
- Pierce, A. J., Johnson, R. D., Thompson, L. H., Jasin, M. XRCC3 promotes homology-directed repair of DNA damage in mammalian cells. Genes Dev. 13 (20), 2633-2638 (1999).
- Luzhna, L., Kathiria, P., Kovalchuk, O. Micronuclei in genotoxicity assessment: from genetics to epigenetics and beyond. Front Genet. 4, (2013).
- Jasin, M. Homologous repair of DNA damage and tumorigenesis: the BRCA connection. Oncogene. 21 (58), 8981-8993 (2002).
- Wallace, N. A., Robinson, K., Howie, H. L., Galloway, D. A. β-HPV 5 and 8 E6 Disrupt Homology Dependent Double Strand Break Repair by Attenuating BRCA1 and BRCA2 Expression and Foci Formation. PLOS Pathog. 11 (3), e1004687 (2015).
- Wallace, N. A., Robinson, K., Howie, H. L., Galloway, D. A. HPV 5 and 8 E6 Abrogate ATR Activity Resulting in Increased Persistence of UVB Induced DNA Damage. PLoS Pathog. 8 (7), (2012).
- Mah, L. J., Vasireddy, R. S., Tang, M. M., Georgiadis, G. T., El-Osta, A., Karagiannis, T. C. Quantification of γH2AX Foci in Response to Ionising Radiation. J Vis Exp. (38), e1957 (2010).
- Popp, H. D., Brendel, S., Hofmann, W. K., Fabarius, A. Immunofluorescence Microscopy of γH2AX and 53BP1 for Analyzing the Formation and Repair of DNA Double-strand Breaks. J Vis Exp. (129), e56617 (2017).
- Burma, S., Chen, B. P., Murphy, M., Kurimasa, A., Chen, D. J. ATM Phosphorylates Histone H2AX in Response to DNA Double-strand Breaks. J Biol Chem. 276 (45), 42462-42467 (2001).
- Britton, S., Coates, J., Jackson, S. P. A new method for high-resolution imaging of Ku foci to decipher mechanisms of DNA double-strand break repair. J Cell Biol. 202 (3), 579-595 (2013).
- Landthaler, M., Shen, B. W., Stoddard, B. L., Shub, D. A. I-BasI and I-HmuI: two phage intron-encoded endonucleases with homologous DNA recognition sequences but distinct DNA specificities. J Mol Biol. 358 (4), 1137-1151 (2006).
- Lackner, D. H., et al. A generic strategy for CRISPR-Cas9-mediated gene tagging. Nat Commun. 6, 10237 (2015).
- Chan, S. H., Stoddard, B. L., Xu, S. Y. Natural and engineered nicking endonucleases--from cleavage mechanism to engineering of strand-specificity. Nucleic Acids Res. 39 (1), 1-18 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados