Method Article
Un enfoque basado en espectrometría de Tandem Liquid Chromatography-Mass para el análisis de metabolitos de
En este artículo
Resumen
Aquí se describe un protocolo para la extracción de metabolitos a partir de Staphylococcus aureus y su posterior análisis por cromatografía de líquidos y espectrometría de masas.
Resumen
En un esfuerzo para frustrar los patógenos bacterianos, anfitriones a menudo limitan la disponibilidad de nutrientes en el sitio de la infección. Esta limitación puede alterar las abundancias de metabolitos clave a los que factores reguladores responden, ajustando el metabolismo celular. En los últimos años, un número de proteínas y ARN han surgido como importantes reguladores de la expresión del gen de virulencia. Por ejemplo, la proteína CodY responde a niveles de aminoácidos de cadena ramificada y GTP y es ampliamente conservada en bajo de G + C bacterias Gram-positivas. Como un regulador global en Staphylococcus aureus, Cody controla la expresión de la virulencia y de genes metabólicos decenas. Se postula que el S. aureus utiliza Cody, en parte, para alterar su estado metabólico en un esfuerzo para adaptarse a las condiciones de nutrientes limitantes potencialmente encontradas en el entorno de acogida. Este manuscrito describe un método para la extracción y el análisis de metabolitos a partir de S. aureus utilizando cromatografía líquida acoplada con espectrometría de masastrometry, un protocolo que fue desarrollado para probar esta hipótesis. El método también destaca las mejores prácticas que garanticen el rigor y la reproducibilidad, tales como mantener el estado de equilibrio biológico y aireación constante y sin el uso de cultivos continuos quimiostato. Con relación a la USA200 susceptibles a la meticilina de S. aureus aislar UAMS-1 cepa parental, la isogénica mutante Cody exhibió incrementos significativos en aminoácidos derivados de aspartato (por ejemplo, treonina e isoleucina) y disminuye en sus precursores (por ejemplo, aspartato y O -acetilhomoserina ). Estos hallazgos se correlacionan bien con los datos de la transcripción obtenidos con el análisis de RNA-seq: genes en estas vías fueron reguladas entre 10 y 800 veces en el mutante nulo Cody. El acoplamiento de los análisis globales del transcriptoma y el metaboloma puede revelar cómo las bacterias alteran su metabolismo cuando se enfrentan a estrés ambiental o nutricional, proporcionando una visión potencial en los Physcambios IOLÓGICA asociados con el agotamiento de nutrientes experimentaron durante la infección. Tales descubrimientos pueden allanar el camino para el desarrollo de nuevos agentes antiinfecciosos y la terapéutica.
Introducción
Los patógenos bacterianos deben enfrentarse a muchos desafíos en el entorno de acogida. Además de ataque directo por las células inmunes, el anfitrión también secuestra nutrientes esenciales para la supervivencia y replicación bacteriana, generando inmunidad nutricional 1, 2. Para sobrevivir a estos ambientes hostiles, los patógenos bacterianos se despliegan factores de virulencia. Algunos de estos factores permiten que las bacterias para evadir la respuesta inmune; Otros factores incluyen las enzimas digestivas, tales como hialuronidasa, termonucleasa, y lipasa, que pueden permitir a las bacterias para reponer los nutrientes que faltan por el consumo de constituyentes derivadas de tejido 3, 4, 5 secretadas. De hecho, las bacterias han evolucionado los sistemas de regulación que atan el estado fisiológico de la célula para la producción de factores de virulencia 6, 7, hasta class = "xref"> 8, 9, 10.
Un cuerpo creciente de evidencia apunta a Cody como un regulador crítico que une el metabolismo y la virulencia. Aunque primero descubierto en Bacillus subtilis como un represor del gen dipéptido permeasa (dpp) 11, Cody ahora se sabe que se produce en casi todas las bacterias G + C Gram-positivos bajos 12, 13 y regula docenas de genes implicados en carbono y nitrógeno metabolismo 14, 15, 16, 17, 18, 19. En las especies patógenas, Cody también controla la expresión de algunos de los genes de virulencia más importantes 20, 21,. ef "> 22, 23, 24, 25, 26, 27 CodY se activa como una proteína de unión a ADN por dos clases de ligandos: aminoácidos de cadena ramificada (BCAA; isoleucina, leucina, y valina [ILV]) y GTP . Cuando estos nutrientes son abundantes, Cody reprime (o en algunos casos, estimula) la transcripción. Como estos nutrientes se vuelven limitado, se reduce progresivamente la actividad Cody, que resulta en una respuesta transcripcional graduada que rutas de re precursores a través de diversas vías metabólicas conectados a metabolismo central 28, 29, 30.
Cromatografía líquida de Tandem acoplada a espectrometría de masas (LC-MS) es una técnica poderosa que puede identificar con precisión y cuantificar de molécula pequeña metabolitos intracelulares 31. Cuando se combina con transcriptasaanálisis riptome (por ejemplo, RNA-Seq), este flujo de trabajo analítico puede dar una idea de los cambios fisiológicos que se producen en respuesta al estrés ambiental o nutricional. A continuación, presentamos un método para la extracción de metabolitos a partir de células de Staphylococcus aureus y el análisis posterior mediante LC-MS. Este enfoque ha sido utilizado para demostrar los efectos pleiotrópicos de CodY en S. aureus fisiología.
Protocolo
1. Preparación de tampón Soluciones
- Prepare la solución salina tamponada con fosfato (PBS; pH 7,4) mediante la dilución de una solución madre de 10x PBS a una concentración final de 1x con ultrapura (destilada y desionizada) agua.
- Preparar la solución de temple mediante la combinación de 2 ml de acetonitrilo, 2 ml de metanol, 1 ml de ultrapura H 2 O, y 19! L (0,1 mM concentración final) de ácido fórmico.
- Prepare LC-MS disolvente A mediante la adición de ácido fórmico (0,2% [v / v] de concentración final) a agua ultrapura.
- Preparar LC-MS disolvente B mediante la adición de ácido fórmico (0,2% [v / v] de concentración final) a acetonitrilo.
NOTA: Todas las soluciones deben prepararse usando los reactivos de la más alta pureza disponible (generalmente de alto rendimiento de grado de cromatografía líquida). Las soluciones deben prepararse fresco antes de cada experimento y se almacenan en hielo antes de su uso.
2. Establecimiento de Crecimiento S. aureus en estado estacionario
- Nodriza de S. aureus cepas de interés para el aislamiento en agar tríptico de soja (TSA) de un stock de glicerol congelado. Incubar a 37 ° C durante 16-24 h.
- Inocular 4 ml de caldo de soja tríptico (TSB) u otro medio adecuado en tubos de incubación de vidrio estériles con colonias individuales de cada cepa. Incubar inclinada (~ 70 ° de ángulo) con rotación a 60 revoluciones por min (rpm) a 37 ° C durante 16-20 h.
NOTA: cultivos de una noche son propensos a gradientes de oxígeno cuando se usan métodos estándar, incluyendo los descritos en el paso 2.2, que afectan a la fisiología celular. Por lo tanto, emplear una estrategia de back-dilución múltiple para asegurar el estado de equilibrio biológico (véanse los pasos 2.4-3.2, a continuación). - Use un espectrofotómetro para medir la densidad óptica de los cultivos de la etapa 2.2 a 600 nm (OD 600). Usar medio estéril como una referencia óptica (en blanco). Diluir estas células a un OD 600 de 0,05 en 50 ml de medio TSB estéril (previamente calentada a 37 ° C) en separada, matraces de 250 mL DeLong.
- Incubate los cultivos a 37 ° C en un baño de agua con agitación a 280 rpm.
- Cada 30 min, tomar OD 600 mediciones; a medida que aumentan las densidades ópticas, puede ser necesario diluir los cultivos con TSB para que permanezcan dentro del rango de absorbancia lineal del espectrofotómetro.
- Cuando los cultivos de la etapa 2.5 a alcanzar un OD 600 de ~ 0,8-1,0, subcultivo ellos en 50 ml de 37 ° C TSB a un OD 600 de 0,01-0,05 y repita los pasos 2.4 y 2.5.
3. Configuración de la muestra Colección
- Preparar un lecho de hielo seco triturado en un recipiente apropiado (por ejemplo, el plato de cristal, cubo de hielo, o más fría).
- A medida que las densidades ópticas de los cultivos se aproximan al punto de cosecha se desea, añadir 1 ml de solución a un 35 mm sin tratar placa de Petri y pre-enfriar en hielo seco temple para ≥5 min.
NOTA: El "punto de cosecha deseada" variará en función de los objetivos experimentales. Por ejemplo, si uno fuera a examinar metabolites durante el crecimiento aeróbico, indicadores clave de este estado incluyen la excreción de etilo y re-asimilación del acetato durante la fase de crecimiento post-exponencial 32, 33. En general, este punto debe estar dentro de una etapa de crecimiento específico (por ejemplo, fase exponencial). Los específicos OD 600 valores asociados con esta fase pueden variar entre las diferentes cepas bacterianas y medios de cultivo. - Coloque una frita de filtro de acero inoxidable (enfriado previamente a -20 ° C) en un tapón de goma y colocarlo encima de un frasco de vacío unido a un vacío de la casa o bomba de vacío.
- Aplicar el vacío y colocar una membrana de éster de celulosa mixto (0,22 tamaño micras de poro) en la parte superior.
NOTA: Es crítico utilizar un filtro con un diámetro igual a la de la frita y para centrar correctamente este filtro para asegurar que la muestra se extrae a través del filtro en lugar de sobre el borde. La humectación de la membrana con enfriado con hielo, H 2 O estéril puede ayudarCon el posicionamiento de la membrana.
4. La muestra de la cosecha
- A una OD 600 de ~ 0,4-0,5, utilizar una pipeta serológica para eliminar 13 ml de cultivo del matraz y para aplicar la muestra al filtro.
- Después de toda la muestra se ha filtrado, lavar inmediatamente el filtro con ≥5 ml de PBS enfriado en hielo para lavar metabolitos asociados medianas.
- Desconectar el vacío y el uso de un par de pinzas estériles para quitar el filtro de la frita. Invertir el filtro (-células hacia abajo) en la solución de enfriamiento rápido pre-enfriada.
Nota: Es importante realizar los pasos anteriores rápidamente (es decir, en cuestión de segundos) y tan pronto como el líquido se ha eliminado para asegurar el enfriamiento rápido de las células, la detención de la actividad metabólica. - Incubar el filtro en una solución de enfriamiento rápido en hielo seco durante ≥20 min.
- Utilizando pinzas estériles, invertir el filtro (lado celular up) en la placa de Petri y el uso de una micropipeta para enjuagar las células fuera de tél membrana en la solución de enfriamiento rápido.
- Resuspenden las células en solución de enfriamiento rápido y luego transferir la suspensión celular a un tubo de 2 ml resistente a los impactos estéril que contiene ~ 100! L de perlas de sílice 0,1 mm. Almacenar esta en hielo seco o a -80 ° C.
5. Extracción de metabolitos
- Descongelar las muestras en hielo húmedo y romper las células en un homogeneizador con cuatro 30 s ráfagas a 6.000 rpm, con 2 min periodos de enfriamiento en hielo seco entre ciclos.
- Aclarar los lisados durante 15 minutos en un pre-refrigerado, de microcentrífuga refrigerada a la velocidad máxima (es decir, 18.213 xg en ≤4 ° C).
- Transferir el sobrenadante a un tubo de microcentrífuga limpio.
- Usando una micropipeta, transferir una pequeña porción de la muestra a un tubo de microcentrífuga para la cuantificación del contenido de péptido residual en el paso 6; almacenar el resto a -80 ° C.
NOTA: El volumen de muestra reservado varía, dependiendo del ensayo BCA utilizado en la etapa 6.1. Estamuestra debe ser almacenado en hielo húmedo para su análisis inmediato o se congeló a -80 ° C.
6. del ácido bicinconínico (BCA) Ensayo
- Realizar un ensayo de BCA como se recomienda por el fabricante del kit, usando muestras procedentes de la etapa 5.4 para determinar la concentración de péptido residual para cada muestra.
7. LC-MS
- Mezclar 75 l de extracto de S. aureus con 75 l de LC-MS disolvente B, preparado en la etapa 1.4.
- Vórtice para mezclar y centrifugado a 13.000 xg durante 5 min.
- Coloque 100 l de sobrenadante en un vial de cromatografía líquida (LC) y la tapa él. Asegúrese de que no hay burbujas de aire atrapadas en la muestra.
- Cargar los viales en el inyector automático LC-MS y LC editar la lista actualizada en el software "Desconectado lista de trabajo Editor".
- Rellene la "Nombre de ejemplo" (por ejemplo, de tipo salvaje-1), "Posición de ejemplo" (por ejemplo, P1-A1), "método" (por ejemplo, fórmico Acid-Negativo Método), y (por ejemplo, de tipo salvaje-1) columnas "del archivo de datos". Haga clic en el botón en el botón "Guardar lista de trabajo". Abra el software de "Espectrometría de Masas de adquisición de datos de estaciones de trabajo" y la entrada de la lista de trabajo previamente guardado. Haga clic en el botón "Inicio Lista de trabajo Ejecutar" para iniciar la medición continua de LC-MS.
- Separar las muestras en una columna, la columna vincular a un tiempo de vuelo espectrómetro (TOF), y acoplar el espectrómetro de TOF con el sistema LC. Utilice un gradiente de fase móvil de la siguiente manera: 0-2 min, 85% de disolvente B; 3-5 min, 80% de disolvente B; 6-7 min, 75% de disolvente B; 8-9 min, 70% de disolvente B; 10-11.1 min, 50% de disolvente B; 11,1 a 14 min, 20% de disolvente B; y 14,1 a 24 min, 5% de disolvente B; terminar con un período de re-equilibrado de 10 min a 85% de disolvente B y un caudal de 0,4 ml min -1.
- El uso de una bomba isocrática, infundir una solución masa de referencia con la carrera para permitir la calibración del eje masa simultánea.
NOTA: Este pasose basa en el manual de espectrómetro de TOF estándar.- Usar la mezcla de ácido acético y D4 hexakis (1H, 1H, 3H-tetrafluoropropoxi) fosfazina como la solución masa de referencia para realizar la calibración en tiempo real. Utilice la bomba isocrática con la velocidad de flujo de 2,5 ml min-1 durante la infusión.
8. lotes Corrección de Counts Ion
- Designe a cualquier muestra para servir como una muestra de referencia para la corrección por lotes (por ejemplo, de tipo salvaje, replicar 1).
- Calcular la suma de los recuentos de iones para todos los metabolitos dentro de la muestra de referencia. Repetir este cálculo para todas las muestras.
- Divida el recuento de iones total de cada muestra por el recuento total de iones de la muestra de referencia para generar una relación.
- Divida el recuento de iones para cada metabolito dentro de una muestra por la relación de muestra / referencia para obtener un recuento de iones lote corregido para cada metabolito.
9. Péptido Normalización
- reivide los valores de recuento de iones de lote corregido para cada muestra obtenida en la etapa 8 por la concentración de péptido determinado con el ensayo de BCA en el paso 6 para producir un valor normalizado para cada metabolito.
NOTA: Los recuentos de iones, corregido por lotes por lotes normalizados para cada metabolito obtenido en la etapa 9.1 se puede comparar directamente entre cepas y se sometió a análisis estadístico (por ejemplo, una U de Mann-Whitney-test). Alternativamente, un metabolito conocido de ser sin cambios, ya sea por el tratamiento o fondo genético puede ser utilizado como un normalizador para detectar cambios debidos a la metabolito descomposición. La inclusión de una cantidad conocida de L-norvalina o ácido gluraric en el tampón de extracción se puede utilizar para corregir la pérdida durante el procesamiento de la muestra 34.
Resultados
Hemos analizado piscinas de metabolitos intracelulares en S. aureus durante el crecimiento in vitro en un medio rico y complejo. Como prueba de principio, se compararon los perfiles de metabolitos entre el S. osteomielitis aureus sensible a la meticilina aíslan UAMS-1 (de tipo salvaje [WT]) y una cepa isogénica que carece del regulador global transcripcional CodY (Δ Cody) 26. El estado de equilibrio, se establecieron cultivos exponenciales de las cepas WT y Cody en medio TSB, como se describe en el paso 2 del protocolo. El comportamiento de crecimiento de la de tipo salvaje y mutantes culturas Cody -null fueron similares, con sólo diferencias leves en el rendimiento de crecimiento y la tasa (Figura 1). Utilizando la tecnología de microarray RNA-Seq y, nosotros y otros reveló que varios genes que codifican para enzimas implicadas en la biosíntesis de aminoácidos derivados de aspartato se de-reprimidos en el Cody -null mutante compared para células WT durante el crecimiento in vitro en TSB (Figura 2) 25, 27, 30. Por otra parte, brnQ1 y brnQ2, que codifican para permeasas de aminoácidos de cadena ramificada, se sobreexpresa en el mutante nulo codY- 30, 35.
Para determinar el grado en que el estado estacionario abundancias intracelulares de metabolitos asociados con esta vía se alteran en el mutante nulo, se realizó metabolito de perfiles basada en LC-MS. Células mutantes nulos WT y codY- se hicieron crecer hasta el estado de equilibrio biológico y se tomaron muestras como se describe en el paso 4 del protocolo. Se determinó la abundancia de metabolitos mediante la integración del área del pico de iones de intensidad para cada metabolito cromatográficamente resuelto usando un paquete de software de análisis (ver Lista de Materiales). Hemos corregido para ver diferenciaserences en la biomasa por la normalización de las abundancias de metabolitos a la concentración de péptido residual de cada muestra. Hemos corregido aún más estos valores para posibles efectos de lote entre las muestras mediante el cálculo del recuento medio de iones para todos los metabolitos dentro de cada muestra y mediante el uso de la muestra de tipo salvaje como el valor de referencia. Este enfoque permitió las comparaciones entre la muestra de la abundancia de metabolitos a través de condiciones. comparaciones Inter-metabolitos dentro de una muestra dada de manera similar se puede lograr mediante la conversión primero abundancias de metabolitos normalizados de los recuentos de iones a molar cantidades utilizando el método de adición estándar.
Hemos comparado los niveles de intermedios clave en la vía de aspartato en UAMS-1 y su mutante nulo codY-. Como se ve en la Figura 3, los productos finales de esta vía (por ejemplo, treonina y (iso) -leucina) son más abundantes en las células mutantes Cody -null, mientras que los precursores (por ejemplo,aspartato y O -acetil homoserina) son más abundantes en las células WT. La regulación por incremento combinado de BrnQ permeasas 36 y la ruta biosintética ILV probable conduce a aumentos en la isoleucina y leucina 30. Aunque las diferencias son relativamente pequeñas (<4 veces), LC-MS-basado cuantificación y corrección lote revelan cambios robustos y estadísticamente significativas que son consistentes con alteraciones transcripcionales mediadas por Cody.
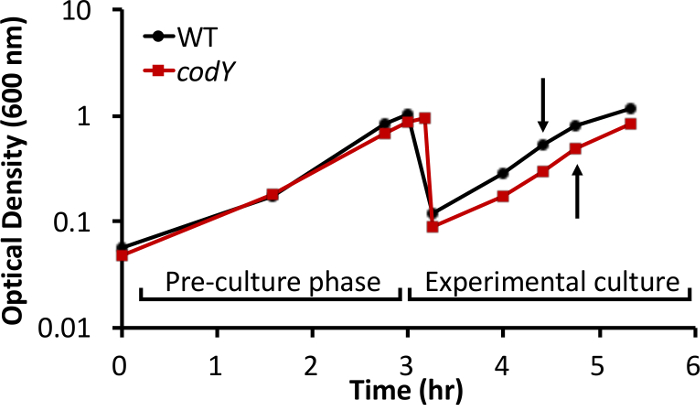
Figura 1: Comportamiento de crecimiento de S. aureus UAMS-1 y un mutante -null Cody isogénica en TSB. Para prolongar el tiempo las células pasados en crecimiento exponencial en estado estacionario, los cultivos se volvieron a diluyeron a una densidad óptica de 0,05 en medio fresco después de la pre-cultivos alcanzaron una DO600 de ~ 1. Se recogieron muestras para el análisis de metabolitos LC-MSa partir de cultivos experimentales a una densidad óptica de ~ 0,5 (flechas). Los datos mostrados son representativos de tres réplicas biológicas.

Figura 2: Representación esquemática de los metabolitos seleccionados derivados de aspartato. Los genes y operones cuyos productos catalizar la síntesis de los aminoácidos aspartato-familia son indicadas en cursiva; el aumento en la abundancia de transcripción en un mutante -null Cody comparación con el tipo salvaje, como se determina por análisis de RNA-seq 29, se observó también. DHP, 2,6-diaminoheptanedioate (2,6-diaminopimelato).

Figura 3: abundancias de metabolitos en la familia aspartato se alteran en un mutante Cody. El registro de 2 cambios -fold de metabolitos seleccionados en else muestran codY- mutante nulo en comparación con UAMS-1 (WT). El cambio se determina dividiendo la abundancia media de tres réplicas biológicas de la cepa nula codY- por la abundancia media de tres réplicas biológicas de la WT strain. El error estándar entre las réplicas biológicas para cada metabolito era <35%. Las líneas discontinuas indican un registro de cambios 2 1,5 veces, el punto de corte usado en este experimento.
Discusión
Todos los metabolitos de molécula pequeña se conectan entre sí a través de sus orígenes comunes en las vías metabólicas centrales. Durante el crecimiento exponencial, las células bacterianas están en el estado de equilibrio biológico y metabólica, proporcionando una instantánea del estado fisiológico en condiciones específicas. CodY supervisa la suficiencia de nutrientes al responder a ILV y GTP. Como ILV y GTP piscinas gota, la actividad Cody es probable reduce progresivamente, el ajuste de la expresión de sus genes diana para adaptarse a la creciente agotamiento de los nutrientes 30. Una cepa deficiente en CodY se comporta como si ILV y GTP se agotan desde el medio ambiente, pero sólo exhibe una diferencia muy suave en el comportamiento de crecimiento relativo a la cepa CodY-competente (Figura 1). Por lo tanto, la comparación de las piscinas en estado estacionario de metabolitos en estas cepas nos proporciona una oportunidad única para revelar el grado en que el metabolismo se reconfigura cuando los nutrientes son escasos. Cabe señalar que our experimentos investigan abundancias de metabolitos cuando la actividad CodY se maximiza (ILV y GTP son más abundantes en la fase exponencial). Sin embargo, otras preguntas pueden ser dirigidas en otras fases de crecimiento. Por ejemplo, en la fase exponencial, ácido tricarboxílico (TCA) la actividad del ciclo es muy bajo; en la fase post-exponencial, el ciclo TCA se activa 36. Un número de fenotipos, incluyendo abundancias de metabolitos, son dependientes de esta activación. Además, el sistema de percepción de quórum agr se activa durante la transición de crecimiento exponencial a la fase estacionaria 37. Recolección de muestras en fase post-exponencial o estacionario puede ser más relevante para los estudios que abordan estos temas. Independientemente de cuando se cosechan las muestras, es importante para recoger, lavar y transferir las muestras al tampón de extracción lo más rápidamente posible (es decir, dentro de s) para reducir al mínimo el volumen de negocios metabolito que se produce cuando el estado de equilibrio es perturbado.
El método cromatográfico descrito aquí es especialmente adecuado para el análisis de aminoácidos polares y no polares y compuestos centrales del metabolismo de carbono. Sin embargo, los métodos adicionales específicos de clase se puede utilizar para cuantificar compuestos específicos de interés no resuelto cromatográficamente por este método. Por ejemplo, alterando el pH de la fase móvil cromatográfica mediante la sustitución de ácido fórmico con ácido acético como aditivo ha sido reportado para permitir la resolución de isoleucina y leucina 38. Modificar el procedimiento de extracción puede permitir de manera similar la recuperación y cuantificación de metabolitos lábiles que son sensibles al método de extracción utilizado aquí. Por ejemplo, compuestos tales como cisteína que son propensos a la formación de enlaces de disulfuro potencialmente se pueden recuperar después de la derivatización con reactivo de Ellman 39. Tampones de extracción de burbujeo con gas nitrógeno pueden preservar NAD + y NADHlas proporciones, lo que permite una evaluación del estado redox celular 40. trifosfatos de nucleósidos pueden descomponerse en soluciones básicas o no tamponadas; la acidificación de la solución de extracción puede mejorar la recuperación de estas moléculas 41.
En un cultivo bacteriano en crecimiento exponencial, el agotamiento de uno o más nutrientes conduce a la transición a las fases de post-exponencial y estacionaria de crecimiento. Estas fases de crecimiento se caracterizan por estados metabólicos distintos 42. culturas basados en quimiostato generan un estado estacionario ideales biológica virtualmente continua para la expresión génica y los estudios fisiológicos. Sin embargo, el método requiere un equipo especializado, es técnicamente exigente, y requiere que una limitación de nutrientes imponerse para mantener una población estable de células bajo flujo. El último requisito provoca perturbaciones de la transcripción y fisiológicos debido a factores distintos de la variable o regulator siendo analizado. Para asegurar que nuestros resultados basados en frasco son representativos de células en crecimiento exponencial en el estado estacionario y no de una transición entre dos fases, empleamos una estrategia de dilución doble de nuevo con un frasco consistente: relación de volumen (ligeros cambios en los niveles de oxígeno pueden conducir a metabolismo alterado 36, 42). Después de una dilución inicial de cultivos de una noche, estas células se hacen crecer hasta una DO 600 de ~ 1,0, back-diluyó hasta una DO 600 de ~ 0,05, y se cosechan cuando alcanzan una DO 600 de ~ 0,5. Tal método también diluye moléculas citoplasmáticas que se acumulan durante el crecimiento durante la noche, incluyendo los ARN estables. De hecho, ARNIII, el efector del sistema de detección de quórum agr, es uno de tales ARN y regula la expresión de algunos de los mismos objetivos de genes como CodY 25, 43, 44. ARNIII acumulada puede enmascarar CodY-depregulación endent, que conduce a una subestimación de la fuerza de represión o estimulación por Cody (Sharma y Brinsmade, resultados no publicados).
Una limitación de este análisis es que proporciona una visión instantánea de abundancias de metabolitos dentro de la célula; no se pueden sacar conclusiones a partir de los resultados con respecto a cambios en el flujo a través de cualquier vía determinada. Por ejemplo, las abundancias de lisina y metionina entre las dos cepas examinadas no cambió, a pesar de la represión de las enzimas biosintéticas en la cepa nula codY- (Figuras 2 y 3). La cepa -null Cody puede de hecho ser la generación de más de lisina y metionina, pero que se puede convertir rápidamente en otros compuestos; Por lo tanto, estas moléculas no se acumulan. Fuentes de carbono o de nitrógeno marcadas con N Uso 13 C- o 15 nos permitiría seguimos esqueletos de carbono y nitrógeno a través de los cruces principales metabólicos 45 , 46.
Hemos utilizado el método descrito para dilucidar los cambios en piscinas de metabolitos en S. aureus, B. subtilis 29, Mycobacterium tuberculosis 47, y Enterococcus faecium 48, pero el método se puede aplicar a otras bacterias Gram-positivas y Gram-negativas, incluyendo otra patógenos humanos fácilmente cultivadas en el laboratorio. De hecho, la integración de la información y la metabolómica transcriptómica puede revelar conexiones inesperadas entre el metabolismo y la virulencia, lo que podría conducir a nuevas estrategias para el tratamiento de infecciones.
Divulgaciones
Los autores declaran que no tienen intereses financieros en competencia.
Agradecimientos
Este trabajo fue financiado en parte por un NIH Camino a la concesión de la Independencia (GM conceder 099.893) y los fondos de inicio facultad de SRB, así como un proyecto de investigación de Grant (subvención GM 042219). Los proveedores de fondos no tuvieron ningún papel en el diseño del estudio, la recogida de datos e interpretación, o la decisión de presentar el trabajo para su publicación.
Materiales
| Name | Company | Catalog Number | Comments |
| Material/Equipmenta | |||
| DeLong Culture Flask (250 mL) | Belco | 2510-00250 | |
| Sidearm Flask, 500 mL | Pyrex | 5340 | |
| 3-hole Rubber Stopper, #7 | Fisher | 14-131E | |
| Stainless Steel Filter holder/frit | VWR | 89428-936 | |
| Petri Dish, 35 mm | Corning | 430588 | Not tissue culture treated |
| Mixed cellulose ester membrane, 0.22 μm pore size | Millipore | GSWP02500 | |
| Impact-resistant tubes, 2 mL | USA Scientific | 1420-9600 | |
| Silica Beads, 0.1 mm | Biospec Products Inc | 11079101Z | |
| Precellys 24 homogenizer | Bertin Instruments | EQ03119-200-RD000.0 | |
| Micro BCA Protein Assay Kit | Pierce (Thermo Scientific) | 23235 | |
| Cogent Diamond hydride type C column | Agilent | 70000-15P-2 | |
| Accurate-Mass Time-of-Flight (TOF) LC-MS, 6200 Series | Agilent | G6230B | |
| Quat Pump, 1290 Series | Agilent | G4204A | |
| Bin Pump, 1290 Series | Agilent | G4220A | |
| Valve Drive, 1290 Series | Agilent | G1107A | |
| Isocratic Pump, 1290 Series | Agilent | G1310B | |
| TCC, 1290 Series | Agilent | G1316C | |
| Sampler, 1290 Series | Agilent | G4226A | |
| Thermostat, 1290 Series | Agilent | G1330B | |
| Chemical | |||
| Tryptic Soy Broth | Becton Dickinson | 211825 | |
| Difco Agar, Granulated | Becton Dickinson | 214530 | Solid media contains 1.5% [w/v] agar |
| Phosphate-buffered saline (pH 7.4) 10x | Ambion | AM9624 | Dilute fresh to 1x with ultra-pure water |
| Acetonitrile | Fisher Scientific | A955-500 | Optima LC-MS |
| Methanol | Fisher Scientific | A456-500 | Optima LC-MS; toxic |
| Formic Acid | Sigma Aldrich | 94318 | For mass spectrometry, 98% |
| Software | |||
| MassHunter | Agilent | G3337AA | |
| Bacterial Strain | Species | Strain | Genotype |
| SRB 337 | Staphylococcus aureus | USA200 MSSA UAMS-1 | wild type |
| SRB 372 | Staphylococcus aureus | USA200 MSSA UAMS-1 | ΔcodY::erm |
| aChemicals and materials listed are specific to the method described and do not include standard laboratory chemicals or supplies. | |||
Referencias
- Hood, M. I., Skaar, E. P. Nutritional immunity: transition metals at the pathogen-host interface. Nat. Rev. Microbiol. 10 (8), 525-537 (2012).
- Weinberg, E. D. Clinical enhancement of nutritional immunity. Comp. Ther. 1 (5), 38-40 (1975).
- Ibberson, C. B., et al. Staphylococcus aureus hyaluronidase is a CodY-regulated virulence factor. Infect. Immun. 82 (10), 4253-4264 (2014).
- Lee, C. Y., Iandolo, J. J. Mechanism of bacteriophage conversion of lipase activity in Staphylococcus aureus. J. Bacteriol. 164 (1), 288-293 (1985).
- Olson, M. E., et al. Staphylococcus aureus nuclease is an SaeRS-dependent virulence factor. Infect. Immun. 81 (4), 1316-1324 (2013).
- Somerville, G. A., Proctor, R. A. At the crossroads of bacterial metabolism and virulence factor synthesis in Staphylococci. Microbiol. Mol. Biol. Rev. 73 (2), 233-248 (2009).
- Seidl, K., et al. Staphylococcus aureus CcpA affects virulence determinant production and antibiotic resistance. Antimicrob. Agents Chemother. 50 (4), 1183-1194 (2006).
- Richardson, A. R., Somerville, G. A., Sonenshein, A. L. Regulating the intersection of metabolism and pathogenesis in Gram-positive bacteria. Microbiol. Spectr. 3 (3), 1-27 (2015).
- Geiger, T., et al. Role of the (p)ppGpp synthase RSH, a RelA/SpoT homolog, in stringent response and virulence of Staphylococcus aureus. Infect. Immun. 78 (5), 1873-1883 (2010).
- Gaupp, R., et al. RpiRc is a pleiotropic effector of virulence determinant synthesis and attenuates pathogenicity in Staphylococcus aureus. Infect. Immun. 84 (7), 2031-2041 (2016).
- Serror, P., Sonenshein, A. L. Interaction of CodY, a novel Bacillus subtilis DNA-binding protein, with the dpp promoter region. Mol. Microbiol. 20 (4), 843-852 (1996).
- Sonenshein, A. L. CodY, a global regulator of stationary phase and virulence in Gram-positive bacteria. Curr. Opin. Microbiol. 8 (2), 203-207 (2005).
- Brinsmade, S. R. CodY, a master integrator of metabolism and virulence in Gram-positive bacteria. Curr. Genet. , (2016).
- Molle, V., et al. Additional targets of the Bacillus subtilis global regulator CodY identified by chromatin immunoprecipitation and genome-wide transcript analysis. J. Bacteriol. 185 (6), 1911-1922 (2003).
- Moses, S., et al. Proline utilization by Bacillus subtilis: Uptake and catabolism. J. Bacteriol. 194 (4), 745-758 (2012).
- Lobel, L., Herskovits, A. A. Systems level analyses reveal multiple regulatory activities of CodY controlling metabolism, motility, and virulence in Listeria monocytogenes. PLoS Genet. 12 (2), 1-27 (2016).
- Belitsky, B. R., Sonenshein, A. L. CodY-mediated regulation of guanosine uptake in Bacillus subtilis. J. Bacteriol. 193 (22), 6276-6287 (2011).
- den Hengst, C. D., Buist, G., Nauta, A., Van Sinderen, D., Kuipers, O. P., Kok, J. Probing direct interactions between CodY and the oppD promoter of Lactococcus lactis. Microbiol. 187 (2), 512-521 (2005).
- Fisher, S. H. Regulation of nitrogen metabolism in Bacillus subtilis: vive la différence. Mol. Microbiol. 32 (2), 223-232 (1999).
- Dineen, S. S., McBride, S. M., Sonenshein, A. L. Integration of metabolism and virulence by Clostridium difficile CodY. J. Bacteriol. 192 (20), 5350-5362 (2010).
- Dineen, S. S., Villapakkam, A. C., Nordman, J. T., Sonenshein, A. L. Repression of Clostridium difficile toxin gene expression by CodY. Mol. Microbiol. 66 (1), 206-219 (2007).
- Hendriksen, W. T., et al. CodY of Streptococcus pneumoniae: Link between nutritional gene regulation and colonization. J. Bacteriol. 190 (2), 590-601 (2008).
- Bennett, H. J., et al. Characterization of relA and codY mutants of Listeria monocytogenes: Identification of the CodY regulon and its role in virulence. Mol. Microbiol. 63 (5), 1453-1467 (2007).
- Stenz, L., Francois, P., Whiteson, K., Wolz, C., Linder, P., Schrenzel, J. The CodY pleiotropic repressor controls virulence in Gram-positive pathogens. FEMS Immunol. and Med. Microbiol. 62 (2), 123-139 (2011).
- Majerczyk, C. D., et al. Direct targets of CodY in Staphylococcus aureus. J. Bacteriol. 192 (11), 2861-2877 (2010).
- Majerczyk, C. D., Sadykov, M. R., Luong, T. T., Lee, C., Somerville, G. A., Sonenshein, A. L. Staphylococcus aureus CodY negatively regulates virulence gene expression. J. Bacteriol. 190 (7), 2257-2265 (2008).
- Pohl, K., et al. CodY in Staphylococcus aureus: A regulatory link between metabolism and virulence gene expression. J. Bacteriol. 191 (9), 2953-2963 (2009).
- Sonenshein, A. L. Control of key metabolic intersections in Bacillus subtilis. Nat. Rev. Microbiol. 5 (12), 917-927 (2007).
- Brinsmade, S. R., et al. Hierarchical expression of genes controlled by the Bacillus subtilis global regulatory protein CodY. Proc. Nat. Acad. Sci. U.S.A. 111 (22), 2-7 (2014).
- Waters, N. R., et al. A spectrum of CodY activities drives metabolic reorganization and virulence gene expression in Staphylococcus aureus. Mol. Microbiol. 101 (3), 495-514 (2016).
- Zhou, B., Xiao, J. F., Tuli, L., Ressom, H. W. LC-MS-based metabolomics. Mol. Biosyst. 8 (2), 470-481 (2012).
- Somerville, G. A., et al. Staphylococcus aureus aconitase inactivation unexpectedly inhibits post-exponential-phase growth and enhances stationary-phase survival. Infect. Immun. 70 (11), 6373-6382 (2002).
- Somerville, G. A., Said-Salim, B., Wickman, J. M., Raffel, S. J., Kreiswirth, B. N., Musser, J. M. Correlation of acetate catabolism and growth yield in Staphylococcus aureus: Implications for host-pathogen interactions. Infect. Immun. 71 (8), 4724-4732 (2003).
- Brinsmade, S. R., Kleijn, R. J., Sauer, U., Sonenshein, A. L. Regulation of CodY activity through modulation of intracellular branched-chain amino acid pools. J. Bacteriol. 192 (24), 6357-6368 (2010).
- Kaiser, J. C., Omer, S., Sheldon, J. R., Welch, I., Heinrichs, D. E. Role of BrnQ1 and BrnQ2 in branched-chain amino acid transport and virulence in Staphylococcus aureus. Infect. Immun. 83 (3), 1019-1029 (2015).
- Ledala, N., Zhang, B., Seravalli, J., Powers, R., Somerville, G. A. Influence of iron and aeration on Staphylococcus aureus growth, metabolism, and transcription. J. Bacteriol. 196 (12), 2178-2189 (2014).
- Novick, R. P. Autoinduction and signal transduction in the regulation of staphylococcal virulence. Mol. Micorbiol. 48 (6), 1429-1449 (2003).
- Pesek, J. J., Matyska, M. T., Fischer, S. M., Sana, T. R. Analysis of hydrophilic metabolites by high-performance liquid chromatography-mass spectrometry using a silica hydride-based stationary phase. J. Chromatog. A. 1204 (1), 48-55 (2008).
- Guan, X., Hoffman, B., Dwivedi, C., Matthees, D. P. A simultaneous liquid chromatography/mass spectrometric assay of glutathione, cysteine, homocysteine and their disulfides in biological samples. J. Pharm. Biomed. Anal. 31 (2), 251-261 (2003).
- Sporty, J. L., Kabir, M. M., Turteltaub, K. W., Ognibene, T., Lin, S. J., Bench, G. Single sample extraction protocol for the quantification of NAD and NADH redox states in Saccharomyces cerevisiae. J. Sep. Sci. 31 (18), 3202-3211 (2008).
- Rabinowitz, J. D., Kimball, E. Acidic acetonitrile for cellular metabolome extraction from Escherichia coli. Anal. Chem. 79 (16), 6167-6173 (2007).
- Somerville, G. A., Powers, R. Growth and preparation of Staphylococcus epidermidis for NMR metabolomic analysis. Methods Mol. Biol. 1106, 71-91 (2014).
- Roux, A., Todd, D. A., Velazquez, J. V., Cech, N. B., Sonenshein, A. L. CodY-Mediated regulation of the Staphylococcus aureus Agr system integrates nutritional and population density signals. J. Bacteriol. 196 (6), 1184-1196 (2014).
- Guillet, J., Hallier, M., Felden, B. Emerging functions for the Staphylococcus aureus RNome. PLoS Pathog. 9 (12), 1003767 (2013).
- Sauer, U., et al. Metabolic flux ratio analysis of genetic and environmental modulations of Escherichia coli central carbon metabolism. J. Bacteriol. 181 (21), 6679-6688 (1999).
- Niittylae, T., Chaudhuri, B., Sauer, U., Frommer, W. B. Comparison of Quantitative Metabolite Imaging Tools and Carbon-13 Techniques for Fluxomics. Methods Mol. Biol. 553 (1), 355-372 (2009).
- de Carvalho, L. P. S., Fischer, S. M., Marrero, J., Nathan, C., Ehrt, S., Rhee, K. Y. Metabolomics of Mycobacterium tuberculosis reveals compartmentalized co-catabolism of carbon substrates. Chem. Biol. 17 (10), 1122-1131 (2010).
- Weisenberg, S. A., Butterfield, T. R., Fischer, S. M., Rhee, K. Y. Suitability of silica hydride stationary phase, aqueous normal phase chromatography for untargeted metabolomic profiling of Enterococcus faecium and Staphylococcus aureus. J. Sep. Sci. 32 (13), 2262-2265 (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados