Method Article
De un solo paso de purificación de proteínas etiquetadas-Twin-Strep y sus complejos de Strep-Tactin resina reticulado con Bis (sulfosuccinimidil) suberato (BS3)
En este artículo
Resumen
Se describe un método para la purificación eficiente de proteínas de fusión doble-Strep-etiquetados y sus complejos específicos sobre estreptavidina modificada (Strep-Tactin) de resina covalentemente reticulado con bis (sulfosuccinimidil) suberato (BS3). El método tiene las ventajas de velocidad rápida, buena recuperación proteína diana y alta pureza, y es compatible con el análisis posterior por espectrometría de masas.
Resumen
La purificación por afinidad de proteínas de fusión Strep-etiquetados en resinas que llevan una estreptavidina de ingeniería (Strep-Tactin) se ha convertido en un método ampliamente utilizado para el aislamiento de complejos de proteínas en condiciones fisiológicas. Las proteínas de fusión que contienen dos copias de Strep-tag II, designado doble-Strep-tag o SIII-etiqueta, tienen la ventaja de una mayor afinidad por Strep-Tactin en comparación con los que contienen sólo un único marcador Strep, permitiendo de este modo la purificación de proteínas más eficiente. Sin embargo, esta ventaja se ve compensada por el hecho de que la elución de las proteínas marcadas con doble Strep con biotina puede ser incompleta, lo que lleva a la recuperación baja en proteínas. La recuperación se puede mejorar drásticamente mediante el uso de elución desnaturalizante con dodecil sulfato de sodio (SDS), pero esto conduce a la contaminación de la muestra con Strep-Tactin liberado de la resina, por lo que el ensayo incompatible con el análisis proteómico de aguas abajo. Para superar esta limitación, se ha desarrollado un método por el cual tetrámero acoplado a la resina de Strep-Tactinprimero se estabiliza mediante la reticulación covalente con bis (sulfosuccinimidil) suberato (BS3) y la resina reticulada resultante se utiliza a continuación para purificar los complejos de proteína diana en una única etapa de purificación por lotes. Elución eficiente con SDS asegura una buena recuperación de proteína, mientras que la ausencia de contaminación de Strep-Tactin permite el análisis de proteínas aguas abajo por espectrometría de masas. Como una prueba de concepto, se describe aquí un protocolo para la purificación de la proteína viral SIII etiquetada VPg-Pro a partir de núcleos de N. infectados por el virus benthamiana plantas utilizando la resina de polimetacrilato de Strep-Tactin reticulado con BS3. El mismo protocolo se puede utilizar para purificar cualquier proteína marcada-Strep-gemelo de interés y caracterizar sus parejas de unión fisiológicos.
Introducción
En los últimos años, la tecnología Strep-tag ha sido ampliamente utilizado en muchas áreas de la investigación biomédica, incluida la proteómica y la biología estructural. Esta tecnología de purificación de proteínas, que se basa en la fusión de proteínas recombinantes a un péptido Strep-tag corto, ha madurado con el advenimiento de matrices de afinidad que llevan Strep-Tactin, una variante modificada genéticamente de la estreptavidina con la mejora de la capacidad de unión al péptido. 1, 2 Las proteínas de fusión que contienen dos copias de Strep-tag II, designado twin-Strep-tag o SIII-tag, muestran una mayor afinidad por matrices Strep-Tactin que las que contienen sólo un único Strep-tag, asegurando la purificación más eficiente de las proteínas recombinantes y sus parejas de unión asociados. Sin embargo, la mayor afinidad de las proteínas marcadas con doble Strep a Strep-Tactin también tiene su lado negativo. Elución competitiva de estas proteínas con el exceso de biotina puede ser incompleta, lo que lleva a la disminución de la producción de proteína objetivo. Una mmineral eficiente alternativa es elución con SDS, pero conduce a contaminación de la muestra no deseado con Strep-Tactin liberado de la resina, por lo que el ensayo incompatible con el análisis proteómico. Este trabajo presenta una técnica para superar esta limitación por primera estabilizar el tetrámero unido a resina de Strep-Tactin por entrecruzamiento químico y luego utilizando SDS para eluir proteínas etiquetadas-twin-Strep y sus complejos asociados de la resina reticulada resultante. Por lo tanto, suficiente rendimiento de proteína se puede lograr sin contaminación de la muestra con Strep-Tactin, permitiendo de este modo su posterior análisis por espectrometría de masas.
El método es adecuado para la purificación de cualquier proteína de fusión recombinante con una SIII-tag expuesto a la superficie 3 o twin-Strep-tag (secuencia de aminoácidos WSHPQFEK (GGGS) 3 WSHPQFEK y SAWSHPQFEK (GGGS) 2 GGSAWSHPQFEK, respectivamente). La proteína puede ser de origen animal, vegetal o bacteriana y puede ser aislado a partir de ya sea total de célulaslisado o fracción enriquecida orgánulo. Como un ejemplo, se describe aquí la purificación de una proteína-SIII etiquetado VPg-Pro de la patata de virus A (PVA) 4 de la fracción nuclear de plantas de Nicotiana benthamiana infectadas por el PVA. La fracción nuclear se aisló tal como se describe anteriormente 5, con las siguientes modificaciones: las células no fueron tratados con formaldehído, butirato de sodio fue sustituido en todos los tampones con fluoruro de sodio 5 mM, inhibidor de proteasa completo fue sustituido con PMSF, Triton X-100 en la extracción de concentración tampón # 2 se redujo a 0,3% (v / v) y el sedimento nuclear obtenido por centrifugación a través de colchón de sacarosa (tampón de extracción # 3) se resuspendió en 1,45 ml de tampón de unión enfriado previamente y se hace girar durante 1,5 horas a 4 ° C. El extracto nuclear resultante que contiene la proteína cebo-etiquetados SIII y complejos asociados (muestra cebo de proteína) se procesó de acuerdo con el protocolo descrito a continuación (véase la sección 2).
Protocolo
1. La reticulación de Strep-Tactin polimetacrilato de resina con Bis (sulfosuccinimidil) suberato (BS3)
- Equilibrar un microtubo sellado que contiene 2 mg de BS3 agente de reticulación a temperatura ambiente. PRECAUCIÓN: BS3 es una sustancia peligrosa. Utilice guantes y gafas protectoras.
- Resina de polimetacrilato Resuspender Strep-Tactin (suspensión al 50% en 100 mM de Tris-HCl, pH 8,0; EDTA 1 mM; NaCl 150 mM) mediante una breve agitación vigorosa y transferir inmediatamente 600 l de la suspensión a una columna de centrifugación usando una punta de pipeta con la extremo cortado.
- Centrifugar a 1500 xg durante 30 segundos a temperatura ambiente. Desechar el flujo a través y añadir 450 l de tampón fosfato salino (PBS) a la columna.
- Repita el paso anterior 2 veces más para reemplazar completamente el tampón Tris con PBS y dejar la resina en 430 l de PBS, ajustada a pH 8,0 después de la última centrifugación.
- Perfore el papel aluminio de la microtubo con BS3 con una punta de pipeta que contiene 1001; l de agua ultrapura. Disuelva el polvo en agua BS3 pipeteando suavemente hacia arriba y hacia abajo y añadir inmediatamente 20 l de la solución a la columna de centrifugación. La concentración final de BS3 en la reacción de reticulación es de ~ 1,2 mM.
- Girar la columna durante 30 min a temperatura ambiente. Compruebe que la resina se mezcla adecuadamente con la solución de BS3.
- Para interrumpir la reacción, añadir 6 l de 3M Tris-HCl, pH 7,5 y girar la columna durante otros 15 min a temperatura ambiente.
- Centrifugar a 1500 xg durante 30 segundos a temperatura ambiente. Desechar el flujo a través y resuspender la resina reticulada en 450 l de solución salina tamponada con Tris con Tween 20 (TBST). Repetir la centrifugación y etapas de lavado dos veces más. En el último paso, resuspender la resina en 450 l de TBS.
- Transferir la suspensión de resina a un tubo nuevo usando una punta de pipeta con el extremo cortado. Resuspender la resina que queda en la columna con otros 450 l de TBS y transferir al mismo tubo. Repita ªe último paso una vez más para asegurar la transferencia máxima de la resina de la columna al tubo.
- Deje que el tubo de reposar durante 10 minutos a temperatura ambiente y ajustar el volumen a 600 l, eliminando el exceso de TBS. La resina está lista para su uso inmediato (recomendado) o se puede almacenar a 4 ° C, sin congelar.
2. Unión de la proteína cebo etiquetados-Twin-Strep y Associated Complejos para el reticulado Strep-Tactin Polimetacrilato Resina
- Centrifugar 1 ml de la muestra de proteína cebo en tampón de unión a 17000 xg durante 10 min a 4 ° C y transferir el sobrenadante a un tubo nuevo.
- Para minimizar la unión de proteínas biotiniladas endógenos a la resina de Strep-Tactin, añadir avidina a una concentración final de 100 mg / ml y girar durante 15 minutos a 4 ° C.
- Si la resina reticulada de la etapa 1.10 ha sido almacenado durante un período prolongado de tiempo a 4 ° C, recoger la resina por centrifugación a 400 xg FOR 1 min a 4 ° C, descartar el sobrenadante y lavar la resina con 1 ml de TBST. Repetir la centrifugación y etapas de lavado dos veces más, primero con TBST y después con TBS. Centrifugar el tubo a 400 xg durante 1 min a 4 ° C y ajustar hasta el volumen original mediante la eliminación de exceso de TBS.
- Resuspender la resina Strep-Tactin reticulado mediante agitación. Inmediatamente después, añadir 50 l de la suspensión de resina al tubo que contiene la muestra de proteína cebo usando una punta de pipeta de corte y girar durante otros 30 min a 4 ° C.
- Mientras espera, establecer termomezclador a 55 ° C y precalentar 500 l de tampón de elución para el uso en el paso 3.1.
- Centrifugar a 400 xg durante 1 min a 4 ° C. Eliminar el sobrenadante y lavar la resina en un aparato rotatorio durante 5 min a 4 ° C con 1 ml de pre-refrigerada tampón de lavado # 1. Repetir la centrifugación y etapas de lavado tres veces. En el último paso, resuspender la resina en 250 l de tampón de lavado # 2.
- Tr ansfer la suspensión de resina a una columna de giro fresco. Resuspender la resina que queda en el tubo en otros 250 l de tampón de lavado # 2 y transferir a la misma columna.
- Se centrifuga a 400 g durante 3 min a 4 ° C, desechar el flujo a través y la transferencia de la columna a un tubo ml delfines nariz 2 fresco. Proceda inmediatamente a la etapa de elución a continuación.
3. La elución de los complejos de proteínas específicas
- Añadir 150 l de tampón de elución precalentado desde el paso 2.4 a la columna de centrifugación.
- Incubar en termomezclador durante 5 min a 55 ° C, agitando a 1400 rpm.
- Centrifugar a 1500 xg durante 1 min a temperatura ambiente.
- Deseche la columna y almacenar las proteínas diana purificadas a ≤ -20 ° C.
| Tampón fosfato salino (PBS) | |
| Na 2 HPO 4 | 10 mM |
| KH 2 PO 4 | 2 mM |
| NaCl | 137 mM |
| KCl | 2,7 mM |
| pH ajustado a 7,4 a menos que se indique lo contrario (de pH 8,0 en la sección 1.4) | |
| Solución salina tamponada con Tris (TBS) | |
| Tris-HCl, pH 7,4 | 50 mM |
| ght = estilo "20" = "altura: 20px; ancho: 299px;"> NaCl | 150 mM |
| Tris amortiguada salina con Tween 20 (TBST) | |
| Tris-HCl, pH 7,4 | 50 mM |
| NaCl | 150 mM |
| De Tween 20 | 0,1% (v / v) |
| El tampón de unión | |
| Tris-HCl, pH 8,0 | 25 mM |
| NaCl | |
| NaF | 5 mM |
| EDTA | 0,5 mM |
| Glicerol | 10% (v / v) |
| PMSF | 0,1 mM |
| Tampón de lavado # 1 | |
| Tris-HCl, pH 8,0 | 25 mM |
| NaCl | 500 mM |
| NaF td> | 5 mM |
| EDTA | 0,4 mM |
| Igepal CA-630 | 0,2% (v / v) |
| Glicerol | 5% (v / v) |
| PMSF | 0,1 mM |
| Tampón de lavado # 2 | |
| Tris-HCl, pH 8,0 | 25 mM |
| NaCl | 150 mM |
| Tris-HCl, pH 8,0 | 25 mM |
| SDS | 1% (w / v) |
Tabla 1. Tampones utilizados en el presente estudio.
Resultados
El procedimiento de purificación se ilustra esquemáticamente en la Figura 1, junto con una representación de los problemas asociados con otros métodos de purificación existentes.
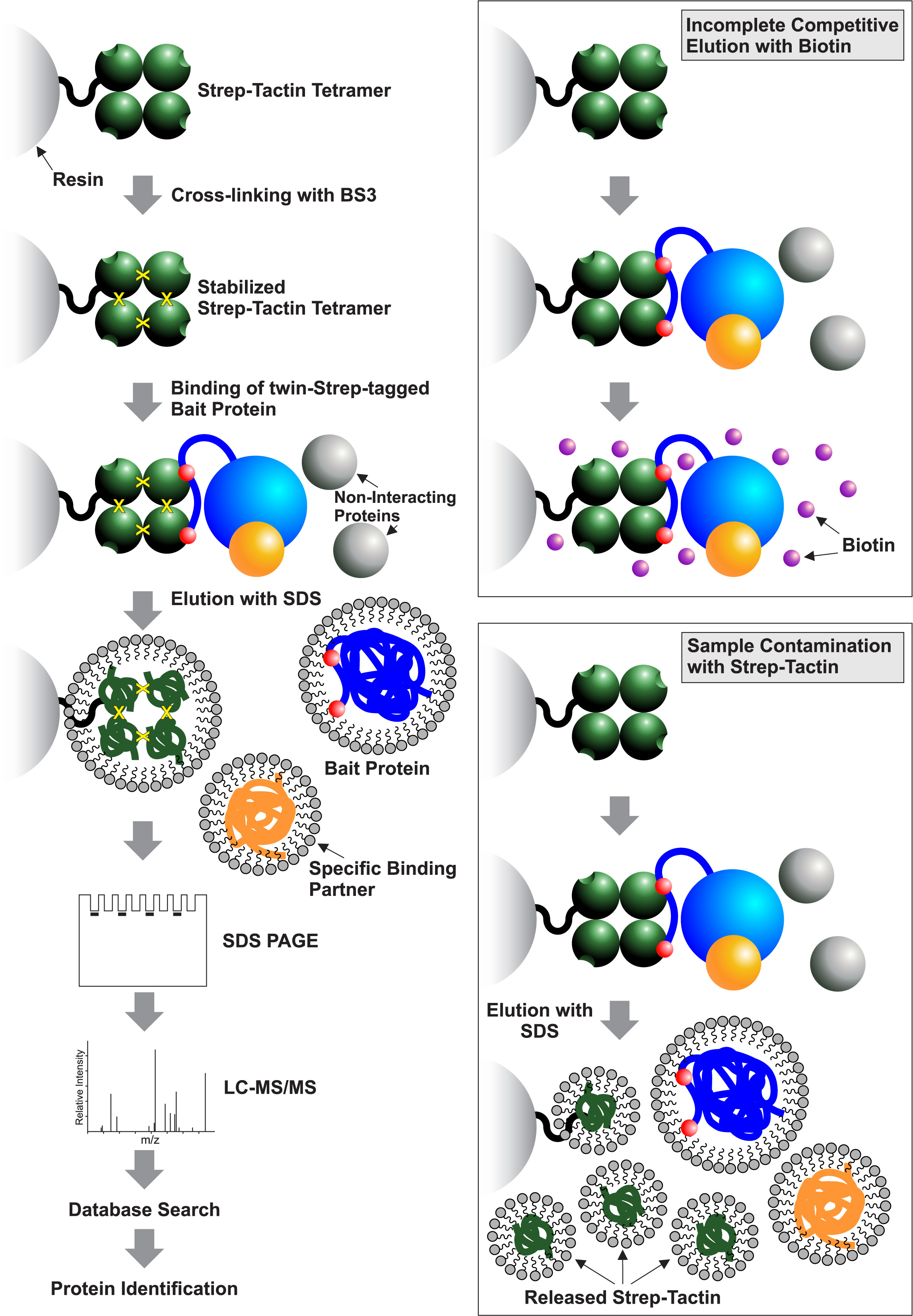
Figura 1. Representación esquemática del procedimiento de purificación. El flujo de trabajo incluye la estabilización de Strep-Tactin tetrámero acoplado a la resina a través de la reticulación covalente con BS3, unión de la proteína cebo de etiquetado de doble Strep y complejos asociados a la resina reticulada , lavando las proteínas no unidas, la desnaturalización de elución de los complejos de proteínas unidos específicamente con SDS al 1% y su análisis por cromatografía-espectrometría de masa líquida (LC-MS/MS). Dos esferas de color rojo en la proteína cebo denotan el gemelo-Strep-tag. Las limitaciones de otros métodos de purificación se muestran en la insercióncuadros de la derecha. Ellos incluyen elución incompleta de proteínas diana con biotina y contaminación de la muestra con Strep-Tactin después de la elución desnaturalización con SDS al 1%.
Los resultados típicos de la purificación de proteínas SIII-etiquetados utilizando la resina de polimetacrilato de Strep-Tactin-reticulado BS3 y desnaturalización de elución con 1% de SDS se muestran en la Figura 2. 10% (15 l) del eluato de la columna de centrifugado que contiene purificada-SIII etiquetados proteínas de PVA VPg-Pro y asociadas se analizaron por SDS-PAGE seguido por tinción con plata. La ausencia de la banda de 52 kDa correspondiente a la de PVA VPg-Pro en el carril de control negativo confirmó la especificidad de la desplegable. 40 l de eluato de la columna de centrifugado restante se aplicaron a un gel de SDS-queratina libre y el carril correspondiente se cortó y se digirieron las proteínas contenidas con tripsina y se analizaron por cromatografía-espectrometría de masas tándem líquido (LC-MS/MS). El análisis de espectrometría de masas identificado viral RNA-dependent RNA polimerasa (replicasa) NIb como la interacción socio de VPg-Pro. Se han detectado múltiples péptidos trípticos que corresponden a NIb en todas las cuatro réplicas biológicas de la purificación por afinidad y ninguno en los cuatro controles que expresan VPg-Pro sin la SIII-etiqueta, lo que confirma la especificidad de la interacción. Debido a que el peso molecular de la punta (59 kDa) es cercana a la de etiquetado-SIII VPg-Pro (52 kDa), las dos proteínas aparecieron como una banda doble durante el análisis de SDS PAGE (Figura 2, punta de flecha). Tomados en conjunto, los resultados descritos anteriormente proporcionan evidencia experimental de que la purificación por afinidad en resinas de Strep-Tactin covalentemente reticulado con BS3 se puede emplear con éxito para aislar proteínas etiquetadas-Strep-doble e identificar sus parejas de unión fisiológicos por espectrometría de masas.

Figura 2. Purificación de SIII-etiquetado de PVA VPg-Pro y su pareja de unión de PVA NIb en resina de polimetacrilato de Strep-Tactin reticulado con BS3. La figura muestra un gel de SDS-poliacrilamida teñido con plata de eluatos de reticulado la perlas. NIb fue identificado por espectrometría de masas como una pareja de unión de VPg-Pro utilizando una parte alícuota de la misma muestra. SIII-etiquetados PVA VPg-Pro (MW prevista: 52 kDa) y punta (MW prevista: 59 kDa) migran como una doble banda, indicado por una punta de flecha. Control negativo (carril central) corresponde a la purificación a partir de células infectadas con el virus que expresa VPg-Pro sin la SIII-etiqueta.
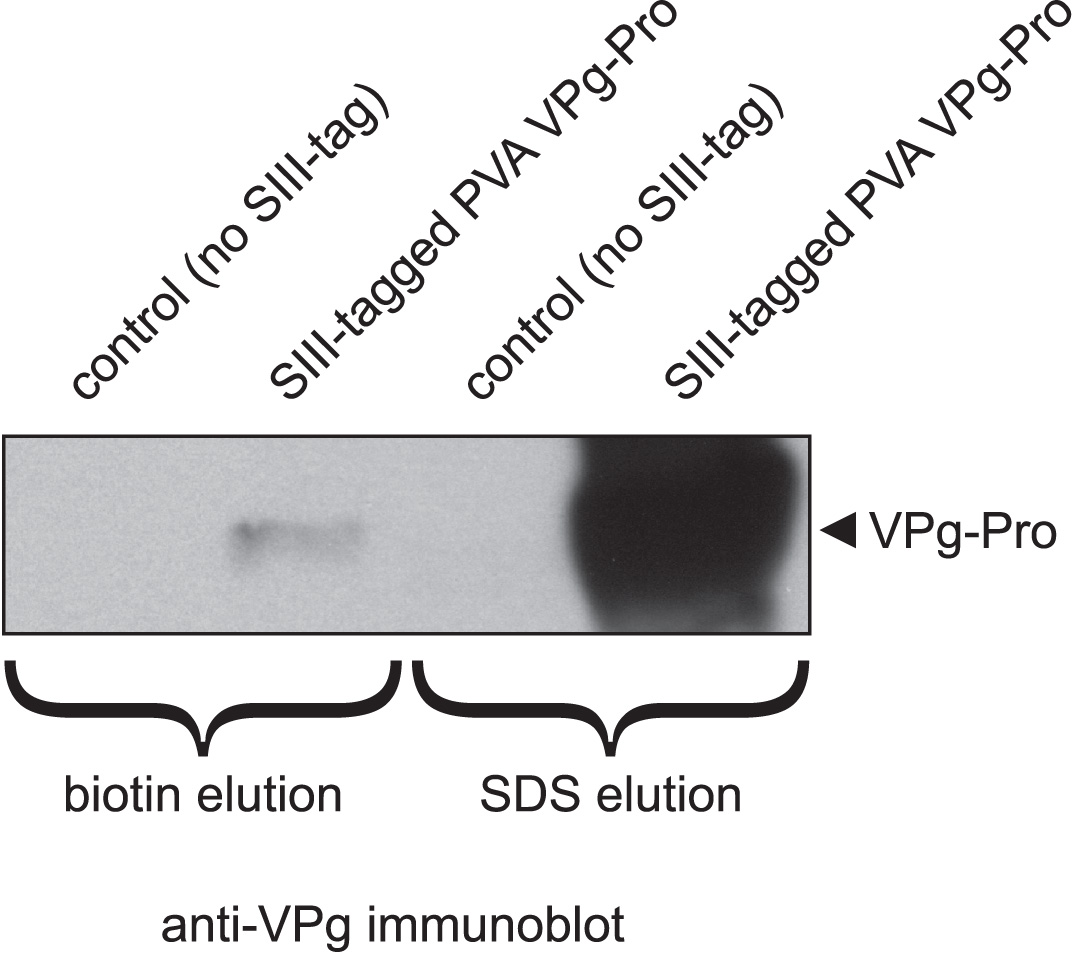
Figura 3. Elución de biotina-etiquetados SIII PVA VPg-Pro a partir de resina de polimetacrilato Strep-Tactin seincompleta en comparación con elución con 1% de SDS. La figura muestra una inmunotransferencia anti-VPg de eluatos de perlas de Strep-Tactin. Las perlas se eluyeron ya sea con biotina 15 mM o con 1% de SDS. La diferencia en la intensidad de la señal refleja diferentes rendimientos de proteína obtenidos con las dos técnicas de elución. Los controles negativos corresponden a purificaciones a partir de células infectadas con el virus que expresa VPg-Pro sin la SIII-etiqueta.

Figura 4. Covalente entrecruzamiento con BS3 previene la liberación de Strep-Tactin de la resina durante la SDS elución. La figura muestra un gel teñido con plata de SDS eluidos de-no reticulado (carril izquierdo) y reticulado BS3 (carril derecho) de resina de polimetacrilato Strep-Tactin. Cabe destacar la presencia de Strep-Tactin lanzado en el carril de la izquierda, pero su ausencia en el carril de la derecha.

Figura 5. Estructura química de bis (sulfosuccinimidil) suberato (BS3) y su reacción de reticulación con los grupos amino épsilon de residuos de lisina en Strep-Tactin.

Figura 6. Purificación de la proteína marcada con doble Strep verde fluorescente (GFP) en la no-reticulado y BS3-reticulado resina de polimetacrilato Strep-Tactin. Dos cantidades iguales de células embrionarias humanas de riñón 293 que expresan GFP-etiquetados de doble Strep se lisaron y se procesan de manera similar utilizando el protocolo anterior, excepto que en un caso la resina se ha reticulado con BS3 y en el otro BS3 ha seres sustituido con agua en la reacción de reticulación. La figura muestra un gel de SDS-poliacrilamida teñido con plata e inmunotransferencia anti-GFP de proteínas purificadas. Tenga en cuenta que una cierta disminución en el rendimiento de proteína es inherente a los métodos de purificación que implican la reticulación química, pero esto se compensa por la mayor pureza de la muestra debido a la ausencia de Autorizaciones de Strep-Tactin.
Discusión
El protocolo anterior se puede utilizar para purificar cualquier proteína cebo de etiquetado de doble Strep de interés y sus complejos asociados en cualquier tampón adecuado que no contiene biotina o desnaturalizantes fuertes. En la versión actual del protocolo, de unión y lavado se llevan a cabo bajo condiciones relativamente estrictas en presencia de altas concentraciones de sal y detergente no iónico. Aunque esto da como resultado en menos de fondo, los complejos de proteínas frágiles pueden disociarse bajo estas condiciones. Para preservar tales complejos de menor afinidad, la concentración de sal se puede bajar y un detergente no iónico puede ser reducido o omitido de unión y de lavado tampones.
La principal ventaja del sistema de doble-Strep-tag es la alta afinidad de la pequeña (3kDa) etiqueta tándem para estreptavidina ingeniería (Strep-Tactin) 1, 2, permitiendo eficiente purificación de una etapa de proteínas diana y sus complejos incluso en lotes modo. Debido a la fuerte unión de doble-Strep-taga Strep-Tactin, la resina se puede lavar en condiciones moderadamente rigurosas, tales como alta detergente y / o concentraciones de sal, lo que resulta en una mayor pureza proteína diana. Sin embargo, tales fuerte unión también tiene sus inconvenientes. Incluso en el caso de las proteínas con un solo marcador Strep II (secuencia de aminoácidos WSHPQFEK), elución competitiva de la resina puede ser a veces difícil. Por ejemplo, la elución de un Strep (II)-etiquetados proteína quinasa, NtCDPK2, de la resina de polimetacrilato de Strep-Tactin con 10 mM de destiobiotina resultó ser infructuoso, lo que requiere la desnaturalización de la elución con tampón de muestra SDS 6. El problema se resolvió sólo después de destiobiotina fue reemplazado con biotina más fuerte que compiten (10 mM) y la elución se llevó a cabo durante 5 minutos con agitación vigorosa 7. En el caso de la doble-Strep-tag, que tiene mayor afinidad por Strep-Tactin, elución proteína diana puede ser incompleta incluso a concentraciones de biotina de hasta 15 mM. Como se muestra en la Figura 3,sólo una pequeña fracción de etiquetado-SIII de PVA VPg-Pro podría ser eluida de la resina de Strep-Tactin con biotina 15 mM en comparación con la cantidad eluida con 1% de SDS. Es importante tener en cuenta que la eficiencia de elución competitiva probable que también depende de la naturaleza de la proteína se eluyó o complejo de proteínas. Las proteínas más grandes y más hidrófobos pueden obstaculizar estéricamente la accesibilidad de la cavidad de unión a biotina en Strep-Tactin, haciendo así elución competitiva menos eficiente. Los inconvenientes anteriores se evitan mediante el uso de elución con SDS, pero tales elución tiene sus propios problemas, que gira principalmente alrededor de el hecho de que la exposición a detergentes conduce a la disociación de los tetrámeros de Strep-Tactin acoplados a resina y contaminación de la muestra con lanzado Strep-Tactin 8. Esto se demuestra en la Figura 4 (carril de la izquierda), que muestra una cantidad considerable de Strep-Tactin liberado después de la incubación de la resina de Strep-Tactin en tampón de elución que contiene 1% de SDS. Tal contaminación es inaceptable sila muestra debe analizarse además por LC-MS/MS.
Una solución eficaz al problema de la contaminación de arriba es la estabilización de la acoplado a la resina de Strep-Tactin tetrámero 9 por reticulación covalente. Para este propósito, se empleó un, no escindible, homobifuncional reticulante BS3-amina reactivo soluble en agua con una historia de la utilización con éxito en la estabilización de estructuras de proteínas 10-13. La figura 5 muestra la fórmula química de BS3 y su reticulación reacción con grupos amino libres épsilon de lisina en Strep-Tactin. Cuando el Strep-Tactin tetrámero acoplado a la resina se estabilizó por reticulación con BS3, el problema de la contaminación de la muestra con lanzado Strep-Tactin prácticamente se eliminó (Figura 4, carril de la derecha). Debido a que el entrecruzamiento con BS3 no causa extensa agregación de proteínas 14, que no reduce la accesibilidad de la cavidad de unión de estreptavidina biotina, por lo que laresina reticulado adecuado para la purificación de proteínas de fusión etiquetadas-Strep. Cabe señalar, sin embargo, que la reticulación química de las proteínas con actividad biológica (anticuerpos, etc) estabiliza ambas conformaciones de proteínas activas e inactivas, que conduce a alguna pérdida de actividad. Por ejemplo, el método de inmunoprecipitación bien establecida usando anticuerpos BS3-reticulado produce un objetivo más bajo rendimiento de proteína en comparación con la obtenida con anticuerpos no reticulados 15. Sin embargo, una cierta pérdida de actividad biológica se considera que es un compromiso aceptable para la pureza de la muestra mejorada. Se observaron resultados similares con la resina de Strep-Tactin BS3-reticulada. El rendimiento de proteína algo más bajo obtenido con la resina reticulada fue más que compensado por el muy mejorado pureza de la muestra (Figura 6).
Otra aplicación potencial del método propuesto es en la purificación de proteínas de membrana Strep-etiquetados solubilized con detergentes. Los detergentes son esenciales para mantener dichas proteínas hidrófobas y sus complejos asociados en la solución, pero su presencia puede dar lugar a la contaminación de la muestra con Strep-Tactin liberado de la resina de afinidad. Un buen ejemplo de esto es la purificación de una proteína de membrana (II) etiquetados-Strep A NTT1 sobre perlas de Strep-Tactin para el propósito de cristalización de proteínas 8. La presencia de la laurylamidodimethylpropylaminoxide detergente (Lapão) en el tampón de elución dio lugar a la liberación de Strep-Tactin de las perlas y el crecimiento posterior de no deseados cristales de Strep-Tactin 8. Estabilización del acoplado a la resina Strep-Tactin tetrámero por reticulación covalente puede proporcionar un medio eficaz para superar este problema.
En resumen, reticulación química se ha empleado con éxito para superar las limitaciones asociadas con la desnaturalización de elución de la resina de Strep-Tactin. Este enfoque ayudó a conseguir una buena proteína diana rerecuperación sin introducir contaminación de la muestra. El método propuesto por lo tanto se puede utilizar para identificar parejas de unión específicas de las proteínas de cebo de etiquetado de doble Strep por espectrometría de masas. Además, el método se puede aplicar para aislar y caracterizar los complejos de nucleoproteína, incluidos los reversiblemente reticulado con formaldehído, así como proteínas de membrana solubilizadas con detergentes. Finalmente, reticulación química no cambia significativamente las propiedades físicas de la resina de Strep-Tactin, haciendo que la resina reticulada potencialmente adecuado para aplicaciones de alto rendimiento.
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
Agradecemos el apoyo técnico de Sini Miettinen, Minna Pöllänen y Taru Rautavesi. Agradecemos Helka Nurkkala para proporcionar células HEK 293 que expresan de etiquetado doble Strep GFP y Pekka Evijärvi para el suministro de equipos de grabación de sonido. Este trabajo fue financiado por la Academia de Finlandia, conceder los números 138329, 134684 y 258978.
Materiales
| Name | Company | Catalog Number | Comments |
| Bis(sulfosuccinimidyl) suberate (BS3), No-Weigh format, 8 x 2 mg | Pierce/Thermo Scientific | 21585 | www.fishersci.com CAUTION: Hazardous substance. Causes serious respiratory, skin and eye irritation. Wear protective gloves and eye protection. |
| Strep-Tactin MacroPrep resin (50% suspension) | IBA | 2-1505 | www.iba-lifesciences.com |
| Spin-X centrifuge tube filter, cellulose acetate membrane, pore size 0.45 μm, non-sterile | Costar (Corning) | 8163 | www.corning.com/lifesciences/ |
| Dolphin-nose tubes | Costar (Corning) | 3213 | www.corning.com/lifesciences/ |
| Avidin | IBA | 2-0204 | www.iba-lifesciences.com |
Referencias
- Voss, S., Skerra, A. Mutagenesis of a flexible loop in streptavidin leads to higher affinity for the Strep-tag II peptide and improved performance in recombinant protein purification. Protein Eng. 10 (8), 975-982 (1997).
- Schmidt, T. G., Skerra, A. The Strep-tag system for one-step purification and high-affinity detection or capturing of proteins. Nat. Protoc. 2 (6), 1528-1535 (2007).
- Junttila, M. R., Saarinen, S., Schmidt, T., Kast, J., Westermarck, J. Single-step Strep-tag purification for the isolation and identification of protein complexes from mammalian cells. Proteomics. 5 (5), 1199-1203 (2005).
- Hafren, A., Hofius, D., Ronnholm, G., Sonnewald, U., Makinen, K. HSP70 and its cochaperone CPIP promote potyvirus infection in Nicotiana benthamiana by regulating viral coat protein functions. Plant Cell. 22 (2), 523-535 (2010).
- Jaskiewicz, M., Peterhansel, C., Conrath, U. Detection of histone modifications in plant leaves. J. Vis. Exp. (55), (2011).
- Witte, C. P., Noel, L. D., Gielbert, J., Parker, J. E., Romeis, T. Rapid one-step protein purification from plant material using the eight-amino acid StrepII epitope. Plant Mol. Biol. 55 (1), 135-147 (2004).
- Werner, A. K., Sparkes, I. A., Romeis, T., Witte, C. P. Identification, biochemical characterization, and subcellular localization of allantoate amidohydrolases from Arabidopsis and soybean. Plant Physiol. 146 (2), 418-430 (2008).
- Panwar, P., Deniaud, A., Pebay-Peyroula, E. Contamination from an affinity column: an encounter with a new villain in the world of membrane-protein crystallization. Acta Crystallogr. D. Biol. Crystallogr. 68 (10), 1272-1277 (2012).
- Hendrickson, W. A., et al. Crystal structure of core streptavidin determined from multiwavelength anomalous diffraction of synchrotron radiation. Proc. Natl. Acad. Sci. U. S. A. 86 (7), 2190-2194 (1989).
- Bernot, K. M., Lee, C. H., Coulombe, P. A. A small surface hydrophobic stripe in the coiled-coil domain of type I keratins mediates tetramer stability. J. Cell Biol. 168 (6), 965-974 (2005).
- Singh, I., et al. Solution structure of human von Willebrand factor studied using small angle neutron scattering. J. Biol. Chem. 281 (50), 38266-38275 (2006).
- Weldon, W. C., et al. Enhanced immunogenicity of stabilized trimeric soluble influenza hemagglutinin. PLoS One. 5 (9), (2010).
- Wang, W., Barger, S. W. Roles of quaternary structure and cysteine residues in the activity of human serine racemase. BMC Biochem. 12, 63 (2011).
- Rappsilber, J. The beginning of a beautiful friendship: cross-linking/mass spectrometry and modelling of proteins and multi-protein complexes. J. Struct. Biol. 173 (3), 530-540 (2011).
- Sousa, M. M., Steen, K. W., Hagen, L., Slupphaug, G. Antibody cross-linking and target elution protocols used for immunoprecipitation significantly modulate signal-to noise ratio in downstream 2D-PAGE analysis. Proteome Sci. 9, 45 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados