Method Article
Avidez detección basada en la interacción extracelular (AVEXIS) para la detección escalable de extracelular de baja afinidad interacciones ligando-receptor
En este artículo
Resumen
AVEXIS es una proteína de alto rendimiento ensayo de la interacción desarrollada para detectar sistemáticamente nuevos ligando extracelular del receptor de los pares que intervienen en los procesos de reconocimiento celular. Se ha diseñado específicamente para detectar las interacciones proteína transitorios que son difíciles de identificar el uso de otros métodos de alto rendimiento.
Resumen
Extracelular de la proteína: proteína interacciones entre las proteínas secretadas o de membrana atados son fundamentales para la cohesión de iniciar la comunicación intercelular y la garantía dentro de los organismos multicelulares. Las proteínas predicho para formar interacciones extracelulares son codificadas por aproximadamente una cuarta parte de los genes humanos 1, pero a pesar de su importancia y la abundancia, la mayoría de estas proteínas han documentado ninguna pareja de unión. Principalmente, esto es debido a su insolubilidad bioquímicos: proteínas de membrana incrustados son difíciles de solubilizar en su conformación nativa y contienen modificaciones post-estructuralmente importantes. Además, las afinidades de interacción entre las proteínas de los receptores se caracterizan a menudo por fuerzas de interacción extremadamente bajas (vida media de <1 segundo) que impiden su detección con muchos de uso común los métodos de alto rendimiento 2.
A continuación, describimos un ensayo, AVEXIS (avidez basada extracelularlar la interacción de la pantalla) que supera estos retos técnicos que permitan la detección de interacciones proteína muy débiles (t 1/2 ≤ 0,1 segundos) con una baja tasa de falsos positivos 3. El ensayo se implementa normalmente en un formato de alto rendimiento para permitir la detección sistemática de muchos miles de interacciones en un formato de placa de microtitulación conveniente (Fig. 1). Se basa en la producción de solubles bibliotecas de proteínas recombinantes que contienen los fragmentos ectodominio de receptores de la superficie celular o proteínas secretadas dentro del cual la pantalla para interacciones, por lo tanto, este enfoque es adecuado para el tipo I, tipo II, IPM-enlazadas receptores de la superficie de células y secretada proteínas, pero no para las proteínas de membrana multipaso como los canales iónicos o transportadores.
Las bibliotecas de proteínas recombinantes se producen mediante un sistema de expresión de mamíferos cómoda y de alto nivel-4, para asegurar que las importantes modificaciones postraduccionales como glycosylabonos ción y el disulfuro se añaden. Proteínas recombinantes expresadas son secretadas en el medio y se produce en dos formas: un cebo con biotina que se puede capturar en una fase sólida recubiertas de estreptavidina adecuado para el cribado, y una enzima-etiquetado pentamerised presa (β-lactamasa). El cebo y proteínas presa se presentan entre sí en una forma binaria para detectar la interacción directa entre ellos, de forma similar a un ELISA convencional (fig. 1). El pentamerisation de las proteínas en la presa se logra a través de una secuencia de péptido de la proteína matriz oligomérica de cartílago (COMP) y aumenta la concentración local de los ectodominios proporcionando así beneficios significativos avidez para permitir interacciones incluso muy transitorios para ser detectado. Por la normalización de las actividades tanto de la carnada y la presa a niveles predeterminados anteriores a la selección, hemos demostrado que las interacciones que tienen monoméricas una vida media de 0,1 segundos se pueden detectar con bajas tasas de falsos positivos 3.
Protocolo
1. Compilación de una biblioteca de cebo y presa vectores plásmidos de expresión
- Compile una lista de receptores de superficie celular y las proteínas secretadas de interés para ser seleccionados para las interacciones y recuperar sus secuencias de una base de datos de secuencias de proteínas adecuada. La mayoría de las proteínas que contienen un péptido secretor N-terminal de señal son adecuados (que incluyen el tipo I, GPI-enlazadas y las proteínas secretadas). Típicamente, una familia de proteínas (por ejemplo superfamilia de inmunoglobulinas o todos los receptores restringidas a un tipo específico de células tales como los eritrocitos) serían seleccionados.
- Determinar la extensión de las regiones extracelulares mediante la identificación de la ubicación del péptido señal y la región transmembrana utilizando la función de la proteína software de predicción. Por lo general utilizan v3.0 SignalP 5 y 6 TMHMM v2.0.
- Conjuntos de diseño de la cartilla que amplifican el fragmento ectodominio entero para cada receptor. Tenemos un paquete de cebo y vectores de presa que son adecuados para este fin que son unaisponible de Addgene ( http://www.addgene.org/~~V ). Diseñar el primer sentido alrededor del inicio metionina para incluir un sitio de enzima de restricción NotI y una secuencia de Kozak óptima, que es seguido por 25 pares de bases de coincidencia exacta de genes específicos de secuencia (Fig. 2A).
- Diseñar el cebador antisentido para truncar tipo I proteínas receptoras justo antes de la región de transmembrana predicha para expresar el ectodominio entero como una proteína recombinante soluble. Incluir un sitio de restricción AscI en este cebador que luego deberá traducirse en el marco de una rata C-terminal Cd4d3 4 etiqueta proteína 7 (fig. 2A).
- Amplificar los fragmentos de ectodominio de todos los genes con estos cebadores y clonarlos en el cebo o el vector de la presa. A veces resulta útil a la primera a clonar en vectores lanzadera antes de subclonación en un vector de expresión de mamíferos apropiada, se utiliza un derivado del vector pTT3 3,4. Tanto el cebo y los vectores de la presa conten C-terminal de la proteína etiquetas como sigue (Fig. 2B).
- Los cebos: Una etiqueta que contiene dominios 3 y 4 de los CD4 de rata (Cd4d3 +4) de proteínas seguido por una secuencia peptídica que es un sustrato para la enzima de E. coli BirA y por lo tanto se puede monobiotinylated en un residuo de lisina específico (Fig. 2B) . El Cd4d3 4 etiqueta es reconocida por el anticuerpo monoclonal OX68 y se utiliza para cuantificar la expresión de las proteínas de cebo por ELISA. Las proteínas son, por tanto cebo monomérica y monobiotinylated. Otros vectores de la proteína cebo, donde se sustituye el péptido biotinylatable por una etiqueta 6-su para la purificación están disponibles.
- Presas: Las presas también contienen una rata Cd4d3 4 etiqueta seguido por una secuencia de péptido de la proteína de rata cartílago matriz oligomérica (COMP) y una enzima C-terminal de β-lactamasa. El péptido COMP asegura que las formas de presas pentámeros expresó una vez (Fig. 2B).
2. Presa y Bait-expresión de la proteína
Usamosun práctico sistema de alto nivel de expresión mediante el cultivo de la suspensión de la línea celular HEK293E 4 para producir nuestras bibliotecas de proteínas, pero cualquier sistema de expresión de mamíferos debe ser adecuado. Una alternativa disponible en el mercado es el sistema de Freestyle. Las células se cultivaron rutinariamente en una plataforma de agitación (125 rpm) a 37 ° C, 5% de CO 2 a 70% de humedad relativa. Proteínas de control tanto positivos como negativos también debe ser expresado. La rata Cd4d3 cuatro etiquetas de sólo fragmento está disponible como un cebo y la presa y son adecuadas para los controles negativos. Del mismo modo, los vectores de cebo y presa que codifican un control positivo se encuentran disponibles (Addgene), que suelen utilizar la rata-CD200 CD200R la interacción 8.
- Semillas de las células HEK293E el día antes de la transfección a una densidad de 2,5 x 10 5 células ml -1. En forma rutinaria cultivo de las células en volúmenes de 50 ml en Freestyle293 medios de comunicación tras las recomendaciones del fabricante. Para garantizar la biotinilación eficiente, completar tél medio de cultivo celular utilizada para producir proteínas de cebo con D-biotina a una concentración final de 100 mM. Medio utilizado para expresar las proteínas presa no necesita ser suplementado con D-biotina adicional.
- El día siguiente, las células usando un reactivo de transfección de las transfecciones adecuado (por ejemplo 293fectin). Utilizar 25 g del cebo o presa construcciones de plásmidos para transfectar un cultivo de 50 ml. Para producir los cebos biotinilados, co-transfectar el plásmido que codifica el cebo construir con un plásmido que codifica una forma secretada de la proteína de E. coli-biotina-ligasa ( BirA) que está disponible de Addgene. Co-transfección de plásmidos de cebo en una proporción de 10:1 (2,5 g) del plásmido que codifica la proteína secretada Bira.
- Cultivos de cosecha cinco días después de la transfección por primera granulación de las células por centrifugación a 3000 x g durante 20 minutos, seguido por filtración del sobrenadante a través de un filtro de 0,22 mM.
Consejo [: cebo y presa de proteínass deben ser almacenados sin diluir a 4 ° C y, como norma general se debe utilizar dentro de un año. Añadir bacteriostáticos tales como 50 mg / ml de polimixina B sulfato o 10 mM de NaN 3 para evitar la contaminación bacteriana de los sobrenadantes.]
3. La normalización de la carnada y las muestras de presas
Una de las características clave del ensayo AVEXIS es que debido a las proteínas recombinantes son secretados pueden ser diluida o concentrada (normalizado) dentro de las actividades de umbral establecidos antes de ser utilizado en el ensayo. Hemos encontrado que los niveles en los que las proteínas recombinantes se expresan abarcan una amplia gama - hasta 4 órdenes de magnitud - y por lo que el paso de normalización reduce significativamente el número de falsos positivos durante las pantallas.
3.1 Cebo de normalización
Nota: no conjugado de D-biotina debe ser retirado de los medios (típicamente por diálisis), ya que competirá con la biotina bait proteína para unirse a la estreptavidina sitios de unión.
- Traslado sobrenadantes de cebo en tubos de diálisis, la etiqueta, enganchada de manera segura y dializa extensamente frente a un tampón adecuado tal como PBS para eliminar la biotina libre.
[Consejo: Se ha encontrado que la diálisis se logra normalmente suficiente para 6 x 50 mL sobrenadantes de cebo contra un volumen de 4,5 l de PBS con 4 cambios durante un período de 24 horas. Diálisis insuficiente puede ser observado por una inhibición de la proteína de unión a diluciones bajas durante la etapa de normalización cebo (fig. 3A).
- Realizar un ELISA para determinar los niveles relativos a la que las proteínas se expresan de cebo. Preparar una serie de dilución de las proteínas biotiniladas cebo y la captura de ellos en una placa de microtitulación revestidos con estreptavidina. Use la rata Cd4d3 cuatro etiqueta para detectar y cuantificar las proteínas cebo capturados utilizando un anticuerpo monoclonal (OX68). Concentrar o diluir las proteínas cebo a la cantidad mínima necesaria para saturar todo el bioten sitios de unión en el pocillo de una placa de estreptavidina-revestido.
- Bloque los pocillos de una placa de estreptavidina-revestido con tampón fosfato saline/0.1% de Tween-20 (PBST) / 2% de seroalbúmina bovina (BSA) durante 15 minutos.
- En una placa separada, hacer una serie de dilución (cuatro diluciones 1:3 son generalmente suficientes) de las proteínas de cebo en PBST BSA / 2% y la transferencia a los pocillos de una placa de estreptavidina-revestido. Incubar durante 1 hora a temperatura ambiente.
- Lave 3 veces en PBST. Añadir 100 l de 2 mg / ml de ratón anti-rata Cd4d3 4 IgG (OX68) incubar durante 1 hora a temperatura ambiente.
- Lave 3 veces en PBST. Añadir 100 l de anti-ratón de fosfatasa alcalina en dilución 1:5000 en PBST / 2% de BSA y se incuba durante 1 hora a temperatura ambiente.
- Lavar 3x en PBST y realizar un lavado final en PBS para eliminar cualquier detergente residual. Añadir 100 l de 1 mg / ml de sustrato 104 en tampón de dietanolamina (10% de dietanolamina (v / v), 0,5 mM MgCl 2, 10 mM de NaN 3, pH9.8, se almacena protegido de la luz a 4° C).
- Después de una hora, la absorbancia medida a 405 nm. Representativos resultados normalización de cebo se muestran en la Figura 3A.
- Cebo proteínas que se expresan en niveles que son insuficientes para saturar todos los sitios de unión de biotina-estreptavidina una placa recubierta, cuando se utiliza sin diluir, se debe concentró usando concentradores Vivaspin (Vivascience, 10kDa MWCO). Como una guía, estimamos que ~ 5 g de una proteína cebo típico (~ 80 kDa) es suficiente para saturar una placa de 96 pocillos.
- Proteínas cebo expresadas a niveles altos puede ser diluida en BSA PBST / 2% a una concentración suficiente para saturar la placa de estreptavidina-revestido. La actividad de los cebos, deberá comprobarse después de la dilución o concentración para garantizar que se saturará todos los sitios de unión de biotina en la placa de estreptavidina-revestido.
3.2 Presa de normalización
- Cuantificar los niveles relativos a la que las proteínas de la presa se expresan utilizando el β-lactamasala actividad enzimática.
- En primer lugar, hacer una serie de diluciones del sobrenadante que contiene las proteínas de presa en PBST / 2% de tampón BSA. Entonces se añaden 20 l de las diluciones de 60 l de unos 242 micras (0,125 mg / ml) de solución de nitrocefina (Calbiochem).
- Inmediatamente transferir la placa a un lector de placas de microtitulación que toma una medida de absorbancia a 485 nm por minuto durante 20 minutos a temperatura ambiente. Representativos resultados normalización presa se muestran en la Figura 3B.
- Concentrar las muestras utilizando concentradores centrífugos o diluirlas en PBST / BSA al 2% para llegar a un umbral de actividad de ~ 2 nmol min -1 volumen de negocios de nitrocefina. Prácticamente, esto se logra si toda la nitrocefina se hidroliza dentro ~ 7 minutos.
4. AVEXIS procedimiento de detección
- Lave una placa revestida con estreptavidina en PBST.
- Añadir 100 l de PBST / 2% de BSA a cada pocillo y se incuba durante 30 minutos para bloquear los sitios de proteínas espurios de unión dentro de cada pocillo.
[Consejo: para eliminar el tampón de lavado después de las etapas de lavado, la placa se golpea ligeramente - con fuerza - en toallas de papel]
- Retire la solución de bloqueo con un movimiento inteligente de la placa sobre una pileta y añadir 100 ml normalizado cebo monomérico con biotina a los pocillos apropiados y se incuba durante 1 hora a temperatura ambiente.
- Retirar las muestras de cebo de la placa por volviendo hacia arriba de la placa y elegantemente agitando sobre un lavabo, y lavar 3 veces con PBST.
- Añadir 100 l de la presa normalizada pentamerized construir a cada pocillo y se incuba a temperatura ambiente durante 1 hora, lavar como en el paso 4,4 y realizar un lavado final con PBS solo.
- Añadir 60 l de 242 mM solución de nitrocefina (disolver 5 mg de nitrocefina en 500 l de DMSO, mezclar bien y diluir aún más la nitrocefina en 40 ml de PBS y se pasan a través de un filtro de 0,22 antes de añadir a los platos) y se incuban a temperatura ambiente . Las interacciones positivas se pueden observarpor el ojo desde la nitrocefina amarillo se hidroliza a un color rojo. Cuantificar mediante la lectura de la absorbancia a 485 nm utilizando un lector de placas. Un plato típico y la cuantificación se muestra en la fig. 4.
[Consejo: Las interacciones positivas se observan generalmente dentro de 2 horas a temperatura ambiente aunque las placas deben colocarse a 4 ° C durante 16 horas para asegurar que todas las interacciones potenciales se detectan. También hemos encontrado que nitrocefina precipitado y las imperfecciones en el plástico de la placa de microtitulación como arañazos o huellas dactilares en ocasiones puede dar lugar a falsos positivos artefactuales a 485 nm a pesar de no hidrólisis nitrocefina manifiesta. Por tanto, es recomendable tomar fotografías de las placas para grabar visualmente el volumen de negocios nitrocefina en los pozos.]
5. Los resultados representativos
Un ejemplo de una placa de detección representante AVEXIS se muestra en la Figura 4. Uno debe observar la hidrólisis (de amarillo a rojo) del sustrato nitrocefina en elpositiva pocillos de control (tanto la mediada por anticuerpos de control de captura de presas y también la interacción control positivo) dentro de unos diez minutos. Algunas interacciones dentro de la pantalla también se observan dentro de esta escala de tiempo, pero otros pueden tardar más tiempo (algunas horas) en aparecer. Típicamente, un golpe de tasa de alrededor de 0,4-0,6% (una interacción positiva por cada 150-250 interacciones siendo examinados) es típica, dependiendo de las bibliotecas de proteínas siendo examinados.
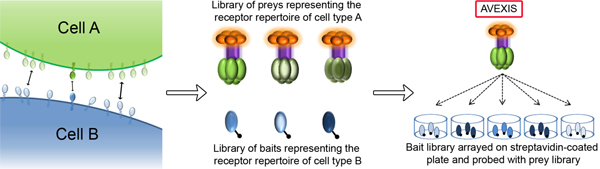
Figura 1. La identificación de las interacciones ligando-receptor que median procesos celulares de reconocimiento utilizando AVEXIS. El esquema muestra dos células interactúan entre sí (A y B) el intercambio de información a través de las interacciones realizadas entre las proteínas del receptor de membrana integradas. AVEXIS es un ensayo de proteínas interacción escalable específicamente diseñado para identificar nuevos pares ligando-receptor que se caracterizan por fuerzas de interacción muy débiles. Recombinante paraluble bibliotecas de proteínas que representan a la totalidad de la ectodominio repertorio de células del receptor de la superficie de las células, se compilan ya sea como pentaméricas presas β-lactamasa de etiquetado (tipo de célula A) o monómeros de cebos biotina-etiquetados (para el tipo de células B). El pentamerisation de la presa aumenta la concentración local de los ectodominios para efectuar una ganancia avidez general por lo que las interacciones incluso muy transitorios pueden ser detectados. La biblioteca de cebo se vistió de streptavidina revestida de placas y sistemáticamente investigado por las interacciones con las proteínas de la presa. Después de un breve lavado, las presas capturadas son detectados gracias a la actividad β-lactamasa asociado a la presa.

Figura 2. El cebo y el diseño de construcción de la presa. (A) La rata CD200 proteína del receptor se proporciona como un ejemplo de cómo los ectodominios enteras de proteínas de las células receptoras de la superficie se determinan y cebadores están diseñados para hacer tanto ceboy construcciones de presas de expresión. La secuencia de la proteína traducida cDNA y se muestran a partir del péptido señal N-terminal a la membrana que abarca la región transmembrana de rata CD200. Un primer sentido está diseñado para incluir un 5 'de restricción NotI sitio de la enzima y la secuencia de Kozak óptimo seguido de 25 bases de la secuencia coincidencia exacta con la secuencia de cDNA CD200. El cebador antisentido contiene un sitio AscI y 25 bases de secuencia coincidencia exacta situado inmediatamente aguas arriba del residuo ectodominio truncamiento seleccionado. Para mantener el ectodominio en marco con la rata Cd4d3 4 etiqueta, el sitio AscI debe traducir como Gly-Ala-Pro. (B) representaciones esquemáticas de la carnada y las construcciones de presas de expresión. Cada uno contiene ectodominios receptores flanqueados por sitios NotI y ascos y una etiqueta Cd4d3 4. Los cebos contienen una secuencia de péptido C-terminal que puede ser específicamente monobiotinylated por cotransfección con un plásmido que expresa una enzima secretada Bira. Las presas son seguidos por la secuencia pentamerisationa partir de la proteína matriz del cartílago oligomérica (COMP) y la enzima β-lactamasa.
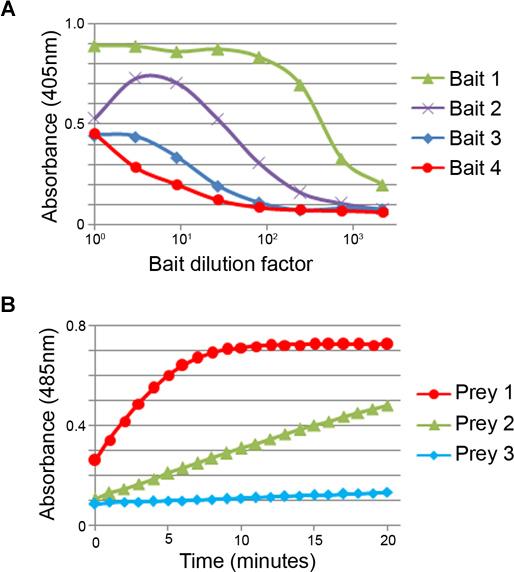
Figura 3. La normalización de la carnada y las proteínas de presa. (A) los ejemplos representativos de la normalización de cebo. Una serie de dilución de cuatro diferentes sobrenadantes dializados cebo se detectaron por ELISA utilizando un anticuerpo anti-CD4 etiqueta monoclonales. Los cuatro cebos se expresan en diferentes niveles 1> 2> 3> 4. Los cebos deben ser normalizados a un nivel de umbral que satura los sitios de unión de biotina en cada pocillo. Cebos altamente expresadas se puede diluir a conservar la proteína (por ejemplo cebo 1 podría ser diluido ~ 1:100), y se concentró cebos pobremente expresadas. Lecturas disminuyó a bajas diluciones a veces se observan en algunos cebos (cebo por ejemplo 2) que es atribuible a la presencia de D-biotina no conjugado debido a diálisis incompleta. (B) Los niveles en los que se aprovechan de las proteínas son exprensada se cuantifica mediante la velocidad de hidrólisis de un sustrato β-lactamasa, nitrocefina. En el ejemplo mostrado, las tres presas se expresan en niveles diferentes: 1 presa> 2> 3. Presas debe ser normalizado a una actividad de ~ 2 -1 nmol min, lo que corresponde para completar la hidrólisis de 14,5 nmol nitrocefina en ~ 7 minutos (por ejemplo, presas 1). Las otras presas en este ejemplo debe concentrarse antes de su uso en el cribado.
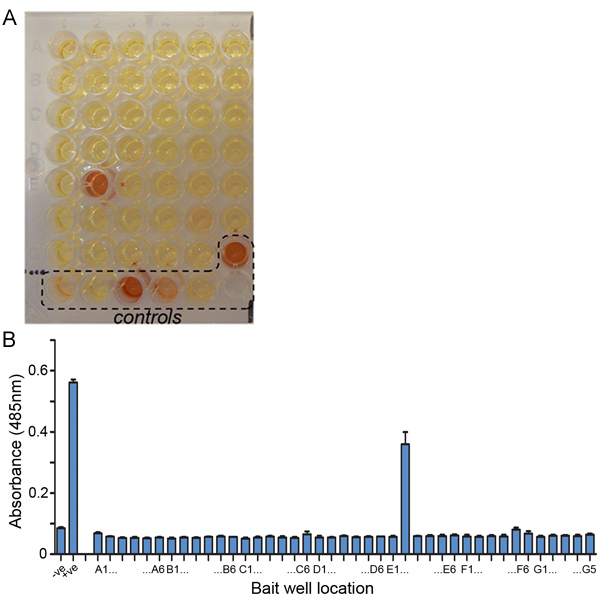
Figura 4. Representante AVEXIS resultados de la prueba. (A) Una fotografía de una placa representativa de detección AVEXIS. Una presa de proteínas se probaron contra 41 diferentes cebos y una interacción positiva se detecta en E2 también. Los controles utilizados en las placas de evaluación incluyen: un biotina anticuerpo anti-CD4 para capturar toda la presa de proteína añadido al pozo (G6), la interacción de control: la rata CD200 (cebo)-CD200R (presa), con la presa CD200R utilizado en la dilución del umbral (H3), 1:10 (H4) y 1:100 (H5). Los controles negativos fueron: a Cd4d3 4 de cebo determinada con la presa de proteínas que están siendo revisadas (H1) y la presa CD200R (H2). (B) Cuantificación de los valores de absorbancia de la misma pantalla que realizaron por triplicado. Los puntos de datos son la media ± desviación estándar.
Discusión
Cuando las células vivas se observan en vivo, sus membranas plasmáticas presentan comportamientos muy dinámicos: la ampliación y procesos de retracción de la membrana como se ponen en contacto y comunicarse con sus 2 vecinos. Las interacciones físicas entre las proteínas receptoras incrustados membrana que median muchos de estos contactos han evolucionado para ser extremadamente débil para permitir este comportamiento muy móviles. Se ha estimado que las fortalezas de monómeros de interacción tan débiles como 50 micras, podría ser suficiente para impulsar las interacciones espontáneas en las densidades de los receptores fisiológicos 9 lo que hace que la detección de nuevas interacciones extremadamente difíciles 2,10. El método descrito aquí AVEXIS proporciona un método sensible para la detección de la proteína extracelular transitoria: las interacciones de proteínas que pueden ser implementadas de una manera escalable con una baja tasa de falsos positivos. Mientras que otros métodos escalables se han desarrollado para detectar las interacciones extracelulares 11-15,Una ventaja de AVEXIS es que los parámetros experimentales, como el cebo y las actividades de las presas han sido cuantificados para detectar hasta el más débil de las interacciones (t 1/2 ≤ 0,1 s), manteniendo una baja tasa de falsos positivos 3. Además, los procedimientos simplificados y la muestra sólida preparación han permitido que este ensayo se realizará a una escala mucho más grande que otros ensayos y se ha descrito recientemente una pantalla de interacción sistemática total de más de 16.500 interacciones potenciales del ~ 16.
El ensayo puede realizarse en cualquier proteína de las que es posible expresar un fragmento soluble de proteína recombinante. Esto incluye tanto las proteínas que contienen un péptido señal N-terminal como el tipo I, IPM-enlazadas y proteínas secretadas. Recientemente, hemos diseñado un conjunto de vectores que ahora nos permiten expresar proteínas de tipo II como cebos 17. El ensayo no es generalmente adecuado para las proteínas de membrana multipaso como transportador de ioness o bombas, ya que es probable que se correctamente plegada fuera del contexto de una membrana plasmática. Hemos aplicado este a una variedad de sistemas diferentes y han expresado con éxito proteínas extracelulares de pez cebra 3,16,18, humano, ratón y P. falciparum para las pantallas de interacción.
En términos de los recursos necesarios para configurar una pantalla AVEXIS, hemos encontrado que un cuello de botella importante es la selección, la construcción, y la secuenciación completa de los plásmidos de expresión. Este aspecto del método puede tomar hasta un año para hacer ~ 100 cebo y construcciones de presas, sin embargo, con el costo de la disminución de la síntesis de genes, que ahora están a favor comercialmente la externalización de este aspecto del proyecto. El sistema de expresión de mamíferos, junto con una gran agitación incubadora de cultivo de tejido (se utiliza una célula Multitron Infors HT) permite que un solo investigador de expresar y normalizar hasta ~ 100 proteínas cebo y presa de un mes. Una vez que las proteínas se producen y se normalizaron, screening para interacciones es rápido con hasta doce placas 96-y siendo convenientemente procesados por día. El único equipo a medida que hemos desarrollado para facilitar la producción de un número tan grande de las proteínas es un aire comprimido impulsado pistón que se utiliza para pasar hasta cinco sobrenadantes a través de un filtro de 0.22μm en paralelo.
La clase principal de las interacciones que pueden perderse si se compara con las interacciones que se esperan de la literatura (falsos negativos) son las interacciones homophilic, aunque algunas de estas interacciones pueden ser detectadas 3. En los casos en que las interacciones esperadas no son detectados, creemos que esto se debe a la formación de las interacciones de las proteínas homophilic presa (presa de "enmascaramiento" efecto) lo que impide que las interacciones con otras proteínas cebo inmovilizados. La frecuencia a la cual se detectan las interacciones depende del contenido de la biblioteca de proteína siendo examinados. Como una guía para el lector, una pantalla de> 30, 000 interacciones dentro de la superfamilia de las inmunoglobulinas pez cebra como resultado 188 positivos - una frecuencia de 0,6% 3,16,18. Una comprobación muy rápido que se puede realizar para verificar cualquier jugado positivos es determinar si la interacción puede ser detectada en ambas presas cebo-orientaciones; es decir, la interacción puede ser detectada misma independientemente de si las proteínas de unión están presentes ya sea como un cebo o una presa. Siempre hemos observado este comportamiento alternativo, la unión ha sido posteriormente demostró ser específico mediante la demostración de la unión directa saturable de proteínas purificadas mediante resonancia de plasmón superficial. Cabe destacar que debido a que aumenta la avidez de unión artificialmente pentamerising las proteínas de la presa, no consideramos adecuado para el análisis cuantitativo comparando fuerzas de interacción. Para obtener los parámetros biofísicos de unión de las interacciones que utilizamos proteínas monoméricas y la resonancia de plasmones de superficie. Finalmente, una vez una interacción ha sido identificado, el hila naturaleza rendimiento gh de la prueba hace que sea relativamente fácil de adaptar el ensayo para la detección de los reactivos, como los anticuerpos o pequeñas moléculas que bloquean la interacción.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue financiado por el Wellcome Trust, el número de subvención [077108] otorgado a GJW.
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre de reactivo | Empresa | Cat. Número | Comentarios |
| Freestyle medios | Invitrogen | 12338018 | |
| Nitrocefina | Calbiochem | 484400 | |
| SA recubiertos con placas de microtitulación | Nunc | 734-1284 | |
| OX68 anticuerpos | AbDSerotec | MCA1022R | |
| Diálisis tubo | Atravesar | PN68100 | |
| Polimixina B | Sigma | P4932 | |
| D-biotina | Sigma | B4639-5G | |
| Sustrato-104 | Sigma | 1040-506 | |
| Vivaspin-20 concentradores centrífugos | Sartorio | VS2002 | |
| Filtros de 0,22 micras | Nalgene | 190-2520 |
Referencias
- Fagerberg, L., Jonasson, K., von Heijne, G. Prediction of the human membrane proteome. Proteomics. 10, 1141-1141 (2010).
- Wright, G. J. Signal initiation in biological systems: the properties and detection of transient extracellular protein interactions. Molecular bioSystems. 5, 1405-1405 (2009).
- Bushell, K. M., Sollner, C., Schuster-Boeckler, B. Large-scale screening for novel low-affinity extracellular protein interactions. Genome research. 18, 622-622 (2008).
- Durocher, Y., Perret, S., Kamen, A. High-level and high-throughput recombinant protein production by transient transfection of suspension-growing human 293-EBNA1 cells. Nucleic acids research. 30, e9-e9 (2002).
- Bendtsen, J. D., Nielsen, H., von Heijne, G. Improved Prediction of Signal Peptides: SignalP 3.0. Journal of molecular biology. 340, 783-783 (2004).
- Krogh, A., Larsson, B., von Heijne, G. Predicting transmembrane protein topology with a hidden markov model: application to complete genomes. Journal of molecular biology. 305, 567-567 (2001).
- Brown, M. H., Barclay, A. N. Expression of immunoglobulin and scavenger receptor superfamily domains as chimeric proteins with domains 3 and 4 of CD4 for ligand analysis. Protein engineering. 7, 515-515 (1994).
- Wright, G. J., Puklavec, M. J., Willis, A. C. Lymphoid/neuronal cell surface OX2 glycoprotein recognizes a novel receptor on macrophages implicated in the control of their function. Immunity. 13, 233-23 (2000).
- Dustin, M. L., Golan, D. E., Zhu, D. M. Low affinity interaction of human or rat T cell adhesion molecule CD2 with its ligand aligns adhering membranes to achieve high physiological affinity. The Journal of biological chemistry. 272, (1997).
- Wright, G. J., Martin, S., Bushell, K. M. High-throughput identification of transient extracellular protein interactions. Biochemical Society transactions. 38, (2010).
- de Wildt, R. M., Tomlinson, I. M., Ong, J. L. Isolation of receptor-ligand pairs by capture of long-lived multivalent interaction complexes. Proceedings of the National Academy of Sciences of the United States of America. 99, (2002).
- Jiang, L., Barclay, A. N. Identification of leucocyte surface protein interactions by high-throughput screening with multivalent reagents. Immunology. 129, 55-55 (2010).
- Urech, D. M., Lichtlen, P., Barberis, A. Cell growth selection system to detect extracellular and transmembrane protein interactions. Biochimica et biophysica acta. 1622, 117-117 (2003).
- Voulgaraki, D., Mitnacht-Kraus, R., Letarte, M. Multivalent recombinant proteins for probing functions of leucocyte surface proteins such as the CD200 receptor. Immunology. 115, 337-337 (2005).
- Wojtowicz, W. M., Wu, W., Andre, I. A vast repertoire of Dscam binding specificities arises from modular interactions of variable Ig domains. Cell. 130, 1134-1134 (2007).
- Martin, S., Sollner, C., Charoensawan, V. Construction of a Large Extracellular Protein Interaction Network and Its Resolution by Spatiotemporal Expression Profiling. Mol. Cell. Proteomics. 9, 2654-2654 (2010).
- Crosnier, C., Bustamante, L. Y., Bartholdson, S. J., Bei, A. K., Theron, M., Uchikawa, M., Mboup, S., Ndir, O., Kwiatkowski, D. P., Duraisingh, M. T., Rayner, J. C., Wright, G. J. Basigin is a receptor essential for erythrocyte invasion by Plasmodium falciparum. Nature. 480, 534-537 (2011).
- Sollner, C., Wright, G. J. A cell surface interaction network of neural leucine-rich repeat receptors. Genome biology. 10, R99-R99 (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados