Method Article
El genotipado de muestras de plantas y animales, sin previa purificación del ADN
En este artículo
Resumen
El enfoque directo de PCR que aquí se presenta facilita la amplificación por PCR directamente a partir de pequeñas cantidades de planta no purificado y el tejido animal.
Resumen
El enfoque de PCR directo facilita la amplificación por PCR directamente a partir de pequeñas cantidades de muestras no purificadas y se demuestra aquí por varias plantas y tejidos animales (Figura 1). PCR directa se basa en el diseño especial Phusion Thermo Scientific y polimerasas Phire de ADN, que incluyen un dominio de ADN de doble hebra de unión que les da características únicas, tales como una alta tolerancia de los inhibidores.
PCR basado en la detección de ADN diana tiene numerosas aplicaciones en la investigación de plantas, incluyendo análisis de plantas genotipo y la verificación de los transgenes. PCR de tejidos de la planta tradicionalmente implica una etapa inicial de aislamiento de ADN, que puede requerir reactivos caros o tóxicos. El proceso requiere mucho tiempo y aumenta el riesgo de contaminación cruzada 1, 2. A la inversa, mediante el uso de Thermo Scientific Phire Plant Direct Kit de PCR del ADN diana se puede detectar fácilmente, sin extracción previa de ADN. En el modelo se demuestra aquí, unaejemplo de derivado troceados análisis de secuencia polimórfica amplificada (dCAPS) 3,4 se realiza directamente a partir de hojas de plantas de Arabidopsis. dCAPS ensayos de genotipificación se puede utilizar para identificar polimorfismos de un solo nucleótido (SNP) por SNP específico de alelo digestión con endonucleasa de restricción 3.
Algunas muestras de plantas tienden a ser más difícil cuando se utilizan métodos de PCR directos, ya que contienen componentes que interfieren con la PCR, tales como compuestos fenólicos. En estos casos, una etapa adicional para eliminar los compuestos se requerido tradicionalmente 2,5. Aquí, este problema se supera mediante el uso de un protocolo de dilución rápida y fácil seguido por amplificación por PCR directa (Figura 1). Quince años de edad, las hojas del roble se utilizan como modelo para las plantas desafiantes como la muestra contiene altas cantidades de compuestos fenólicos incluyendo taninos.
La transferencia de genes en ratones se utiliza ampliamente para estudiar las funciones de los genes en desallo, la fisiología y la enfermedad humana. El uso de estos animales requiere la detección de la presencia del transgén, por lo general con PCR. Tradicionalmente, esto implica una pérdida de tiempo etapa de aislamiento de ADN, durante la cual el ADN para el análisis de PCR se purificó a partir de los tejidos del oído, la cola o del pie 6,7. Sin embargo, con el Thermo Scientific Phire tejido animal directos ratones transgénicos Kit de PCR se puede genotipo sin purificación previa del ADN. En este protocolo de ratón transgénico genotipificación se consigue directamente de los tejidos del oído de ratón, como se demuestra aquí para un ejemplo desafiante donde solamente se utiliza un conjunto de cebadores para la amplificación de dos fragmentos diferentes mucho en tamaño.
Protocolo
1. El genotipado de individuos de plantas de Arabidopsis con Direct Protocol
- Para comenzar el ensayo de genotipado dCAPS directamente en la hoja de la planta Arabidopsis, primero formular 20 o 50 mu l reacciones de PCR utilizando el Phire Plant Direct Kit de PCR como se describe en la Tabla 1.
- A continuación, corte un golpe de 0,50 mm a partir de una hoja de la planta Arabidopsis mediante el Harris Uni-Core y Mat Harris Cutting. Sosteniendo el pegador con firmeza, presione el borde de corte en el tejido y gire el pegador de ida y vuelta.
- Pulsar el émbolo para expulsar el disco punzón en una mezcla de reacción PCR. Asegúrese de que la muestra cae dentro de la solución de PCR y no se pegue a las paredes del tubo.
- Limpie el borde de corte de la perforadora entre cada muestra para evitar la contaminación cruzada por inmersión en solución al 2% NaClO. Presione el émbolo hacia arriba y hacia abajo unas cuantas veces y limpie la punta con una toalla de papel limpia. La estera de corte también debe estarenjuagarse entre muestras.
- A continuación, emplean un Thermo Scientific Piko 24-así Thermal Cycler y Thermo Scientific Plate Piko PCR para llevar a cabo las reacciones de PCR usando las condiciones de ciclos descritas en la Tabla 2. Las reacciones de PCR también se puede realizar en termocicladores de PCR convencionales.
- Después de la PCR, centrifugar el material vegetal. Transferir 5 l del sobrenadante a un nuevo tubo de microcentrífuga. Añadir 4 l de agua y 1 l de enzima de restricción, SspI. Mezclar suavemente y centrifugar brevemente para recoger el contenido en la parte inferior del tubo. Incubar la mezcla de reacción durante una hora a 37 ° C. Inactivar la enzima de restricción por incubación a 65 ° C durante 20 min.
- Cuando amplificando el ADN directamente a partir de tejidos vegetales, los productos de PCR incluyen componentes de la planta y de PCR derivados-que pueden interferir con la enzima de digestión de restricción. Por lo tanto, puede ser necesario diluir o bien (por ejemplo, 1:02 o 1:03 en agua) o purificar el producto de PCR antes de tque la digestión posterior, por ejemplo mediante el uso de un kit comercial adecuado tal como Thermo Scientific kit de purificación de PCR GeneJET.
- Después de la digestión con enzimas de restricción, analizar los fragmentos resultantes en un gel de agarosa. Añadir 2,5 l de 5x tampón de carga a la reacción y realizar la electroforesis en gel de agarosa con 10 l de la mezcla resultante.
2. Amplificar fragmentos específicos de ADN de Oak 15-años de edad, hojas usando el protocolo de dilución
- Para empezar, cortar un punzón de 2 mm de hoja de roble.
- Coloque la muestra en 20 l de tampón de dilución se suministra con la Planta Phire Kit PCR directa. Limpie el borde de corte de la perforadora y una base como antes.
- Triturar la muestra de hoja con una punta de pipeta 100 l pulsando brevemente contra la pared del tubo. Dependiendo del material de la planta, el protocolo de dilución a veces funciona mejor sin aplastar la muestra.
- Haga girar las muestras brevemente en una microcentrífuga hasta tél reacciones son en el fondo de los tubos.
- El sobrenadante puede ser utilizado directamente en la PCR, o puede ser diluido 1:10 o 1:100 en agua estéril, dependiendo del tipo de muestra. Usar 0,5 l a 1 l del sobrenadante o dilución como una plantilla para una reacción de 20 l de PCR. Preparar la reacción como se describe en la Tabla 1.
- Una vez que las reacciones se preparan, emplear un Thermo Scientific Piko 24-así Thermal Cycler y Thermo Scientific Plate Piko PCR para llevar a cabo las reacciones de PCR usando las condiciones de ciclos descritas en la Tabla 2. Las reacciones de PCR también se puede realizar en termocicladores de PCR convencionales.
- Después de la PCR, añadir 5 l de 5x tampón de carga a la reacción y realizar la electroforesis en gel de agarosa con 15 l de la mezcla resultante.
3. Genotipado de ratones transgénicos utilizando el protocolo de dilución
- Empezar el protocolo de dilución en ratones transgénicos mediante la colocación de un punzón de 2 mm de la mouse oreja en 20 l de tampón de dilución que contiene 0,5 l de aditivo DNARelease suministrado con el kit de Animal Tissue Phire directo PCR.
- Se incuban las muestras a temperatura ambiente durante 2 min, seguido de una incubación de 2 minutos a 98 ° C.
- Girar las muestras brevemente en una microcentrífuga hasta que las reacciones son en el fondo de los tubos.
- Usar 1 l de sobrenadante como molde para una reacción de 20 l de PCR, preparado como se describe en la Tabla 3. Cualquier sobrenadante que no se va a utilizar de inmediato puede ser transferido a un nuevo tubo y se almacenó a -20 ° C.
- A continuación, emplean un Thermo Scientific Piko 24-así Thermal Cycler y Thermo Scientific Plate Piko PCR para llevar a cabo las reacciones de PCR usando las condiciones de ciclos descritas en la Tabla 4. Una vez más, las reacciones de PCR también se puede realizar en cicladores PCR convencionales.
- Tras la PCR, añadir 5 l de 5x tampón de carga a la reacción y realizar la electroforesis en gel de agarosa with 15 l de la mezcla resultante.
4. Los resultados representativos
En la técnica de dCAPS el sitio de restricción en el SNP de interés o bien se introduce o se destruye usando un cebador de PCR con uno o más desajustes en el ADN diana. La genotipificación de los individuos de plantas de Arabidopsis se realizó con la técnica de dCAPS directamente de golpes hoja. Los productos amplificados se digirieron los fragmentos resultantes y revelando el genotipo de cada individuo se analizaron por electroforesis en gel (Figura 2).
En este ejemplo, los 160 pb productos PCR que contienen el sitio de SNP de interés (G o alelo A) en el genoma de Arabidopsis fueron amplificados de golpes hoja de la planta con la planta de Phire Kit PCR directa. El cebador directo contenía un error de emparejamiento en el extremo 3 ', la creación de un sitio de restricción SspI-específico en el ADN diana, que incluye el SNP de interés (alelo A), pero noen el otro alelo del SNP (Figura 2B).
Protocolos de dilución son necesarias para material vegetal difíciles, como la hoja de roble, debido a su alta concentración de compuestos fenólicos. Usando el protocolo de dilución descrito aquí, el fragmento de 297 pb de ADN cloroplástico se amplificó utilizando el protocolo 3-paso. Especímenes de hoja de roble con diferentes diluciones se muestra en la Figura 3, además de los controles positivos y negativos.
El Animal Tissue Phire Direct Kit de PCR se empleó con un protocolo de desafío en el que se utiliza sólo un conjunto de cebadores para la amplificación de dos fragmentos con una diferencia de tamaño grande, resultando en rendimientos abundantes de ambos productos del tamaño correcto de 1.500 pb para ratones transgénicos y 200 pb para los los ratones de tipo salvaje (Figura 4). La banda más débil superior con ratones heterocigotos es debido a la competencia de las dos plantillas (alelos) para el mismo primer par, sin embargo, la genotypción los resultados son ambiguos.
La estabilidad de las muestras preparadas usando el protocolo de dilución también fue probado. Los resultados muestran que las muestras de dilución se pueden almacenar a -20 ° C durante al menos un año. Además, repetidos ciclos descongelación / congelación no afectó significativamente la reacción (Figura 5). También se muestra la amplificación robusta de las muestras de tejido de la oreja de ratón usando la polimerasa de ADN Phire y el método de PCR directo en comparación con arranque en caliente polimerasa Taq y el ADN purificado (Figura 6).
| Componentes | 20 l de reacción | 50 l de reacción | Conc. final. |
| H 2 O | añadir hasta 20 l | agregar a 50 l | |
| 2x Phire Plant PCR Buffer | 10 l | 25 l | 1x |
| Un cebador | X l | X l | 0,5 mM |
| cebador B | X l | X l | 0,5 mM |
| Phire Hot Start II DNA Polymerase | 0,4 l | 1 l | |
| Muestra / Direct Protocol | 0,50 mm ponche | ||
| Muestra / dilución protocolo | 0.5-1 l |
Tabla 1. Las condiciones de reacción para la Planta de PCR utilizado en este protocolo.
| Ciclo de Paso | Temperatura | Tiempo | Ciclos |
| La desnaturalización inicial | 98 ° C | 5 min | 1 |
| La desnaturalización Recocido * Extensión | 98 ° C X ° C 72 ° C | 5 sec 5 sec 20 sec | 40 |
| Extensión Final | 72 ° C 4 ° C | 1 min mantener | 1 |
Tabla 2. Ciclismo condiciones para la Planta de PCR utilizado en este protocolo.
* La Tm calculadora enoscientific.com / pcrwebtools "target =" _blank "> www.thermoscientific.com / pcrwebtools se recomienda para determinar la Tm de los cebadores que se utilizarán con Phire Hot Start ADN polimerasa II. La temperatura recomendada de recocido para primers ≤ 20 nt es igual a la Tm más baja dada por la herramienta web. Para cebadores> 20 nt, utilizar una temperatura de recocido 3 ° C más alta que la Tm más baja dada por la herramienta web.
| Componentes | 20 l de reacción | Conc. final. |
| H 2 O | añadir hasta 20 l | |
| 2x Phire Animal Tissue PCR Buffer | 10 l | 1x |
| Un cebador | X l | 0,5 mM |
| cebador B | X l | 0,5 mM |
| Phira Hot Start II ADN polimerasa | 0,4 l | |
| Muestra / dilución protocolo | 1 l |
Tabla 3. Las condiciones de reacción para la PCR tejido animal utilizado en este protocolo.
| Ciclo de Paso | Temperatura | Tiempo | Ciclos |
| La desnaturalización inicial | 98 ° C | 5 min | 1 |
| La desnaturalización Recocido * Extensión | 98 ° C X ° C 72 ° C | 5 sec 5 sec 20 sec ≤ 1 kb 20 sec / kb> 1 kb | 40 |
| Extensión Final | 72 ° C 4 ° C | 1 min Mantener | 1 |
Tabla 4. Ciclismo condiciones para la PCR de tejidos animales utilizados en este protocolo.
* La calculadora Tm en www.thermoscientific.com / pcrwebtools se recomienda cuando la determinación de la Tm de los cebadores para ser utilizados con Phire Hot Start ADN polimerasa II. La temperatura recomendada de recocido para cebadores ≤ 20 nt es igual a la menor Tm propuesta por la herramienta web. Para cebadores> 20 nt, utilizar una temperatura de recocido 3 ° C mayor que la Tm más baja dada por la herramienta web.
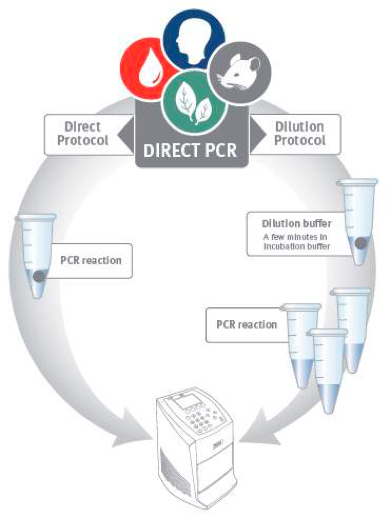
Figura 1. PCR directa de flujo de trabajo. Directo PCR se puede realizar utilizando dos protocolos alternativos. En el protocolo directa de una pequeña cantidad de muestra se añade directamente a la PCR reacción. El protocolo de dilución emplea una breve preincubación antes de la etapa de PCR para liberar el ADN de la muestra de material.
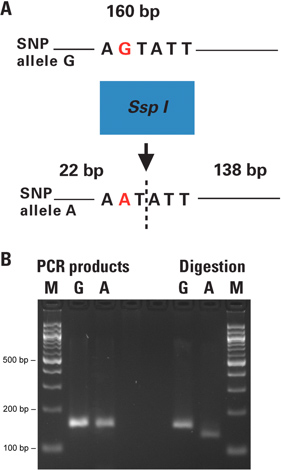
Figura 2. El genotipado de individuos de plantas de Arabidopsis con la técnica dCAPS directamente de golpes hoja. A) La Figura 2A muestra el principio del ensayo realizado dCAPS. El sitio de restricción en el SNP de interés está bien introducido o eliminado por PCR utilizando un cebador con uno o más desajustes en el ADN diana. Los productos de PCR se digieren y los fragmentos resultantes revelan el genotipo de cada individuo cuando se analizó por electroforesis. B) 0,50 mm punzones de hojas de las plantas se colocaron directamente en 50 mu l reacciones de PCR. Los 160 bp productos de PCR que contiene el sitio SNP (G o alelo A) se amplificaron con los cebadores introduciendo un único sitio SspI para el alelo A-. Los productos de PCR fueron purificados digestiónTed con enzima de restricción SspI. Fragmentos resultantes se analizaron en un gel de agarosa al 3%. Carril M es el marcador de tamaño; carriles A y G corresponden a los alelos de SNP de cada muestra. Los resultados obtenidos fueron confirmados por análisis convencional de extracción de ADN seguido de PCR y digestión de restricción (datos no mostrados). Las reacciones de control negativo sin ADN molde se incluyeron en la prueba de configurar y ejecutar en un gel de agarosa separado en paralelo con las reacciones reales de PCR antes de realizar las digestiones (datos no mostrados).
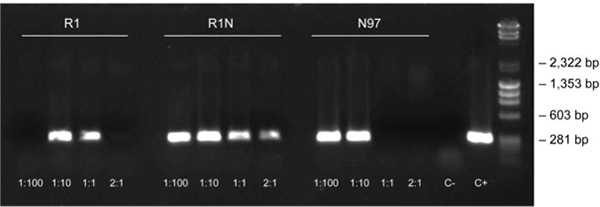
Figura 3. Protocolo de dilución utilizando muestras de hoja de roble. El protocolo de dilución se utilizó para amplificar el fragmento de 297 pb de ADN cloroplástico en muestras de hoja de roble (3-paso protocolo, hibridación a 62 ° C) a diferentes diluciones (1:100, 1:10, 1 : 1 y 2:1) y con positivo y negativocontroles [C +: control positivo de ADN purificado (Arabidopsis), C-: control negativo sin ADN plantilla]. La nomenclatura de la muestra representa el punto de recogida de la muestra (las ciudades en Finlandia) y la muestra de numeración.

Figura 4. Genotipado de los ratones transgénicos con par de cebadores uno utilizando el protocolo de dilución. Tejidos del oído de nueve ratones individuales (carriles 1-9) se colocaron en 20 l de tampón de dilución incluyendo Aditivo DNARelease. Después de las incubaciones a temperatura ambiente y 98 ° C, 1 l de sobrenadante se utilizó como plantilla en la reacción de 20 l de PCR. Tamaños de los fragmentos: 1.500 pb (transgénicos) y 200 pb (tipo salvaje).

Figura 5. Muestras preparadas de acuerdo con el protocolo de dilución son estables durante el almacenamiento a largo plazo. Las muestras de los tejidos del oído de ratón fueron inc ubated en 20 l de tampón de dilución incluyendo Aditivo DNARelease y se sometieron a congelación repetida / descongelación (carril 1), almacenados a -20 ° C durante un año (carril 2), o se utiliza inmediatamente para la PCR (carril 3). Los tamaños de los fragmentos amplificados fueron 900 pb, 1.500 pb y 3.200.
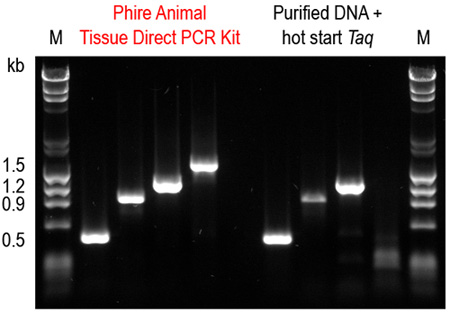
Figura 6. Phire Animal Tissue Kit de PCR directo supera a un sistema de purificación de ADN comercial combinado con una polimerasa de ADN Taq de arranque en caliente. Cuatro amplicones (0.5-1.5 kb) se amplificaron a partir de los tejidos del oído de ratón utilizando el protocolo de dilución de Animal Phire Tissue Kit PCR directa. Para la comparación, el ADN se purificó primero utilizando un kit de extracción de ADN comercial y los mismos fragmentos se amplificaron usando un arranque en caliente, la ADN polimerasa Taq de acuerdo con las recomendaciones del fabricante. Sólo con la Phire Animal Tissue PCR Kit Direct fueron amplificados con éxito los cuatro amplificados.
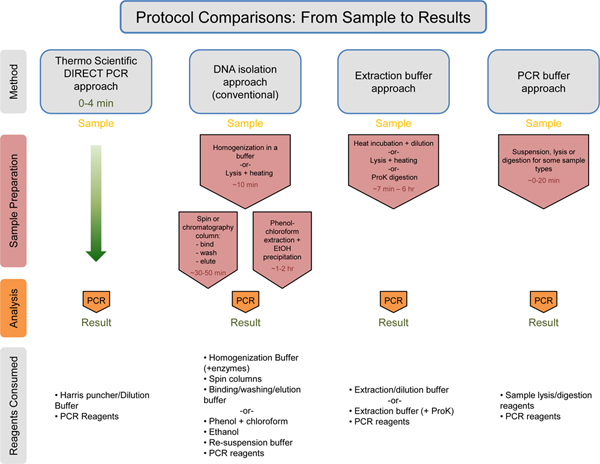
Figura 7. Comparación universal del flujo de trabajo con la estimación de tiempos / reactivos consumidos. Thermo Scientific directo enfoque de PCR se compara con el enfoque de aislamiento de ADN, el enfoque de tampón de extracción, y el enfoque de tampón de PCR. El tiempo estimado para cada método antes de la etapa de PCR se muestra en rojo. El Thermo Scientific enfoque directo PCR tarda sólo 0-4 minutos para la preparación de muestras antes de la PCR. Debajo de cada método, los reactivos consumidos se enumeran. El Thermo Scientific enfoque directo PCR utiliza la menor cantidad de reactivos en comparación con los otros métodos. Haga clic aquí para ampliar la cifra .
Discusión
El enfoque directo PCR demostró aquí permite la amplificación PCR directamente a partir de pequeñas cantidades de muestras no purificadas, reduciendo el tiempo necesario y simplificar el flujo de trabajo para la planta y el genotipo de animales (Figura 7). También se ha demostrado aquí es el uso del protocolo de dilución en combinación con el enfoque directo de PCR (Figura 3). El protocolo de dilución se recomienda con muestras difíciles (por ejemplo, hojas de plantas de edad, especies de plantas que contienen compuestos de interferencia) o largos (con o ricos en GC) amplicones. Este protocolo es particularmente útil cuando se inicia un nuevo experimento PCR directa, lo que permite la optimización de la reacción. El protocolo puede servir como una herramienta para abordar las dificultades ocasionales, tales como bajos rendimientos del producto debido a diferencias en el material tejido y / o iniciadores utilizados. Además, cuando se ejecuta múltiples reacciones de la misma muestra, el uso del protocolo de dilución garantiza toda la muestra no se consume en un soloreacción.
Con el fin de purificar el ADN de las plantas de la PCR, la convención fue realizar la lisis celular, seguido por el aislamiento de ADN utilizando diversos componentes que potencialmente podrían interferir con el análisis de PCR 8. En particular, las plantas desafiante con alto contenido de compuestos fenólicos, además de polivinilpirrolidona se ha utilizado tradicionalmente para eliminar los compuestos que se unen al ADN tras la lisis celular 2,5. Como es evidente aquí, estos complicados, intensivas en tiempo pasos se pueden evitar mediante el uso de la Phire Plant Direct Kit de PCR para detectar el ADN diana. La lisis celular y los pasos de aislamiento de ADN se puede omitir utilizando las técnicas dCAPS sensibles directamente a partir de hojas de plantas de Arabidopsis (Figura 2). Como se demuestra aquí, el protocolo de dilución administra de manera eficaz la presencia de componentes problemáticos que pueden interferir con PCR (Figura 3).
Tradicionalmente, un requisito previo para la determinación del genotipo de animales era isolatien el ADN genómico a partir de tejido animal a través de un tóxico, proceso que consume tiempo caracterizado por una incubación prolongada con proteinasa K para digerir el tejido conectivo y la piel, seguido por extracción con disolvente orgánico, precipitación con alcohol, y las etapas de centrifugación 6,9,10. Como se ha demostrado aquí, la simplificación de este proceso se logra a través del uso de la Phire Animal Tissue Kit de PCR directo, permitiendo que los ratones transgénicos para determinar el genotipo de ADN sin purificación previa (Figura 4). En el ejemplo mostrado en este artículo es particularmente difícil ya que sólo un conjunto de cebadores se utilizó para la amplificación de dos fragmentos con una diferencia de tamaño grande. A pesar de dificultad innata del protocolo, el uso del enfoque de PCR directo en conjunción con el protocolo simple dilución dio lugar a altos rendimientos de los dos productos de PCR (Figura 4).
PCR basado en la detección de ADN de destino tiene muchas aplicaciones en la investigación, incluyendo el análisis del genotipo para identificarlas funciones de los genes en el desarrollo, la fisiología y la enfermedad. Debido a la tolerancia inhibidor de las polimerasas de DNA especializados, los protocolos se puede completar en un tiempo mínimo sin purificación de ADN antes.
Divulgaciones
La producción y el libre acceso a este artículo es patrocinado por Thermo Fisher Scientific, Inc.
Agradecimientos
Para las muestras de Arabidopsis y los interesados cebadores de PCR que agradecer al profesor Jaakko Kangasjarvi y su grupo, el Grupo de Estrés Vegetal, Biología Vegetal, Departamento de Ciencias Biológicas de la Universidad de Helsinki. Los experimentos con el método tradicional se llevaron a cabo por la Sra. Airi Lamminmäki.
Muestras de ratones transgénicos fueron proporcionadas por el Dr. Jaana Vesterinen, Instituto de Biomedicina / Bioquímica de la Universidad de Helsinki.
Harris Uni-Core y Mat Harris Cutting son marcas comerciales de Shunderson Communications Inc. Todas las demás marcas comerciales son propiedad de Thermo Fisher Scientific Inc. y sus filiales.
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | Comentarios |
| Phire Plant Direct PCR Kit | Thermo Fisher Scientific | F-130 | 200 reacciones (50 l cada uno) |
| Phire Animal Tissue PCR Kit Direct | Thermo Fisher Scientific | F-140 | 200 reations (50 l cada uno) |
| Harris Uni-Core 0,35 mm (rosa) | Thermo Fisher Scientific | F-180S / L | Cantidad: 5/25 |
| Harris Uni-Core 0,50 mm (azul) | Thermo Fisher Scientific | F-185S / L | Cantidad: 5/25 |
| Harris Cutting Mat 6,4 x 7,6 cm | Thermo Fisher Scientific | F-190 | Cantidad: 5 |
| SspI | Thermo Fisher Scientific | ER0771 | 100 l (100 reacciones) |
| Piko 24-Bueno Thermal Cycler | Thermo Fisher Scientific | TCP0024 | |
| 24-Bueno Piko placas PCR | Thermo Fisher Scientific | SLP0241 | |
| GeneJET PCR Purificación Kit | Thermo Fisher Scientific | K0701 K0702 | 50 preps 250 preps |
Referencias
- Doyle, J. J., Doyle, J. L. A rapid DNA isolation procedure for small quantities of fresh leaf tissue. Phytochemical Bulletin. 19, 11-15 (1987).
- Kim, C. S., Lee, C. H., Shin, J. S., Chung, Y. S., Hyung, N. I. A simple and rapid method for isolation of high quality genomic DNA from fruit trees and conifers using PVP. Nucleic Acids Research. 25, 1085-1086 (1997).
- Neff, M. M., Neff, J. D., Chory, J., Pepper, A. E. dCAPS, a simple technique for the genetic analysis of single nucleotide polymorphisms: experimental applications in Arabidopsis thaliana genetics. The Plant Journal. 14, 387-392 (1998).
- Michaels, S. D., Amasino, R. M. A robust method for detecting single-nucleotide changes as polymorphic markers by PCR. The Plant Journal. 14, 381-385 (1998).
- John, M. E. An efficient method for isolation of RNA and DNA from plants containing polyphenolics. Nucleic Acids Research. 20, 2381-23 (1992).
- Wang, Z., Storm, D. R. Extraction of DNA from mouse tails. BioTechniques. 41, 410-412 (2006).
- Malumbres, M., Mangues, R., Ferrer, N., Lu, S., Pellicer, A. Isolation of High Molecular Weight DNA for Reliable Genotyping of Mice. BioTechniques. 22, 1114-1119 (1997).
- Yang, Y., Kim, J. Y., Soh, M. S., Kim, D. S. A simple and rapid gene amplification from Arabidopsis Leaves Using AnyDirect system. Journal of Biochemistry and Molecular Biology. 40, 444-447 (2007).
- Sambrook, J., Fritsch, E., Maniatis, T. . Molecular Cloning: A Laboratory. , (1989).
- Meldgaard, M., Bollen, P. J. A., Finsen, B. Non-invasive method for sampling and extraction of mouse DNA for PCR. Laboratory Animals. 3, 413-441 (2004).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados