Method Article
Identificación de los socios interacción de proteínas en tándem de purificación de afinidad
* Estos autores han contribuido por igual
En este artículo
Resumen
Tandem purificación de afinidad es un enfoque sólido para la identificación de socios de la unión a proteínas. Como prueba de concepto, esta metodología se aplicó a la traducción factor eIF4E bien caracterizado iniciación a co-precipitado los factores implicados en la célula huésped iniciación de la traducción. Este método se adapta fácilmente a cualquier proteína celular o viral.
Resumen
Un paso crítico y la limitación de frecuencia en la comprensión de la función de acogida y las proteínas virales es la identificación de la interacción de proteínas asociados celulares o virales. Existen muchos enfoques que permiten la identificación de socios que interactúan, incluyendo la levadura de dos sistema híbrido, así como derribar ensayos utilizando proteínas recombinantes y de inmunoprecipitación de las proteínas endógenas seguido por espectrometría de masa de identificación 1. Estudios recientes han puesto de relieve la utilidad de la doble purificación por afinidad mediada por la etiqueta, junto con dos pasos de elución específicos en la identificación de proteínas que interactúan. Este enfoque, denominado tándem Purificación por afinidad (TAP), se utilizó inicialmente en la levadura 2,3 pero más recientemente se ha adaptado para usar en células de mamífero 4-8.
Como prueba de concepto que hemos establecido una purificación por afinidad en tándem (TAP), utilizando la bien caracterizada iniciación de la traducción eucariótica factortor eIF4E 9,10. La traducción eIF4E factor celular es un componente crítico del complejo eIF4F celular implicada en la tapa que dependen de iniciación de traducción 10. La etiqueta TAP utilizado en el presente estudio se compone de dos unidades de proteína G y un péptido de unión de estreptavidina separados por un virus del Tabaco Etch (VET) secuencia de escisión de la proteasa. La etiqueta TAP utilizado en el presente estudio se compone de dos unidades de proteína G y un péptido de unión de estreptavidina separados por un virus del Tabaco Etch (VET) secuencia proteasa escisión 8. Para renunciar a la necesidad de que la generación de líneas celulares clonales, hemos desarrollado un sistema rápido que se basa en la expresión de la proteína TAP-etiquetados cebo de un plásmido episomal mantenido sobre la base de pMEP4 (Invitrogen). Expresión de eIF4E etiquetados murino de este plásmido se controla mediante el cloruro de cadmio promotor inducible metalotioneína.
La lisis de las células que expresan y purificación de afinidad posterior a través de la unión a Rabbit IgG de agarosa, la escisión VET proteasa, la unión a estreptavidina ligada agarosa y elución biotina posterior identificado numerosas proteínas aparentemente específicas para el eIF4E desplegable (en comparación con el control de líneas celulares que expresan la etiqueta TAP solo). Las identidades de las proteínas se obtuvieron mediante la escisión de las bandas de espectrometría 1D tándem SDS-PAGE y posterior masa. Los componentes identificados incluyen las proteínas de unión eIF4E conocidos eIF4G y 4EBP 1. Además, otros componentes del complejo eIF4F, de los cuales eIF4E es un componente fueron identificados, a saber eIF4A y Poly-Una proteína de unión. La capacidad de identificar no sólo se conocen los socios directos de unión, así como secundarias proteínas que interactúan, destaca aún más la utilidad de este enfoque en la caracterización de proteínas de función desconocida.
Protocolo
1. Generación de líneas celulares: pMEP4 transfección / expresión
- Transfectar células con el vector de expresión pMEP4 y seleccionar con 100 g de higromicina B / ml (Roche) hasta que todas las células transfectadas simuladas se han matado. El plásmido pMEP4 se mantiene episomalmente lo que no hay necesidad de aislar clones específicos.
- Las células que contienen el vector pMEP4 puede ser inducida por tratamiento con 10 CdCl 2 mM durante 16 horas. Expresión y la inducibilidad debe ser confirmada en una pequeña escala antes de la amplificación de las líneas celulares. La proteína TAP-etiquetados puede ser fácilmente detectado como los dominios de la proteína G se unen a los anticuerpos de casi todas las especies. Para los grandes purificaciones normalmente 10 confluentes 175 cm 2 frascos de células son obligatorios. Esto equivale a aproximadamente 2 X10 8 células que expresan.
2. De preparación de células lisado
- Raspe las células en PBS y se combinan en un tubo de 50 ml de una sola. Spin 1200X gfo 5 minutos (esto debería resultar en 2-3 ml de células empaquetadas para empezar, lo que reduce a aproximadamente 1,5 ml después del lavado).
- Se lavan las células tres veces en PBS enfriado en hielo (50 ml cada vez).
- Lisar las células en 5 ml de tampón de lisis (50 mM Tris-HCl (pH 7,5), NaCl 125 mM, glicerol al 5%, 0,2% de NP-40, 1,5 mM de MgCl 2, 25 mM de NaF, 1 mM Na 3 VO 4 y el inhibidor de proteasa ). Nota: NaF, Na 3 VO 4 y los inhibidores de la proteasa debe añadirse fresco. Pipetear arriba y hacia abajo 10 veces antes de dejar durante 5 minutos en hielo. Veces jeringa hacia arriba y hacia abajo 5-10 usando una aguja roma antes de salir de nuevo en hielo durante 5 minutos en hielo. Repetir su inyección y se deja durante otros 5 minutos en hielo.
- Congelación-descongelación del lisado dos veces (hielo líquido N / 2 o seco y etanol). No permita que la muestra para llegar a más de 4 ° C. Nota: se puede almacenar la muestra a -80 ° C hasta que tenga tiempo para procesar.
- Alícuotas de la muestra en tubos de 1,5 ml y eliminar las células no lisadas y debris por centrifugación (10 minutos a 4 ° C, 16.000 xg).
- Recuperar sobrenadante, se combinan en un tubo Falcon de 15 ml y (opcionalmente) pasan a través de unos 0,45 m de filtro antes de tomar una muestra de 20 l para la cuantificación de proteínas rendimiento. Eliminar un ulterior 50 l alícuota para su posterior análisis (Muestra 1).
3. La unión a IgG de conejo-agarosa
(Nota: Todos los giros deben llevarse a cabo a 1200X g en una centrífuga refrigerada a 4 ° C durante 1 minuto, a menos que se indique lo contrario)
- Resuspender la IgG de conejo-agarosa solución (Sigma-Aldrich) por agitación botella. Tomar 380 l de IgG de agarosa (utilizando un corte de punta de la pipeta 1 ml) y retirar el envío / solución conservante por centrifugación durante 1 minuto. Lavar la agarosa tres veces en tampón de lisis frío (4 ° C) usando centrifugación para aclarar. El rendimiento final de agarosa debe ser de aproximadamente 250 l de perlas envasados.
- Añadir el lisado celular desaparecido de 2,6 (en una tina de 15 ml Falcone) el lavado de conejo IgG de agarosa y se incuba durante 3 horas (o durante la noche) a 4 ° C usando un mezclador giratorio.
- Derivado establecen las perlas de agarosa durante 5 minutos a 4 ° C y separar el sobrenadante para su posterior análisis (Muestra 2). Nota: El TAP etiquetado de proteínas debe ahora estar asociados con las cuentas.
4. Escisión de la proteasa TEV
(Nota: Todos los giros deben llevarse a cabo a 1200X g en una centrífuga refrigerada a 4 ° C durante 1 minuto, a menos que se indique lo contrario)
- Lavar las perlas de agarosa IgG de conejo tres veces en tampón de lisis frío (4 ° C) mediante centrifugación para aclarar (nota: el tampón de lisis no debe contener inhibidores de la proteasa). Se debe tener cuidado de no eliminar o perder los granos durante los pasos de lavado.
- Se lavan las perlas de otras dos veces con tampón de escisión VET-proteasa (10 mM Tris-HCl (pH 7,5), NaCl 100 mM, y 0,2% NP-40) utilizando centrifugación para aclarar. Después del último lavado retire con cuidado todas las liquid de las perlas.
- Prepare una mezcla de la proteasa TEV división, para cada muestra se incluyen 467,5 l H 2 O, 25 l 20x TEV buffer (Invitrogen), 5 ml DTT 0,1 M y 2,5 l (25 U) de la proteasa TEV (Invitrogen). Añadir 500 l de esta mezcla de escisión VET a cada muestra de perlas envasados y transferir toda la mezcla a un 1,7 ml de pre-lubricado tubo (Costar). Antes de la incubación eliminar una parte alícuota de 30 l para su posterior análisis (Muestra 3).
- Incubar la reacción proteasa TEV durante la noche a 4 ° C usando un mezclador giratorio. Nótese que tiempos más cortos incubaciones se pueden utilizar dependiendo de la naturaleza de la proteína cebo y la accesibilidad del sitio de escisión de la proteasa VET pero el tiempo de incubación mínimo debe ser determinado empíricamente.
5. La unión a estreptavidina inmovilizada Ultralink Plus cuentas
(Nota: Todos los giros deben llevarse a cabo a 1200X g en una centrífuga refrigerada a 4 ° C durante 1 minuto, a menos que se indique lo contrario)
- Centrifugar la reacción de escisión VET que contiene las perlas de agarosa IgG de conejo durante 5 minutos a 1200X g (4 ° C). Retirar una parte alícuota de 20 l del sobrenadante clarificado para el análisis (Ejemplo 4)
- Transferir el sobrenadante restante (aproximadamente 480 l) a un nuevo máximo de 1,5 ml de pre-lubricadas tubo (Costar) y dejar este en el hielo. No se deshaga de este sobrenadante - contiene la proteína de TEV cebo hendidas y las proteínas de unión asociados.
- Añadir 500 l de tampón de lisis frío (Sección 2.3) a las cuentas restantes de IgG de conejo de agarosa y volver a suspender. Centrifugar esta mezcla para aclarar las perlas antes de retirar el sobrenadante y combinándolo con el de paso 5,2. Deje este tubo en hielo (que ahora debe contener ~ 980 ml de muestra).
- Conserve las IgG de conejo perlas de agarosa para su posterior análisis (muestra 5). Nota: si se ejecuta este ejemplo en un gel de SDS-PAGE (o Western Blot) que le dará un montón de fondo debido a la presencia de la pesada de IgG y de la luzcadenas.
- Mientras tanto Resuspender el Ultralink estreptavidina inmovilizada Plus perlas de agarosa (Pierce), haciendo girar la botella. Con una punta de pipeta de 200 ml con el corte final, alícuota de 70 l de cuentas estreptavidina por muestra en pre-lubricadas tubos de 1,5 ml y eliminar el envío / solución conservante por centrifugación. Nota: estas cuentas son muy pequeñas y por lo tanto "pico de pato" o plana / estrecho de composición consejos pueden ser utilizados para minimizar la pérdida de grano durante el lavado.
- Lávese las cuentas estreptavidina tres veces en tampón de lisis frío (4 ° C) mediante centrifugación para aclarar. Esto debería dejar de aproximadamente 45-50 l de bolas llenas por tubo.
- Transferir el sobrenadante escisión VET-proteasa (es decir, el l de la muestra 980 de paso 5,3) al tubo que contiene las perlas de estreptavidina lavadas e incubar durante 3 horas (o durante la noche) a 4 ° C usando un mezclador giratorio.
- Derivado establecen las perlas de estreptavidina durante 5 minutos a 4 ° C y separar el sobrenadante para su posterior análisis (Samejemplo 6). Lávese las cuentas estreptavidina tres veces en tampón de lisis frío (4 ° C) mediante centrifugación para aclarar. Después del lavado final, cuidadosamente eliminar el tampón de lavado restante.
6. Biotina elución del péptido y proteína de unión de estreptavidina cebo
(Nota: Todos los giros deben llevarse a cabo a 1200X g en una centrífuga refrigerada a 4 ° C durante 1 minuto, a menos que se indique lo contrario)
- Eluir proteínas etiquetadas de las perlas de estreptavidina mediante la adición de 500 l de PBS: biotina (1 mM de D-biotina) y la incubación a 4 ° C durante 3 horas (o durante la noche) usando un mezclador giratorio.
- Gira por las perlas de estreptavidina durante 5 minutos a 4 ° C y retirar el sobrenadante a un tubo de 1,5 ml de pre-lubricadas (Nota: esta es la muestra final / de elución (Ejemplo 7).
- Añadir otra Biotina 500 l: PBS a las perlas de estreptavidina restantes y se incuba a 4 ° C durante 2-3 horas (o durante la noche) usando un mezclador giratorio.
- Returba el paso 6.2 y combinar los dos eluciones (Ejemplo 7). Este elución final puede ser almacenado a -80 ° C.
- Para evaluar la eficiencia de la elución de la biotina, congelar las cuentas de estreptavidina restantes y hervir más tarde en tampón de muestra SDS (Muestra 8) (recomendaciones: LaneMarker 5x tampón de muestra de la reducción de Fisher).
7. La concentración de proteínas
- El eluido final (Ejemplo 7) tiene que ser concentrado antes de su análisis, debido a la baja concentración de proteínas y el volumen relativamente alto. Esto se puede lograr utilizando de bajo peso molecular (menos de 5 kDa) columnas Vivaspin spin (Vivascience). Por objeto concentrar el volumen a menos de 100 l. Esta muestra final se puede hervir y se almacena en el búfer de la proteína de la muestra (recomendaciones: LaneMarker 5x tampón de muestra de la reducción de Fisher).
8. Análisis
- Las muestras 1-8 se puede analizar por Western blot para determinar la eficacia de tirar hacia abajo. Nota: La mayoría de los anticuerpos secundarios seuna reacción cruzada con los dominios de la proteína G de la proteína de fusión, pero esto se elimina después de la escisión de la proteasa VET.
- Ejemplo 7 pueden ser analizados por 1D o 2D SDS-PAGE. La tinción puede realizarse usando plata o de Coomassie (recomendaciones: tinción SilverQuest plata coloidal y kits de tinción con azul Coomassie de Invitrogen). Identificación de proteínas se puede realizar utilizando espectrometría de masas.
9. Los resultados representativos
Un ejemplo de 1D análisis SDS-PAGE de la elución final (Ejemplo 7) de este protocolo para identificar parejas de unión de eIF4E TAP etiquetado se proporciona en la Figura 2. Este gel representativo refleja la naturaleza compleja y abundante de las interacciones con otras proteínas eIF4E en la célula. La comparación con el control negativo también se muestra en la Figura 2, generada a partir de una línea celular que expresa sólo la etiqueta TAP, ilustra la especificidad de este eIF4E cebo desplegable. En este ejemplo el 15% de la elución final concentrada (Muestra7) se analizó por SDS-PAGE 1D utilizando comercialmente disponibles prefabricados geles de gradiente antes de ser teñidos utilizando el kit de tinción de plata Silverquest de Invitrogen.
Típicamente 50-85% de la restante elución concentrado final (Ejemplo 7) se analiza con Coomassie coloidal mancha (Invitrogen). Las muestras (rodajas de gel o bandas) de todo el carril se extrajeron luego y se analizaron por espectrometría de masas.
Las proteínas identificadas en el eIF4E pull-down se filtraron en contra de las parejas de unión del control negativo para identificar no específicos asociados vinculantes. Las proteínas finales identificados utilizando este proceso de eIF4E TAP etiquetado puede verse en la Figura 2, que es representativo de las proteínas normales identificados utilizando esta técnica. Además, un número de las subunidades eIF3 También se han detectado (datos no presentados).

Figura 1. Esquema de laconjuntamente procedimiento de purificación por afinidad. El tándem de seis pasos de purificación de afinidad (TAP) de protocolo implica la generación de líneas celulares, la lisis celular, IgG de conejo de agarosa inmuno-precipitación, TEV división de la proteasa, estreptavidina purificación por afinidad de cuentas y, finalmente elución biotina.
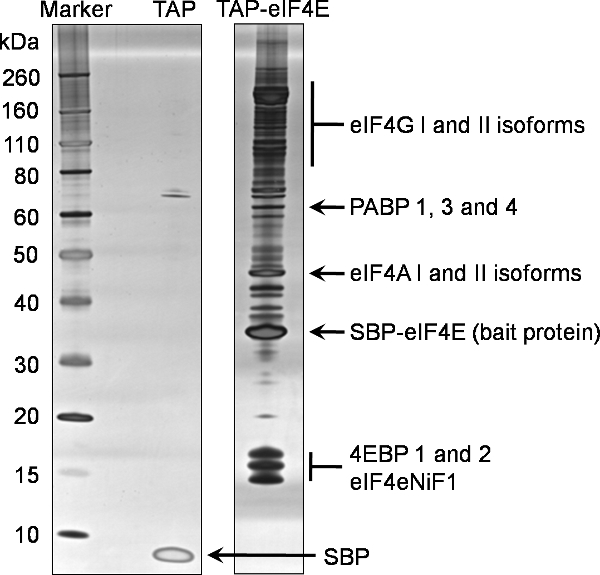
Figura 2. Tándem purificación por afinidad de la proteína murina eIF4E. Interactuar socios de la N terminal TAP etiquetados eIF4E se purificaron a partir de células eucariotas HEK293 utilizando el protocolo adjunto. Una fracción del 20% de la elución final (Ejemplo 7) se analizó por SDS-PAGE en un gel de gradiente prefabricado 4-12% (PAT-eIF4E carril). Un análisis equivalente se llevó a cabo para la etiqueta por sí solo (carril TAP). Las proteínas se identificaron mediante la tinción de plata. Las proteínas posteriormente identificados por espectrometría de masas de la misma muestra se destacan en este gel.
Abreviaturas: eIF4G; iniciación de la traducción eucariótica factor de 4 gamma, PABP, proteína poli vinculante, eIF4A: factor de iniciación de la traducción eucariótica 4 alfa, SBP-eIF4E, la proteína PAT cebo restante que contiene el péptido estreptavidina vinculante fusionado con el factor 4E traducción eucariótico de iniciación, 4EBPs; eucariótico de iniciación de traducción 4E factor de unión proteínas, eIF4eNiF1l eucariótico de iniciación de traducción factor de factor nuclear de importación 4E 1, PAS, el péptido estreptavidina restante de unión del péptido TAP contenga restos de TEV.
Discusión
La técnica de marcado TAP ilustrado aquí demuestra un método altamente específico y estrictas para aislar las parejas de unión de proteínas de cebo en las células eucariotas. Este enfoque puede ser aplicado a las proteínas celulares y virales. A nuestro entender, esta es la primera vez que esta técnica aplicada a la traducción eIF4E factor de iniciación. Identificación de la eIF4G eIF4E conocido proteínas vinculantes y los 4EBPs uso de esta técnica se confirma la validez de este enfoque. Además, la identificación del componente restante del complejo eIF4F, a saber eIF4A, y PABP confirma que la interacción indirecta y complejos terciarios permanecer intacto durante el proceso de purificación. La identificación de múltiples isoformas de las proteínas de unión eIF4E canónicos era también evidente. Estos se describen con más detalle en la Figura 2.
En cuanto a las limitaciones del enfoque, la atención de algunos se debe tomar con respecto a la elecciónde proteína cebo y si o no para colocar la etiqueta TAG en el extremo N-o C-terminal. Podría ser conveniente para llevar a cabo un ensayo funcional o examinar la localización de la proteína de fusión con el microscopio antes de la purificación a gran escala para asegurar el derivado de etiquetado es funcional. Integral de la membrana o proteínas nucleares no necesariamente puede ser liberado por las condiciones relativamente suaves detergentes descritas en la etapa de lisis. Como con todas las inmunoprecipitaciones y similares ensayos desplegable, se podría hacer modificaciones a la naturaleza y concentración del detergente en el tampón de lisis para aumentar la eficiencia de la lisis. La concentración iónica de los tampones de lisis y lavado también puede modificarse para aumentar o disminuir la severidad de la purificación. También es posible llevar a cabo la purificación inicial bajo condiciones suaves estándar (descrito anteriormente), pero posteriormente dividir las perlas de estreptavidina (sección 5,8) en alícuotas de 4-5, que posteriormente se pueden lavar bajo con iónica crecientecondiciones. Esto puede permitir al usuario identificar las condiciones óptimas que eliminan proteínas no específicas que interactúan. Todo el proceso puede realizarse también utilizando la transfección transitoria cuando las distintas partes que interactúan son conocidos y su presencia o ausencia determinó por transferencia subsiguiente occidental solamente. En este caso, le sugerimos dos 100cm 2 placas de células transfectadas con cada uno de 8 g de plásmido de expresión. Este procedimiento modificado es de uso particular, al examinar la capacidad de los derivados mutantes de la proteína TAP fusión para interactuar con parejas de unión conocidas.
El análisis de las muestras 1 a 8 en la teñidos con plata geles SDS-PAGE (o por Western blot, si un anticuerpo está disponible) es una excelente forma de solución de problemas, si la técnica de no generar resultados aceptables. La eficacia de cada precipitación basado en la afinidad y la elución específica puede ser analizado utilizando este método de muestreo sistemático. Las muestras de uno a ocho se debe tomar durante Optimzación del protocolo para cada nuevo cebo.
La técnica de marcado TAP ofrece una alternativa potente y robusta con otros enfoques, tales como GST pull-downs, la levadura de dos ensayos de híbridos, etc La purificación por afinidad de matrimonio y los pasos específicos de elución (TEV división y la elución biotina) proporcionan la especificidad y el rigor que se mantiene a un alto nivel durante todo el procedimiento de purificación. Críticamente esta técnica se puede aplicar a cualquier proteína y por lo tanto representa un método excelente para la identificación de parejas de unión para una proteína diana de interés.
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
Esta investigación fue financiada por una beca de Wellcome Trust Superior otorgado al Dr. Ian Goodfellow.
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | |
| Higromicina B | Roche | 10843555001 | |
| IgG de conejo de agarosa | Sigma | A2909 | |
| AC-VET proteasa | Invitrogen | 12575-015 | |
| Cóctel de inhibidores de la proteasa | Calbiochem | 539134 | |
| Ultralink estreptavidina inmovilizada cuentas Plus | Atravesar | 53116 | |
| Vivaspin 500 concentrador centrífugo (5KDa) | Vivaspin | V50112 | |
| SilverQuest kit de tinción de plata | Invitrogen | LC6070 | |
| Novex azul de Coomassie coloidal kit de tinción | Invitrogen | LC6025 | |
| 1,7 ml tubos prelubricados | Costar | 3207 | |
| Microcapilar puntas de pipeta | VWR | 37001-150 | |
| NuPage 4-12% Bis-Tris gradiente de geles | Invitrogen | NP0322BOX | |
| CdCl 2 | Sigma | 202908 | |
| 5x tampón de muestras SDS | Pescador | PN39000 |
Referencias
- Williamson, M. P., Sutcliffe, M. J. Protein-protein interactions. Biochem. Soc. Trans. 38 (4), 875-878 (2010).
- Rigaut, G. A generic protein purification method for protein complex characterization and proteome exploration. Nat. Biotechnol. 17 (10), 1030-1032 (1999).
- Blackwell, C., Brown, J. D. The application of tandem-affinity purification to Candida albicans. Methods Mol. Biol. 499, 133-148 (2009).
- Cochrane, A. Stable complex formation between HIV Rev and the nucleosome assembly protein, NAP1, affects Rev function. Virology. 388 (1), 103-111 (2009).
- Fernandez, E. Targeted tandem affinity purification of PSD-95 recovers core postsynaptic complexes and schizophrenia susceptibility proteins. Mol. Syst. Biol. 5, 269-269 (2009).
- Gloeckner, C. J. Tandem affinity purification of protein complexes from mammalian cells by the Strep/FLAG (SF)-TAP tag. Methods Mol. Biol. 564, 359-372 (2009).
- Holowaty, M. N. Protein profiling with Epstein-Barr nuclear antigen-1 reveals an interaction with the herpesvirus-associated ubiquitin-specific protease HAUSP/USP7. J. Biol. Chem. 278 (32), 29987-29994 (2003).
- Burckstummer, T. An efficient tandem affinity purification procedure for interaction proteomics in mammalian cells. Nat. Methods. 3 (12), 1013-1019 (2006).
- Goodfellow, I. Calicivirus translation initiation requires an interaction between VPg and eIF 4 E. EMBO Rep. 6 (10), 968-972 (2005).
- Goodfellow, I. G., Roberts, L. O. Eukaryotic initiation factor 4E. Int. J. Biochem. Cell Biol. 40 (12), 2675-2680 (2008).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados