Method Article
Identifizierung von Protein Interaktionspartner mittels Tandem Affinity Purification
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Tandem Affinitätsreinigung ist ein robustes Verfahren zur Identifizierung von Protein Bindungspartner. Als Proof of Concept wurde die Methode auch auf die gut charakterisierte Translationsinitiationsfaktors eIF4E zu Copräzipitat der Wirtszelle Faktoren in Initiation der Translation beteiligt angewendet. Dieses Verfahren ist einfach einem der zellulären oder viralen Proteins.
Zusammenfassung
Eine kritische und oft limitierende Schritt zum Verständnis der Funktion des Host und viralen Proteinen ist die Identifizierung von interagierenden zellulären oder viralen Proteins Partner. Es gibt viele Ansätze, die die Identifizierung von interagierenden Partner, einschließlich der Hefe-Zwei-Hybrid-System, sowie Pulldown-Assays unter Verwendung von rekombinanten Proteinen und Immunpräzipitation von endogenen Proteinen durch Massenspektrometrie Kennzeichnung 1 gefolgt ermöglichen. Neuere Studien haben die Nützlichkeit von Doppel-Affinitäts-Tag vermittelten Reinigung mit zwei spezifischen Elutionsschritte bei der Identifizierung von wechselwirkenden Proteinen gekoppelt ist hervorgehoben. Dieser Ansatz, genannt Tandem Affinity Purification (TAP), wurde ursprünglich in der Hefe 2,3 verwendet, aber in jüngerer Zeit wurde angepasst, um in Säugetierzellen verwenden 8.4.
Als Proof-of-Konzept haben wir eine Tandem-Affinitätsreinigung (TAP)-Methode etabliert mit dem gut charakterisierten eukaryotischen Translations-Initiations-factor eIF4E 9,10. Die zelluläre Übersetzung Faktor eIF4E ist eine kritische Komponente der zellulären eIF4F Komplex in Cap-abhängige Translation Initiation 10 beteiligt. Der TAP-Tag in der vorliegenden Studie verwendet wird, von zwei Protein G-Bausteinen bestehen und ein Streptavidin-Bindungspeptid von einem Tabak Etch Virus (TEV) Protease-Schnittstelle Sequenz getrennt ist. Der TAP-Tag in der vorliegenden Studie verwendet wird, von zwei Protein G-Bausteinen bestehen und ein Streptavidin-Bindungspeptid durch eine Tabakätzvirus getrennt (TEV) Protease-Schnittstelle Sequenz 8. Um die Notwendigkeit der Erzeugung von klonalen Zelllinien zu verzichten, entwickelten wir ein schnelles System, die auf der Expression des TAP-markierten Proteins aus einem Köder episomal aufrechterhalten Plasmid auf pMEP4 (Invitrogen) auf der Grundlage beruht. Die Expression von Maus-tagged eIF4E aus diesem Plasmid wurde unter Verwendung der Cadmiumchlorid induzierbaren Metallothionein-Promotor.
Lysis der exprimierenden Zellen und die nachfolgende Affinitätsreinigung durch die Bindung an rabbit IgG Agarose, TEV-Protease-Spaltung, die Bindung an Streptavidin gebunden Agarose und anschließende Elution Biotin wurden zahlreiche Proteine scheint spezifisch für die eIF4E Pull-down (im Vergleich zu Zelllinien, die das TAP-Tag alleine zu steuern). Die Identität der Proteine wurden durch Ausschneiden der Bänder aus 1D SDS-PAGE und anschließende Tandem-Massenspektrometrie. Die identifizierten Komponenten enthalten die bekannten eIF4E bindende Proteine eIF4G und 4EBP-1. Zusätzlich können andere Komponenten des eIF4F Komplex, bestehend aus dem eIF4E ist eine Komponente identifiziert wurden, nämlich eIF4A und Poly-A-bindendes Protein. Die Fähigkeit, nicht nur bekannt direkten Bindungspartnern sowie sekundäre interagierende Proteine identifizieren weiterer Beleg für die Nützlichkeit dieses Ansatzes in der Charakterisierung von Proteinen mit unbekannter Funktion.
Protokoll
1. Generierung von Zelllinien: pMEP4 Transfektion / expression
- Transfektion von Zellen mit dem Expressionsvektor pMEP4 und wählen Sie mit 100 ug Hygromycin B / ml (Roche), bis alle mock-transfizierten Zellen getötet wurden ausgeschaltet. Die pMEP4 Plasmid episomal so gibt es keine Notwendigkeit, spezifische Klone zu isolieren gehalten.
- Zellen, welche die pMEP4 Vektor kann durch Behandlung mit 10 uM CdCl 2 für 16 Stunden induziert werden. Expression und Induzierbarkeit sollte im kleinen Maßstab vor der Verstärkung der Zelllinien bestätigt werden. Der TAP-markierte Protein kann leicht nachgewiesen werden, da die Protein-G-Domänen an Antikörper aus fast allen Arten. Für großflächige Reinigungen in der Regel 10 konfluente 175 cm 2-Kolben von Zellen erforderlich sind. Dies entspricht etwa 2 X10 8 exprimierenden Zellen.
2. Zelllysate Herstellung
- Kratzen Sie Zellen in PBS und kombinieren in einem einzigen 50 ml Tube. Spin 1200X gfoder 5 Minuten (dies sollte in 2-3 ml gepackten Zellen zur Folge haben, mit zu beginnen, das reduziert sich auf etwa 1,5 ml nach dem Waschen).
- Waschen Sie die Zellen dreimal in eiskaltem PBS (50 ml jedes Mal).
- Lyse Zellen in 5 ml Lysepuffer (50 mM Tris-HCl (pH 7,5), 125 mM NaCl, 5% Glycerin, 0,2% NP-40, 1,5 mM MgCl 2, 25 mM NaF, 1 mM Na 3 VO 4 und Protease-Inhibitor ). Hinweis: NaF, Na 3 VO 4 und Protease-Inhibitoren sollte hinzugefügt werden frisch. Und Abpipettieren 10-mal, bevor er nach 5 Minuten auf Eis. Spritze auf und ab 5-10 mal mit einer stumpfen Nadel, bevor er wieder auf Eis für 5 Minuten auf Eis. Wiederholen Sie Ausspritzen und lassen für weitere 5 Minuten auf Eis.
- Frost-Tau-Lysat zweimal (flüssigem N 2 / oder Trockeneis und Ethanol). Nicht Probe mehr als 4 ° C zu erreichen Hinweis: Sie können dieses Beispiel bei -80 ° C lagern, bis Sie Zeit zu verarbeiten haben.
- Aliquoten in 1,5 ml Röhrchen und entfernen lysierten Zellen und debris durch Zentrifugation (10 Minuten bei 4 ° C, 16.000 × g).
- Recover Überstand, in einem 15 ml Falcon Röhrchen verbinden und (optional) durch einen 0,45 um Filter zu passieren, bevor sie eine 20 ul Probe für die Ausbeute an Protein Quantifizierung. Entfernen Sie weitere 50 ul-Aliquot für die anschließende Analyse (Probe 1).
3. Die Bindung an Kaninchen-IgG-Agarose
(Hinweis: Alle Spins sollte bei 1200X g durchgeführt werden in einer gekühlten Zentrifuge bei 4 ° C für 1 Minute, wenn nicht anders angegeben)
- Vorsichtig resuspendieren Kaninchen-IgG-Agarose-Lösung (Sigma-Aldrich) durch Verwirbelung Flasche. Nehmen Sie 380 ul IgG Agarose (bei einem Cutoff 1ml Pipettenspitze) lösen und Versand / Konservierungsmittel Lösung durch Zentrifugation für 1 Minute. Waschen der Agarose dreimal in gekühltem Lysepuffer (4 ° C) durch Zentrifugation geklärt. Die endgültige Ausbeute von Agarose sollte etwa 250 ul verpackt Perlen sein.
- Fügen Sie die gelöscht Zelllysat von 2,6 (in einem 15 ml-Falcon-Wannee) zur gewaschenen Kaninchen-IgG Agarose und Inkubieren für 3 Stunden (oder über Nacht) bei 4 ° C unter Verwendung eines rotierenden Mischers.
- Spin-Down der Agaroseperlen für 5 Minuten bei 4 ° C und entfernen Sie den Überstand für die anschließende Analyse (Probe 2). Hinweis: Die TAP-markierte Protein sollte nun mit den Perlen in Verbindung gebracht werden.
4. TEV-Protease-Spaltung
(Hinweis: Alle Spins sollte bei 1200X g durchgeführt werden in einer gekühlten Zentrifuge bei 4 ° C für 1 Minute, wenn nicht anders angegeben)
- Waschen Sie die Kaninchen-IgG-Agarose-Beads dreimal in gekühltem Lysepuffer (4 ° C) durch Zentrifugation geklärt (Hinweis: Die Lyse-Puffer sollten keine Protease-Inhibitoren). Es sollte darauf geachtet nicht zu entfernen oder verlieren alle Perlen während der Waschschritte werden.
- Waschen der Kügelchen noch zweimal mit TEV-Protease Spaltpuffer (10 mM Tris-HCl (pH 7,5), 100 mM NaCl und 0,2% NP-40) durch Zentrifugation geklärt. Nach dem letzten Waschen sorgfältig entfernen liquid von den Kügelchen.
- Bereiten Sie eine TEV-Protease-Spaltung Mix; für jede Probe enthalten 467,5 ul H 2 O, 25 ul 20x TEV Buffer (Invitrogen), 5 ul 0,1 M DTT und 2,5 ul (25 U) TEV-Protease (Invitrogen). Fügen Sie 500 ul dieser TEV-Spaltung Mix zu jeder Probe von Kügelchen verpackt und überweisen Sie den gesamten Mix zu einer 1,7-ml-Tube geschmiert (Costar). Vor der Inkubation Entfernen eines 30 ul Aliquot für die anschließende Analyse (Probe 3).
- Inkubieren Sie die TEV-Protease-Reaktion über Nacht bei 4 ° C unter Verwendung eines rotierenden Mischers. Beachten Sie, dass kürzere Zeiten verwendet Inkubationen können je nach Art des Bait-Protein und die Zugänglichkeit der TEV-Protease-Spaltstelle, aber der minimale Inkubationszeit werden sollte empirisch bestimmt werden.
5. Bindung an immobilisiertes Streptavidin Plus-Perlen Ultralink
(Hinweis: Alle Spins sollte bei 1200X g durchgeführt werden in einer gekühlten Zentrifuge bei 4 ° C für 1 Minute, wenn nicht anders angegeben)
- Zentrifugieren der TEV-Spaltung, die die Kaninchen-IgG Agarosekügelchen für 5 Minuten bei 1200X g (4 ° C). Nehmen Sie einen 20 ul Aliquot der geklärte Überstand zur Analyse (Probe 4)
- Übertragen Sie die verbleibende Überstand (etwa 480 ul) in ein frisches 1,5-ml-Tube geschmiert (Costar) und lassen Sie diese auf Eis. Werfen Sie diesen Überstand - das enthält Ihre TEV gespalten Köderprotein und alle zugehörigen bindenden Proteinen.
- Fügen Sie 500 ul gekühlt Lysepuffer (Abschnitt 2.3) zu den übrigen Kaninchen-IgG-Agarose-Beads und resuspendieren. Zentrifuge dieser Mischung, um die Kügelchen vor dem Entfernen des Überstandes und die Kombination mit dem aus Schritt 5.2 klären. Lassen Sie dieses Röhrchen auf Eis (es sollte nun ~ 980 ul Probe).
- Behalten Sie die Kaninchen-IgG-Agarose-Beads für die anschließende Analyse (Probe 5). Hinweis: Wenn Sie dieses Beispiel auf einem SDS-PAGE-Gel (oder Western-Blot) startet, benötigt es eine Menge Hintergrund aufgrund der Anwesenheit der schweren und leichten IgG gebenKetten.
- Inzwischen vorsichtig resuspendieren Ultralink immobilisiertes Streptavidin Plus-Agarose-Beads (Pierce) durch Schwenken der Flasche. Mit Hilfe eines 200 pl Pipettenspitze mit dem Ende geschnitten, 70 ul Aliquot Streptavidinbeads pro Probe in die Vor-geschmiert 1,5 ml Röhrchen und entfernen Versand / Konservierungsmittel Lösung durch Zentrifugieren. Hinweis: diese Perlen sind sehr klein und so "Duck-billed" oder flach / spitz zulaufenden Spitzen können verwendet werden, um Beadverlust beim Waschen zu minimieren.
- Waschen der Streptavidin-Kügelchen dreimal in Lysepuffer gekühlt (4 ° C) durch Zentrifugation geklärt. Dies sollte zu verlassen ca. 45-50 ul verpackt Perlen pro Röhre.
- Übertragen der TEV-Protease-Schnittstelle Überstand (dh der 980 ul Probe aus Schritt 5.3) auf das Rohr mit der gewaschenen Streptavidin-Kügelchen und Inkubation für 3 Stunden (oder über Nacht) bei 4 ° C unter Verwendung eines rotierenden Mischers.
- Spin-Down der Streptavidinbeads für 5 Minuten bei 4 ° C und entfernen Sie den Überstand für die anschließende Analyse (SamPLE 6). Waschen der Streptavidin-Kügelchen dreimal in Lysepuffer gekühlt (4 ° C) durch Zentrifugation geklärt. Nach dem letzten Waschen, sorgfältig entfernen Sie alle verbleibenden Waschpuffer.
6. Biotin Elution der Streptavidin bindende Peptid-und Protein-Köder
(Hinweis: Alle Spins sollte bei 1200X g durchgeführt werden in einer gekühlten Zentrifuge bei 4 ° C für 1 Minute, wenn nicht anders angegeben)
- Eluiert markierte Proteine aus den Streptavidin-Kügelchen durch Zugabe von 500 ul PBS: Biotin (1 mM D-Biotin) und Inkubieren bei 4 ° C für 3 Stunden (oder über Nacht) unter Verwendung eines rotierenden Mischers.
- Spin-Down der Streptavidinbeads für 5 Minuten bei 4 ° C und entfernen Sie den Überstand in ein 1,5-ml-Tube geschmiert (Hinweis: Dies ist Ihre letzte Probe / Elution (Probe 7).
- Fügen Sie eine weitere 500 ul Biotin: PBS zu den übrigen Streptavidinbeads und inkubieren Sie bei 4 ° C für 2-3 Stunden (oder über Nacht) mit Hilfe eines rotierenden Mischers.
- ReTorf Schritt 6,2 und verbindet die beiden Elutionen (Probe 7). Dieser abschließende Elution kann bei -80 ° C gelagert werden
- Um die Effizienz der Biotin Elution beurteilen, frieren die restlichen Streptavidinbeads und später in SDS-Probenpuffer (Sample 8) (Empfehlungen: LaneMarker 5x reduzierendem Probenpuffer von Fisher) kochen.
7. Proteinkonzentration
- Die letzten Eluats (Beispiel 7) muss vor der Analyse wegen der geringen Proteinkonzentration und relativ hohen Volumen konzentriert werden. Dies kann mit niedrigem Molekulargewicht (<5 kDa) Vivaspin Spin Columns (Vivascience). Ziel, um das Volumen zu konzentrieren auf weniger als 100 ul. Diese letzte Probe gekocht und gelagert werden Protein-Probenpuffer (Empfehlungen: LaneMarker 5x reduzierendem Probenpuffer von Fisher).
8. Analyse
- Beispiele 1-8 können durch Western-Blot analysiert werden, um die Effizienz der Pull-Down-zu bestimmen. Hinweis: Die meisten sekundären AntikörperProtein mit den G-Domänen des Fusionsproteins kreuzreagieren aber nach TEV-Protease-Spaltung entfernt.
- Probe 7 kann durch 1D oder 2D-SDS-PAGE analysiert werden. Die Färbung kann mit Silber oder Coomassie (Empfehlungen: SilverQuest Silberfärbung und Kolloidale Coomassie-Färbung blau-Kits von Invitrogen) werden. Proteinidentifizierung kann mit Hilfe der Massenspektrometrie werden.
9. Repräsentative Ergebnisse
Ein Beispiel 1D SDS-PAGE-Analyse der endgültigen Elution (Beispiel 7) von diesem Protokoll, um Bindungspartner der TAP markiert eIF4E zu identifizieren, ist in 2 vorgesehen. Dieser Vertreter Gel spiegelt das komplexe und reiche Natur des eIF4E Interaktionen mit anderen Proteinen in der Zelle. Vergleich mit der Negativkontrolle auch in 2 gezeigt, aus einer Zelllinie, die nur die TAP-Tag generiert, zeigt die Spezifität dieses eIF4E Pull-Down-Köder. In diesem Fall 15% der konzentrierten endgültige Elution (Probe7) wurde durch 1D SDS-PAGE unter Verwendung von kommerziell erhältlichen vorgefertigten Gradientengelen, bevor sie gefärbt mit dem Silverquest Silberfärbung Kit von Invitrogen analysiert.
Typischerweise 50-85% des verbleibenden konzentrierten endgültige Elution (Beispiel 7) mit kolloidalem Coomassie-Färbung (Invitrogen) analysiert. Die Proben (Gelscheiben / Bänder) aus dem gesamten Bahn wurden dann extrahiert und mittels Massenspektrometrie analysiert.
Proteine in dem Pull-Down-eIF4E identifiziert wurden gegen die Bindungspartner aus der negativen Kontrolle auf nicht-spezifische Bindungspartner zu identifizieren filtriert. Die endgültigen Proteine identifiziert mit diesen Verfahren TAP Tagging eIF4E kann in 2, die repräsentativ für die normale Proteine identifiziert unter Verwendung dieser Technik ist, aufgefasst werden. Darüber hinaus wurden eine Reihe von eIF3 Untereinheiten ebenfalls identifiziert (Daten nicht gezeigt).

Abbildung 1. Schematische Darstellung derTandem-Affinitätsreinigung Verfahren. Die sechs Schritt Tandem-Affinitätsreinigung (TAP)-Protokoll beinhaltet Zelllinie Generation, Zelllyse, Kaninchen IgG Agarose Immunpräzipitation, TEV-Protease-Spaltung, Streptavidin Wulst Affinitätsreinigung und schließlich Biotin Elution.
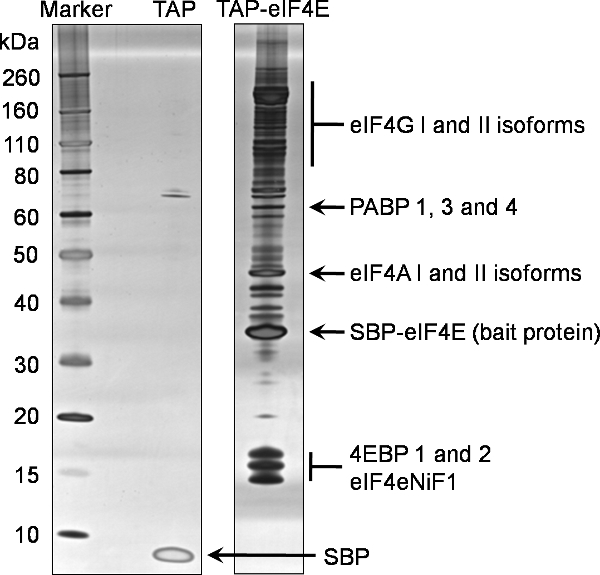
Abbildung 2. Tandem Affinitätsreinigung des murinen Proteins eIF4E. Interagierenden Partner des N terminal markierte TAP eIF4E wurden aus eukaryontischen HEK293-Zellen mit dem beigefügten Protokoll gereinigt. Ein 20% Anteil der endgültigen Elution (Beispiel 7) wurde durch SDS-PAGE auf einem vorgefertigten 4-12% Gradientengel (TAP-eIF4E Spur) analysiert. Eine entsprechende Analyse wurde für das Tag allein (TAP Spur) durchgeführt. Die Proteine wurden identifiziert mittels Silberfärbung. Proteine im Massenspektrometer der gleichen Probe identifiziert werden auf diesem Gel hervorgehoben.
Abkürzungen: eIF4G; eukaryotischen Translations-Initiations-fSchauspieler Gamma 4, PABP; polyA-bindendes Protein, eIF4A: eukaryotischen Translations-Initiations-Faktor 4 alpha, SBP-eIF4E, die restlichen TAP Köderprotein mit dem Streptavidin-bindende Peptid fusioniert mit dem eukaryotischen Translations-Initiations-Faktor 4E, 4EBPs; eukaryotischen Translations-Initiations-Faktor 4E verbindlich Proteine, eIF4eNiF1l Eukaryoteninitia-4E Kernimport Faktor 1, SBP, die restlichen Streptavidin-Bindungspeptid aus der TEV-fusionierten TAP-Peptid.
Diskussion
Die TAP-Tagging-Technik hier dargestellten demonstriert eine hochspezifische und strengen Verfahren zur Isolierung die Bindungspartner der Köder Proteine in eukaryotischen Zellen. Dieser Ansatz kann sowohl zelluläre und virale Proteine angewandt werden. Nach unserem Wissen ist dies das erste Mal eine solche Technik zum Translationsinitiationsfaktors eIF4E aufgebracht worden ist. Bezeichnung des bekannten eIF4E bindende Proteine eIF4G und den 4EBPs mit dieser Technik bestätigt die Gültigkeit eines solchen Ansatzes. Darüber hinaus ist die Identifizierung der verbleibende Anteil des komplexen eIF4F bestätigt nämlich eIF4A und PABP, dass indirekte Wechselwirkung und tertiären Komplexe intakt während des Reinigungsprozesses bleiben. Die Identifizierung von mehreren Isoformen der kanonischen eIF4E bindenden Proteinen zeigte sich auch. Diese werden im Detail in 2 beschrieben.
Im Hinblick auf die Grenzen des Ansatzes, sollte gewisse Sorgfalt im Hinblick auf die Wahl genommen werdender Bait-Protein und ob die TAG-Tag am N-Terminus oder C-zu platzieren. Es könnte sinnvoll sein, um einen funktionellen Test durchführen oder untersuchen die Lokalisation des Fusionsproteins mittels Mikroskopie vor dem Endwert Reinigung, um sicherzustellen, das markierte Derivat ist funktional. Integrale Membranproteine oder nukleare Proteine können nicht unbedingt von den relativ milden Bedingungen in der Lyse-Schritt beschrieben freigegeben werden. Wie bei allen Immunpräzipitationen und ähnliche Pulldown-Assays, unbefugte Änderungen der Art und Konzentration des Waschmittels in dem Lyse-Puffer, um die Effizienz der Lyse zu erhöhen werden. Die Ionenkonzentration der Lyse und Waschpuffer kann auch modifiziert werden, um zu erhöhen oder zu verringern, die Stringenz der Reinigung werden. Es ist auch möglich, die anfängliche Reinigung unter Standardbedingungen milden Bedingungen (oben beschrieben) durchzuführen, aber dann teilt die Streptavidin-Kügelchen (Abschnitt 5.8) in 4-5 Aliquots, die anschließend unter zunehmender Ionenstärke con gewaschen werden könnenbedingungen. Dies kann dem Benutzer ermöglichen, die optimalen Bedingungen, welche die nicht-spezifische interagierende Proteine zu entfernen, zu identifizieren. Der gesamte Prozess kann auch unter Verwendung transienter Transfektion, wenn die interagierenden Partner sind bekannt und ihre Anwesenheit oder Abwesenheit von nachfolgenden Western-Blot nur bestimmt werden. In diesem Fall würden wir vorschlagen, zwei 100cm 2 Gerichte von Zellen mit je 8 pg Expressionsplasmid transfiziert. Dieses modifizierte Verfahren ist von besonderem Nutzen bei der Prüfung der Fähigkeit der mutierten Derivate des TAP-Fusionsprotein mit den bekannten Bindungspartner wechselwirken.
Analyse von Proben 1 bis 8 auf Silber gefärbten SDS-PAGE-Gelen (oder durch Western-Blot, wenn ein Antikörper zur Verfügung steht) ist eine hervorragende Möglichkeit der Fehlerbehebung, sollte die Technik versagen, um akzeptable Ergebnisse zu generieren. Die Effizienz der einzelnen Affinität basierte Niederschlag und spezifische Elution kann unter Verwendung dieses systematischen Stichprobenverfahren werden. Die Proben 1-8 sollte während der Optim genommen werdensierung des Protokolls für jede neue Köder.
Die TAP-Tagging-Technik bietet eine leistungsstarke und robuste Alternative zu anderen Ansätzen wie GST-Pull-downs, Hefe-Zwei-Hybrid-Assays usw. Die doppelte Affinitätsreinigung und spezifische Elutionsschritte (TEV-Spaltung und Biotin Elution) Spezifität und Stringenz sorgen bei einer aufrecht erhalten werden hohem Niveau über die Reinigungsprozedur. Kritisch kann diese Technik auf jedes Protein angewendet werden und stellt somit eine ausgezeichnete Methode zur Identifizierung von Bindungspartnern für ein Zielprotein von Interesse.
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Diese Arbeit wurde durch eine Wellcome Trust Senior Fellowship verliehen an Dr. Ian Goodfellow finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | |
| Hygromycin B | Roche | 10843555001 | |
| Rabbit IgG Agarose | Sigma | A2909 | |
| AC-TEV-Protease | Invitrogen | 12575-015 | |
| Protease-Inhibitor-Cocktail | Calbiochem | 539134 | |
| Ultralink immobilisiertes Streptavidin Plus-Perlen | Durchstechen | 53116 | |
| Vivaspin 500 Zentrifugenkonzentrator (5 kDa) | Vivaspin | V50112 | |
| SilverQuest Silberfärbungskit | Invitrogen | LC6070 | |
| Novex Kolloidale Coomassie Blau Färbekit | Invitrogen | LC6025 | |
| 1,7 ml Röhrchen vorgeschmiert | Costar | 3207 | |
| Mikrokapillare Pipettenspitzen | VWR | 37001-150 | |
| NuPage 4-12% Bis-Tris-Gradientengelen | Invitrogen | NP0322BOX | |
| CdCl 2 | Sigma | 202908 | |
| 5x SDS-Probenpuffer | Fischer | PN39000 |
Referenzen
- Williamson, M. P., Sutcliffe, M. J. Protein-protein interactions. Biochem. Soc. Trans. 38 (4), 875-878 (2010).
- Rigaut, G. A generic protein purification method for protein complex characterization and proteome exploration. Nat. Biotechnol. 17 (10), 1030-1032 (1999).
- Blackwell, C., Brown, J. D. The application of tandem-affinity purification to Candida albicans. Methods Mol. Biol. 499, 133-148 (2009).
- Cochrane, A. Stable complex formation between HIV Rev and the nucleosome assembly protein, NAP1, affects Rev function. Virology. 388 (1), 103-111 (2009).
- Fernandez, E. Targeted tandem affinity purification of PSD-95 recovers core postsynaptic complexes and schizophrenia susceptibility proteins. Mol. Syst. Biol. 5, 269-269 (2009).
- Gloeckner, C. J. Tandem affinity purification of protein complexes from mammalian cells by the Strep/FLAG (SF)-TAP tag. Methods Mol. Biol. 564, 359-372 (2009).
- Holowaty, M. N. Protein profiling with Epstein-Barr nuclear antigen-1 reveals an interaction with the herpesvirus-associated ubiquitin-specific protease HAUSP/USP7. J. Biol. Chem. 278 (32), 29987-29994 (2003).
- Burckstummer, T. An efficient tandem affinity purification procedure for interaction proteomics in mammalian cells. Nat. Methods. 3 (12), 1013-1019 (2006).
- Goodfellow, I. Calicivirus translation initiation requires an interaction between VPg and eIF 4 E. EMBO Rep. 6 (10), 968-972 (2005).
- Goodfellow, I. G., Roberts, L. O. Eukaryotic initiation factor 4E. Int. J. Biochem. Cell Biol. 40 (12), 2675-2680 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten