Method Article
Termodinámica del plegado de proteínas de membrana medido por espectroscopia de fluorescencia
En este artículo
Resumen
Este artículo detalla el procedimiento de vídeo experimental para la obtención de la energía libre de Gibbs del plegamiento de las proteínas de membrana de triptófano fluorescencia.
Resumen
Plegamiento de proteínas de membrana es un tema emergente con un significado fundamental y relacionadas con la salud. La abundancia de proteínas de membrana en las células subyace la necesidad de un estudio integral del plegamiento de esta familia de proteínas en todas partes. Además, los avances en nuestra capacidad para caracterizar las enfermedades asociadas con las proteínas mal plegadas, han motivado importantes esfuerzos experimentales y teóricos en el campo de plegamiento de proteínas. Los rápidos avances en este campo tan importante es, por desgracia obstaculizado por los problemas inherentes asociados con las proteínas de membrana y de la complejidad del mecanismo de plegado. Aquí, se describe un procedimiento experimental para la medición de la propiedad termodinámica de la energía libre de Gibbs se desarrolla en ausencia de desnaturalizante, Δ G ° H 2 O, una proteína de membrana integral representante de E. coli. Este protocolo se centra en la aplicación de la espectroscopia de fluorescencia para determinar las poblaciones de equilibrio de los estados plegado y desplegado en función de la concentración de desnaturalizante. Consideraciones experimentales para la preparación de vesículas de lípidos sintéticos, así como pasos clave en el procedimiento de análisis de datos se destacan. Esta técnica es muy versátil y puede llevarse a cabo con diferentes tipos de desnaturalizante, incluyendo la temperatura y pH, así como en diversos entornos de plegado de los lípidos y las micelas. El protocolo actual es el que puede ser generalizado a cualquier membrana o proteínas solubles que se reúne el conjunto de los criterios expuestos.
Protocolo
1. Preparación de ~ 50 nm de diámetro pequeño vesículas unilaminares (SUV) de plegado de proteínas de membrana
- Una solución de 1,2-dimiristoil-sn-glicero-3-fosfocolina (DMPC) lípidos en cloroformo se compra y alícuotas en viales de vidrio limpia en cantidades de 20 mg por vial para el almacenamiento. Una capa de gas nitrógeno, se añade a cada vial para evitar la oxidación de lípidos, y los viales están sellados con tapas y parafina. Los viales se almacenan en un congelador a -20 ° C hasta su uso.
- Un solo frasco que contiene una alícuota de 20 mg de lípidos en cloroformo se utiliza para cada experimento. El contenido de la ampolla se seca con una corriente de gas nitrógeno durante 1 hora hasta que ya no sigue siendo solvente.
- Lípidos secos se resuspendieron en 1 ml de 20 mM de tampón fosfato de potasio (pH = 7,3) con un baño de ultrasonido de agua para aproximadamente 30 segundos. Esta solución de lípidos suspendido aparecerá nublado, y se transfiere a un plástico de 15 ml del tubo con un fondo cónico. Otra alícuota de 1 ml de solución amortiguadora de fosfato se añade a la ampolla de vidrio vacía que contenía originalmente los lípidos, y sonicación se repite, la solución se añade a la misma de 15 ml tubo. Este proceso se repite un total de 4 veces hasta que el volumen final en el tubo es de 4 ml, resultando en una concentración de lípidos de 5 mg / mL de esta solución de vesículas de valores.
- El volumen de 4 ml de solución de lípidos vesícula se coloca en un lugar cálido (30 ° C) baño de agua, y se sonicaron utilizando un micropunta ultrasonicador durante 1 hora a 50% del ciclo. El propósito del baño de agua es doble. En primer lugar, evita que la solución de lípidos se vuelva demasiado caliente debido al proceso de sonicación. En segundo lugar, el baño caliente asegura que la temperatura de la solución acuosa de lípidos se mantiene por encima de la temperatura durante la fase de transición de dos capas sonicación, por DMPC, esta temperatura de transición es de ~ 23 ° C.
- La solución sonicado se pasa a través de una jeringa de 0,22 micras filtro para eliminar los restos de la punta sonicador, y esta solución filtrada se equilibra la noche a 37 ° C.
2. Preparación de muestras para la curva inicial de fluorescencia Despliegue
- Una solución stock de 10 M de urea en 20 mM tampón fosfato (pH 7,3) se hace. Es esencial que el agua se añade a la sólida (y no al revés), ya que la gran cantidad de urea ocupa un volumen importante en la solución. Esta solución de urea se puede hacer mediante la adición de solutos a una botella limpia, vacía y añadir agua hasta el volumen final previsto. Esta solución puede requerir un calentamiento en un baño de agua o sobre una plancha caliente con agitación para disolver el soluto. La urea es higroscópica y por lo tanto, la concentración real de la solución de urea debe ser determinada mediante el índice de refracción 1.
- Una solución de reserva de estabilización de fosfato 20 mM (pH 7,3) se hace.
- Una solución de reserva de proteínas desplegadas se prepara. Esta solución de proteínas de reserva debe tener una concentración de proteína M ~ 200 en urea 8 M, 20 mM tampón fosfato.
- Las muestras para los estudios de fluorescencia se preparan de la siguiente manera. Los volúmenes adecuados de proteína de valores (sección 2.3), solución de reserva de lípidos (5 mg / mL de lípidos, la sección 1), las acciones de solución de urea 10 M (sección 2.1), y la Reserva de Estabilización de fosfato (sección 2.2) se combinaron para hacer de las muestras que contienen ~ 4 proteína M, 1 mg / mL de lípidos, y de 0 a 8 M urea en incrementos de 1 M. El volumen total de cada muestra es de 200 mL. Una hoja de ejemplo (tabla 1) se incluye que los volúmenes de listas requiere el uso de 200 acciones M solución de proteína.
- Muestras en blanco se hacen como se describe en la sección 2.4, sin embargo, la proteína no se añade. En lugar de proteínas, 4 l de una solución de urea 8 M se puede añadir que las concentraciones de otros componentes son idénticos a los de la sección 2.4. Estos espacios en blanco se utiliza para restar la dispersión y la señal de fondo otra que aparece en el espectro de fluorescencia de la proteína.
- Las muestras de las secciones 2.4 y 2.5 se permite que se incuba a 37 ° C durante al menos 2 horas antes de la medición de los espectros de fluorescencia para asegurar plegado completo y equilibrado.
3. Medición de los espectros de fluorescencia
Fluorescencia de triptófano de cada muestra y el blanco se mide utilizando un fluorómetro de estado estacionario. Espectros de fluorescencia debe registrarse con longitud de onda de excitación de 290 nm para evitar la excitación de los residuos de tirosina, y escaneado 305 a 500 nm. Usual de ingreso y paso de banda de salida es de 3 nm. El incremento de longitud de onda y el tiempo de integración puede ser optimizado para la relación señal-ruido. Los valores típicos para el incremento de longitud de onda y el tiempo de integración 1 nm / paso y 0.5 seg / paso, respectivamente. Proteínas de la membrana en general, se pliegan en lípidos sintéticos sólo cuando la temperatura está por encima de la temperatura de la fase de transición de dos capas. Por lo tanto, en este caso de DMPC, la temperatura de la muestra se mantiene constante a 30 º C. Una cubeta microvolumen sílice fundido con capacidad de 160 l se utiliza en estos exexperimentos.
4. Generación de la curva inicial de despliegue y la estimación de la energía libre de Gibbs de desplegamiento de proteínas de membrana
- Espectros de fluorescencia en la forma de un conjunto de datos xy se cargan en el software como Igor Pro, Matlab, origen, o Excel.
- Señal desde el fondo de la vesícula urea y de los lípidos se debe restar mediante el siguiente procedimiento:
Espectro de A = espectro de fluorescencia de la proteína cruda en la presencia de vesículas de lípidos y la concentración específica de la urea.
Espectro B = espectro de fluorescencia de crudo de la muestra en blanco correspondiente que contiene vesículas de lípidos y la concentración de urea mismo que en el espectro de A, no es la proteína en las muestras en blanco.
Espectro corregido = Espectro A - C * espectro B. C es un escalar por lo general igual a 1,0. En algunos casos, C es inferior o superior a 1,0 debido a problemas de dispersión que causa sobre o sub-resta cerca de la región 305-320 nm ~. - La longitud de onda del máximo de fluorescencia, λ max, está tabulada de los espectros corregidos para cada concentración de urea. Un valor típico para desplegada completamente proteína de la membrana es de 350 nm, mientras que la proteína plegada es de ~ 330 nm. Longitudes de onda tabulados se convierten en la fracción se desarrolló mediante la conversión de la gama de valores λ max (~ 330 a ~ 350 nm) para el rango de 0,0 a 1,0 para correlacionar el valor de λ max a la fracción de la población se desarrolló. Por ejemplo, un valor máximo de λ de 330 nm corresponde a 0,0, 350 nm corresponde a 1,0 y 340 nm corresponde a 0,5 en términos de la fracción se desarrolló. Esta fracción de la proteína desplegada se traza contra la concentración de urea. Como se mencionó anteriormente, la concentración de urea se debe determinar experimentalmente midiendo el índice de refracción.
- Suponemos que la proteína puede existir en dos estados: plegar o desplegar. La trama de la fracción se desarrolló, f, frente a la concentración de urea, C, puede estar en forma para la ecuación: 2

El coeficiente m corresponde a la tasa de cambio de la energía libre con respecto a la concentración de desnaturalizante, y C m corresponde a la concentración de urea en el punto medio en el que la población es igual a doblado la población se desarrolló. Estos valores son las variables de ajuste obtenidos a partir de los mínimos cuadrados instalación de software de análisis de datos. R (0.001987 kcal / mol • K) es la ley constante de los gases, y T es la temperatura (303 K). - Los coeficientes obtenidos del ajuste se utilizan para determinar la energía libre de Gibbs (kcal / mol) de desarrollo en ausencia de urea:
 .
.
5. Despliegue optimizado para la curva de medición más precisa de la energía libre de Gibbs de despliegue.
- La curva de desarrollo descritas anteriormente revela el rango de concentraciones de urea que provoca el mayor cambio en el máximo de λ. Este rango es generalmente pequeña, y la mayoría de despliegue se produce dentro de este rango pequeño. Con el fin de determinar con precisión la energía libre de desarrollo, se analizan muestras adicionales en esta región para obtener una gráfica que contiene muchos puntos de datos necesarios para la optimización de mínimos cuadrados de montaje. Por ejemplo, si la proteína se desarrolla entre 2 y 4 M de urea, las muestras se puede decir que contiene 2 a 4 M de urea en incrementos de 0,2 M a revelar con mayor precisión la forma de esta importante región de la curva de desarrollo. No es necesario medir el espectro en blanco en cada concentración de urea, sino que es suficiente para medir un espectro blanco cada paso ~ 0,5 M.
- La energía libre de desarrollo obtenidos a partir de esta trama puede diferir ligeramente de los obtenidos en la curva original desarrollo, sino que refleja un valor más preciso.
6. Los resultados representativos:
| Muestra | final [urea], M | proteína de acciones (l) | de valores de lípidos (l) | stock 10 M urea (l) | reserva de estabilización (l) |
| 1 | ~ 0,16 | 4 | 40 | 0 | 156 |
| 2 | 1 | 4 | 40 | 20 | 136 |
| 3 | 2 | 4 | 40 | 40 | 116 |
| 4 | 3 | 4 | 40 | 60 | 96 |
| 5 | 4 | 4 | 40 | 80 | 76 |
| 6 | 5 | 4 | 40 | 100 | 56 |
| 7 | 6 | 4 | 40 | 120 | 36 |
| 8 | 7 | 4 | 40 | 140 | 16 |
Tabla 1. Volumen de solución madre necesaria para hacer muestras de fluorescencia

Figura 1. Triptófano espectros de fluorescencia de la proteína de ~ 5 M de membrana representante que contiene un único residuo de triptófano. (A) de los espectros de fluorescencia de la proteína cruda (espectro A partir de 4.2), (B) de los espectros de fluorescencia de crudo en blanco (espectro B de 4,2), (C) Se ha corregido espectro de 4,2.
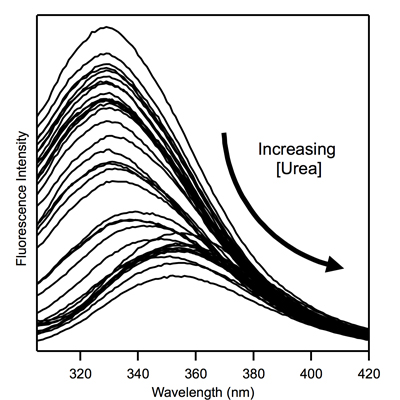
Figura 2. Corregido triptófano espectros de fluorescencia de la proteína de la membrana de la Figura 1 para las concentraciones de urea numerosas.
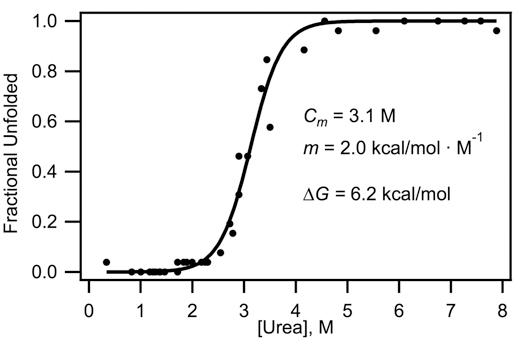
Figura 3. Despliegue curva obtenida a partir de datos en la figura 2, con ajuste a la ecuación en el 4,4. La energía libre de Gibbs se calcula de acuerdo con la sección 4.5.
Discusión
El actual protocolo describe la generación de curvas de desarrollo asociados a la membrana de las proteínas y los péptidos que contienen residuos de triptófano. En este caso, se asume que la fluorescencia de triptófano refleja si la proteína se dobla y se introduce en las vesículas de lípidos sintéticos, o se desarrolló en la solución. Supuestos adicionales, como el plegado de dos estados y la dependencia lineal de energía libre con la concentración de desnaturalizante, se hacen en el presente informe;. Modificación de estos supuestos, en consecuencia diferentes ecuaciones 2 El protocolo experimental se pueden modificar fácilmente para utilizar diferentes observables espectroscópicas, tales como dicroísmo circular, absorción o fluorescencia de un colorante extrínseca, 3, así como la electroforesis en gel. 4 Además, desnaturalizantes diferentes, tales como guanidinio, soluciones ácidas, o la temperatura, así como los lípidos alternativa / vesículas, como los lípidos aniónicos, grandes vesículas unilamelares, o micelas, pueden ser utilizados. 5,6 Finalmente, el protocolo puede ser aplicado a cualquier proteína de la membrana, el péptido asociado a membrana, y el péptido proteína soluble / que cumple con los criterios de plegado reversible, y muestra que la espectroscopia o de otro tipo marcadores observables de diferentes estados conformacionales. En el caso de las proteínas solubles, las vesículas son, evidentemente, omitido en el protocolo.
Energía libre de Gibbs de desarrollo ofrece información sobre las interacciones no covalentes que estabilizan las biomoléculas. En nuestro laboratorio, hemos medido las curvas de desarrollo para los mutantes de la proteína de membrana externa A (OmpA) que contienen un solo residuos de triptófano en diferentes lugares, e informó de que los mutantes se desestabilizaron en relación con la proteína de tipo salvaje que contiene cinco residuos de triptófano nativa 7. Estos estudios se complementan con desarrollo de otras técnicas, como la espectroscopia vibracional, para aclarar los detalles moleculares de, por ejemplo, la vinculación del hidrógeno y las interacciones de pares que subyacen a la variación en la estabilidad. En resumen, la técnica relativamente sencilla de las curvas de desarrollo se pueden aplicar a las membranas y proteínas solubles para revelar la termodinámica asociada con el plegamiento de proteínas.
Divulgaciones
Agradecimientos
Damos las gracias a Beijing Wu para el uso de sus datos. Este trabajo fue apoyado por un premio CARRERA NSF a JEK
Materiales
| Material Name | Type | Company | Catalogue Number | Comment |
|---|---|---|---|---|
| Name | Company | Catalog Number | Comments | |
| DMPC | Avanti Polar Lipids | 850345C | ||
| Urea | MP Biochemicals | 04821527 | ||
| Potassium Phosphate Dibasic | Fisher | P288 | ||
| Potassium Phosphate Monobasic | Fisher | P285 |
Referencias
- Shirley, B. A., Shirley, B. A. . Protein Folding and Stability. , 177-190 (1995).
- Pace, C. N. Determination and Analysis of Urea and Guanidine Hydrochloride Denaturation Curves. Methods Enzymol. 131, 266-279 (1986).
- Lau, F. W., Bowie, J. U. A Method for Assessing the Stability of a Membrane Protein. Biochemistry. 36, 5884-5892 (1997).
- Burgess, N. K., Dao, T. P., Stanley, A. M., Fleming, K. G. β-Barrel Proteins that Reside in the Escherichia coli Outer Membrane in Vivo Demonstrate Varied Folding Behavior in Vitro. J. Biol. Chem. 283, 26748-26758 (2008).
- Booth, P. J., Curnow, P. Folding scene investigation: membrane proteins. Curr. Opin. Struct. Biol. 19, 8-13 (2009).
- Hong, H., Tamm, L. K. Elastic coupling of integral membrane protein stability to lipid bilayer forces. Proc. Natl. Acad. Sci. U.S.A. 101, 4065-4070 (2004).
- Sanchez, K. M., Gable, J. E., Schlamadinger, D. E., Kim, J. E. Effects of Tryptophan Microenvironment, Soluble Domain, and Vesicle Size on the Thermodynamics of Membrane Protein Folding: Lessons from the Transmembrane Protein OmpA. Biochemistry. 47, 12844-12852 (2008).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados