JoVE Science Education
Microbiology
Sie haben vollen Zugriff auf diesen Inhalt durch
Nanyang Technological UniversityWachstumskurven: Erzeugen von Wachstumskurven mit Hilfe von koloniebildenden Einheiten und Messungen der optischen Dichte
Überblick
Quelle: Andrew J. Van Alst1, Rhiannon M. LeVeque1, Natalia Martin1, und Victor J. DiRita1
1 Department of Microbiology and Molecular Genetics, Michigan State University, East Lansing, Michigan, Vereinigte Staaten von Amerika
Wachstumskurven liefern wertvolle Informationen über die bakterielle Wachstumskinetik und Zellphysiologie. Sie ermöglichen es uns zu bestimmen, wie Bakterien unter variablen Wachstumsbedingungen reagieren, sowie optimale Wachstumsparameter für ein bestimmtes Bakterium zu definieren. Eine archetypische Wachstumskurve schreitet durch vier Wachstumsstadien: Verzögerung, Exponential, Stationär und Tod (1).
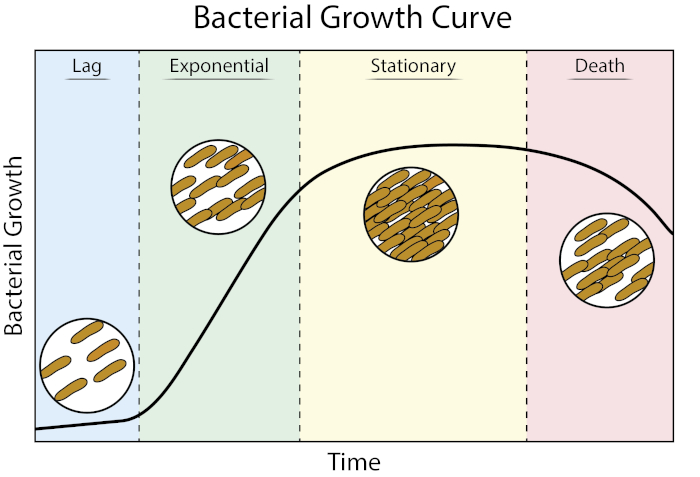
Abbildung 1: Bakterielle Wachstumskurve. Bakterien, die in der Batchkultur angebaut werden, schreiten durch vier Wachstumsphasen voran: Verzögerung, Exponential, stationär und Tod. Lag-Phase ist die Zeit, die es dauert, bis die Bakterien einen physiologischen Zustand erreichen, der in der Lage ist, schnelles Zellwachstum und Division zu erreichen. Die exponentielle Phase ist die Phase des schnellsten Zellwachstums und der schnellsten Zellteilung, in der DIE DNA-Replikation, die RNA-Transkription und die Proteinproduktion mit konstanter, schneller Geschwindigkeit erfolgen. Die stationäre Phase ist durch eine Verlangsamung und Auftimon des Bakterienwachstums aufgrund von Nährstoffbegrenzung und/oder toxischer Zwischenakkumulation gekennzeichnet. Die Todesphase ist das Stadium, in dem die Zelllyse als Folge einer starken Nährstoffbeschränkung auftritt.
Lag-Phase ist die Zeit, die es dauert, bis die Bakterien einen physiologischen Zustand erreichen, der in der Lage ist, schnelles Zellwachstum und Division zu erreichen. Diese Verzögerung tritt auf, weil es Zeit braucht, bis sich Bakterien an ihre neue Umgebung anpassen. Sobald die notwendigen zellulären Komponenten in der Verzögerungsphase erzeugt sind, treten Bakterien in die exponentielle Wachstumsphase ein, in der DNA-Replikation, RNA-Transkription und Proteinproduktion auftreten.
Verfahren
1. Einrichtung
- Erforderliche Labormaterialien: flüssige Medien, erstarrte Agarmedien, Erlenmeyerkolben, 15 ml Reagenzgläser, Phosphatgepufferte Saline (PBS), Bakteriellezellstreuer, 70% Ethanol und ein Spektralphotometer. Alle Lösungen und Glaswaren müssen vor der Verwendung sterilisiert werden.
- Bereiten Sie den Arbeitsplatz vor, indem Sie mit 70% Ethanol sterilisieren. Arbeiten Sie in der Nähe eines Bunsenbrenners, um eine Kontamination der Medien zu verhindern.
- Bei der Arbeit mit Bakterien sollten geeignete persönliche Schutzausrüstung und aseptische Technik verwendet werden. Bei der Arbeit mit Bakterienkulturen sind ein Labormantel und Handschuhe erforderlich.
- Rezepte für Puffer, Lösungen und Reagenzien
- Phosphatgepufferte Saline (PBS) (8).
- Luria-Bertani Broth (LB) (9).
2. Protokoll
- Vorbereitung von Medien
- Identifizieren Sie die Wachstumsmedien, mit denen die Bakterien wachsen und bereiten Sie sowohl flüssige Brühe als auch feste Agar (1,5% w/v Agar) Medien in separaten autoklavierbaren Flaschen. Hier wurden LB-Brühe und LB-Agar für das Wachstum von Escherichia colivorbereitet.
- Sterilisieren Sie die Medien mit einer halbfestgezogenen Kappe in einem Autoklaven, der 35 min auf 121 °C eingestellt ist.
- Für Agar-Medien, nach dem Autoklavieren, in einem Wasserbad auf 50 °C für 30 Minuten eingestellt, um abzukühlen. Nach dem Abkühlen 20-25 ml Agarmedien in 100x15mm runde Petrischalen gießen. Lassen Sie die Platten vor Gebrauch 24 Stunden bei Raumtemperatur einstellen.
- Erstvorbereitung von Bakterien
- Aus gefrorenen Beständen, StreifenBakterien für die Isolierung auf ausgewählten Medien Agar, um einzelne Kolonie Isolate zu erhalten. Inkubieren sie in Wachstumsbedingungen, die für die ausgewählten Bakterien zulässig sind. Hier wird E. coli auf LB-Agar gestreift und bei 37 °C über Nacht (16-18h) inkubiert.
- Wählen Sie mit einer sterilen Impfschleife eine einzelne Kolonie aus der Streifenplatte aus und impfen Sie 4 ml flüssige Medien in einem 15 ml Reagenzglas und wachsen unter Bedingungen, die für die ausgewählten Bakterien zulässig sind. Hier wird E. coli bei 37 °C angebaut, wobei über Nacht (16-18h) bei 210 U/min geschüttelt wird.
- Aufbau der Wachstumskurve
- Wachstumskolbenvorbereitung
- Autoklaven entsprechend dimensioniert Erlenmeyer Kolben. In der Regel wird ein Verhältnis von 1:5 von Medien zu Gesamtkolbenvolumen verwendet. Hier werden 100 ml LB-Medien in einem 500 ml Kolben verwendet.
- Mit einer serologischen Pipette sterile Medien auf den Erlenmeyerkolben übertragen.
- Vorbereitung der Verdünnungsserie
- Etikett ieren 15 ml Reagenzgläser: -1, -2, -3, -4, -5, -6, -7, -8 und -9, verteilen jeweils 9 ml PBS. Diese Zahlen entsprechen dem Verdünnungsfaktor, der zur Berechnung der KBE/ml verwendet wird. Für jeden Sammelzeitpunkt wird ein neuer Satz von Rohren benötigt. (Abbildung 2)
- Agar-Plattenvorbereitung
- Etikettenplatten mit Abholzeit und Verdünnungsfaktor. Für jeden Zeitpunkt gibt es eine Platte für jede Verdünnung.
- Wachstumskolbenvorbereitung
- Wachstumskurvenprotokoll
- Impfung der Medien
- Mit der über Nacht flüssigen Kultur im Rahmen von Schritt 2.2.2 vorbereitet, impfen Sie die Kolbenmedien mit 1:1000 Volumen der Kultur. Hier bei 100 ml LB-Medien werden 100 l über Nacht Flüssigkeitskultur zugegeben.
- Wirbeln Sie die Medien, um die Bakterien gleichmäßig zu verteilen.
- Timepoint-Auflistung
- Aufbau des Wachstumszustands
- Platzkolben in experimentellen Wachstumsbedingungen für die gegebenen Bakterien gewählt. Zeitpunkte sollten häufig für schnell wachsende Bakterien genommen werden und können in längeren Intervallen für langsam wachsende Bakterien eingenommen werden. Hier wird E. coli bei 37°C angebaut, wobei das Schütteln bei 210 Umdrehungen pro Minute (Rpm) und zeitpunkte alle 1 Stunde genommen wird.
- Messung der optischen Dichte (OD600)
- Zu jedem Zeitpunkt, einschließlich des Startzeitpunkts (t = 0), 1 ml Bakterienkultur zurückziehen und in eine Spektralphotometer-Küvette geben.
- Wischen Sie die Küvette sauber und zeichnen Sie die optische Dichte bei 600 nm Wellenlänge auf. Wenn die optische Dichtemessung größer als 1,0 ist, verdünnen Sie 100 l Kultur 1:10 mit 900 l Frischmedien, erfassen Sie die optische Dichte, und multiplizieren Sie diesen Wert mit 10 für die OD600-Messung.
- Messung der Kolonieformeinheit (CFU/mL)
- Zu jedem Zeitpunkt 1 ml Bakterienkultur zurückziehen und in das -1 Glas-Reagenzglas mit 9 ml PBS abgeben.
- Für die Verdünnungsserie 1 ml von der -1 Röhre seriell nach unten in die -9 übertragen, die nach jeder Übertragung wirbelt. (Abbildung 2)
- Geben Sie für jede Verdünnung 100 l Zellsuspension auf die entsprechend beschriftete Feststoff-Agarplatte aus. (Abbildung 2)
- Mit einem Zellstreuer, der in Ethanol sterilisiert, durch eine Bunsen-Brennerflamme geleitet und durch Berühren der Oberfläche des Agars gekühlt wurde, verteilen Sie die 100 l Zellsuspension, bis die Oberfläche der Agarplatte trocken wird.
- Inkubieren Sie die Streuplatten auf dem Kopf bei einer Temperatur, die das Wachstum der Bakterien unterstützt. Hier wird E. coli bei 37°C inkubiert.
- Nach der Inkubation, sobald sichtbare Kolonien entstehen, zählen Sie die Anzahl der Bakterienkolonien auf jeder Platte und erfassen Sie diese Werte zusammen mit dem zugehörigen Verdünnungsfaktor für alle Platten zu jedem Zeitpunkt.
- Aufbau des Wachstumszustands
- Impfung der Medien
3. Datenanalyse und Ergebnisse
- Wachstumskurvendiagramm (OPTICAL Density( OD600)
- Zeichnen Sie die optische Dichte (OD600) im Vergleich zur Zeit auf einer Halbprotokollskala. (Abbildung 3)
- Colony Forming Unit (CFU/mL) Wachstumskurvendiagramm
- Wählen Sie für jeden Zeitpunkt die Verdünnungsplatte aus, bei der die Koloniezählungen in den Bereich von 30-300 Bakterien fielen. Multiplizieren Sie die Koloniezahl mit dem Verdünnungsfaktor und dann mit 10, da der Spread von 100 l bei der Berechnung der KBE/ml als zusätzliche Verdünnung von 1:10 betrachtet wird.
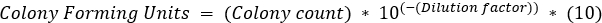
- Plotten Sie die Kolonie bildenden Einheiten im Vergleich zur Zeit auf einer Halblogskala. (Abbildung 4)
- Wählen Sie für jeden Zeitpunkt die Verdünnungsplatte aus, bei der die Koloniezählungen in den Bereich von 30-300 Bakterien fielen. Multiplizieren Sie die Koloniezahl mit dem Verdünnungsfaktor und dann mit 10, da der Spread von 100 l bei der Berechnung der KBE/ml als zusätzliche Verdünnung von 1:10 betrachtet wird.
- Beziehen von optischen Dichte- und Koloniebildungseinheiten
- Zeichnen Sie die koloniebildenden Einheiten im Vergleich zur optischen Dichte auf einer linearen Skala für OD600-Werte kleiner oder gleich 1,0 OD600, da die Beziehung zwischen OD600 und CFU/mL weniger genau als 1,0 OD600 ist. Hier werden die ersten sechs Zeitpunkte dargestellt. (Abbildung 5)
- Generieren Sie eine lineare Regressionstrendlinie, die die Gleichung und denR2-Wert anzeigt.
- Bestimmung der Bakterienverdoppelungszeit
- Identifizieren Sie mithilfe des Koloniebildenden Einheitenwachstumskurvendiagramms während der Exponentialphase zwei Punkte im Diagramm mit der steilsten Neigung zwischen ihnen, um die Verdoppelungszeit zu berechnen.
- Berechnung der Verdoppelungszeit
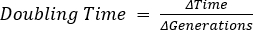
- Zeit = t2 - t1, wobei t1 = Timepoint 1 und t2 = Timepoint 2
-
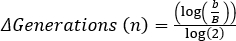 , wobei b = Anzahl der Bakterien bei t2, B = Anzahl der Bakterien bei t1und n = Anzahl der Generationen. Abgeleitet von:
, wobei b = Anzahl der Bakterien bei t2, B = Anzahl der Bakterien bei t1und n = Anzahl der Generationen. Abgeleitet von: .
- Berechnen Sie die Verdoppelungszeit mit:
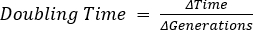
Ergebnisse
Plots von koloniebildenden Einheiten und optische Dichte sind zwei Möglichkeiten, wachstumskinetik zu visualisieren. Durch die Bestimmung der Beziehung zwischen CFU/mL und OD600 liefert das optische Dichtediagramm auch eine Schätzung von CFU/mL im Zeitverlauf. Bedingungen, die in der kürzesten Verdoppelungszeit führen, gelten als optimal für das Wachstum der gegebenen Bakterien.
Anwendung und Zusammenfassung
Wachstumskurven sind wertvoll für das Verständnis der Wachstumskinetik und Physiologie von Bakterien. Sie ermöglichen es uns zu bestimmen, wie Bakterien unter variablen Wachstumsbedingungen reagieren, sowie die optimalen Wachstumsparameter für ein bestimmtes Bakterium zu definieren. Koloniebildnende Einheit und optische Dichte Diagramme enthalten beide wertvolle Informationen, die die Dauer der Verzögerungsphase, maximale Zelldichte erreicht, und ermöglicht die Berechnung der bakteriellen Verdoppelungszeit. Wachstumskurven ermöglichen auch den Vergleich zwischen verschiedenen Bakterien unter den gleichen Wachstumsbedingungen. Darüber hinaus bietet die optische Dichte eine Möglichkeit, anfängliche Inokulume zu standardisieren und so die Konsistenz in anderen Experimenten zu verbessern.
Die Bestimmung, welcher Ansatz beim Entwerfen eines Wachstumskurvenexperiments verwendet werden soll, erfordert Überlegungen. Als bevorzugte Methode zur Erzeugung von Wachstumskurven spiegeln koloniebildende Einheitendie zahlbare Zellzahlen in der Chargenkultur genauer wider. Koloniebildnende Einheiten-Plots ermöglichen auch die Messung des bakterienwachstums unter Bedingungen, die andernfalls eine optische Dichtemessung stören würden. Es ist jedoch ein zeitaufwändigerprozesslicher Prozess, der eine umfangreiche Verwendung von Reagenzien erfordert und manuell durchgeführt werden muss. Optische Dichtediagramme sind weniger genau und liefern nur eine Schätzung der Koloniebildenden Einheiten, die eine Standardkurve für jedes einzelne Bakterium erfordern. Die optische Dichte wird in erster Linie für seine Bequemlichkeit verwendet, da sie weit weniger zeitaufwändig ist und nicht viele Reagenzien erfordert. Was für die optische Dichte am attraktivsten ist, ist, dass spektrophotometrische Inkubatoren automatisch Wachstumskurven erzeugen können, was die Anzahl der Kulturbedingungen, die gleichzeitig getestet werden können, erheblich erhöht und die Notwendigkeit entfällt, ständig an der Kultur teilzunehmen.
Referenzen
- R. E. Buchanan. 1918. Life Phases in a Bacterial Culture. J Infect Dis 23:109-125.
- CAMPBELL A. 1957. Synchronization of cell division. Bacteriol Rev 21:263-72.
- Wang P, Robert L, Pelletier J, Dang WL, Taddei F, Wright A, Jun S. 2010. Robust growth of Escherichia coli. Curr Biol 20:1099-103.
- Goldman E, Green LH. 2015. Practical Handbook of Microbiology, Third Edition. CRC Press.
- Ben-David A, Davidson CE. 2014. Estimation method for serial dilution experiments. J Microbiol Methods 107:214-221.
- Koch AL. 1968. Theory of the angular dependence of light scattered by bacteria and similar-sized biological objects. J Theor Biol 18:133-156.
- Sezonov G, Joseleau-Petit D, D'Ari R. 2007. Escherichia coli physiology in Luria-Bertani broth. J Bacteriol 189:8746-9.
pringen zu...
Videos aus dieser Sammlung:

Now Playing
Wachstumskurven: Erzeugen von Wachstumskurven mit Hilfe von koloniebildenden Einheiten und Messungen der optischen Dichte
Microbiology
297.9K Ansichten

Erstellen einer Winogradsky-Säule: Eine Methode zur Anreicherung der mikrobiellen Spezies in einer Sedimentprobe
Microbiology
129.9K Ansichten

Serielle Verdünnungen und Ausplattieren: Auszählung von Mikrobiom
Microbiology
317.1K Ansichten

Anreicherungskulturen: Kultivieren von aeroben und anaeroben Mikroben auf selektiven und differenziellen Medien
Microbiology
132.2K Ansichten

Reinkulturen und Streak-Plating: Isolierung einzelner Bakterienkolonien aus einer gemischten Probe
Microbiology
166.4K Ansichten

16S rRNA-Sequenzierung: Eine PCR-basierte Technik zur Identifizierung bakterieller Arten
Microbiology
189.7K Ansichten

Antibiotika-Empfindlichkeitstests: Epsilometertests zur Bestimmung der MHK-Werte von zwei Antibiotika und zur Bewertung der Antibiotika-Synergie
Microbiology
93.9K Ansichten

Mikroskopie und Färbungen: Gram-, Kapsel- und Endosporenfärbung
Microbiology
364.1K Ansichten

Plaque-Assay: Eine Methode zur Bestimmung des Virustiters als Plaque bildende Einheiten (PFU)
Microbiology
186.5K Ansichten

Transformation von E. coli-Zellen mit einem adaptierten Calciumchlorid-Verfahren
Microbiology
87.1K Ansichten

Konjugation: Eine Methode zur Übertragung der Ampicillinresistenz vom Spender auf den Empfänger E. coli
Microbiology
38.4K Ansichten

Phagen-Transduktion: Eine Methode zur Übertragung der Ampicillinresistenz von einem Spender auf den Empfänger E. coli
Microbiology
29.1K Ansichten
ISSN 2689-3657
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten
Wir verwenden Cookies, um Ihre Erfahrung auf unserer Website zu verbessern.
Indem Sie unsere Website weiterhin nutzen oder auf „Weiter“ klicken, stimmen Sie zu, unsere Cookies zu akzeptieren.