Method Article
Primärkultur von Stammzellen der Zahnpulpa
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Dieser Artikel enthält eine schrittweise Anleitung zur Etablierung einer Primärkultur von Zahnpulpa-Stammzellen unter Verwendung der Explantatkulturmethode und zur Charakterisierung dieser Zellen auf der Grundlage der ICSCRT-Richtlinien. Die durch dieses Protokoll isolierten Zellen können als mesenchymale Stammzellen für weitere Anwendungen betrachtet werden.
Zusammenfassung
Die humane Zahnpulpa stellt ein vielversprechendes multipotentes Stammzellreservoir mit herausragender regenerativer Kompetenz dar, das aus einem extrahierten Zahn gewonnen werden kann. Der aus der Neuralleiste gewonnene ekto-mesenchymale Ursprung von Zahnpulpa-Stammzellen (DPSCs) verleiht ein hohes Maß an Plastizität, das auf seine vielfältigen Vorteile bei der Gewebereparatur und -regeneration zurückzuführen ist. Es gibt verschiedene praktische Möglichkeiten, adulte Stammzellen zu gewinnen, zu erhalten und zu vermehren, die für ihren Einsatz in der regenerativen Medizin untersucht werden. In dieser Arbeit zeigen wir die Etablierung einer primären mesenchymalen Stammzellkultur aus Zahngewebe mit Hilfe der Explantatkulturmethode. Die isolierten Zellen waren spindelförmig und hafteten an der Kunststoffoberfläche der Kulturplatte. Die phänotypische Charakterisierung dieser Stammzellen zeigte eine positive Expression der von der International Society of Cell Therapy (ISCT) empfohlenen Zelloberflächenmarker für MSC, wie CD90, CD73 und CD105. Darüber hinaus bestätigte die vernachlässigbare Expression von hämatopoetischen (CD45) und endothelialen Markern (CD34) und weniger als 2% der HLA-DR-Marker die Homogenität und Reinheit der DPSC-Kulturen. Des Weiteren haben wir ihre Multipotenz anhand der Differenzierung zu adiogenen, osteogenen und chondrogenen Linien veranschaulicht. Wir haben diese Zellen auch dazu gebracht, sich durch Zugabe entsprechender Stimulationsmedien in leberähnliche und neuronale Zellen zu differenzieren. Dieses optimierte Protokoll wird bei der Kultivierung einer hochgradig erweiterbaren Population von mesenchymalen Stammzellen helfen, die im Labor oder für präklinische Studien verwendet werden sollen. Ähnliche Protokolle können in klinische Setups für die Durchführung von DPSC-basierten Behandlungen integriert werden.
Einleitung
Adulte Stammzellen haben sich aufgrund ihrer Plastizität, ihrer parakrinen Mechanismen und immunmodulatorischen Eigenschaften zu einem leistungsfähigen therapeutischen Werkzeug für zellgerichtete Behandlungen und Therapien entwickelt 1,2,3. Die ermutigenden Daten aus stammzellbasierten präklinischen Studien haben Forscher dazu inspiriert, an der Labor-zu-Krankenbett-Translation zu arbeiten. Die Art der Stammzellen, die für die Stammzelltherapie verwendet werden, spielt eine wichtige Rolle für erfolgreiche Ergebnisse. In präklinischen und klinischen Studien ist das Knochenmark nach wie vor die am häufigsten berichtete Quelle für mesenchymale Stammzellen (MSCs) 4,5. Zu den größten Nachteilen bei der Verwendung von Stammzellen (BMSCs) aus dem Knochenmark gehören jedoch ihre seltene Population, hochinvasive Verfahren zur Isolierung und ihre begrenzte Fähigkeit zur Expansion. Daher werden alternative Quellen für MSC untersucht. In dieser Hinsicht wurden Zahngewebe mit ihrer leichten Zugänglichkeit, enormen Plastizität, ihrem hohen Regenerationspotenzial und ihrer hohen Proliferationsfähigkeit nun als reichhaltige und potenzielle alternative Quelle für Stammzellen angesehen 6,7,8,9,10.
Zahnpulpa-Stammzellen (DPSCs) waren die erste Art von dentalen Stammzellen, die im Jahr 2000 von Gronthos isoliert und charakterisiert wurden11. DPSCs haben aufgrund ihrer hohen Proliferationsrate, ihres signifikanten Differenzierungspotenzials, ihrer einfachen Zugänglichkeit bei müheloser Kultivierung und vor allem ihrer Fähigkeit, ohne ethische Bedenken aus einem verworfenen Zahn gewonnen zu werden, die Aufmerksamkeit für Tissue-Engineering-Anwendungen auf sich gezogen12. Die Einschränkungen, die andere Stammzellquellen, wie z. B. BMSCs und aus Fettgewebe gewonnene Stammzellen (ADSCs), in ihrer Isolierung und unzureichenden Selbsterneuerungsfähigkeit mit sich bringen, werden durch DPSCsumgangen 13. Humane DPSCs können aus menschlichen Milchzähnen, bleibenden Zähnen, Weisheitszähnen, exfolierten Milchzähnen (SHEDs) und apikalen Papillen gewonnen werden. Darüber hinaus können DPSCs auch aus überzähligen Zähnen isoliert werden, die in der Regel verworfen werden14. DPSCs exprimieren Neuralleisten-assoziierte Marker und haben das Potenzial, sich sowohl in vitro als auch in vivo in neuronale Zellen zu differenzieren 15. Zusätzlich zu ihrem neurogenen Potenzial können sich DPSCs unter bestimmten Differenzierungsbedingungen in andere Zelllinien differenzieren, wie z. B. osteogene, chondrogene, adipogene, hepatische und myogene Zelllinien13. Somit bergen diese multipotenten Zellen ein großes Potenzial für die zellbasierte Therapie und können für die Regeneration verschiedener Gewebe eingesetzt werden. Studien haben auch über die potenzielle Rolle von DPSCs bei der Rekonstruktion der Hornhaut16, die Reparatur des Myokardinfarkts17 und ihre potenzielle therapeutische Rolle bei Krankheiten wie Ischämie der Gliedmaßen18, Alzheimer 19, Parkinson 20 und Alternberichtet 21. Daher können aus Zahngewebe gewonnene Stammzellen nicht nur für die Zahnregeneration verwendet werden, sondern auch für die Reparatur und Regeneration von nicht-zahnärztlichen Organen wie Augen16, Herzen17, Leber22, Knochen23 usw.
Es gibt zwei besondere Methoden für die Isolierung einer MSC-Population aus Pulpagewebe - enzymatischer Verdau und Explantatkultur24,25. Bei beiden Methoden wurde über eine erfolgreiche Etablierung von Primärkulturen ohne signifikante Unterschiede in der Menge und den Eigenschaften von DPSC berichtet26. In dieser Studie haben wir uns auf die Isolierung von DPSCs durch die Explantatmethode27 konzentriert, da diese Methode DPSCs ohne Kontamination von hämatopoetischen und endothelialen Zellen erzeugt, im Vergleich zum enzymatischen Verdau, der zu einer Fibroblastenkontamination führen kann28.
Protokoll
Alle in der Studie beschriebenen Verfahren wurden von der Ethikkommission des Instituts (IEC# 9195/PG-12 ITRG/2571-72) von PGIMER, Chandigarh, genehmigt. Alle Experimente im Zusammenhang mit der Zellkultur müssen in einer biologischen Sicherheitswerkbank (BSC) der Klasse II nach aseptischer Technik durchgeführt werden. Die Zahnpulpa wurde von gesunden Zähnen von drei Patienten (F/14, M/14 und M/20) gewonnen, die sich aus kieferorthopädischen Gründen einer Extraktion des dritten Molaren unterzogen hatten. Vor der Probenentnahme wurde eine schriftliche Einverständniserklärung des Patienten/Erziehungsberechtigten gemäß den Richtlinien der Ethikkommission von PGIMER, Chandigarh, eingeholt.
1. Etablierung einer Primärkultur von Stammzellen der Zahnpulpa (DPSCs) aus menschlichem Zahngewebe
HINWEIS: Kariöse Zähne sollten nicht verwendet werden.
- Entnahme von Zahnpulpaproben und Transport zum Gewebekulturlabor
- Führen Sie eine Zahnextraktion unter nicht-traumatischen und aseptischen Bedingungen durch. Spülen Sie den extrahierten Zahn gründlich mit einer sterilen Kochsalzlösung aus, um Blutreste zu entfernen.
- Schneiden Sie den extrahierten Zahn in Längsrichtung in zwei Teile, indem Sie einen Hartmetall-Fissurenbohrer mit ständiger Spülung mit steriler normaler Kochsalzlösung verwenden, um die Pulpa freizulegen.
- Transportieren Sie den extrahierten Zahn zusammen mit der intakten Pulpa in sterilen α-Minimum essentiellen Medien (α-MEM), die auf Eis aufbewahrt werden, zum Zell- und Gewebekulturlabor.
HINWEIS: Idealerweise werden alle Proben, die innerhalb von 2 Stunden eingehen, für die Zellkultur verarbeitet. Ein kürzlich veröffentlichter Bericht deutet jedoch darauf hin, dass die Verarbeitung der Zahnprobe auch nach 24 Stunden die Lebensfähigkeit und Proliferation von DPSCs nicht beeinflusste, jedoch ihre Funktionalität verringerte29.
2. Entnahme von Pulpagewebe aus dem Zahn und Zellkultur von DPSCs (Zeit: 60-120 min)
HINWEIS: Alle Schritte nach dem Zahntransport wurden im Zell- und Gewebekulturlabor in einer Biosicherheitswerkbank der Stufe 2 durchgeführt.
- Legen Sie die Zahnprobe in sterile Petrischalen und spülen Sie sie gründlich (drei- bis viermal) mit 1x steriler phosphatgepufferter Kochsalzlösung (PBS) aus.
- Entfernen Sie das Pulpagewebe vorsichtig mit Hilfe eines scharfen Baggers und einer Pinzette aus der Pulpahöhle.
- Sobald die Pulpa entfernt ist, reinigen Sie sie gründlich mit sterilem PBS, um alle Blutreste zu entfernen.
- Schneiden Sie das Pulpagewebe mit Hilfe einer chirurgischen Klinge in einer Petrischale in kleine Stücke von ca. 2-3 mm.
- Teilen Sie die Oberfläche der Schale in vier Teile, indem Sie 1-2 ml steriles PBS in jeden Bereich geben. Geben Sie die Pulpagewebeprobe in einen Bereich mit PBS und schneiden Sie das Gewebe mit einer chirurgischen Klinge in möglichst kleine Stücke (je kleiner das Stück, desto leichter können Stammzellen aus dem Gewebe wandern). Übertragen Sie danach jedes Stück, indem Sie es in einen anderen Bereich mit PBS heben, und so weiter bis zum vierten Bereich, damit die Gewebestücke richtig gewaschen und frei von Verunreinigungen sind.
- Gießen Sie in der Zwischenzeit etwa 0,8-1 ml α-MEM, das nicht-essentielle Aminosäuren mit 10 % fötalem Rinderserum (FBS) und einem Antibiotika-Cocktail (d. h. Penicillin und Streptomycin) enthält, in die Vertiefungen einer 6-Well-Platte. Verteilen Sie das Medium gleichmäßig, indem Sie die Platte so bewegen, dass sie gleichmäßig auf der Vertiefungsoberfläche verteilt wird.
- Säen Sie die kleinen Stücke von Fruchtfleischgewebe oder Explantaten mit Hilfe einer Pinzette in eine einzelne Vertiefung (vier bis sechs Stück pro Vertiefung) der 6-Well-Platte und bewahren Sie die Platte 5 min lang ungestört in einer Biosicherheitshaube auf.
HINWEIS: Dieser Schritt ermöglicht bzw. hilft bei der Adhärenz von Gewebe/Explantaten auf den behandelten Oberflächen von Kulturplatten. - Bewahren Sie die Kulturplatte in einer befeuchteten Atmosphäre mit einer Temperatur von 37 °C und einem CO2 -Gehalt von 5 % auf.
- Am nächsten Tag saugen Sie das Medium vorsichtig aus den Vertiefungen ab, ohne die Explantate zu stören, und fügen Sie frisches Medium hinzu. Wechseln Sie das Medium bei diesem Schritt jeden zweiten Tag, um ein Austrocknen zu verhindern. Stellen Sie sicher, dass der Inkubator genügend Wasser in der Wanne hat, damit zusätzliche Verdunstung nicht dazu führt, dass das Medium in den Plattenvertiefungen austrocknet.
HINWEIS: Dies ist ein sehr wichtiger Schritt, und Störungen können dazu führen, dass sich die Explantate aus ihrer Position lösen. Wenn sich die Explantatstücke während des Medientransfers aus ihrer Position lösen, versuchen Sie, sie wieder anzubringen, indem Sie das Medium vollständig entfernen und an der Well-Oberfläche haften lassen. Das Medium sollte vorsichtig an der Wand der Vertiefung entlang gegeben werden, ohne die Explantate zu stören, da sie sich sonst wieder ablösen könnten. Beobachten Sie die Zellkulturplatten regelmäßig unter einem inversen Mikroskop (4- oder 10-fache Vergrößerung). Identifizieren Sie DPSCs an ihrer Fähigkeit, an der Kunststoffoberfläche zu haften, und an ihrer spindelförmigen Morphologie. - Sobald genügend Zellen aus den Explantaten migrieren, entfernen Sie das Gewebe und lassen Sie die Zellen vermehren und die optimale Zellkonfluenz von 70 % bis 75 % erreichen, bevor Sie subkultivieren. Das Volumen von α-MEM, das 10 % FBS enthält, kann in diesem Schritt auf 2 ml erhöht werden, damit sich die Zellen schneller teilen können. Wechseln Sie zu diesem Zeitpunkt jeden dritten Tag das Medium.
3. DPSC-Erweiterung
- Waschen Sie den konfluenten T75-Kulturkolben (Konfluenz: 70 %-80 %) der DPSCs zweimal mit PBS (10 ml).
- Geben Sie 2,5 ml 0,25 % Trypsin-Ethylendiamintetraessigsäure (EDTA) an die Seite des T75-Kolbens und schwenken Sie ihn vorsichtig, um sicherzustellen, dass das Trypsin-EDTA die gesamte Zellfalte oder Monoschicht der DPSCs bedeckt.
- Den Kolben in der Haube 3-5 min bei 37 °C halten. Nach 3 Minuten klopfen Sie vorsichtig auf den Kolben, um die Dissoziation der Zellen sicherzustellen.
- Fügen Sie 5 mL vollständiges Medium (α-MEM mit 10 % FBS) oder 250 μl FBS (10 % des verwendeten Trypsinvolumens) als Neutralisationsmittel hinzu, um die Trypsinaktivität zu inaktivieren. Übertragen Sie die Ganzzellsuspension der dissoziierten DPSCs in ein frisches konisches 15-ml-Zentrifugenröhrchen.
- Zentrifugieren Sie die dissoziierten Zellen bei 500 x g für 2 min bei Raumtemperatur, um das Zellpellet zu erhalten.
- Saugen Sie den gesamten Überstand mit der Pipette ab, geben Sie 5-10 ml PBS in den Kolben, um die Restzellen zu sammeln, waschen Sie die Zelle in PBS durch vorsichtiges Pipettieren ab und zentrifugieren Sie die Zellen erneut bei 500 x g für 2 Minuten. Wiederholen Sie den Vorgang bei Bedarf.
- Resuspendieren Sie das Zellpellet in 1 ml vollständigem Medium (α-MEM mit 10 % FBS), nachdem Sie den Überstand vorsichtig entfernt und vorsichtig pipettiert haben, um eine Einzelzellsuspension zu erhalten.
- Teilen Sie die Zellen und säen Sie sie (gleiches Volumen) in zwei T75-Kulturflaschen aus, die jeweils 10 ml vollständiges Medium enthalten. Halten Sie den Kolben in der Haube bei 37 °C.
- Saugen Sie am nächsten Tag das Medium ab und ersetzen Sie es durch das frische vollständige Medium.
HINWEIS: Eine längere Behandlung mit Trypsin kann die Zellintegrität, Lebensfähigkeit und Zellfunktion beeinträchtigen, indem das Zelloberflächenprotein und das extrazelluläre Matrixprotein gespalten werden.
4. Identifizierung von phänotypischen Markern für Stammzellen (Zeit: 90 - 120 min)
HINWEIS: Für die Charakterisierung von Zellen, die aus dem Pulpagewebe entnommen wurden, sind Zellen zwischen der dritten und fünften Passage zu verwenden.
- Verwenden Sie zwei 70%-80% (~3-4 x 106 Zellen; zählen Sie die Zellen mit einem bildbasierten Zytometer oder Hämozytometer) konfluenten T75-Kulturflaschen mit DPSCs und waschen Sie die Zellen zweimal mit 10 ml PBS.
- Fügen Sie 2,5 ml 0,25 % Trypsin-EDTA hinzu und halten Sie den Kolben in der Haube 3-5 Minuten lang bei 37 °C. Fügen Sie 5 ml des vollständigen Mediums oder 250 μl FBS (10 % des verwendeten Trypsinvolumens) als Neutralisationsmittel hinzu, um die Trypsinaktivität zu inaktivieren.
- Sammeln Sie die Zellen mit einer Pipette in einem konischen 15-ml-Zentrifugenröhrchen. Zentrifugieren Sie die dissoziierten Zellen bei 500 x g für 2 min bei Raumtemperatur, um das Zellpellet zu erhalten.
- Saugen Sie den gesamten Überstand mit der Pipette ab, geben Sie 5 ml PBS in den Kolben, um die Restzellen zu sammeln, waschen Sie die Zelle in PBS durch vorsichtiges Pipettieren ab und zentrifugieren Sie die Zellen erneut bei 500 x g für 2 Minuten.
- Resuspendieren Sie das Pellet in 1 ml PBS, nachdem Sie den Überstand vorsichtig entfernt haben, und führen Sie ein vorsichtiges Pipettieren durch, um eine einzellige Suspension zu erhalten.
- Zählen Sie die Zellen je nach Verfügbarkeit mit einem bildbasierten Zytometer oder Hämozytometer und verteilen Sie sie in Mikrozentrifugenröhrchen bei 1 x 104 Zellen/Röhrchen. Inkubieren Sie die Zellen 0,5-1% Rinderserumalbumin (BSA) für 0,5-1 h.
- Zu jedem Röhrchen werden 1-2 μl Fluoresceinisothiocyanat (FITC)- und Phycoerythrin (PE)-konjugierte Antikörper für Cluster von Differenzierungsmarkern (CD) (d. h. CD90 [0,4 μg], CD73 [0,8 μg], CD105 [0,4 μg], CD34 [0,8 μg], CD45 [0,4 μg] und HLA-DR [0,08 μg])6,7,8,9 und 0,5 h im Dunkeln bei Raumtemperatur inkubiert.
- Nach 0,5 h werden die Zellen durch Zentrifugieren bei 500 x g für 5 min pelletiert. Den Überstand ansaugen, das Kügelchen der Zellen mit PBS abwaschen und erneut zentrifugieren.
- Nach der Zentrifugation resuspendieren Sie das Pellet in PBS (200 μl/Röhrchen), überführen Sie die Zellsuspension in Durchflusszytometrieröhrchen und entnehmen Sie diese mit einem Durchflusszytometer. Jede kommerzielle Software kann verwendet werden, um den Prozentsatz positiver Zellen für verschiedene Antikörper weiter zu analysieren.
HINWEIS: Die Schritte 4.7 bis 4.9 sind im Dunkeln bei Raumtemperatur durchzuführen. Geeignete Isotypkontrollen und ungefärbte Proben sollten verwendet werden, um die Hintergrundfluoreszenz der Zellen zu vermeiden.
5. DPSC-Differenzierung in mehrere Linien
HINWEIS: Verwenden Sie 75%-80% konfluente Kulturen von DPSCs an der dritten bis fünften Passage zur Beurteilung der Multipotenz. Eine Kontrollzellgruppe, die α-MEM enthält, sollte für alle unten beschriebenen Arten von Differenzierungen verwendet werden.
- Osteogene Differenzierung von DPSCs, Alizarinrotfärbung und Quantifizierung
- Stellen Sie eine einzellige Suspension mit 0,25 % Trypsin-EDTA her, wie oben in Schritt 4.2 beschrieben, und resuspendieren Sie das Zellpellet vollständig α-MEM.
- Zählen Sie die Zellen und säen Sie sie in 24-Well-Platten mit Gewebekulturen bei einer Zelldichte von 2 x 104 Zellen/Well, die 0,4-0,5 ml vollständiges Medium für 48 Stunden bei 37 °C und 5 % CO2 enthält.
- Entfernen Sie nach 48 Stunden das Medium aus den Vertiefungen und fügen Sie 0,4-0,5 ml osteogenes induktives Medium hinzu (d. h. α-MEM mit osteogenen induktiven Faktoren wie Beta-Glycerophosphat [5 mM], Monokaliumphosphat [1,8 mM] und Dexamethason-Dinatriumphosphat [0,01 mM])8. Präparation von Kontrollvertiefungen (nicht induzierte Zellen), die nur vollständiges α-MEM enthalten, ohne osteogene induktive Faktoren (Kontrollmedien).
HINWEIS: Es wird auch berichtet, dass die Zugabe von 50 μM Ascorbat die osteogene Differenzierung verbessert 30,31. Ersetzen Sie nach jedem dritten Tag die Induktionsmedien und Kontrollmedien für 21 Tage (3 Wochen) durch frische Medien und Kulturen. Entfernen Sie am Ende der Induktionsphase das Medium von den Kulturplatten, fügen Sie 0,5 ml PBS hinzu und waschen Sie es zweimal. - Fixieren Sie die Zellen nach dem PBS-Waschen 20-30 Minuten lang in 0,5 ml 10%iger neutraler gepufferter Formalinlösung (NBF) bei Raumtemperatur. Führen Sie die Fixierung in einem Abzug/einer chemischen Haube durch.
- Alizarinrot-Färbung
- Bereiten Sie den Alizarinrot-S-Farbstoff (2%) vor, indem Sie 1 g Alizarinrot S in 50 mL doppelt destilliertem Wasser (ddH2O) auflösen und den pH-Wert mit Hilfe von Salzsäure (HCL) oder Ammoniumhydroxid (NH4OH) auf 4,0-4,2 einstellen. Filtern Sie den Fleck mit Filterpapier. Lagern Sie den Fleck im Dunkeln (4 °C).
- Waschen Sie die Zellen zweimal mit doppelt destilliertem Wasser (ddH2O), fügen Sie Alizarinrot S hinzu und inkubieren Sie sie 30-45 Minuten lang bei Raumtemperatur.
- Entfernen Sie den Fleck aus allen Vertiefungen und waschen Sie die Zellen vier- bis fünfmal mit ddH2O, um den ungebundenen Fleck zu entfernen. Nehmen Sie die Bilder mit einem Hellfeldmikroskop auf.
- Extraktion von Alizarin-Rotflecken für die quantitative Analyse
- Geben Sie Cetylpyridiniumchlorid (CPC)-Puffer (pH 7,0) in die gefärbten Vertiefungen (0,4 ml/Vertiefung) und inkubieren Sie die Zellen 2 Stunden lang bei 37 °C. Zusätzlich können die Zellen auch mit 10%iger Essigsäure inkubiert werden, um die Alizarinrotkristalle ordnungsgemäß aufzulösen32. Bewahren Sie die Platten auf einer Tellerwippe oder einem Shaker auf.
- Nach der Inkubation wird der Überstand (extrahierter Farbstoff) entnommen, auf eine 96-Well-Platte übertragen und die Platte bzw. Proben bei 405 nm abgelesen. Um einen leeren Wert zu erhalten, fügen Sie nur den CPC-Puffer (gleiches Volumen) hinzu und lesen Sie32,33 .
6. Adipogene Differenzierung von DPSCs, Ölrot-O-Färbung und Quantifizierung
HINWEIS: Die ersten Schritte der Aussaat sind die gleichen wie oben (d.h. osteogener Differenzierungsschritt 5.1).
- Entfernen Sie nach 48 Stunden das Medium aus den Vertiefungen und fügen Sie 0,4-0,5 mL adipogenes induktives Medium hinzu (d. h. α-MEM mit adipogenen induktiven Faktoren wie Isobutylmethylxanthin [IBMX; 0,5 mM], Dexamethason [1 μM], Indomethacin [200 μM] und Insulin [10 μM])8. Bereiten Sie Kontrollvertiefungen vor, die nicht induzierte Zellen enthalten (d.h. nur in α-MEM ohne induktive Faktoren kultiviert (Kontrollmedien)).
- Füllen Sie das Medium jeden dritten Tag auf und kultivieren Sie die Zellen 3 Wochen lang. Nach 3 Wochen aspirieren Sie das Medium aus den Kulturplatten und fügen Sie 0,5 ml PBS hinzu, um die Zellen zweimal zu waschen.
- Fixieren Sie die Zellen nach dem PBS-Waschen 20-30 Minuten lang bei Raumtemperatur in 10%iger NBF-Lösung.
- Waschen Sie die Zellen zweimal mit doppelt destilliertem Wasser (ddH2O), fügen Sie ölrote O-Beize hinzu und inkubieren Sie sie 30-40 Minuten lang bei Raumtemperatur.
- Entfernen Sie den Fleck aus allen Vertiefungen und spülen Sie die Vertiefungen mit Zellen wiederholt mit ddH2O aus, bis sie klar sind (zwei- bis viermal). Beobachten Sie unter dem Mikroskop und nehmen Sie Bilder auf.
- Extraktion von ölrotem O für die quantitative Analyse
- Geben Sie 0,4 ml 100% 2-Propanol in die gefärbten Vertiefungen und inkubieren Sie bei Raumtemperatur, indem Sie die Platte 30 Minuten lang richtig abdecken. Bewahren Sie die Platte auf einer Tellerwippe oder einem Shaker auf.
- Nach der Inkubation wird der Überstand entnommen, auf eine 96-Well-Platte übertragen, die Platte oder die Proben bei 510 nm abgelesen. Verwenden Sie 100% 2-Propanol zum Ausblenden.
7. Chondrogene Differenzierung von DPSCs, Alcianblau-Färbung und Quantifizierung
HINWEIS: Die chondrogene Differenzierung wurde in der Monolayer-Kultur von DPSCs induziert. Die ersten Schritte der Aussaat sind die gleichen wie oben (d.h. osteogener Differenzierungsschritt 5.1).
- Nach 48 Stunden nehmen Sie das Medium aus den Vertiefungen und fügen Sie 0,4-0,5 ml chondrogenes Induktionsmedium hinzu (d. h. α-MEM mit induktiven Faktoren, die für die chondrogene Differenzierung spezifisch sind, Ascorbinsäure [0,2 M], Natriumpyruvat [1 mM], transformierender Wachstumsfaktor-beta 3 [TGF-β3; 10 μM], Dexamethason [1 mM] und 1x ITS-Premix [Insulin, Transferrin und selenöse Säure)10. Bereiten Sie Kontrollvertiefungen vor, die nicht induzierte Zellen enthalten (d. h. nur in α-MEM ohne induktive Faktoren [Kontrollmedien] kultiviert).
- Füllen Sie das Medium jeden dritten Tag auf und kultivieren Sie die Zellen 3 Wochen lang. Nach dem Ende der Induktionsphase aspirieren Sie das Medium aus den Kulturplatten und fügen Sie 0,5 ml PBS hinzu, um die Zellen zweimal zu waschen.
- Fixieren Sie die Zellen nach dem PBS-Waschen 15-20 min lang in 10%iger NBF-Lösung bei Raumtemperatur. Waschen Sie die Zellen zwei- bis dreimal mit doppelt destilliertem Wasser (ddH2O), fügen Sie Alcian-Blau-Beize hinzu und inkubieren Sie sie 45 Minuten lang bei Raumtemperatur.
- Entfernen Sie den Fleck aus allen Vertiefungen und spülen Sie anschließend zwei- bis dreimal mit 0,1 N HCL nach. Waschen Sie die Zellen viermal oder geben Sie sie mal mit ddH2O.
- Beobachten Sie die Zellen auf Proteoglykane (blaue Farbe) unter dem Mikroskop und nehmen Sie die Bilder auf.
- Für die Quantifizierung von Alcianblau extrahieren Sie den Farbstoff mit 6 M Guanidin-HCl und lesen Sie die Proben bei einer optischen Dichte (OD) von 595 nm ab. Das in dieser Studie verwendete Alcian-Blue-Kit färbt saure Proteoglykane im Knorpel und bindet speziell sulfatierte Glykosaminoglykane (GAGs), die in der Matrix der Chondrozyten10 vorhanden sind.
8. Differenzierung von DPSCs in Richtung hepatisch-ähnlicher Abstammungslinie und Charakterisierung
- Stellen Sie eine einzellige Suspension von Zellen durch Trypsin-Behandlung her, wie oben beschrieben, und resuspendieren Sie das Zellpellet vollständig α-MEM.
- Zählen Sie die Zellen und säen Sie sie in 24-Well-behandelten Kulturplatten bei 2 x 104 Zellen/Well mit 0,4-0,5 ml vollständigem Medium für 48 Stunden.
- Nach 48 Stunden nehmen Sie das Medium aus den Vertiefungen und inkubieren Sie die Zellen 14 Tage lang mit hepatischem Induktionsmedium. Das Induktionsmedium besteht aus Dulbecco's modifiziertem Eagle-Medium (DMEM) mit niedrigem Glukosegehalt, das Dexamethason (0,5 μM), den epidermalen Wachstumsfaktor (EGF; 2 ng/ml), den ITS+-Premix (50 mg/ml) und den Hepatozyten-Wachstumsfaktor (HGF; 20 ng/ml) enthält 6.
- Nach 14 Tagen wird das Medium für weitere 14 Tage durch Reifemedium ersetzt (α-MEM ergänzt mit Oncostatin M [OSM; 20 ng/ml], ITS-Premix [50 mg/ml] und Dexamethason [0,5 μM]) 6. Füllen Sie die Medien jeden dritten Tag mit frischen Medien auf.
- Führen Sie eine immunhistochemische Analyse und einen LDL-Assay (Low-Density-Lipoprotein) durch, um die leberähnliche Differenzierung zu bestätigen.
- Aufnahme von LDL und Immunfluoreszenzfärbung für den LDL-Rezeptor
HINWEIS: Hepatozyten-ähnliche Zellen nach der Differenzierung wurden durch das LDL-Aufnahme-Assay-Kit gemäß den Anweisungen des Kits charakterisiert. LDL-550 Fluorochrom wurde verwendet, um die LDL-Aufnahme in differenzierten Zellen im lebenden Zustand zu bestimmen, und der LDL-Rezeptor-Antikörper wurde zum Nachweis von LDL-Rezeptoren in differenzierten Zellen nach der Fixierung verwendet.- Nach der Differenzierung färben Sie die lebenden Zellen 3-4 h lang mit LDL-550 (1:100). Ersetzen Sie nach 3-4 Stunden die Färbelösung durch Nährmedien und beobachten Sie die LDL-Aufnahme unter einem Fluoreszenzmikroskop, das in der Lage ist, Propidiumiodid (PI)-Anregung und Emissionswellenlängen bei 537 bzw. 617 nm zu erzeugen.
- Fixieren Sie die Zellen für 10 min in der Fixierlösung. Nach der Zellfixierung die Zellen 30 Minuten lang in der Blockierungslösung blockieren.
- Inkubieren Sie die Zellen mit einem primären LDL-Rezeptor-Antikörper für 1 h, waschen Sie sie dreimal mit 1x PBS und inkubieren Sie dann mit 488 sekundären Antikörpern (1:100) für 1 h im Dunkeln.
- Waschen Sie die Zellen dreimal mit PBS, um überschüssige Sekundärantikörper zu entfernen. Fügen Sie 4′,6-Diamidino-2-phenylindol (DAPI) als Kernfärbung hinzu.
- Beobachten Sie die Zellen unter einem Fluoreszenzmikroskop. Nehmen Sie Bilder mit einem Fluoreszenzmikroskop auf, das Wellenlängen im Anregungs-/Emissionsbereich von DAPI, PI und FITC messen kann.
HINWEIS: Einzelzell-Intensitätsparameter können aufgezeichnet werden, um die LDL-Rezeptor-Antikörperexpression und die LDL-Aufnahme zu quantifizieren. Für die Immunfluoreszenzfärbung sollten geeignete Sekundärantikörperkontrollen verwendet werden, um die Hintergrundfluoreszenz, die nur aus dem Sekundärantikörper resultiert, zu vermeiden.
- Aufnahme von LDL und Immunfluoreszenzfärbung für den LDL-Rezeptor
9. Neuronale Differenzierung von DPSCs in Richtung neuronaler Abstammung und Charakterisierung
- Nach der Beschichtung der Zellen, wie oben beschrieben, kultivieren Sie die Zellen 48 h lang in α-MEM.
- Aspirieren Sie das Medium und ersetzen Sie es durch neurobasale Medien, die Wachstumsfaktoren enthalten, wie z. B. 0,5 % G5-Supplement, 0,2 % N2-Supplement, 20 ng/ml epidermaler Wachstumsfaktor, 20 ng/ml basischer Fibroblasten-Wachstumsfaktor und B27-Supplement (1%). Kultivieren Sie die Zellen 25-41 Tage lang in diesem Medium7.
- Wechseln Sie das Medium jeden zweiten oder dritten Tag, je nachdem, wie es von den Zellen verstoffwechselt wird. Um eine effiziente neuronale Reifung und mehr Mikrotubuli-assoziierte Protein-2 (MAP-2+) Neuronen zu erreichen, entfernen Sie EGF und Fibroblasten-Wachstumsfaktor 2 (FGF2) nach der neuronalen Induktionsphase (14 Tage).
HINWEIS: Im Laufe der Zeit und nach wiederholtem Medienwechsel können sich differenzierte Neuronen aufgrund geringer Adhärenz von den Plattenvertiefungen lösen, was in Langzeitkulturen zu einem Neuronenverlust führt. Um dies zu vermeiden, beschichten Sie die Plattenvertiefungen mit Laminin oder Fibronektin (optional). - Durchführung immunhistochemischer Analysen zur Bestätigung der neuronalen Differenzierung.
- Immunfärbung von differenzierten DPSCs
HINWEIS: Um die neuronale Differenzierung von DPSCs zu bestätigen, wurde in den differenzierten Zellen Immunzytochemie unter Verwendung von MAP-2 und Neurofilament (NFM)-Antikörpern durchgeführt, bei denen es sich um neurogene linienspezifische Proteine handelt7.- Entfernen Sie den Nährboden, nachdem die Differenzierung abgeschlossen ist. Fügen Sie PBS hinzu, schwenken Sie die Platte vorsichtig und entfernen Sie das PBS mit Hilfe einer Pipette. Wiederholen Sie den Schritt.
- Fixieren Sie die Zellen mit 10% NBF oder 4% Paraformaldehyd für 20-30 min bei Raumtemperatur.
- Entfernen Sie das Fixiermittel und waschen Sie die Zellen zwei- bis dreimal mit 1x PBS. Geben Sie Permeabilisierungslösung (Triton-X-100; 0,1%) und inkubieren Sie die Zellen für 5 Minuten.
- Führen Sie zwei- bis dreimal eine PBS-Wäsche durch und blockieren Sie die Zellen mit 0,5 %-1 % Blockierungsmittel (BSA) für 1 h bei 37 °C oder über Nacht bei 4 °C. Entfernen Sie das Blockierungsmittel und waschen Sie die Zellen mit 1x PBS.
- Inkubieren Sie die Zellen über Nacht mit einem primären Antikörper (hergestellt in 0,1 % BSA, NFM [1:50] und MAP-2 [1:200]).
- Entfernen Sie den primären Antikörper und waschen Sie die Zellen drei- bis viermal mit 1x PBS, um ungebundene Antikörper zu entfernen.
- Sekundärantikörper (Kaninchen Anti-Maus 1:2.000) zugeben und die Zellen bei Raumtemperatur 1,5-2 h im Dunkeln inkubieren. Waschen Sie die Zellen mit PBS (zwei- oder dreimal).
- Inkubieren Sie die Zellen 10 Minuten lang in DAPI und waschen Sie sie erneut mit PBS. Bewahren Sie das Restvolumen auf, um ein Austrocknen der Probe zu vermeiden. Unter dem Fluoreszenzmikroskop beobachten.
HINWEIS: Zusätzlich können auch die Karyotypstabilität10 und die Seneszenzcharakterisierung durchgeführt werden, um eine bessere Vorstellung von der Reinheit und Funktionalität der Zellen zu erhalten.
- Immunfärbung von differenzierten DPSCs
Ergebnisse
Hier beschreiben wir, wie Forscher eine Reinkultur von DPSCs mit der Explantatmethode 6,7,8,9,10 etablieren und sie zu mehreren Linien induzieren können, um die Reinheit der Kultur für nachgelagerte Anwendungen zu etablieren.
Wir etablierten eine Primärkultur von DPSCs aus dem kleinen Gewebe der Pulpa, das aus dem dritten Backenzahn von Patienten extrahiert wurde, wie in Abbildung 1A dargestellt. Zunächst wurden am vierten und fünften Tag nach der Explantataussaat nur sehr wenige abgerundete Zellen in unmittelbarer Nähe von Explantaten oder Geweben beobachtet. Am 12. Tag stieg jedoch die Anzahl der Zellen, die aus den Explantaten hervorgingen, an, und am Ende der zweiten Woche (14.-16. Tag) waren die meisten Explantate von vielen adhärenten Zellen mit spindelförmiger Morphologie umgeben. Zu diesem Zeitpunkt konnte das Explantat von den Platten entfernt werden und die Zellen konnten in Gegenwart des vollständigen Mediums wachsen. Nach Erreichen einer Konfluenz von 70%-75% (dritte Woche) wurden die Zellen subkultiviert und für den weiteren Versuchsaufbau expandiert. Durch die Explantatmethode erhielten wir eine einheitliche Kultur von Stammzellen mit einer Positivität der MSC-CD-Marker CD90, CD105, CD73 und einer fast vernachlässigbaren Zellpopulation eines anderen Zelltyps, des Endothelzellmarkers CD34 und des hämatopoetischen Zellmarkers CD45 (Abbildung 1B).
Wir differenzierten DPSCs auch erfolgreich in Zellen osteogener Abstammung, wie die positive Aufnahme von Alizarinrot-Färbungen in verkalkten Granulaten in Osteozyten, die adipogene Abstammung, wie die Aufnahme von Ölrot-O-Färbung durch Lipidtröpfchen in differenzierten Adipozyten zeigt, und die chondrogene Abstammung, wie durch eine positive Alcian-Blau-Färbung gezeigt wird (Abbildung 2). DPSCs zeigten auch eine effiziente Differenzierung in Richtung hepatisch-ähnlicher Abstammungslinie, wie die typische quaderförmige Morphologie und die Aufnahme von LDL-550 durch die Färbung lebender Zellen (Abbildung 3A) zeigt. Die differenzierten hepatozytenähnlichen Zellen wurden fixiert und für die Färbung mit dem LDL-Rezeptor-Antikörper aufbereitet. Abbildung 3B zeigt die positive Expression des LDL-Rezeptors in differenzierten hepatozytenähnlichen Zellen zusammen mit LDL-550. DPSCs zeigten auch eine ausgeprägte Fähigkeit, sich in neuronale Zellen zu differenzieren, wie die positive Färbung von MAP-2 und NFM, typischen neuronalen Proteinen, zeigt (Abbildung 4). Kurz gesagt, unsere Studie zeigt, dass DPSCs als reine Population mit einer Reinheit von >95 % für Stammzellmarker isoliert und kultiviert und in verschiedene Abstammungslinien für weitere nachgelagerte Anwendungen wie Zelltherapie, Medikamententests und Krankheitsmodellierung differenziert werden können.

Abbildung 1: Primärkultur von DPSCs. (A). Bilder des differentiellen Interferenzkontrasts (DIC), die verschiedene Entwicklungsstadien der DPSC-Primärkultur zeigen: 1) Explantation im Anfangsstadium der Knospenbildung mit abgerundeten blasenartigen Zellen, die am zweiten Tag der Aussaat aus dem Zahngewebe kommen; 2) Etablierung eines chaotischen Netzwerks von Zellen, die am vierten Tag aus dem Gewebe kommen; 3) Zellen mit voller spindelförmiger Morphologie bei Konfluenz am 13. Tag und Zellen, die nach der Gewebeentnahme die gesamte Oberfläche der Kulturschale bedecken. Maßstabsleiste = 100 μm, (B). Charakterisierung von Stammzellmarkern mittels Durchflusszytometrie mit Histogrammen und einem Balkendiagramm, das die positive Expression von Stammzellmarkern und die negative Expression von Nicht-Stammzellmarkern in DPSCs zeigt (n = 3). Jedes Experiment wurde mindestens dreimal in dreifacher Ausfertigung wiederholt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Multilineage-Differenzierung von DPSCs. Hellfeldmikroskopische Bilder, die die Färbung von Alizarinrot nach osteogener Differenzierung, die Färbung von Ölrot O nach der adipogenen Differenzierung und die Färbung von Alcianblau nach chondrogener Differenzierung bei DPSCs zeigen. Maßstabsbalken = 100 μm (n = 3). Jedes Experiment wurde mindestens dreimal in dreifacher Ausfertigung wiederholt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
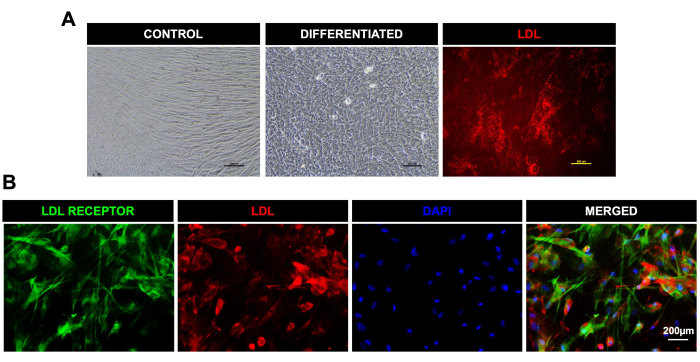
Abbildung 3: Differenzierung von DPSCs zu hepatozytenähnlichen Zellen und Aufnahme von LDL. (A). Lichtmikroskopische Aufnahmen für Kontrollzellen und leberähnliche Zellen, die von DPSCCs differenziert werden; Das letzte Bild ganz rechts zeigt die LDL-Aufnahme in den lebenden differenzierten Zellen. (B). Fluoreszierende mikroskopische Bilder, die die Expression des LDL-Rezeptors (grün) und die Aufnahme von LDL-550 (rot) (fixierte Zellen) in DPSCs nach hepatischer Differenzierung zeigen. DAPI wurde als nuklearer Farbstoff verwendet. Maßstabsleiste = 200 μm (n = 3). Jedes Experiment wurde mindestens dreimal in dreifacher Ausfertigung wiederholt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
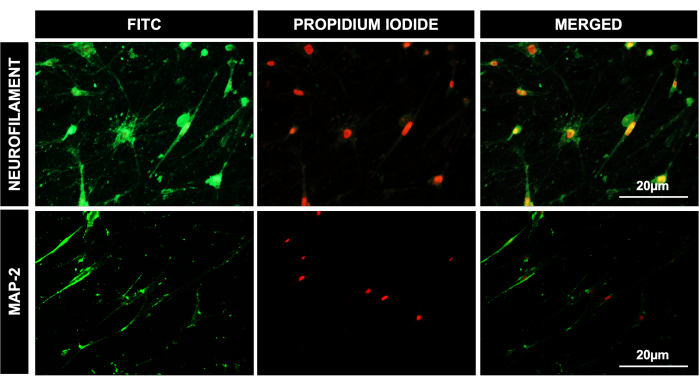
Abbildung 4: Differenzierung von DPSCs zur neuronalen Abstammung. Fluoreszierende mikroskopische Bilder für DPSCs nach 41 Tagen neuronaler Differenzierung, die eine positive Expression des Neurofilaments und von MAP-2 der neuronalen Abstammungslinie spezifischen Antikörper zeigen. PI wurde als nukleare Färbung verwendet. Maßstabsleiste = 20 μm (n = 3). Jedes Experiment wurde mindestens dreimal in dreifacher Ausfertigung wiederholt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Stammzellen haben aufgrund ihrer Plastizität, Robustheit, immunmodulatorischen Eigenschaften, parakrinen Mechanismen und Homing-Effizienz die Hoffnung auf die Heilung zahlreicher Krankheiten geknüpft. Zahnpulpagewebe gilt als die potenteste und wertvollste Quelle für Stammzellen, mit herausragender Plastizität und regenerativer Fähigkeit. Hier demonstrieren wir die Isolierung von DPSCCs unter Verwendung der weit verbreiteten Explantatkulturmethode, bei der die Zellen aus Pulpagewebestücken oder Explantaten migrieren, um zu einer homogenen Zellkultur zu wachsen, die morphologisch spindelförmigen Fibroblasten-ähnlichen Zellen ähnelt. Das Explantatverfahren führt zu einheitlicheren Kulturen von DPSCs, die frei von anderen Zelltypen wie Endothelzellen und Perizyten sind, die während des enzymatischen Verfahrens zur Etablierung der DPSC-Kultur verbleiben34. Wir zeigten, dass DPSCs, die mit der explantativen Methode abgeleitet wurden, ISCT-empfohlene mesenchymale Zelloberflächen-CD-Marker wie CD90, CD73 und CD105 exprimieren. Sie sind auch frei von CD45-, CD34- und HLA-DR-Marker-exprimierenden Zellen, was auf die Homogenität und Reinheit der DPSC-Kultur hinweist. Das Fehlen von HLA-DR macht sie auch für Transplantationszwecke geeignet, mit minimalen Abstoßungsmöglichkeiten. Das Protokoll enthält einige wichtige Schritte, um die Effizienz und Reinheit der DPSC-Kultur zu gewährleisten. Die Explantate sollten mindestens viermal mit PBS gewaschen werden, bevor sie in die Kulturplatten gegeben werden, um eine mögliche Kontamination zu vermeiden. Außerdem sollten die Explantate gut an der Oberfläche der Kulturvertiefung haften, da abgelöste Explantate keine Zellen ergeben würden. Daher ist beim Umgang mit Explantaten während des Medienwechsels äußerste Vorsicht geboten. Das Medium sollte vorsichtig tropfenweise entlang der Ecken der Vertiefung hinzugefügt werden, um die Explantate nicht zu stören. Sobald die Zellen in den Plattenvertiefungen wachsen, sollten Gewebestücke vorsichtig mit der Spitze entfernt werden, um die umliegenden Zellen nicht zu beschädigen. Bei der Entnahme von DPSCs beim ersten Durchgang aus den Plattenvertiefungen kann es sein, dass es nur sehr wenige Zellen gibt, so dass nach der Zentrifugation keine sichtbaren Zellpellets im Röhrchen zu sehen sind. Einige Zellen können auch an den Wänden des Zentrifugenröhrchens kleben, was es schwierig macht, während des Upscaling-Prozesses Zellen zu erhalten. Es sollte darauf geachtet werden, die Kulturplatte und das 15-ml-Röhrchen gut mit PBS zu spülen, um so viele Zellen wie möglich zu gewinnen. Alle zellbasierten Differenzierungsexperimente sollten in DPSCs unterhalb der achten Passage durchgeführt werden, da höhere Passagen zur Akkumulation genetischer Aberrationen und spontaner Differenzierung führen und somit die Ergebnisse beeinflussen können.
Es gibt auch einige Einschränkungen des Protokolls. Die Explantatkultur ist ein langsames Protokoll und liefert nach nur 15-20 Tagen Kultur genügend Zellen. Dazwischen besteht die Gefahr einer Kontamination, und das Explantat (Zahnpulpastücke) kann sich von der Plattenoberfläche lösen, was später keine Zellen mehr hervorbringen würde. Daher ist beim Wechsel des Mediums äußerste Vorsicht geboten. Außerdem sind wiederholte PBS-Waschungen wichtig, bevor die Explantate ausgesät werden, da die Zähne reich an oraler Mikroflora sind und die Wahrscheinlichkeit einer Kontamination der Kultur sehr hoch ist, wenn nicht ordnungsgemäß gewaschen wird. Humane DPSCs sind ektodermalen Ursprungs und während der Zahnentwicklung; Sie wandern aus der Neuralleiste und differenzieren sich später zu mesenchymalen Zellen. Dies verleiht ihnen besondere Eigenschaften sowohl der mesodermalen als auch der ektodermalen Linie34. Arthur et al. postulierten, dass die von der Neuralleiste abgeleiteten Zellen für die neuronale Differenzierung prädisponiert sind35; die spontane neuronale Differenzierung von DPSCs wurde auch von Kim et al. berichtet36. Eine andere Studie hat die potenzielle Rolle des distalen C-Terminus des L-Typ-spannungsgesteuerten Kalziumkanals bei der Ausrichtung von DPSCs auf das Erreichen eines neuronalen Phänotyps37 gezeigt. Zuvor hat unser Labor die Wirksamkeit der Kryokonservierung dieser DPSCs nachgewiesen, indem es sie 5 Jahre lang bei -80 °C konservierthat 10. Diese Zellen waren in der Lage, die Stammheitseigenschaften, die Proliferation und die Differenzierungsfähigkeit sowie einen stabilen Karyotyp ohne strukturelle oder numerische Anomalien beizubehalten. Darüber hinaus zeigte unser Labor in einer vergleichenden Analyse auch die überlegene Tendenz von DPSCs zu neurogenen7 - und osteogenen8-Linien im Vergleich zu BMSCs. Ihre überlegene Kompetenz und ihre vererbte Tendenz zu osteogenen und neuralen Abstammungslinien machen diese Zellen zum am besten geeigneten Kandidaten für Stammzelltherapien bei Knochenerkrankungen und neurodegenerativen Erkrankungen.
Insgesamt beschreiben wir hier ein effizientes Protokoll zur Gewinnung einer reinen Population von DPSCs durch die Explantatmethode, die sich in ihrer Stammzellmarkerexpression und ihrer Fähigkeit zur Differenzierung mehrerer Linien zeigt.
Offenlegungen
Die Autoren erklären, dass keine finanziellen oder nichtfinanziellen Interessenkonflikte bestehen.
Danksagungen
Wir danken für die finanzielle Unterstützung der AK durch das Department of Health Research (DHR), ICMR, Regierung von Indien (DHR-NRI Grant # R.12015/01/2022-HR). SR hat eine Finanzierung von ICMR, Regierung von Indien (Grant # 2020-7593/SCR-BMS) erhalten, und PS hat ein Stipendium von CSIR, Regierung von Indien, erhalten. Wir danken auch Frau Sandhya Tokhi und Frau Bhupinder Kaur für ihre Unterstützung bei der Durchflusszytometrie sowie dem Central Sophisticated Instrumentation Core (CSIC) und PGIMER, Chandigarh, für die Bereitstellung von infrastruktureller Unterstützung.
Materialien
| Name | Company | Catalog Number | Comments |
| 6 well cell culture plate | Costar | 3516 | For cell culture |
| Alcian blue stain | EZstain chondrocyte staining kit, HiMedia | CCK029 | |
| alizarin red S stain | Sigma-Aldrich | TMS-008 | Osteogenic stain |
| Antibiotic cocktail | Himedia | A002-5X50ML | To prevent culture contamination |
| Ascorbic Acid | Himedia | TC094-25G | Chondrogenic induction |
| B27 supplement | Gibco | 17504044 | For neural induction |
| bFGF ( basic Fibroblast Growth Factor) | Gibco | PHG0024 | For neural induction |
| CD 105 | BD-Pharmingen | 560839 | |
| CD 35 | Biolegend | 343604 | |
| CD 45 | Biolegend | 304006 | |
| CD 73 | Biolegend | 344016 | |
| CD 90 | Biolegend | 328107 | Characterization |
| cetyl pyridinium chloride (CPC) | Sigma-Aldrich | 1104006 | For Alizarin Red extraction |
| Dexamethasone 21-phosphate disodium | Sigma-Aldrich | D1159-100MG | |
| Dulbecco's Phosphate Buffered Saline | Himedia | TS1006-5L | For washing purpose |
| EGF (Epidermal Growth Factor) | Gibco | PHG0311 | For hepatic and neural induction |
| EVOS LED microscope | Invitrogen | For fluorescence imaging | |
| EZ stain Chondrocyte staining kit | Himedia | CCK029-1KT | Chondro stain Kit |
| FACS Canto flow cytometer | BD Biosciences | For cell characterization | |
| Fetal Bovine Serum | Gibco | 16000044 | For primary culture |
| Fetal Bovine Serum | Sigma-Aldrich | F2442 | For cell culture |
| G5 supplement | Gibco | 17503012 | For neural induction |
| HGF( Hepatocyte Growth Factor) | Sigma-Aldrich | H1404 | For hepatic Induction |
| HLA-DR | Biolegend | 307605 | |
| Human TGF-β3 | Peprotech | #100-36E-10U | |
| Insulin-Transferrin-Selenous acid premix | Sigma-Aldrich | I3146 | For hepatic Induction |
| ITS premix | Corning | 354350 | |
| LDL Uptake Assay kit | Abcam | ab133127 | For hepatic characterization |
| Low glucose DMEM | Gibco | 11885-084 | For hepatic induction |
| MAP2 antibody | Sigma-Aldrich | M4403 | For neural characterization |
| N2 supplement | Gibco | 17502048 | For neural induction |
| Neural Basal Media | Gibco | 21103049 | For neural induction |
| NFM antibody | Sigma-Aldrich | N4142 | For neural characterization |
| Nikon Elipse TS100 microscope | Nikon | For fluorescence imaging | |
| Oil Red O | Sigma-Aldrich | 01391-250Ml | Adipogenic stain |
| Oncostatin M | R&D Systems | 295-OM-010/CF | For hepatic Induction |
| Petridish | Tarson | 460090-90MM | For tissue cutting |
| Potassium phosphate monobasic | Sigma-Aldrich | 15655-100G | Osteogenic induction |
| Propan-2-ol | Thermo Fisher | Q13827 | For Oil Red O extraction |
| Sodium pyruvate solution | Sigma life sciences | S8636-100ML | |
| Trypsin-EDTA | Sigma-Aldrich | T4049 | For cell passaging |
| Whatman filter paper | merck | WHA1001325 | filter paper |
| α- Minimum Essential Media (α-MEM) | Sigma-Aldrich | M0643-10X 1L | Media for primary culture |
| β-glycerophosphate disodium salt hydrate | Sigma-Aldrich | G9422-50G |
Referenzen
- Bhattacharyya, S., Kumar, A., Lal Khanduja, K. The voyage of stem cell toward terminal differentiation: a brief overview. Acta Biochimica et Biophysica Sinica. 44 (6), 463-475 (2012).
- Prentice, D. A. Adult stem cells. Circulation Research. 124 (6), 837-839 (2019).
- Raik, S., Kumar, A., Bhattacharyya, S. Insights into cell-free therapeutic approach: Role of stem cell "soup-ernatant". Biotechnology and Applied Biochemistry. 65 (2), 104-118 (2018).
- Rodriguez-Fuentes, D. E., et al. Mesenchymal stem cells current clinical applications: a systematic review. Archives of Medical Research. 52 (1), 93-101 (2021).
- Yamazaki, K., Kawabori, M., Seki, T., Houkin, K. Clinical trials of stem cell treatment for spinal cord injury. International Journal of Molecular Sciences. 21 (11), 3994 (2020).
- Kumar, A., et al. Molecular spectrum of secretome regulates the relative hepatogenic potential of mesenchymal stem cells from bone marrow and dental tissue. Scientific Reports. 7 (1), 15015 (2017).
- Kumar, A., Kumar, V., Rattan, V., Jha, V., Bhattacharyya, S. Secretome cues modulate the neurogenic potential of bone marrow and dental stem cells. Molecular Neurobiology. 54 (6), 4672-4682 (2017).
- Kumar, A., Kumar, V., Rattan, V., Jha, V., Bhattacharyya, S. Secretome proteins regulate comparative osteogenic and adipogenic potential in bone marrow and dental stem cells. Biochimie. 155, 129-139 (2018).
- Kumar, A., Bhattacharyya, S., Rattan, V. Effect of uncontrolled freezing on biological characteristics of human dental pulp stem cells. Cell Tissue Bank. 16 (4), 513-522 (2015).
- Raik, S., et al. Assessment of post-thaw quality of dental mesenchymal stromal cells after long-term cryopreservation by uncontrolled freezing. Applied Biochemistry and Biotechnology. 191 (2), 728-743 (2020).
- Gronthos, S., Mankani, M., Brahim, J., Robey, P. G., Shi, S. Postnatal human dental pulp stem cells (DPSCs) in vitro and in vivo. Proceedings of the National Academy of Sciences. 97 (25), 13625-13630 (2000).
- Tsutsui, T. W. Dental pulp stem cells: advances to applications. Stem Cells and Cloning. 13, 33-42 (2020).
- Huang, G. T. -. J., Gronthos, S., Shi, S. Mesenchymal stem cells derived from dental tissues vs. those from other sources: their biology and role in regenerative medicine. Journal of Dental Research. 88 (9), 792-806 (2009).
- La Noce, M., et al. Dental pulp stem cells: state of the art and suggestions for a true translation of research into therapy. Journal of Dentistry. 42 (7), 761-768 (2014).
- Xiao, L., Tsutsui, T. Characterization of human dental pulp cells-derived spheroids in serum-free medium: stem cells in the core. Journal of Cellular Biochemistry. 114 (11), 2624-2636 (2013).
- Kumar, A., Yun, H., Funderburgh, M. L., Du, Y. Regenerative therapy for the cornea. Progress in Retinal and Eye Research. 87, 101011 (2022).
- Gandia, C., et al. Human dental pulp stem cells improve left ventricular function, induce angiogenesis, and reduce infarct size in rats with acute myocardial infarction. Stem Cells. 26 (3), 638-645 (2008).
- Yong, Z., et al. Comparison of the angiogenic ability between SHED and DPSC in a mice model with critical limb ischemic. Tissue Engineering and Regenerative Medicine. 19 (4), 861-870 (2022).
- Zhang, X. M., et al. Therapeutic potential of dental pulp stem cell transplantation in a rat model of Alzheimer's disease. Neural Regeneration Research. 16 (5), 893-898 (2021).
- Kabir, R., et al. Imperative role of dental pulp stem cells in regenerative therapies: a systematic review. Nigerian Journal of Surgery. 20 (1), 1-8 (2014).
- Kumar, A., et al. Dental pulp stem cell secretome ameliorates d-galactose induced accelerated aging in rat model. Cell Biochemistry and Function. 40 (5), 535-545 (2022).
- Hirata, M., et al. Multifaceted therapeutic benefits of factors derived from dental pulp stem cells for mouse liver fibrosis. Stem Cells Translational Medicine. 5 (10), 1416-1424 (2016).
- Fujii, Y., et al. regeneration by human dental pulp stem cells using a helioxanthin derivative and cell-sheet technology. Stem Cell Research & Therapy. 9 (1), 24 (2018).
- Ferrua, C. P., et al. How has dental pulp stem cells isolation been conducted? A scoping review. Brazilian Oral Research. 31, e87 (2017).
- Pilbauerova, N., Soukup, T., Suchankova Kleplova, T., Suchanek, J. Enzymatic isolation, amplification and characterization of dental pulp stem cells. Folia Biologica. 65 (3), 124-133 (2019).
- Tatullo, M., Marrelli, M., Shakesheff, K. M., White, L. J. Dental pulp stem cells: function, isolation and applications in regenerative medicine. Journal of Tissue Engineering and Regenerative Medicine. 9 (11), 1205-1216 (2015).
- Hilkens, P., et al. Effect of isolation methodology on stem cell properties and multilineage differentiation potential of human dental pulp stem cells. Cell and Tissue Research. 353 (1), 65-78 (2013).
- Hendijani, F. Explant culture: An advantageous method for isolation of mesenchymal stem cells from human tissues. Cell Proliferation. 50 (2), e12334 (2017).
- Aryal Ac, S., Islam, M. S., Samsudin, A. R. Investigation of the effect of a time delay on the characteristics and survival of dental pulp stem cells from extracted teeth. Archives of Oral Biology. 119, 104896 (2020).
- Langenbach, F., Handschel, J. Effects of dexamethasone, ascorbic acid and β-glycerophosphate on the osteogenic differentiation of stem cells in vitro. Stem Cell Research & Therapy. 4, 117 (2013).
- Jaiswal, N., Haynesworth, S. E., Caplan, A. I., Bruder, S. P. Osteogenic differentiation of purified, culture-expanded human mesenchymal stem cells in vitro. Journal of Cellular Biochemistry. 64 (2), 295-312 (1997).
- Bernar, A., Gebetsberger, J. V., Bauer, M., Streif, W., Schirmer, M. Optimization of the alizarin red S assay by enhancing mineralization of osteoblasts. International Journal of Molecular Sciences. 24 (1), 723 (2022).
- Coe, C. L., Lubach, G. R., Schneider, M. L., Dierschke, D. J., Ershler, W. B. Early rearing conditions alter immune responses in the developing infant primate. Pediatrics. 90, 505-509 (1992).
- Bakopoulou, A., et al. Assessment of the impact of two different isolation methods on the osteo/odontogenic differentiation potential of human dental stem cells derived from deciduous teeth. Calcified Tissue International. 88 (2), 130-141 (2011).
- Arthur, A., Rychkov, G., Shi, S., Koblar, S. A., Gronthos, S. Adult human dental pulp stem cells differentiate toward functionally active neurons under appropriate environmental cues. Stem Cells. 26 (7), 1787-1795 (2008).
- Kim, B. C., et al. Osteoblastic/cementoblastic and neural differentiation of dental stem cells and their applications to tissue engineering and regenerative medicine. Tissue Engineering. Part B, Reviews. 18 (3), 235-244 (2012).
- Ge, J., et al. Distal C terminus of CaV1.2 channels plays a crucial role in the neural differentiation of dental pulp stem cells. PLoS One. 8 (11), e81332 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten