Method Article
Quantitative Echtzeit-Polymerase-Kettenreaktions-Bewertung der MicroRNA-Expression in Niere und Serum von Mäusen mit altersabhängiger Nierenfunktionsstörung
In diesem Artikel
Zusammenfassung
Wir stellen eine Methode zur Bewertung der microRNA-Expression in Niere und Serum von Mäusen mit altersabhängiger Nierenfunktionsstörung durch quantitative Reverse-Transkriptions-Polymerase-Kettenreaktion vor.
Zusammenfassung
MicroRNAs (miRNAs) sind kleine, nicht-kodierende RNAs, die aus 21-25 Basen bestehen. Sie werden nicht in Proteine übersetzt, sondern behindern die Funktion ihrer Zielboten-RNAs (mRNAs), indem sie sie destabilisieren und ihre Translation stören. Obwohl die miRNA-Expressionsprofile in verschiedenen Mausorganen und -geweben untersucht wurden, gab es keine Standardmethoden zur Reinigung und Quantifizierung von Mausnieren- und Serum-miRNAs. Wir haben eine effektive und zuverlässige Methode zur Extraktion und Bewertung der miRNA-Expression im Serum und in der Niere von Mäusen mit altersabhängiger Nierenfunktionsstörung etabliert.
Die Methode verwendet die quantitative Reverse-Transkriptions-Polymerase-Kettenreaktion (qRT-PCR), und das Protokoll erfordert sechs Schritte: (1) Vorbereitung von Seneszenz-beschleunigten Mauswiderstands-1 (SAMR1) Mäusen und Seneszenz-beschleunigten Maus-anfälligen (SAMP1) Mäusen; (2) Entnahme von Serumproben aus diesen Mäusen; (3) Entnahme einer Nierenprobe aus jeder Maus; (4) Extraktion der Gesamt-RNA (einschließlich miRNA) aus Nieren- und Serumproben jeder Maus; (5) die Synthese komplementärer DNA (cDNA) mit reverser Transkription aus der miRNA; (6) Durchführung einer qRT-PCR unter Verwendung der erhaltenen cDNA.
Dieses Protokoll wurde verwendet, um zu bestätigen, dass im Vergleich zu den Kontrollen die Expression von miRNA-7218-5p und miRNA-7219-5p in der Niere und im Serum eines Mausmodells mit altersabhängiger Nierenfunktionsstörung signifikant verändert war. Dieses Protokoll klärte auch die Beziehung zwischen der Niere und dem Serum des Mausmodells der altersabhängigen Nierenfunktionsstörung. Dieses Protokoll kann verwendet werden, um die miRNA-Expression in der Niere und im Serum von Mäusen mit altersabhängiger Nierenfunktionsstörung zu bestimmen.
Einleitung
Es ist bekannt, dass die Expression verschiedener mRNAs, die sowohl in der Physiologie als auch in der Krankheit eine wichtige Rolle spielen (z. B. Entzündungen, Fibrose, Stoffwechselstörungen und Krebs), durch miRNAs reguliert wird, bei denen es sich um kurze, nicht-kodierende RNAs handelt, die den Abbau verursachen und die Transkription von mRNA1 hemmen. Es ist daher möglich, dass bestimmte miRNAs als neue Kandidatenbiomarker und/oder therapeutische Ziele für verschiedene Krankheitendienen könnten 2,3,4,5. Es wurden Untersuchungen zu miRNA-Expressionsprofilen in einer Vielzahl von Mausorganen und -geweben (einschließlich Gehirn6, Herz7, Lunge8, Leber9 und Niere10) durchgeführt. Es gibt jedoch keine Standard- oder etablierten Methoden zur Extraktion und Bewertung von miRNAs in den Nieren oder im Serum von Mäusen mit altersabhängiger Nierenfunktionsstörung.
Daher haben wir ein Protokoll etabliert, mit dem die miRNA-Expression im Serum und in der Niere von Mäusen mit altersabhängiger Nierenfunktionsstörung zuverlässig gereinigt und nachgewiesen werden kann. Es gibt sechs Hauptschritte im Protokoll: (1) Vorbereitung sowohl von 50 Wochen alten männlichen SAMR1-Mäusen als auch von männlichen SAMP1-Mäusen; (2) Entnahme von Blutproben aus der unteren Hohlvene beider Mäusestämme mit anschließender Verwendung eines Spitzröhrchens mit Heparin, gefolgt von Zentrifugation, um eine Serumprobe zu erhalten; (3) Nierenprobenextraktion aus den Mäusen - ein Siliziumhomogenisator wird verwendet, um die Nierenprobe separat zu homogenisieren, und die Probe wird dann in ein Biopolymer-Zerkleinerungssystem auf einer Mikrozentrifugenspinsäule11 überführt; (4) Gesamt-RNA-Extraktion (miRNA) aus den Serumproben unter Verwendung einer Siliciumdioxidmembran-basierten Spin-Säule12 und Gesamt-RNA-enthaltende miRNA-Extraktion aus den Nierenproben unter Verwendung einer Siliciumdioxidmembran-basierten Spin-Säule11; (5) Synthese komplementärer DNA (cDNA) aus der Gesamt-RNA unter Verwendung von reverser Transkriptase, Poly(A)-Polymerase und Oligo-dT-Primer13,14; und (6) schließlich Bestimmung der miRNA-Expression mittels qRT-PCR und eines interkalierenden Farbstoffs13,14.
Dieses neue Protokoll basierte auf Studien, die es geschafft haben, miRNAs in verschiedenen Gewebearten zu extrahieren undzu bewerten 11,12,13. Es wurde gezeigt, dass das Biopolymer-Zerkleinerungssystem des Protokolls in der Lage ist, hochwertige Gesamt-RNA aus Gewebenzu reinigen 11. Die Genauigkeit und Sensitivität von Aspekten dieses Protokolls, die für die Bewertung der miRNA-Expression mittels qRT-PCR mit einem interkalierenden Farbstoff verwendet werden, wurden13,14 nachgewiesen, beispielsweise die cDNA-Synthese mit reverser Transkriptase, Poly(A)-Polymerase und Oligo-dT-Primern aus der extrahierten Gesamt-RNA. Das neue Protokoll hat mehrere Vorteile: Einfachheit, Zeitersparnis und reduzierte technische Fehler. Es kann daher für Untersuchungen verwendet werden, die eine genaue und sensitive Identifizierung von Nieren- und Serum-miRNA-Profilen erfordern. Studien zu vielen pathologischen Zuständen können auch das neue Protokoll verwenden.
Die miRNA-Expressionsprofile in SAMP1-Mäusen, die ein Modell für eine altersabhängige Nierenfunktionsstörung darstellen, können wie unten gezeigt bestimmt werden. Beim Menschen ist eine altersabhängige Nierenfunktionsstörung mit dem Fortschreiten der Niereninsuffizienz assoziiert und sowohl durch eine Zunahme des Bereichs der renalen interstitiellen Fibrose als auch durch das Fortschreiten der Glomerulosklerose gekennzeichnet15,16. Die altersabhängige Nierenfunktionsstörung ist auch ein wichtiges und häufiges Merkmal chronischer Nierenerkrankungen und terminaler Niereninsuffizienz15,16.
Protokoll
Das Versuchsprotokoll wurde von der Tierethikkommission der Jichi Medical University genehmigt und in Übereinstimmung mit dem Jichi Medical University Guide for Laboratory Animals und seinen Richtlinien für die Verwendung und Pflege von Versuchstieren durchgeführt. Dieses Protokoll verwendet vier 50 Wochen alte männliche SAMR1-Mäuse und männliche SAMP1-Mäuse (40-45 g).
1. Entnahme von Serumproben
- Bereiten Sie für jede Maus Folgendes vor: 30 G-Nadeln mit einer 1,0-ml-Spritze, ein 1,0-ml-Spitzzentrifugationsröhrchen mit Heparin, 1,5-ml-Mikrozentrifugenröhrchen, Isofluran-Anästhetikum, ein Korktuch, 70% Ethanol, zwei mit phosphatgepufferter Kochsalzlösung (PBS) angefeuchtete Wattestäbchen, eine Petrischale mit PBS, Pinzette und chirurgische Schere.
- Betäuben Sie die Maus mit 1,5% Isofluran und halten Sie sie bei 1,5%. Verabreichen Sie ein Analgetikum (Meloxicam 5 mg / kg) subkutan, injizieren Sie dann 1,0 ml 70% Ethanol in den Bauch und legen Sie es in Rückenlage auf das Korkblatt.
- Bestätigen Sie die Tiefe der Anästhesie durch das Verschwinden des Pedalrückzugsreflexes. Schneiden Sie mit der Pinzette und der chirurgischen Schere die Haut des Bauches ein. Schneiden Sie die Muskeln und die Peritonealmembran von der Blase bis zum unteren linken Rand der Rippen.
- Verwenden Sie die Pinzette, um die Peritonealmembran anzuheben, und machen Sie mit der chirurgischen Schere einen seitlichen Schnitt in der Oberkante der Peritonealmembran. Setzen Sie den Schnitt entlang der untersten Kante der Rippen fort.
- Identifizieren Sie die untere Hohlvene mit den beiden PBS-befeuchteten Wattestäbchen. Führen Sie eine der 30 G-Nadeln mit der 1,0-ml-Spritze in die Vena cava inferior ein und ziehen Sie dann an der Spritze. Ziehen Sie die Nadel langsam aus der unteren Hohlvene, um eine Hämolyse zu vermeiden. Übertragen Sie das Blut in die 1,0 ml Spitzröhre mit Heparin und mischen Sie es dann, indem Sie das Röhrchen mehrmals umkehren.
HINWEIS: Die Maus wird durch zervikale Dislokation eingeschläfert. - Drehen Sie das Spitzrohr bei Raumtemperatur (RT), 3.000 × g für 10 min.
- Saugen Sie den Überstand langsam ab, stellen Sie sicher, dass er kein Sediment enthält, und geben Sie ihn dann in ein unbenutztes 1,5-ml-Mikrozentrifugenröhrchen.
- Lagern Sie das Röhrchen vor Gebrauch bei −80 °C.
HINWEIS: Wenn Sie sofort mit Schritt 3 fortfahren, muss das Röhrchen nicht bei −80 °C gelagert werden.
2. Entnahme von Nierenproben
- Bereiten Sie für jede Maus Folgendes vor: 2,0 ml Kryoröhrchen, Isofluran-Anästhetikum, ein Korktuch, 70% Ethanol, eine Petrischale mit PBS, Pinzette und chirurgische Schere.
- Betäuben Sie die Maus mit 1,5% Isofluran und halten Sie sie bei 1,5%. Verabreichen Sie ein Analgetikum (Meloxicam 5 mg / kg) subkutan, injizieren Sie dann 1,0 ml 70% Ethanol in den Bauch und legen Sie es in Rückenlage auf das Korkblatt.
- Bestätigen Sie die Tiefe der Anästhesie durch das Verschwinden des Pedalrückzugsreflexes. Verwenden Sie die Pinzette und die chirurgische Schere, um einen Schnitt in die Bauchhaut zu machen. Schneiden Sie die Muskeln und die Peritonealmembran von der Blase bis zum unteren linken Rand der Rippen.
- Verwenden Sie die Pinzette, um die Peritonealmembran anzuheben und mit der chirurgischen Schere einen seitlichen Schnitt in der Oberkante der Peritonealmembran zu machen. Setzen Sie den Schnitt entlang der untersten Kante der Rippen fort.
- Identifizieren Sie die linke Niere. Refluxen Sie es mit PBS, um das Blut aus den Gefäßen zu spülen, bis die Niere eine gelblich-weiße Farbe annimmt. Schneiden Sie zuerst die gesamte Niere mit einer chirurgischen Schere aus, um die linke Nierenarterie und Vene zu durchtrennen. Legen Sie die Niere in eine Petrischale und waschen Sie sie vorsichtig mit PBS.
HINWEIS: Die Maus wird durch zervikale Dislokation eingeschläfert. - Verwenden Sie die Pinzette und die chirurgische Schere, um die Niere in 10 mg Proben zu schneiden (10 mg ist eine geeignete Probengröße für den nächsten Schritt). Legen Sie jede Probe der Niere in ein eigenes 2,0 ml Kryoröhrchen. Schließen Sie den Verschluss der Röhre.
- Für die Langzeitlagerung wird jedes Kryoröhrchen in flüssigen Stickstoff umgewandelt und bei −80 °C gelagert.
3. Gesamt-RNA-Extraktion aus einer Serumprobe
- Bereiten Sie zuerst die folgenden Elemente vor: einen Wirbelmischer, ein Phenol/Guanidin-basiertes Lysereagenz, 80% Ethanol, 100% Ethanol, 100% Chloroform, Biopolymer-Spinsäulen (in 2,0-ml-Sammelröhrchen 11), membranverankerte Spinsäulen (in 2,0-ml-Sammelröhrchen11), Waschpuffer #1 (d. h. Waschpuffer, der Guanidin und100% Ethanol im Verhältnis 1:2 enthält), Waschpuffer #2 (d. h. Waschpuffer mit Guanidin und 100% Ethanol im Verhältnis 1:4), RNase-freiem Wasser, 1,5-ml-Mikrozentrifugenröhrchen und 2,0-ml-Mikrozentrifugenröhrchen.
- Nehmen Sie zuerst eine 200-μL-Serumprobe in einem 1,5-ml-Mikrozentrifugenröhrchen und fügen Sie dann 1.000 μl des Phenol / Guanidin-basierten Lysereagenz hinzu. Wirbeln Sie die Mischung für 5 s.
- Inkubieren Sie die Probe bei RT für 5 min.
- Geben Sie 200 μL Chloroform in die Serumprobe im Röhrchen und schließen Sie die Röhrchenkappe fest. Drehen Sie das Röhrchen 15x um, um das Chloroform und die Serumprobe zu mischen.
- Jede Probe wird bei RT für 3 min inkubiert. Als nächstes drehen Sie die Probe bei 4 °C für 15 min bei 12.000 x g.
- Die 300 μL Überstand werden in ein neues 1,5 mL Mikrozentrifugenröhrchen überführt, ohne das Pellet zu stören. Fügen Sie 450 μL 100% Ethanol hinzu und wirbeln Sie das Röhrchen für 5 s.
HINWEIS: In allen folgenden Schritten legen Sie die membranverankerte Spinsäule für die Trennung von RNA und DNA in ein 2,0 ml Sammelröhrchen, um sie zu zentrifugieren. - Entfernen Sie dann 700 μL der Probe, laden Sie sie auf eine membranverankerte Spinsäule und schließen Sie die Kappe. Drehen Sie die Säule bei RT für 15 s bei 8.000 × g und lassen Sie den Überstand in der Säule. Entsorgen Sie das im Auffangrohr verbleibende Pellet.
- Fügen Sie 700 μL Waschpuffer #1, der Teil des Serum-/Plasma-Kits ist (siehe Materialtabelle), in die membranverankerte Spinsäule ein, um die Probe gründlich zu reinigen. Schließen Sie die Säulenkappe und drehen Sie die Säule bei RT für 15 s bei 8.000 × g und lassen Sie den Überstand in der Säule. Entsorgen Sie das im Auffangrohr verbleibende Pellet.
- Zur Entfernung von Spurensalzen nehmen Sie 500 μL Waschpuffer #2 und laden ihn auf eine membranverankerte Schleudersäule. Nach dem Schließen der Säulenkappe drehen Sie die Säule bei RT für 15 s bei 8.000 × g und lassen den Überstand in der Säule. Entsorgen Sie das Pellet im Auffangrohr.
- Für die Entfernung von Spurensalzen nehmen Sie 500 μL 80% Ethanol und laden es auf eine membranverankerte Spinsäule. Nach dem Schließen der Säulenkappe drehen Sie die Säule bei RT für 15 s bei 8.000 × g und lassen Sie den Überstand in der Säule. Entsorgen Sie das Pellet im Auffangrohr.
- Drehen Sie die membranverankerte Spinsäule erneut bei RT für 5 min bei 15.000 × g.
- Nach dem Transfer der membranverankerten Spinsäule in ein neues 1,5-ml-Sammelröhrchen werden 14 μL RNase-freies Wasser in die Säule gegeben, um die gesamte RNA aufzulösen. Nach dem Schließen der Säulenkappe warten Sie 5 Minuten mit dem Röhrchen bei RT. Drehen Sie die Säule erneut für 1 Minute bei 15.000 × g RT.
- Lagern Sie die Röhrchen mit Proben vor Gebrauch bei −80 °C.
4. Extraktion der Gesamt-RNA aus einer Nierenprobe
- Bereiten Sie zuerst die folgenden Elemente vor: einen Siliziumhomogenisator, Eis, einen Wirbelmischer, 100% Ethanol, 100% Chloroform, Biopolymer-Spinsäulen (in 2,0-ml-Sammelröhrchen 11), membranverankerte Spinsäulen (in 2,0-ml-Sammelröhrchen11), Phenol/Guanidin-basiertes Lysereagenz, Waschpuffer #1 (derselbe Puffer wie Schritt 3.1.), Waschpuffer #2 (derselbe Puffer wie Schritt 3.1.), RNase-freies Wasser, 1,5-ml-Mikrozentrifugenröhrchen und 2,0-ml-Mikrozentrifugenröhrchen.
- Geben Sie eine 10-mg-Nierenprobe in den Siliziumhomogenisator und fügen Sie 700 μL des Phenol/Guanidin-basierten Lysereagenz hinzu.
- Stellen Sie den Homogenisator auf. Drücken und drehen Sie den Stößel des Homogenisators vorsichtig gegen die Nierenprobe, um die Probe zu homogenisieren. Drücken und drehen Sie den Stößel weiter, bis die Nierenprobe im Phenol/Guanidin-basierten Lysereagenz vollständig homogenisiert ist.
- Zur weiteren Homogenisierung nehmen Sie das homogenisierte Lysat (in einem 2,0-ml-Sammelröhrchen) und übertragen es in eine Biopolymer-Spinsäule.
- Zentrifugieren Sie das homogenisierte Lysat bei RT für 3 min bei 14.000 × g und geben Sie dann das gesamte gefällte Pellet in ein unbenutztes 1,5-ml-Mikrozentrifugenröhrchen, um das gefällte Pellet umzukehren.
- Mischen Sie das Pellet mit 140 μL Chloroform im Röhrchen; Schließen Sie dann die Schlauchkappe fest. Drehen Sie das Röhrchen 15x um, um Lysat und Chloroform zu mischen.
HINWEIS: Das Chloroform kann sicher ohne Haube verwendet werden. - Inkubieren Sie die Probe bei RT für 2-3 min. Zentrifugieren Sie die Probe bei 4 °C für 15 min bei 12.000 × g.
- Übertragen Sie den Überstand (normalerweise ~ 300 μL) in ein neues 1,5 ml Mikrozentrifugenröhrchen, ohne den Niederschlag zu stören. Fügen Sie das 1,5-fache seines Volumens (das normalerweise ~ 450 μL beträgt) von 100% Ethanol hinzu. Wirbeln Sie die Mischung für 5 s.
HINWEIS: Legen Sie in allen folgenden Schritten die membranverankerte Spinsäule zur Trennung von RNA und DNA in ein 2,0-ml-Sammelröhrchen, um sie zu zentrifugieren. - Laden Sie 700 μL der Probe auf eine der membranverankerten Spinsäulen. Schließen Sie die Kappe und zentrifugieren Sie die Säule bei 15.000 × g für 15 s. Verwerfen Sie das im Sammelröhrchen verbleibende gefällte Lysat.
- Fügen Sie 700 μL Waschpuffer #1 in die Schleudersäule hinzu, um sie gründlich zu waschen. Schließen Sie die Kappe und zentrifugieren Sie die Säule bei 15.000 × g für 15 s. Verwerfen Sie das im Sammelröhrchen verbleibende gefällte Lysat.
- Zur Entfernung von Salzspuren 500 μL Waschpuffer #2 auf die membranverankerte Schleudersäule laden. Nach dem Schließen der Säulenkappe zentrifugieren Sie die Säule bei 15.000 × g für 15 s. Verwerfen Sie das gefällte Lysat im Sammelröhrchen.
- Wiederholen Sie Schritt 4.11.
- Zentrifugieren Sie die membranverankerte Spinsäule erneut für 1 min bei 15.000 × g. Verwerfen Sie das gefällte Lysat im Sammelröhrchen.
- Nehmen Sie die membranverankerte Spinsäule und geben Sie sie in ein neues 1,5-ml-Sammelrohr. Geben Sie 30 μL RNase-freies Wasser in die Säule, um die gesamte RNA aufzulösen. Nachdem Sie die Kappe der Säule geschlossen haben, warten Sie 5 Minuten mit dem Röhrchen bei RT und zentrifugieren Sie dann die Säule für 1 Minute bei 15.000 × g.
- Das Gesamtvolumen der Probe mit der Gesamt-RNA wird in ein neues Mikrozentrifugenröhrchen überführt. Legen Sie das Röhrchen auf Eis und messen Sie die Gesamt-RNA-Konzentration durch Spektrophotometrie. Stellen Sie sicher, dass die Gesamt-RNA-Konzentration ~ 300-1.500 ng / μL beträgt.
- Lagern Sie Röhrchen mit Proben vor Gebrauch bei −80 °C.
5. Synthese von cDNA mit der reversen Transkription der Gesamt-RNA im Serum
HINWEIS: Die Mindestinformationen für die Veröffentlichung der Richtlinien für quantitative real-time PCR-Experimente (MIQE) empfehlen die Verwendung besserer experimenteller Praktiken, um zuverlässige, eindeutige Ergebnisse zu erhalten17. In diesem Protokoll wird cDNA aus der gesamten RNA synthetisiert, die in einem zweistufigen Verfahren unter Verwendung von reverser Transkriptase, Poly(A)-Polymerase und Oligo-dT-Primern gereinigt wird.
- Bereiten Sie zuerst Folgendes vor: einen Wirbelmischer, einen Thermocycler, Acht-Well-Streifenröhrchen, die Kappe jedes Acht-Streifen-Röhrchens, destilliertes Wasser, Eis, das Reverse-Transkriptase-Kit (siehe Materialtabelle)13,14 im geschmolzenten Zustand und 1,5-ml-Mikrozentrifugenröhrchen.
- Starten Sie den Thermocycler.
- Bereiten Sie die Master-Mix-Lösung vor; Um insgesamt 8,0 μL Master-Mix pro Acht-Well-Streifenröhrchen zu erhalten, fügen Sie 2,0 μL Reverse-Transkriptase-Mix (im Kit enthalten) und 2,0 μL 10x Nukleinsäuremischung zu 4,0 μL Reverse-Transkriptionspuffer in einem 1,5-ml-Mikrozentrifugenröhrchen hinzu.
- Geben Sie 8,0 μl der Master-Mix-Lösung in jedes Röhrchen eines Acht-Well-Streifenröhrchens.
- Geben Sie ein Aliquot von 12 μL Gesamt-RNA in jedes Röhrchen des Acht-Well-Streifenröhrchens und schließen Sie die Kappe des Röhrchens. Zentrifugieren Sie das Röhrchen für 15 s bei RT und 2.000 × g.
- Das Röhrchen in den Thermocycler geben und 60 min bei 37 °C inkubieren. Inkubieren Sie die Probe für weitere 5 Minuten bei 95 °C, um die cDNA zu synthetisieren.
- Nach der Inkubation wird die cDNA in ein neues 1,5-ml-Mikrozentrifugenröhrchen überführt. Verdünnen Sie die cDNA zehnfach (1:10) mit destilliertem Wasser. Vortex und Zentrifugieren Sie das Röhrchen für 5 s bei RT und 2.000 × g.
- Lagern Sie die verdünnte cDNA vorübergehend auf Eis. Lagern Sie die verdünnten Proben vor Gebrauch bei −80 °C.
6. Synthese von cDNA mit der reversen Transkription der Gesamt-RNA in der Niere
ANMERKUNG: Die MIQE-Leitlinien fördern bessere experimentelle Praktiken, um zuverlässige und eindeutige Ergebnisse zu gewährleisten17. Dieses Protokoll verwendet Reverse-Transkriptase, Poly(A)-Polymerase und Oligo-dT-Primer, um cDNA aus 1,0 μg gereinigter Gesamt-RNA in einem zweistufigen Verfahren zu synthetisieren.
- Bereiten Sie zunächst Folgendes vor: einen Wirbelmischer, einen Thermocycler, 1,5-ml-Mikrozentrifugenröhrchen, Acht-Well-Streifenröhrchen, die Kappe jedes Acht-Streifen-Röhrchens, destilliertes Wasser, Eis und ein Reverse-Transkriptase-Kit (siehe Materialtabelle)13,14 im geschmolzenten Zustand.
- Starten Sie den Thermocycler.
- Bereiten Sie die Master-Mix-Lösung vor; Um insgesamt 8,0 μL Master-Mix-Lösung pro Röhrchen zu erhalten, fügen Sie 2,0 μL der im Kit enthaltenen reversen Transkriptasemischung und 2,0 μL 10x Nukleinsäuremischung zu 4,0 μL des reversen Transkriptionspuffers hinzu.
- Fügen Sie 8,0 μL der Master-Mix-Lösung zu jedem Röhrchen eines Acht-Well-Streifenrohrs hinzu.
- Stellen Sie die Gesamt-RNA-Dichte wie folgt ein. Für die Trennung von 1,0 μg Gesamt-RNA aus einer Nierenprobe mit 12 μL RNase-freiem Wasser wird eine geeignete Menge Gesamt-RNA entnommen und unter Verwendung der oben (in Schritt 4.15) gemessenen Konzentration in destilliertes Wasser überführt.
HINWEIS: In Gegenwart einer DNA-Kontamination wird die kontaminierte DNA durch qRT-PCR co-amplifiziert. - Führen Sie den gleichen Vorgang wie oben in den Schritten 5.5.-5.8 beschrieben durch.
7. Die qRT-PCR der miRNA
HINWEIS: Für die qRT-PCR der miRNAs wird eine Intercalator-Methode verwendet. Primer werden für RNA verwendet: U6 small nuclear 2 (RNU6-2), miRNA-223-3p, miRNA-423-5p, miRNA-7218-5p und miRNA-7219-5p.
- Bereiten Sie Folgendes vor: einen Wirbelmischer, ein real-time PCR-System, eine 96-Well-Reaktionsplatte für qRT-PCR, Klebefolie für die 96-Well-Reaktionsplatte, Klebefilmapplikator, einen 96-Well-Zentrifugenrotor, miRNA-spezifische Primer, ein grünes Farbstoff-basiertes PCR-Kit mit 2x PCR-Mastermix und 10x Universalprimer (siehe Materialtabelle)13,14, und ein 1,5 ml Mikrozentrifugenröhrchen.
- Nach dem Mischen in einem 1,5-ml-Mikrozentrifugenröhrchen werden folgende Infusionen durchgeführt: 6,25 μL destilliertes Wasser, je 1,25 μL 5 μM miRNA-Primer, gelöst in nukleasefreiem Wasser, 12,5 μL 2x PCR-Mastermix und 2,5 μL 10x Universalprimer.
- Bereiten Sie die synthetisierte cDNA vor und schmelzen Sie sie, wie in Schritt 5 (Serum) oder Schritt 6 (Niere) beschrieben. Vortex und zentrifugieren Sie die cDNA für 5 s.
- Nehmen Sie 22,5 μL Aliquots des Reagenzes (wie in Schritt 7.2. oben beschrieben) und legen Sie sie separat in jede Vertiefung der 96-Well-Platte.
- Fügen Sie jeder Vertiefung der Platte ein Aliquot von 2,5 μL cDNA hinzu.
- Um die Klebefolie auf der Platte zu befestigen, verwenden Sie den Klebefolienapplikator. Zentrifugieren Sie die Platte für 30 s bei 1.000 x g im 96-Well-Zentrifugenrotor. Stabilisieren Sie die Reaktion am Boden jeder Vertiefung.
8. Verwendung des Real-Time-PCR-Systems und der Software zum Ausführen des PCR-Zyklusprogramms
- Starten Sie das real-time PCR-System. Legen Sie die erstellte Platte wie in Schritt 7.6 beschrieben ein. im real-time PCR-System. Ändern Sie die Einstellungen; Geben Sie einen Namen für das Experiment an, und wählen Sie dann 96-well (0,2 ml ) als Experimenttyp des Systems, vergleichende CT (ΔΔCT) als Quantifizierungsmethode, Standard als Systemlaufmodus und SYBR Green Reagents als Reagenzien zum Nachweis der Zielsequenz aus.
- Geben Sie Namen für die Probe und die Ziel-miRNAs an, und benennen Sie die Probe und die Ziel-miRNA in jeder Vertiefung. Weisen Sie doppelte Proben zu, um Daten zu erhalten, die zur Bestätigung der Ergebnisse geeignet sind, und wählen Sie eine Referenzprobe und die endogene Kontrolle. Damit der Farbstoff als passive Referenz verwendet werden soll, wählen Sie Keine. Um eine Reagenzkreuzkontamination zu eliminieren, richten Sie eine negative Reverse-Transkriptase und eine Nicht-Template-Kontrolle für die miRNA-Expression ein.
- Als nächstes stellen Sie sicher, dass das Reaktionsvolumen auf 20 μL eingestellt ist und die PCR-Zyklusbedingungen wie folgt eingestellt sind: 95 °C für 15 min, dann 40 Denaturierungszyklen bei 94 °C für 15 s, Glühen bei 55 °C für 30 s und schließlich Verlängerung bei 70 °C für 30 s.
- Klicken Sie im Softwareprogramm des Systems auf Analysieren , um die qRT-PCR-Daten nach Abschluss des Vorgangs zu analysieren. Vergewissern Sie sich, dass die vom Programm automatisch ausgewählte Schwellenwertlinie für jede Vertiefung geeignet ist.
- Überprüfen Sie den Schwellenwert für den Zyklus (CT) der endogenen Kontroll- und Ziel-miRNAs, die in jeder Probe analysiert wurden. Bestimmen Sie die CT-Werte durch den Schnittpunkt der Verstärkungskurve und der Schwellenwertlinie.
HINWEIS: In der vorliegenden Studie wurden RNU6-2 und miRNA-423-5p als endogene Kontrollen für Ziel-miRNA-Expressionsniveaus verwendet, und die ΔΔCT-Methode wurde verwendet, um die relativen Expressionsniveaus jeder Ziel-miRNA18 zu bestimmen.
Ergebnisse
Für das Mausmodell mit altersabhängiger Nierenfunktionsstörung verwendeten wir 50 Wochen alte männliche SAMP1-Mäuse mit einem Gewicht von 40-45 g. Ungefähr 0,8 ml Blut wurden pro Maus gesammelt und in ein 1,0 ml Spitzröhrchen mit Heparin überführt, invertiert und zentrifugiert. Jede Niere wurde mit PBS gespült, seziert und zur weiteren Analyse in flüssigem Stickstoff gelagert. Fünfzig Wochen alte SAMR1-Mäuse dienten als Kontrollen. Basierend auf den miRNA qRT-PCR-Daten, die mit diesem altersabhängigen Nierenfunktionsmodell gewonnen wurden, beobachteten wir, dass der Nierenspiegel von miRNA-7219-5p signifikant erhöht und der Nierenspiegel von miRNA-7218-5p bei den SAMP1-Mäusen im Vergleich zu den Kontrollen signifikant erniedrigt war (Abbildung 1). Die Serumspiegel von miRNA-7219-5p und miRNA-7218-5p waren bei den SAMP1-Mäusen im Vergleich zu den Kontrollen erheblich erhöht (Abbildung 2). Die Expressionsniveaus von miRNA-223-3p änderten sich in keinem der beiden Stämme und zwischen Niere und Serum (Abbildung 1 und Abbildung 2).

Abbildung 1: Differentiell exprimierte microRNAs in den Nieren von SAMP1-Mäusen. qRT-PCR-Analyse der Expression von miRNA-223-3p, miRNA-7218-5p und miRNA-7219-5p in SAMR1-Mäusen (Kontrolle, n = 4) und SAMP1-Mäusen (n = 4). Die Daten sind Mittelwert ± Standardfehler (Fehlerbalken); t-Tests wurden verwendet, um Unterschiede zwischen Gruppen zu analysieren; p < 0,05 wurde als signifikant (*p < 0,05), n.s.: nicht signifikant angesehen. Abkürzungen: miRNA = microRNA; SAMP1 = seneszenzbeschleunigte Maus anfällig; SAMR1 = seneszenzbeschleunigter Mauswiderstand 1; qRT-PCR = quantitative Reverse-Transkription-Polymerase-Kettenreaktion. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
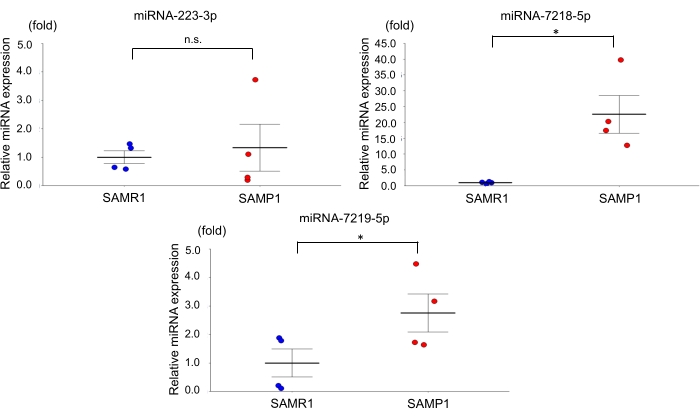
Abbildung 2: Differentiell exprimierte miRNAs im Serum von SAMP1-Mäusen. qRT-PCR-Analyse der Expression von miRNA-223-3p, miRNA-7218-5p und miRNA-7219-5p in SAMR1-Mäusen (Kontrolle, n = 4) und SAMP1-Mäusen (n = 4). Die Daten sind Mittelwert ± SE (Fehlerbalken); T-Tests wurden verwendet, um signifikante Unterschiede zwischen den Gruppen zu untersuchen. *P < 0,05 durch t-Test. Abkürzungen: miRNA = microRNA; SAMP1 = seneszenzbeschleunigte Maus anfällig; SAMR1 = seneszenzbeschleunigter Mauswiderstand 1; qRT-PCR = quantitative Reverse-Transkription-Polymerase-Kettenreaktion Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Die Expressionsniveaus der Ziel-miRNAs wurden erfolgreich durch das oben beschriebene Protokoll mittels qRT-PCR bestimmt. Die Auswertung der extrahierten miRNAs ist ein wichtiger Schritt, um aussagekräftige qRT-PCR-Daten zu erhalten. Um die adäquate Qualität der miRNAs vor der Durchführung der qRT-PCR zu bestätigen, sollte mittels Spektrophotometrie das Verhältnis der Absorption bei 260 nm zu der bei 280 nm bestimmt werden. DNA-Verunreinigungen können auftreten, und/oder Primer-Dimere in jeder Vertiefung der Reaktionsplatte können vorhanden sein, wenn die qRT-PCR keine einzige PCR-Amplifikation der erwarteten Länge und Schmelztemperatur liefert oder wenn sie eine monomodale Schmelzkurve liefert.
MiRNA-Expressionsniveaus können mit verschiedenen Methoden als qRT-PCR bewertet werden, einschließlich Northern Blotting, einem Microarray und Ribonuklease-Schutzassays. Die qRT-PCR-Methode ist jedoch ein empfindliches, genaues, einfaches und reproduzierbares Verfahren, das ein kleineres Probenvolumen erfordert als für Northern Blotting und Ribonuklease-Schutzassays19. Da Microarrays die Expression von Zehntausenden von miRNAs gleichzeitig messen können, können sie verwendet werden, um miRNA-Markerkandidaten zu identifizieren. Microarray-Daten zeigen auch eine hohe Gesamtkorrelation mit Daten, die mit qRT-PCR20 gewonnen wurden. Es wurde jedoch kein Konsens über die optimale Methodik für den Vergleich von Microarray-Daten erzielt, die in verschiedenen Studien gewonnen wurden21.
Die Auswertung von Serum-miRNAs weist folgende Merkmale auf. Erstens ist es einfach, Serum zu sammeln, und da Serum-miRNAs stabil gegen Einfrieren und Auftauen, Temperatur und Säure sind, können die miRNAs gute Biomarker sein. Zweitens gibt es eine hohe Homologie von miRNAs zwischen den Arten, und die Ergebnisse von Tierversuchen lassen sich leicht auf den Menschen übertragen. Drittens haben Serum-miRNAs Potenzial für den Einsatz als therapeutische Medikamente gezeigt3. Mehrere Studien haben auch gezeigt, dass der Grad der Expression von miRNA in Organen mit miRNA im Serumkorreliert ist 22,23,24. In der vorliegenden Studie zeigte miRNA-223-3p, deren Nierenspiegel keinen signifikanten Unterschied zwischen den SAMR1- und SAMP1-Mäusen zeigten, ebenfalls keinen signifikanten Unterschied zwischen den Stämmen im Serum. Im Gegensatz dazu zeigten miRNA-7218-5p und miRNA-7219-5p, deren Nierenspiegel einen signifikanten Unterschied zwischen den SAMR1- und SAMP1-Mäusen zeigten, erhebliche Unterschiede zwischen den Stämmen im Serum.
Für dieses Protokoll gelten die folgenden Einschränkungen. Erstens wurde seine Nützlichkeit in anderen Organen wie Leber und Lunge nicht nachgewiesen, und zweitens wurde es nicht an anderen Labortieren wie Ratten, Hunden und Schweinen getestet. Mehrere Forschungsgruppen haben dieses Protokoll für die Aufreinigung und den Nachweis von miRNAs mittels qRT-PCR verwendet und berichtet, dass dieses Protokoll die Reinigung von hochwertiger RNA aus Geweben und Serumermöglicht 13,14,22,23,24. Diese Methode hat nachweislich eine hohe Genauigkeit und Empfindlichkeit zum Nachweis der Expression von miRNAs 13,14,22,23,24. Die Ergebnisse der vorliegenden Studie zeigen, dass dieses Protokoll die miRNA-Expression im Serum und in der Niere von Mäusen erfolgreich nachweisen kann. Daher kann das Protokoll verwendet werden, um die Serum- und Nieren-miRNA-Expressionsprofile bei Mäusen mit einer Vielzahl von Pathologien zu bestimmen. Aufgrund der Einfachheit des Protokolls kann eine große Anzahl von Proben gleichzeitig verarbeitet werden. Analysen der Expression vieler miRNAs bei verschiedenen pathologischen Zuständen der Niere können daher das hierin beschriebene Protokoll verwenden.
Es gibt bestimmte Aspekte des Protokolls zu beachten. Um den Abbau der gereinigten miRNAs zu vermeiden, der bei Raumtemperatur auftreten würde, müssen die miRNAs auf Eis gehalten werden. Die Nierenproben müssen homogenisiert werden, bis sie vollständig im Lysereagenz gelöst sind. Mausniere enthält eine beträchtliche Menge an Bindegewebe, das in Lysereagenz unlöslich ist, und daher ist ein Säulenschredder für die weitere Homogenisierung erforderlich. Darüber hinaus sollte die geeignete endogene Kontroll-miRNA (mit stabiler Expression unter den Proben) während des Aufbaus eines qRT-PCR-Experiments validiert werden. Dies liegt daran, dass die Interferenz verschiedener Substanzen während der Durchführung dieses Protokolls die Expressionsniveaus endogener Kontroll-miRNAs verändern kann, was möglicherweise die Ergebnisse beeinträchtigt. Zusammenfassend beschreibt dieser Artikel ein qRT-PCR-Protokoll zum Nachweis, zur Aufreinigung und Bewertung der miRNA-Expression im Serum und in der Niere von Mäusen mit altersabhängiger Nierenfunktionsstörung.
Offenlegungen
Die Autoren erklären, dass sie keine Interessenkonflikte haben.
Danksagungen
Nichts.
Materialien
| Name | Company | Catalog Number | Comments |
| 1.0 mL spitz with heparin | Greiner-bio-one | 450534 | |
| Buffer RPE (wash buffer #2 containing guanidine and ethanol in ratio of 1:4) | Qiagen | 79216 | Wash buffer 2 |
| Buffer RWT (wash buffer #1 containing guanidine and ethanol in ratio of 1:2) | Qiagen | 1067933 | Wash buffer 1 |
| MicroAmp Optical 96-well reaction plate for qRT-PCR | Thermo Fisher Scientific | 4316813 | 96-well reaction plate |
| MicroAmp Optical Adhesive Film | Thermo Fisher Scientific | 4311971 | Adhesive film for 96-well reaction plate |
| miRNA-223-3p primer | Qiagen | MS00003871 | 5'-CGUGUAUUUGACAAGCUGAGUU G-3' |
| miRNA-423-5p primer | Qiagen | MS00012005 | 5'-UGAGGGGCAGAGAGCGAGACU UU-3' |
| miRNA-7218-5p primer | Qiagen | MS00068067 | 5'-UGCAGGGUUUAGUGUAGAGGG -3' |
| miRNA-7219-5p primer | Qiagen | MS00068081 | 5'-UGUGUUAGAGCUCAGGGUUGA GA-3' |
| miRNeasy Mini kit | Qiagen | 217004 | Membrane anchored spin column in a 2.0 mL collection tube |
| miRNeasy Serum/Plasma kit | Qiagen | 217184 | Membrane anchored spin column in a 2.0 mL collection tube |
| miScript II RT kit (reverse transcription buffer) | Qiagen | 218161 | Reverse transcriptase kit |
| miScript SYBR Green PCR kit | Qiagen | 218073 | Green dye-based PCR kit |
| QIA shredder | Qiagen | 79654 | Biopolymer spin columns in a 2.0 mL collection tube |
| QIAzol Lysis Reagent (phenol/guanidine-based lysis reagent) | Qiagen | 79306 | Phenol/guanidine-based lysis reagent |
| QuantStudio 12K Flex Flex Real-Time PCR system | Thermo Fisher Scientific | 4472380 | Real-time PCR instrument |
| QuantStudio 12K Flex Software version 1.2.1. | Thermo Fisher Scientific | 4472380 | Real-time PCR instrument software |
| RNase-free water | Qiagen | 129112 | |
| RNU6-2 primer | Qiagen | MS00033740 | Not disclosed due to confidentiality |
| SAMP1 male mice | Nippon SLC Corporation | Not assigned | |
| SAMR1 male mice | Nippon SLC Corporation | Not assigned | |
| Takara biomasher standard | Takara Bio | 9790B | Silicon homogenizer |
Referenzen
- Huang, Y. The novel regulatory role of lncRNA-miRNA-mRNA axis in cardiovascular diseases. Journal of Cellular and Molecular Medicine. 22 (12), 5768-5775 (2018).
- Yang, C., Dou, R., Yin, T., Ding, J. MiRNA-106b-5p in human cancers: diverse functions and promising biomarker. Biomedicine and Pharmacotherapy. 127, 110211 (2020).
- Lu, T. X., Rothenberg, M. E. MicroRNA. The Journal of Allergy and Clinical Immunology. 141 (4), 1202-1207 (2018).
- McGuire, A., Brown, J. A., Kerin, M. J. Metastatic breast cancer: the potential of miRNA for diagnosis and treatment monitoring. Cancer and Metastasis Reviews. 34 (1), 145-155 (2015).
- Bjorkman, S., Taylor, H. S. MicroRNAs in endometriosis: biological function and emerging biomarker candidates. Biology of Reproduction. 100 (5), 1135-1146 (2019).
- Zhou, C. X., et al. miRNA and circRNA expression patterns in mouse brain during toxoplasmosis development. BMC Genomics. 21 (1), 46 (2020).
- Jing, R., Zhong, Q. Q., Long, T. Y., Pan, W., Qian, Z. X. Downregulated miRNA-26a-5p induces the apoptosis of endothelial cells in coronary heart disease by inhibiting PI3K/AKT pathway. European Review for Medical and Pharmacological Sciences. 23 (11), 4940-4947 (2019).
- Xie, W., et al. miR-34b-5p inhibition attenuates lung inflammation and apoptosis in an LPS-induced acute lung injury mouse model by targeting progranulin. Journal of Cellular Physiology. 233 (9), 6615-6631 (2018).
- Bala, S., et al. Circulating microRNAs in exosomes indicate hepatocyte injury and inflammation in alcoholic, drug-induced, and inflammatory liver diseases. Hepatology. 56 (5), 1946-1957 (2012).
- Ishii, H., et al. MicroRNA expression profiling in diabetic kidney disease. Translational Research. 237, 31-52 (2021).
- Mastropasqua, R., et al. Serum microRNA levels in diabetes mellitus. Diagnostics (Basel). 11 (2), 284 (2021).
- Gordanpour, A., Nam, R. K., Sugar, L., Bacopulos, S., Seth, A. MicroRNA detection in prostate tumors by quantitative real-time PCR (qPCR). Journal of Visualized Experiments: JoVE. (63), e3874 (2012).
- Ahn, J. H., Kwak, J., Lee, J. H., Lee, S. S. Efficient and accurate analysis of microRNA using a specific extension sequence. Molecular Biology Reports. 45 (4), 611-619 (2018).
- Denic, A., Glassock, R. J., Rule, A. D. Structural and functional changes With the aging kidney. Advances in Chronic Kidney Disease. 23 (1), 19-28 (2016).
- Weinstein, J. R., Anderson, S. The aging kidney: physiological changes. Advances in Chronic Kidney Disease. 17 (4), 302-307 (2010).
- Bustin, S. A., et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Clinical Chemistry. 55 (4), 611-622 (2009).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Rajeevan, M. S., Vernon, S. D., Taysavang, N., Unger, E. R. Validation of array-based gene expression profiles by real-time (kinetic) RT-PCR. The Journal of Molecular Diagnostics. 3 (1), 26-31 (2001).
- Chen, Y., Gelfond, J. A., McManus, L. M., Shireman, P. K. Reproducibility of quantitative RT-PCR array in miRNA expression profiling and comparison with microarray analysis. BMC Genomics. 10, 407 (2009).
- Dallas, P. B., et al. Gene expression levels assessed by oligonucleotide microarray analysis and quantitative real-time RT-PCR -- how well do they correlate. BMC Genomics. 6, 59 (2005).
- Petriella, D., et al. miRNA profiling in serum and tissue samples to assess noninvasive biomarkers for NSCLC clinical outcome. Tumour Biology. 37 (4), 5503-5513 (2016).
- Skrzypa, M., et al. miRNA-146a-5p is upregulated in serum and cartilage samples of patients with osteoarthritis. Polski Przeglad Chirurgiczny. 91 (3), 1-5 (2019).
- Farzanehpour, M., et al. Serum and tissue miRNAs: potential biomarkers for the diagnosis of cervical cancer. Journal of Virology Journal. 16 (1), 116 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten