Method Article
Quantifizierung von Zell-Substrat-Adhäsionsflächen und Zellformverteilungen in MCF7-Zell-Monolayern
In diesem Artikel
Zusammenfassung
Der Artikel beschreibt die Quantifizierung von 1) der Größe und Anzahl der fokalen Verklebungen und 2) Zellformindex und seine Verteilung von konfokalen Bildern der konfluenten Monolayer von MCF7-Zellen.
Zusammenfassung
Die hier vorgestellten Methoden quantifizieren einige Parameter von konfluent haftenden Zellmonolayern aus mehreren entsprechend gefärbten konfokalen Bildern: Haftung auf dem Substrat in Abhängigkeit von Anzahl und Größe der Fokaladesionen und Zellform, gekennzeichnet durch den Zellformindex und andere Formdeskriptoren. Fokale Adhäsionen wurden durch Paxillin-Färbung visualisiert und Zellzellränder wurden durch Kreuzung Plakoglobin und Actin gekennzeichnet. Die Methoden für Zellkultur und Färbung waren Standard; Bilder stellen einzelne Fokalebenen dar; Die Bildanalyse wurde mit einer öffentlich zugänglichen Bildverarbeitungssoftware durchgeführt. Die vorgestellten Protokolle werden verwendet, um die Anzahl und Größe der fokalen Adhäsionen und die Unterschiede in der Zellformverteilung in den Monolayern zu quantifizieren, aber sie können für die Quantifizierung der Größe und Form jeder anderen unterschiedlichen Zellstruktur, die gefärbt werden kann (z. B. Mitochondrien oder Kerne), umfunktioniert werden. Die Bewertung dieser Parameter ist wichtig für die Charakterisierung der dynamischen Kräfte in der anhaftenden Zellschicht, einschließlich Zellhaftung und Actomyosin-Kontraktilität, die sich auf die Zellform auswirkt.
Einleitung
Epithelzellmonolayer fungieren als Kollektiv, in dem Zell-Zell- und Zellsubstratadhäsion sowie kontraktile Kräfte und Spannungen wichtige Parameter darstellen und ihr richtiges Gleichgewicht zur Gesamtintegrität der Einheit1,2,3beiträgt. Die Bewertung dieser Parameter stellt daher eine Möglichkeit dar, den aktuellen Status der Zellschicht festzulegen.
Die beiden hier beschriebenen Methoden stellen eine zweidimensionale Analyse der konfluenten Monolayer von anhaftenden Epithelzellen dar (in diesem Fall MCF7 Brustkrebszelllinie). Die Analyse erfolgt mit konfokalen Bildern (einzelne Z-Slices) aus verschiedenen Regionen auf der Z-Achse; Basalbereich in der Nähe eines Substrats für fokale Adhäsionsmessungen (FA) und apikaler Bereich für Zellformmessungen. Die vorgestellten Methoden sind relativ einfach und erfordern Standardlabortechniken und Open-Source-Software. Die konfokale Mikroskopie ist für dieses Protokoll ausreichend, so dass sie ohne speziellere TIRF-Mikroskopie (Total Internal Reflection Fluorescence) durchgeführt werden kann. Somit könnte das Protokoll in einer relativ standardisierten Laborumgebung implementiert werden. Obwohl die Genauigkeit der Methoden begrenzt ist, können sie grundlegende Unterschiede in der fokalen Haftung und Zellform unterscheiden.
Beide hier beschriebenen Methoden bestehen aus den üblichen experimentellen Verfahren wie Zellkultivierung, Immunfärbung, konfokale Bildgebung und Bildanalyse, die mit ImageJ durchgeführt werden. Es kann jedoch jede Bildverarbeitungssoftware mit den entsprechenden Funktionen verwendet werden. Die vorgestellten Methoden können Veränderungen nachverfolgen und vergleichen, die durch pharmakologische Behandlung oder minimale genetische Veränderung verursacht werden. Aufgrund der begrenzten Genauigkeit dieser Methoden wird nicht empfohlen, bestimmte Werte zu erhalten. Zwei automatisierte Makros wurden aufgenommen, um die Messung vieler Bilder zu erleichtern.
Protokoll
1. Vorbereitende Schritte
- Zellaussaat zur Gewinnung von konfluenten Monolayern
- Vor der Aussaat die Brunnen einer 4-Wells-Kammerrutsche mit Kollagen I (oder einer anderen ECM-Komponente nach Wahl) beschichten. Für die Kollagen-I-Beschichtung folgen Sie einem kommerziellen Protokoll: https://www.sigmaaldrich.com/technical-documents/articles/biofiles/collagen-product-protocols.html bei einer Konzentration von 8 g/cm2.
- Samen 400.000 Zellen zu einem Brunnen einer 4-Well-Kammerrutsche.
- Kultur die Zellen für 24 h (oder länger, je nach den experimentellen Endpunkten) vor der Färbung, in einem Inkubator bei 37 °C, 5%CO2. Dieser Schritt ermöglicht die Reifung von Zell-Zell-Kontakten und die Bildung von Monolayern.
- Verwenden Sie ein optisches, invertiertes Mikroskop, um die Konfluenz (ca. 90% erforderlich) und einen allgemeinen Zustand von Monolayern zu überprüfen. Gehen Sie nicht vor, wenn Zellen schweben oder gestresst aussehen.
- Immunfluoreszierende Färbung
HINWEIS: Zellen können mit einem Protokoll Ihrer Wahl befleckt werden. Hier wurde die Immunfluoreszenz wie zuvor beschrieben4durchgeführt.- Für die fokale Adhäsionsanalyse färben Sie die Zellen mit einem fokalen Adhäsionsprotein der Wahl (in diesem Protokoll paxillin). Für die Zellformanalyse Fleck mit Zell-Zell-Knoten-Protein der Wahl (in diesem Protokoll desmosomalProtein Plakoglobin).
- Verwenden Sie für das Verfahren 0,5 ml der angegebenen Lösungen, sofern nicht anders angegeben.
- Fix Zellen in 4% Formaldehyd in PBS für 30 min auf Eis.
- Mit 0,1 M Ammoniumchlorid (in PBS) für 10 min zur Abschreckung der Autofluoreszenz inkubieren.
- Fügen Sie 0.5% Triton X-100 (in PBS) für 30 min (Permeabilisierung).
- Block mit 5% Milch (oder 1% BSA) in TBS-T für 1 h.
- Inkubieren mit primärem Antikörper bei 4 °C über Nacht (Anti-Paxillin: Kaninchen, 1:250, Antiplakoglobin: Kaninchen, 1:400)
- Inkubat mit Sekundärantikörper für 30 min (Alexa Fluor 594 Ziege Anti-Kaninchen, 1:500, 1:500)
- Fleck mit 300 nM DAPI für 1-5 min, lichtgeschützt.
HINWEIS: Optional kann eine zusätzliche Färbung von Actin mit fluoreszierenden Phalloidin-Konjugaten (konz. 1:400) durchgeführt werden; Phalloidin sollte im selben Schritt wie der sekundäre Antikörper hinzugefügt werden.
- Konfokale Bildgebung
- Nehmen Sie Einzelne Z-Scheiben mit einem konfokalen Mikroskop (z. B. Zeiss LSM800) auf.
HINWEIS: Optionale Aktinfärbung kann helfen, die richtige Brennebene zu beurteilen. Die kortikale Aktinfärbung ist im apikalen Bereich vorhanden, während Actin-Spannungsfasern im Basalbereich in der Nähe des Substrats vorhanden sind, wie in Abbildung 1dargestellt. - Fokale Adhäsions-Bildgebung
- Wählen Sie Z-Slices für die fokale Haftungsanalyse aus dem Basalbereich, in der Nähe des Substrats.
- Verwenden Sie ein Objektiv mit der höchsten verfügbaren numerischen Blende (vorzugsweise 63x N.A.: 1.4).
HINWEIS: Die Form von FA ist sehr spezifisch und leicht erkennbar. Daher wird empfohlen, den Scan von einer Brennebene unter den Zellen zu starten und dann langsam in Richtung zu ihnen zu scannen, bis FAs deutlich sichtbar sind. Die Bildanalyse wird aus kleineren Sichtfeldern genauer, die tendenziell einheitlicher sind, aber dies impliziert, dass weniger Zellen in einem einzigen Sichtfeld berechnet werden.
- Zell-Zell-Kontakte-Bildgebung
- Verwenden Sie ein 40x- oder 63x-Objektiv.
- Wählen Sie Kanäle für die Kernfärbung und die bevorzugte Knotenproteinfärbung aus.
- Wählen Sie Z-Slices für die Zellformanalyse aus dem apikalen Bereich der Monolayer aus.
- Nehmen Sie Bilder für mindestens 3 verschiedene Sichtfelder (200-400 Zellen) auf.
- Nehmen Sie Einzelne Z-Scheiben mit einem konfokalen Mikroskop (z. B. Zeiss LSM800) auf.
2. Bildanalyse
HINWEIS: Vorausgesetzt, Makros funktionieren optimal auf ImageJ Version 1.50f oder neuer. Verwendung nur zur Quantifizierung von Bildern mit einem hohen Signal-Rausch-Verhältnis und ohne unter- oder übersättigte Pixel. Die beschriebenen Methoden umfassen Schritte, die eine manuelle Parameteranpassung erfordern. Daher wird eine blinde Analyse/blinde Experimenteinrichtung empfohlen. Zum Verschlüsseln von Bilddateinamen können ImageJ-Plugins wie "Blind Analysis Tool" (verfügbar unter: https://imagej.net/Blind_Analysis_Tools) verwendet werden.
- Fokale Haftungsanalyse
HINWEIS: Die empfohlenen Eingabedateien für die folgenden Methoden sind: Bilder von FAs, die in 8-Bit-Graustufen im .tiff-Format gespeichert sind.- Öffnen Sie das Bild mit ImageJ.
- Festlegen des Maßstabs eines Bildes auf Pixel(Analysieren | Set Skala; Entfernen Sie Scale, und aktivieren Sie die Option Global).
- Den Dateinamen und den ROI-Bereich in die Messoptionen einbeziehen (Analysieren | Messen einstellen...); Optionen Bereich und Anzeigebeschriftung aktivieren.
- Hintergrund subtrahieren (Prozess | Hintergrund subtrahieren; Rollkugelradius-Parameter auf 50 Pixel setzen; Option Schiebeparaboloid prüfen). Bei pseudofarbenen RGB-Bildern: Geteilte RGB-Kanäle, lassen Sie den Kanal mit FAs geöffnet, schließen Sie die verbleibenden Kanäle (Bild | Farbe | Split-Kanäle).
- Bestimmen Sie die Fläche der kleinsten Interessenregion (ROI). Mit Freihand- oder Polygonauswahl umreißt die kleinste einzelne Fokushaftung und misst deren Fläche (Analysieren | Maßnahme). Wiederholen Sie diesen Schritt für verschiedene ROIs (FAs) aus einigen zufällig ausgewählten Bildern (für insgesamt 20 ROIs). Berechnen und speichern Sie den Mittelwert der erhaltenen Ergebnisse.
HINWEIS: Dieser Schritt ist nur erforderlich, wenn ein bestimmter Satz von Bildern zum ersten Mal analysiert wird (spezifische Zelllinie, Beschichtungsschlitten mit spezifischen extrazellulären Matrixkomponenten, unterschiedliche Kulturbedingungen). - Konvertieren Sie Image in Binärdatei mit einer der folgenden Methoden:
- Festlegen des globalen Schwellenwerts(Bild | Anpassen | Schwellenwert; Überprüfen Sie die Optionen Standard, B&W und Dark Background, passen Sie den Schwellenwert manuell an oder legen Sie ihn automatisch fest).
- Legen Sie den lokalen Schwellenwert fest(Bild | Anpassen | Auto Lokaler Schwellenwert...; Legen Sie Methode auf Phansalkar fest und aktivieren Sie Weiße Objekte auf schwarzem Hintergrund. Als Nächstes invertieren Sie das Bild (Bearbeiten | Invertieren).
- Messen Sie die Anzahl und die Fläche von ROIs. Analysieren auswählen | Analysieren von Partikeln; Überprüfen Sie Pixeleinheiten, Anzeigeergebnisse, Ergebnisse löschen und Optionen zusammenfassen, legen Sie Größenparameterfest , als untere Grenze verwenden Sie den Mittelwert der kleinsten ROIs aus Schritt 2.1.5. Die obere Grenze kann auf 25 % einer typischen Zellfläche festgelegt werden.
- Übertragen Sie Daten (einschließlich Bildname, Anzahl der FAs, Gesamt- und Mittelfläche von FAs; bzw. Slice, Count, Total Area, Average Size) aus dem Fenster Zusammenfassung in das Datenverwaltungsprogramm Ihrer Wahl.
- Bestimmen Sie die Anzahl der Zellen pro Bild, indem Sie DAPI-befleckte Kerne zählen. Zählen kann manuell durchgeführt werden (Plugins | Analysieren | Zellenzähler) oder wie in verfügbaren Protokollen wie: https://imagej.net/Nuclei_Watershed_Separation.
- Alternativ können Sie das angefügte ImageJ-Makro (FAs.ijm) verwenden, um die Zählung von FAs zu erleichtern.
- Verschieben Sie .ijm Datei mit dem Makro in Plugins oder Makros Ordner in ImageJ Quelldateien Ordner befindet.
- Bestimmen Sie einen Bereich mit dem kleinsten ROI, wie in Schritt 2.1.4 beschrieben.
- Makrodatei öffnen (Plugins | Makros | Bearbeiten...).
- Vor dem Ausführen des Makros legen Sie den Wert von drei Variablen fest: Füllwert von area_of_the_smallest_region_of_interest mit einer Zahl, die während Schritt 2.1.4 erworben wurde. Legen Sie threshold_type Wert auf manuell oder automatischfest.
- Änderungen speichern (das Makro sollte einsatzbereit sein).
- Rufen Sie das Makro aus dem ImageJ-Bedienfeld auf, oder erstellen Sie eine Verknüpfung dazu. Das Makro beginnt mit dem standardmäßig geöffneten Dialogfenster. Wählen Sie das zu verarbeitende Bild aus.
HINWEIS: Im Falle einer manuellen Schwellenwertanpassung ist eine manuelle Bestätigung des Schwellenwerts erforderlich (vermeiden Sie das Akzeptieren von Änderungen mithilfe der Schaltfläche Anwenden im Dialogfenster Schwellenwert, verwenden Sie stattdessen das benutzerdefinierte Dialogfeld Aktion erforderlich). Die Ergebnisse, die durch arbeiten mit dem Makro erzielt werden, sind die gleichen wie in Schritt 2.1.8 beschrieben (im Dialogfenster Zusammenfassung enthalten). Darüber hinaus wird bei der manuellen Schwellenwertanpassung der untere Schwellenwertpegel in einem Log-Dialogfenster angezeigt, da dieser Wert es ermöglicht, erhaltene Ergebnisse bei Bedarf in der Zukunft zu reproduzieren. Die Zusatzfiguren S1 und S2 wurden als Trainingsdatensatz für das Makro FAs.ijm aufgenommen.
- Zellformanalyse
- Manuell
- Öffnen Sie ein Bild in ImageJ oder einer anderen Bildverarbeitungssoftware mit einem ähnlichen Satz von Funktionen (weitere Anweisungen zu ImageJ). Wählen Sie die zu messenden Parameter aus, indem Sie im Menü Analyse | Legen Sie Messungen fest und kreuzen Sie Shape-Deskriptoren im angezeigten Feld an.
- Mit dem Freehand-Auswahlsymbol können Sie Zellränder, die durch Junction-Proteine der Wahl gekennzeichnet sind, manuell abgrenzen. Die ausgewählten Parameter werden automatisch für jede Zelle berechnet. Speichern Sie die Ergebnisse nach der Gliederung der einzelnen Zellen, indem Sie auf Bearbeiten | Auswahl | Zum Manager hinzufügen. Es sollten nur vollständige, vollständig sichtbare Zellen mit ununterbrochenen Rahmen gezählt werden.
- Wenn alle Zellen im Sichtfeld umrissen sind, führen Sie die Messung durch Markieren aller Zahlen aus, die im linken Feld des ROI-Managers (den Zellen entsprechen) und auf Messen klicken Die Ergebnisse werden im Feld Ergebnisse angezeigt und können in die Kalkulationstabelle ihrer Wahl übertragen werden.
- Automatisierte
ANMERKUNG: Um die Quantifizierung von Zellformdeskriptoren (CSI, Seitenverhältnis, Rundheit, Solidität) zu erleichtern, wurde ein ImageJ-Makro vorbereitet und diesem Artikel (CSI.ijm) beigefügt. Das Makro basiert hauptsächlich auf DemImageJ-Plugin namens MorphoLibJ (https://imagej.net/MorphoLibJ)5. Das Makro führt die folgenden Schritte aus: 1) Erweiterung jedes Bildrahmens um 10 schwarze Pixel [MorpholibJ]; 2) Runden von Dilatationen und Erosionen - morphologischer Filter [MorpholibJ]; 3) Erzeugung von binären Bild von Zellgrenzen durch morphologische Segmentierung [MorpholibJ]; 4) Dilatation von Zellgrenzen; 5) Umkehrung des Pixelwertes; 6) Generierung von Auswahlen und Messung von Fläche und Umfang der Zellen auf dem Bild; und 7) Speichern des Bildes mit umrissenen Zellen und ImageJ ROI-Auswahl in einer neuen Datei.- Verschieben Sie die .ijm-Datei mit dem Makro in den Plugins- oder Makroordner, der sich in den ImageJ-Quelldateien befindet. Rufen Sie das Makro aus dem ImageJ-Bedienfeld auf, oder erstellen Sie eine Verknüpfung dazu.
- Bestimmen Sie vor der Quantifizierung eines neuen Datasets die Werte der kleinsten und größten Interessengebiete. Gliederung (Freihand- oder Polygonauswahl) einige (3-10) Beispiele der kleinsten und größten Zellen auf dem Bild und dann deren Fläche messen (Analysieren | Maßnahme).
- Alternativ können Sie das Makro mit den Standardeinstellungen ausführen (die untere Größenbeschränkung ist auf 0 und die obere Grenze auf unendlich festgelegt), warten Sie, bis das Makro beendet ist, und wählen Sie die Option Zellengrößengrenzen festlegen aus. Messen Sie die Fläche der kleinsten und größten Zellen, indem Sie auf ihr Etikett klicken und dann im ImageJ ROI Manager auf Messen drücken. Legen Sie den Wert von the_smallest_cell und the_biggest_cell Variablen fest. Speichern Sie Änderungen, schließen Sie alle Makrodialogfenster, und führen Sie das Makro erneut aus.
HINWEIS: Das Makro kann ohne Festlegen von ROI-Größengrenzen verwendet werden, wird jedoch nicht empfohlen, da es die Wahrscheinlichkeit, ungeeignete Zellfragmente oder Zellcluster zu messen, erheblich erhöht. - Starten Sie das Makro mit dem standarddialogfeld Öffnen. Wählen Sie das zu verarbeitende Bild (Graustufen).
- Analysieren Sie die Ergebnisse. Die Ausgabe des Makros besteht aus: Tabelle der Ergebnisse (Zellbeschriftung, Bildbeschriftung, Zellbereich [Pixel2], Zellperimeter [Pixel2], Zirkularität [CSI], Seitenverhältnis, Rundheit, Solidität), Bild mit umrissenen Zellen und ROI-Auswahlliste (die auch in einer neuen Datei im Unterordner Ergebnisse gespeichert wird). Die Ergebnistabelle wird automatisch in die Zwischenablage des Benutzers kopiert.
HINWEIS: Die Zusatzzahlen S3 und S4 wurden als Trainingsdatensatz für das CSI.ijm-Makro aufgenommen.
- Manuell
3. Quantifizierung
- Quantifizierung von FAs
- Berechnen Sie die durchschnittliche FAs-Zahl und die durchschnittliche FA-Größe pro Zelle/Kerne.
HINWEIS: Bei einigen Zelllinien ist es möglich, FAs getrennt in verschiedenen Zellen zu zählen. Bei Zelllinien, die starke Zellzellenkontakte haben und als Monolayer wie MCF7 wachsen, können Anzahl und Größe der FAs pro Zelle berechnet werden, indem die aus fAs erhaltenen Werte durch die Anzahl der Kerne im gesamten Bild dividiert werden. - Bewerten Sie die statistische Signifikanz potenzieller Unterschiede zwischen Populationen (experimentelle Gruppen). Je nach Verteilung und Varianz der Daten verwenden Sie für den Vergleich der beiden verschiedenen Gruppen Student t-test (Normalverteilung) oder nicht-parametrischen U-Test (Mann-Whitney). Zum Vergleich mehrerer Gruppen verwenden Sie einwegs ANOVA oder Kruskal-Wallis in Verbindung mit den entsprechenden Post-hoc-Tests.
- Berechnen Sie die durchschnittliche FAs-Zahl und die durchschnittliche FA-Größe pro Zelle/Kerne.
- Zellformanalyse
- Berechnung von Formdeskriptoren
- Manuelle Analyse: Berechnen Sie den Zellformindex (CSI, auch Zirkularität oder Zellform genannt) in der Kalkulationstabelle der Wahl für jede gemessene Zelle aus der entsprechenden Fläche und dem entsprechenden Umfang mit der Formel:

HINWEIS: CSI geht von Werten zwischen 1 (kreisförmig) und 0 (langgestreckt) aus. Die Beispiele verschiedener Zellformen (mit demselben Bereich) und deren jeweiligen CSIs sind in Abbildung 2dargestellt. In der automatisierten Analyse werden die Werte von Formdeskriptoren (eingetragen und unten definiert) automatisch berechnet und erscheinen im Ergebnisfeld:
(1) CSI = 4x*Fläche/(Perimeter)2
(2) AR = Hauptachse/Nebenachse
(3) Rundheit = 4*Fläche/-*(Hauptachse)2
(4) Festigkeit = Fläche/Konvexfläche
- Manuelle Analyse: Berechnen Sie den Zellformindex (CSI, auch Zirkularität oder Zellform genannt) in der Kalkulationstabelle der Wahl für jede gemessene Zelle aus der entsprechenden Fläche und dem entsprechenden Umfang mit der Formel:
- Histogramme der Zellformverteilung
- Plotzellenformverteilung als Histogramm der Zirkularität (CSI). Klassifizieren Sie Zellen nach ihrem CSI-Wert (berechnet für mindestens 200–400 Zellen) in einen der zehn gleichmäßigen Intervalle (Bereich: 0-1, Lagerplatzbreite: 0,1). Das Histogramm zeigt die Anzahl der Zellen in jeder Kategorie an.
HINWEIS: Das Histogramm, das die Formverteilung der typischen MCF7-Zellschicht anzeigt, zeigt einen Spitzenwert von etwa 0,7-0,8 CSI. Wenn die Form der Zellen durch einen Faktor verzerrt wird (z. B. Paclitaxel-Behandlung, die einen G2/M-Phasenstillstand verursacht und in der Folge mehr Zellen rund sind), sollte sie auf dem Histogramm reflektiert werden.
- Plotzellenformverteilung als Histogramm der Zirkularität (CSI). Klassifizieren Sie Zellen nach ihrem CSI-Wert (berechnet für mindestens 200–400 Zellen) in einen der zehn gleichmäßigen Intervalle (Bereich: 0-1, Lagerplatzbreite: 0,1). Das Histogramm zeigt die Anzahl der Zellen in jeder Kategorie an.
- Kumulierte Verteilungsdiagramme
- Vergleichen Sie die kumulativen CSI-Verteilungen für jede Zelllinie, da dies die beste Möglichkeit ist, statistisch wichtige Unterschiede in Zellformänderungen (oder anderen Änderungen in der Verteilung) zu bewerten. Sie kann z. B. angewendet werden, um die sich ändernde Verteilung von FAs nachzuverfolgen.
- Berechnen Sie die kumulative Verteilungsfunktion (CDF), um Verteilungen zu vergleichen. CDF weist für einen bestimmten CSI-Wert (auf der X-Achse dargestellt) den Prozentsatz (oder die relative Anzahl) zu, für den alle Werte kleiner oder gleich diesem CSI-Wert (auf der Y-Achse geplottet) sind. Wenn also der CSI-Wert höher wird, wird auch der Prozentsatz der Werte, die kleiner oder gleich diesem Wert sind, höher. CDF kann durch die statistische Software der Wahl oder manuell berechnet werden.
- Für die statistische Analyse verwenden Sie Kolgomorov-Smirnov nichtparametrischen Test.
- Berechnung von Formdeskriptoren
Ergebnisse
Fokale Haftungsanalyse
Der Knockdown des HAX1-Gens hatte zuvor gezeigt, dass er fokale Adhäsionen beeinflusst6. Die Zellen wurden auf kollageni-beschichteter Oberfläche für 48 h kultiviert. Bilder der MCF7-Kontrollzellen und MCF7-Zellen mit einem HAX1-Knockdown (HAX1 KD) aus drei unabhängigen Experimenten, die mit fokalem Adhäsionsprotein Paxillin gefärbt waren, wurden mit einem konfokalen Mikroskop (Bild von einer einzigen Fokalebene/Z-Scheibe aus dem Basalbereich) gewonnen. FAs aus etwa 2.000-2.500 Zellen aus jeder Zelllinie wurden mit dem beschriebenen Protokoll quantifiziert. Der Mittelwert für die kleinste Brennpunkthaftung wurde auf 50 (Pixel2)gesetzt. Repräsentative Bilder von FAs zählen mit ImageJ, einschließlich der endgültigen, nummerierten Umrisse und der Überlagerung der FAs-Umrisse mit dem Originalbild, werden für beide Zelllinien in Abbildung 3Aangezeigt. Die Unterschiede in Anzahl und Größe der FAs in beiden Zelllinien sind in Abbildung 3Bdargestellt.
Zellformanalyse
Manuelle Bewertung: MCF7-Zellen wurden für 24 h kultiviert, das Medium gegen das gleiche frische Medium (unbehandelt) oder das Medium mit 0,1 M Paclitaxel (PTX) ausgetauscht – um Zellrundung zu induzieren – und für weitere 24 h kultiviert. Bilder der konfluenten MCF7-Monolayer, die mit Anti-Plakoglobin-Antikörpern und DAPI gefärbt waren, wurden mit einem konfokalen (Mikroskop- Etwa 200-400 Zellen (2-3 Sichtfelder) aus jedem Experiment (unbehandelt/behandelt) wurden dokumentiert (40x objektiv) und die Bilder wurden mit der ImageJ Bildverarbeitungssoftware bewertet (Abbildung 4A). Repräsentative Bereiche jeder Zellschicht mit umrissenen Zellen sind in Abbildung 4A dargestellt (unbehandelte und PTX-behandelte Zellen). Alle Zellen wurden nach ihren CSI-Werten (10 Intervalle, Bin-Breite 0,1) kategorisiert und in den jeweiligen Histogrammen (Abbildung 4B) dargestellt, die eine Zunahme des letzten Behälters (0,9-1) und die Abflachung der Hauptspitzen (0,6-0,9) für die PTX-behandelten Zellen zeigen, im Vergleich zur unbehandelten Steuerung. Unterschiede in der kumulativen Verteilung von CSI wurden für die statistische Signifikanz mit Dem Kolgomorov-Smirnov-Test (K-S) berechnet, einem nichtparametrischen Test für die Gleichheit eindimensionaler Wahrscheinlichkeitsverteilungen. Frequenzverteilungshistogramme und kumulative Verteilungsdiagramme (Abbildung 4C) wurden mit Software generiert.
Automatisierte Bewertung: MCF7-Zellen wurden für 24 h kultiviert, fixiert und mit fluorophorkonjugiertem Phalloidin gefärbt, um Actin zu visualisieren. Die Bilder wurden mit einem konfokalen Mikroskop (einzelne Z-Scheibe aus dem apikalen Bereich) aufgenommen. Die Graustufenbilder der Monolayer wurden mit der angehängten Makrodatei analysiert, entsprechend dem mitgelieferten Protokoll (Abbildung 5A-C). Insgesamt wurden 512 Zellen aus 12 Sichtfeldern quantifiziert. Die Ergebnisse wurden als Histogramm dargestellt, das die Verteilung der Zirkularität darstellt (Abbildung 5D).
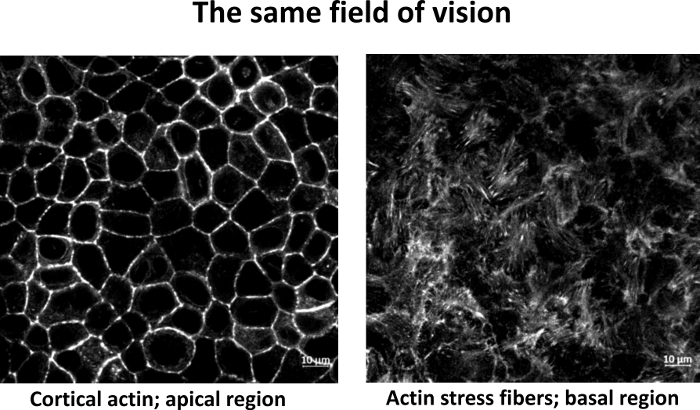
Abbildung 1: Actin-Färbung mit fluorophorkonjugiertem Phalloidin, zwei verschiedene Z-Scheiben aus demselben Sichtfeld. (A) Der apikale Bereich mit kortikdem Aktin. (B) Der Basalbereich mit Actin-Spannungsfasern. Bar: 10 m. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Beispiele für verschiedene CSI-Werte (Cell Shape Index) für die Shapes mit unterschiedlichen Perimetern, aber demselben Bereich. Die sehr längige Form auf der linken Seite hat CSI nahe 0, während der ideale Kreis auf der rechten Seite CSI von 1 hat. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
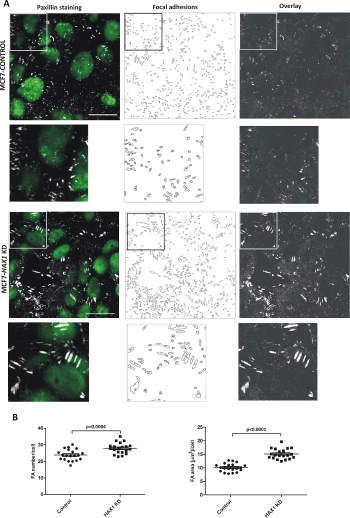
Abbildung 3: Fokale Adhäsionsquantifizierung. (A) Repräsentative Bilder von MCF7-basierten Zelllinien: CONTROL und HAX1 KD mit Paxillin gefärbt; von links nach rechts: (1) Paxillin und Kerne (2) umrissene und nummerierte FAs (3) Überlagerung von FAs-Umrissen und dem Originalbild. Bar: 20 m. Eintsets (unter jedem Bild) zeigen vergrößerte Feldbereiche an. (B) Diagramme, die den Unterschied in fAs Größe und Zahl zwischen den beiden Zelllinien anzeigen. Die statistische Signifikanz wurde anhand des Student t-Tests bewertet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
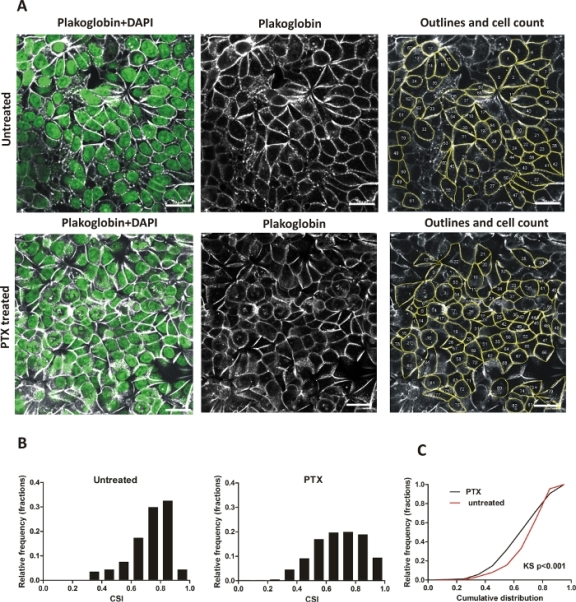
Abbildung 4: Veränderungen der Zellform, die durch Paclitaxel (PTX) in der MCF7-Zellschicht induziert werden; manuelle Anzahl. (A) Repräsentative Regionen von MCF7-Monolayern, zellen unbehandelt und mit PTX behandelt, gefärbt mit Junction-Protein Plakoglobin und DAPI, bar: 20 m. Das Panel auf der rechten Seite zeigt das in ImageJ verarbeitete Bild; Zellenkanten werden umrissen, und alle gezählten Zellen werden nummeriert. (B) Histogramme mit CSI-Verteilung in unbehandelten und PTX-behandelten Zellen, 200-400 Zellen in jedem Experiment (unbehandelt/behandelt), Behälterbreite: 0,1. Plots wurden mittels Frequenzverteilungsanalyse mit relativen Frequenzen als Bruchteil erzeugt. (C) Das Diagramm der kumulativen Verteilung für die unbehandelten und PTX-behandelten Monolayer. Statistische Differenz berechnet mit Kolgomorov-Smirnov nonparametrischen Test (KS). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
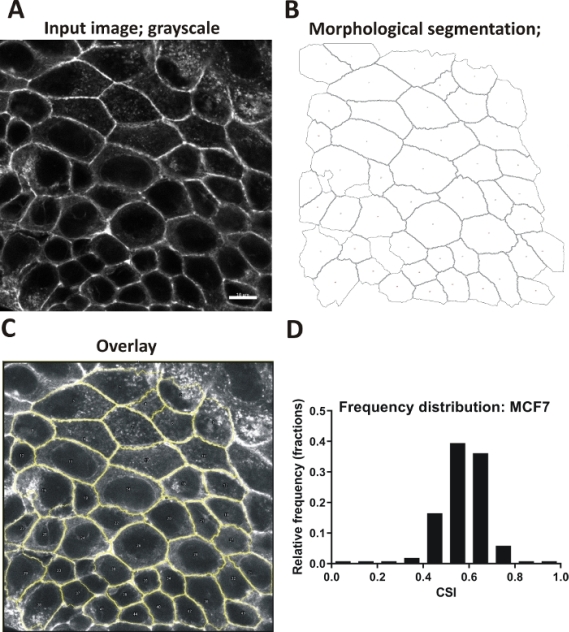
Abbildung 5: Zellenform in MCF7-Monolayern, die nach der automatisierten Methode bewertet werden. (A-C) Ein Beispiel für die automatisierte Bildanalyse, die nachfolgende Schritte veranschaulicht, die vom angehängten Makro ausgeführt werden. (A) Eingabebild; kortikaler Aktin; Graustufen, Maßstabsleiste 10 m (zu informationsinformativen Zwecken; Skalenbalken sollten nicht in analysierte Bilder eingebettet werden). (B) Zellschicht nach Segmentierung; Zellrahmen umrissen; Zellen ohne vollständige Grenzen eliminiert. (C) Überlagerung der Zellumrisse auf dem Originalbild. (D) Ein Histogramm, das die Verteilung von CSI im analysierten Datensatz zeigt, 512 Zellen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Figur S1, S2: MCF7-Zellen, Paxillin und DAPI-gefärbt, um FAs und Kerne zu visualisieren. Als Trainingsdatensatz für das FAs.ijm-Makro bereitgestellt.
Abbildung S1: Bitte klicken Sie hier, um diese Abbildung herunterzuladen.
Abbildung S2: Bitte klicken Sie hier, um diese Abbildung herunterzuladen.
Ergänzende Figur S3, S4: MCF7-Zellen, Plakoglobin und DAPI-gefärbt, um Zell-Zell-Verbindungen und Kerne zu visualisieren. Als Trainings-Dataset für das CSI.ijm-Makro bereitgestellt.
Abbildung S3: Bitte klicken Sie hier, um diese Abbildung herunterzuladen.
Abbildung S4: Bitte klicken Sie hier, um diese Abbildung herunterzuladen.
FAs.txt: Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Die Zellzell- und Zellsubstratadhäsion sind inhärente Attribute der Epithelzellen und spielen die entscheidende Rolle bei der Gewebemorphogenese und Embriogenese. In erwachsenen Geweben ist die richtige Regulierung der mechanischen Eigenschaften der Zellschicht entscheidend für die Aufrechterhaltung der Homöostase und die Verhinderung pathologischer Reaktionen wie Tumorprogression und Metastasierung. Die Größe und Anzahl der Fokaladesionen hängt von der Stärke der Zellsubstrathaftung ab, während die Zellform von kontraktilen Kräften abhängt und mit dem Status von Zellzellenkontakten zusammenhängt.
Hier beschreiben wir zwei einfache Methoden der quantitativen Analyse der Fläche, Anzahl und Form von zellulären Strukturen, die durch Immunfluoreszenz gefärbt sind, in diesem Fall fokale Adhäsionen und die ganzen Zellen in der Zellschicht. Die vorgeschlagenen Instrumente können jedoch für die Quantifizierung jeder ausgewählten Struktur umgewidmet werden. Das zentrale Thema für diese Analysen ist die Qualität der immunfluoreszierenden Färbung und konfokalen Bildgebung. Diese Methoden können in jedem Standardlabor eingesetzt werden, das mit einer Zellkultureinheit und einem konfokalen Mikroskop ausgestattet ist. Sie sind so konzipiert, dass sie Zelllinien vergleichen, insbesondere wenn die Unterschiede (natürlich oder durch spezifische Behandlung induziert) in den gemessenen Parametern erheblich sind. Es wird nicht empfohlen, Minutenunterschiede zu messen oder absolute Messungen festzulegen, da sie auf minimale Änderungen in den anfänglichen willkürlichen Einstellungen empfindlich sind, insbesondere bei Fokaladesionen. Diese Methode der FA-Quantifizierung ist schlechter als fortgeschrittene rebevorgefeilte und spezifischere Methoden wie die TIRF-Mikroskopie, hat aber den Vorteil, dass keine ausgeklügelte Ausrüstung erforderlich ist.
Ähnliche Methoden der fokalen Haftungsmessungen wurden vor7,8,9,10beschrieben. Hier haben wir die Einstellungen angegeben und ein kostenloses ImageJ-Makro mit mehreren Optionen erstellt, um die Messungen zu erleichtern.
Die Zellformanalyse wurde mehrfach beschrieben, einschließlich sehr komplexer und detaillierter Methoden11,12. Hier stellen wir eine einfache Methode vor, um die Veränderungen in Epithelzellmonolayern nachzuverfolgen, was für den Vergleich von Zellmorphologie oder Entwicklungsveränderungen sehr wichtig sein könnte. Die manuelle Methode der Zellformanalyse in den in diesem Protokoll vorgestellten Monolayern wurde in einem früheren Bericht6beschrieben. Die CSI-Formel als eine Möglichkeit, die Form eines Objekts zu beschreiben und zu vergleichen, wird in verschiedenen Disziplinen13,14häufig verwendet, einschließlich der Geologie, aus der es stammt. Die Darstellung der Ergebnisse in Form eines Histogramms und/oder als kumulative Verteilungsfunktion wird häufig für Vergleiche von Verteilungen jeder Art8,10,15verwendet.
Insbesondere präsentieren wir hier ein Tool für die automatisierte Zellformanalyse basierend auf dem ImageJ Plugin MorhpLibJ (https://imagej.net/MorphoLibJ). Wir stellen eine Makrodatei bereit, die diese Analyse schnell und effizient durchführen kann. Diese Methode erkennt nicht immer die richtigen Zellränder, aber der Prozentsatz der fehlerhaften Messungen (die in jeder automatisierten Analyse vorhanden sind) ist minimal und sollte das Endergebnis nicht signifikant beeinflussen, insbesondere wenn genügend Zellen analysiert werden. Zellen ohne vollständige Grenzen werden eliminiert. Automatisierte Zellformanalyse hat unbestreitbare Vorteile und wir präsentieren diese Methode, so dass sie von der wissenschaftlichen Gemeinschaft geschätzt werden kann.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Diese Arbeit wurde vom Polnischen National Science Center unter der Fördernummer 2014/14/M/NZ1/00437 unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Alexa Fluor 594 | ThermoFisher Scientific | A32740 | goat anti-rabbit, 1:500 |
| Ammonium chloride | Sigma | A9434 | |
| BSA | BioShop | ALB001.500 | |
| Collagen from calf skin | Sigma | C9791-10MG | |
| DAPI | Sigma | D9542 | 1:10000 (stock 1 mg/mL in H2O), nucleic acid staining |
| DMEM + GlutaMAX, 1 g/L D-Glucose, Pyruvate | ThermoFisher Scientific | 21885-025 | |
| FBS | ThermoFisher Scientific | 10270-136 | |
| Junction plakoglobin | Cell Signaling | 2309S | rabbit, 1:400 |
| Laminar-flow cabinet class 2 | Alpina | standard equipment | |
| MCF7-basedHAX1KD cell line | Cell line established in the National Institute of Oncology, Warsaw, described in Balcerak et al., 2019 | MCF7 cell line withHAX1knockdown | |
| MCF7 cell line (CONTROL) | ATCC | ATCC HTB-22 | epithelial, adherent breast cancer cell line |
| Olympus CK2 light microscope | Olympus | ||
| Paxillin | Abcam | ab32084 | rabbit, 1:250, Y113 |
| PBS | ThermoFisher Scientific | 10010023 | |
| Phalloidin-TRITC conjugate | Sigma | P1951 | 1:400 (stock 5 mg/mL in DMSO), actin labeling |
| PTX | Sigma | T7402-1MG | |
| TBST – NaCl | Sigma | S9888 | |
| TBST – Trizma base | Sigma | T1503 | |
| Triton X-100 | Sigma | 9002-93-11 | |
| Zeiss LSM800 Confocal microscope | Zeiss |
Referenzen
- Li, D. S., Zimmermann, J., Levine, H. Modeling closure of circular wounds through coordinated collective motion. Search Results. 13 (1), 016006 (2016).
- Ilina, O., Friedl, P. Mechanisms of collective cell migration at a glance. Journal of Cell Science. 122, 3203-3208 (2009).
- Ladoux, B., Mege, R. M. Mechanobiology of collective cell behaviours. Nature Reviews Molecular Cell Biology. 18 (12), 743-757 (2017).
- Stossi, F., et al. High throughput microscopy identifies bisphenol AP, a bisphenol A analog, as a novel AR down-regulator. Oncotarget. 7 (13), 16962-16974 (2016).
- Legland, D., Arganda-Carreras, I., Andrey, P. MorphoLibJ: integrated library and plugins for mathematical morphology with ImageJ. Bioinformatics. 32 (22), 3532-3534 (2016).
- Balcerak, A., et al. HAX1 impact on collective cell migration, cell adhesion, and cell shape is linked to the regulation of actomyosin contractility. Molecular Biology of the Cell. 30 (25), 3024-3036 (2019).
- Buskermolen, A. B. C., Kurniawan, N. A., Bouten, C. V. C. An automated quantitative analysis of cell, nucleus and focal adhesion morphology. PLoS One. 13 (3), 0195201 (2018).
- Fokkelman, M., et al. Cellular adhesome screen identifies critical modulators of focal adhesion dynamics, cellular traction forces and cell migration behaviour. Scientific Reports. 6, 31707 (2016).
- Horzum, U., Ozdil, B., Pesen-Okvur, D. Step-by-step quantitative analysis of focal adhesions. MethodsX. 1, 56-59 (2014).
- Kim, D. H., Wirtz, D. Focal adhesion size uniquely predicts cell migration. FASEB Journal. 27 (4), 1351-1361 (2013).
- Pincus, Z., Theriot, J. A. Comparison of quantitative methods for cell-shape analysis. Journal of Microscopy. 227, 140-156 (2007).
- Tsygankov, D., et al. CellGeo: a computational platform for the analysis of shape changes in cells with complex geometries. Journal of Cell Biology. 204 (3), 443-460 (2014).
- Tiryaki, V. M., Adia-Nimuwa, U., Ayres, V. M., Ahmed, I., Shreiber, D. I. Texture-based segmentation and a new cell shape index for quantitative analysis of cell spreading in AFM images. Cytometry A. 87 (12), 1090-1100 (2015).
- Tong, J., et al. Cell micropatterning reveals the modulatory effect of cell shape on proliferation through intracellular calcium transients. Biochimica et Biophysica Acta. 1864 (12), 2389-2401 (2017).
- Vartanian, K. B., Kirkpatrick, S. J., Hanson, S. R., Hinds, M. T. Endothelial cell cytoskeletal alignment independent of fluid shear stress on micropatterned surfaces. Biochemical and Biophysical Research Communications. 371 (4), 787-792 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten