Method Article
Exon Springt in direkt umprogrammierten Myotubes aus menschlichen Urin-abgeleiteten Zellen
In diesem Artikel
Zusammenfassung
In diesem Artikel beschreiben wir ein detailliertes Protokoll zur effizienten Modellierung des Duchenne-Muskeldystrophie-Muskels mit MYOD1-konvertiertenUrin-abgeleiteten Zellen, um die Wiederherstellung von Dystrophin mRNA und Proteinspiegel nach Exon-Skipping zu bewerten.
Zusammenfassung
Duchenne-Muskeldystrophie (DMD), eine progressive und tödliche Muskelerkrankung, wird durch Mutationen im DMD-Gen verursacht, die zum Fehlen von Dystrophin-Protein führen. Bis heute haben wir eine vom Forscher initiierte Erste-in-Human-Studie am National Center of Neurology and Psychiatry abgeschlossen, die auf der systemischen Injektion der Morpholino-Oligonukleotide basiert, die anfällig für Exon-53-Skipping sind. Für die wirksame Behandlung von DMD wird davon ausgegangen, dass In-vitro-Tests mit Myoblasten von DMD-Patienten zur Untersuchung von Medikamenten und zur Beurteilung der Patientenberechtigung vor der Durchführung klinischer Studien als wesentlich angesehen werden. Vor kurzem berichteten wir über eine neue MYOD1-konvertierteUrin-abgeleitete Zelle (UDC), die mit dem Histon-Methyltransferase-Inhibitor (3-Deazaneplanocin-A-Hydrochlorid) als Zellmodell von DMD behandelt wurde. Die neue autologe UDC könnte eine Phänokopie der krankheitsspezifischen Phänotypen von DMD zeigen, was zur Anwendung von Präzisionsmedizin bei einer Vielzahl von muskelbedingten Erkrankungen führt. In diesem Artikel beschreiben wir ein detailliertes Protokoll zur effizienten Modellierung von DMD-Muskelzellen mit MYOD1-konvertiertenUDCs zusammen mit Reverse-Transkriptase-Polymerase-Kettenreaktion (RT-PCR), Western Blotting und Immunzytochemie zur Bewertung der Wiederherstellung von Dystrophin-mRNA und Proteinspiegeln nach Exon-Skipping.
Einleitung
Duchenne-Muskeldystrophie (DMD), eine progressive, tödliche Muskelerkrankung, wird durch Frame-Shift-Mutationen im DMD-Gen verursacht, die zum Fehlen von Dystrophinprotein1führen. Antisense Oligonukleotid-basierte Exon-Skipping-Therapie wird gedacht, um vielversprechend für DMD sein. Diese Therapie basiert auf der Umwandlung des schwereren DMD-Phänotyps in den milderen Becker-Muskeldystrophie-ähnlichen Phänotyp durch Veränderung der Pre-mRNA-Spleißung, um den DMD-Leserahmen 2wiederherzustellen. Wir haben vor kurzem eine erste studien abgeschlossen, die auf der wiederholten intravenösen Verabreichung des Phosphorodiamid-Morpholino-Oligomers (PMO)-Viltolarsen basiert, das Exon 53-Springen in DMD induzieren kann, und ein ausgezeichnetes Sicherheitsprofil, vielversprechende Wirksamkeit und akzeptable pharmakokinetische Parameter (registriert als UMIN: 000010964 und ClinicalTrials.gov: NCT02081625)3.
Um jedoch kosteneffiziente und effiziente Behandlungen für die Krankheit zu entwickeln, sind In-vitro-Tests mit Primärmuskelzellen, die von DMD-Patienten gewonnen wurden, für das Arzneimittelscreening und die Überprüfung der Patientenberechtigung vor der Durchführung klinischer Studien sowie Für Biomarker, die die Wirksamkeit von Exon-Skipping-Therapien während der Studien am Menschenwiderspiegeln,unerlässlich 4 . Erst vor kurzem berichteten wir über eine neuartige Technologie zur Entwicklung patientenspezifischer MYOD1-konvertierterUrinzellen (UDCs)5,6 als primäres Myoblastenmodell von DMD7. So ist zur Erzeugung der Myoblasten nur die Entnahme von Urin von Patienten erforderlich und es ist kein invasives Verfahren erforderlich. In diesem Artikel beschreiben wir ein detailliertes Protokoll zur effizienten Modellierung von DMD-Muskeln mit MYOD1-konvertiertenUDCs, die mit 3-Deazaneplanocin-A-Hydrochlorid behandelt wurden, um die wiederhergestellte Dystrophin-mRNA und das Protein nach dem Exon-Springen zu bewerten.
Protokoll
Die Ethikkommission des National Center of Neurology and Psychiatry genehmigte diese Studie (Zulassungs-ID: A2017-018, A2018-029). Alle Personen gaben informierte Zustimmung, bevor sie Urin zur Verfügung gestellt. Alle Experimente wurden nach den einschlägigen Richtlinien und Vorschriften durchgeführt.
1. Isolation und Primärkultur von UDCs
HINWEIS: UDCs wurden gemäß einem zuvor veröffentlichten Protokoll8,9,10 mit einigen Änderungen isoliert.
- Sammeln Sie Urinproben während der spontanen Micturition in sterilisierten Plastikflaschen.
HINWEIS: Eine Sterilisation der äußeren Harnröhrenöffnung ist nicht erforderlich. Midstream-Urin ist wünschenswert, um das Risiko einer Viruskontamination zu reduzieren. Wenn die Lagerzeit vor dem nächsten Eingriff >1 h beträgt, sollten Urinproben auf 4 °C übertragen werden, um die Zelllebensfähigkeit zu erhalten. Eine Temperatur von <4 °C sollte jedoch vermieden werden, da unlösliche Ausscheidungen auftreten können. - Zentrifugieren Sie die gesamte Urinprobe bei 400 x g für 10 min bei Raumtemperatur.
- Aspirieren Sie den Überstand und lassen Sie 1 ml in der Röhre.
- Setzen Sie die Pellets einzeln in den restlichen 1 ml Urin aus und sammeln Sie diese dann in einem einzigen 50 ml-Rohr.
- Fügen Sie 10 ml Waschpuffer, bestehend aus 99 ml PBS ohne Kalzium und Magnesium, 1% Penicillin/Streptomycin (P/S), 0,5 g/ml Amphotericin B, und Zentrifuge die Proben bei 200 x g für 10 min bei Raumtemperatur.
- Aspirieren Sie den Überstand und lassen Sie 0,2 ml in der Röhre.
- Die Zellpellets in 4,5 ml Primärmedium, bestehend aus einem 1:1-Gemisch aus dem modifizierten Eagle-Medium (DMEM) ohne Natriumpyruvat und Hams F-12-Nährstoffmix, ergänzt durch rekombinanten humanen epidermalen Wachstumsfaktor (EGF), Insulin, Hydrocortison, Adrenalin, T3, Transferrin, 10% Tetracyclinfreies fetales Rinderserum (FBS), 1% P/S und 0,5 g/ml Amphotericin B.
- Säen Sie die Zellen in drei Brunnen mit Gelatine beschichtet sechs Brunnenplatten (Gesamtvolumen jedes Brunnens, 1,5 ml). Kultur befeuchtet bei 37 °C und 5%CO2 für 24 h.
- Fügen Sie 1,5 ml des primären Mediums täglich für die nächsten 3 Tage hinzu.
- Ersetzen Sie am 4. Tag das Medium durch 1,5 ml Wachstumsmedium, ergänzt durch rekombinanten menschlichen EGF, Insulin, Hydrocortison, Epinephrin, T3, Transferrin, 15% Tetracyclin-freies FBS, 0,5% L-Alanin-L-Glutamin, 0,5% nicht essentielle Aminosäuren und 2,5 ng/mL Fibroblasten-Wachstumsfaktor-Basic (bFGF), rekombinanter humaner Thrombozyten-abgeleiteter Wachstumsfaktor (PDGF), EGF und 1% P/S.
- Ändern Sie das Wachstumsmedium jeden zweiten Tag.
HINWEIS: UDC-Kolonien erscheinen innerhalb einer Woche. - Wenn die UDC-Kultur zu 80 bis 90 % konfluent wird, entfernen Sie das Medium und die Waschzellen mit PBS, teilen Sie alle Zellen mit 0,25% Trypsin-EDTA und Samen bei 3.000-5.000 Zellen/cm2 auf eine neue gelatinebeschichtete 60-mm-Schale (Durchgang 1).
HINWEIS: Die UDCs können in flüssigem Stickstoff gespeichert werden. UDCs werden in der Regel bei 60 bis 70 % Konfluenz in 60 mm Kulturschale in drei Stockrohre unterteilt.
2. Retrovirales Konstrukt
- Verstärken Sie den Kodierungsbereich von MYOD1 (NM_002478.4) Plasmid durch Polymerase-Kettenreaktion (PCR).
ANMERKUNG: Die Mischung für die MYOD1-Verstärkung und die Bedingungen für den thermischen Cycler sind in Tabelle 1 bzw. Tabelle 2dargestellt. - Erkennen Sie ein einzelnes Band von etwa 1.000 bp Größe durch 0,7% Agarose-Gel-Elektrophorese mit 1 l des verstärkten PCR-Produkts, um zu bestätigen, dass die MYOD1-Sequenz erfolgreich verstärkt wurde.
- Reinigen Sie das PCR-Produkt mit dem Clean-up-Kit und bestimmen Sie seine Konzentration mit einem Spektralphotometer.
- Inkubieren Sie die Mischung, wie in Tabelle 3 gezeigt, bei 37 °C über Nacht, um retroviralen Vektor mit einem Tet-on-System und einem Puromycin-resistenten Gen in restriktionsenzym-gezielten Regionen am Mehrfachklonstandort zu verdauen.
- Detektieren Sie ein einzelnes Band um 0,7% Agarose-Gel-Elektrophorese mit 1 l des verdauten Produkts, um zu bestätigen, dass der retrovirale Vektor erfolgreich verdaut wurde.
- Reinigen Sie das verdaute Produkt mit einem Reinigungsset und bestimmen Sie seine Konzentration per Spektralphotometer.
- Um das verstärkte MYOD1-Fragment (produziert durch die Schritte 2.1-2.3) in den verdauten retroviralen Vektor (produziert durch die Schritte 2.4-2.6) zu klonen, führen Sie eine In-Fusion-Klonreaktion durch. Richten Sie die Reaktion wie in Tabelle 4dargestellt ein, inkubieren Sie die Reaktion 15 min bei 50 °C, und legen Sie sie dann auf Eis.
- Durchführung der Transformation mit E. coli kompetenten Zellen gemäß den Anweisungen des Herstellers (Materialtabelle).
- Wählen Sie die transformierten kompetenten Zellen aus, indem Sie auf einer LB-Kulturplatte kultivieren, die aus 10 g/L Bacto Trypton, 5 g/L Bacto-Hefe-Extrakt, 5 g/L NaCl, 15 g/L-Bacto-Agar und 50 mg/L Ampicillin besteht.
- Die ausgewählte Kolonie und Kultur im LB-Kulturmedium ohne Bacto-Agar bei 200 Umdrehungen pro Minute bei 37 °C über Nacht abholen.
- Reinigen Sie die MYOD1-inserted retroviralen Vektoren mit einem Plasmid-Reinigungskit (Tabelle der Materialien) und quantifizieren Sie mit einem Spektralphotometer.
- Vergewissern Sie sich, dass MYOD1 korrekt in den retroviralen Vektor eingeführt wird, indem das PCR-Produkt direkt sequenziert wird, das durch die auf beiden Seiten der eingefügten MYOD1-Sequenz ausgerichteten Vorwärts- und Rückwärtsprimer verstärkt wird.
HINWEIS: Die MYOD1-Sequenz kann zwischen retroviralen Vektorsequenzen nachgewiesen werden, wenn das Klonen in der Fusion erfolgreich ist. - Für die retrovirale Produktion die Verpackungszellen bei 50.000 Zellen/cm2 auf 10 cm kollagenbeschichteten Platten und Kultur in DMEM mit 10% FBS bei 37 °C und 5%CO2 für 24 h befeuchtet säen.
- Wenn sich die Verpackungszellen zu 80 % Koninfluenza vermehren, mischen Sie 30 g MYOD1-insertedretrovirale Vektoren, 30 g Verpackungsvektoren und Transfektionsreagenz, das zelldurchdringendes Peptid (Materialtabelle) durch Wirbeln enthält, und inkubieren Sie sie für 10 min.
- Fügen Sie die inkubierte Mischung in das Medium der Verpackungszellen und Kultur bei 37 °C und 5%CO2befeuchtet.
HINWEIS: Kollagenbeschichtung ist notwendig. Die Transfektion kann nach der Aussaat bei 24 h oder mehr besser sein, da sich Verpackungszellen leicht von der Kulturplatte lösen. - Nach 4 h oder über Nacht, ändern Sie das Medium zu frischem Wachstumsmedium.
- Sammeln Sie den viralen Überstand und ersetzen Sie mit frischem Medium bei 24 und 48 h nach Derkotransfetion und kombinieren Sie den Überstand.
- Um den viralen Überstand zu konzentrieren, mit dem Konzentratorreagenz mischen und bei 37 °C über Nacht inkubieren, dann bei 1.500 x g für 45 min bei 4 °C zentrieren.
- Filtern Sie den Retrovirus-Überstand durch einen PVDF-Filter mit 0,45 m Poren.
- Überprüfen Sie den Titer des retroviralen Vektors mit einem quantitativen PCR-Kit und einem thermischen Cycler-System gemäß den Anweisungen des Herstellers.
- Teilen Sie den viralen Überstand in kleine Aliquots auf und lagern Sie ihn bei -80 °C.
3. Infektion mit MYOD1-retroviralen Vektor in UDCs
- Säen Sie die UDCs bei 3.000-5.000Zellen/cm2 auf einer gelatinebeschichteten 60 mm Schale.
- Nach 24 h Aussaat das aufgetaute Retrovirus (Schritt 2.21) bei einer Vielzahl von Infektionen von 200 zu infizieren, indem man Hexmethrinbromid in einer Konzentration von 8 g/ml hinzufügt.
- Nach einer 24 h Inkubation, die bei 37 °C und 5%CO2befeuchtet wird, ersetzen Sie das Kulturmedium durch ein frisches Wachstumsmedium, das 1 g/ml Puromycin enthält, um die MYOD1-transducedZellen auszuwählen. Ändern Sie das Medium jeden zweiten Tag.
HINWEIS: Bei der Auswahl für MYOD1-positiveZellen wird in der Regel 1 g/ml Puromycin zur Auswahl verwendet. Die entsprechende Dosis sollte bestimmt werden. Verwenden Sie eine Platte, die untransfizierte Zellen enthält, und wählen Sie die Dosis, die alle Zellen in 3-5 Tagen abtötet. MYOD1-positive Zellen sollten innerhalb von 7 bis 10 Tagen nach Zugabe von Puromycin ausgewählt werden. Die MYOD1-transduced UDCs können in flüssigem Stickstoff gespeichert werden.
4. Myogene Differenzierung von MYOD1-transducedUDCs, die mit 3-Deazaneplanocin A Hydrochlorid (DZNep) behandelt wurden
HINWEIS: Kürzlich wurde berichtet, dass DZNep, ein Histon-Methyltransferase-Inhibitor, die Expression von MYOGENIN, einem der späten Muskelregulierungsfaktoren, signifikant fördern und auch zu einer Myotube-Differenzierung führen könnte7.
- Platte MYOD1-transduced UDCs in den kollagenbeschichteten Brunnen mit einer Dichte von 3,5 x 104 Zellen/cm2. Kultur befeuchtet bei 37 °C und 5%CO2.
- Nach 24 h, ändern Sie das Wachstumsmedium zu Differenzierungsmedium bestehend aus hoher Glukose DMEM mit L-Alanin-L-Glutamin, 5% Pferdeserum, ITS-Ergänzung, 1 g/ml Doxycyclin, und 5 m DZNep.
HINWEIS: Die 10 mM DZNep Lösung kann 3 Monate lang bei -80 °C gelagert werden. Verwenden Sie einen Gefrierschrank und vermeiden Sie wiederholte Gefrierzyklen. Sowohl MYOD1 durch Doxycyclin aktiviert und DZNep unterdrücken die Proliferation und fördern die myogene Differenzierung von UDCs. Daher wird empfohlen, Doxycyclin und DZNep nach der Induktion der myogenen Differenzierung hinzuzufügen. DZNep fördert die myogene Differenzierung der MYOD1-UDCsdosisabhängig. Auf der anderen Seite zeigt es Zytotoxizität in einer hohen Konzentration. Bestimmen Sie daher die geeignete Konzentration von DZNep, die je nach Wirkung auf die myogene Differenzierung und zelluläre Bioverfügbarkeit von 1 bis 10 M reicht. - Nach 3 Tagen das Differenzierungsmedium ohne DZNep in frisches Differenzierungsmedium umstellen. Wechseln Sie dann das Medium alle 3 Tage.
HINWEIS: UDCs verschmelzen miteinander und bilden Myotuben innerhalb von 1 bis 2 Wochen nach der Differenzierung.
5. Exon-Springen in MYOD1-konvertiertenUDCs
HINWEIS: Hier werden drei Protokolle beschrieben, um das Exon-Springen in patientenabgeleiteten Zellen zu bewerten: 1) Reverse-Transkriptase-Polymerase-Kettenreaktion (RT-PCR) von Dystrophin mRNA; 2) Semiquantifizierung des wiederhergestellten Dystrophin-Proteinsignals durch Western Blot; und 3) Semiquantifizierung des wiederhergestellten Dystrophinfluoreszenzsignals durch Immunzytochemie. Alle Methoden können Exon-Springen auf dosisabhängige Weise erkennen.
- Bewertung der Exon-Skipping-Effizienz durch RT-PCR
- Um Antisense-Oligonukleotid (ASO) in MYOD1-konvertierte UDCs zu transfekten, die von DMD-Patienten am Tag 7 nach der Differenzierung erhalten wurden, Mischen Sie ASO, Transfektionsreagenz (Materialtabelle), und Differenzierungsmedium bis zu einer Endkonzentration von 1 bis 10 m. Kultur bei 37 °C und 5%CO2befeuchtet.
- Nach 72 h Inkubation mit ASO das Medium ohne ASO in frisches Differenzierungsmedium ändern.
- Von 3 bis 7 Tage nach der ASO-Transfektion, entfernen Sie Differenzierungsmedium und waschen 1x mit PBS. Fügen Sie den Zelllysepuffer hinzu, lysieren Sie die UDCs und ernten Sie die gesamte RNA mit einem RNA-Extraktionskit.
- Messen Sie die RNA-Konzentration mit einem Spektralphotometer.
- Kombinieren Sie die erforderlichen Reagenzien für eine stufe RT-PCR-Reaktion in PCR-Röhren gemäß Tabelle 5.
- Legen Sie die PCR-Rohre mit dem Gemisch in einen Thermocycler. Führen Sie den Thermocycler gemäß Tabelle 6aus.
- Führen Sie die Mikrochip-Elektrophorese durch und berechnen Sie die Exon-Skipping-Effizienz mit der Molkonzentration wie unten.
Exon-Skipping-Effizienz (%) = übersprungenes Band / (übersprungenes Band + nicht übersprungenes Band) x 100
HINWEIS: Bewahren Sie das PCR-Produkt bei 4 °C bei 4 °C für die kurzfristige Lagerung oder -20 °C für die Langzeitlagerung auf.
- Nachweis von Dystrophin nach Exon-Springen durch Western Blotting
- Transfekte ASO und Kultur MYOD1-UDCs nach den Schritten 5.1.1 und 5.1.2.
- Ändern Sie das Medium alle 3 Tage.
- Nach 2 Wochen der Differenzierung, extrahieren Sie das gesamte Protein aus den kultivierten Zellen mit Radioimmunopräzipitations-Assay (RIPA) Puffer mit Protease-Inhibitoren.
- Die Lysate auf Eis und Zentrifuge bei 14.000 x g 15 min bei 4 °C beschallen.
- Sammeln Sie den Überstand und bestimmen Sie die Proteinkonzentrationen mit einem BCA-Protein-Assay-Kit.
- Fügen Sie 15 g Gesamtprotein in ein 0,5 ml-Rohr und verdünnen Sie, indem Sie den RIPA-Puffer, der Protease-Inhibitoren enthält, zu einem Gesamtvolumen von 10 l hinzufügen. Führen Sie 15 g Gesamtprotein pro Lane aus.
- Fügen Sie Probenpuffer, Reduktionsmittel und entionisiertes Wasser hinzu, wie in Tabelle 7dargestellt. Denaturieren der Zelle bei 70 °C für 10 min.
- Bereiten Sie den Trisacetat-Laufpuffer vor, der 8,95 g/L Tricin, 6,06 g/L Tris-Basis, 1,0 g/L Natriumdodecylsulfat (SDS) enthält.
- Die Probe (20 l) auf Trisacetat 3-8% Gel laden und 75 min Elektrophorese bei 150 V durchführen.
- Bereiten Sie den Blotting-Puffer ohne Methanol vor.
- Die PVDF-Membran 20 s in Methanol und dann im Fleckpuffer bis zum Gebrauch (mindestens 10 min) einweichen. Schneiden Sie die PVDF-Membran mit Minigelen auf eine Größe von 6 x 8 cm und mit Midi-Gelen auf 8 x 12 cm.
- Schneiden Sie die Blotting-Papiere auf die gleiche Größe wie die PVDF-Membran und tränken Sie die im Blotting-Puffer bis zum Gebrauch.
- Nach der Elektrophorese das Gel auf die gleiche Größe wie die PVDF-Membran schneiden und in destilliertem Wasser einweichen.
- Legen Sie die Blotting-Papiere, PVDF-Membran und Gel auf das semidry Transfergerät (Abbildung 1). Transfer bei 4 mA/cm2 für 30 min.
- Spülen Sie die Membran 2x mit destilliertem Wasser.
- Bereiten Sie Antidystrophin (1:500) und Anti-S-Tubulin (1:1,200) Antikörper als primäre Antikörper vor.
- Bereiten Sie HRP-konjugierte Anti-Maus-Antikörper (1:100) als sekundären Antikörper vor.
- Die Membranen mit den primären Antikörpern bebrüten, mit Waschpuffer waschen und dann mit dem sekundären Antikörper mit einem automatisierten Western-Verarbeitungsgerät (Materialtabelle) bei Raumtemperatur inkubieren.
HINWEIS: Anti-A-Tubulin-Antikörper wird in der Regel als Ladekontrolle verwendet. Die gemischten primären Antikörperlösungen, einschließlich 1:500 Anti-Dystrophin und 1:1.200 Anti-A-Tubulin-Antikörper funktionieren gut, wenn die Antikörperreaktion gleichzeitig durchgeführt wird. - Spülen Sie die Membran in destilliertem Wasser.
- Erkennen Sie die Proteine mit einem chemilumineszierenden Detektionsreagenz und einem auf Ladung gekoppelten Geräte(CCD) Kamera-basierten Imager.
- Analysieren Sie die Daten mit entsprechender Software.
- Nachweis von Dystrophin nach Exon-Überspringen durch Immunzytochemie
- Die UDCs direkt in die Myotuben in kollagenbeschichteter 96-Wellplatte nach den Schritten 4.1-4.3 umprogrammieren.
- Transfedien Sie die ASO und Kultur MYOD1-UDCs nach den Schritten 5.1.2 und 5.1.3.
- Nach 2 Wochen Differenzierung die Zellen mit PBS waschen und in 4% Paraformaldehyd für 10 min bei 4 °C fixieren.
- MyOD1-UDCsin 0,1% nichtionischem Reinigungsmittel für 10 min bei Raumtemperatur permeimisieren und diejenigen mit 10% Ziegenserum für 15 min bei 37 °C blockieren.
- Inkubieren Sie die Zellen mit einem primären Antikörper über Nacht bei 4 °C.
- Waschen Sie die Zellen mit PBS und inkubieren Sie diejenigen mit dem sekundären Antikörper für 30 min bei Raumtemperatur.
HINWEIS: Hier wird Maus-Antidystrophin (1:30) als primärer Antikörper, Anti-Maus-IgG als sekundärer Antikörper und Hoechst (1:10.000) für die Kernfärbung verwendet. - Stellen Sie die Platten mit einem Fluoreszenzmikroskop ab und verwenden Sie einen Analysator, um das Fluoreszenzsignal automatisch in jedem Brunnen unter demselben Zustand zu halbquantifizieren.
Ergebnisse
Wir konnten die UDCs einfach und nicht-invasiv sammeln. UDCs bildeten Kolonien innerhalb einer Woche nach Beginn der primären Zellkultur beobachteten wir eine ausgeprägte proliferative Fähigkeit. Die Kultur der UDCs war einfach, und bakterielle oder Pilzkontamination war selten, wenn das Verfahren korrekt durchgeführt wurde.
Abbildung 2 zeigt repräsentative Phasenkontrastbilder der UDC-Kolonie eine Woche nach der Primärkultur (Abbildung 2A) und MYOD1-UDCs eine Woche nach der Differenzierung (Abbildung 2B). Abbildung 3 zeigt den erfolgreichen Nachweis von Exon-Skipping bei UDCs, die von DMD-Patienten durch RT-PCR erhalten wurden. Abbildung 3A zeigt die RT-PCR-Analyse von Dystrophin nach Antisense-Oligonukleotid-Behandlung in DZNep-behandelten MYOD1-UDCs,die von einem 6-jährigen Männchen mit einer Exon-45-54-Deletion im DMD-Gen abgeleitet wurden. Der offene Leserahmen wurde durch exon 44 skipping wiederhergestellt. Am 14. Tag nach der Differenzierung bestätigten wir die induktion des Exonspringens in dosisabhängiger Weise (Abbildung 3B). Die oberen Bänder bezeichnen native Produkte, und die unteren Bänder bezeichnen exon 44-übersprungene Produkte, die den offenen Leserahmen wiederhergestellt haben.
Abbildung 4 zeigt den erfolgreichen Nachweis von Dystrophin nach Exon-Überspringen in den UDCs, die von DMD-Patienten durch Western-Blotting in dosisabhängiger Weise erhalten wurden. Wir haben auch die wiederhergestellte Dystrophin-Expression mittels Immunzytochemie nachgewiesen (Abbildung 5). Wir haben die Intensitäten von Dystrophin mit einem Fluoreszenzmikroskop 1 Woche nach der Antisense-Oligonukleotid(ASO) Transfektion auf einer 96-Well-Platte gemessen (Abbildung 5A). Deutlich höhere Fluoreszenzsignale wurden bei MYOD1-UDCsbeobachtet, die mit ASO behandelt wurden, als bei MYOD1-UDCs,die mit Kontroll-ASO behandelt wurden (Abbildung 5B).
Diese Ergebnisse deuten darauf hin, dass unser neuer Assay Exon-Springen effizient in MYOD1-UDCsbewerten kann, die von DMD-Patienten auf mRNA- und Proteinebene erhalten wurden.
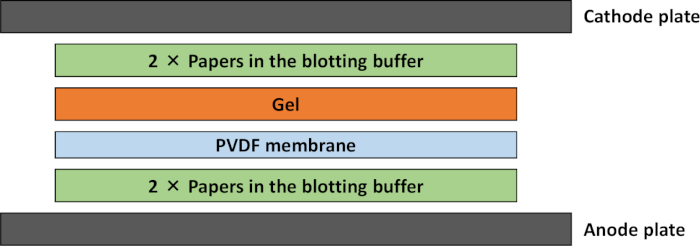
Abbildung 1: Schematische Darstellung des Übertragungsstapels für semidry Western Blot. Zwei Papiere, die in den Blotting-Puffer eingeweicht waren, wurden am negativen Terminal gelegt, und zwei Papiere, die in den Puffer eingeweicht wurden, wurden darüber gestapelt. Das Gel, das im Puffer eingeweicht wurde, wurde sanft über die PVDF-Membran gelegt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Repräsentative Bilder der UDCs. (A) Phasenkontrastbild von UDCs eine Woche nach der Primärkultur. Maßstabsleiste = 200 m. Einschub: Ein vergrößertes Bild des Bereichs im weißen Rechteck. (B) Phasenkontrastbild von MYOD1-UDCs eine Woche nach der Differenzierung. Skalenbalken = 50 m. Diese Zahl wurde von Takizawa et al.7geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Erfolgreiche Bewertung des Exon-Springens in Urin-abgeleiteten Zellen (UDCs), die von DMD-Patienten durch RT-PCR erhalten wurden. (A) RT-PCR-Analyse von Dystrophin nach Antisense-Oligonukleotid-Behandlung in 3-Deazaneplanocin A Hydrochlorid (DZNep)-behandelten MYOD1-UDCs,die von Duchenne-Muskeldystrophie (DMD) Patienten mit einer Exon 45-54 Deletion abgeleitet wurden. DZNep-behandelte MYOD1-UDCswurden auch mit dem Kontrollantisense bei 1-10 M Konzentration als Kontrolle behandelt. Die oberen Bänder waren ungeklappbare Produkte (Ex 45-54 Löschung), die aus dem Leserahmen blieben. Die unteren Bänder waren die exon 44-übersprungenen Produkte (Ex 44-54 Löschen und Ex 44 übersprungen), die den offenen Leserahmen wiederhergestellt. (B) Der Übersprungswirkungsgrad wurde mit einem Mikrochip-Elektrophoresesystem als (exon 44-skipped transcript molarity) x 100% berechnet. Einweg-ANOVA gefolgt von Bonferronis Post-hoc-Test wurde verwendet, um die Springeffizienzzustände zu vergleichen (n = 3 für jede Gruppe, ****P < 0.0001). Die Daten werden als Mittelwert SEM ausgedrückt. Diese Zahl wurde von Takizawa et al.7geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Erfolgreiche Auswertung des Exon-Springens in Urin-abgeleiteten Zellen (UDCs) von DMD-Patienten durch Western Blot. (A) Repräsentativer Westlicher Blot für Dystrophin in DZNep-behandelten MYOD1-UDCsvon DMD-Patienten mit exon 45-54 Deletion nach Exon 44 Skipping. Zur Dystrophindetektion wurde Antidystrophin (gegen C-Terminal) verwendet. (B) Die relativen Intensitäten der auf die Expression von A-Tubulin normalisierten Bänder wurden in patientenabgeleiteten Zellen mit und ohne Antisense-Oligonukleotid-Behandlung durch einseitige ANOVA verglichen, gefolgt von Bonferronis Post-hoc-Test (n = 3 für jede Gruppe, **P < 0,01, ***P < 0,001, HI = gesund). Diese Zahl wurde von Takizawa et al.7geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Heatmaps der Immunzytochemie für Dystrophin nach Antisense-Oligonukleotid-Behandlung in DZNep-behandelten MYOD1-UDCs,die von DMD-Patienten mit Exon 45-54-Deletion erhalten wurden. (A) Das Löschen von exon 45-54 stellte den offenen Leserahmen auf der Grundlage des Exon-Überspringens von exon 44 wieder her. (B) Die Signalintensität wurde mit einem Fluoreszenzmikroskop nach 1 Woche Antisense-Oligonukleotid-Transfektion auf einer 96-Well-Platte quantifiziert. Für den Vergleich wurde eine einwegige ANOVA gefolgt von Bonferronis Post-hoc-Test verwendet (n = 3-4 für jede Gruppe, ****P < 0,0001). Diese Zahl wurde von Takizawa et al.7geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Reagenz | Volumen | Endgültige Konzentration |
| 2x PCR-Vormischung | 12,5 l | 1x |
| Vorwärtsgrundierung | 5 pmol | 0,2 m |
| Reverse Primer | 5 pmol | 0,2 m |
| Vorlage | 80 ng | |
| Sterilisiertes destilliertes Wasser | bis zu 25 l | |
| Gesamtvolumen pro Reaktion | 25 l |
Tabelle 1: Mischung zur MYOD1-Verstärkung durch RT-PCR.
| 98 °C | 10 s | |
| 55 °C | 10 s | • 35 Zyklen |
| 72 °C | 10 s |
Tabelle 2: Bedingungen für den thermischen Cycler für die MYOD1-Verstärkung.
| Reagenz | Volumen |
| 10x K Puffer | 2 l |
| Retroviraler Vektor (500 ng/l) | 2 l |
| Restriktionsenzym 1 (2-15 U) | 1 L |
| Restriktionsenzym 2 (2-15 U) | 1 L |
| Sterilisiertes destilliertes Wasser | 14 l |
| Gesamtvolumen | 20 l |
Tabelle 3: Mischung für ein Rohr, um retroviralen Vektor zu verdauen.
| Reagenz | Volumen |
| Gereinigtes MYOD1-Fragment | 100 ng |
| Verdauter retroviraler Vektor | 100 ng |
| 5x Enzym-Premix | 4 l |
| Sterilisiertes destilliertes Wasser | bis zu 20 l |
| Gesamtvolumen | 20 l |
Tabelle 4: Mischung für die Infusions-Klonreaktion.
| Lösung | Volumen/Reaktion (L) | Endgültige Konzentration |
| RNasefreies Wasser | Variable | - |
| Einstufiger RT-PCR-Puffer | 4 | 1x |
| dNTP-Mischung (enthält 10 mM jedes dNTP) | 0.8 | 400 mM von jedem dNTP |
| Vorwärtsgrundierung (10 mM) | 1.2 | 0,6 mM |
| Reverse Primer (10 mM) | 1.2 | 0,6 mM |
| Einstufiger RT-PCR-Enzymmix | 0.8 | - |
| RNase-Inhibitor (optional) | Variable | 5 x 10 Einheiten/Reaktion |
| Template-RNA | 50 x 400 ng | |
| Gesamtvolumen | 20 |
Tabelle 5: Notwendige Verbindungen für eine Reaktion der einstufigen RT-PCR.
| 1 Zyklus | Umgekehrte Transkription | 30 Min. | 50 °C |
| 1 Zyklus | Erster PCR-Aktivierungsschritt | 15 Min. | 95 °C |
| 1 Zyklus | Denaturierung | 1 Min. | 94 °C |
| Glühen | 1 Min. | 60 °C | |
| Erweiterung | 1 Min. | 72 °C | |
| 1 Zyklus | Endgültige Verlängerung | 7 Min. | 72 °C |
| Halten | ∞ | 4 °C |
Tabelle 6: Thermische Cycler-Bedingung für einstufige RT-PCR.
| Reagenz | Volumen |
| Protein (15 g) | 10 l |
| Beispielpuffer (4x) | 5 l |
| Reduktionsmittel (10x) | 2 l |
| Deionisiertes Wasser | 3 l |
| Gesamtvolumen | 20 l |
Tabelle 7: Herstellung von Proben für Natriumdodecylsulfat Polyacrylamid-Gelelektrophorese (SDS-PAGE).
Diskussion
Hier beschreiben wir ein detailliertes Protokoll des Exon-Springens in MYOD1-konvertiertenUDCs, die von DMD-Patienten erhalten wurden. Mit dem Assay-System haben wir optimale Antisense-Sequenzen effizient gescreent. Wir gehen davon aus, dass MYOD1-konvertierteUDCs für die Untersuchung der Pathophysiologie der Krankheit nützlich sein können.
Die Bewertung des Exon-Überspringens unter Verwendung von Patienten-abgeleiteten Zellen auf mRNA-Ebene ist für das Screening neuer Medikamente und die Beurteilung der Patientenberechtigung vor der Durchführung klinischer Studien unerlässlich. Die Berechnung der Exon-Skipping-Effizienz kann nur auf mRNA-Ebene ausgewertet werden.
Die Bewertung des Exon-Springens auf Proteinebene ist auch deshalb wichtig, weil die Dystrophin-Wiederherstellung als Ersatz-Biomarker wichtig ist, um die Vorteile des Exon-Springens vorherzusagen. Bis heute wird das Screening von Antisense-Oligonukleotid-Sequenzen oft mit primären Muskelzelllinien oder verewigten Myoblast-Zelllinien einschließlich menschlicher Rhabdomyosarkom (RD)-Zellen durchgeführt, aber wir können die Erholung des Dystrophinspiegels nicht mit Muskelzelllinien oder RD-Zelllinien messen, da sie dieses Protein endogene ausdrücken. Wir können die Wiederherstellung von Dystrophin in von DMD-Patienten abgeleiteten MYOD1-UDCsauf dosisabhängige Weise eindeutig erkennen. In unserem neuen Test sind wir der Ansicht, dass die Bewertung des wiederhergestellten Proteins durch Western Blotting in der Quantifibilität überlegen ist. Andererseits ist die Auswertung durch Immunzytochemie mit 96 Wellplatten ideal, um viele Kandidatenverbindungen gleichzeitig zu screening.
In diesem Artikel beschreiben wir ein detailliertes Protokoll für eine effiziente Modellierung von DMD-Muskeln mit MYOD1-konvertiertenUDCs zusammen mit RT-PCR, Western Blotting und Immunzytochemie, um die wiederhergestellte Dystrophin auf mRNA- und Proteinspiegel nach dem Exon-Springen zu bewerten. UDCs können nicht invasiv und einfach gesammelt werden. Daher gehen wir davon aus, dass der brandneue In-vitro-Assay unabhängig von der Art der Muskelstörungen auf eine Breite von Basis- und Translationsstudien angewendet werden kann.
Offenlegungen
Das National Center of Neurology and Psychiatry entwickelt jetzt NS-065/NCNP-01, ein Exon 53 Skipping-Medikament für DMD, mit Nippon Shinyaku Co., Ltd.
Danksagungen
Diese Arbeit wurde von der Japan Society for the Promotion of Science Grant-in-Aid for Scientific Research (C) unterstützt [Grant-Nr. 18K07544 an Y.A.], Grants-in-Aid for Research on Nervous and Mental Disorders [Grant No. 28-6 to Y.A.] und the Japan Agency for Medical Research and Development [grant nos. 18ek0109239h0002, 18lm0203066h0001, and 18lm0203069h0001 to Y.A.].
Materialien
| Name | Company | Catalog Number | Comments |
| 1% P/S Solution Stabilized | Thermo Fisher | 15070-063 | Cell culture |
| Amphotericin B | Sigma Aldrich | A2942 | |
| Anti-dystrophin | Abcam | ab15277 | Western blot (WB) |
| Anti-dystrophin | Leica | NCL-DYS1 | Immunocytochemistry(ICC) |
| Anti-mouse IgG, Dylight 488 | Vector Laboratories | DK-2488 | ICC |
| Anti-α-tubulin | Sigma | T6199 | Western bot and ICC |
| BZ-X800 | KEYENCE | BZ-800 | Fluorescent microscope |
| CELLBANKER | ZENOAQ | CB011 | Cell stock in liquid nitrogen |
| ChemiDoc MP Imaging System | Bio-Rad | 170-8280J1 | WB |
| CloneAmp HiFi PCR premix | Clontech | 639298 | Retroviral production |
| cOmplete Protease Inhibitor Cocktail | Roche | 4693116001 | Protein extraction for WB |
| E.coli DH5 α Competent Cells | TAKARA | 9057 | |
| ECL Prime Western Blotting Detection Reagent | GE healthcare | RPN2232 | WB |
| EGF | Peprotech | AF-100-15 | Cell culture |
| Endo-Porter | GeneTools | 2922498000 | ASO transfection |
| Extra Thick Blot Filter Paper | Bio-Rad | 1703965 | WB |
| EzFastBlot HMW | Atto | AE-1460 | WB |
| fibroblast growth factor-basic | Sigma-Aldrich | F0291 | Cell culture |
| Glutamax | Thermo Fisher Scientific | 35050-061 | Cell culture |
| GP2-293 packaging cells | Clontech | 631458 | Retroviral production |
| Ham's F-12 Nutrient Mix | Thermo Fisher Scientific | 11765-054 | Cell culture |
| High glucose DMEM with GlutaMAX-I | Thermo Fisher Scientific | 10569-010 | Cell culture |
| High glucose DMEM without sodium pyruvate | GE Healthcare | SH30022.01 | Cell culture |
| HiSpeed Plasmid Purification Kit | QIAGEN | 12643 | Retroviral production |
| Histofine Simple Stain MAX PO | NICHIREI BIOSCIENCE INC. | 424151 | WB |
| Hoechst 33342 | Thermo Fisher Scientific | H3570 | ICC |
| Human PDGF-AB | Peprotech | 100-00AB-10UG | Cell culture |
| iBind Flex Solution | Thermo Fisher Scientific | SLF2020 | WB |
| iBind Flex Western Device | Thermo Fisher Scientific | SLF2000 | WB (Automated mestern-processing device) |
| Immobilon-P Transfer Membrane (PVDF) | MERCK | IPVH304F0 | WB |
| In-Fusion HD cloning Kit | Clontech | 639648 | Retroviral production |
| ITS Liquid Media Supplement | Sigma-Aldrich | I3146 | Cell culture |
| MILTEX HV 0.45 μm filter | MERCK | SLHV033RS | Retroviral production |
| MultiNA | SHIMADZU | MCE-202 | Microchip electrophoresis |
| MYOD1 (GFP-tagged) | ORIGENE | RG209108 | Retroviral production |
| NanoDrop | Thermo Fisher | ND-ONE-W | Spectrophotometer |
| Nonessential amino acids | Thermo Fisher | 11140-050 | Cell culture |
| NucleoSpin Gel and PCR Clean-Up Kit | Clontech | 740986.20 | PCR clean up |
| NuPAGE 3-8% Tris-Acetate Protein Gels | Invitrogen | EA03785BOX | WB |
| NuPAGE Antioxidant | Invitrogen | NP0005 | WB |
| NuPAGE LDS Sample Buffer | Invitrogen | NP0007 | WB |
| NuPAGE Sample Reducing Agent | Invitrogen | NP0009 | WB |
| NuPAGE Tris-Acetate SDS Running Buffer | Invitrogen | LA0041 | WB |
| One Step TB Green PrimeScript RT-PCR Kit | TAKARA | RR066A | Titer check of retroviral vector |
| PBS | Thermo Fisher Scientific | 14190-250 | Cell culture |
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23227 | WB |
| Polybrene Infection / Transfection Reagent | Sigma-Aldrich | TR-1003 | Retroviral infection |
| pRetroX-TetOne-Puro Vector | Clontech | 634307 | Retroviral vecor |
| Puromycin | Clontech | 631305 | Cell culture |
| QIAGEN OneStep RT-PCR Kit | Qiagen | 210212 | PCR |
| REGM Bullet Kit | Lonza | CC-3190 | Material for growth medium of UDCs |
| REGM SingleQuots | Lonza | CC-4127 | Material for primary medium of UDCs |
| Retrovirus Titer Set | TAKARA | 6166 | Titer check of retroviral vector |
| Retro-X Concentrator | Clontech | 631455 | Retroviral production |
| RIPA buffer | Thermo Fisher Scientific | 89901 | WB |
| RNeasy kit | Qiagen | 74104 | RNA extraction for PCR |
| Tetracycline-free foetal bovine serum | Clontech | 631106 | Cell culture |
| Triton-X | MP Biomedicals | 9002-93-1 | ICC |
| Trypsin-EDTA (0.05%) | Gibco | 25300054 | Cell culture |
| XCell SureLock Mini-Cell | Invitrogen | EI0001 | WB |
| Xfect transfection reagent | Clontech | 631317 | Transfection of plasmids into packaging cells |
Referenzen
- Hoffman, E. P., Brown, R. H., Kunkel, L. M. Dystrophin: the protein product of the Duchenne muscular dystrophy locus. Cell. 51, 919-928 (1987).
- Cirak, S., et al. Exon skipping and dystrophin restoration in patients with Duchenne muscular dystrophy after systemic phosphorodiamidate morpholino oligomer treatment: an open-label, phase 2, dose-escalation study. Lancet. 378, 595-605 (2011).
- Komaki, H., et al. Systemic administration of the antisense oligonucleotide NS-065/NCNP-01 for skipping of exon 53 in patients with Duchenne muscular dystrophy. Science Translational Medicine. 10 (437), (2018).
- Antoury, L., et al. Analysis of extracellular mRNA in human urine reveals splice variant biomarkers of muscular dystrophies. Nature Communications. 9, 3906 (2018).
- Rahmoune, H., et al. Glucose transporters in human renal proximal tubular cells isolated from the urine of patients with non-insulin-dependent diabetes. Diabetes. 54, 3427-3434 (2005).
- Zhang, Y., et al. Urine derived cells are a potential source for urological tissue reconstruction. The Journal of Urology. 180, 2226-2233 (2008).
- Takizawa, H., et al. Modelling Duchenne muscular dystrophy in MYOD1-converted urine-derived cells treated with 3-deazaneplanocin A hydrochloride. Scientific Reports. 9, 3807 (2019).
- Zhou, T., et al. Generation of human induced pluripotent stem cells from urine samples. Nature Protocols. 7, 2080-2089 (2012).
- Chen, W., et al. Skeletal myogenic differentiation of human urine-derived cells as a potential source for skeletal muscle regeneration. Journal of Tissue Engineering and Regenerative Medicine. 11, 334-341 (2017).
- Kim, E. Y., Page, P., Dellefave-Castillo, L. M., McNally, E. M., Wyatt, E. J. Direct reprogramming of urine-derived cells with inducible MyoD for modeling human muscle disease. Skeletal Muscle. 6, 32 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten