Method Article
Floresan Protein İşaretleyicileri ile Maya Organellerinin Görselleştirilmesi
Bu Makalede
Özet
Burada, tomurcuklanan maya Saccharomyces cerevisiae'nin canlı hücre görüntülemesinde bir dizi floresan protein bazlı organel markörünün kullanımını açıklıyoruz.
Özet
Tomurcuklanan maya, Saccharomyces cerevisiae, organel işlevi ve dinamiği üzerinde çalışan klasik bir model sistemdir. Önceki çalışmalarımızda, çekirdek, endoplazmik retikulum (ER), Golgi aygıtı, endozomlar, vakuoller, mitokondri, peroksizomlar, lipid damlacıkları ve otofagozomlar dahil olmak üzere ana organeller ve endomembran yapılar için floresan protein bazlı belirteçler oluşturduk. Burada sunulan protokol, maya dönüşümü için DNA hazırlığı, transformantların seçimi ve değerlendirilmesi, floresan mikroskobik gözlem ve beklenen sonuçlar dahil olmak üzere bu belirteçlerin mayada kullanılmasına ilişkin prosedürleri açıklamaktadır. Metin, maya organeli çalışması alanına başka geçmişlerden giren araştırmacılara yöneliktir. Temel adımların yanı sıra mikroskop donanımı hususları ve birkaç yaygın tuzak hakkında teknik notlar ele alınmaktadır. İnsanların canlı hücre floresan mikroskobu ile maya hücre altı varlıklarını gözlemlemeleri için bir başlangıç noktası sağlar. Bu araçlar ve yöntemler, protein hücre altı lokalizasyonunu tanımlamak ve hızlandırılmış görüntülemede ilgilenilen organelleri izlemek için kullanılabilir.
Giriş
Zara bağlı organellere hücre altı bölümlere ayırma, ökaryotik hücrelerin organizasyonunda yaygın bir ilkedir. Her organel belirli işlevleri yerine getirir. Ökaryotik biyolojinin diğer birçok alanında olduğu gibi, tomurcuklanan maya, Saccharomyces cerevisiae, organel organizasyonu ve dinamiğinin temel ilkelerini aydınlatmada klasik bir model sistem olmuştur. Örnekler arasında protein salgılama yolu, peroksizomal protein ithalat yolu ve otofaji yolu 1,2,3'teki ufuk açıcı keşifler yer alır.
Tipik besin açısından zengin koşullarda, hızlı büyüyen maya hücreleri endoplazmik retikulum (ER), erken Golgi, geç Golgi/erken endozomlar, geç endozomlar, vakuoller ve mitokondri içerir. Bazı peroksizomlar, lipid damlacıkları ve otofagozomlar (ilk ikisinden bile daha az, esas olarak besin açısından zengin koşullarda bulunan Cvt vezikül tipinden4) de mevcuttur, ancak belirli kültür koşullarında (lipid açısından zengin ortam, açlık ortamı, vb.) Diğer yaygın ökaryotik modellerle karşılaştırıldığında, maya hücreleri oldukça küçüktür; Tipik bir maya hücresinin çapı, çoğu hayvan ve bitki hücresi için onlarca mikrometreye kıyasla yaklaşık 5 μm'dir. Sonuç olarak, normalde tek bir yapışık hayvan hücresi içeren aynı görüntüleme alanında, normalde çeşitli hücre döngüsü aşamalarında onlarca maya hücresi görülür. Boyut farkının yanı sıra, maya organel morfolojisinin de bazı kendine özgü özellikleri vardır. Ultrastrüktürel düzeyde, maya ER, diğer sistemlerde olduğu gibi tabaka ve tübüllerden oluşur. Floresan mikroskobu altında, maya ER, aralarında bazı birbirine bağlı yapılar bulunan iki halka olarak kendini gösterir. İç halka, nükleer zarf ile sürekli olan nükleer ER'dir ve dış halka, plazma zarının5 altında yatan boru şeklinde bir ağ olan periferik ER'dir. Bitki hücrelerine benzer, ancak hayvan hücrelerinden farklı olarak, melez bir organel olan geç Golgi/erken endozom, salgı yolu ile endositik yol 6,7 arasındaki kesişme noktasında yer alır. Morfolojik olarak, maya Golgi aygıtları sitoplazmada dağılır. Vakuoller fonksiyonel olarak hayvan hücrelerindeki lizozomlara benzer. Genellikle sitoplazmanın büyük bölümlerini işgal ederler ve sık sık fisyon ve füzyona uğrarlar. Floresan kolokalizasyon belirteçlerinin kullanılmasının yanı sıra, vakuolar membran nükleer ER'den en az iki kriter ile ayırt edilebilir: Vakuolar membran genellikle nükleer ER'den daha yuvarlaktır ve DIC'deki vakuolün içbükey görünümü de çekirdeğinkinden daha belirgindir.
Rutin olarak, canlı maya hücrelerinde yukarıda belirtilen organelleri görselleştirmek için bir dizi floresan protein bazlı işaretleyici kullanırız (Tablo 1). Bu organel belirteçlerinin aslına uygunluğu ve işlevselliği deneysel olarak doğrulanmıştır 7,8. Bu belirteç yapıları, floresan protein kimera kasetlerini maya genomuna sokmak için tasarlanmıştır. Aşağıda özetlendiği gibi, maya transformasyonuna hazırlık olarak, doğrusal DNA fragmanları ya enzimatik sindirim ya da PCR amplifikasyonu 7,8 ile üretilir. Doğrusal DNA parçaları, homolog rekombinasyon yoluyla genoma entegre edilir. Bu protokolde açıklanan plazmitler için üç tip tasarım kullanılır. Plazmitlerin çoğunu kapsayan ilk tipte, yapının birden fazla kopyasını taşıyan dönüştürücüler elde etmek genellikle mümkündür. Bu genellikle istenmeyen bir durumdur çünkü dönüştürücüler arasında ifadesel ve muhtemelen işlevsel farklılıklar sunar. Tek kopya transformantların, bu protokolde tarif edildiği gibi görüntüleme, immünoblotlama veya dikkatlice tasarlanmış PCR testleri yoluyla tanımlanması gerekir. GFP-Sed5, GFP-Pep12 ve GFP-Atg8'i kapsayan ikinci tipte, haploid maya hücrelerinde sadece tek kopya entegrasyonu üretilir. Hem birinci tip hem de ikinci tip, işaretleyici genin endojen kopyasını genomda bozulmadan tutar. Sec7-2GFP ve Vph1-2GFP'yi kapsayan üçüncü bir plazmit tasarımı tipinin, kimeraların karşılık gelen işaretleyici genin tek kopyası olmasına yol açan C-terminal knock-in'leri tanıtması amaçlanmıştır.
Burada, bu organel belirteçlerini kullanma prosedürünü açıklıyoruz, örnek mikroskopi görüntüleri sağlıyoruz ve maya organel görüntülemeye yeni başlayan araştırmacılara yönelik önlemleri tartışıyoruz.
Protokol
1. Maya suşu yapısı
- İşaretleyici plazmitler ve uygun bir maya suşu elde edin.
NOT: Plazmitler Addgene'den temin edilebilir. Bu protokol örnek olarak TN124 (MATa ura3 trp1 pho8Δ60 pho13Δ::LEU2), BY4741(MATa leu2Δ0 ura3Δ his3Δ1 met15Δ0) ve DJ03 (BY4741 trp1Δ::MET15) kullanır. Bilimsel sorunun doğası dışında, gerinim seçimi için önemli bir husus, seçim belirteçlerinin uyumluluğudur. Burada açıklanan organel işaretleyici plazmitleri, seçim belirteçleri olarak URA3 ve TRP1'i kullanır. Bu nedenle, alıcı suşun genotipinin ura3 ve trp1 olması gerekir. Aksi takdirde, suşu veya plazmitleri değiştirmek gerekir. - Maya ortamını aşağıdaki tariflere göre hazırlayın.
NOT: SMD (sentetik minimal dekstroz; %2 glikoz, amino asit içermeyen %0.67 maya azot bazı (YNB), 30 mg/L adenin, 30 mg/L lizin, 30 mg/L metiyonin, 20 mg/L histidin, 20 mg/L urasil, 50 mg/L triptofan, 50 mg/L lösin). SMD + CA (% 0.5 kasamino asit ilavesi ile SMD). YPD (Maya özütü pepton dekstroz; %1 maya özütü, %2 pepton, %2 glikoz). SD-N (azot içermeyen sentetik dekstroz; amino asit ve amonyum sülfat içermeyen %0.17 YLB, %2 glikoz). Ortam seçimiyle ilgili düşünceler için lütfen Tartışma bölümüne bakın. - Dönüşüm adımından önce, bir organel işaretleyici plazmidinin enzimatik sindirimi veya PCR amplifikasyonu ile doğrusallaştırılmış DNA fragmanları oluşturun.
NOT: Tablo 1 , organel işaretleyici plazmitlerinden doğrusal fragmanlar oluşturmak için kısıtlama enzim bölgelerini ve PCR primerlerini listeler. - Sıvı YPD ortamında maya hücrelerini kültürleyin ve mayayı doğrusal DNA fragmanı ile dönüştürün.
NOT: Maya dönüşümü, geleneksel LiAc bazlı yöntem9 veya tercih edilen diğer yöntemler kullanılarak gerçekleştirilebilir. Transformasyon için uygun kontrollerin, özellikle plazmit veya plazmidden türetilmiş DNA içermeyen negatif bir kontrolün dahil edilmesi tavsiye edilir. - Uygun bir seçim plakasında (örneğin, URA3 seçimi için SMD-Ura ortamı kullanın) 30 ° C'de 2-3 gün inkübe edin.
NOT: Tek kolonilerin ortaya çıkması yaklaşık 2 gün sürer. - Floresan kimera ekspresyonunu ve entegrasyon kopya numarasını doğrulamak için, seçim plakasından sekiz koloni seçin, yeni seçim plakalarına yeniden çizin ve 30 °C'de inkübe edin.
2. Floresan mikroskobu: genel prosedürler ve tek zaman noktası görüntüleme
- Kültür mayası hücreleri, SMD sıvı ortamda gece boyunca 30 ° C'de çalkalanarak bekletilir.
- Ertesi sabah, maya kültürünün optik yoğunluğunu bir spektrofotometre veya plaka okuyucuda 600 nm'de (bundan sonra OD600 olarak anılacaktır) ölçün.
NOT: Mayanın aşılanması ile ilgili bazı uygulamalarla, genellikle bu aşamada tüm numunelerin OD600'ünün 2'den düşük olduğundan emin olunabilir. Daha yüksek biterse, bir sonraki adımda daha fazla seyreltilmesi ve maya hücrelerinin iyileşmesi için daha fazla zaman tanınması tavsiye edilir. - Taze bir ortam kullanarak maya kültürünü yaklaşık 0.2 OD600'e seyreltin.
- OD600 yaklaşık 0.8-1.0'a ulaşana kadar kültürlemeye devam edin.
NOT: Mayanın iki katına çıkma süresi yaklaşık 1.5-2 saattir. - Düz bir yüzeye kağıt mendilin üzerine bir kapak camı koyun, kapak camının üst tarafına 5 μL 1 mg / mL concanavalin A yayın ve 5 dakika bekleyin (Şekil 1A)10.
NOT: Bu hazırlık adımı önceden yapılabilir. - 100 μL maya kültürünü kapak camının üst tarafına aktarın ve 5 dakika bekleyin.
- Kapak camını, maya hücreleri arasına sıkıştırılmış olarak destekleyici bir cam sürgü ile birleştirin; Ataşmanı sabitlemek için uygun kuvvetle bastırın.
NOT: Doğru basıncı bulmak biraz pratik gerektirir, böylece maya hücreleri hareketsiz tek bir tabaka oluşturur ancak ezilmez (Şekil 1B). Bu adım sırasında sıvı ortam, alttaki kağıt mendil tarafından dışarı itilecek ve emilecektir. - Numune slaytını mikroskop tablasına monte edin. Diferansiyel girişim kontrastı (DIC) veya faz kontrastı (PC) aydınlatması kullanarak bir maya hücresi yamasını bulun ve odaklanın.
NOT: Hücreleri bulmak için floresan kullanmayın; aksi takdirde, veri toplanmadan önce sinyal ağartılabilir. - Görüntü z yığınlarını toplamak için üç parametre kümesini manuel olarak yapılandırın: z kesiti, görüntüleme kanalları ve pozlama parametreleri.
NOT: Bir ticari yazılımdaki parametre ayarlarının GUI örnekleri için Ek Şekil 1'e bakın. Tam görünüm, yazılım platformlarına göre farklılık gösterir, ancak seçenekler aşağı yukarı aynıdır.- Z kesiti: 15 dilim için 0,5 μm'lik adımlarla dilimleri toplayın (7 μm derinliği kaplar, genellikle normal haploid maya hücreleri için yeterlidir).
- Görüntüleme kanalları: Hücre konturları ve gerektiğinde uygun floresan kanalları için DIC veya PC'yi seçin.
- Uyarma ışığı yoğunluğu ve maruz kalma süresi: Uyarma ışığı yoğunluğunu ve maruz kalma süresini örnek için uygun şekilde ayarlayın.
NOT: Başlangıç noktası olarak, uyarma ışığı yoğunluğu için %100 ve maruz kalma süresi için 100 ms kullanın; Z-yığını toplamanın ilerlemesiyle fotoağartma belirgin hale gelirse veya sinyal kamera kayıt kapasitesini aşıyorsa (yani sinyal doygunluğu) ışık yoğunluğunu azaltın; Sinyal-gürültü oranı düşükse ışık yoğunluğunu artırın.
- Görüntüleme ayarlarını not edin ve karşılaştırılacak tüm örnekler için aynı ayarları kullanın.
NOT: Veri toplama ve görüntü görselleştirme için "otomatik" ayarını kullanmayın. Ayarları bir laboratuvar notuna kaydetmek önemlidir, böylece aynı parametreler gelecekteki deneysel tekrarlarda yeniden uygulanabilir. Bazı mikroskop kontrol yazılımı uygulamaları, ayarları kaydetme ve yeniden uygulama yeteneğine sahiptir; Yine de, yazılım profilleri iş arkadaşları tarafından istemeden silinebileceğinden veya değiştirilebileceğinden, ayarların bağımsız olarak kaydedilmesi tavsiye edilir. - Verileri 16 bit çok kanallı yığın biçiminde kaydedin.
NOT: 8 bit RGB resimler (veya üç rengi de sayarsanız 24 bit) olarak kaydetmeyin. 8 bit formatın sınırlamaları için görüntü görselleştirme bölümüne (Bölüm 4) bakın. - Bir sonraki görüntü yığını koleksiyonu için tamamen farklı bir alana gidin.
NOT: Bitişik alanlarda foto ağartma ve fototoksisite yaşanmış olabilir. Sonuç olarak, bu alanlardan toplanan görüntü yığını yanıltıcı olabilir.
3. Hızlandırılmış görüntüleme
NOT: Hızlandırılmış görüntüleme prosedürü, tek zaman noktası görüntüleme prosedüründen iki alanda farklılık gösterir: numune hazırlama ve görüntüleme parametreleri.
- Numune hazırlama
- 35 mm'lik cam tabanlı bir tabağı 1 mg / mL concanavalin A ile kaplayın ve 5 dakika veya daha fazla bekleyin.
- Tabağa 1.5 mL maya sıvı kültürü ekleyin ve maya hücrelerinin cam yüzeye çökmesi için 5 dakika bekleyin.
- Bir pipet kullanarak, sıvı ortamı tabağın kenarından aspire edin, ardından güvenli olmayan bir şekilde bağlanmış hücreleri çıkarmak için maya tortusu yamasını yaklaşık 1 mL taze ortamla nazikçe durulayın.
- Durulamayı 2-3 kez tekrarlayın. Bir pipet ile aspire edin, ardından yavaşça 2 mL taze kültür ortamı ekleyin.
- Görüntüleme parametreleri
- Z-kesiti: 15 dilim için dilimleri 0,5 μm'lik adımlarla toplayın.
- Görüntüleme kanalları: Gerektiği gibi seçin.
- Uyarma ışığı yoğunluğu ve maruz kalma süresi: İncelenen hücre altı yapıyı ayırt etmek için gereken minimum değeri kullanın.
NOT: Hızlandırılmış görüntüleme için, tekrarlanan aydınlatmadan kaynaklanan foto ağartma ve fototoksisite, toplanabilecek toplam zaman noktası sayısını sınırlar. Bu nedenle, uyarma yoğunluğu ve maruz kalma süresi genellikle düşük değerlere ayarlanır, böylece daha fazla zaman noktası görüntülenebilir. - Görüntüleme aralığı: İncelenmekte olan biyolojik süreç için uygun zamanlama aralıklarını ayarlayın.
NOT: Örneğin, açlık koşullarında bir otofagozomun oluşumu yaklaşık 5-10 dakika sürer; Dinamiklerini izlemek için 1 dakika veya daha kısa bir aralık tercih edilir. Besin açısından zengin koşullar altında, maya hücresi döngüsü saat ölçeğinde gerçekleşir; 10-20 dk gibi daha uzun bir aralık kullanılabilir.
- Uygun donanım mevcutsa, yemek sıcaklığını 30 °C'de tutun.
NOT: Maya hücrelerindeki çoğu biyolojik süreç, genel olarak daha yavaş bir hız dışında, oda sıcaklığında (RT) da görüntülenebilir.
4. Görüntü yığınlarının görselleştirilmesi ve entegrasyon kopya numarasının değerlendirilmesi
- Görüntü görselleştirme ve analizi için Fiji veya ImageJ yazılımını yükleyin11,12.
NOT: Fiji, genel amaçlı biyolojik görüntü verilerinin görselleştirilmesi ve işlenmesi için uygun, ImageJ tabanlı bir araç koleksiyonudur. Bu protokolde listelenen görüntü işleme için, ek eklentiler olmadan ImageJ yeterlidir. - Görüntü görüntüleme ve karşılaştırma için uygun parametreleri seçin.
- Fiji'de birkaç z-stack görüntüsü açın.
- Her yığındaki z konumunu orta bölüme (veya incelenen yapı orada daha iyi görselleştiriliyorsa diğer konumlara) sürükleyin.
- Görüntü > Parlaklığı/Kontrastı Ayarla > gidin ve Sıfırla'ya tıklayın.
- Parlaklık/Kontrast (B&C) penceresinde, incelenen yapıyı net bir şekilde ayırt etmek için Minimum ve Maksimum değerler olmak üzere iki parametreyi değiştirmek için kaydırma çubuklarını kullanın.
NOT: Genel olarak, Minimum değeri boş alanlarda ortalama değere yakın olacak şekilde ayarlanır ve Maksimum değeri görüntüdeki maksimum değere yakın olacak şekilde ayarlanır. Bu değerler görüntülenen histogramdan çıkarılabilir (Şekil 2A). Görüntüleme iş istasyonu renk kalibrasyonluysa, daha fazla arka plan sinyalini gizlemek için minimum değer biraz daha yükseğe ayarlanabilir. - Brightness / Contrast (B&C) penceresinde, Set'e tıklayın ve açılır pencerede Set Display Range (Ekran Aralığını Ayarla) seçeneğiyle Proparate all other Open Images (Diğer Tüm Açık Görüntülere Yay) onay kutusunu seçin.
NOT: Bu işlem, görüntülerin çapraz karşılaştırılabilmesi için açılan tüm görüntülere aynı parlaklık/kontrast ayarlarını (minimum ve maksimum değerler dahil) uygular. - Farklı görüntülere bakın ve her birindeki z yığınında gezinin. Görüntüler uygun bir koyu arka plan ve biraz abartılı doygunluklarla iyi görünüyorsa, Minimum ve Maksimum ayarları not edin.
- Çok kanallı görüntüler için, her kanal için görüntü görüntüleme ayarlarını seçin:
- Görüntü > Renkli > Kanallar Aracı'na gidin, açılır pencerede Gri tonlama modunu seçin Kanallar.
- Her kanalı gözden geçirin, o kanal için uygun Parlaklık/Kontrastı seçin ve ayarları not edin.
- Parlaklık/Kontrast ayarlandıktan sonra, Kanallar penceresine geri dönün ve birden çok kanalın aynı anda görselleştirilmesi için Bileşik moda geçin.
NOT: Her kanal için sahte renk, kişisel tercihe uyacak şekilde Kanallar'da manuel olarak değiştirilebilir. Onay kutuları, belirli kanalları göstermek/gizlemek içindir.
- Gerekirse, belirli kanalları göstermek veya gizlemek için Kanallar penceresindeki onay kutularını kullanın.
- İsterseniz, Görüntü > Yığınları > Z Projesi'ne giderek yığın projeksiyonları oluşturun.
NOT: Çeşitli projeksiyon modları mevcuttur. Maksimum yoğunluk, hızlı değerlendirme için iyi bir seçimdir. Niceleme için belirli bir projeksiyon modunun mu yoksa tek tek dilimlerin mi kullanılması gerektiğini belirlemek için araştırmanın doğası göz önünde bulundurulmalıdır. Projeksiyon, odak dışı ışığın girişimini azaltmak için belirli z dilimleriyle de sınırlandırılabilir. - Tek kopya tümleştirme dönüştürücüleri için ekran.
- Açılan tüm görüntülere aynı Parlaklık/Kontrast ayarları uygulandıktan sonra, plazmit entegrasyon numarasını çıkarmak için numuneler arasındaki görüntü yoğunluklarını karşılaştırın.
NOT: Görüntüler, sıvı bir ortamda gece boyunca kültürlemeden başlayarak, tek zaman noktası görüntüleme için genel prosedür izlenerek elde edilmelidir. Çoklu kopya entegrasyonları (örnekler için Şekil 2B'deki koloni 2 ve 3'e bakınız) tek kopya olanlardan ( Şekil 2B'deki koloni 1) önemli ölçüde daha parlak görünür. Buna karşılık, tek kopya olanlar birbirine benzer (Şekil 2B). - Aynı Parlaklık/Kontrast ayarlarını kullandığınızdan emin olarak her gerinim için sekiz dönüştürücüden gelen görüntüleri inceleyin.
- Sonraki çalışma için üç veya daha fazla tek kopya dönüştürücüyü yeniden çizin ve kaydedin.
- Açılan tüm görüntülere aynı Parlaklık/Kontrast ayarları uygulandıktan sonra, plazmit entegrasyon numarasını çıkarmak için numuneler arasındaki görüntü yoğunluklarını karşılaştırın.
- Görüntüleri sunum için dışa aktarın.
- İlgilendiğiniz görüntünün bir kopyasını oluşturmak için Görüntü > Çoğalt'a gidin ve ona tercih edilen bir ad verin.
- Yinelenen kopyayı 8 bit> ten 16 bit'e dönüştürmek ve gerektiği gibi kaydetmek için Görüntü > Türüne gidin ve gerektiği gibi kaydedin.
NOT: Bu işlem uygulandıktan sonra, Parlaklık/Kontrast ayarları daha önce yazılmamışsa, bu bilgileri almanın kolay bir yolu olmadığını ve dolayısıyla sonraki görüntü görselleştirmelerinde aynı ayarı yeniden uygulamanın kolay bir yolu olmadığını unutmayın. Bu aynı zamanda önceki çoğaltma işleminin önerilmesinin nedenidir. - Bir z yığınını tek tek görüntülerden oluşan bir koleksiyona bölmek için Görüntü > Yığınları > Görüntülere Yığınla'ya gidin ve gerektiği gibi kaydedin.
NOT: Görüntüler Fiji'de daha küçük bir alana da kırpılabilir.
Sonuçlar
Organel morfolojisi ve dinamiği, maya hücreleri dış ve iç sinyallere yanıt verdikçe değişebilir. Burada, orta log fazındaki maya organellerinin temsili görüntülerini sunuyoruz (Şekil 3A,B). Daha önce de belirtildiği gibi, birkaç organelin kendine özgü morfolojik özellikleri vardır, bu nedenle diğer organel belirteçleri ile kapsamlı bir karşılaştırma yapmadan tanınması kolaydır. Bunlara ER, mitokondri ve vakuoller dahildir. Burada gösterdiklerimiz de dahil olmak üzere bazı laboratuvar suşlarında, vakuollerin fisyon-füzyon dengesinin, birçok log faz hücresinin tek bir vakuol içerdiği ölçüde çarpık olduğuna dikkat edin. Golgi aygıtları, endozomlar, lipid damlacıkları ve peroksizomlar genellikle normal ışık mikroskobunun konturlarını çözmesi için çok küçüktür. Böylece hepsi sitoplazmik noktalar olarak görünür. Kimliklerini belirlemek, log faz hücrelerinde7 mevcut işaretleyici seti için gerçekleştirdiğimiz diğer organel belirteçleri ile çapraz karşılaştırma gerektirir.
Giriş bölümünde sunulduğu gibi, belirteç yapılarımızın çoğu genomda birden çok kez entegre edilme potansiyeline sahiptir. Gereksiz komplikasyonları önlemek için, tek kopya entegrasyonuna sahip dönüştürücüler, işaretleyici ekspresyonları floresan mikroskobu ile doğrulanırken taranabilir (Şekil 2B). Birden fazla kopya taşıyan transformatörlerdeki floresan sinyali, tek bir kopyaya sahip olanlardan çok daha parlaktır.
Canlı maya hücreleri içeren mikroskop lamlarının hazırlanması, tek bir deneyde onlarca numuneyi taramak için hızlı bir şekilde yapılabilen basit bir işlemdir. Bununla birlikte, slaytlar üzerinde tek bir hücre katmanını ezmeden sabitleyen "doğru" basıncı bulmak biraz pratik gerektirir (Şekil 1B). Çok az kuvvet, hücreler birden fazla katmanda kalır ve muhtemelen etrafta yüzer; Çok fazla kuvvet, hücreler esasen mekanik olarak parçalanır. Tecrübeyle, vahşi tip log faz popülasyonlarında nadiren ortaya çıkması gereken ölü hücreler ve eski hücreler de tespit edilebilir.
Hızlandırılmış görüntüleme, membran dinamiklerini araştırmak için güçlü bir tekniktir. Burada, GFP-Atg8 ile etiketlenmiş aç maya hücrelerinde otofagozomların sık oluşumunu ve tüketimini gösteriyoruz (Şekil 4). Literatürde, bir maya hücresinin otofagozom oluşturan tek bir hücre altı bölgeye sahip olduğuna dair iddialar vardır. Ortalama olarak hücre başına yaklaşık bir nokta olduğundan, yalnızca tek zaman noktalı anlık görüntüleri kontrol ediyorsa kulağa doğru geliyor. Bununla birlikte, hızlandırılmış görüntülemede, tek bir maya hücresinde, otofagozom biyogenezinin farklı örneklerinin zaman içinde farklı hücre altı konumlarda bağımsız olarak meydana gelebileceği kolayca görülebilir.
Her bir organelin dinamiklerini izlemenin yanı sıra, organel belirteçlerinin bir başka yaygın kullanımı, ilgilenilen bir proteinin hücre altı lokalizasyonunu belirlemektir. Pratikte, hücrelerdeki proteinler genellikle birden fazla organele lokalizedir. Burada örnek olarak Sft1'i kullanıyoruz; Sft1, hücre içi taşımada çalışan bir v-SNARE'dir13. GFP etiketli Sft1, çoklu sitoplazmik noktalar olarak ortaya çıkar (Şekil 5). Birlikte ifade edilen kırmızı organel yapıcılarla çapraz karşılaştırma yaparak, Sft1'in erken Golgi ve geç Golgi/erken endozom belirteçleri ile kısmen kolokalize olduğu görülebilir, bu da Sft1 fonksiyonunu anlamak için önemli ipuçları sağlar.

Şekil 1: Örnek slayt hazırlama. (A) Hazırlığın karton çizimi. (1) Kağıt mendil üzerine bir kapak camı yerleştirin, concanavalin A solüsyonunu kapak camının üst tarafına yayın ve 5 dakika veya daha uzun süre bekleyin. (2) Kapak camının üst tarafına ~ 100 μL maya kültürü ortamı ekleyin ve hücrelerin oturması için 5 dakika bekleyin. (3) Kapak camını bir cam sürgü ile örtün ve kapak camıyla birleştirmek için bastırın. Fazla sıvı, alttaki kağıt mendil tarafından emilir. (B) İstenmeyen durumlarda maya hücrelerinin ortaya çıkması. Soldan sağa: slayt hazırlamada yetersiz kuvvet uygulandığında hücre katmanlarının oluşumu; lam hazırlamada aşırı kuvvet uygulandığında ezilmiş maya hücreleri; oklarla işaretlenmiş potansiyel olarak ölü iki hücre; oklarla işaretlenmiş çok büyük bir vakuole sahip eski bir hücre. Ölçek çubuğu: 2 μm. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 2: Görüntü gösterimini ayarlama ve tek kopya entegrasyon dönüştürücülerini tanımlama. (A) Minimum görüntüleme değerini ayarlamanın etkisi. ImageJ/Fiji'de 16 bitlik bir görüntünün görsel görünümü, kullanıcı tarafından belirlenen görüntü aralığına bağlıdır. Burada, minimum değerin ayarlanmasının endoplazmik retikulumun görünümü üzerindeki etkisi gösterilmektedir. Değer, Parlaklık ve Kontrast (B &C) penceresinde ayarlanabilir. Aynı 16 bit görüntünün görünümü, bu ayar değiştikçe değişir. Solda, 0 değeriyle, kontrast düşüktür ve arka plan sinyali hücre olmayan alanlarda görünür. Sağ tarafta, 400 değeri arka plandan önemli ölçüde daha yüksektir; Sonuç olarak, periferik endoplazmik retikulum (yani plazma zarının altındaki ağ) gizlenir. Parlaklık ve Kontrast (B&C) penceresindeki histogram, sinyal yoğunluğunun görüntü boyunca dağılımı hakkında bilgi sağlar ve bu, uygun görüntüleme aralığının seçilmesinde bir kılavuz olarak kullanılabilir. Ölçek çubuğu: 2 μm. (B) Floresan sinyal yoğunluğunu karşılaştırarak transformatörler arasında yapı entegrasyon sayısının çıkarılması. Görüntüleri çekerken aynı pozlama parametrelerinin kullanılması gerektiğini unutmayın. Bu örnekte, üç farklı dönüştürücüden gelen görüntüler karşılaştırılmaktadır. Koloni 1, tek kopya entegrasyonlu bir dönüştürücüdür; Koloni 2 ve Koloni 3 çok kopya integrasyonlu dönüştürücülerdir. En üst satırda, her görüntüye farklı minimum ve maksimum eşikler uygulandı (sırasıyla 80-1400, 80-3000, 80-4000), daha parlak örneklere daha yüksek maksimum değerler uygulandı. Bu görünümde, her üç örnekte de benzer hücre altı dağılım modelleri görülebilir. Alt satırda, her üç görüntüye de aynı minimum ve maksimum eşikler (80-2000) uygulandı. Bu görüşe göre, koloni 2 ve koloni 3'teki daha yüksek sinyal seviyeleri belirgindir ve bu da genomlarında Rtn1-msGFP yapısının birden fazla kopyasını taşıdıkları gerçeğini yansıtır. Ölçek çubuğu: 2 μm. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 3: Ana maya organellerinin tipik morfolojisi. (A) Temsili anlık görüntü görüntüleri. Floresan protein yapıları ve temsil ettikleri organeller görüntülerin solunda belirtilmiştir. DIC: diferansiyel girişim kontrastı, tek bir dilim. Dilim: Floresan kanalının z yığınındaki tek bir dilim. Projeksiyon: Floresan kanalın maksimum yoğunluk projeksiyonu. Burada, organellerin dilimlerdeki görünürlüğünü artırmak için 16 bitten 8 bite eşlemenin dilimler ve projeksiyonlar için farklı şekilde yapıldığını unutmayın. Ölçek çubuğu: 2 μm. (B) Nükleer endoplazmik retikulum/nükleer zarf ve vakuol arasındaki morfolojik farklılıklar. Hem nükleer endoplazmik retikulum hem de vakuoller, hücrenin merkezinde görüntülendiğinde dairesel yapılar olarak kendini gösterir. Vakuollerle karşılaştırıldığında, nükleer endoplazmik retikulum genellikle daha az yuvarlaktır ve DIC'deki karşılık gelen alan da daha az belirgindir. Ölçek çubuğu: 2 μm. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
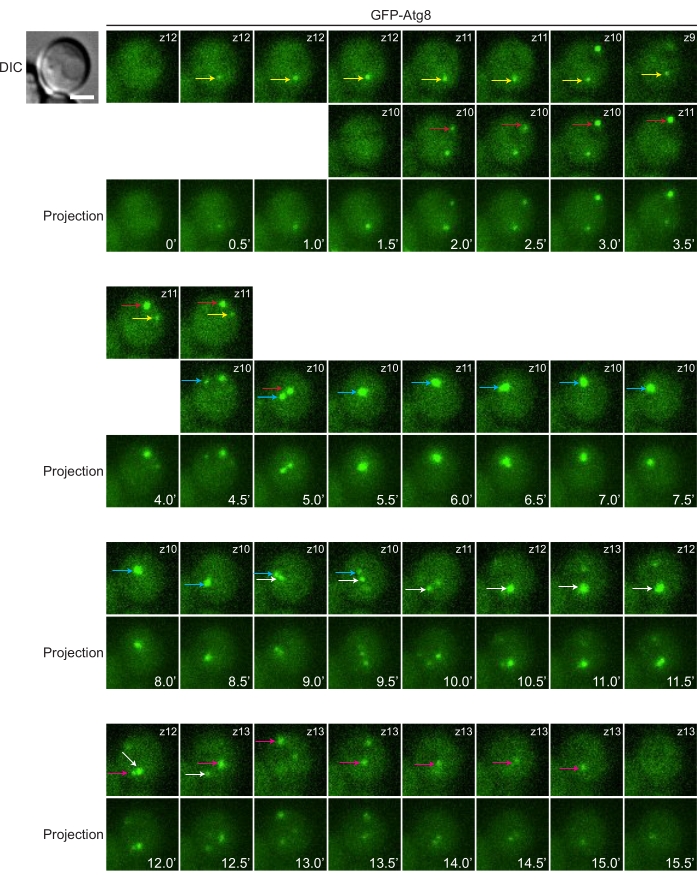
Şekil 4: Hızlandırılmış görüntüleme ile otofagozomların dinamiklerini takip etmek. GFP-Atg8 eksprese eden maya hücreleri, YPD ortamında orta log fazına kadar büyütüldü, daha sonra açlık için SD-N ortamına kaydırıldı. Ortam geçişinden 45 dakika sonra, maya hücreleri cam tabanlı tabaklara monte edildi ve hızlandırılmış görüntüleme için SD-N ile inkübe edildi. Görüntü yığınları 30 sn aralıklarla toplanmıştır. Bu örnekte, beş tur otofagozom oluşumu ve kaybolması görülebilir. Atg8 pozitif otofajik yapıları temsil eden noktalar zaman içinde manuel olarak izlendi ve farklı renkteki oklarla etiketlendi (yani, aynı renkteki oklar takip edilen aynı noktayı gösterir). Herhangi bir belirli zaman noktasında, farklı z konumlarında farklı noktaların (birden fazla varsa) görünebileceğini unutmayın. Odakta izlenen noktaları içeren tek tek dilimler, sağ üst köşede belirtilen z konumu ile üstteki bir veya iki satırda gösterilir (yani, z10 10. dilimi ve z12 12. dilimi belirtir). Maksimum yoğunluklu projeksiyonun kullanılması, tüm yapılara göz atmak için uygun bir yaklaşımdır. Bununla birlikte, her bir yapının konturu, tek dilimlerdeki kadar net değildir. Maksimum yoğunluk projeksiyonları, dilimlerin altındaki satırda gösterilir. Ölçek çubuğu: 2 μm. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 5: Protein hücre altı lokalizasyonunu belirlemek için organel belirteçlerinin kullanılması. GFP-Sft1'i Sec2-DuDre (geç Golgi / erken endozom belirteci) veya Anp1-mCherry (erken Golgi belirteci) ile birlikte eksprese eden maya hücrelerinin temsili dilimleri. Sft1, her iki organel markörü ile kısmi kolokalizasyon gösterdi. Beyaz oklar, kolokalizasyon insidansı; kırmızı oklar, kolokalizasyon insidansı yok. Ölçek çubuğu: 2 μm. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Ek Şekil 1: VisiView yazılımında görüntüleme parametrelerinin seçimini gösteren ekran görüntüleri. (A) Uyarma ışığı yoğunluğunun ayarlanması. Başlangıç noktası olarak, tüm kanallar için %100'ü ayarlayın. Uyarma parametrelerinin ayarlanmasında göz önünde bulundurulması gereken genel hususlar için metne bakın. (B) Görüntülenecek ışık kanallarının seçilmesi. Bu örnek, iki floresan kanalının seçimini gösterdi: GFP ve mCherry. VisiView'da her kanalın bir açılır menüden seçilmesi gerekir. Diğer birçok yazılım uygulamasında, tüm kanallar elektronik tablo benzeri tek bir grafik arayüzde yapılandırılabilir. Bu özel yazılımda, her kanal için benzersiz pozlama süresine izin vermek için işaretlenmemesi gereken Tüm Dalga Boyları için Aynı Pozlama / Kazanç onay kutusu olduğunu unutmayın. (C) Dilim sayısını ayarlama ve z-yığınlarını toplamak için adımlama. Her yönde 3,5 μm, 0,5 μm'lik adımlarla 7 μm derinliği kapsar. (D) Çok kanallı görüntülemede kanal değiştirme ve z-stack ilerlemesinin sırasını seçmek. Çoğu mikroskop kontrol yazılımında, bir sonraki dilime geçmeden önce tüm kanalları z konumunda bitirmeyi veya bir sonraki kanala geçmeden önce her kanalda tam bir z yığınını bitirmeyi seçebilirsiniz. Bu seçim, hem görüntü alımındaki zaman tüketimini hem de hareketli nesneler için kolokalizasyonun doğruluğunu etkiler. (E) Hızlandırılmış görüntüleme için zaman noktalarının ve aralıkların sayısını ayarlama. Bu dosyayı indirmek için lütfen buraya tıklayın.
Tablo 1: Organel işaretleyici plazmitlerinin listesi. Bu Tabloyu indirmek için lütfen buraya tıklayın.
Tartışmalar
Burada açıklanan protokol, görüntüleme mayası organellerini keşfetmek için diğer araştırma alanlarından giren kişiler için basit bir başlangıç sağlar. Belli konulara geçmeden önce, görüntüleme yazılımlarında otomatik özelliklerin aşırı kullanımından kaçınılması gerektiğini bir kez daha vurgulamak isteriz. Mikroskopi görüntüleri sadece güzel resimler değil, aynı zamanda bilimsel verilerdir ve bu nedenle elde edilmeleri ve yorumlanmaları buna göre ele alınmalıdır. Görüntü toplama parametrelerinin dikkatli bir şekilde seçilmesi ve 16 bit ve 8 bit görüntü kavramlarının aşina olması özellikle önemlidir.
Bu protokol dört farklı maya ortamını listeler: SMD, SMD + CA, YPD ve SD-N. SMD, "sentetik minimal dekstroz" anlamına gelir. SMD'nin avantajı, düşük otofloresan üretmesidir. Bu nedenle, SMD floresan görüntüleme için tercih edilen ortamdır. SMD + CA, artık tamamen sentetik olmadığı sınırlaması ile SMD'den daha zengin beslenme sağlayan kasamino asitler içerir. Besin açısından zengin olan YPD, maya hücrelerinin kültürlenmesi için genel bir ortam olarak iyidir, ancak getirdiği yüksek otofloresan nedeniyle görüntüleme için daha az idealdir. SD-N, otofajiyi ve lipit damlacıklarının çoğalmasını indüklemek için kullanılabilen bir nitrojen açlığı ortamıdır. SD-N ayrıca sentetiktir ve düşük otofloresan üretir.
Bu protokolde kullanılan SMD tarifindeki amino asit ve nükleobaz takviyesinin birçok yaygın laboratuvar türü için uygun olduğunu, ancak hepsi için uygun olmadığını unutmayın. SMD'nin, kapsamı suş genotipine bağlı olan temel besinleri sağlaması gerekir. Tamamlanan bir oksotrofik gen aleli olan transformatörler için seçim yapılırken, bırakma ortamını oluşturmak için karşılık gelen besin bileşeni ihmal edilir (yani, Ura+ hücreleri için seçim yapmak üzere urasil dışarıda bırakılır). Daha fazla bilgi için lütfen bu konudaki diğer yayınlara bakın14. Ayrıca, ortam sterilizasyonu için amino asit ve nükleobaz takviyelerinin otoklavlanmaması gerektiğini unutmayın. Filtrelenmesi gerekiyor. Glikoz uzun süre otoklavlandığında kararır, bu da kaçınılmalıdır.
Organel dinamiklerini ve hücre içi protein trafiğini incelerken, floresan protein yapılarının aşırı eksprese edilmesinden kaçınmak önemlidir. Aşırı ekspresyon, protein kompleksi stokiyometrisini bozma ve araştırılan orijinal hücre altı dağılım modelini gizleyebilecek veya ortadan kaldırabilecek beklenmedik işlev kazanımı sonuçları ortaya çıkarma eğilimindedir.
Bu düşünceyi göz önünde bulundurarak, endojen seviyelerinde eksprese edilen proteinleri gözlemlemek için iyi bir ters çevrilmiş geniş alan floresan mikroskobu kullanmanızı öneririz. Tavandaki tüm aydınlatmalar kapatılarak çalışma ortamının loş tutulması gerekir. Nokta taramalı lazer konfokal mikroskopları kullanmaktan kaçının. Yazma sırasında, çoğu nokta taramalı lazer konfokal mikroskop, düşük floresan sinyallerini tespit etmede yüksek kaliteli geniş alanlı floresan mikroskopları kadar hassas değildir. Mevcut geniş alanlı floresan mikroskopları arasından, iyi bir CMOS kameraya ve yüksek sayısal açıklık objektifine sahip birini seçin. Yüksek büyütmeli objektifler (100x gibi), esas olarak maya hücrelerinin küçük olması nedeniyle tercih edilir. İdeal olarak, kameranın piksel aralığı, tercih edilen hedefle eşleştirildiğinde Nyquist örnekleme frekansını karşılamalıdır. Mikroskop bunun yerine son derece düşük sinyallerin tespiti için daha büyük piksel aralığına sahip bir kamera ile donatılmışsa, hesaplamalı işleme ile uzamsal detay kurtarma denemeye değer15. Bu mikroskopi prosedüründeki sarf malzemeleri doğası gereği jeneriktir. Olası bir sorun, bazı tedarikçilerin kapak camının yüksek standartta temizlenmemesidir, bu nedenle numune gözlemini engelleyen floresan üretebilir. Farklı bir tedarikçiye geçmek basit bir çözümdür.
Görüntüleme için maya hücrelerinin sağlıklı bir durumda tutulması esastır. Agar plakalarındaki maya, 4 °C'de aylarca hayatta kalabilir. Bununla birlikte, sıvı bir kültürü aşılamak için eski bir plaka kullanılırsa, görüş alanında sıklıkla çirkin yaşlanan hücreler görünecektir. Bu, yeni çizgili bir plaka kullanılarak önlenir.
Bir kimeranın floresan sinyali tespit edilemiyorsa, immünoblotlama ile ifadesini kontrol edin. Yapı ifade edilirse ve moleküler ağırlık makul görünüyorsa, büyük olasılıkla bir donanım yapılandırma sorunudur. Bir mikroskop uzmanına danışın. Herhangi bir ifade algılanmazsa, sorun büyük olasılıkla gerinim yapısındaki bir hatadır. Plazmidin doğru olduğunu ve amaçlanan şekilde kullanıldığını doğrulayın (yani, enzim sindirilmiş veya PCR amplifiye edilmiş).
Maya hücreleri mikroskobik gözlem için cam yüzeylere tutturulduğunda, sıvı bir ortam mevcut olsa ve sıcaklık korunsa bile, ortam, çalkalama ile bir test tüpündekiyle aynı değildir. Havaya erişim sınırlıdır ve mekanik ipucu da farklıdır. Bu farklılıkların araştırılan biyolojik süreci değiştirme olasılığı vardır16. Bu tür nadir durumlarda, belirli bir süreci devam ettirmek için daha ustaca yollar bulmak gerekir.
Teşekkürler
Yazarlar, el yazması hazırlamadaki cömert yardımları için Xie laboratuvarı üyelerine teşekkür eder. Bu çalışma, Çin Ulusal Doğa Bilimleri Vakfı (hibe 91957104), Şanghay Belediye Eğitim Komisyonu (hibe 2017-01-07-00-02-E00035) ve Şanghay Belediye Bilim ve Teknoloji Komisyonu (hibe 22ZR1433800) tarafından desteklenmiştir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Adenine | Sangon Biotech | A600013 | |
| Casaminoacid | Sangon Biotech | A603060 | |
| Concanavalin A from canavalia ensiformis (Jack bean) | Sigma Aldrich | L7647 | |
| D-Glucose | Sangon Biotech | A501991 | |
| Fiji | https://fiji.sc/ | ||
| Glass-bottom petri dish | NEST | 706001 | Φ35 mm |
| ImajeJ | https://imagej.net/ | ||
| Inverted florescence microscope | Olympus | IX83 equipped with UPLXAPO 100X oil immersion objective, Lumencor Spectra X light source, and Hamamatsu Orca Flash4.0 LT camera. | |
| L-Histidine | Sangon Biotech | A604351 | |
| L-Leucine | Sangon Biotech | A100811 | |
| L-Lysine | Sangon Biotech | A602759 | |
| L-Methionine | Sangon Biotech | A100801 | |
| L-Tryptophan | Sangon Biotech | A601911 | |
| Microscope cover glass | CITOTEST | 10222222C | 22 mm x 22 mm, 0.16–0.19 mm |
| Microscope slides | CITOTEST | 1A5101 | 25 mm x 75 mm, 1–1.2 mm |
| Peptone | Sangon Biotech | A505247 | |
| Uracil | Sangon Biotech | A610564 | |
| Visiview | Visitron System GmbH | https://www.visitron.de/products/visiviewr-software.html | |
| Yeast extract | Sangon Biotech | A100850 | |
| Yeast nitrogen base without amino acids | Sangon Biotech | A610507 | |
| YNB without amino acids and ammonium sulfate | Sangon Biotech | A600505 |
Referanslar
- Levine, B., Klionsky, D. J. Autophagy wins the 2016 Nobel prize in physiology or medicine: Breakthroughs in baker's yeast fuel advances in biomedical research. Proceedings of the National Academy of Sciences of the United States of America. 114 (2), 201-205 (2017).
- Spang, A. Anniversary of the discovery of sec mutants by Novick and Schekman. Molecular Biology of the Cell. 26 (10), 1783-1785 (2015).
- Walter, T., Erdmann, R. Current advances in protein import into peroxisomes. The Protein Journal. 38 (3), 351-362 (2019).
- Farre, J. C., Subramani, S. Mechanistic insights into selective autophagy pathways: lessons from yeast. Nature Reviews. Molecular Cell Biology. 17 (9), 537-552 (2016).
- West, M., Zurek, N., Hoenger, A., Voeltz, G. K. A 3D analysis of yeast ER structure reveals how ER domains are organized by membrane curvature. Journal of Cell Biology. 193 (2), 333-346 (2011).
- Day, K. J., Casler, J. C., Glick, B. S. Budding yeast has a minimal endomembrane system. Developmental Cell. 44 (1), 56-72 (2018).
- Zhu, J., et al. A validated set of fluorescent-protein-based markers for major organelles in yeast (Saccharomyces cerevisiae). mBio. 10 (5), 19(2019).
- Li, D., et al. A fluorescent tool set for yeast Atg proteins. Autophagy. 11 (6), 954-960 (2015).
- Gietz, R. D., Woods, R. A. Yeast transformation by the LiAc/SS Carrier DNA/PEG method. Methods in Molecular Biology. 313, Clifton, N.J. 107-120 (2006).
- Caloca, B., et al. Comparison of concanavalin A and poly-l-lysine as cell adhesives for routine yeast microscopy applications. Yeast. , (2021).
- Rueden, C. T., et al. ImageJ2: ImageJ for the next generation of scientific image data. BMC Bioinformatics. 18 (1), 529(2017).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Banfield, D. K., Lewis, M. J., Pelham, H. R. B. A SNARE-like protein required for traffic through the Golgi complex. Nature (London). 375 (6534), 806-809 (1995).
- Curran, B. P., Bugeja, V. Basic investigations in Saccharomyces cerevisiae. Methods in Molecular Biology. 1163, Clifton, N.J. 1-14 (2014).
- Zhao, W., et al. Sparse deconvolution improves the resolution of live-cell super-resolution fluorescence microscopy. Nature Biotechnology. , (2021).
- He, C. W., et al. Membrane recruitment of Atg8 by Hfl1 facilitates turnover of vacuolar membrane proteins in yeast cells approaching stationary phase. BMC Biology. 19 (1), 117(2021).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır