Method Article
Yüzlerce C. albicans Hücresinde Biyo-Atomik Kuvvet Mikroskop Ölçümlerinin Otomasyonu
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
Bu protokol, yüzlerce mikrobiyal hücre üzerinde AFM ölçümlerini otomatikleştirmeyi amaçlamaktadır. İlk olarak, mikroplar PDMS damga mikroyaplarına hareketsiz hale getirilir ve daha sonra yüzlerce hareketsiz hücre üzerinde kuvvet spektroskopisi ölçümleri otomatik olarak yapılır.
Özet
Bu makalede sunulan yöntem, Bio-AFM deneylerini ve kuvvet eğrilerinin kaydedilini otomatikleştirmeyi amaçlamaktadır. Bu yöntemi kullanarak, kuvvetleri 1000 hücreye otomatik olarak 4 saatte kaydetmek mümkündür. 4 saatlik analiz süresini korumak için hücre başına kuvvet eğrisi sayısı 9 veya 16'ya düşürülür. Yöntem, Jython tabanlı bir programı ve tanımlanmış desenler üzerinde hücreleri birleştirmek için bir stratejiyi birleştirir. Ticari bir Bio-AFM üzerinde uygulanan program, ucu dizinin ilk hücresine ortalayabilir ve ardından her hücrede kuvvet eğrileri kaydederken otomatik olarak hücreden hücreye hareket edebilir. Bu metodolojiyi kullanarak, hücrelerin sertlikleri, yapışkan özellikleri vb. Otomasyon ve analiz edilen çok sayıda hücre ile hücre popülasyonunun davranışına erişilebilir. Bu, biyo-AFM alanında şimdiye kadar verilerin sadece birkaç on hücreye kaydedildiği bir atılımdır.
Giriş
Bu çalışma, atomik kuvvet mikroskobu (AFM) kullanarak yüzlerce canlı hücre üzerinde otomatik kuvvet ölçümleri yapmak için bir metodoloji sağlar. Ayrıca, sıvı bir ortamda yapılan AFM deneyleriyle uyumlu bir PDMS mikro yapılandırılmış damga üzerindeki mikropları hareketsiz hale getirmek için bir yöntem sağlar.
Bio-AFM, biyolojideki uygulamalar için tasarlanmış ve daha sonra canlı hücreleri incelemek için kullanılan son derece uzmanlaşmış bir teknolojidir. O anda bir hücreyi analiz edebilecek eğitimli bir mühendise ihtiyaç var. Bu koşullarda, analiz edilebilen farklı hücrelerin sayısı oldukça küçüktür, tipik 5 ila 10 hücre 4-5 saat içinde. Bununla birlikte, tek bir hücrede kaydedilen kuvvet ölçümlerinin miktarı genellikle çok yüksektir ve kolayca 1000'e ulaşabilir. Bu nedenle, AFM kuvvet ölçümlerinin canlı hücreler üzerindeki mevcut paradigması, yüzlerce kuvvet eğrisini (FC) ancak sınırlı sayıda hücre üzerinde kaydetmektir.
İstatistiksel olarak, bu yaklaşım şüphelidir ve örneğin temsili konusunu gündeme getirmektedir. Aslında, örneğin, bu birkaç hücreye yüzlerce ölçüm kaydedilse bile, sadece birkaç hücreyi ölçerek bir hücre popülasyonunun heterojenliğini değerlendirmek zordur. Bununla birlikte, bu paradigma temelinde biyofizik, mikrobiyoloji ve nanotıp 1 ,2,3'te büyük ilerlemeler yapılmıştır. Gerçekten de, tek hücre ölçeğinde nanometre analizi hücresel nanomekanik, transmembran proteinlerinin organizasyonu veya antimikrobiyal veya antikanser ilaçların etki mekanizması hakkında yeni bilgiler sağlamıştır4,5,6,7. Bununla birlikte, son zamanlarda, hücreler üzerinde yapılan birkaç yüksek verimli biyomekanik test ortaya çıkmıştır8Bilimsel topluluğun bu paradigmayı değiştirmeye ve hücre popülasyonu heterojenliğine erişmeye olan ilgisini gösteren. Bu testlerin tümü, hücreleri deforme etmek ve genel yüzey elastikiyetlerinin dolaylı bir ölçüsünü elde etmek için stres altındaki deformasyonlarını optik olarak ölçmek için mikroakışkan sistemlere dayanır8. Bununla birlikte, bu yöntemlerle ilgili önemli bir sorun mono-parametrik olmalarıdır: sadece hücre esnekliği araştırılabilir. Ayrıca, örneğin memeli hücrelerinin veya biyofilmlerin sirkülasyonsuz çalışmaları için sınırlayıcı olabilen yapışık hücrelerin mekanik parametrelerinin ölçülmesine izin vermezler.
AFM'yi içeren yaklaşımlar S. Scheuring9 ve M. Favre10ekipleri tarafından geliştirilmiştir. Scheuring ve ark. fibronektin desenleri üzerinde hareketsiz hücreler9, tek tek hücreleri desen şeklini almaya zorlamak9. Daha sonra bu ekip, ortalama verileri tanımlamak için birkaç hücrenin mekanik özelliklerini haritaladı, 14 ila 18 hücreyi temsil etti. AfM cantilevers10paralelleştirerek ölçümleri çoklamayı amaçlayan Farve ve ark. Bilgimize göre, çoklama yönünde yapılan bu çalışma canlı hücreler üzerinde ölçümlere yol etmemektedir.
Dujardin'in ekibi tarafından önerilen ilginç bir yaklaşım, hücreleri tanımlayabilen ve özel yapım kuyuların dibinde görüntüleyebilen otomatik bir AFM sunar. Bu yöntem büyük bir hücre popülasyonunun analizine izin vermese de, her kuyuda farklı koşulların otomatik olarak testine izin verir11.
Ortalama bir hücreye değil, aksine hücreler arasındaki heterojenliğe erişmek için en az 1000 hücreyi ölçmek istediğimiz için bu çalışmadaki hedefimiz daha iddialı. Burada AFM kullanarak hücre popülasyon heterojenliğine erişmek için geliştirdiğimiz strateji, sınırlı sayıda kuvvet eğrisinin kaydedildiği yüzlerce hücrenin analizine dayanmaktadır. Sınırlı sayıda hücre üzerinde çok sayıda kuvvet eğrisi kaydetmenin "klasik" yaklaşımı ile karşılaştırıldığında, aynı bilgileri sağlamadığı için bu yaklaşım tamamlayıcı olarak düşünülmelidir. Gerçekten de, tipik yöntem, yaklaşımımızı kullanarak, bireysel hücre yüzeyi heterojenliğini araştırmamıza izin verirken, tüm hücre popülasyonu heterojenliğine erişebiliyoruz. Bu amaca ulaşmak için, mikropları (burada maya türü Candida albicans)doğrudan bir PDMS mikro yapılandırılmış damga12kuyularına indiren ve AFM ucunu otomatik olarak hücreden hücre13'e taşımak ve her hücrenin mekanik özelliklerini ölçmek için orijinal bir program geliştiren bir yöntem geliştirdik.
Protokol
1. Mikrobiyal hücre kültürü
- Gliserol stoğundan hücreleri canlandır.
NOT: C. albicans gliserol stoklarında, mermerlerde -80 °C'de saklanır.- -80 °C stokta bir mermer seçin ve maya pepton dekstroz (YPD) agar üzerine sürün. Sıvı ekimden önce hücreleri 30 ° C'de 2 gün boyunca yetiştirin.
- Sıvı kültürleri hazırlayın.
- Bir kültür tüpünü 5 mL steril YPD suyu ile doldurun ve YPD agar plakasında yetişen tek bir C. albicans hücresi kolonisi ekleyin.
- Santrifüjleme ile hasat etmeden önce 20 saat boyunca 30 °C'de statik koşullarda kültürü büyütün (4000 x g, 5 dk). Süpernatantı atın ve biyolojik tehlike atığı olarak ortadan kaldırın.
- Peletleri 10 mL asetat tamponu (8 mM sodyum asetat, 1 mM CaCl2, 1 mM MnCl 2 , pH5.2) ile 2xyıkayın. Yıkamalar arasında santrifüj (4000 x g, 5 dk).
- Peletin 2 mL asetat arabelleğine yeniden kullanın ve PDMS damgasında hücre hareketsizleştirme için bu çözümü kullanın.
NOT: Bu süspansiyon saklanamaz ve bölüm 3 için taze olarak hazırlanmalıdır.
2. PDMS damga hazırlama
- Silikon ana kalıp hazırlama
- Bilgisayar destekli tasarım (CAD) yazılımını kullanarak istediğiniz mikro yapıları çizin.
NOT: Tasarlanan kuyular, tuzaklanacak mikropla benzer boyutta olmalıdır. Tasarım, listede 100 x 100 kuyuların büyük bir matrisini sağlamalıdır. Mikrobun ortalama boyutu etrafında biraz farklı boyutlarda birkaç dizi yapmak en iyisidir. - Temiz bir oda varsa, daha önce yayımlanan protokol 12'nin 2 ile12arasında olan adımlarını izleyin. Aksi takdirde, silikon ana kalıp ticari temiz oda tesislerinden edinilebilir.
- Bilgisayar destekli tasarım (CAD) yazılımını kullanarak istediğiniz mikro yapıları çizin.
- PDMS damga kalıplama
- PDMS oligomerleri ve kürleme maddesinin (Malzeme Masası) 10 ila 1, kütle oranı karışımını içeren 55 g PDMS prepolimerçözeltisi hazırlayın.
- Bu çözeltiyi vakum altında (10-1 -10-2 bar aralığında) pdms çözeltisinden (5−10 dk) tüm sıkışmış kabarcıklar çıkarılana kadar karıştırın ve gazdan arındırın.
- Gazdan arındırılmış çözeltinin 20 g'ını silikon ana kalıbına dökün ve tekrar gaz çözün (10-1 -10-2 bar aralığında).
NOT: Damga kalınlığı yaklaşık 2−3 mm olmalıdır. - Tüm kabarcıklar çıkarıldığında, PDMS'yi 1 saat boyunca 80 °C'de yeniden ifade edin.
- PDMS mikroyapılı damgayı görünür mikroyapı dizilerine paralel bir yönde neşterle(0,5 x 1,5 cm 2)kesin.
- Damgayı silikon ana kalıbından soyun.
- Üst tarafındaki mikro yapıları sergilemek için damgayı iade edin ve cam bir kaydırağa yatırın. Mikroyapıların cam kaydıraktan uzağa dönük olduğundan emin olun. Damgada görülebilen mikro yapıları, daha sonra AFM otomasyon prosedürü için referans görevi görecek olan cam slaydın yanıyla hizalayın.
NOT: Bu aşamada, PDMS damgası hücre hareketsiz hale getirilmesi için hazırdır. PDMS pulları silikon ana kalıpta birkaç ay saklanabilir. Tüm PDMS ana kalıptan çıkarıldığında, ana kalıba tekrar yeni bir PDMS damgası atılabilir (ana kalıbı güvende tutmak için poliüretanda çoğaltmak mümkündür)14.
3. Örnek hazırlama
- Hücre hareketsizleştirme
- Tamponu hücrelerden ayırmak için yeniden sulanan hücre çözeltisinin santrifüjü (500 x g, 5 dk) 600 μL.
- 3.1.1 adımından PDMS damgasına kadar süpernatantın pipet 200 μL'si ve vakum altında (10-1 -10-2 bar aralığında) yaklaşık 40 dakika boyunca gaz.
NOT: Bu adım kuyuların içindeki hücre hareketsizleştirmesini iyileştirmek için önemlidir. Üst tarafta bulunan maya hücresi duvarından moleküller, muhtemelen bu ön ıslatma adımı sırasında PDMS yüzeyinde birikir. Bu moleküller, büyük olasılıkla, hücrelerin yapışmasını arttırır ve damga dolum oranındaki artışa katkıda bulunur. - 40 dakika sonra, bir pipetle, tamponu PDMS yüzeyinden çıkarın ve bir pipetle, oda sıcaklığında 15 dakika boyunca 1.2.4 adımdan 200 μL hücre çözeltisi biriktirin.
- Hücreleri konvektif/kılcal montaj ile damganın mikroyapılarına yerleştirin. Bunun için, 30 ila 50 ° arasında bir açıya sahip her iki yönde de bir cam slayt kullanarak 200 μL hücre süspansiyonunu damgaya manuel olarak yayın. Yüksek dolum oranı elde etmek için cam kaydırağı pulda birkaç kez geçirmek gerekebilir.
NOT: Bu yöntemin tam açıklaması mevcuttur13. - Hücre süspansiyonu pipetle çıkarın. Damgayı 3x 1 mL asetat tamponu ile yıkayın, pH 5.2 sıkışmamış hücreleri çıkarmak için.
- Damganın kuru Petri kabına yapışmasını sağlamak için damganın arkasını azot akışı kullanarak kurutun.
- Son olarak, hücrelerle dolu PDMS damgasını bir Petrikabına (Malzeme Masası) yatırınve hücreleri sıvı ortamda tutmak için 2 mL asetat tamponu ile doldurun.
- AFM sahnesine damgayı ayarlama
- AFM işlemlerine başlarken sahneyi 0:0'da ortala.
- Unsay ve ark.15'te açıklandığı gibi, kantilatıcının cam üzerindeki ve sudaki hassasiyetini ve yay sabitini kalibre edin.
- Petri kabını damga ile birlikte alın ve AFM Petri kabı tutucusuna yerleştirin.
- Damga kenarını Petri kabı tutucu Y eksenine dik olarak hizalayın.
NOT: Şekil 1'degösterildiği gibi kabul edilebilir bir eğim açısı 5°'nin altındadır. - AFM kafasını sahneye yerleştirin ve step motorlarının ucun damgaya çarpmasını önlemek için yeterince uzatıldıklarına dikkat edin.
4. AFM programını çalıştırma
NOT: AFM programı Ek Malzeme olarak sağlanmıştır (AutomatipSoftware2019.pdf). Motorlu bir sahne ve JPK masaüstü yazılım sürümü 4.3 ile donatılmış bir JPK-Bruker AFM Nanowizard II veya III gerektirir. Program Jython altında geliştirilmiştir (python 2.7 tabanlı sürüm)
- Veri toplama
- AFM ucunu, AFM optik mikroskobu kullanarak 4,5 x 4,5 μm2 kuyunun (hücre boyutuna karşılık gelen) sol köşesinin üzerine ortalayın. Başka bir kuyu boyutu gerekiyorsa, istediğiniz kuyuların sol üst köşesine ortalanın.
- 100 x 100 μm² alan üzerinde 64 x 64 kuvvet haritası (Z aralığı =4μm, uç hızı = 90 μm·s -1 , uygulanan kuvvet 3 ila 5 nN) gerçekleştirin. Ölçüm modu açılan kutusundan Eşlemeyi Zorla modunu seçin. Kuvvet denetimi eşleme paneline aşağıdaki parametreler girilir: Rel. Setpoint = 3 ila 5 nN; z uzunluğu 4 μm; Z hareketi: sabit süre; uzatma süresi: 0.01s; ek gecikme:0; Retr gecikmesi: 0, Gecikme modu: Sabit Kuvvet, Örnek hızı 2048 Hz; Z kapalı döngü işaretini kaldırın; Izgara: Kare görüntüyü kontrol edin, Hızlı 100 μm, yavaş: 100μm, X ofset: 0 μm; Y ofset: 0 μm; ızgara açısı: 0 derece; Piksel: 64x64; piksel oranı: 1:1
NOT: Tipik bir sonuç Şekil 2'de gösterilmiştir. Bu görüntü, iki kuyu arasındaki perdeyi ölçmeye ve doğrulamaya yardımcı olacaktır. - Sol üst kuyunun (W1) ve sol alt kuyunun (Şekil 2'deW2 olarak adlandırılır) merkezinin koordinatlarına dikkat edin. Bunu yapmak için kuyunun etrafına kare bir kutu yapın. Kutunun merkezinin koordinatı AFM yazılımının sol panelinde x,y koordinat kutularında görünür.
- Otomasyon yazılımını açmak için (Automatip_scan.py): JPK masaüstü yazılımında üst çubuk menüsünden ileriye tıklayın ve komut dosyasını açın'ı seçin. Açılan pencerede, Tamamlayıcı Veriler (Automatip_scan.py) içinde sağlanan komut dosyasına giden yolu seçin.
- Jython komut dosyasının Girişler kutusu bölümünde W1 ve W2 koordinat değerlerini uygulayın (Şekil 3). Komut dosyasının P1 değişken satırı 239'daki W1 koordinatlarını ve P2 değişken satırı 241'deki W2 koordinatlarını girin.
NOT: İlk koordinatlar (W1 ve W2) olarak seçilen kuyular tarama alanı kenarından çok yakın olmamalıdır. Aksi takdirde, merkezi algoritma, kuyunun her iki tarafındaki PDMS yüzeyindeki yüksekliği ölçmesi gerektiğinden doğru çalışmaz. Örneğin, bkz. - Pitch değerini komut dosyasının perde değişken satırı 245'e bağlayın.
- Kuyu boyutunu Ws değişken satırı 248'e girin. Bu kuyu desenlerinin tasarımından bilinir ve perdeyi doğrulamak için kullanılanla aynı görüntüde kontrol edilebilir (Şekil 2).
- Verileri istenen yere kaydetmek için kaydetme dizininin yolunu satır 251'e yazın.
- TotalArea değişken satırı 254'i 100 μm'lik birden fazla "n" isteğine ayarlayın (bu, kullanılan AFM'nin maksimum tarama alanıdır). Araştırılacak toplam kuyu sayısı bu değer ve perde kullanılarak hesaplanabilir: maksimum tarama alanı / perde*n2.
NOT: Şekil 3örneğinde, 100 x 100 μm2'nin 9 alanı analiz edilecektir. - NumScans değişken satırı 257'de kuyu başına kaydedilen kuvvet eğrileri matrisini, satır ve sütunu (3, 3 veya 4, 4) ayarlayın.
NOT: Şekil 3örneğinde, her kuyu için 3 x 3 = 9 VC matrisi kaydedilecektir. - Programı çalıştırın. Başlat düğmesine tıklayın.
NOT: Program, W1 ve W2 kuyularının merkezini daha iyi belirlemek için önce otomatik olarak bir merkezleme algoritması yürütür (adım 1). Daha sonra ilk tarama alanının her kuyusunda (adım 2) Kuvvet Eğrileri (IC) matrisini otomatik olarak alır. Bu alanın tüm kuyuları araştırıldığında, komut dosyası AFM ucunu otomatik olarak bir sonraki tarama alanının ilk kuyusuna taşır. Uç geri çekilir, mikroskop aşaması bir sonraki alana taşınır, uç damgaya tekrar yaklaşıldı ve merkezleme algoritması, o alanın ilk kuyusunda (1') otomatik olarak yeniden ortalamak için tekrar yürütülür (adım 3). İlk alan kullanıcı tarafından tanımlanır, ikincisi, n'ye ulaşılana kadar sağda vb. n+1 alanı n'nin altında, n+1'in solunda n+2 vb. 2n+1 2n'nin altında, 2n+2 ise 2nn vb. Küresel olarak, uç toplam alandan geçer. Tarama alanlarının toplam sayısı "n 2" araştırılana kadaradım 2ve 3 otomatik olarak tekrarlanır. Şekil 5 programın akış çizelgesini sunar. Programın tamamlanması ~4 saat sürer.
- Veri analizi
- FCS dosyalarını tek bir klasörde düzenlemek için "Dosyaları kopyala" python komut dosyasını(Copy_files_L.py, Tamamlayıcı Verilerdesağlanır) yürütün. Bu komut dosyası Python 2.7 ve SciPy modülü ile geliştirilmiştir. Python komut dosyasını açmak için Visual Studio Code yazılımını kullanın. Genel klasörün giriş yolu (ek verilerde sağlanan komut dosyasının satır 67'si) ve depolanacağı yer (satır 73).
- Kuvvet eğrilerini analiz etmek için AFM üreticisi veri işleme yazılımını açın. Üst menüde Dosya,spektroskopi eğrileri grubuaç 'ı seçin.
- Toplu işleme penceresinde, Tamamlayıcı Veriler 'de (StiffnessProcess.jpk-proc-force) sağlanan işlemi seçin. İşlemin son adımını seçin ve Tüme Sakla ve Uygula 'yıtıklatın. Tüm kuvvet eğrileri aynı tedaviyi görecektir.
NOT: İşlem, sapma eğrilerini Newton'da kalibre edilmiş kuvvet eğrilerine dönüştürmek için FCS dosyalarından kalibrasyonu kullanır; veri yumuşatma algoritması uygulanır (ortalama 3 ardışık nokta); taban çizgisi sıfır ekseninde duracak şekilde çevrilir; temas noktası tahmin edilir ve FC, temas noktasını koordinatlara yerleştirmek için dengelenir (0,0); yaverin bükülmesi FCS'ye çıkarılır, geri çekme eğimi takılır. Veri işlemenin sonunda, yazılım her GC için bir tablo içeren bir dosya oluşturur: adı, Genç Modül, temas noktası, yapıştırma kuvveti, eğimler, vb. - Tüm denemeler için 4.2.1 ile 4.2.3 adımlarını yineleyin. Verileri farklı klasörlere kaydetmeye dikkat edin (örneğin: "...\TREATED\" ve "...\UNTREATED\")
- Histogramları ve kutu çizimlerini çizmek ve ANOVA istatistiksel tedavileri gerçekleştirmek için Tamamlayıcı Veriler'de sağlanan R komut dosyasını kullanın.
- R komut dosyasını (DataAnalisys.R) açmak için R studio yazılımını kullanın ve veri işleme yazılımı (.tsv) ile ayıklanan bilgileri içeren dosyaları yükleyin.
- Ortam penceresinde Veri Kümesini İçeri Aktar düğmesini kullanın, görüntülenen listeden metinden (readr) seçin ve yeni pencerede Tarayıcı düğmesini seçin ve .tsv dosyasını bulun.
- Dosya yüklendikten sonra, analiz için eklenecek sütunları (sertlik ve yapışkanlık) seçin. Tüm kodu çalıştırmak için Ctrl+Alt+R tuşlarınabasın.
NOT: Komut dosyası 4 veri kümesiyle çalışır, hem işlenmemiş hem de tedavi edilmiş hücrelere sahip iki denemeyi göz önünde bulundurun. Komut dosyasının bloklarını yürütmek ve değişkenlerin yürütülen işlevlere göre nasıl değiştiğini görmek mümkündür.
Sonuçlar
Açıklanan protokolü, caspofungin'in maya formundaki fırsatçı insan patojeni C. albicans'ın biyofiziksel özellikleri üzerindeki etkisini analiz etmek için kullandık. Caspofungin, hücrelerin antifungallere doğru geliştirdiği direnç mekanizmaları nedeniyle diğer ilaçlar etkisiz olduğunda kullanılan son şans antifungal molekülüdür. Etki mekanizması, ß glukan sentezi sorumlu karmaşık fks1/Fks2 alt birimi Fks2'nin inhibisyonunu temel almaktadır. ß glukanlar mantar hücre duvarının önemli bir bileşeni olduğundan16,17, hücre duvarının biyofiziksel özelliklerinin değiştirilmesini bekliyorduk: sertlik ve yapışma.
Şekil 6, yukarıda sunulan tüm protokol uygulandığında elde edilen tipik histogramları sunar. Kırmızı histogram, 957 yerel hücrede kaydedilen sertlik repartisyonunu ve 574 caspofungin tedavi edilmiş hücredeki mavi olanı temsil eder. İlk ilginç gözlem, her iki histogramın da değerlerin bimodal dağılımını gösterdiğidir. Bu gözlem sadece yüzlerce hücreyi ölçtüğümiz için mümkündür. Daha küçük örneklerde, araştırmacılar genellikle tek bir dağılım gözlemler ve popülasyon heterojenliğini kaçırırlar. 17,18
İkinci gözlem caspofungin etkisi ile ilgilidir. Hala iki alt nüfus varken küresel olarak hücrelerin sertliğini azaltır. Son adımda, önerilen protokol, Şekil 7'desunulan yerel ve işlenmiş hücrelerin ANOVA karşılaştırmasını sağlar. İki koşulun farklı bir sertliğe sahip olduğunu ve bu farkın son derece önemli olduğunu gösterir (p değeri < 0.001). Analiz edilen çok sayıda hücre sayesinde bu değere ulaşılır ve elde edilen sonuçlara daha fazla güven sağlar.
Otomatik olarak kaydedilen verilerden de yapıştırma çıkarılmıştır ve çıplak uç ile yerli hücreler arasındaki yapıştırma kuvvetinin 0,64 ± 0,6 nN olduğunu tespit ettik. Bu durumda ayrıca iki alt nüfus bulundu: birincisi 0.7 ± 1.4 nN ortalama yapıştırma kuvvetine sahipken, ikincisi 4.5 ± 1.5 nN. Caspofungin ile tedavinin yapışma üzerinde öngörülemeyen etkileri vardı. Bir deneyde hiçbir etki gözlenmedi, ancak başka bir deneyde, caspofungin uçtaki yapışmada bir azalmaya ve popülasyon yapışma heterojenliğinin azalmasına neden oldu. Bu sonuçlar Proa ve ark.13,totalite olarak sunuldukları yerden çıkarılır.

Şekil 1: Mikro yapılandırılmış damganın AFM aşamasındaki kabul edilebilir konumu.
Sol resimlerdeki eğim açısı (5°'ye kadar) program tarafından işlenebilir, ancak sağdaki eğim önemlidir (10°). Bu rakam13'ten değiştirilmiştir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
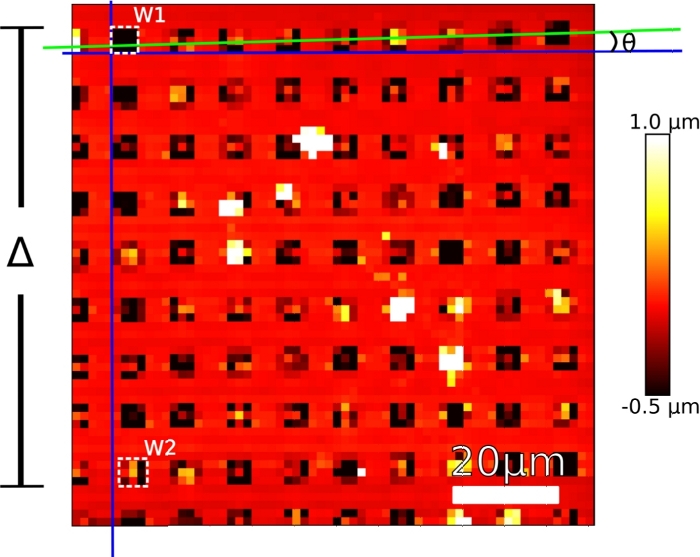
Şekil 2: İlk koordinatları W1 ve W2 olarak gösteren doldurulmuş pdms damgalarının tipik AFM görüntüsü, tarama alanının boyutu (Δ2),eğim açısı (φ).
Bu Şekil13'ten değiştirilmiştir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 3: Komut dosyasının kullanıcı giriş bölümü.
P1 ve P2, Şekil 2'nin kuyu 1 (W1) ve kuyu 2 (W2) koordinatlarını ifade eder. Diğer parametreler μm'deki perde, μm'deki kuyu boyutu (Ws), verileri kaydetmek için dizin yolu, otomatik AFM tarafından araştırılacak toplam kare alan (totalArea, toplam kare alanın μm'deki uzunluğudur) ve kuyu başına kuvvet eğrisi sayısıdır (numScans). Tüm birimler μm cinsindendir.

Şekil 4: Değerli (yeşil noktalar) ilk kuyulara bir örnek sağlayan optik görüntü.
Siyah kare, tarama alanını ve kırmızı noktaları, atılması gereken ilk kuyuları temsil eder. Bu rakam13'ten değiştirilmiştir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 5: AFM tarafından otomatik olarak yürütülen 5 adımı gösteren program akış çizelgesi. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
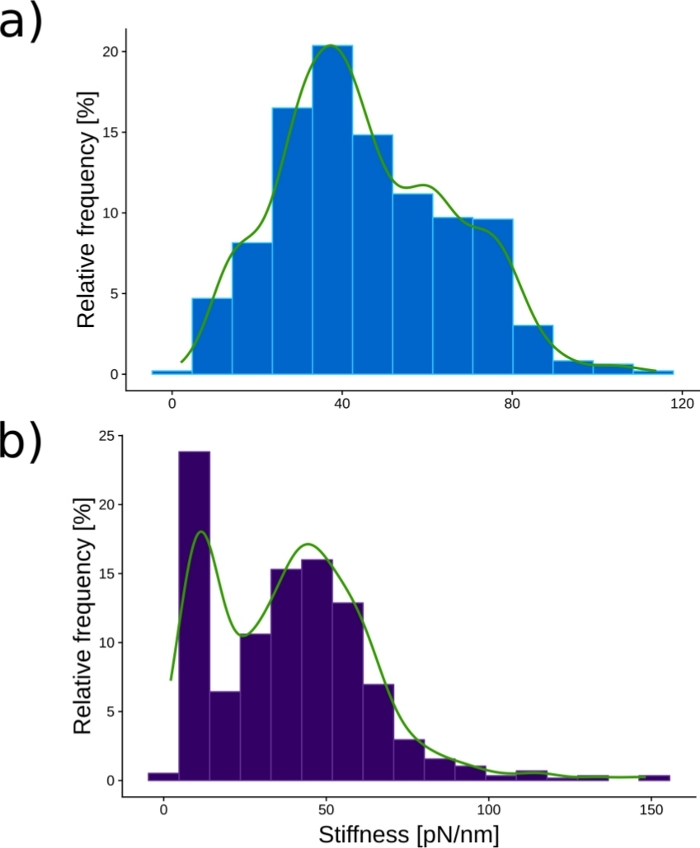
Şekil 6: Ortanca sertlik değerlerinin histogramları.
(A, B) Yerel ve caspofungin tedavi edilen hücre için hücre başına ortanca sonuçları gösterin. Bu rakam13'ten değiştirilmiştir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
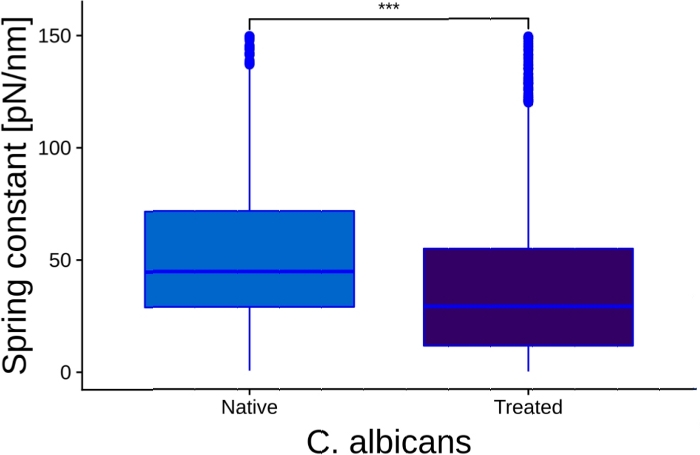
Şekil 7: Yerel ve caspofungin hücreleri ile tedavi karşılaştıran kutu çizimleri.
3 yıldız, p<0.001'in önemini temsil eder. Kutu sonuçların% 90'ını temsil eder, orta çizgi ortanca değerdir ve dikey çubuklar tüm verilerin aralığını temsil eder. Bu rakam13'ten değiştirilmiştir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 8: Değerlerin zaman konumu bağımlılığı.
Merkezdeki histogramlar, kurulan alt nüfuslara (mavi/sarı) karşılık gelen farklı alt gruplara ayrılan orijinal verilerdir. (A,B) Deneyde her saat iki alt popülasyonun varlığını gösterin. (C,D) girinti pozisyonlarını gösterir; her pozisyonda alt nüfusların (mavi/sarı) varlığını görmek mümkündür. Alt grup organizasyonu k-means algoritması kullanılarak yapıldı. Bu rakam13'ten değiştirilmiştir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
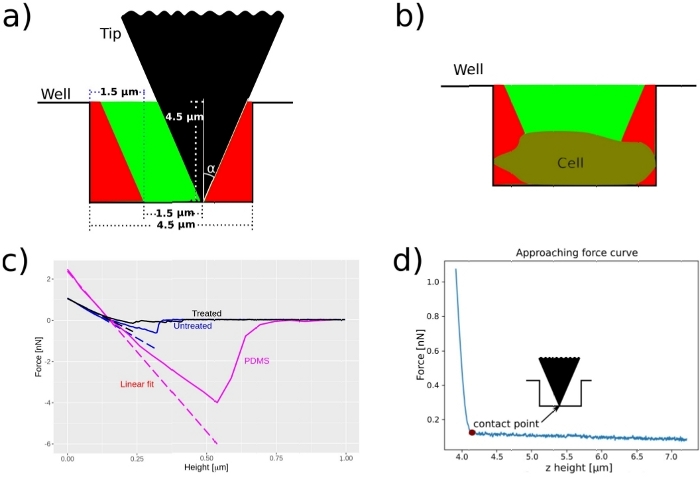
Şekil 9: Güvenli alan.
PDMS kuyusunun içindeki bir alan, piramit ucunun kuyu dibine ulaşırken kuyu kenarına dokunmadığı alan olarak tanımlanmıştır (boş bir kuyu durumunda). Bu Şekil13'ten değiştirilmiştir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Tamamlayıcı Veriler. Bu dosyaları aşağı çekmek için lütfen buraya tıklayın.
Tartışmalar
Bu metodolojinin sağladığı temel gelişme, ölçülen hücrelerin sayısında belirlenen sürede önemli bir artıştır. Muadili, hücre başına ölçülen puan sayısının azaltılmasıdır. Bu yöntemin tek bir hücrenin ayrıntılı bir analizini sağlamak için tasarlanmadığı anlamına gelir. Yöntem yalnızca PDMS damgasının kuyularına sığabilen hücreler için geçerlidir. Damga oldukça çok yönlüdür, 1.5 x 1.5 μm 2 ila 6 x6 μm2kuyular içerir. Yine de basili veya çok daha büyük hücreleri hareketsiz hale getirmek imkansızdır. Pul ve kılcal konvektif biriktirme, çok daha büyük (yaklaşık 100 μm uzunluğunda) memeli hücrelerini hareketsiz hale getirmek için kullanılamaz.
Bu bağlamda Peric ve arkadaşları Escherichia coli ve bacillus subtilis gibi basilleri hareketsiz hale getirmek için akıllı bir mikroakışkan cihaz geliştirdiler. Bu cihaz, bacillus'un tanımlanmış pozisyonlarda ve fizyolojik koşullar altında hareketsiz hale getirsini mümkün kılar. Yazılımı bu cihazın belirli boyutuna uyarlamak çok ilginç olurdu.
Uç kirlenmesi de bu otomatik sistemde bir sorun olabilir. Mikrobiyal hücreler söz konusu olduğunda, çok yaygın değildir, ancak memeli hücreleri durumunda yüksek öneme sahiptir. Dujardin ve ark.11, otomatik protokollerine bir temizlik adımı ekleyerek bu sorunu ele aldı. Bu adım, lazer toplamını kontrol etmekten ve toplam çok düşükse temizleme prosedürünü etkinleştirmekten oluşur. Temiz adım, ucu su veya etanol ile dolu bir kuyuya batırmaktan oluşur.
Sistematik olarak bu otomasyon çalışmasından kaynaklanan bir soru şu olmuştur: "heterojenlik deney sırasında hücrelerin evriminden mi geliyor?". Bu soruyu cevaplamak için, sertlik sonuçlarını Şekil 8A,B'desunulduğu gibi zamanın bir işlevi olarak çizdik. Heterojen sertlik değerlerinin deney sırasında herhangi bir zamanda kaydedildiğini açıkça göstermektedir.
Aynı bağlamda, önlem sırasındaki uç pozisyonu sorusu ortaya çıktı. Bir hücrenin kenarına kaydedilen kuvvet eğrilerinin, hücrelerin üst kısmında kaydedilen FC'den farklı bir sertliğe sahip olması mümkün olabilir. Bu rahatsızlıktan kaçınmak için güvenli alan dediğimiz alanı tanımladık. Şekil 9A,B'de tasvir edilir ve kuvvet ölçümü sırasında ucun kuyu kenarlarına temas etmeyeceği kuyuların içindeki bir alanı temsil eder. Bu "güvenli alanı" kullanarak FC'yi sadece hücrelere ve üstlerine kaydedebiliriz. Şekil 8C,D'de gösterildiği gibi, güvenli alandaki uç pozisyonu, ucun her pozisyonu için her iki fenotipi de bulduğumuz için sonuçların heterojenliğinden sorumlu değildir.
Her pozisyonda kaydedilen değerlerin homojen olduğundan emin olmak için, şekil 9C,D'desunulduğu gibi pozisyonun bir işlevi olarak sertlik değerlerini çizdik. Kuyudaki her pozisyonda heterojen sertlik değerlerinin kaydedildiğini gösterir, bu da gözlenen heterojenliğin kuyulardaki uç pozisyonu nedeniyle bir eser olmadığı anlamına gelir.
Bu makalede sunulan protokol, yaşam biliminde uygulanan AFM alanında kavramsal ve metodolojik bir atılımı temsil eder. Üretilen büyük miktarda veri, şüphesiz hücre popülasyonlarının yeni kriterlere göre sınıflandırılmasına izin verecek otomatik analiz ile uyumludur. Bu protokolün protein veya şeker dizilerine uygulanması tamamen mümkündür ve ilgi alanları arasındaki aralığı göz önünde bulundurmak için sadece birkaç uyarlama gerektirir.
Bu nedenle, güçlü disiplinler arası işbirliğinin sonucu olan çok yönlü bir protokoldür.
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
Conacyt (Meksika), Fransa Dışişleri Bakanlığı ve Université Paris 13'ün FONCYCYT'ini kabul etmek istiyoruz, ancak tanı için Nano palpation adlı uluslararası işbirlikçi ECOS-NORD projesinin finansal desteği, 263337 Numara (Meksika) ve MI5P02 (Fransa). AMR, 20195489 No'yla SIP-IPN'nin finansal desteğine teşekkür eder. SPC, çift doktora sertifikası (IPN-UPS) almak için cotutelle anlaşması aracılığıyla CONACYT (No. 288029) ve IPN'den bir doktora bursu ile desteklenmektedir. ED ve CFD, Centre National de la Recherche Scientifique'de (CNRS) araştırmacıdır.
Malzemeler
| Name | Company | Catalog Number | Comments |
| AFM cantilever | Bruker AFM probes | MLCT | The cantilevers used were the labeled “C” with resonant frequency of 7 to 10 kHz and k: 0.01 N/m |
| AFM data analysis | JPK-Bruker | JPK Data processing version minimum 5.1.8 | Can be downloaded from a JPK-Bruker user acount |
| AFM Petri dishes | WPI | FluoroDish FD35-100 | The heater was used to monitor the temperature changes during the experiment |
| Atomic force Microscope (AFM) | JPK-Bruker | Nanowizard II or III | the AFM should be mounted on an inverted optical microscope with a motorized stage |
| Caspofungin | Sigma-Aldrich | SML0425-5MG | Caspofungin was used with a concentration of 4 MIC (Minimum Inhibitor Concentration) |
| Code editor | Microsoft | Visual Studio Code version 1.40.1 | https://code.visualstudio.com/ |
| Cryobeads | IFU | CB12 | |
| Dessicator/Degassing chamber | Fisherbrand | 15594635 | The equipment is used to degassing the PDMS stamps for about 50 minutes any dessicator coupled with a vaccum pump will do. |
| Petri dish heater | JPK-Bruker | PetriDishHeater | This is an add-on to the JPK/Bruker AFM. The heater was used to monitor the temperature changes during the experiment |
| Sodium acetate buffer pH 5.2 | Sigma-Aldrich | S7899 | The solution contains 18 mM sodium acetate, 1 mM CaCl2, and 1 mM MnCl2. Adjust the pH with glacial acetic acid. The solution can be stored at 4 °C for 2 months |
| Statistical analysis language | https://www.r-project.org | R version 3.6.1 | R is a language and environment for statistical computing and graphics. It is a GNU project which is similar to the S language and environment |
| Statistical analysis software | https://rstudio.com | R studio version 1.1.463 | collaboration between the R Foundation, RStudio, Microsoft, TIBCO, Google, Oracle, HP and others. RStudio and Shiny are affiliated projects of the Foundation for Open Access Statistics |
| Sylgard 184 | Sigma-Aldrich | 761028 | Polydimethylsiloxane (PDMS) and curing agent in one set |
| Yeast Peptone D Broth | Difco | 242820 | |
| YPD Agar | Difco | DF0427-17-6 |
Referanslar
- Cross, S. E., Jin, Y. S., Rao, J., Gimzewski, J. K. Nanomechanical analysis of cells from cancer patients. Nature Nanotechnology. 2 (12), 780-783 (2007).
- Dague, E., et al. Atomic force and electron microscopic-based study of sarcolemmal surface of living cardiomyocytes unveils unexpected mitochondrial shift in heart failure. Journal of Molecular and Cellular Cardiology. 74, 162-172 (2014).
- Muller, D. J., Helenius, J., Alsteens, D., Dufrene, Y. F. Force probing surfaces of living cells to molecular resolution. Nature Chemical Biology. 5 (6), 383-390 (2009).
- Dague, E. Atomic Force Microscopy to Explore Electroporation Effects on Cells. Handbook of Electroporation. , 1-13 (2016).
- Puntheeranurak, T., Neundlinger, I., Kinne, R. K. H., Hinterdorfer, P. Single-molecule recognition force spectroscopy of transmembrane transporters on living cells. Nature Protocols. 6 (9), 1443-1452 (2011).
- Formosa, C., et al. Nanoscale analysis of the effects of antibiotics and CX1 on a Pseudomonas aeruginosa multidrug-resistant strain. Scientific Reports. 2, (2012).
- Pillet, F., Chopinet, L., Formosa, C., Dague, &. #. 2. 0. 1. ;. Atomic Force Microscopy and pharmacology: From microbiology to cancerology. Biochimica et Biophysica Acta (BBA) - General Subjects. 1840 (3), 1028-1050 (2014).
- Wu, P. H., et al. A comparison of methods to assess cell mechanical properties. Nature Methods. 15 (7), 491-498 (2018).
- Rigato, A., Rico, F., Eghiaian, F., Piel, M., Scheuring, S. Atomic Force Microscopy Mechanical Mapping of Micropatterned Cells Shows Adhesion Geometry-Dependent Mechanical Response on Local and Global Scales. ACS Nano. 9 (6), 5846-5856 (2015).
- Favre, M., et al. Parallel AFM imaging and force spectroscopy using two-dimensional probe arrays for applications in cell biology. Journal of Molecular Recognition. 24 (3), 446-452 (2011).
- Dujardin, A., Wolf, P. D., Lafont, F., Dupres, V. Automated multi-sample acquisition and analysis using atomic force microscopy for biomedical applications. PLOS ONE. 14 (3), 0213853 (2019).
- Formosa, C., et al. Generation of living cell arrays for atomic force microscopy studies. Nature Protocols. 10 (1), 199-204 (2015).
- Proa-Coronado, S., Séverac, C., Martinez-Rivas, A., Dague, E. Beyond the paradigm of nanomechanical measurements on cells using AFM: an automated methodology to rapidly analyse thousands of cells. Nanoscale Horizons. , (2019).
- Foncy, J., et al. Comparison of polyurethane and epoxy resist master mold for nanoscale soft lithography. Microelectronic Engineering. 110, 183-187 (2013).
- Unsay, J. D., Cosentino, K., García-Sáez, A. J. Atomic Force Microscopy Imaging and Force Spectroscopy of Supported Lipid Bilayers. Journal of Visualized Experiments. (101), e52867 (2015).
- Schiavone, M., et al. A combined chemical and enzymatic method to determine quantitatively the polysaccharide components in the cell wall of yeasts. FEMS Yeast Research. 14 (6), 933-947 (2014).
- Formosa, C., et al. Nanoscale Effects of Caspofungin against Two Yeast Species, Saccharomyces cerevisiae and Candida albicans. Antimicrobial Agents and Chemotherapy. 57 (8), 3498-3506 (2013).
- El-Kirat-Chatel, S., et al. Nanoscale analysis of caspofungin-induced cell surface remodelling in Candida albicans. Nanoscale. 5 (3), 1105-1115 (2013).
- Peric, O., Hannebelle, M., Adams, J. D., Fantner, G. E. Microfluidic bacterial traps for simultaneous fluorescence and atomic force microscopy. Nano Research. 10 (11), 3896-3908 (2017).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır