Method Article
Automatisierung von Bio-Rasterkraftmikroskop-Messungen an Hunderten von C. albicans-Zellen
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Dieses Protokoll zielt darauf ab, AFM-Messungen an Hunderten von mikrobiellen Zellen zu automatisieren. Zuerst werden Mikroben in PDMS-Stempelmikrostrukturen immobilisiert und dann werden kraftspektroskopische Messungen automatisch an Hunderten von immobilisierten Zellen durchgeführt.
Zusammenfassung
Die in diesem Artikel vorgestellte Methode zielt darauf ab, Bio-AFM-Experimente und die Aufzeichnung von Kraftkurven zu automatisieren. Mit dieser Methode ist es möglich, Kräftekurven auf 1000 Zellen in 4 Stunden automatisch aufzuzeichnen. Um eine Analysezeit von 4 Stunden einzuhalten, wird die Anzahl der Kraftkurven pro Zelle auf 9 oder 16 reduziert. Die Methode kombiniert ein Jython-basiertes Programm und eine Strategie zum Zusammensetzen von Zellen auf definierten Mustern. Das Programm, das auf einem kommerziellen Bio-AFM implementiert ist, kann die Spitze auf der ersten Zelle des Arrays zentrieren und sich dann automatisch von Zelle zu Zelle bewegen, während Kraftkurven auf jeder Zelle aufgezeichnet werden. Mit dieser Methodik ist es möglich, auf die biophysikalischen Parameter der Zellen wie ihre Steifigkeit, ihre Hafteigenschaften usw. zuzugreifen. Mit der Automatisierung und der großen Anzahl der analysierten Zellen kann man auf das Verhalten der Zellpopulation zugreifen. Dies ist ein Durchbruch im Bio-AFM-Bereich, wo bisher nur Daten an wenigen Dutzend Zellen aufgezeichnet wurden.
Einleitung
Diese Arbeit bietet eine Methodik, um automatische Kraftmessungen an Hunderten von lebenden Zellen mit einem Rasterkraftmikroskop (AFM) durchzuführen. Es bietet auch eine Methode zur Immobilisierung von Mikroben auf einem mikrostrukturierten PDMS-Stempel, die mit AFM-Experimenten kompatibel ist, die in einer flüssigen Umgebung durchgeführt werden.
Bio-AFM ist eine hochspezialisierte Technologie, die für Anwendungen in der Biologie konzipiert und dann zur Untersuchung lebender Zellen verwendet wird. Es erfordert einen ausgebildeten Ingenieur, der eine Zelle zu diesem Zeitpunkt analysieren kann. Unter diesen Bedingungen ist die Anzahl der verschiedenen Zellen, die analysiert werden können, eher klein, typische 5 bis 10 Zellen in 4-5 Stunden. Die Menge der an einer einzelnen Zelle aufgezeichneten Kraftmessungen ist jedoch in der Regel sehr hoch und kann leicht 1000 erreichen. Daher besteht das derzeitige Paradigma der AFM-Kraftmessungen an lebenden Zellen darin, Hunderte von Kraftkurven (FCs) aufzuzeichnen, jedoch auf einer begrenzten Anzahl von Zellen.
Statistisch gesehen ist dieser Ansatz fragwürdig und wirft die Frage nach der Repräsentativität der Stichprobe auf. Tatsächlich ist es beispielsweise schwierig, die Heterogenität einer Zellpopulation zu bewerten, indem man nur wenige Zellen misst, selbst wenn hunderte von Messungen an diesen wenigen Zellen aufgezeichnet werden. Auf der Grundlage dieses Paradigmas wurden jedoch große Fortschritte in der Biophysik, Mikrobiologie und Nanomedizin erzielt1,2,3. Tatsächlich hat die Nanometeranalyse auf der Skala einzelner Zellen neue Informationen über die zelluläre Nanomechanik, über die Organisation von Transmembranproteinen oder den Wirkmechanismus antimikrobieller oder krebshemmender Medikamente geliefert4,5,6,7. In jüngster Zeit sind jedoch mehrere biomechanische Hochdurchsatztests an Zellen aufgetaucht8, die das Interesse der wissenschaftlichen Gemeinschaft zeigen, dieses Paradigma zu ändern und auf die Heterogenität der Zellpopulation zuzugreifen. Diese Tests basieren alle auf mikrofluidischen Systemen, um Zellen zu verformen und ihre Verformung unter Belastung optisch zu messen, um ein indirektes Maß für ihre gesamte Oberflächenelastizität zu erhalten8. Ein wichtiges Problem bei diesen Methoden ist jedoch, dass sie monoparametrisch sind: Nur die Zellelastizität kann untersucht werden. Darüber hinaus erlauben sie keine Messung der mechanischen Parameter von adhärenten Zellen, was beispielsweise für die Untersuchung von nicht zirkulierenden Säugetierzellen oder Biofilmen einschränkend sein kann.
Ansätze mit AFM wurden von den Teams von S. Scheuring9 und M. Favre10entwickelt. Scheuring et al. immobilisierten Zellen auf Fibronektinmustern9, die einzelne Zellen zwangen, die Form des Musters anzunehmen9. Dann kartierte dieses Team die mechanischen Eigenschaften einiger Zellen, um durchschnittliche Daten zu definieren, die für 14 bis 18 Zellen repräsentativ sind. Die von Farve et al. durchgeführte Entwicklung zielte darauf ab, die Messungen durch Parallelisierung der AFM-Ausleger10 zumultiplexen. Unseres Wissens hat diese Arbeit in Multiplexrichtung nicht zu Messungen an lebenden Zellen geführt.
Ein interessanter Ansatz, der von Dujardins Team vorgeschlagen wurde, stellt ein automatisiertes AFM vor, das in der Lage ist, Zellen zu identifizieren und sie am Boden von maßgeschneiderten Vertiefungen abbilden zu können. Obwohl diese Methode nicht die Analyse einer großen Population von Zellen ermöglicht, ermöglicht sie das automatische Testen verschiedener Bedingungen in jederVertiefung 11.
Unser Ziel in dieser Arbeit ist ehrgeiziger, da wir mindestens 1000 Zellen messen wollten, um nicht auf eine durchschnittliche Zelle zuzugreifen, sondern im Gegenteil auf die Heterogenität zwischen den Zellen. Die Strategie, die wir hier entwickelt haben, um mit AFM auf Zellpopulationsheterogenität zuzugreifen, basiert auf der Analyse von Hunderten von Zellen, auf denen eine begrenzte Anzahl von Kraftkurven aufgezeichnet wird. Im Vergleich zum "klassischen" Ansatz, eine große Anzahl von Kraftkurven auf einer begrenzten Anzahl von Zellen aufzuzeichnen, sollte dieser Ansatz als komplementär betrachtet werden, da er nicht die gleichen Informationen liefert. Während die typische Methode es ermöglicht, die Heterogenität einzelner Zelloberflächen zu untersuchen, sind wir mit unserem Ansatz in der Lage, auf die gesamte Heterogenität der Zellpopulation zuzugreifen. Um dieses Ziel zu erreichen, haben wir eine Methode kombiniert, die Mikroben (hier die Hefeart Candida albicans)direkt in die Vertiefungen eines mikrostrukturierten PDMS-Stempels12immobilisiert und ein originelles Programm entwickelt, um die AFM-Spitze automatisch von Zelle zu Zelle13 zu bewegen und die mechanischen Eigenschaften jeder Zelle zu messen.
Protokoll
1. Mikrobielle Zellkultur
- Belebung von Zellen aus einem Glycerinvorrat.
HINWEIS: C. albicans werden bei -80 °C in Glycerinvorräten auf Murmeln gelagert.- Wählen Sie eine Murmel in der -80 °C-Brühe und reiben Sie sie auf Hefepeptrose (YPD) Agar. Züchten Sie die Zellen für 2 Tage bei 30 °C, bevor Sie die Flüssigkeit kultivieren.
- Bereiten Sie flüssige Kulturen vor.
- Füllen Sie ein Kulturröhrchen mit 5 ml steriler YPD-Brühe und fügen Sie eine einzelne Kolonie von C. albicans-Zellen hinzu, die auf der YPD-Agarplatte gezüchtet wurden.
- Die Kultur unter statischen Bedingungen bei 30 °C für 20 h vor der Ernte durch Zentrifugation (4000 x g,5 min) züchten. Entsorgen Sie den Überstand und beseitigen Sie als biogefährliche Abfälle.
- Waschen Sie die Pellets 2x mit 10 mL Acetatpuffer (8 mM Natriumacetat, 1 mM CaCl2,1 mM MnCl2,pH 5,2). Zentrifuge (4000 x g, 5 min) zwischen den Waschgängen.
- Resuspend das Pellet in 2 mL Acetatpuffer und verwenden Sie diese Lösung für die Zellimmobilisierung auf dem PDMS-Stempel.
HINWEIS: Diese Suspension kann nicht gelagert werden und sollte für Abschnitt 3 frisch zubereitet werden.
2. PDMS-Stempelvorbereitung
- Silizium-Master-Formvorbereitung
- Zeichnen Sie die gewünschten Mikrostrukturen mit CAD-Software (Computer Assisted Design).
HINWEIS: Die entworfenen Brunnen sollten eine ähnliche Größe wie die zu fangenden Mikroben haben. Das Design sollte eine große Matrix von Brunnen 100 x 100 bei Liste bieten. Es ist am besten, mehrere Arrays mit leicht unterschiedlichen Größen um die durchschnittliche Größe der Mikrobe herum zu erstellen. - Wenn ein Reinraum verfügbar ist, führen Sie die Schritte 2 bis 12 des zuvor veröffentlichten Protokolls12 aus. Andernfalls kann Silizium-Master-Form aus gewerblichen Reinraumeinrichtungen erworben werden.
- Zeichnen Sie die gewünschten Mikrostrukturen mit CAD-Software (Computer Assisted Design).
- PDMS Stempelformen
- 55 g PDMS-Prepolymerlösung mit einer Mischung aus 10 zu 1 Massenverhältnis aus PDMS-Oligomeren und Härter(Materialtabelle) werden 55g PDMS-Prepolymerlösung zubereitet.
- Mischen und entgasen Sie diese Lösung unter Vakuum (im Bereich von 10-1-10-2 bar), bis alle eingeschlossenen Blasen aus der PDMS-Lösung entfernt sind (5-10 min).
- Gießen Sie 20 g der entgasten Lösung auf die Silizium-Masterform und entgasen Sie erneut (im Bereich von 10-1-10-2 bar).
HINWEIS: Die Stempeldicke sollte etwa 2−3 mm beträgen. - Wenn alle Blasen entfernt sind, setzen Sie das PDMS bei 80 °C während 1 h.
- Schneiden Sie den mikrostrukturierten PDMS-Stempel mit einem Skalpell (0,5 x 1,5 cm2)in eine Richtung parallel zu den sichtbaren Mikrostrukturarrays.
- Schälen Sie den Stempel aus der Silikon-Masterform.
- Geben Sie den Stempel zurück, um die Mikrostrukturen auf der Oberseite auszustellen, und legen Sie ihn auf einen Glasträger. Stellen Sie sicher, dass die Mikrostrukturen vom Glasobjektträger nach oben gerichtet sind. Richten Sie die Mikrostrukturen, die auf dem Stempel zu sehen sind, an der Seite des Glasträgers aus, der später als Referenz für das AFM-Automatisierungsverfahren dienen wird.
HINWEIS: Zu diesem Zeitpunkt ist der PDMS-Stempel bereit für die Zellimmobilisierung. Die PDMS-Stempel können mehrere Monate auf der Silizium-Masterform gelagert werden. Wenn das gesamte PDMS aus der Master-Form entfernt ist, kann ein neuer PDMS-Stempel wieder auf die Master-Form gegossen werden (um die Master-Form sicher zu halten, ist es möglich, sie in Polyurethan zu replizieren)14.
3. Probenvorbereitung
- Zellimmobilisierung
- Zentrifuge (500 x g,5 min) 600 μL der resuspendierten Zelllösung, um den Puffer von den Zellen zu trennen.
- 200 μL des Überstandes aus Schritt 3.1.1 auf den PDMS-Stempel pipetieren und unter Vakuum (im Bereich von 10-1 -10-2 bar) für ca. 40 min entgasen.
HINWEIS: Dieser Schritt ist wichtig, um die Zellimmobilisierung in den Vertiefungen zu verbessern. Moleküle aus der Hefezellwand, die im Überstand vorhanden sind, lagern sich während dieses Vorbenetzungsschritts wahrscheinlich auf der PDMS-Oberfläche ab. Diese Moleküle verbessern höchstwahrscheinlich die Adhäsion der Zellen und tragen zur Erhöhung der Stempelfüllrate bei. - Nach 40 min mit einer Pipette den Puffer von der PDMS-Oberfläche entfernen und mit einer Pipette 200 μL der Zelllösung aus Schritt 1.2.4 für 15 min bei Raumtemperatur ablagern.
- Platzieren Sie die Zellen durch konvektive/kapillare Anordnung in die Mikrostrukturen des Stempels. Verteilen Sie dazu manuell 200 μL Zellsuspension über den Stempel mit einem Glasschieber in beide Richtungen mit einem Winkel zwischen 30 und 50°. Es kann notwendig sein, den Glasträger mehrmals auf dem Stempel zu führen, um eine hohe Füllrate zu erreichen.
HINWEIS: Eine vollständige Beschreibung dieser Methode ist verfügbar13. - Entfernen Sie die Zellsuspension mit einer Pipette. Waschen Sie den Stempel 3x mit 1 ml Acetatpuffer, pH 5,2, um die nicht eingeschlossenen Zellen zu entfernen.
- Trocknen Sie die Rückseite des Stempels mit Stickstoffstrom, um sicherzustellen, dass der Stempel an der trockenen Petrischale haftet.
- Zum Schluss den mit Zellen gefüllten PDMS-Stempel in einer Petrischale (Materialtabelle) ablegen und mit 2 mL Acetatpuffer füllen, um die Zellen in flüssigem Medium zu halten.
- Setzen des Stempels auf der AFM-Bühne
- Zentrieren Sie die Bühne bei 0:0, wenn AFM-Vorgänge gestartet werden.
- Kalibrierung der Empfindlichkeit und Federkonstante des Auslegers auf Glas und wasser wie in Unsay et al.15 beschrieben
- Nehmen Sie die Petrischale mit dem Stempel und legen Sie sie in den AFM Petrischalenhalter.
- Richten Sie die Stempelkante senkrecht zur Y-Achse des Petrischalenhalters aus.
HINWEIS: Ein akzeptabler Neigungswinkel liegt unter 5°, wie in Abbildung 1 dargestellt. - Legen Sie den AFM-Kopf auf die Bühne und achten Sie darauf, dass die Schrittmotoren ausreichend ausgefahren sind, um zu vermeiden, dass die Spitze auf den Stempel stürzt.
4. Ausführen des AFM-Programms
HINWEIS: Das AFM-Programm wird als Ergänzendes Material (AutomatipSoftware2019.pdf) bereitgestellt. Es erfordert einen JPK-Bruker AFM Nanowizard II oder III, der mit einer motorisierten Bühne und JPK Desktop-Software Version 4.3 ausgestattet ist. Das Programm wurde unter Jython entwickelt (Version basierend auf Python 2.7)
- Datenerfassung
- Zentrieren Sie die AFM-Spitze oben in der linken Ecke der 4,5 x 4,5 μm2 Wells (entsprechend der Zellgröße) mit dem AFM-Lichtmikroskop. Wenn eine andere Bohrplatzgröße benötigt wird, zentrieren Sie in der oberen linken Ecke der gewünschten Vertiefungen.
- Führen Sie eine 64 x 64 Kraftkarte (Z-Bereich = 4 μm, Spitzengeschwindigkeit = 90 μm·s-1,aufgebrachte Kraft 3 bis 5 nN) über eine Fläche von 100 x 100 μm² durch. Wählen Sie im Dropdown-Feld Messmodus die Option Zuordnungsmodus erzwingen aus. Geben Sie im Force Control Mapping Panel folgende Parameter ein: Rel. Sollwert = 3 bis 5 nN; z Länge 4 μm; Z-Bewegung: konstante Dauer; Verlängerungszeit: 0,01s; ext. Verzögerung:0; Retr-Verzögerung: 0, Verzögerungsmodus: Konstante Kraft, Abtastrate 2048 Hz; Z geschlossene Schleife deaktivieren; Raster: Quadratisches Bild prüfen, Schnell 100 μm, langsam: 100μm, X-Offset: 0 μm; Y-Offset: 0 μm; Gitterwinkel: 0 Grad; Pixel: 64x64; Pixelverhältnis: 1:1
HINWEIS: Ein typisches Ergebnis ist in Abbildung 2 dargestellt. Dieses Bild hilft, die Steigung zwischen zwei Bohrbrunnen zu messen und zu überprüfen. - Beachten Sie die Koordinaten der Mitte der oberen linken Vertiefung (W1) und der unteren linken Vertiefung (in Abbildung 2als W2 bezeichnet). Um dies zu tun, machen Sie eine quadratische Box um den Brunnen. Die Koordinate der Mitte des Felds wird im linken Bereich der AFM-Software in x,y-Koordinatenfeldern angezeigt.
- Um die Automatisierungssoftware (Automatip_scan.py): klicken Sie in der JPK-Desktop-Software auf Weiter im menü der oberen Leiste und wählen Sie Skript öffnen. Wählen Sie im sich öffnenden Fenster den Pfad zu der Skriptdatei aus, die unter Ergänzende Daten (Automatip_scan.py) bereitgestellt wird.
- Implementieren Sie W1- und W2-Koordinatenwerte im Abschnitt Eingaben des Jython-Skripts (Abbildung 3). Geben Sie die W1-Koordinaten in die P1-Variablenlinie 239 des Skripts und die W2-Koordinaten in die P2-Variablenlinie 241 ein.
HINWEIS: Die als Anfangskoordinaten ausgewählten Vertiefungen (W1 und W2) sollten nicht zu nahe am Rand des Scanbereichs liegen. Andernfalls würde der Zentrierungsalgorithmus nicht korrekt ausgeführt, da er die Höhe auf der PDMS-Oberfläche auf jeder Seite des Bohrplatzes messen muss. Ein Beispiel finden Sie in Abbildung 4. - Ordnen Sie den Pitch-Wert der Pitch-Variablen Zeile 245 des Skripts zu.
- Geben Sie die Bohrliniendimension in die Ws-Variable Zeile 248 ein. Dies ist aus dem Design der Bohrbrunnenmuster bekannt und kann auf demselben Bild überprüft werden, das zur Überprüfung der Steigung verwendet wurde (Abbildung 2).
- Schreiben Sie den Pfad zum Speicherverzeichnis in Zeile 251, um die Daten an der gewünschten Stelle zu speichern.
- Setzen Sie die totalArea-Variable Zeile 254 auf das gewünschte Vielfache "n" von 100 μm (das ist die maximale Scanfläche des verwendeten AFM). Die Gesamtzahl der bohrten Bohrungen kann mit diesem Wert und der Steigung berechnet werden: maximale Scanfläche/Steigung*n2.
HINWEIS: Im Beispiel von Abbildung 3werden 9 Bereiche von 100 x 100 μm2 analysiert. - Legen Sie die Kraftkurvenmatrix, Zeile und Spalte (3, 3 oder 4, 4) fest, die pro Vertiefung in der numScans-Variablenlinie 257 aufgezeichnet wurden.
HINWEIS: Im Beispiel von Abbildung 3wird für jede Bohrung eine Matrix von 3 x 3 = 9 FCs aufgezeichnet. - Führen Sie das Programm aus. Klicken Sie auf die Schaltfläche Start.
HINWEIS: Das Programm führt zunächst automatisch einen Zentrierungsalgorithmus aus, um das Zentrum der W1- und W2-Bohrungen besser zu bestimmen (Schritt 1). Es erfasst dann automatisch die Force Curves (FCs) -Matrix auf jeder Vertiefung des ersten Scanbereichs (Schritt 2). Wenn alle Vertiefungen dieses Bereichs untersucht werden, verschiebt das Skript die AFM-Spitze automatisch zur ersten Vertiefung des nächsten Scanbereichs. Die Spitze wird zurückgezogen, der Mikroskopstand bewegt sich zum nächsten Bereich, die Spitze wird wieder auf dem Stempel angefahren und der Zentrierungsalgorithmus wird erneut ausgeführt, um automatisch auf der ersten Vertiefung (1') dieses Bereichs neu zu zentrieren (Schritt 3). Der erste Bereich wird vom Benutzer definiert, der zweite, befindet sich auf der rechten Seite usw., bis n erreicht ist. n+1 Bereich ist unter n, n+2 links von n+1, etc. bis 2n erreicht ist. 2n + 1 ist unter 2n und 2n + 2 ist auf der rechten Seite auf 2n usw. Global serpentinen die Spitze durch die Gesamtfläche. Schritt 2 und 3 werden automatisch wiederholt, bis die Gesamtzahl "n2" der Scanbereiche untersucht wurde. Abbildung 5 zeigt das Flussdiagramm des Programms. Es dauert ~ 4 Stunden, um das Programm abzuschließen.
- Datenanalyse
- Führen Sie das Python-Skript "Dateien kopieren" aus (Copy_files_L.py, bereitgestellt in Supplementary Data), um die FCs-Dateien in einem Ordner zu organisieren. Dieses Skript wurde mit Python 2.7 und dem SciPy-Modul entwickelt. Verwenden Sie Visual Studio Code-Software, um das Python-Skript zu öffnen. Eingabepfad zum allgemeinen Ordner (Zeile 67 des in den ergänzenden Daten bereitgestellten Skripts) und wo es gespeichert wird (Zeile 73).
- Öffnen Sie die AFM-Herstellerdatenverarbeitungssoftware, um die Kraftkurven zu analysieren. Wählen Sie im oberen Menü Dateidie Option'Batch of spectroscopy curvesöffnen .
- Wählen Sie im Stapelverarbeitungsfenster den unter Ergänzende Daten (StiffnessProcess.jpk-proc-force) bereitgestellten Prozess aus. Wählen Sie den letzten Schritt des Prozesses aus und klicken Sie auf Behalten und auf Alle anwenden. Alle Kraftkurven werden gleich behandelt.
HINWEIS: Der Prozess verwendet die Kalibrierung aus den FCs-Dateien, um die Durchbiegungskurven in in Newton kalibrierte Kraftkurven umzuwandeln. ein Datenglättungsalgorithmus angewendet wird (Durchschnitt von 3 aufeinanderfolgenden Punkten); die Basislinie wird so übersetzt, dass sie auf der Nullachse ruht. der Kontaktpunkt wird extrapoliert und der FC wird versetzt, um den Kontaktpunkt an der Koordinate (0,0) zu platzieren. die Biegung des Auslegers wird von den FCs abgezogen, die Rückzugsneigung wird eingebaut. Am Ende der Datenbehandlung generiert die Software eine Datei, die eine Tabelle enthält, die für jedes FCs folgende Angaben enthält: Name, Young Modulus, Kontaktpunkt, Adhäsionskraft, Steigungen usw. - Wiederholen Sie die Schritte 4.2.1 bis 4.2.3 für alle Experimente. Achten Sie darauf, die Daten in verschiedenen Ordnern zu speichern (z. B.: "...\TREATED\" und "...\UNBETREATED\")
- Verwenden Sie das R-Skript in Supplementary Data, um Histogramme und Box-Plots zu zeichnen und statistische ANOVA-Behandlungen durchzuführen.
- Um das R-Skript (DataAnalisys.R) zu öffnen, verwenden Sie die R Studio-Software und laden Sie die Dateien, die die mit der Datenverarbeitungssoftware (.tsv) extrahierten Informationen enthalten.
- Verwenden Sie im Umgebungsfenster die Schaltfläche Datensatz importieren, wählen Sie in der angezeigten Liste aus Text aus (Readr) und wählen Sie im neuen Fenster die Schaltfläche Browser aus und suchen Sie die TSV-Datei.
- Sobald die Datei geladen ist, wählen Sie die Spalten (Steifigkeit und Haftung) aus, die für die Analyse einbezogen werden sollen. Um den gesamten Code auszuführen, drücken Sie STRG+ALT+R.
HINWEIS: Das Skript arbeitet mit 4 Datensätzen, betrachten Sie zwei Experimente mit unbehandelten und behandelten Zellen. Es ist möglich, Blöcke des Skripts auszuführen und zu sehen, wie sich die Variablen entsprechend den ausgeführten Funktionen ändern.
Ergebnisse
Wir haben das beschriebene Protokoll verwendet, um die Wirkung von Caspofungin auf die biophysikalischen Eigenschaften des opportunistischen Humanpathogens C. albicans in seiner Hefeform zu analysieren. Caspofungin ist ein Antimykotikum der letzten Chance, das verwendet wird, wenn andere Medikamente aufgrund der Resistenzmechanismen, die Zellen gegenüber Antimykotika entwickeln, unwirksam sind. Sein Wirkmechanismus beruht auf der Hemmung der Untereinheit Fks2 des komplexen fks1/Fks2, der für die ß-Glucansynthese verantwortlich ist. Da ß-Glucane ein Hauptbestandteil der Pilzzellwand16,17sind, erwarteten wir eine Modifikation der biophysikalischen Eigenschaften der Zellwand: Steifigkeit und Adhäsion.
Abbildung 6 zeigt typische Histogramme, die erhalten werden, wenn das gesamte oben dargestellte Protokoll angewendet wird. Das rote Histogramm stellt die Steifigkeitsaufteilung dar, die auf 957 nativen Zellen aufgezeichnet wurde, und das blaue auf 574 caspofunginbehandelten Zellen. Die erste interessante Beobachtung ist, dass beide Histogramme eine bimodale Verteilung der Werte zeigen. Diese Beobachtung ist nur möglich, weil wir Hunderte von Zellen gemessen haben. Bei kleineren Stichproben beobachten Forscher in der Regel eine einzelne Verteilung und übersehen die Populationsheterogenität. 17,18
Die zweite Beobachtung betrifft die Wirkung von Caspofungin. Es reduziert global die Steifigkeit der Zellen, während noch zwei Subpopulationen existieren. In einem letzten Schritt bietet das vorgeschlagene Protokoll einen ANOVA-Vergleich der nativen und behandelten Zellen, wie in Abbildung 7 dargestellt. Es zeigt, dass die beiden Bedingungen eine unterschiedliche Steifigkeit haben und dass dieser Unterschied sehr signifikant ist (p-Wert < 0,001). Dieser Wert wird dank der großen Anzahl der analysierten Zellen erreicht und bietet ein größeres Vertrauen in die erhaltenen Ergebnisse.
Die Adhäsion wurde auch aus den automatisch aufgezeichneten Daten extrahiert und wir fanden heraus, dass die Adhäsionskraft zwischen der nackten Spitze und den nativen Zellen 0,64 ± 0,6 nN betrug. In diesem Fall wurden auch zwei Subpopulationen gefunden: Die erste hat eine mittlere Adhäsionskraft von 0,7 ± 1,4 nN, während die zweite von 4,5 ± 1,5 nN. Die Behandlung mit Caspofungin hatte unvorhersehbare Auswirkungen auf die Adhäsion. In einem Experiment wurde kein Effekt beobachtet, aber in einem anderen Experiment induzierte Caspofungin eine Abnahme der Adhäsion an der Spitze und eine Verringerung der Populationsadhärentexterogenität. Diese Ergebnisse stammen aus Proa et al.13, wo sie in der Gesamtheit präsentiert werden.

Abbildung 1: Akzeptable Position des mikrostrukturierten Stempels auf der AFM-Stufe.
Der Neigungswinkel auf den linken Bildern (bis zu 5°) kann vom Programm gehandhabt werden, aber die Neigung auf der rechten Seite ist wichtig (10°). Diese Zahl wurde von13geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
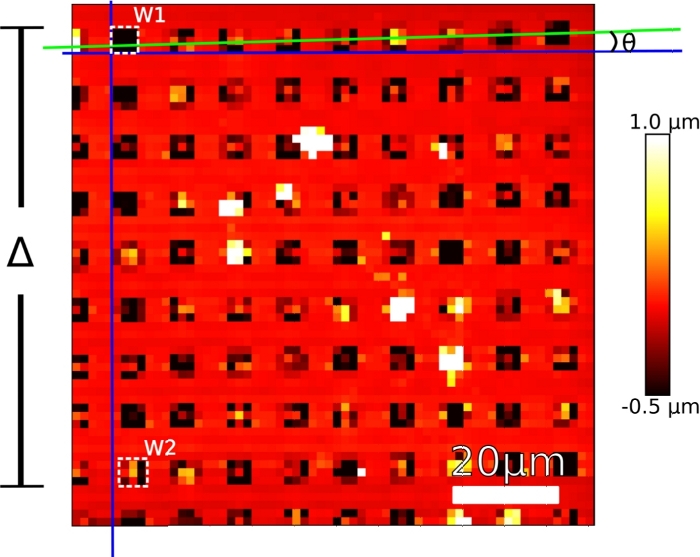
Abbildung 2: Typisches AFM-Bild eines gefüllten PDMS-Stempels mit den Anfangskoordinaten als W1 und W2, der Größe des Scanbereichs (Δ2),dem Neigungswinkel (θ).
Diese Abbildung wurde von13geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Benutzereingabeabschnitt des Skripts.
P1 und P2 beziehen sich auf die Koordinaten von Bohrung 1 (W1) und Bohrung 2 (W2) in Abbildung 2. Die anderen Parameter sind die Steigung in μm, die Bohrplatzgröße in μm (Ws), der Verzeichnispfad zum Speichern der Daten, die gesamte quadratische Fläche, die vom automatisierten AFM untersucht wird (totalArea ist die Länge in μm der Seite der gesamten quadratischen Fläche) und die Anzahl der Kraftkurven pro Bohrplatz (numScans). Alle Einheiten sind in μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Optisches Bild als Beispiel für wertvolle (grüne Punkte) Anfangsbohrungen.
Das schwarze Quadrat stellt den Scanbereich und die roten Flecken dar, anfängliche Vertiefungen, die besser verworfen werden sollten. Diese Zahl wurde von13geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Programmflussdiagramm mit den 5 Schritten, die vom AFM automatisch ausgeführt werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
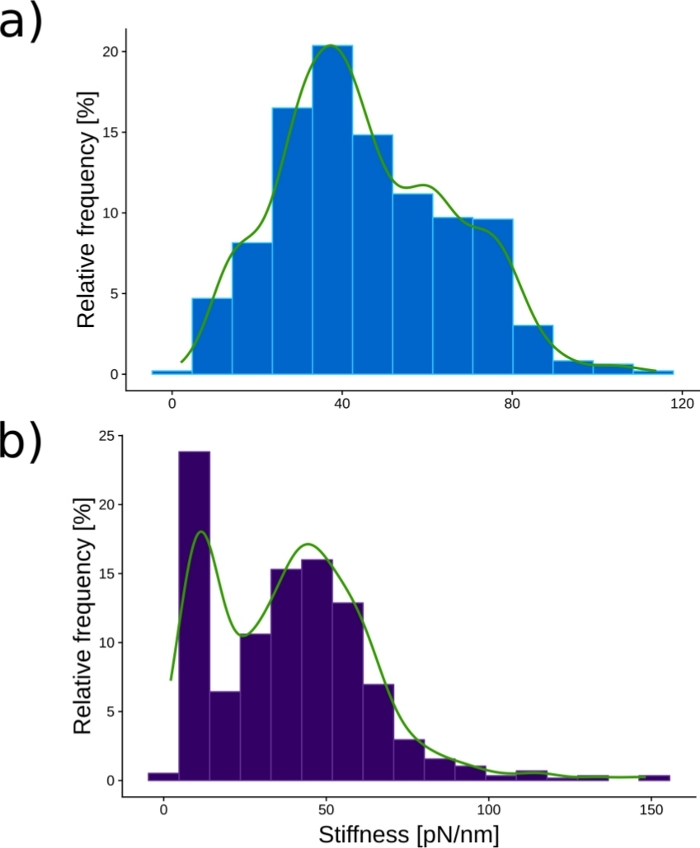
Abbildung 6: Histogramme der mittleren Steifigkeitswerte.
(A, B) Zeigen Sie die medianen Ergebnisse pro Zelle für native und mit Caspofungin behandelte Zellen an. Diese Zahl wurde von13geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
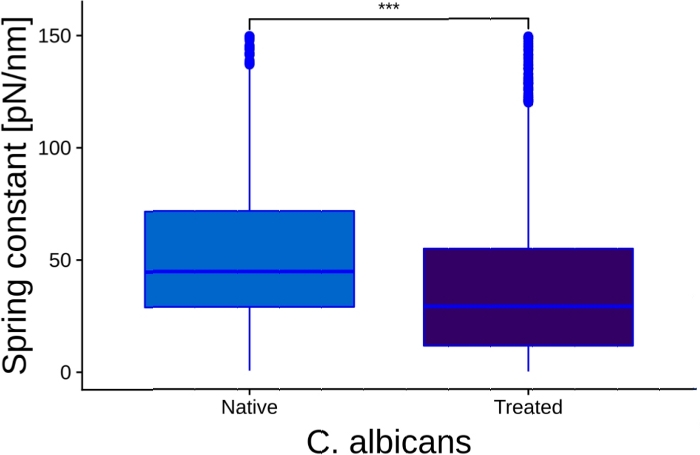
Abbildung 7: Box-Diagramme, die native und mit Caspofungin-Zellen behandelte Zellen vergleichen.
Die 3 Sterne repräsentieren eine Bedeutung von p<0,001. Das Feld repräsentiert 90% der Ergebnisse, die zentrale Linie ist der Medianwert und die vertikalen Balken repräsentieren den Bereich aller Daten. Diese Zahl wurde von13geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 8: Zeit-Positions-Abhängigkeit von Werten.
Histogramme in der Mitte sind die Originaldaten, die in die verschiedenen Untergruppen unterteilt sind, die den gegründeten Subpopulationen entsprechen (blau/gelb). (A,B) Zeigen Sie das Vorhandensein der beiden Subpopulationen zu jeder Stunde im Experiment. (C,D) zeigen die Positionen der Einrückung; An jeder Position ist es möglich, das Vorhandensein der Subpopulationen (blau / gelb) zu sehen. Die Organisation der Untergruppe erfolgte mit dem k-means-Algorithmus. Diese Zahl wurde von13geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
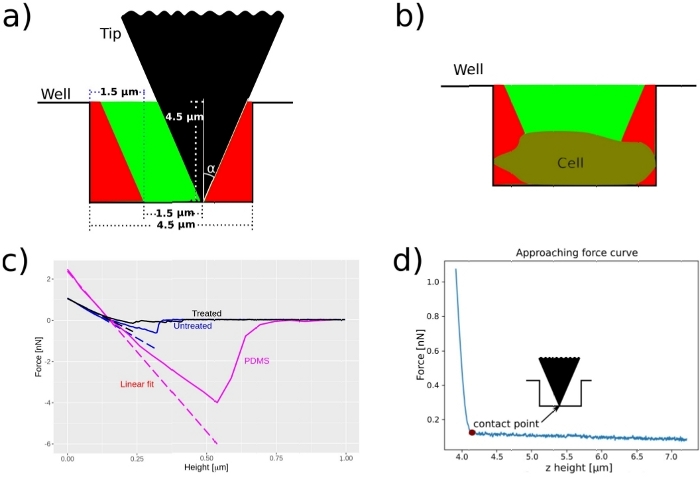
Abbildung 9: Der sichere Bereich.
Ein Bereich innerhalb des PDMS-Brunnens wurde als der Bereich definiert, in dem die pyramidenförmige Spitze die Brunnenkante nicht berührt, während sie den Bohrplatzboden erreicht (im Falle eines leeren Brunnens). Diese Abbildung wurde von13geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Daten. Bitte klicken Sie hier, um diese Dateien herunterzukleinern.
Diskussion
Die wichtigste Verbesserung, die diese Methodik bietet, ist eine signifikante Erhöhung der Anzahl der gemessenen Zellen in einem bestimmten Zeitraum. Das Gegenstück ist eine Reduzierung der Anzahl der pro Zelle gemessenen Punkte. Dies bedeutet, dass diese Methode nicht für eine detaillierte Analyse einer einzelnen Zelle ausgelegt ist. Die Methode gilt nur für Zellen, die in die Vertiefungen des PDMS-Stempels passen. Der Stempel ist sehr vielseitig, während er Vertiefungen von 1,5 x 1,5 μm2 bis zu 6 x 6 μm2enthält. Dennoch ist es unmöglich, Bazillus oder viel größere Zellen zu immobilisieren. Die Stempel- und kapillare konvektive Ablagerung kann nicht verwendet werden, um Säugetierzellen zu immobilisieren, die viel größer sind (etwa 100 μm lang).
In diesem Zusammenhang entwickelten Peric et al.19 ein intelligentes mikrofluidisches Gerät, um Bazillus wie Escherichia coli und Bacillus subtilis zu immobilisieren. Dieses Gerät ermöglicht es, Bacillus an definierten Positionen und unter physiologischen Bedingungen zu immobilisieren. Es wäre sehr interessant, die Software an die jeweilige Größe dieses Gerätes anzupassen.
Auch die Kontamination der Spitzen kann in diesem automatisierten System ein Problem darstellen. Im Falle von mikrobiellen Zellen ist es nicht so weit verbreitet, aber es ist von hoher Bedeutung im Fall von Säugetierzellen. Dujardin et al.11 haben dieses Problem behoben, indem sie ihrem automatisierten Protokoll einen Reinigungsschritt hinzugefügt haben. Dieser Schritt besteht darin, die Lasersumme zu überprüfen und den Reinigungsvorgang zu aktivieren, wenn die Summe zu niedrig ist. Der saubere Schritt besteht darin, die Spitze in einen mit Wasser oder Ethanol gefüllten Brunnen zu tauchen.
Eine Frage, die sich aus dieser Automatisierungsarbeit systematisch ergibt, lautet: "Kommt die Heterogenität von der Evolution der Zellen während des Experiments?". Um diese Frage zu beantworten, haben wir die Steifigkeitsergebnisse als Funktion der Zeit dargestellt, wie in Abbildung 8A,Bdargestellt. Es zeigt deutlich, dass heterogene Steifigkeitswerte jederzeit während des Experiments erfasst werden.
Im gleichen Zusammenhang stellte sich die Frage nach der Spitzenposition während der Maßnahme. Es könnte möglich sein, dass Kraftkurven, die am Rand einer Zelle aufgezeichnet werden, eine andere Steifigkeit haben als FC, die auf der Oberseite der Zellen aufgezeichnet wurden. Um diese Unannehmlichkeiten zu vermeiden, haben wir den sogenannten sicheren Bereich definiert. Es ist in Abbildung 9A,B dargestellt und stellt einen Bereich innerhalb der Vertiefungen dar, in dem die Spitze die Bohrplatzkanten während der Kraftmessung nicht berührt. Mit diesem "sicheren Bereich" konnten wir FC nur auf Zellen und an der Spitze von ihnen aufzeichnen. Wie in Abbildung 8C,D gezeigt, ist die Spitzenposition innerhalb des sicheren Bereichs nicht für die Heterogenität der Ergebnisse verantwortlich, da wir beide Phänotypen für jede Position der Spitze im sicheren Bereich gefunden haben.
Um sicherzustellen, dass die an jeder Position aufgezeichneten Werte homogen sind, haben wir die Steifigkeitswerte als Funktion der Position dargestellt, wie in Abbildung 9C,Ddargestellt. Es zeigt, dass an jeder Position im Bohrplatz heterogene Steifigkeitswerte aufgezeichnet werden, was bedeutet, dass die beobachtete Heterogenität aufgrund der Spitzenposition in den Vertiefungen kein Artefakt ist.
Das in diesem Artikel vorgestellte Protokoll stellt einen konzeptionellen und methodischen Durchbruch auf dem Gebiet der AFM in den Lebenswissenschaften dar. Die großen Datenmengen, die generiert werden, sind mit der automatischen Analyse kompatibel, die zweifellos die Klassifizierung von Zellpopulationen nach neuen Kriterien ermöglichen wird. Die Anwendung dieses Protokolls auf Protein- oder Zuckerarrays ist durchaus machbar und erfordert nur wenige Anpassungen, um den Abstand zwischen den Interessengebieten zu berücksichtigen.
Es ist daher ein vielseitiges Protokoll, das das Ergebnis einer starken interdisziplinären Zusammenarbeit ist.
Offenlegungen
Die Autoren haben nichts preiszugeben.
Danksagungen
Wir möchten FONCYCYT von CONACYT (Mexiko), dem französischen Außenministerium und der Université Paris 13 dank der finanziellen Unterstützung des internationalen Kooperationsprojekts ECOS-NORD mit dem Namen Nano-Palpation for diagnosis, No. 263337 (Mexiko) und MI5P02 (Frankreich) anerkennen. AMR bedankt sich für die finanzielle Unterstützung von SIP-IPN durch das Projekt Nr. 20195489. SPC wird durch ein PhD-Stipendium von CONACYT (Nr. 288029) und IPN durch die Cotutelle-Vereinbarung unterstützt, um ein doppeltes PhD-Zertifikat (IPN-UPS) zu erhalten. ED und CFD sind Forscher am Centre National de la Recherche Scientifique (CNRS).
Materialien
| Name | Company | Catalog Number | Comments |
| AFM cantilever | Bruker AFM probes | MLCT | The cantilevers used were the labeled “C” with resonant frequency of 7 to 10 kHz and k: 0.01 N/m |
| AFM data analysis | JPK-Bruker | JPK Data processing version minimum 5.1.8 | Can be downloaded from a JPK-Bruker user acount |
| AFM Petri dishes | WPI | FluoroDish FD35-100 | The heater was used to monitor the temperature changes during the experiment |
| Atomic force Microscope (AFM) | JPK-Bruker | Nanowizard II or III | the AFM should be mounted on an inverted optical microscope with a motorized stage |
| Caspofungin | Sigma-Aldrich | SML0425-5MG | Caspofungin was used with a concentration of 4 MIC (Minimum Inhibitor Concentration) |
| Code editor | Microsoft | Visual Studio Code version 1.40.1 | https://code.visualstudio.com/ |
| Cryobeads | IFU | CB12 | |
| Dessicator/Degassing chamber | Fisherbrand | 15594635 | The equipment is used to degassing the PDMS stamps for about 50 minutes any dessicator coupled with a vaccum pump will do. |
| Petri dish heater | JPK-Bruker | PetriDishHeater | This is an add-on to the JPK/Bruker AFM. The heater was used to monitor the temperature changes during the experiment |
| Sodium acetate buffer pH 5.2 | Sigma-Aldrich | S7899 | The solution contains 18 mM sodium acetate, 1 mM CaCl2, and 1 mM MnCl2. Adjust the pH with glacial acetic acid. The solution can be stored at 4 °C for 2 months |
| Statistical analysis language | https://www.r-project.org | R version 3.6.1 | R is a language and environment for statistical computing and graphics. It is a GNU project which is similar to the S language and environment |
| Statistical analysis software | https://rstudio.com | R studio version 1.1.463 | collaboration between the R Foundation, RStudio, Microsoft, TIBCO, Google, Oracle, HP and others. RStudio and Shiny are affiliated projects of the Foundation for Open Access Statistics |
| Sylgard 184 | Sigma-Aldrich | 761028 | Polydimethylsiloxane (PDMS) and curing agent in one set |
| Yeast Peptone D Broth | Difco | 242820 | |
| YPD Agar | Difco | DF0427-17-6 |
Referenzen
- Cross, S. E., Jin, Y. S., Rao, J., Gimzewski, J. K. Nanomechanical analysis of cells from cancer patients. Nature Nanotechnology. 2 (12), 780-783 (2007).
- Dague, E., et al. Atomic force and electron microscopic-based study of sarcolemmal surface of living cardiomyocytes unveils unexpected mitochondrial shift in heart failure. Journal of Molecular and Cellular Cardiology. 74, 162-172 (2014).
- Muller, D. J., Helenius, J., Alsteens, D., Dufrene, Y. F. Force probing surfaces of living cells to molecular resolution. Nature Chemical Biology. 5 (6), 383-390 (2009).
- Dague, E. Atomic Force Microscopy to Explore Electroporation Effects on Cells. Handbook of Electroporation. , 1-13 (2016).
- Puntheeranurak, T., Neundlinger, I., Kinne, R. K. H., Hinterdorfer, P. Single-molecule recognition force spectroscopy of transmembrane transporters on living cells. Nature Protocols. 6 (9), 1443-1452 (2011).
- Formosa, C., et al. Nanoscale analysis of the effects of antibiotics and CX1 on a Pseudomonas aeruginosa multidrug-resistant strain. Scientific Reports. 2, (2012).
- Pillet, F., Chopinet, L., Formosa, C., Dague, &. #. 2. 0. 1. ;. Atomic Force Microscopy and pharmacology: From microbiology to cancerology. Biochimica et Biophysica Acta (BBA) - General Subjects. 1840 (3), 1028-1050 (2014).
- Wu, P. H., et al. A comparison of methods to assess cell mechanical properties. Nature Methods. 15 (7), 491-498 (2018).
- Rigato, A., Rico, F., Eghiaian, F., Piel, M., Scheuring, S. Atomic Force Microscopy Mechanical Mapping of Micropatterned Cells Shows Adhesion Geometry-Dependent Mechanical Response on Local and Global Scales. ACS Nano. 9 (6), 5846-5856 (2015).
- Favre, M., et al. Parallel AFM imaging and force spectroscopy using two-dimensional probe arrays for applications in cell biology. Journal of Molecular Recognition. 24 (3), 446-452 (2011).
- Dujardin, A., Wolf, P. D., Lafont, F., Dupres, V. Automated multi-sample acquisition and analysis using atomic force microscopy for biomedical applications. PLOS ONE. 14 (3), 0213853 (2019).
- Formosa, C., et al. Generation of living cell arrays for atomic force microscopy studies. Nature Protocols. 10 (1), 199-204 (2015).
- Proa-Coronado, S., Séverac, C., Martinez-Rivas, A., Dague, E. Beyond the paradigm of nanomechanical measurements on cells using AFM: an automated methodology to rapidly analyse thousands of cells. Nanoscale Horizons. , (2019).
- Foncy, J., et al. Comparison of polyurethane and epoxy resist master mold for nanoscale soft lithography. Microelectronic Engineering. 110, 183-187 (2013).
- Unsay, J. D., Cosentino, K., García-Sáez, A. J. Atomic Force Microscopy Imaging and Force Spectroscopy of Supported Lipid Bilayers. Journal of Visualized Experiments. (101), e52867 (2015).
- Schiavone, M., et al. A combined chemical and enzymatic method to determine quantitatively the polysaccharide components in the cell wall of yeasts. FEMS Yeast Research. 14 (6), 933-947 (2014).
- Formosa, C., et al. Nanoscale Effects of Caspofungin against Two Yeast Species, Saccharomyces cerevisiae and Candida albicans. Antimicrobial Agents and Chemotherapy. 57 (8), 3498-3506 (2013).
- El-Kirat-Chatel, S., et al. Nanoscale analysis of caspofungin-induced cell surface remodelling in Candida albicans. Nanoscale. 5 (3), 1105-1115 (2013).
- Peric, O., Hannebelle, M., Adams, J. D., Fantner, G. E. Microfluidic bacterial traps for simultaneous fluorescence and atomic force microscopy. Nano Research. 10 (11), 3896-3908 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten