Method Article
Сравнение прогностической эффективности трех систем стадирования лимфатических узлов при колоректальной кольцевой карциноме на основе модели машинного обучения
* Эти авторы внесли равный вклад
В этой статье
Резюме
В этом исследовании оцениваются прогностические системы у пациентов с колоректальным кольцевым раком с использованием моделей машинного обучения и конкурирующего анализа риска. Он определяет логарифмическую вероятность положительного результата в лимфатических узлах как превосходный предиктор по сравнению со стадией pN, демонстрируя высокую прогностическую эффективность и помогая принимать клинические решения с помощью надежных инструментов прогнозирования выживаемости.
Аннотация
Состояние лимфатических узлов является критическим прогностическим предиктором для пациентов; тем не менее, прогноз колоректальной кольцевой карциномы (SRCC) привлек ограниченное внимание. В этом исследовании изучается прогностическая прогностическая способность логарифмических шансов положительных лимфатических узлов (LODDS), соотношения лимфатических узлов (LNR) и стадирования pN у пациентов с SRCC с использованием моделей машинного обучения (Random Forest, XGBoost и Neural Network) наряду с конкурирующими моделями риска. Соответствующие данные были извлечены из базы данных Surveillance, Epidemiology, and End Results (SEER). Для моделей машинного обучения были определены прогностические факторы для специфической для рака выживаемости (CSS) с помощью одномерного и многомерного регрессионного анализа Кокса с последующим применением трех методов машинного обучения — XGBoost, RF и NN — для определения оптимальной системы стадирования лимфатических узлов. В конкурирующей модели риска для выявления прогностических факторов были использованы одномерный и многомерный конкурирующие анализы риска, а также была построена номограмма для прогнозирования прогноза пациентов с SRCC. Для оценки характеристик модели использовалась область под кривой рабочих характеристик приемника (AUC-ROC) и калибровочные кривые. Всего в исследование было включено 2409 пациентов с SRCC. Для подтверждения эффективности модели была включена дополнительная когорта из 15 122 пациентов с колоректальным раком, за исключением случаев SRCC, для внешней валидации. Как модели машинного обучения, так и конкурирующая номограмма риска продемонстрировали высокую производительность в прогнозировании результатов выживания. По сравнению со стадией pN, системы стадирования LODDS продемонстрировали превосходные прогностические возможности. После оценки модели машинного обучения и конкурирующие модели рисков показали превосходную прогностическую производительность, характеризующуюся хорошей дискриминацией, калибровкой и интерпретируемостью. Наши результаты могут помочь в принятии клинических решений для пациентов.
Введение
Колоректальный рак (КРР) занимает третье место среди наиболее распространенных злокачественных опухолей в мире 1,2,3. Кольцевая карцинома (SRCC), редкий подтип КРР, составляет примерно 1% случаев и характеризуется обильным внутриклеточным муцином, вытесняющим ядро клетки 1,2,4. SRCC часто ассоциируется с более молодыми пациентами, имеет более высокую распространенность у женщин и имеет поздние стадии опухоли при постановке диагноза. По сравнению с колоректальной аденокарциномой, SRCC демонстрирует более слабую дифференцировку, более высокий риск отдаленного метастазирования и 5-летнюю выживаемость всего 12%-20%5,6. Разработка точной и эффективной прогностической модели для SRCC имеет решающее значение для оптимизации стратегий лечения и улучшения клинических результатов.
Данное исследование направлено на построение надежной прогностической модели для пациентов с синдромом SRCC с использованием передовых статистических подходов, включая машинное обучение (ML) и конкурирующие модели риска. Эти методологии могут учитывать сложные взаимосвязи в клинических данных, предлагая индивидуальную оценку рисков и превосходя традиционные методы по точности прогнозирования. Модели машинного обучения, такие как Random Forest, XGBoost и Neural Networks, превосходны в обработке многомерных данных и выявлении сложных закономерностей. Исследования показали, что модели ИИ эффективно прогнозируют результаты выживаемости при колоректальном раке, подчеркивая потенциал машинного обучения в клинических приложениях 7,8. Дополняя машинное обучение, конкурирующие модели риска учитывают несколько типов событий, таких как смертность от рака по сравнению с другими причинами смерти, для уточнения анализа выживаемости. В отличие от традиционных методов, таких как оценка Каплана-Мейера, конкурирующие модели риска точно оценивают предельную вероятность событий при наличии конкурирующих рисков, обеспечивая болееточную оценку выживаемости. Интеграция машинного обучения и конкурирующего анализа рисков повышает эффективность прогнозирования, предлагая мощную основу для персонализированных прогностических инструментов в SRCC 9,10,11.
Метастазирование в лимфатические узлы существенно влияет на прогноз и рецидив у пациентов с КРР. В то время как оценка N-стадии в классификации TNM имеет решающее значение, недостаточное обследование лимфатических узлов, о котором сообщалось в 48-63% случаев, может привести к недооценке заболевания. Чтобы решить эту проблему, были введены альтернативные подходы, такие как коэффициент лимфатических узлов (LNR) и логарифмические шансы положительных лимфатических узлов (LODDS). LNR, отношение положительных лимфатических узлов (PLN) к общему количеству лимфатических узлов (TLNs), меньше зависит от количества TLN и служит прогностическим фактором при КРР. LODDS, логарифмическое отношение PLN к отрицательным лимфатическим узлам (NLNs), показал превосходную прогностическую способность как при SRCC желудка, так и при колоректальном раке10,11. Машинное обучение все чаще применяется в онкологии, с моделями, улучшающими стратификацию риска и прогностическое прогнозирование различных видов рака, включая рак молочной железы, предстательной железы и легких 12,13,14. Тем не менее, его применение при колоректальном SRCC остается ограниченным.
Данное исследование направлено на преодоление этого разрыва путем интеграции LODDS с ML и конкурирующими моделями риска для создания комплексного прогностического инструмента. Оценивая прогностическую ценность LODDS и используя передовые методы прогнозирования, это исследование направлено на улучшение процесса принятия клинических решений и улучшение результатов для пациентов с SRCC.
протокол
В данном исследовании не говорится об этическом одобрении и согласии на участие. Данные, использованные в данном исследовании, были получены из баз данных. В исследование были включены пациенты с диагнозом колоректальная карцинома с 2004 по 2015 гг., а также другие виды колоректального рака. Критерии исключения включали пациентов со временем выживаемости менее одного месяца, пациентов с неполной клинико-патологической информацией, а также случаи, когда причина смерти была неясна или не указана.
1. Сбор данных
- Скачать SEER. Получение статистики 8.4.3 программного обеспечения из базы данных SEER (http://seer.cancer.gov/about/overview.html). После входа в программное обеспечение нажмите Сеанс списка дел > Данные и выберите базу данных Incidence SEER Research Plus, 17 реестров, ноябрь 2021 г. Sub (2000-2019).
- Нажмите на Выбор > Редактировать и выберите {Раса, Пол, Год Dx. Год диагноза} = '2004', '2005', '2006', '2007', '2008', '2009', '2010', '2011', '2012', '2013', '2014', '2015' И {Сайт и морфология. Код сайта ICD-O-3/WHO 2008} = '8490/3'.
- Затем нажмите на «Таблица» и в интерфейсе доступных переменных выберите «Возраст с одиноким возрастом и 100+», «Пол, Супружеский», «Код места ICD-O-3/WHO 2008», «Размер опухоли CS», «Региональный nodes_examined(1988+)), Региональный nodes_positive(1988+)), Производный AJCC Stage Group, 6-е издание (2004-2015), Производный AJCC T, 6-е издание (2004-2015), Производный AJCC N, 6-е издание (2004-2015), Производный AJCC M, 6-е издание (2004-2015), CEA, Перекодирование лучевой терапии, Перекодирование химиотерапии (да, нет/unk), Классификация смертности по причинам SEER, Перекодировка жизненного статуса (используется предельный срок исследования), Месяцы выживаемости, Год постановки диагноза.
- Наконец, нажмите « Вывод», присвойте данные имя и нажмите « Выполнить », чтобы вывести и сохранить данные. Подробный процесс включения показан на рисунке 1.
- Загрузка данных пациентов с колоректальным раком, за исключением случаев SRCC, для последующей внешней валидации. Нажмите на Выбор > Редактировать и выберите {Раса, Пол, Год Dx. Год постановки диагноза} = '2004', '2005', '2006', '2007', '2008', '2009', '2010', '2011', '2012', '2013', '2014', '2015' И {Основной сайт - помечен} = 'C18-C20'. Повторите шаги 1.3 и 1.4 для получения клинической патологической информации и исключения образцов с {Сайт и морфология. Перекодировка сайта ICD-O-3/WHO 2008} = '8490/3' из загруженного файла.
- Для сравнения обработайте несколько переменных. Классифицируйте состояние лимфатических узлов с помощью коэффициента лимфатических узлов (LNR) и логарифма вероятности положительных лимфатических узлов (LODDS).
- Определите LNR как отношение положительных лимфатических узлов (PLN) к общему количеству лимфатических узлов (TLN). Рассчитаем значение LODDS по формуле:
loge(количество PLN + 0,5) / (количество отрицательных лимфатических узлов (NLNs) + 0,5)
где 0.5 было добавлено, чтобы предотвратить бесконечный результат. Пороговые значения для LNR, LODDS и размера опухоли определяли с помощью программного обеспечения X-tile (версия 3.6.1) на основе метода минимального P-значения.
- Определите LNR как отношение положительных лимфатических узлов (PLN) к общему количеству лимфатических узлов (TLN). Рассчитаем значение LODDS по формуле:
- Откройте программу X-tile, нажмите «Файл» > «Открыть» и выберите файл данных, чтобы импортировать его в программу. После загрузки данных сопоставьте переменные: Цензор соответствует статусу выживания, Время выживания соответствует времени выживания, а marker1 — это переменная для анализа, чтобы обеспечить правильное совпадение данных.
- Затем нажмите « Выполнить > Каплана-Мейера > маркер1 », чтобы выполнить анализ выживаемости по Каплану-Мейеру и построить кривую выживаемости. Основываясь на разделении кривых выживаемости Каплана-Мейера, статистической значимости (например, p-значения) и клинической значимости, определите оптимальное пороговое значение и, наконец, запишите или экспортируйте результаты анализа.
- Разделите ЛНР на три группы: ЛНР 1 (≤0,16), ЛНР 2 (0,16 - 0,78) и ЛНР 3 (≥ 0,78). Разделите пациентов на три группы на основе LODDS: LODDS 1 (≤ -1,44), LODDS 2 (-1,44 - 0,86) и LODDS 3 (≥ 0,86).
- Классифицируют размер опухоли на три категории: ≤ 3,5 см, 3,5 - 5,5 см и ≥ 5,5 см. Преобразуйте возраст из непрерывной в категориальную переменную. Классифицируйте возраст пациентов на момент постановки первоначального диагноза как ≥60 лет и <60 лет. Классифицируйте локализацию опухоли на основе распределения опухолей кольцевой карциномы (SRCC) как опухоли правой ободочной кишки, левой ободочной кишки и прямой кишки. Правая ободочная кишка включает слепую кишку, восходящую ободочную кишку, печеночный изгиб и поперечную ободочную кишку, в то время как левая ободочная кишка включает селезеночный изгиб, нисходящую ободочную кишку, сигмовидную кишку и ректосигмоидное соединение.
- Для этого исследования случайным образом распределите в общей сложности 2409 подходящих данных пациентов с SRCC в учебную когорту (N = 1686) и проверяющую когорту (N = 723) в соотношении 7:3. Используйте следующий код для случайного разбиения и исходите data.csv из базы данных SEER. Файлы, сгенерированные после случайного разделения, будут использоваться для дальнейшего анализа.
Библиотека (каретка)
данные <- read.csv("data.csv")
set.seed(123)
train_indices <- createDataPartition(data$variable, p = 0.7, list = FALSE)
train_data <- данные[train_indices, ]
test_data <- данные[-train_indices, ]
write.csv(train_data, "traindata.csv", row.names = FALSE)
write.csv(test_data, "testdata.csv", row.names = FALSE)
2. Разработка и верификация ML-моделей
- Загрузите RStudio (2024.04.2+764) и программное обеспечение R (4.4.1). Откройте RStudio для запуска программного обеспечения R. Нажмите « Новый файл » и выберите «Скрипт R », чтобы создать новый программный интерфейс R. Введите соответствующий код в редакторе кода и нажмите « Выполнить », чтобы выполнить код.
- Используйте следующий код для скрининга переменных, включенных в модели машинного обучения, с помощью регрессионного анализа Кокса. Кроме того, изучите влияние стадирования LODDS, LNR и pN на выживаемость при раке (CSS) у пациентов с SRCC. traindata.csv – это данные, полученные из базы данных SEER.
библиотека («выживание»)
библиотека("survminer")
библиотека("RMS")
библиотека("dplyr")
данные <- read.csv("traindata.csv")
data$time=as.numeric(data$time)
data$status=as.numeric(data$status)
переменные <- c("Пол", "Возраст", "Раса", "Брачный", "Стадия", "T", "N", "M","Tumor_size", "LNR", "LODDS", "CEA","Radiation", "Chemotherapy", "Site")
данные <- данные %>%
mutate(across(all_of(variable), as.factor))
cox=coxph(Surv(время, статус) ~ data$T, data = данные)
коэффициенты cox$
pval=anova(cox)$Pr[2]
clean_data=data[,c(1:12, 14:18)]
get_coxVariable=function(your_data,index){cox_list=c() k=1
for (i in 1:index) {mod=coxph(Surv(time, status) ~ your_data[,i],data=your_data) pval=anova(mod)$Pr[2] print(pval) print(colnames(your_data)[i]) if (pval<0.05) {cox_list[k]=colnames(your_data))[i] k=k+1}}return(cox_list)}
variable_select=get_coxVariable(clean_data,15)
for(i в 1:15){print(variable_select[i])}
for (var in variable_select) {formula <- as.formula(paste("Surv(time, status) ~", var))cox_model <- coxph(formula, data = data) print(summary(cox_model))
GGFOREST (Кокс)
переменные <- c("Пол", "Возраст", "Раса", "Супружеский", "Стадия", "Т", "Н", "М", "Tumor_size", "LNR", "ЛОДДС", "Химиотерапия")
данные <- данные %>%
mutate(across(all_of(variable), as.factor))
cox=coxph(Surv(время, статус) ~ Пол+Возраст+Раса+Брак+T+N+M+Tumor_size+LNR+
LODDS+Химиотерапия, данные = данные)
ggforest(cox,data = данные)
ggplot_forest <- ggforest(cox, data = data) - Используйте следующий код для сравнения прогностических возможностей прогнозирования трех систем LN (LODDS, LNR и pN staging) в когортах обучения, валидации и внешней валидации.
Библиотека (RMS)
Библиотека (выживание)
Библиотека (Survminer)
библиотека(рискРегрессия)
Библиотека(gt)
train_data <- read.csv("train_data123.csv")
validation_data <- read.csv("test_data123.csv")
dd <- datadist(train_data)
options(datadist = "дд")
model_LNR <- cph(Surv(время, статус) ~ LNR, data = train_data, x = TRUE, y = TRUE)
model_LODDS <- cph(Surv(time, status) ~ LODDS, data = train_data, x = TRUE, y = TRUE)
model_pN <- cph(Surv(время, статус) ~ N, data = train_data, x = TRUE, y = TRUE)
calculate_performance <- функция(модель, данные) {pred <- predict(model, newdata = data) c_index_result <- concordance(Surv(data$time, data$status) ~ pred) c_index <- c_index_result$concordance aic <- AIC(model) bic <- BIC(model) return(c(C_index = round(c_index, 3), AIC = round(aic, 2), BIC = round(bic, 2)))}
calculate_performance <- function(model, data) {pred <- predict(model, newdata = data, type = "lp") concordance_result <- concordancefit(Surv(data$time, data$status), x = pred) c_index <- concordance_result$concordance ci_lower <- c_index - 1.96 * sqrt(concordance_result$var) ci_upper <- c_index + 1.96 * sqrt(concordance_result$var) aic <- AIC(model) bic <- BIC(model) return(c(C_Index = round(c_index, 3), CI_Lower = round(ci_lower, 3), CI_Upper = round(ci_upper, 3), AIC = round(aic, 2), BIC = round(bic, 2)))}
train_LNR <- calculate_performance(model_LNR, train_data)
train_LODDS <- calculate_performance(model_LODDS, train_data)
train_pN <- calculate_performance(model_pN, train_data)
model_LNR_val <- cph(Surv(время, статус) ~ LNR, data = validation_data, x = TRUE, y = TRUE)
model_LODDS_val <- cph(Surv(время, статус) ~ LODDS, data = validation_data, x = TRUE, y = TRUE)
model_pN_val <- cph(Surv(время, статус) ~ N, data = validation_data, x = TRUE, y = TRUE)
val_LNR <- calculate_performance(model_LNR_val, validation_data)
val_LODDS <- calculate_performance(model_LODDS_val, validation_data)
val_pN <- calculate_performance(model_pN_val, validation_data)
results <- data.frame(Variable = c("LNR", "LODDS", "pN"), Training_C_Index = c(paste(train_LNR["C_Index"], "(", train_LNR["CI_Lower"], ", ", train_LNR["CI_Upper"], ")", sep = ""), paste(train_LODDS["C_Index"], "(", train_LODDS["CI_Lower"], ", ", train_LODDS["CI_Upper"], ")", sep = ""), paste(train_pN["C_Index"], "(", train_pN["CI_Lower"], ", ", train_pN["CI_Upper"], ")", sep = "")), Training_AIC = c(train_LNR["AIC"], train_LODDS["AIC"], train_pN["AIC"]), Training_BIC = c(train_LNR["BIC"], train_LODDS["BIC"], train_pN["BIC"]), Validation_C_Index = c(paste(val_LNR["C_Index"], "(", val_LNR["CI_Lower"], ", ", val_LNR["CI_Upper"], ")", sep = ""), paste(val_LODDS["C_Index"], "(", val_LODDS["CI_Lower"], ", ", val_LODDS["CI_Upper"], ")", sep = ""), paste(val_pN["C_Index"], "(", val_pN["CI_Lower"], ", ", val_pN["CI_Upper"], ")", sep = "")), Validation_AIC = c(val_LNR["AIC"], val_LODDS["AIC"], val_pN["AIC"]), Validation_BIC = c(val_LNR["BIC"], val_LODDS["BIC"], val_pN["BIC"]))
results_table <- GT(результаты) %>%
tab_header(title = "Прогнозирование эффективности трех лимфатических узловых систем стадии") %>%
cols_label(Переменная = "Переменная",Training_C_Index = "С-индекс (95% ДИ) (Обучение)", Training_AIC = "АИК (Обучение)", Training_BIC = "БИК (Обучение)", Validation_C_Index = "С-индекс (95% ДИ) (Валидация)", Validation_AIC = "АИК (Валидация)", Validation_BIC = "БИК (Валидация)")
write.csv(результаты, "prediction_performance.csv", row.names = FALSE) - Используйте следующий код для построения модели XGBoost и создания столбчатых диаграмм относительной важности переменных, чтобы сравнить важность трех систем LN. Аналогичным образом создайте кривые ROC и калибровочные кривые. Данные получены из базы данных SEER.
Библиотека (xgboost)
Библиотека (каретка)
библиотека (pROC)
train_data <- read.csv("train_data.csv")
test_data <- read.csv("test_data.csv")
train_matrix <- xgb. DMatrix(data = as.matrix(train_data[, c('Возраст', 'T', 'N', 'M', 'LODDS', 'Химиотерапия')]), label = train_data$status)
test_matrix <- xgb. DMatrix(data = as.matrix(test_data[, c('Возраст', 'T', 'N', 'M', 'LODDS', 'Химиотерапия')]), label = test_data$status)
params <- list(booster = "gbtree", objective = "binary:logistic", eval_metric = "auc", eta = 0.1, max_depth = 6, подвыборка = 0.8, colsample_bytree = 0.8)
xgb_model <- xgb.train(params = params, data = train_matrix, nrounds = 100, watchlist = list(train = train_matrix), verbose = 1)
pred_probs <- predict(xgb_model, newdata = test_matrix)
pred_labels <- ifelse(pred_probs > 0.5, 1, 0)
conf_matrix <- confusionMatrix(as.factor(pred_labels), as.factor(test_data$status))
roc_curve <- roc(test_data$status, pred_probs)
auc_value <- auc(roc_curve)
ci_auc <- ci.auc(roc_curve)
чувствительность <- conf_matrix$byClass["Чувствительность"]
специфичность <- conf_matrix$byClass["Специфичность"]
точность <- conf_matrix$overall["Точность"]
ppv <- conf_matrix$byClass["Значение Pos Pred"]
npv <- conf_matrix$byClass["Отрицание значения преда"]
result_table <- data.frame(Model = "XGBoost", AUC = sprintf("%.3f (%.3f-%.3f)", auc_value, ci_auc[1], ci_auc[3]), Чувствительность = sprintf("%.3f", чувствительность), Специфичность = sprintf("%.3f", специфичность), Точность = sprintf("%.3f", точность), PPV = sprintf("%.3f", ppv), NPV = sprintf("%.3f", npv))
write.csv(result_table, "xgboost_model_performance.csv", row.names = FALSE)
roc_df <- data.frame(FPR = 1 - roc_curve$особенности, TPR = roc_curve$чувствительность)
roc_plot <- ggplot(roc_df, aes(x = FPR, y = TPR)) +geom_line(color = "steelblue", size = 1.2) + geom_abline(intercept = 0, slope = 1, linetype = "пунктирный", color = "серый") + annotate("text", x = 0.9, y = 0.2, label = paste("AUC =", round(auc_value, 3)), size = 5, color = "черный") + labs(title = "Кривая ROC для модели XGBoost", x = "Коэффициент ложных срабатываний", y = "Коэффициент истинного срабатывания") + theme_minimal() + theme(panel.border = element_rect(color = "черный", fill = NA, размер = 1))
calibration_data <- data.frame(Status = as.factor(test_data$status), pred_probs = pred_probs)
calib_model <- калибровка(Статус ~ pred_probs, данные = calibration_data, класс = "1", разрезы = 5)
ggplot(calib_model$data, aes(x = середина, y = процент)) + geom_line(color = "steelblue", size = 1) + geom_point(color = "красный", size = 2) + geom_abline(intercept = 0, slope = 1, linetype = "пунктирный", color = "черный") +labs(title = "Калибровочная кривая для модели XGBoost", x = "прогнозируемая вероятность", y = "наблюдаемая пропорция") + theme_minimal() + theme(panel.border = element_rect(color = "черный", fill = NA, размер = 0.5)) - Используйте следующий код для построения радиочастотной модели и создания гистограмм относительной важности переменных, тем самым сравнивая важность трех систем LN. Аналогичным образом создайте кривые ROC и калибровочные кривые. Данные получены из базы данных SEER.library(randomForest)
Библиотека (DPLYR)
библиотека(ggplot2)
библиотека (pROC)
Библиотека (каретка)
Библиотека (RMS)
Поезд <- read.csv("train_data.csv")
Испытано <-read.csv("test_data.csv")
trainset$status=factor(trainset$status)
variables1 <- c("Возраст", "T", "N", "M", "LODDS", "Химиотерапия")
Trainset <- Trainset %>%
mutate(across(all_of(variables1), as.numeric))
testsed$status=factor(tested$status)
протестировано <- протестировано %>%
mutate(across(all_of(variables1), as.numeric))
RF=randomForest(trainset$status ~ Возраст + T + N + M + LODDS + Химиотерапия, data=trainset,ntree=100,importance=TRUE,proximity=TRUE)
imp=важность(РЧ)
varImpPlot(РЧ)
impvar=rownames(imp)[order(imp[,4],decreasing = TRUE)]
importance_df <- as.data.frame(imp)
importance_df$Variables <- rownames(importance_df)
importance_plot <- ggplot(importance_df, aes(x = reorder(Variables, MeanDecreaseAccuracy), y = MeanDecreaseAccuracy)) +geom_bar(stat = "identity", fill = "steelblue") +coord_flip() + labs(title = "Переменная важность", x = "Переменные", y = "Среднее снижение точности") + theme_minimal()
pred_probs <- predict(RF, testset, type = "prob")[,2]
roc_obj <- roc(testset$status, pred_probs)
auc_value <- auc(roc_obj)
roc_plot <- ggplot() +geom_line(aes(x = 1 - roc_obj$specificities, y = roc_obj$sensitivities), color = "steelblue", size = 1.2) +geom_abline(intercept = 0, slope = 1, linetype = "пунктирный", color = "серый") + annotate("text", x = 0.8, y = 0.2, label = paste("AUC =", round(auc_value, 3)), color = "черный", размер = 5, hjust = 0) + labs(title = "кривая ROC для модели случайного леса", x = "Процент ложных срабатываний", y = "Процент истинных срабатываний") +theme_minimal() + theme(panel.border = element_rect(color = "черный", fill = NA, размер = 1))
calibration_data <- data.frame(pred_probs = pred_probs, status = testsed$status)
calib_model <- калибровка (статус ~ pred_probs, данные = calibration_data, класс = "1", обрезки = 5)
calib_df <- as.data.frame(calib_model[["data"]])
calib_df$mid <- calib_df$midpoint
calib_df$% <- calib_df$%
calibration_plot <- ggplot(calib_df, aes(x = mid, y = Percent)) + geom_line(color = "steelblue", size = 1.2) + geom_point(color = "steelblue", size = 3) + geom_abline(intercept = 0, slope = 1, linetype = "пунктирный", color = "черный", размер = 0.8) + labs(title = "Калибровочная кривая для случайного леса", x = "Прогнозируемая вероятность", y = "Фактическая вероятность") + theme_minimal() + theme(panel.border = element_rect(color = "черный", fill = NA, размер = 1), plot.title = element_text(hjust = -0.05, vjust = -1.5, face = "жирный", размер = 12) )
rf_probs <- predict(RF, newdata=tested, type="prob")[, 2]
rf_auc <- roc(проверено$status, rf_probs)
auc_value <- auc(rf_auc)
ci_auc <- ci.auc(rf_auc)
rf_predictions <- predict(RF, newdata=протестировано)
conf_matrix <- confusionMatrix(rf_predictions, testsed$status)
чувствительность <- conf_matrix$byClass["Чувствительность"]
специфичность <- conf_matrix$byClass["Специфичность"]
точность <- conf_matrix$overall["Точность"]
ppv <- conf_matrix$byClass["Значение Pos Pred"]
npv <- conf_matrix$byClass["Отрицание значения преда"]
result_table <- data.frame(Model = "RF", AUC = sprintf("%.3f (%.3f-%.3f)", auc_value, ci_auc[1], ci_auc[3]), Чувствительность = sprintf("%.3f", чувствительность), Специфичность = sprintf("%.3f", специфичность), Точность = sprintf("%.3f", точность), PPV = sprintf("%.3f", ppv), NPV = sprintf("%.3f", npv))
write.csv(result_table, "RF_model_performance.csv", row.names = FALSE) - Используйте следующий код для построения модели NN и создания столбчатых диаграмм относительной важности переменных, тем самым сравнивая важность трех систем LN. Аналогичным образом создайте кривые ROC и калибровочные кривые. Данные получены из базы данных SEER.library(nnet)
Библиотека (каретка)
библиотека (pROC)
библиотека(ggplot2)
train_data <- read.csv("train_data.csv")
test_data <- read.csv("test_data.csv")
train_data$status <- as.factor(train_data$status)
test_data$status <- as.factor(test_data$status)
особенности <- c ("Возраст", "Т", "Н", "М", "ЛОДДС", "Химиотерапия")
x_train <- train_data[, функции]
y_train <- train_data$статус
x_test <- test_data[, функции]
y_test <- test_data$статус
nn_model <- nnet(status ~ Age + T + N + M + LODDS + Химиотерапия, data = train_data, size = 5, decay = 0.01, maxit = 200)
pred_probs <- predict(nn_model, newdata = x_test, type = "raw")
pred_labels <- ifelse(pred_probs > 0.5, 1, 0)
roc_curve <- roc(as.numeric(y_test), pred_probs)
auc_value <- auc(roc_curve)
auc_ci <- ci.auc(roc_curve)
auc_text <- paste0(round(auc_value, 3), " (", round(auc_ci[1], 3), "-", round(auc_ci[3], 3), ")")
conf_matrix <- confusionMatrix(as.factor(pred_labels), y_test)
точность <- conf_matrix$overall["Точность"]
чувствительность <- conf_matrix$byClass["Чувствительность"]
специфичность <- conf_matrix$byClass["Специфичность"]
ppv <- conf_matrix$byClass["Значение Pos Pred"]
npv <- conf_matrix$byClass["Отрицание значения преда"]
performance_table <- data.frame(Metric = c("AUC (95% CI)", "Точность", "Чувствительность", "Специфичность", "PPV", "NPV"),Value = c(auc_text, round(accuracy, 3), round(sensitivity, 3), round(specificity, 3), round(ppv, 3), round(npv, 3)))
write.csv(performance_table, "NN_performance_table.csv", row.names = FALSE)
roc_curve <- roc(y_test, pred_probs)
auc_value <- auc(roc_curve)
roc_plot <- ggplot() + geom_line(aes(x = 1 - roc_curve$specificities, y = roc_curve$sensitivities), color = "steelblue", size = 1.2) +geom_abline(intercept = 0, slope = 1, linetype = "пунктирный", color = "серый") + annotate("text", x = 0.8, y = 0.2, label = paste("AUC =", round(auc_value, 3)), color = "черный", размер = 5, hjust = 0) + labs(title = "ROC-кривая для модели нейронной сети", x = "Частота ложных срабатываний", y = "Истинная положительная скорость") + theme_minimal() + theme(panel.border = element_rect(color = "черный", fill = NA, размер = 1))
calibration_data <- data.frame(pred_probs = pred_probs, status = as.numeric(y_test) - 1)
calibration_data$pred_probs <- as.numeric(calibration_data$pred_probs)
calibration_data$calibration_bin <- cut(calibration_data$pred_probs, breaks = seq(0, 1, by = 0.2), include.lowest = TRUE)
calibration_summary <- aggregate(status ~ calibration_bin, data = calibration_data, FUN = mean)
calibration_summary$pred_mean <- aggregate(pred_probs ~ calibration_bin, data = calibration_data, FUN = mean)$pred_probs
calibration_plot <- ggplot(calibration_summary, aes(x = pred_mean, y = статус)) + geom_line(color = "steelblue", size = 1.2) + geom_point(color = "red", size = 3) + geom_abline(intercept = 0, slope = 1, linetype = "пунктирный", color = "черный", размер = 0.8) + labs(title = "калибровочная кривая для нейронной сети", x = "прогнозируемая вероятность", y = "фактическая вероятность") + theme_minimal() + theme(panel.border = element_rect(color = "черный", fill = NA, размер = 1))
nn_var_importance <- varImp(nn_model)
importance_df <- data.frame(Feature = rownames(nn_var_importance), Importance = nn_var_importance$Overall )
importance_plot <- ggplot(importance_df, aes(x = reorder(Feature, Importance), y = Importance)) + geom_bar(stat = "identity", fill = "steelblue") + coord_flip() + labs(title = "Переменная важность для нейронной сети", x = "Функции", y = "Важность") + theme_minimal()
3. Разработка и верификация конкурирующей модели риска
- Используйте следующий код для выполнения одномерного анализа и построения кривой кумулятивной функции заболеваемости (CIF). data.csv – это данные, полученные из базы данных SEER. Способ сохранения последующих изображений будет таким же, как и на этом шаге. Заменяйте Site в коде один за другим на другие факторы, чтобы выполнить одномерный анализ по всем факторам.
Библиотека (Tidycmprsk)
библиотека(gtsummary)
библиотека(ggplot2)
Библиотека (ggsurvfit)
Библиотека (GGPRISM)
AA <- read.csv("data.csv")
cif2 <- tidycmprsk::cuminc(Surv(time, Status1) ~Site, data = aa)
tidy(cif2,times = c(12,24,36,48,60))
tbl_cuminc(cif2, times =c(12,24,36,48,60), outcomes = c("CSS", "OSS"),estimate_fun = NULL, label_header = "**{time/12}-year cuminc**") %>%
add_p() %>%
add_n(location = "уровень")
cuminc_plot <- ggcuminc(cif2, result = c("CSS", "OSS"), size = 1.5) + labs(x = "time") +add_quantile(y_value = 0.20, size = 1) + scale_x_continuous(breaks = seq(0, 84, by = 12), limits = c(0, 84)) +scale_y_continuous(label = scales::p ercent, breaks = seq(0, 1, by = 0.2), limits = c(0, 1)) + theme_prism() + theme(legend.position = c(0.2, 0.8), panel.grid = element_blank(),panel.grid.major.y = element_line(цвет = "серый80")) + theme(legend.spacing.x = unit(0.1, "cm"), legend.spacing.y = unit(0.01, "cm")) + theme(axis.ticks.length.x = unit(-0.2, "cm"), axis.ticks.x = element_line(цвет = "черный", размер = 1, конец линии = 1)) + theme(axis.ticks.length.y = unit(-0.2, "см"), axis.ticks.y = element_line(цвет = "черный", размер = 1, конец линии = 1)) - Используйте следующий код для выполнения многомерного анализа и визуализации. Этот data1.csv исходит из результатов предыдущего кода. После выполнения кода нажмите «Экспорт», затем нажмите «Сохранить как PDF» и, наконец, нажмите «Сохранить », чтобы сохранить изображение.
Библиотека (Tidycmprsk)
библиотека(gtsummary)
AA <-read.csv('data1.csv')
for (i in names(aa)[c(1:16, 19)]){aa[,i] <- as.factor(aa[,i])}
mul1Таблица 2 <- моль1 %>%
gtsummary::tbl_regression(exponentiate = TRUE) %>%
add_n(location = "уровень"); Таблица 2
table_df <-as_tibble (таблица 2)
Вкладка <- стол2$table_body
tab1 <- tab[,c(12,19,20,22:29)] - Используйте следующий код для построения номограммы, кривой ROC и калибровочной кривой. После обучения модели с использованием данных из обучающей когорты используйте данные когорт проверки и внешней проверки для проверки model.library(QHScrnomo). Данные внешней когорты состоят из образцов колоректального рака, отличных от кольцевоклеточного рака, которые были отобраны на этапе 1.4.
Библиотека (RMS)
библиотека(timeROC)
Библиотека (выживание)
AA <-read.csv('data3.csv')
for (i in names(aa)[c(1:16, 19)]){aa[,i] <- as.factor(aa[,i])}
dd <- datadist(aa)
options(datadist = "дд")
mul <- cph(Surv(time, Status1 == 1) ~ T + N + M + LODDS + Site, data = aa, x = TRUE, y = TRUE, surv = TRUE)
m3 <- crr.fit(моль, failcode = 1, cencode = 0)
nomo <-Newlabels(fit = m3, labels =c(T="T", N= "N", M = "M", LODDS = "LODDS", Site = "Site"))
nomoc("N0","N1","N2"),M=c("M0","M1"),LODDS=c
("LODDS1","LODDS2","LODDS3"),Site=
c("RSC","LSC","Rectum")))
nomogram.crr(fit =nomo , lp = F, xfrac = 0.3, fun.at =seq(from=0, to=1, by= 0.1) , failtime =c(12,36,60), funlabel = c("1-летний CSS Cumulative Incigence","3-летний CSS Cumulative Incigence","5-летний CSS Cumulative Incigence"))
time_points <- c(12, 36, 60)
pred_risks_list <- lapply(time_points, function(time_point) {predict(m3, newdata = aa, type = "risk", time = time_point)})
pred_risks_df <- data.frame(do.call(cbind, pred_risks_list))
colnames(pred_risks_df) <- paste("risk_at", time_points, "месяцев", sep = "_")
roc_1year <- timeROC(T = aa$time, delta = ifelse(aa$Status1 == "CSS", 1, 0), marker = pred_risks_df$risk_at_12_months, cause = 1, times = 12, iid = TRUE)
roc_3year <- timeROC(T = aa$time, delta = ifelse(aa$Status1 == "CSS", 1, 0), marker = pred_risks_df$risk_at_36_months, cause = 1, times = 36, iid = TRUE)
roc_5year <- timeROC(T = aa$time, delta = ifelse(aa$Status1 == "CSS", 1, 0), marker = pred_risks_df$risk_at_60_months, cause = 1,times = 60, iid = TRUE)
legend("bottomright",legend = c("1 год CSS", "3 год CSS", "5 год CSS"), col = c("#BF1D2D", "#262626", "#397FC7"), lwd = 2)
sas.cmprsk(m3,время = 36)
set.seed(123)
aa$pro <- tenf.crr(m3,time = 36)
cindex(prob = aa$pro, fstatus = aa$Status1, ftime = aa$time, type = "crr", failcode = 1, cencode = 0, tol = 1e-20)
groupci(x=aa$pro, ftime = aa$time, fstatus = aa$Status1, failcode = 1, cencode = 0, ci = TRUE, g = 5, m = 1000, u = 36, xlab = "Прогнозируемая вероятность", ylab = "Фактическая вероятность", lty=1, lwd=2, col="#262626",xlim=c(0,1.0), ylim=c(0,1.0), add =TRUE)
Результаты
Характеристика пациентов
Это исследование было сосредоточено на пациентах с диагнозом колоректальный SRCC с использованием данных из базы данных SEER за период с 2004 по 2015 год. Критерии исключения включали пациентов со временем выживаемости менее одного месяца, пациентов с неполной клинико-патологической информацией, а также случаи, когда причина смерти была неясна или не указана. В общей сложности 2409 пациентов с колоректальным SRCC, которые соответствовали критериям включения, были случайным образом разделены на обучающую когорту (N = 1686) и валидационную когорту (N = 723). Демографические и клинические параметры когорт обучения и валидации были проанализированы с использованием программного обеспечения R, как показано в таблице 1. Среди всех включенных пациентов большинство были старше 60 лет, с одинаковым количеством пациентов мужского и женского пола. Большинство пациентов были белыми. Более половины пациентов (56%) состояли в браке. Большинство опухолей были классифицированы как III-IV (76%). У большинства пациентов (82%) размеры опухоли превышали 3,5 сантиметра, а большинство пациентов относились к группе LODDS1 (42%). Во всей когорте высокая доля пациентов (53%) получала химиотерапию. Первичные опухоли в основном располагались в правой толстой кишке (67%). После рандомизации не было существенных различий в исходных характеристиках между двумя группами статистически.
Идентификация прогностических клинических факторов, входящих в ML-модели
Сначала мы проверили значимые переменные для включения в модель машинного обучения с помощью регрессионного анализа Кокса. Результаты одномерной регрессии Кокса показали, что время выживаемости в значительной степени коррелировало с определенными клиническими переменными, включая пол, возраст, расу, семейное положение, стадию AJCC, стадию pT, стадию pN, стадию pM, размер опухоли, уровень CEA, классификацию LNR, классификацию LODDS, а также то, получал ли пациент лучевую терапию или химиотерапию (Таблица 2). Примечательно, что стадирование LNR, LODDS и pN продемонстрировали статистически значимые отношения рисков (HR), что указывает на то, что эти три системы стадирования LN связаны с прогнозом. Последующий многофакторный регрессионный анализ Кокса был проведен для дальнейшего определения связи между стадированием pN, LODDS, LNR и CSS у пациентов с SRCC. Результаты показали, что LODDS, LNR и pN статус значительно влияют на CSS у пациентов с SRCC (рис. 2).
Сравнение систем LN
Прогностические способности трех систем LN были схожими как в когорте обучения, валидации, так и во внешней валидации (образцы колоректального рака, отличные от кольцевой карциномы, которые были отобраны на этапе 1.4; Таблица 3). В обучающей когорте индексы C для LNR, LODDS и pN составили 0,309, 0,308 и 0,337 соответственно, в то время как в валидационной когорте индексы C составили 0,288, 0,279 и 0,319 соответственно. В когорте внешней валидации С-индексы для LNR, LODDS и pN составили 0,419, 0,420 и 0,424 соответственно. Кроме того, значения AIC для каждой системы в обучающей когорте составили 12667,56, 12670,57 и 12731,89, а в валидационной когорте — 4575,36, 4559,13 и 4613,20 соответственно. В когорте внешней валидации значения AIC для каждой системы были следующими: 106554,68 для LODDS, 106581,85 для LNR и 106915,45 для стадии pN. Эти результаты свидетельствуют о том, что различия в качестве дискриминации между тремя системами минимальны. Поэтому мы использовали методы машинного обучения — RF, XGBoost и NN — для дальнейшего определения оптимальной системы LN с точки зрения прогностических возможностей. Этот анализ включал переменные, которые были значимы в одномерной регрессии Кокса и, по крайней мере, одну многомерную модель (pN, LODDS или LNR) для построения моделей машинного обучения, включая pN, pT, pM, возраст, расу, классификацию LNR, классификацию LODDS, а также то, получал ли пациент лучевую терапию или химиотерапию.
Мы построили модели RF, XGBoost и NN с использованием обучающего набора данных. Значения важности для каждой переменной показаны на рисунке 3. В RF и XGBoost наибольшее значение продемонстрировал ЛНР, в то время как LODDS также продемонстрировал значительное значение. Тем не менее, в модели NN LODDS продемонстрировали лучшую прогностическую способность по сравнению с pN и LNR. Рассмотрев объединенные результаты трех подходов к машинному обучению, мы пришли к выводу, что система LODDS может быть лучшей системой для оценки статуса ЛУ у пациентов с SRCC.
Производительность моделей машинного обучения
Как показано в таблице 4 и на рисунке 4A-C, все три модели смогли эффективно прогнозировать прогноз, а AUC трех моделей варьировались от 0,777 до 0,851 в тестовом наборе данных (XGBoost: AUC = 0,820, 95% ДИ =0,789-0,851; РФ: AUC = 0,819, 95% ДИ = 0,788-0,850; NN: AUC = 0,809, 95% ДИ = 0,777-0,841). Модели XGBoost, RF и NN показали большую специфичность (0,82, 0,825, 0,815) и точность (0,762, 0,763, 0,757). Калибровочные кривые показаны на рисунке 5D-F.
Построение и валидация модели конкурентных рисков
Учитывая, что модели машинного обучения не учитывали влияние конкурирующих факторов риска, мы построили конкурирующую модель риска для дальнейшего выявления LN-системы, которая показала наилучшие результаты с точки зрения прогностических возможностей. Специфическая для рака выживаемость (CSS) представляет собой смерть от рака, в то время как общая выживаемость (OSS) учитывает смертность от других причин, выступая в качестве конкурирующих событий риска. Мы использовали одномерные и многомерные конкурирующие модели риска для анализа прогностических факторов для CSS в обучающей когорте. Одномерная конкурирующая модель риска показала, что прогностические факторы для CSS включали пол, возраст, расу, семейное положение, стадию AJCC, стадию TNM, размер опухоли, классификацию LNR, классификацию LODDS, уровень CEA, получал ли пациент лучевую терапию или химиотерапию, а также расположение первичной опухоли. Наконец, многофакторный конкурирующий анализ риска, стадия T, стадия N, стадия M, классификация LODDS и расположение первичной опухоли были идентифицированы как пять независимых прогностических маркеров для пациентов с колоректальным SRCC. Результаты одномерного и многомерного конкурирующего анализа риска представлены в таблице 5, а соответствующие кривые кумулятивной функции заболеваемости (CIF) для независимых факторов риска представлены на рисунке 5. Основываясь на пяти значимых переменных (стадия T, стадия N, стадия M, классификация LODDS и локализация первичной опухоли), мы разработали прогностическую номограмму (рис. 6A). Мы обнаружили, что по сравнению с pN, LODDS показал более высокий вес. Этот вывод согласуется с предыдущими результатами, предполагая, что система LODDS является лучшей системой для оценки статуса ЛУ у пациентов с SRCC.
Для оценки точности модели были построены калибровочные кривые (рис. 6B-D). Результаты показали, что модель показала хорошие результаты в прогнозировании общей выживаемости пациентов через 1, 3 и 5 лет. Подгонка кривых к линии 45° демонстрирует сильную согласованность модели. Результаты оценки ROC-кривой для номограммы (рис. 5E-G) показали, что площадь под кривой (AUC) для прогнозов через 1, 3 и 5 лет была больше 0,75. Эти результаты свидетельствуют о том, что кривые прогнозирования на 3 и 5 лет продемонстрировали значительные преимущества, указывающие на то, что номограмма имеет ценное клиническое применение и референсную ценность.

Рисунок 1: Блок-схема, представляющая процесс скрининга в базе данных SEER. В соответствии с нашими критериями включения и исключения мы успешно отобрали пациентов с колоректальным раком из базы данных SEER для последующей серии анализов на основе R. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.

Рисунок 2: Ассоциация стадий LNR, LODDS и pN с CSS в обучающей когорте. На этом рисунке показаны результаты многомерного регрессионного анализа Кокса для стадирования (A) LNR, (B) LODDS и (C) pN, оцениваемые наряду с другими независимыми прогностическими факторами. Анализ включает отношения рисков (ОР) и 95% доверительные интервалы (ДИ). Результаты показывают, что LNR, LODDS и pN статус являются значимыми прогностическими факторами для специфической для рака выживаемости у пациентов с SRCC, при этом все значения HR демонстрируют статистическую значимость (p < 0,05). *p < 0,05, **p < 0,01, ***p < 0,001. Полосы погрешностей представляют 95% ДИ. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.

Рисунок 3: Относительная важность переменных. (A) модель XGBoost, (B) радиочастотная модель и (C) модель NN. На этом рисунке показана относительная важность переменных. В моделях XGBoost и RF наибольшее значение имел LNR, а LODDS также продемонстрировал значительную значимость. И наоборот, в модели NN LODDS продемонстрировал превосходную прогностическую способность по сравнению с pN и LNR. На основании объединенных результатов трех моделей предполагается, что система LODDS является наиболее эффективной для оценки статуса ЛУ у пациентов с СРКК. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
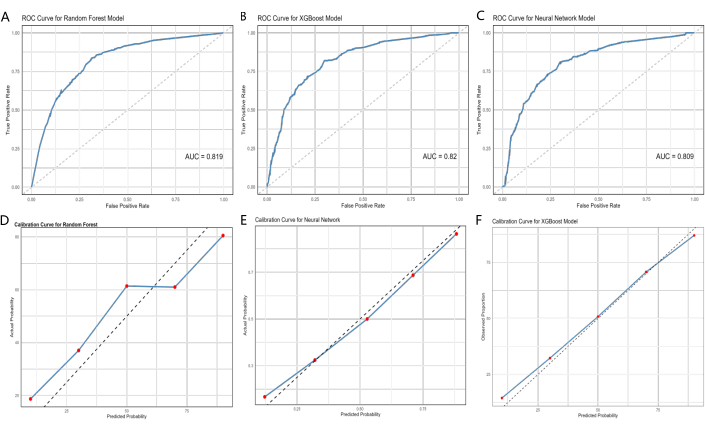
Рисунок 4: ROC-кривые и калибровочные кривые моделей машинного обучения, XGBoost и eXtreme градиентного бустинга. (A, D) обучающая когорта, (B, E) валидационная когорта и (C, F) внешняя валидационная когорта. Значение площади под кривой (AUC), близкое к 1, указывает на более высокую производительность классификации модели. Полосы погрешностей представляют 95% доверительный интервал для прогнозируемой вероятности фактического наступления события. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
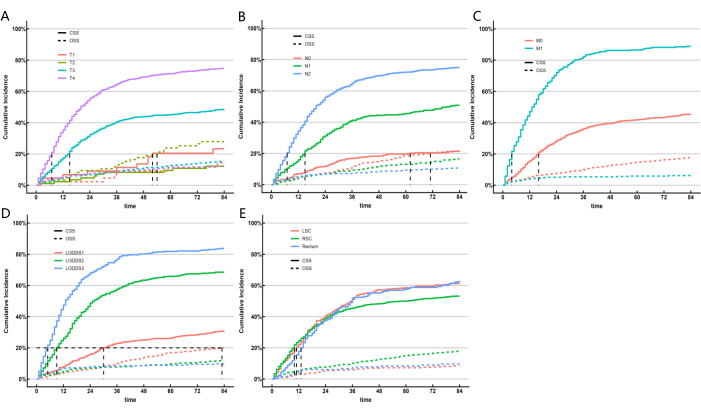
Рисунок 5: Кумулятивные оценки заболеваемости смертностью в соответствии с характеристиками пациента на колоректальном SRCC. КИФ подгрупп. Был применен тест Грея. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
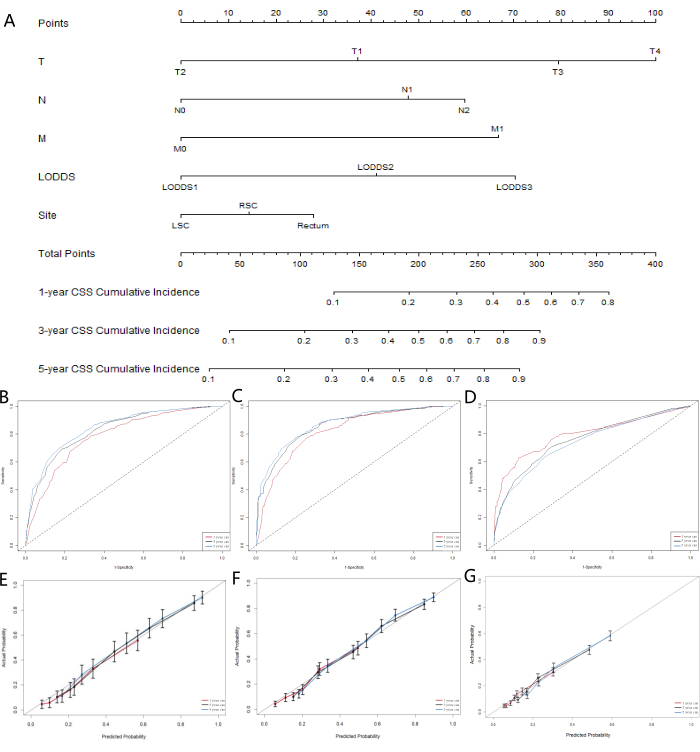
Рисунок 6: Разработка и валидация номограммы для CSS у пациентов с колоректальным SRCC. (A) Конкурирующая номограмма риска, прогнозирующая 1-летнюю, 3-летнюю и 5-летнюю вероятность выживаемости пациентов с колоректальным SRCC при раке. Калибровочные кривые номограмм для прогнозирования 1-, 3-, 5-летней выживаемости при раке в (B) обучающей когорте, (C) валидационной когорте и (D) внешней валидационной когорте. Кривые рабочих характеристик приемника для прогнозирования 1-, 3- и 5-летней выживаемости при раке в (E) обучающей когорте, (F) валидационной когорте и (G) внешней валидационной когорте. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Таблица 1: Клиническая характеристика пациентов с колоректальным SRCC. Пожалуйста, нажмите здесь, чтобы скачать эту таблицу.
Таблица 2: Одномерный регрессионный анализ CSS по Коксу в обучающей когорте. Пожалуйста, нажмите здесь, чтобы скачать эту таблицу.
Таблица 3: Прогностическая эффективность трех систем стадирования лимфатических узлов. Пожалуйста, нажмите здесь, чтобы скачать эту таблицу.
Таблица 4: Прогностическая эффективность моделей в когорте валидационной проверки и когортах внешней валидации. Пожалуйста, нажмите здесь, чтобы скачать эту таблицу.
Таблица 5: Одномерный и многомерный конкурирующий анализ риска смертности от колоректального SRCC при раке в обучающей когорте. Пожалуйста, нажмите здесь, чтобы скачать эту таблицу.
Обсуждение
Колоректальный рак (КРР) SRCC является редким и особым подтипом колоректального рака с плохим прогнозом. Поэтому необходимо уделять больше внимания прогнозу пациентов с СРКК. Точное прогнозирование выживаемости пациентов с SRCC имеет решающее значение для определения их прогноза и принятия индивидуальных решений о лечении. В данном исследовании мы изучили взаимосвязь между клиническими особенностями и прогнозом у пациентов с SRCC и определили оптимальную систему стадирования LN для пациентов с SRCC из базы данных SEER. Насколько нам известно, это первое исследование, в котором была определена подходящая система LN для пациентов с колоректальным SRCC путем комплексного использования машинного обучения и конкурирующих методов анализа риска, а также построена номограмма для прогностического прогнозирования.
Количество метастатических ЛУ у пациентов с КРР является важным показателем прогноза и рецидива. Точное определение стадии ЛУ играет ключевую роль в определении стратегии лечения и прогноза для пациентов с СРКК. LNR и LODDS являются альтернативными методами, используемыми для оценки участия LN в GC, улучшения систем стадирования и предоставления более точной прогностической информации10,13. Мы выявили корреляцию между стадией LODDS, LNR и pN с CSS у пациентов с SRCC с использованием базы данных SEER. Прогностические способности этих трех систем LN (LNR, LODDS и pN) сравнивались с использованием AUC, AUC, BIC и индексов C. Однако различия между ними были минимальными. Поэтому мы использовали три метода машинного обучения — Xgboost, RF и NN — для выбора наиболее важных функций в качестве оптимальной LN-системы. Основываясь на объединенных результатах трех методов, мы определили LODDS как подходящую LN-систему.
Тем не менее, OSS является конкурирующим событием риска, которое влияет на прогноз пациентов с CSS. В процессе скрининга переменных на предмет включения в модель машинного обучения с использованием метода регрессии Кокса не учитывалось влияние OSS, что может привести к неточной оценке отношений рисков12. Таким образом, для дальнейшего определения оптимальной системы оценки ЛУ для пациентов с SRCC мы построили конкурирующую модель риска. Полученные результаты еще раз подтвердили, что система стадирования LODDS предоставляет более точную прогностическую информацию по сравнению с системой pN. За время наблюдения из 2409 пациентов 1339 (56%) умерли от ПСС, а 464 (19%) умерли от СОСС. Кроме того, мы также разработали конкурирующую диаграмму риска для прогнозирования смертности от рака через 1 год, 3 года и 5 лет. Мы считаем, что эта модель имеет существенное значение для клинических исследований с участием пациентов с колоректальным SRCC. Хотя Американский объединенный комитет по раку рекомендует систему TNM в качестве системы определения стадии для всех гистологических типов колоректального рака, она в основном используется для определения стадии колоректальной аденокарциномы. Стадия AJCC N ограничена TLN, в то время как LNR не учитывает влияниеNLN 13,14,15,16. Отчеты указывают на то, что LODDS меньше подвержен влиянию TLN и считает числоNLN 10,17. Scarinci et al. продемонстрировали, что LODDS предсказывает ОВ у пациентов с КРР лучше, чем стадирование LNR и pN, и предположили, что будущие исследования должны подтвердить его роль в различных подтипах КРР18. В этом исследовании мы обнаружили, что LODDS оказывает значительное прогностическое прогностическое влияние на ССС при колоректальном SRCC. Таким образом, LODDS может быть ценным инструментом для оценки диссекции лимфатических узлов и прогноза у пациентов с колоректальным SRCC. Несмотря на то, что оптимальный порог для LODDS еще не установлен, он зарекомендовал себя как самая надежная система этапирования LN. В связи с растущим вниманием к ЛОДДС, принято считать, что в обозримом будущем он получит широкое признание в клинических условиях.
Наше исследование показало, что расположение первичной опухоли является важным прогностическим фактором для ЦСС, со значительно худшим прогнозом для ректального SRCC, что согласуется с предыдущими исследованиями 12,19,20. Ректальный SRCC может иметь уникальные клинические, патологические и молекулярные характеристики21,22, требующие дальнейшего изучения. Диаграммы, полученные на основе прогностических моделей, являются ключевыми и эффективными инструментами для принятия клинических решений и консультирования пациентов. Насколько нам известно, это исследование является первым, в котором интегрированы модели ML и конкурирующие модели риска для изучения оптимальной системы стадирования LN для пациентов с SRCC. Мы разработали и валидировали три модели машинного обучения для прогнозирования прогноза пациентов с SRCC. В тестовом наборе данных модели XGBoost, RF и NN показали хорошую прогностическую прогностическую прогностическую эффективность на основе значений AUC и соответствующих метрик. Таким образом, ML-модели могут помочь в принятии решений о лечении пациентов с SRCC путем прогнозирования прогноза. Кроме того, мы создали конкурирующие диаграммы риска на основе моделей пропорционального риска для анализа прогностических факторов для колоректального SRCC и оценки роли LODDS в нем. Мы использовали индекс С и калибровочные кривые для оценки прогностической эффективности номограммы. На диаграмме отображались общие клинические переменные, такие как первичный очаг опухоли и группировка LODDS. Кроме того, построенная нами номограмма является эффективным методом прогнозирования CSS на 1, 3 и 5 лет у пациентов с колоректальным SRCC. Этот инструмент может помочь клиницистам в проведении точных, тщательных и своевременных прогностических оценок для каждого пациента с колоректальным SRCC, что позволяет им формулировать персонализированные планы лечения23.
Наконец, это исследование имеет несколько ограничений. Во-первых, пациенты в исследовании были диагностированы в период с 2004 по 2015 год, что привело к относительно короткому периоду наблюдения. Мы ожидаем, что более длительный период наблюдения поможет повысить точность прогнозов моделей. Во-вторых, используемый здесь дизайн исследования является ретроспективным и опирается на данные, полученные из базы данных SEER, что может привести к некоторым врожденным смещениям. Некоторая информация, такая как расположение метастатических LN, не регистрировалась. Наконец, большинство пациентов в этом исследовании были белыми, что потребовало более широких исследований с участием различных групп населения для подтверждения и усиления этих результатов.
Заключение
Исследование показало, что LODDS проявляют сильную прогностическую прогностическую способность к колоректальному SRCC. Основываясь на этом фундаменте, мы разработали номограмму на основе конкурирующей модели риска для прогнозирования общей выживаемости с интервалами 1 год, 3 года и 5 лет для пациентов с колоректальным SRCC. После серии оценок и внутренних валидаций номограмма продемонстрировала значительную клиническую применимость и ценность, послужив руководством для клиницистов при принятии решений о лечении. Кроме того, мы построили три модели машинного обучения. Эти подходы ML могут расширить прогностические прогностические возможности SRCC и помочь врачам понять, как ML может быть использовано для оптимизации стратегий лечения и последующего наблюдения.
Раскрытие информации
Авторы не имеют финансовых конфликтов интересов, которые можно было бы раскрыть.
Благодарности
Никакой
Материалы
| Name | Company | Catalog Number | Comments |
| SEER database | National Cancer institiute at NIH | ||
| X-tile software | Yale school of medicine | ||
| R-studio | Posit |
Ссылки
- Siegel, R. L., Giaquinto, A. N., Jemal, A. Cancer statistics, 2024. CA Cancer J Clin. 74 (1), 12-49 (2024).
- Korphaisarn, K., et al. Signet ring cell colorectal cancer: Genomic insights into a rare subpopulation of colorectal adenocarcinoma. Br J Cancer. 121 (6), 505-510 (2019).
- Willauer, A. N., et al. Clinical and molecular characterization of early-onset colorectal cancer. Cancer. 125 (12), 2002-2010 (2019).
- Watanabe, A., et al. A case of primary colonic signet ring cell carcinoma in a young man which preoperatively mimicked Phlebosclerotic colitis. Acta Med Okayama. 73 (4), 361-365 (2019).
- Kim, H., Kim, B. H., Lee, D., Shin, E. Genomic alterations in signet ring and mucinous patterned colorectal carcinoma. Pathol Res Pract. 215 (10), 152566(2019).
- Deng, X., et al. Neoadjuvant radiotherapy versus surgery alone for stage II/III mid-low rectal cancer with or without high-risk factors: A prospective multicenter stratified randomized trial. Ann Surg. 272 (6), 1060-1069 (2020).
- Buk Cardoso, L., et al. Machine learning for predicting survival of colorectal cancer patients. Sci Rep. 13 (1), 8874(2023).
- Monterrubio-Gómez, K., Constantine-Cooke, N., Vallejos, C. A. A review on statistical and machine learning competing risks methods. Biom J. 66 (2), e2300060(2024).
- Kim, H. J., Choi, G. S. Clinical implications of lymph node metastasis in colorectal cancer: Current status and future perspectives. Ann Coloproctol. 35 (3), 109-117 (2019).
- Xu, T., et al. Log odds of positive lymph nodes is an excellent prognostic factor for patients with rectal cancer after neoadjuvant chemoradiotherapy. Ann Transl Med. 9 (8), 637(2021).
- Chen, Y. R., et al. Prognostic performance of different lymph node classification systems in young gastric cancer. J Gastrointest Oncol. 12 (4), 285-1300 (2021).
- Bouvier, A. M., et al. How many nodes must be examined to accurately stage gastric carcinomas? Results from a population based study. Cancer. 94 (11), 2862-2866 (2002).
- Coburn, N. G., Swallow, C. J., Kiss, A., Law, C. Significant regional variation in adequacy of lymph node assessment and survival in gastric cancer. Cancer. 107 (9), 2143-2151 (2006).
- Li Destri, G., Di Carlo, I., Scilletta, R., Scilletta, B., Puleo, S. Colorectal cancer and lymph nodes: the obsession with the number 12. World J Gastroenterol. 20 (8), 1951-1960 (2014).
- Dinaux, A. M., et al. Outcomes of persistent lymph node involvement after neoadjuvant therapy for stage III rectal cancer. Surgery. 163 (4), 784-788 (2018).
- Sun, Y., Zhang, Y., Huang, Z., Chi, P. Prognostic implication of negative lymph node count in ypN+ rectal cancer after neoadjuvant chemoradiotherapy and construction of a prediction nomogram. J Gastrointest Surg. 23 (5), 1006-1014 (2019).
- Xu, Z., Jing, J., Ma, G. Development and validation of prognostic nomogram based on log odds of positive lymph nodes for patients with gastric signet ring cell carcinoma. Chin J Cancer Res. 32 (6), 778-793 (2020).
- Scarinci, A., et al. The impact of log odds of positive lymph nodes (LODDS) in colon and rectal cancer patient stratification: a single-center analysis of 323 patients. Updates Surg. 70 (1), 23-31 (2018).
- Nitsche, U., et al. Prognosis of mucinous and signet-ring cell colorectal cancer in a population-based cohort. J Cancer Res Clin Oncol. 142 (11), 2357-2366 (2016).
- Kang, H., O'Connell, J. B., Maggard, M. A., Sack, J., Ko, C. Y. A 10-year outcomes evaluation of mucinous and signet-ring cell carcinoma of the colon and rectum. Dis Colon Rectum. 48 (6), 1161-1168 (2005).
- Sung, C. O., et al. Clinical significance of signet ring cells in colorectal mucinous adenocarcinoma. Mod Pathol. 21 (12), 1533-1541 (2008).
- Alvi, M. A., et al. Molecular profiling of signet ring cell colorectal cancer provides a strong rationale for genomic targeted and immune checkpoint inhibitor therapies. Br J Cancer. 117 (2), 203-209 (2017).
- Brownlee, S., et al. Evidence for overuse of medical services around the world. Lancet. 390 (10090), 156-168 (2017).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены
Мы используем файлы cookie для улучшения качества работы на нашем веб-сайте.
Продолжая пользоваться нашим веб-сайтом или нажимая кнопку «Продолжить», вы соглашаетесь принять наши файлы cookie.
