Method Article
Разрешающая Affinity Очищенная Белковые комплексы от Синей Native ПААГ и белка корреляции профилирование
В этой статье
Резюме
Здесь представлены протоколы для аффинной очистки белковых комплексов и их разделения по синим нативного PAGE с последующим профилированием белка корреляции с использованием меток бесплатного количественного масс-спектрометрии. Этот метод полезен для решения интерактомов Into различных белковых комплексов.
Аннотация
Most proteins act in association with others; hence, it is crucial to characterize these functional units in order to fully understand biological processes. Affinity purification coupled to mass spectrometry (AP-MS) has become the method of choice for identifying protein-protein interactions. However, conventional AP-MS studies provide information on protein interactions, but the organizational information is lost. To address this issue, we developed a strategy to unravel the distinct functional assemblies a protein might be involved in, by resolving affinity-purified protein complexes prior to their characterization by mass spectrometry. Protein complexes isolated through affinity purification of a bait protein using an epitope tag and competitive elution are separated through blue native electrophoresis. Comparison of protein migration profiles through correlation profiling using quantitative mass spectrometry allows assignment of interacting proteins to distinct molecular entities. This method is able to resolve protein complexes of close molecular weights that might not be resolved by traditional chromatographic techniques such as gel filtration. With little more work than conventional AP-geLC-MS/MS, we demonstrate this strategy may in many cases be adequate for obtaining protein complex topological information concomitantly to identifying protein interactions.
Введение
В клетках, большинство белков выполняют свои функции через транзиторных белок-белковых взаимодействий, или путем формирования стабильных белковых агрегатов. Характеризуя белковые взаимодействия имеет решающее значение для полного понимания клеточных процессов. Сродство очистки в сочетании с масс-спектрометрией (AP-MS) является одним из наиболее часто применяемых стратегий для выявления нативные белковых взаимодействий. Значительные улучшения в возможностях инструментов, достигнутых в последнее десятилетие сделали этот подход чрезвычайно мощным. Важно отметить, что взаимодействия, выявленные в экспериментах AP-MS включают смесь прямых и косвенных ассоциаций между приманкой и охотится. Кроме того, часто белки принимают участие в нескольких различных комплексов в пределах одной и той же сотовой связи, которые могут иметь различные биологические функции, и, следовательно, interactors, которые определены с помощью AP-MS может представлять собой смесь различных белковых агрегатов или функциональных объектов. Это не возможно получитьтакая топологическая информация априори из моно-мерных списков белков , полученных с помощью простых экспериментов AP-MS. Тем не менее, этот метод может быть использован в дальнейшем, чтобы определить архитектуру белковых комплексов путем объединения его с одним или несколькими способами, чтобы решить эти сборки.
Для решения топологии белковых взаимодействий, определенных с помощью AP-MS, несколько стратегий, которые были применены. Один из подходов заключаются в выполнении итеративных экспериментов AP-MS с использованием охотится , указанные в предыдущем туре экспериментов , как приманки 1. Хотя очень информативный, это довольно интенсивная задача труда экспериментально и аналитически. Белок сшивание в сочетании с масс - спектрометрией все чаще используется для получения топологической информации о белковых комплексов 2, 3, 4, 5. Тем не менее, computatioNAL анализ сшитых пептидов по-прежнему остается сложной задачей, и, следовательно, является узким местом в процессе. Появление МС-расщепляемые сшивающих реагентов должно облегчить отображение аминокислотных остатков , которые находятся в непосредственной близости от взаимодействующих белков в 6, 7. Другой альтернативой является сочетание аффинной очистки с предыдущими методами разделения ортогональных 8, 9. Хроматографическое фракционирование с помощью гель - фильтрации или ионного обмена, или градиенте сахарозы фракционирования также недавно были использованы в сочетании с количественным масс - спектрометрии для описания мультибелковых комплексов на уровне всей системы, минуя сложный этап изоляции 10, 11, 12, 13. Электрофорез Синий гель родной полиакриламид (BN-PAGE) широко применяется дляисследовать нативные белковые взаимодействия, как правило , те , которые вовлекают митохондриальную мембрану белковых комплексов 14. Этот метод разделения был также недавно использован в сочетании с этикеткой , свободной от белков и количественной оценкой корреляции профилирования, а не только на митохондриальных комплексах 15, 16, 17, но и для распутывания других белковых комплексов из целых клеток 18, 19. Мы предположили, что комбинация аффинной очистки с последующими нативными подходами фракционирования и количественным MS должна обеспечивать полезную стратегию для решения нескольких сборок белков, содержащих конкретный белок.
Здесь мы опишем метод, который сочетает в себе общий эпитоп на основе аффинной очистки с синим нативного электрофореза в полиакриламидном геле выделенных комплексов, с последующим количественным массового зрectrometry и белок корреляция профилирование, чтобы решить многочисленные сборки белок, может быть задействованы. Мы используем мышиные эмбриональные стволовые клетки, где интерес белок слит с эпитоп теге экспрессируется эндогенным локус для достижения близких к физиологическому изобилию и обеспечить эффективную уроженку комплекс изоляции. Этот подход распутывает множественные взаимодействия белка участвует в, разрешая их в различные сборки на основе их корреляция профилей, в то время как не требующие больше работы , чем обычные НЕЦЫ-MS 20.
протокол
1. Выделение коренных белковых комплексов с помощью FLAG Affinity очистки
- Препараты
- Настройка С на водяной бане 37 ° для оттаивания осадок клеток.
- Охладить микроцентрифугу до 4 ° C.
- Поместите несколько микроцентрифужных пробирки различных размеров (1,5 мл, 15 мл) и гомогенизатор во льде.
- Подготовьте 10 мл буфера для лизиса (50 мМ Трис-HCl, рН 8, 150 мМ NaCl, 0,1% Нонидет Р-40, 1 мМ ЭДТА) и держать во льду. Непосредственно перед использованием буфера, добавляют 10 мкл 1 М DTT и дробленый таблетку ингибиторов протеазы ЭДТА свободной.
- Получение антител-связанных шариков
Примечание: Шарики, покрытые антителами, могут быть получены до недели заранее и хранить в холодильнике, пока они не потребуются. Белок G имеет более высокое сродство, чем белок А для большинства видов иммуноглобулина подтипов, для кролика IgG, для которых белок А имеет более высокое сродство исключения.- Промыть 50 мкл белка G-покрытием магнитных шариков: Передача 50 &# 956; л суспензии борта к микроцентрифужным трубкам поместите трубку в магните, чтобы собрать шарики на стороне трубки и удаление жидкости. Ресуспендируют бусины в 0,5 мл PBS, 0,01% Tween-20.
- Поместите пробирку в магнит для сбора бусинки на стороне трубки и удаления жидкости. Ресуспендируют бусины в 40 мкл PBS, 0,01% Tween-20. Добавьте 10 мкг (10 мкл 1 мг / мл исходного раствора) М2 анти-FLAG антителом. Инкубируйте с вращением в течение 20 мин при комнатной температуре.
- Поместите пробирку в магнит для сбора бусинки на стороне трубки и удаления жидкости. Промыть шарики с 0,5 мл PBS, 0,01% Tween-20.
- Для сшивания антитело к гранулам, мыть шарики дважды с 1 мл 0,2 М триэтаноламин рН 8,2. Затем, ресуспендирования бисером в 1 мл 20 мМ DMP (диметил pimelimidate) в 0,2 М триэтаноламин рН 8,2 (раствор DMP следует приготовить свежую непосредственно перед использованием). Инкубируйте с вращением при комнатной температуре в течение 30 мин.
- Поместите трубку в магните. Удалить супернатант и ресуспендируют бусины в 0,5 мл 50 мМ Трис-HCl, рН 7,5. Инкубируйте с вращением при комнатной температуре в течение 15 мин.
- Вымойте бисером 3 раза с 0,5 мл PBS-0,1% Tween-20. Оставьте шарики в последней промывке до тех пор, пока это необходимо.
- аффинной очистки на основе ФЛАГ
Примечание: Будьте осторожны в не выходя из бусин без буфера в течение длительного периода времени. Все буферы и трубы должны храниться на льду во все времена. Следующий протокол оптимизирован для очистки белковых комплексов из 2-5 × 10 8 клеток (10-20 мг белка лизата) , выражающей эндогенные уровни FLAG-меченого белка 20.- Оттепель осадок клеток в водяной бане при 37 ° С до тех пор, пока не начнет плавиться. Возьмите трубку из ванны, закружить мягко, пока осадок не будет полностью размораживают, а сразу поместить трубку, содержащую суспензию клеток на льду.
- Добавьте 5 мл охлажденного льдом буфера для лизиса(Содержащей ДТТ и ингибиторы протеаз) к клеточной суспензии, и водоворот перемешать. Инкубируют на льду в течение 10 мин.
- Передача лизата к холодному Доунса гомогенизатора. Lyse с 20-30 ударов с помощью тугого пестика (до тех пор, пока не заметно вязкости). Передача гомогената к холодным микропробирок.
- Центрифуга при 18000 мкг в течение 15 мин при 4 ° С.
- Передача очищенную лизата в чистую холодную трубу. Оставьте 50 мкл лизата позади. Возьмите 35 мкл аликвоты лизата для измерения концентрации белка и контролировать очистку.
- Удалить PBS-0,1% Tween-20 из гранул. Ресуспендируют бусины с 1 мл лизата и смешать с остальной частью лизата. Выдержите смесь во вращающемся колесе при 4 ° С в течение 1-2 ч.
- Собирайте шарики на стороне трубки с магнитом. Сбор 30 мкл аликвоты надосадочной жидкости и отбросить остальную часть надосадочной жидкости. Ресуспендируют бусины в 0,5 мл буфера IPP150 (10 мМ Трис-HCl, рН 8, 150 мМ NaCl, 1 мМ ЭДТА, 0,1% NP-40) с помощью пипетки 4-6 раз. Переход к 1,5 мл холодной трубки.
- Повторите промывки с 0,5 мл буфера IPP150 еще два раза.
- Промывочный бисер три раза 0,5 мл FLAG нативного буфера для элюции (20 мМ Bis-Tris рН 7, 20 мМ NaCl, 0,02% Нонидет Р-40, 1 мМ EDTA, 200 мМ ε-аминокапроновая кислота). С последней промывкой передать бусинки к новой холодной трубе. Удалить буфер тщательно.
- Ресуспендируют бисером в 100 мкл 200 мкг / мл FLAG пептида 3x в нативном буфере для элюирования. Инкубирует при температуре 4 ° С в течение 10 мин при осторожном вращении.
- Собрать шарики с магнитом и переносят супернатант в новую пробирку холодной.
- Повторите шаги 1.3.10-1.3.11 дважды. Бассейн все элюатов.
- Концентрат элюата до 25 мкл в центробежном блоке фильтра (10 кДа номинального предел молекулярной массы отсечка, PES) путем центрифугирования при 10000 х г при 4 ° С. Передача концентрированного элюата к новой холодной трубе и добавить 50% glyceрол для конечной концентрации 5%. Концентрируют элюат можно хранить при температуре 4 ° С в течение ночи, если это необходимо.
2. Синий Native PAGE
- Препараты
- Готовит 1 л 1x нативного PAGE анодного буфера, 1x нативного PAGE Темно-синий катодный буфер и 1x нативного PAGE светло-голубой Катодный буфер.
- Удалите гребенку сборного 3-12% -ном ПААГ родной и промойте лунки дважды 1x нативный PAGE Темно-синий Катодный буфер. Заполните лунки с 1x нативного PAGE Темно-синий Катодный буфером. Установите гель в проточной баке, но не добавляют темно-синий Катодный буфер с катодом (внутри) камеры.
- Электрофорезная пробег
- Подготовка образца: К 25 мкл концентрированных элюат добавляют соответствующий объем 4x родной загрузки образца буфера и 0,5% G-250 образца добавки для конечной концентрации 1х и 0,005% соответственно.
- Загрузите образец и маркеры молекулярной массы родной оставляя пустой колодец betweeп их. Нагрузка 10-20 мкл образца 1x родной загрузочного буфера во всех пустых лунок.
- Заполните (внутри) катодную камеру с 200 мл 1x нативный PAGE темно-синие Катодный буфер осторожно, чтобы не мешать образцы. Заполните камеру анод (снаружи) с 550 мл 1x нативный PAGE анодного буфера.
- Запуск геля при 150 V в течение 30 мин. Остановить бег, удалить темно-синий катодный буфер с серологической пипеткой и заменить 1x светло-голубой Катодный буфер. Продолжайте бежать в течение 60 мин. Гель можно проводить при комнатной температуре или при 4 ° С; температура может оказывать влияние на структуру белка сложной.
- Гель окрашивания
- Открыть гель кассету и выбросить одну из кассет пластин. Геля остается прикрепленной к другой пластине кассеты. Передача геля в лоток или емкость, содержащий достаточное количество Фиксирующего раствора (40% метанол, 2% уксусная кислота), чтобы покрыть гель и инкубировать в течение 30 мин при осторожном встряхивании.
- Удалите закрепляющее Solutионы и добавить достаточное количество воды, чтобы покрыть гель. Гель можно сканировать для дальнейшего использования.
Анализ 3. Масс-спектрометрия
Примечание: Все последующие шаги должны быть выполнены в вытяжном шкафу с ламинарным потоком, если это возможно, чтобы обеспечить чистоту образцов. Все растворы должны быть приготовлены с ВЭЖМ-классом водой. Обсудить этот протокол с лабораторией масс-спектрометрии, которая будет проводить анализ.
- Препараты
- Поместите коническую нижнюю пластину 96-луночного поверх другой 96-луночный планшет для сбора отходов. Пирс дно лунки конического нижнего 96-луночный планшет с иглой 21 G.
- Промыть лунки проколотыми 96-луночного планшета.
- Добавьте 200 мкл 50% ацетонитрила-0,25% муравьиной кислоты в лунки прошитого пластины. Инкубируйте стопку пластин в шейкере в течение 10 мин.
- Центрифуга при 500 мкг в течение 1 мин, так что жидкость протекает через отверстие в пластину сбора отходов. Выбросьте Wасте жидкости.
- Повторите шаги 3.1.2.1-3.1.2.2 еще два раза.
- Заполните лунки проколотой 96-луночный планшет с 150 мкл 50 мМ бикарбоната аммония (свежеприготовленный).
- Промыть лунки центробежной фильтрации 96-луночного планшета.
- Поместите нормальный 96-луночный планшет под действием центробежной фильтрации пластины для сбора отходов. Добавьте 200 мкл 50% ацетонитрила-0,25% муравьиной кислоты в каждую лунку центробежной фильтрации пластины.
- Центрифуга пакета пластин при 200 х г в течение 1 мин. Выбросьте отходы.
- Повторите шаги 3.1.3.1-3.1.3.2 еще два раза.
- Гель иссечение и в геле пищеварения
Примечание: В то время как иссечения геля, идентифицировать и сделать отметку среза геля с выравниванием по каждому из белковых стандартов. Стандарты белка могут быть использованы для оценки приблизительных молекулярных масс для всех срезов геля.- Поместите гель со стеклянной пластиной на чистую поверхность. Нарезать переулок содержатING образец в 48 одинаковых ломтиков (1,5 мм х 5 мм). Вырезать каждый ломтик на 2-3 более мелкие куски (за исключением последних пяти срезов в верхней части геля) и поместить каждый ломтик последовательно в одну лунку проколотой и промывали 96-луночный планшет.
- Добавить 50 мкл ацетонитрила в каждую лунку. Инкубируйте планшет при встряхивании в течение 30-60 мин. Удалить жидкость путем центрифугирования, как на стадии 3.1.2.2.
- Добавляют 200 мкл 2 мМ TCEP в 50 мМ бикарбоната аммония в каждую лунку. Инкубируйте планшет при встряхивании в течение 30 мин при комнатной температуре. Удалить жидкость путем центрифугирования, как на стадии 3.1.2.2.
- Добавляют 200 мкл 4 мМ иодацетамида в 50 мМ бикарбоната аммония в каждую лунку. Инкубируйте планшет при встряхивании в течение 30 мин при 37 ° C в темноте. Удалить жидкость путем центрифугирования, как на стадии 3.1.2.2.
- Добавьте 150 мкл 50 мМ бикарбоната аммони плюс 50 мкл ацетонитрила и инкубируйте при встряхивании в течение 30 мин при комнатной температуре. Повторите эти стиральные стадии аев много раз, как требуется, пока весь синий цвет не будет удален из кусочков геля.
- Высушить гель частей путем добавления 200 мкл чистого ацетонитрила и инкубировать при встряхивании при комнатной температуре в течение 20 мин до тех пор, пока кусочки геля белого цвета и непрозрачным. Удалить ацетонитрил.
- Добавьте 150 мкл 0,001 мг / мл трипсина (секвенирование сорта) в холодном 50 мМ бикарбоната аммония в каждую лунку. Инкубируйте планшет при встряхивании при 37 ° С в течение 2 ч. Через 1 ч, проверьте, чтобы кусочки геля покрыты жидкостью; если это не так, добавить еще 50-100 мкл 50 мМ бикарбоната аммония. Через 2 ч при 37 ° С, продолжают переваривание при температуре 25 ° С в течение ночи.
- Поместите чистую коническую нижнюю пластину под гель, содержащий пластину, обеспечивая правильную ориентацию пластин. Сбор жидкости, содержащей пептиды центрифугированием при 200g в течение 1 мин.
- Поместите пептиды, содержащие пластину в центробежном испарителе и испарения жидкости во время выполнения тон следующий шаг.
- Добавьте 150 мкл 50% ацетонитрила-0,25% муравьиной кислоты на кусочки геля. Инкубируют при встряхивании при 37 ° С в течение 30 мин.
- Накопление жидкости в частично высушенной пластину со стадии 3.2.9 центрифугированием при 200g в течение 1 мин. Продолжить выпаривание пептидов, содержащую пластину во время выполнения следующего шага.
- Повторите шаги 3.2.10-3.2.11.
- Добавьте 200 мкл чистого ацетонитрила в каждую лунку планшета, содержащего кусочки геля. Инкубирует при встряхивании при комнатной температуре в течение 15 мин.
- Накопление жидкости в пептиды, содержащей пластины центрифугированием при 200g в течение 1 мин.
- Убедитесь, что конечная концентрация ацетонитрила во всех скважинах выше 55%. При необходимости добавить дополнительный чистый ацетонитрил.
- Передача пептидных решений промытой центробежной фильтрации пластина. Поместите чистую коническую нижнюю пластину под ней, чтобы собрать пептиды. Центрифуга пакета пластин при 200 х г в течение 1 мин.
- Evaporели жидкость в конической нижней пластине до тех пор, пока скважины полностью не высохнут. Пластина может быть покрыта силиконовой крышкой и хранили при -20 ° С на этой стадии.
- Повторно растворяются пептиды в 32 мкл 0,5% -ной муравьиной кислоты при энергичном встряхивании в течение 15 мин. Добавить 8 мкл 400 мМ бикарбоната аммония и инкубировать при интенсивном встряхивании в течение 15 мин. Центрифуга пластины при 200 х г в течение 1 мин. Покрытие с силиконовой крышкой и хранить пластины при температуре от -20 ° C до анализа масс-спектрометрии.
- Масс-спектрометрии
Примечание: Анализ пептидов на масс-спектрометре высокого разрешения с использованием методов, совместимых с дробовика протеомики. зависят от данных приобретения является наиболее часто используемым LC-тандем МСА установки для этого типа анализа и должны быть оптимизировано для эффективной идентификации пептида, сводя к минимуму избыточной последовательности и выбору кандидатов на фоне шум. Масс-спектры в анализе сканирования первой стадии массового (MS1), должны быть записаны в режиме профиля. Это рекомендуетсяред для предпочтительного выбора двукратно и трехкратно заряженные ионы для второй стадии анализа массы (MS2). Диапазон массы м / г 400-1,600 подходит для идентификации большинства пептидов между 8-30 диапазоном аминокислот. Здесь, протокол для анализа с помощью масс-спектрометра Orbitrap предусмотрен.- Вводят 20 мкл образца на анализ с помощью автоматического пробоотборника системы nanoLC-МС / МС. Обессоливания и концентрат пептидов на обращенной фазе на колонке C18 (100 мкм ID, 2 см).
- Отдельные пептиды на аналитической колонке С18 (75 мкм идентификатором, 15 см) с использованием 30 мин линейный градиент 4-28% ацетонитрила / 0,1% муравьиной кислоты со скоростью 300 л / мин.
- Анализ пептидов в масс-спектрометре с верхним 10 данных зависимого метода сбора данных со следующими параметрами: диапазон м / з 380-1,600, разрешение на 30000 м / з 400, ширина изоляция 2 Th, динамическое исключение при ± 10 частей на миллион в течение 45 сек , автоматическая регулировка усиления целевой при 1 × 10 6 для MS и 5000 для MS / MS, максимальное время впрыска 150 мс для MS и100 мс для MS / MS.
Анализ 4. Данные
- Идентификация и количественное определение белка с использованием MaxQuant
- Используйте свободно доступное программное обеспечение MaxQuant для идентификации и количественного определения белков в каждой гель-фракции из масс-спектрометрии исходных данных.
Примечание: MaxQuant работает на данных, полученных с помощью дробовика приобретения протеомики подходов данных зависящих. Данные МС из голубых нативных фракций гель должны быть проанализированы в пакетном виде отдельных экспериментов, а не в качестве фракций эксперимента, сочетающей все кусочки геля. Проверьте значение окна iBAQ для выполнения расчетов стехиометрии. Подробные протоколы для поиска базы данных и белков с использованием количественной оценки MaxQuant описаны в 21, 22.
- Используйте свободно доступное программное обеспечение MaxQuant для идентификации и количественного определения белков в каждой гель-фракции из масс-спектрометрии исходных данных.
- Анализ профиля белка с помощью Персей
- Используйте свободно доступное программное обеспечение Perseus для отображения профилей миграции идентифицированных белков и сравнить их с помощьюстатистический анализ.
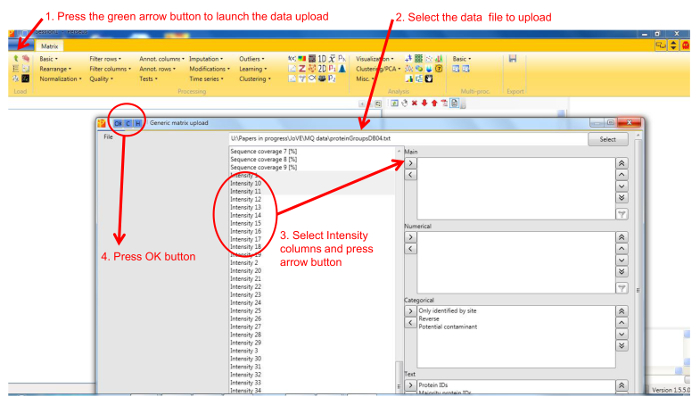
Примечание: Общая документация по использованию Персей доступно на http://www.coxdocs.org/doku.php?id=perseus:user:tutorials. Образец набор данные доступны в качестве дополнительных данных. - Загрузить файл proteingroups.txt, возвращенный MaxQuant анализа, содержащих белка и идентификации значение Количественного для всех фракций геля. Заполнение значений интенсивности в главное окно (рисунок 1). Матрица будет отображаться, содержащей все белковые идентификации рядов, с гелем фракцией как колонно.
- Удалить записи , соответствующие обратный удар, белки только идентифицированного сайтом и потенциальными загрязнителями путем выбора фильтров строк на основе категориального столбца в раскрывающемся меню Фильтра строк (рисунок 2).
- Нормализация интенсивности доли каждого белка через профиль по отношению к суммарной интенсивности белка путем выбора Divide в раскрывающемся меню Normalize, то сумма (рисунок 3). Новый мпоявляется Atrix.
- Отображение миграции профиля участков всех белков, выбрав Профиль Plot в меню Визуализация ниспадающего (рис 4).
- Выбор фильтра строк на основе допустимых значений в раскрывающемся меню строк фильтра. Введите 1 в количестве допустимых значений, требуемых.
- Выполнение иерархической кластеризации идентифицированных белков во всех голубых нативных фракциях геля путем выбора иерархической кластеризации в раскрывающемся меню кластеризации / PCA. Непроверьте Столбцы дерева окно (рисунок 5). Кластеры визуализируются в тепловой карте на вкладке кластеризации рядом с матрицей (рисунок 6).
- Расположить кластер, содержащий белок приманки в дендрограммах. Другие компоненты кластера представляют собой белки совместно мигрирующие с приманкой и, следовательно, потенциально взаимодействующие белки / субъединиц одного и того же комплекса.
- Используйте свободно доступное программное обеспечение Perseus для отображения профилей миграции идентифицированных белков и сравнить их с помощьюстатистический анализ.
 г /> Рисунок 1. Снимок экрана окна загрузки данных Персея. На рисунке показаны шаги для загрузки идентификации белка и данные количественного определения в Персее. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
г /> Рисунок 1. Снимок экрана окна загрузки данных Персея. На рисунке показаны шаги для загрузки идентификации белка и данные количественного определения в Персее. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.

Рисунок 2. Снимок экрана окна Персея для фильтрации записей , соответствующих загрязняющих веществ, обратного удара и белки , определенные сайта. Выбор строк фильтра по категорическому колонке открывает новое окно для выбора типов записей белков, которые будут исключены из набора данных. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
/55498fig3.jpg»/>
Рисунок 3. Скриншот окна нормализации Персея. Выбор Разделить в меню Нормализации открывает новое окно, с различными опциями. Выбор Суммы делит значение интенсивности белка в каждой фракции по общей интенсивности этого белка во всех фракциях. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.

Рисунок 4. Снимок экрана Персеус показывающий участки миграции профилей. Белки могут быть выбраны в матрице ниже профилей, чтобы выделить их соответствующий профиль. Панель инструментов может быть использована для редактирования и экспорта профилей. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.

Рисунок 5. Скриншот Персея окна иерархической кластеризации. На рисунке показаны настройки для иерархической кластеризации белков с использованием Manhattan (L1), показатель расстояния. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
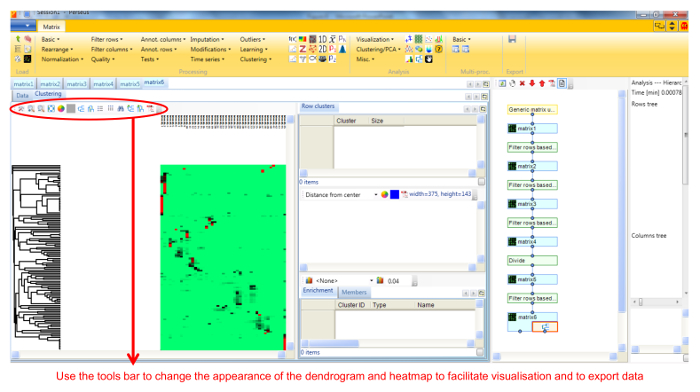
Рисунок 6. Скриншот Персея показывает дендрограммы и интенсивности белка Heatmap. Рабочий процесс на правой стороне основной панели показывает каждый шаг, проведенный и полученную матрицу, и может быть использован для отмены любого шага. Рабочий процесс также может расшириться из любой промежуточной матрицы. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этого еigure.
- Расчет относительной стехиометрии
- Выберите субъединицы белкового комплекса на основе их совместного кластеризация с белком приманки и разграничить пик профиля (ов), где они появляются.
- Используйте значение iBAQ возвращенного MaxQuant анализа для каждой субъединицы в каждой фракции отдельно и нормализуют с помощью соответствующей приманки значения iBAQ в этой фракции.
- Участок нормированных iBAQs для наживки и белковых субъединиц комплекса по фракции выбранного пика и применить линейную регрессию. Вычислить относительную величину комплексной субъединицей травить путем сравнения тенденции их нормированных значений iBAQ.
- Оценка белкового комплекса размера
- Использование белковых стандартов для расчета предсказанных молекулярных масс для центрирующего синих нативные срезов геля из образца полосы. Из них, генерировать модели линейной регрессии путем построения Гелевые срезы в оси х и молекулярные массы в оси у.
- Используйте модель для оценки наблюдаемого диапазона молекулярной массы белка комплекса (ов) на основе синего нативный среза (ы), из которого они были идентифицированы.
Результаты
Рабочий процесс аффинной очистки Синий профилирования нативный белок корреляции методом масс - спектрометрии (АВС-МС) стратегия показана на рисунке 7. Коренные белковые комплексы вокруг интересующего белка изолированы с помощью аффинной очистки с использованием антител против эпитопов тега (в данном случае FLAG) и конкурентного элюирования. Комплексы разрешаются BN-PAGE, и весь гель полосы вырезают на 48 секций, и подготовлены к Ружье LC-MS / MS. Количественные данные МСА используются для создания профиля миграции для каждого идентифицированного белка через голубое родное разделение. Белки, которые взаимодействуют с образованием различных комплексных отображения аналогичных профилей миграции с наложимо пиками. Когда интересующий белок принимает участие в более чем одной сборки, множественные пики наблюдаются в его миграции профиль, учитывая суб-комплексы находятся в пределах разрешающей способности голубого нативного геля. Систематическое сравнение МИГПрофили рацион может быть достигнуто за счет корреляции белка профилирования с использованием иерархической кластеризации. Дендрограммы и интенсивности пиков для всех фракций визуализируются в тепловой карте, что облегчают идентификацию взаимодействующих белков , которые принадлежат к различным белковым комплексам (Рисунок 8).
Мы использовали эту стратегию для анализа взаимодействующих партнеров mta2, основной субъединица NuRD хроматина комплекса 20. Как показано на рисунке 9, профиль миграции mta2 показаны два пика различных интенсивностей между 700 кДа и 1,2 МДа, с меньшей массой пика , где отображаются более высокую численность. Другая основные NuRD субъединица, в том числе mta1 / 3, HDAC1 / 2 и mbd3, показала одинаковую картину разделения, хотя и пики для некоторых из субъединиц, а именно chd4, Gatad2a / б и rbbp4 / 7, имели обратное распределение численности (9, Б). В отличие от этого, профиль Cdk2ap1, регулирующий фактор , который принимает на работу NuRD комплекса к Wnt промоторов генов 23, отображается только более высокий массовый пик (рис 9c), и так же, Sall4 репрессор транскрипции , что также было показано , чтобы связать NuRD 20, 24, 25. Таким образом, фракционирование аффинных очищенных mta2 белков, ассоциированные с синим нативным ПААГОМ удалось решить две различных формы NuRD комплекса.
АВС-МС также позволяет идентифицировать новые interactors в то время присвоения им определенных белковых образований. Через рассмотрения белков кластеров и интенсивностей фракций , представленных в тепловой карте мы обнаружили сильную корреляцию между NuRD субъединиц и WDR5, регуляторной субъединицей комплекса метилтрансферазы MLL 26, с WDR5 , отображающих два пика миграции совпадает с два р NuRDeaks (Фигура 8), предполагая новое взаимодействие между WDR5 и NuRD. Мы подтвердили это взаимодействие путем совместной иммунопреципитации и совместной миграции в вытеснительной хроматографии 20.
Выбор расстояния вычислении метрики в иерархической кластеризации будет влиять на форму кластеров и, следовательно, корреляций. Мы рекомендуем поэкспериментировать с различными метриками для достижения наилучшего соответствия с имеющимися знаниями взаимодействия белка или комплекса интереса. Как правило , мы добились наилучших результатов с Manhattan (L1) расстояния метрики 20. Корреляции Пирсона или евклидовы показатели расстояния также сообщалось для идентификации комплексов на основе методов альтернативы фракционирования 10, 11, 12.
цюаньДанные ственный MS, полученные из синих нативных фракций также может быть использованы для определения стехиометрии белковых комплексов. Значение iBAQ (интенсивность на основе абсолютного Количественная) представляет собой меру относительной численности идентифицированных белков 27, 28. Значения iBAQ для белкового комплекса членов во всех фракциях в пределах пика миграции профиля нормированы к тому, что части белка приманки для получения относительных количеств. Нормированное iBAQs через пик профиля для комплексного элемента данного белка должно следовать горизонтальной тенденции, а значения тренда отражает стехиометрию взаимодействующих белков по сравнению с белком приманки. Для более детального представления расчетов стехиометрии см 20.

Рисунок 7: Схема рабочего процесса обобщения АВС-МС сегии. Этот показатель изменяется от 20. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
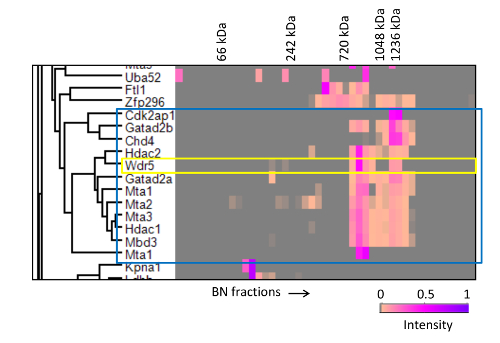
Рисунок 8. Иерархическая кластеризация БН-PAGE миграции профилей mta2 interactors. Mta2-ассоциированные белки были сгруппированы на основе сходства их миграции профилей. Только часть тепловой карты, содержащей комплекс NuRD показана (заключенный в синей коробке). Желтый ящик подчеркивает сильную корреляцию WDR5 с NuRD комплексом. Аннотированные молекулярные массы были оценены по миграции расстояний белковых стандартов работают в том же самом геле. Это исследование было первоначально опубликовано в молекулярной и клеточной протеомике 20 Американского общества биохимии и Мо лярных биология. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
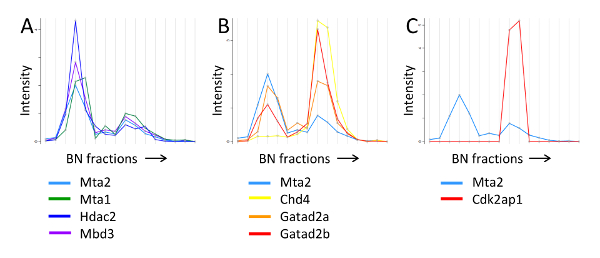
Рисунок 9. БН-ПААГ миграции профиля NuRD субъединиц и связанных с ними белков. А) mta2 и NuRD субъединицы , присутствующие на аналогичную схему интенсивности. Б) mta2 и NuRD субъединиц с рисунком обратной интенсивности. С) mta2 и Cdk2ap1, который присутствует только в более высокой молекулярной массой NuRD сущности. Аннотированные молекулярные массы оценивались по миграции профиля стандартов белка. Это исследование было первоначально опубликовано в молекулярной и клеточной протеомики 20 Американского общества биохимии и молекулярной биологии._blank "> Пожалуйста, нажмите здесь, чтобы посмотреть увеличенную версию этой фигуры.

Справочная данные: Образец набора данных. MaxQuant полученный файл proteingroups.txt, содержащий белковые идентификаций и значения количественного определения из эксперимента АВС-MS. Это исследование было первоначально опубликовано в молекулярной и клеточной протеомики 20 Американского общества биохимии и молекулярной биологии. Пожалуйста , нажмите здесь , чтобы скачать этот файл.
Обсуждение
Здесь мы опишем использование аффинной очистки с последующим синим электрофореза нативного гель в сочетании с количественной масс-спектрометрии для решения белковых комплексов. Этот подход предлагает способ распутать одномерные списки взаимодействия белка в функциональных белковых агрегатов.
Мы демонстрируем метод, основанный на использовании эпитопов меченых белков. Однако, если линия клеток, экспрессирующих белок, помеченный не доступна, альтернативой может быть использование антител против белка, представляющего интерес, при условии, есть пептид, доступный для достижения нативный конкурентного элюирования. Количество исходного материала и количества гранул, возможно, должно быть изменено в зависимости от уровня экспрессии белка-мишени. Мы обычно выполняют этот протокол с 2-5 × 10 8 клеток исходного материала, и этого достаточно даже для белков с низким уровнем экспрессии. Состав буфера для лизиса должен быть выбран эмпирический для достижениярядом с полной солюбилизации приманки. Это может быть сложным для некоторых типов белков, в частности хроматина связывания или мембранных белков. Альтернативные варианты включают в себя увеличение количества соли, при условии , что сложный белок при исследовании устойчиво в высокой концентрации соли, или с помощью ультразвука и / или лечения нуклеазы для хроматина связывающих белков 20, 29. В случае мембранных белков, переключая моющее средство DDM или дигитонину может быть целесообразным 30. В случае ДНК - связывающие белки, это полезно включать нуклеазу , такую как бензоназу во время стадии очистки 20. Полное удаление нуклеиновых кислот гарантирует, что взаимодействие обнаруженное происходит между белками и не опосредованы ДНК.
Критический элемент следует учитывать при использовании этого подхода является стабильность комплекса под следствием. Процедура долго и может включать в себя preservinг белка комплекс в течение ночи. Мы достигли хороших успехов с двух хроматина комплексов (Д. Боде и М. Пардо, данные не показаны), но это должно быть оценено. Стадия аффинной очистки может быть сокращена, если это необходимо.
Альтернативные методы, позволяющие родное фракционирование, такие как гель-проникающая хроматография, которые широко используются в течение более 50 лет, чтобы охарактеризовать белковые комплексы. Мы и другие показали , что разрешение синего нативного PAGE превосходит , что достигается с помощью эксклюзионной хроматографии на 20, 31, 32, 33. Еще одно преимущества голубого нативного ПААГА является то, что он не требует системы хроматографии, которые являются дорогостоящими, а использует белка электрофоретическое оборудование, которое широко распространен в лабораториях. С точки зрения практического времени, этот метод не предполагает больше работы, чем традиционные НЕЛЦ-MS / MS аpproach или отсутствует хроматографического фракционирования. Однако, как и большинство методов фракционирования, она имеет предел их разрешения. Комплексы, которые очень однородна или близки по массе и форме, может быть за разрешением, предложенной синим нативного ПААГ, и, следовательно, протокол, как сообщалось здесь не может быть универсально успешным в решении различных комплексов с общими субъединиц. Обнадеживает то, что мы добились успеха в выделении двух очень похожих тетрамерные комплексов обмена трех субъединиц (М. Пардо, рукописи в процессе подготовки).
Так как масс-спектрометрия становится все более чувствительным, даже незначительные количества неспецифических interactors и загрязняющих веществ могут быть обнаружены в образцах АП-MS. Подход, представленный здесь, может помочь в различении реальных interactors от фонового загрязнения в аффинных очистках, сосредоточив внимание на белках с миграционными пиками, совпадающий с приманкой миграции пиками при более высокой молекулярной массе, чем умономерные белки.
Несколько групп использовали методы фракционирования с последующими профилированием белка корреляции очертить белковые комплексы на клеточном уровне без необходимости предварительного выделения 10, 11, 12, 34. Тем не менее, это может привести к невозможности обнаружить суб-стехиометрических взаимодействия. Включение обогащения, шаг через аффинной очистки может помочь преодолеть это. Описанный здесь подход должен быть в целом полезен для изучения топологии белковых комплексов и разгадывая множество комплексов данного белка принимает участие в пределах одной и той же сотовой связи. Стратегия проста и поддающийся лабораторию, которые не могут иметь дорогое хроматографическое фракционирование оборудования для решения белковых комплексов.
Раскрытие информации
The authors have nothing to disclose.
Благодарности
This work was funded by the Wellcome Trust (WT098051).
Материалы
| Name | Company | Catalog Number | Comments |
| Protein G Dynabeads | Life Technologies | 10003D | |
| FLAG antibody M2 | Sigma-Aldrich | F1804 | |
| Tween-20, protein grade, 10% solution | Millipore | 655206 | |
| Dimethyl pimelimidate | Sigma-Aldrich | 80490 | |
| 0.2 M Triethanolamine buffer, pH 8.2 | Sigma-Aldrich | T0449 | |
| 3x FLAG peptide | Sigma-Aldrich | F4799 | |
| Vivaspin 5K NMWCO, PES | Sartorius | VS0112 | |
| NativePAGE 3-12% Bis-Tris gel | Life Technologies | BN1001 | |
| 20x NativePAGE Running Buffer | Life Technologies | BN2001 | |
| 20x NativePAGE Cathode Additive | Life Technologies | BN2002 | |
| 4x NativePAGE sample loading buffer | Life Technologies | BN2003 | |
| NativeMARK | Life Technologies | LC0725 | |
| NativePAGE 5% G-250 sample additive | Life Technologies | BN2004 | |
| Nunc conical bottom 96-well plate, polypropylene | Thermo | 249944 | |
| Multiscreen HTS 96-well filtration system | Thermo | MSDVN6550 | |
| Nunc 96-well cap natural | Thermo | 276002 | |
| TCEP (Tris(2-carboxyethyl)phosphine hydrochloride) | Sigma-Aldrich | 646547 | |
| Iodoacetamide | Sigma-Aldrich | I1149 | |
| Acetonitrile, HPLC grade | Fisher Scientific | A/0627/17 | |
| Trypsin, sequencing grade | Roche | 11418475001 | |
| MaxQuant (software) | N.A. | N.A. | http://www.biochem.mpg.de/5111795/maxquant |
| Perseus (software) | N.A. | N.A. | http://www.biochem.mpg.de/5111810/perseus |
Ссылки
- Goudreault, M., et al. A PP2A phosphatase high density interaction network identifies a novel striatin-interacting phosphatase and kinase complex linked to the cerebral cavernous malformation 3 (CCM3) protein. Mol Cell Proteomics. 8 (1), 157-171 (2009).
- Leitner, A., et al. Probing native protein structures by chemical cross-linking, mass spectrometry, and bioinformatics. Mol Cell Proteomics. 9 (8), 1634-1649 (2010).
- Rappsilber, J. The beginning of a beautiful friendship: cross-linking/mass spectrometry and modelling of proteins and multi-protein complexes. J Struct Biol. 173 (3), 530-540 (2011).
- Sinz, A. Chemical cross-linking and mass spectrometry to map three-dimensional protein structures and protein-protein interactions. Mass Spectrom Rev. 25 (4), 663-682 (2006).
- Walzthoeni, T., Leitner, A., Stengel, F., Aebersold, R. Mass spectrometry supported determination of protein complex structure. Curr Opin Struct Biol. 23 (2), 252-260 (2013).
- Kaake, R. M., et al. A new in vivo cross-linking mass spectrometry platform to define protein-protein interactions in living cells. Mol Cell Proteomics. 13 (12), 3533-3543 (2014).
- Kao, A., et al. Development of a novel cross-linking strategy for fast and accurate identification of cross-linked peptides of protein complexes. Mol Cell Proteomics. 10 (1), (2011).
- Hughes, M. A., Langlais, C., Cain, K., MacFarlane, M. Isolation, characterisation and reconstitution of cell death signalling complexes. Methods. 61 (2), 98-104 (2013).
- Lim, S., Zou, Y., Friedman, E. The transcriptional activator Mirk/Dyrk1B is sequestered by p38alpha/beta MAP kinase. J Biol Chem. 277 (51), 49438-49445 (2002).
- Havugimana, P. C., et al. A census of human soluble protein complexes. Cell. 150 (5), 1068-1081 (2012).
- Kirkwood, K. J., Ahmad, Y., Larance, M., Lamond, A. I. Characterization of native protein complexes and protein isoform variation using size-fractionation-based quantitative proteomics. Mol Cell Proteomics. 12 (12), 3851-3873 (2013).
- Kristensen, A. R., Gsponer, J., Foster, L. J. A high-throughput approach for measuring temporal changes in the interactome. Nat Methods. 9 (9), 907-909 (2012).
- Liu, F., Heck, A. J. Interrogating the architecture of protein assemblies and protein interaction networks by cross-linking mass spectrometry. Curr Opin Struct Biol. 35, 100-108 (2015).
- Wittig, I., Schagger, H. Native electrophoretic techniques to identify protein-protein interactions. Proteomics. 9 (23), 5214-5223 (2009).
- Heide, H., et al. Complexome profiling identifies TMEM126B as a component of the mitochondrial complex I assembly complex. Cell Metab. 16 (4), 538-549 (2012).
- Wessels, H. J., et al. Analysis of 953 human proteins from a mitochondrial HEK293 fraction by complexome profiling. PLoS One. 8 (7), 68340 (2013).
- Wessels, H. J., et al. LC-MS/MS as an alternative for SDS-PAGE in blue native analysis of protein complexes. Proteomics. 9 (17), 4221-4228 (2009).
- Remmerie, N., et al. Unraveling tobacco BY-2 protein complexes with BN PAGE/LC-MS/MS and clustering methods. J Proteomics. 74 (8), 1201-1217 (2011).
- Sessler, N., Krug, K., Nordheim, A., Mordmuller, B., Macek, B. Analysis of the Plasmodium falciparum proteasome using Blue Native PAGE and label-free quantitative mass spectrometry. Amino Acids. 43 (3), 1119-1129 (2012).
- Bode, D., Yu, L., Tate, P., Pardo, M., Choudhary, J. Characterization of Two Distinct Nucleosome Remodeling and Deacetylase (NuRD) Complex Assemblies in Embryonic Stem Cells. Mol Cell Proteomics. 15 (3), 878-891 (2016).
- Cox, J., Mann, M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nat Biotechnol. 26 (12), 1367-1372 (2008).
- Cox, J., et al. A practical guide to the MaxQuant computational platform for SILAC-based quantitative proteomics. Nat Protoc. 4 (5), 698-705 (2009).
- Kim, J. J., et al. A novel regulatory factor recruits the nucleosome remodeling complex to wingless integrated (Wnt) signaling gene promoters in mouse embryonic stem cells. J Biol Chem. 287 (49), 41103-41117 (2012).
- Lauberth, S. M., Rauchman, M. A conserved 12-amino acid motif in Sall1 recruits the nucleosome remodeling and deacetylase corepressor complex. J Biol Chem. 281 (33), 23922-23931 (2006).
- Miller, A., et al. Sall4 controls differentiation of pluripotent cells independently of the Nucleosome Remodelling and Deacetylation (NuRD) complex. Development. 143 (17), 3074-3084 (2016).
- Dharmarajan, V., Lee, J. H., Patel, A., Skalnik, D. G., Cosgrove, M. S. Structural basis for WDR5 interaction (Win) motif recognition in human SET1 family histone methyltransferases. J Biol Chem. 287 (33), 27275-27289 (2012).
- Schwanhausser, B., et al. Global quantification of mammalian gene expression control. Nature. 473 (7347), 337-342 (2011).
- Smits, A. H., Jansen, P. W., Poser, I., Hyman, A. A., Vermeulen, M. Stoichiometry of chromatin-associated protein complexes revealed by label-free quantitative mass spectrometry-based proteomics. Nucleic Acids Res. 41 (1), 28 (2013).
- Lambert, J. P., Tucholska, M., Pawson, T., Gingras, A. C. Incorporating DNA shearing in standard affinity purification allows simultaneous identification of both soluble and chromatin-bound interaction partners. J Proteomics. 100, 55-59 (2014).
- Wittig, I., Braun, H. P., Schagger, H. Blue native PAGE. Nat Protoc. 1 (1), 418-428 (2006).
- Schagger, H., Cramer, W. A., von Jagow, G. Analysis of molecular masses and oligomeric states of protein complexes by blue native electrophoresis and isolation of membrane protein complexes by two-dimensional native electrophoresis. Anal Biochem. 217 (2), 220-230 (1994).
- Schagger, H., von Jagow, G. Blue native electrophoresis for isolation of membrane protein complexes in enzymatically active form. Anal Biochem. 199 (2), 223-231 (1991).
- Camacho-Carvajal, M. M., Wollscheid, B., Aebersold, R., Steimle, V., Schamel, W. W. Two-dimensional Blue native/SDS gel electrophoresis of multi-protein complexes from whole cellular lysates: a proteomics approach. Mol Cell Proteomics. 3 (2), 176-182 (2004).
- Liu, F., Rijkers, D. T., Post, H., Heck, A. J. Proteome-wide profiling of protein assemblies by cross-linking mass spectrometry. Nat Methods. 12 (12), 1179-1184 (2015).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены