Method Article
Auflösen von Affinity Purified Protein-Komplexen von Blue native PAGE und Protein Correlation Profilieren
In diesem Artikel
Zusammenfassung
Hier stellen wir Protokolle für die Affinitätsreinigung von Proteinkomplexen und deren Trennung durch blaue native PAGE, gefolgt von Proteinkorrelationsprofilierungs freie quantitative Massenspektrometrie Label. Diese Methode ist nützlich Interaktom in verschiedene Proteinkomplexe zu lösen.
Zusammenfassung
Most proteins act in association with others; hence, it is crucial to characterize these functional units in order to fully understand biological processes. Affinity purification coupled to mass spectrometry (AP-MS) has become the method of choice for identifying protein-protein interactions. However, conventional AP-MS studies provide information on protein interactions, but the organizational information is lost. To address this issue, we developed a strategy to unravel the distinct functional assemblies a protein might be involved in, by resolving affinity-purified protein complexes prior to their characterization by mass spectrometry. Protein complexes isolated through affinity purification of a bait protein using an epitope tag and competitive elution are separated through blue native electrophoresis. Comparison of protein migration profiles through correlation profiling using quantitative mass spectrometry allows assignment of interacting proteins to distinct molecular entities. This method is able to resolve protein complexes of close molecular weights that might not be resolved by traditional chromatographic techniques such as gel filtration. With little more work than conventional AP-geLC-MS/MS, we demonstrate this strategy may in many cases be adequate for obtaining protein complex topological information concomitantly to identifying protein interactions.
Einleitung
In Zellen, führen die meisten Proteine ihre Funktionen durch transitorische Protein-Protein-Wechselwirkungen oder durch stabile Proteinanordnungen bilden. Protein-Wechselwirkungen charakterisierenden ist für voll Verständnis zellulärer Prozesse von entscheidender Bedeutung. Affinitätsreinigung in Kombination mit der Massenspektrometrie (AP-MS) ist eine der am häufigsten angewandt Strategien native Protein-Wechselwirkungen zu identifizieren. Signifikante Verbesserungen in den Instrumenten Fähigkeiten in den letzten zehn Jahren erreicht haben, diesen Ansatz extrem leistungsfähig gemacht. Es ist wichtig zu beachten, dass die von AP-MS-Experimenten identifiziert Wechselwirkungen eine Mischung aus direkten und indirekten Verbindungen zwischen Köder und Beute sind. Darüber hinaus oft Proteine nehmen an mehreren verschiedenen Komplexen innerhalb des gleichen zellulären Kontext, die unterschiedliche biologische Rollen haben könnte, und daher sind die Interaktoren, die durch AP-MS identifiziert werden könnte eine Mischung von verschiedenen Protein-Baugruppen oder Funktionseinheiten darstellen. Es ist nicht möglich abzuleitensolche topologischen Informationen a priori aus den mono-dimensional Listen von Proteinen durch einfache AP-MS - Experimente erzeugt. Jedoch kann die Technik ausgenutzt werden, um die Architektur weiter von Proteinkomplexen zu definieren, indem sie mit einer oder mehreren Methoden, die Kombination dieser Baugruppen zu lösen.
Um haben die Topologie von Protein-Wechselwirkungen identifiziert durch AP-MS, verschiedene Strategien angewandt worden zu lösen. Ein Ansatz ist die iterativen AP-MS - Experimente durchzuführen , unter Verwendung des in einer früheren Runde von Experimenten identifiziert preys als Köder 1. Obwohl sehr informativ, ist dies eine Arbeit ganz Aufgabe intensiv experimentell und analytisch. Protein Vernetzung in Kombination mit der Massenspektrometrie wird zunehmend eingesetzt, um 2 auf den Proteinkomplexen topologische Informationen abzuleiten, 3, 4, 5. Allerdings computational Analyse von vernetzten Peptide bleibt nach wie vor eine schwierige Aufgabe, und daher ist der Engpass im Arbeitsablauf. Das Aufkommen von MS-spaltbare Vernetzungsreagenzien sollten Kartierung der Aminosäurereste erleichtern , die 6 Proteine in enger Nähe sind, 7 in zusammenwirkt. Eine weitere Alternative ist die Affinitätsreinigung des Standes der orthogonalen Trenntechniken zu kombinieren 8, 9. Chromatographische Fraktionierung durch Gelfiltration oder Ionenaustausch oder Saccharosegradienten - Fraktionierung wird kürzlich auch in Kombination mit quantitativer Massenspektrometrie verwendet Multiproteinkomplexe in einer systemweiten Ebene zu beschreiben, unter Umgehung des komplexen Isolierungsschrittes 10, 11, 12, 13. Blau nativen Polyacrylamid-Gelelektrophorese (BN-PAGE) wurde in großem Umfang angewendetuntersuchen native Protein - Wechselwirkungen, Protein typischerweise mitochondrialen Membrankomplexe solcher , die 14. Diese Trenntechnik wurde kürzlich auch in Kombination mit markierungsfreien Proteinquantifizierung und Korrelations Profilieren, nicht nur auf die mitochondriale Komplexe 15, 16, 17, sondern auch für andere entwirren Proteinkomplexe aus ganzen Zellen 18, 19 verwendet. Wir stellten die Hypothese, dass die Kombination von Affinitätsreinigung mit anschließenden nativen Fraktionierung Ansätzen und quantitativem MS sollte für die Lösung mehrerer Proteineinheiten eine nützliche Strategie bietet ein bestimmtes Protein enthalten.
Hier beschreiben wir ein Verfahren, das generische Epitop-basierte Affinitätsreinigung mit blauem nativer Polyacrylamidgelelektrophorese der isolierten Komplexe, gefolgt von quantitativen Massen sp vereintectrometry und Proteinkorrelationsprofilerstellung, die mehrere Baugruppen ein Protein lösen könnten beteiligt werden. Wir embryonalen Stammzellen der Maus verwenden, wo ein Protein von Interesse an ein Epitop-Tag fusioniert ist, von dem endogenen Locus exprimiert physiologischen Fülle der Nähe zu erreichen und effizient nativen gewährleisten Komplex Isolation. Dieser Ansatz entwirrt die mehrere Wechselwirkungen ein Protein eingreift, so dass sie in verschiedene Baugruppen auf ihre Korrelationsprofile basierend Lösung, während erfordern keine Arbeit mehr als herkömmliche GELC-MS 20.
Protokoll
1. Isolierung von nativem Protein-Komplexe durch FLAG Affinitätsreinigung
- Die Vorbereitungen
- ein 37 ° C Wasserbad zum Auftauen des Zellpellets aufgebaut.
- Das Abkühlen der einen Mikro bis 4 ° C.
- Platzieren mehrere Reaktionsgefäße in verschiedenen Größen (1,5 ml, 15 ml) und ein Homogenisator in Eis.
- Herstellung von 10 ml Lysepuffer (50 mM Tris-HCl pH 8, 150 mM NaCl, 0,1% Nonidet P-40, 1 mM EDTA) und halten in Eis. Unmittelbar vor den Puffer zu verwenden, werden 10 ul 1 M DTT und ein zerkleinertes Tablette EDTA-freie Proteaseinhibitoren.
- Herstellung von Antikörpern-gebundenen Perlen
HINWEIS: Die Antikörper-beschichteten Beads bis zu einer Woche im Voraus vorbereitet werden kann und im Kühlschrank aufbewahrt, bis sie benötigt. Protein G hat eine höhere Affinität als Protein-A für die meisten Arten Subtypen Immunglobulin, mit Ausnahme von Kaninchen-IgG, für das Protein eine höhere Affinität hat.- Waschen Sie 50 ul Protein G-beschichteten magnetischen Kügelchen: Transfer 50 &# 956; l Kügelchenaufschlämmung zu einem Mikrozentrifugenröhrchen, das Reagenzglas in dem Magneten die Wülste auf der Seite des Röhrchens zu sammeln, und um die Flüssigkeit zu entfernen. Resuspendieren der Kügelchen in 0,5 ml PBS-0,01% Tween-20.
- Das Röhrchen wird in den Magneten die Wülste auf der Seite des Röhrchens zu sammeln, und um die Flüssigkeit zu entfernen. Resuspendieren der Perlen in 40 ul PBS-0,01% Tween-20. In 10 ug (10 ul von 1 mg / ml Stammlösung) von M2 anti-FLAG-Antikörper. Inkubieren mit Rotation für 20 min bei Raumtemperatur.
- Das Röhrchen wird in den Magneten die Wülste auf der Seite des Röhrchens zu sammeln, und um die Flüssigkeit zu entfernen. Waschen der Kügelchen mit 0,5 ml PBS-0,01% Tween-20.
- Für die Vernetzung des Antikörpers an die Beads, Waschen der Kügelchen zweimal mit 1 ml 0,2 M Triethanolamin pH 8,2. Dann Resuspendieren der Kügelchen in 1 ml 20 mM DMP (Dimethylpimelimidat) in 0,2 M Triethanolamin pH 8,2 (DMP-Lösung sollte frisch unmittelbar vor der Verwendung hergestellt werden). Inkubieren mit Rotation bei Raumtemperatur für 30 min.
- Das Röhrchen wird in dem Magneten. Entfernen Sie den Überstand und resuspendiere die Perlen in 0,5 ml 50 mM Tris-HCl pH 7,5. Inkubieren mit Rotation bei Raumtemperatur für 15 min.
- Waschen des Kügelchen 3-mal mit 0,5 ml PBS-0,1% Tween-20. Lassen Sie Perlen in der letzten Waschung bis sie benötigt werden.
- FLAG-basierte Affinitätsreinigung
HINWEIS: Achten Sie darauf, nicht für eine lange Zeit die Kügelchen ohne Puffer zu verlassen. Alle Puffer und Rohre sollten jederzeit in Eis gehalten werden. Das folgende Protokoll wird für die Reinigung von Proteinkomplexen optimieren 2-5 x 10 8 Zellen (10-20 mg Protein - Lysat) 20 endogenen Spiegel eines FLAG-markierten Protein exprimiert.- Auftauen das Zellpellet in einem Wasserbad bei 37 ° C, bis sie zu schmelzen beginnt. Nehmen Sie den Schlauch aus dem Bad, schwenken Sie sie vorsichtig, bis das Pellet vollständig aufgetaut ist, und sofort das Reagenzglas die Zellsuspension auf Eis enthält.
- In 5 ml eiskaltem Lysepuffer(Enthaltend DTT und Proteaseinhibitoren) zu der Zellsuspension und Verwirbelung zu vermischen. Inkubieren auf Eis für 10 min.
- Übertragen Sie das Lysat zu einer kalten Dounce-Homogenisator. Lyse mit 20-30 Hüben die enge Stößel (bis keine merkliche Viskosität). Übertragen Sie das Homogenat zu kalt Mikrozentrifugenröhrchen.
- Zentrifuge bei 18.000 xg für 15 min bei 4 ° C.
- Übertragen Sie das geklärte Lysat auf ein sauberes kaltes Rohr. Lassen Sie 50 ul Lysat hinter. Nehmen Sie ein 35-ul-Aliquot des Lysats Proteinkonzentration zu messen und um die Reinigung zu überwachen.
- Entfernen Sie die PBS-0,1% Tween-20 von den Kügelchen. mit dem Rest des Lysats Resuspendieren der Kügelchen, die mit 1 ml des Lysats und mischen. Inkubieren der Mischung in einem rotierenden Rad bei 4 ° C für 1-2 h.
- Sammelt die Wülste auf der Seite des Rohres mit dem Magneten. Sammeln Sie einen 30-ul-Aliquot des Überstandes und entsorgen Sie den Rest des Überstandes. Resuspendieren der Kügelchen in 0,5 ml IPP150-Puffer (10 mM Tris-HCl pH 8, 150 mM NaCl, 1 mM EDTA, 0,1% NP-40) durch 4-6 mal pipettiert. Transfer in ein 1,5 ml kaltes Rohr.
- Wiederholen Sie die Waschungen mit 0,5 ml Puffer IPP150 zwei weitere Male.
- Wash Kügelchen dreimal mit 0,5 ml FLAG nativen Elutionspuffer (20 mM Bis-Tris pH 7, 20 mM NaCl, 0,02% Nonidet P-40, 1 mM EDTA, 200 mM ε-Aminocapronsäure). Mit der letzten Wäsche, übertragen Sie die Perlen zu einem neuen kalten Rohr. Entfernen Puffer gründlich.
- Resuspendieren der Kügelchen in 100 ul von 200 ug / ml 3x FLAG-Peptid in nativen Elutionspuffer. bei 4 ° C inkubieren für 10 min unter leichter Rotation.
- Sammeln die Kügelchen mit dem Magneten und den Überstand zu einem kalten neuen Röhrchen.
- Wiederholen Sie die Schritte 1.3.10-1.3.11 zweimal. Pool alle Eluate.
- Konzentriere das Eluat bis auf 25 & mgr; l in einer Zentrifugal-Filtereinheit (10 kDa nominale Molekulargewichtsgrenze abgeschnittene, PES) durch Zentrifugation bei 10.000 × g bei 4 ° C. Übertragen Sie das konzentrierte Eluat auf einen neuen kalten Rohr und 50% glycerol für eine Endkonzentration von 5%. Das konzentrierte Eluat kann über Nacht bei 4 ° C gehalten werden, wenn erforderlich.
2. Blaue native PAGE
- Die Vorbereitungen
- Bereiten Sie 1 l 1x native PAGE Anodenpuffer, 1x native PAGE Dark Blue Cathode Buffer und 1x native PAGE Light Blue Cathode Buffer.
- Nehmen Sie den Kamm einer vorgefertigten 3-12% native PAGE-Gel und waschen Sie die Vertiefungen zweimal mit 1x native PAGE Dark Blue Cathode Buffer. Füllen Sie den Wells mit 1x native PAGE Dark Blue Cathode Buffer. Legen Sie das Gel im laufenden Tank, aber nicht Dark Blue Kathodenpuffer zur Kathode (innen) Kammer hinzuzufügen.
- Elektrophoreselauf
- Bereiten Sie die Probe: Zu den 25 ul konzentrierten Eluat geeignete Volumen 4x nativem Probenladungspuffer hinzuzufügen und von 0,5% G-250 Probe Additiv für Endkonzentrationen von 1x und 0,005% betragen.
- Laden Sie die Probe und nativen Molekulargewichtsmarker eine leere gut verlassen between ihnen. Last 10-20 ul 1x nativen Probe Ladepuffer in allen leeren Brunnen.
- Füllen Sie die Kathode (innen) Kammer mit 200 ml 1x native PAGE Dark Blue Kathodenpuffer vorsichtig, um die Proben nicht zu stören. Füllen der Anode (außerhalb) Kammer mit 550 ml 1x native PAGE Anodenpuffer.
- Führen Sie das Gel bei 150 V für 30 min. Stoppen Sie den Lauf, entfernen Sie den dunkelblauen Kathodenpuffer mit einer serologischen Pipette und ersetzt mit 1x Light Blue Cathode Buffer. Weiter den Lauf für 60 min. Das Gel kann bei Raumtemperatur oder bei 4 ° C ausgeführt werden; die Temperatur könnte eine Auswirkung auf die Protein komplexe Struktur haben.
- Gel-Färbung
- Öffnen der Gelkassette und entsorgen eine der Kassettenplatten. Das Gel bleibt an der anderen Kassettenplatte befestigt. Übertragen, um das Gel in eine Schale oder Behälter genügend Fixierlösung (40% Methanol, 2% Essigsäure) enthält, um das Gel zu bedecken, und für 30 min unter leichtem Schütteln inkubieren.
- Entfernen Sie die Fixiermittel solutIon und genug Wasser hinzu, das Gel zu bedecken. Das Gel kann zum späteren Nachschlagen gescannt werden.
3. massenspektrometrischen Analyse
Hinweis: Alle nachfolgenden Schritte sollten in einer laminaren Strömungshaube durchgeführt werden, wenn möglich Sauberkeit der Proben zu gewährleisten. Alle Lösungen sollten mit HPLC-Wasser hergestellt werden. Diskutieren Sie dieses Protokoll mit dem Mass Spectrometry Labor, die die Analyse durchführen wird.
- Die Vorbereitungen
- Legen Sie eine konische Boden 96-Well-Platte auf einer anderen 96-Well-Platte um den Abfall zu sammeln. Pierce der Boden der Vertiefungen der konischen Bodens 96-Well-Platte mit einer 21 G-Nadel.
- Waschen Sie die Vertiefungen der durchbohrten 96-Well-Platte.
- Zugabe von 200 ul 50% Acetonitril-0,25% Ameisensäure in den Vertiefungen der durchbohrten Platte. Inkubieren der Plattenstapel in einem Schüttler für 10 min.
- Zentrifuge bei 500 × g für 1 min, so daß die Flüssigkeit fließt durch die Löcher in die Abfallsammelplatte. Entsorgen Sie die wASTE Flüssigkeit.
- Wiederholen Sie die Schritte 3.1.2.1-3.1.2.2 zwei weitere Male.
- Füllt die Vertiefungen der durchbohrte Platte mit 96 Vertiefungen mit 150 ul 50 mM Ammoniumbicarbonat (frisch hergestellt).
- Wasche die Vertiefungen einer Zentrifugalfiltration 96-Well-Platte.
- Legen Sie eine normale 96-Well-Platte unter der zentrifugalen Filtrationsplatte um den Abfall zu sammeln. Zugabe von 200 ul 50% Acetonitril-0,25% Ameisensäure zu jeder Vertiefung der zentrifugalen Filtrationsplatte.
- Zentrifugiert den Plattenstapel bei 200 × g für 1 min. Entsorgen Sie den Abfall.
- Wiederholen Sie die Schritte 3.1.3.1-3.1.3.2 zwei weitere Male.
- Gel Exzision und in-Gel-Verdauung
HINWEIS: Während das Gel Exzision, identifizieren und eine Notiz von dem Gelstück zu jedem der Protein-Standards ausgerichtet werden. Die Proteinstandards können ungefähre Molekulargewichte für alle Gelschnitten zu schätzen.- Das Kieselgel wird mit der Glasplatte auf einer sauberen Oberfläche. Schneiden Sie die Spur enthaltening der Probe in 48 identische Scheiben (1,5 mm x 5 mm). Schneidet jede Scheibe in 2-3 kleinere Stücke (mit Ausnahme der letzten fünf Scheiben am oberen Ende des Gels) und legt jede Scheibe sequentiell in einer Vertiefung der durchstochenen und wusch 96-Well-Platte.
- In 50 ul Acetonitril zu jeder Vertiefung. Inkubiere die Platte für 30-60 min unter Schütteln. Entfernen Sie die Flüssigkeit durch Zentrifugieren, wie in Schritt 3.1.2.2.
- Zugabe von 200 ul 2 mM TCEP in 50 mM Ammoniumbicarbonat in jede Vertiefung. Inkubiere die Platte für 30 min bei Raumtemperatur unter Schütteln. Entfernen Sie die Flüssigkeit durch Zentrifugieren, wie in Schritt 3.1.2.2.
- Zugabe von 200 ul 4 mM Iodacetamid in 50 mM Ammoniumbicarbonat in jede Vertiefung. Inkubiere die Platte für 30 min bei 37 ° C im Dunkeln unter Schütteln. Entfernen Sie die Flüssigkeit durch Zentrifugieren, wie in Schritt 3.1.2.2.
- Zugabe von 150 ul 50 mM Ammoniumbicarbonat plus 50 & mgr; l Acetonitril und inkubiere die Platte für 30 min bei Raumtemperatur unter Schütteln. Wiederholen Sie diesen Waschschritt eins oft wie erforderlich, bis alles blaue Farbe aus den Gelstückchen entfernt wird.
- Entwässern die Gelstücke durch 200 & mgr; l reinem Acetonitril Zugabe und Inkubation bei Raumtemperatur für 20 min unter Schütteln, bis die Gelstücke weiß und opak sind. Entfernen Acetonitril.
- Zugabe von 150 & mgr; l 0,001 mg / ml Trypsin (sequencing grade) in kaltem 50 mM Ammoniumbicarbonat in jede Vertiefung. Inkubiere die Platte bei 37 ° C für 2 Stunden unter Schütteln. Nach 1 h, zu überprüfen, dass die Gelstücke mit Flüssigkeit bedeckt ist; Ist dies nicht der Fall ist, fügen eine weitere 50-100 ul 50 mM Ammoniumbicarbonat. Nach 2 h bei 37 ° C, über Nacht weiter die Verdauung bei 25 ° C.
- Ein sauberes konisches Bodenplatte unter dem Gel enthaltenden Platte, korrekte Ausrichtung der Platten zu gewährleisten. Sammeln Sie die Flüssigkeit, welche die Peptide durch Zentrifugation bei 200 × g für 1 min enthält.
- Platzieren Sie die Peptide enthaltende Platte in einem Zentrifugalverdampfer verdampft und die Flüssigkeit während der Durchführung ter nächster Schritt.
- Zugabe von 150 ul 50% Acetonitril-0,25% Ameisensäure in die Gelstücke. Inkubieren mit 30 min bei 37 ° C schüttelnd.
- Sammeln Sie die Flüssigkeit in die teilweise getrockneten Platte aus Schritt 3.2.9 durch Zentrifugation bei 200 × g für 1 min. Weiterhin die Peptide enthaltenden Platte Verdampfen während des nächsten Schritt ausführen.
- Wiederholen Sie die Schritte 3.2.10-3.2.11.
- Füge 200 & mgr; l reinen Acetonitril zu jeder Vertiefung der Platte, die Gelstücke enthält. Inkubieren mit 15 min bei Raumtemperatur geschüttelt.
- Sammeln Sie die Flüssigkeit in die Peptide enthaltenden Platte durch Zentrifugation bei 200 × g für 1 min.
- Achten Sie darauf, die endgültige Konzentration von Acetonitril in allen Vertiefungen über 55% liegt. Falls erforderlich, fügen Sie zusätzlichen reinen Acetonitril.
- Überträgt die Peptidlösungen zu der gewaschenen Zentrifugenfiltrationsplatte. Legen Sie eine saubere konische Bodenplatte unter ihm die Peptide zu sammeln. Zentrifugiert den Plattenstapel bei 200 × g für 1 min.
- Evaporaß die Flüssigkeit in der konischen Bodenplatte, bis Vertiefungen vollständig trocken sind. Die Platte kann mit einem Silikondeckel und gelagert bei -20 ° C in diesem Stadium abgedeckt werden.
- Wieder aufzulösen Peptiden in 32 & mgr; l von 0,5% iger Ameisensäure unter heftigem Schütteln für 15 min. Aus 8 & mgr; l 400 mM Ammoniumbicarbonat und inkubiere unter heftigem Schütteln für 15 min. Zentrifugieren der Platte bei 200 × g für 1 min. Deckel mit einem Silikondeckel und speichern die Platte bei -20 ° C bis zur Massenspektrometrieanalyse.
- Massenspektrometer
HINWEIS: Analysieren Peptide auf einem hohen Massenspektrometers Auflösung unter Verwendung von Methoden kompatibel mit Shotgun Proteomics. Datenabhängige Akquisition ist die am häufigsten verwendeten LC-Tandem-MS-Set-up für diese Art von Analyse und soll für eine effiziente Peptid Identifizierung optimiert werden, redundante Sequenzierung und Kandidatenauswahl über Hintergrundrauschen zu minimieren. Massenspektren in der ersten Stufe der Massenanalyse-Scan (MS1) sollten im Profil-Modus aufgezeichnet werden. Es wird empfohlen,ed, um vorzugsweise doppelt und dreifach geladene Ionen für die zweite Stufe der Massenanalyse (MS2) auszuwählen. Ein Massenbereich m / z 400-1,600 ist geeignet, um die meisten Peptide zwischen 8-30 Aminosäuren Bereich zu identifizieren. Hier wird ein Protokoll für die Analyse eines Orbitrap-Massenspektrometer bereitgestellt.- Einzuzuspritzen 20 ul Probe pro Analyse, um den Auto-sampler eines nanoLCs-MS / MS-System. Entsalzen und konzentrieren Peptide auf einem Umkehrphasen-C18-Säule (100 & mgr; m id, 2 cm).
- Separate Peptide auf einer analytischen C18-Säule (75 um ID, 15 cm) mit einem 30 min linearen Gradienten von 4-28% Acetonitril / 0,1% Ameisensäure bei 300 Nl / min verwendet wird.
- Analysieren Peptide in einem Massenspektrometer mit einem Oberseite 10 datenabhängigem Erfassungsverfahren mit den folgenden Parametern: m / z-Bereich 380-1,600, Auflösung 30.000 bei m / z 400, Isolationsbreite von 2 Th, dynamische Ausschlüsse bei ± 10 ppm für 45 s automatisches Verstärkungssteuerziel bei 1 x 10 6 für MS und 5.000 für MS / MS, maximale Einspritzzeit 150 ms für MS und100 ms für MS / MS.
4. Datenanalyse
- Protein Identifizierung und Quantifizierung unter Verwendung MaxQuant
- Verwenden Sie die frei verfügbare MaxQuant Software zu identifizieren und zu quantifizieren Proteine in jeder Gelfraktion von dem Massenspektrometrie Rohdaten.
HINWEIS: MaxQuant arbeitet auf Daten, die von datenabhängige Erfassung Shotgun Proteomics Ansätzen. Die MS-Daten von den blauen nativen Gel Fraktionen sollten in Charge als separate Experimente analysiert werden und nicht als Brüche eines Experiments Kombination aller Gelschnitten. Überprüfen Sie die iBAQ Wertefeld Stöchiometrie Berechnungen durchzuführen. Detaillierte Protokolle für die Datenbanksuche und Proteinquantifizierung MaxQuant verwenden , sind in 21 beschrieben, 22.
- Verwenden Sie die frei verfügbare MaxQuant Software zu identifizieren und zu quantifizieren Proteine in jeder Gelfraktion von dem Massenspektrometrie Rohdaten.
- Proteinanalyse Profil mit Perseus
- Verwenden Sie die frei verfügbare Software Perseus die Migrationsprofile der identifizierten Proteine anzuzeigen, und vergleichen sie mitstatistische Analyse.
HINWEIS: Allgemeine Dokumentation, wie Perseus zu verwenden ist bei http://www.coxdocs.org/doku.php?id=perseus:user:tutorials zur Verfügung. Ein Beispieldatensatz ist, als zusätzliche Daten verfügbar. - Hochladen der Datei durch die proteingroups.txt MaxQuant Analyse enthaltend die Protein-Identifikationen und Quantifizierungswerte für alle Gelfraktionen zurückgegeben. Bevölkern der Intensitätswerte in das Hauptfeld (1). Eine Matrix wird erscheinen alle Protein Identifikationen in Reihen enthält, mit den Gelfraktionen als Spalten.
- Entfernen von Einträgen entsprechende Treffer umkehren, Proteinen nur durch ort identifiziert und potenzielle Verunreinigungen durch Filter Reihen Auswählen basierend auf der Kategorie Spalte in der Filterreihe Dropdown - Menü (Figur 2).
- Normalisieren der Fraktion Intensitäten jedes Proteins über das Profil gegen das Gesamtprotein Intensität durch Dividieren im Normalisieren Dropdown - Menü auswählen, dann Sum (Abbildung 3). Ein neues matrix erscheint.
- Zeigen Sie das Migrationsprofil Plots aller Proteine , die durch die Auswahl Profil Plot im Visualisierungs - Dropdown - Menü (Abbildung 4).
- Wählen Sie Filter Zeilen basierend auf gültigen Werte in der Filterreihen Dropdown-Menü. Geben Sie 1 in der Anzahl der gültigen Werte erforderlich.
- Führen Sie hierarchisches Clustering von identifizierten Proteinen in allen blauen nativen Gel-Fraktionen von Hierarchical Clustering im Clustering / PCA Drop-Down-Menü auswählen. Deaktivieren der Columns Baumkasten (Abbildung 5). Die Cluster werden in einem wärme Karte in der Clustering Lasche neben der Matrix (6) sichtbar gemacht.
- Suchen Sie den Cluster, den Köder-Protein in der dendrogram enthält. Andere Komponenten des Cluster repräsentieren Proteine mit dem Köder Co-Migration und sind daher potentielle Proteine / Untereinheiten des gleichen Komplexes interagiert.
- Verwenden Sie die frei verfügbare Software Perseus die Migrationsprofile der identifizierten Proteine anzuzeigen, und vergleichen sie mitstatistische Analyse.
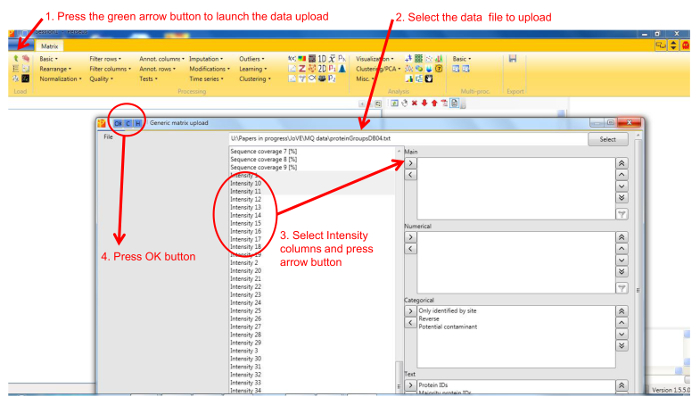 r /> Abbildung 1. Screenshot von Perseus Daten - Upload - Fenster. Die Figur zeigt die Schritte zum Laden Protein-Identifizierung und Quantifizierung von Daten in Perseus. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
r /> Abbildung 1. Screenshot von Perseus Daten - Upload - Fenster. Die Figur zeigt die Schritte zum Laden Protein-Identifizierung und Quantifizierung von Daten in Perseus. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.

Abbildung 2 Screenshot von Perseus Fenstern für die Filterung von Einträgen zu Verunreinigungen entsprechenden, umgekehrte Hits und Proteine durch ort identifiziert. Filter Zeilen Auswählen von kategorischen Spalte öffnet sich ein neues Fenster für die Arten von Protein-Einträge Auswahl aus dem Datensatz eliminiert werden. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
/55498fig3.jpg“/>
Abbildung 3. Screenshot von Perseus Normalisierungsfenster. Auswählen von Teilen im Normalisierungs Menü öffnet sich ein neues Fenster mit verschiedenen Optionen. Sum Auswahl unterteilt den Intensitätswert eines Proteins in jeder Fraktion durch die Gesamtintensität dieses Proteins in allen Fraktionen. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.

Abbildung 4. Screenshot von Perseus Migrationsprofil Plots zeigt. Proteine können unter den Profilen in der Matrix ausgewählt werden, um ihr entsprechendes Profil zu markieren. Die Werkzeugleiste kann verwendet werden, um die Profile zu bearbeiten und exportieren. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.

Abbildung 5. Screenshot von Perseus hierarchischen Clustering - Fenster. Die Figur zeigt die Einstellungen für hierarchische Clustern von Proteinen Manhattan (L1) Distanzmetrik verwendet wird. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
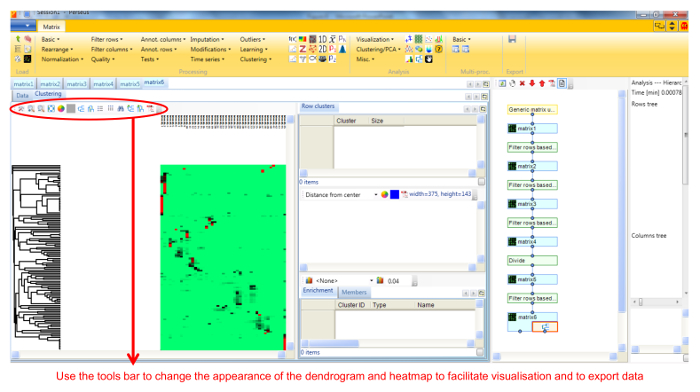
Abbildung 6. Screenshot von Perseus zeigt dendrogram und Proteinintensitäten Heatmap. Der Arbeitsablauf auf der rechten Seite der Hauptplatte zeigt jeden Schritt durchgeführt, und die resultierende Matrix, und kann dazu verwendet werden, die mit Schritt rückgängig zu machen. Der Arbeitsablauf kann auch aus jeder beliebigen Zwischenmatrix verzweigen. Bitte klicken Sie hier , um eine größere Version dieses f anzuzeigenild.
- Berechnung der relativen Stöchiometrie
- Wählen Sie die Untereinheiten eines Proteinkomplexes auf der Grundlage ihrer Co-Clustering mit dem Köderprotein und begrenzen den Profilspitze (n), in dem sie erscheinen.
- Verwenden, um die Werte, die durch iBAQ MaxQuant Analyse für jede Untereinheit in jeder Fraktion getrennt und normalisieren durch den entsprechenden Köder iBAQ Wert in dieser Fraktion zurückgeführt.
- Zeichnen Sie die normalisierte iBAQs für Köder und Proteinkomplex Untereinheiten über die Fraktionen des ausgewählten Peaks und lineare Regression anwenden. Berechnen relative Menge von komplexer Untereinheit Köder durch die Trends ihrer normalisierten iBAQ Werte zu vergleichen.
- Die Einschätzung der Proteinkomplex Größe
- Verwenden, um die Proteinstandards zu vorhergesagten Molekulargewichte für den ausrichtende blauen nativen Gelscheiben aus der Probenspur zu berechnen. Aus diesem erzeugt ein lineares Regressionsmodell Gelscheiben in der x - Achse und die Molekulargewichte in der y - Achse durch Auftragen.
- Verwenden Sie das Modell des beobachtete Molekulargewichtsbereich des Proteinkomplexes zu schätzen (n), basierend auf der blaue nativer slice (s), aus denen sie identifiziert wurden.
Ergebnisse
Der Arbeitsablauf der Affinitätsreinigung Blau native Protein Correlation Profilierung durch Massenspektrometrie (ABC-MS) Strategie ist in Abbildung 7 dargestellt. Native Protein-Komplexe um ein Protein von Interesse werden durch Affinitätsreinigung isoliert Antikörper gegen einen Epitop-Tag (in diesem Fall FLAG) und kompetitive Elution mit. Die Komplexe werden durch BN-PAGE aufgetrennt, und die ganze Gelspur ist in 48 Abschnitte herausgeschnitten und für Schrot LC-MS / MS. Quantitative MS Informationen werden verwendet, um ein Migrationsprofil für jedes identifizierte Protein über die blaue nativen Trennung zu erzeugen. Proteine, die interagieren, ein deutliches komplexe Anzeige ähnliche Migrationsprofile mit deckungs Spitzen bilden. Wenn ein Protein von Interesse an mehr als eine Baugruppe erfolgt, werden mehrere Peaks in ihrem Migrationsprofil beobachtet, da die Teilkomplexe sind innerhalb des Auflösungsvermögens des blauen nativen Gels. Ein systematischer Vergleich der migRation Profile können durch Protein-Korrelation erreicht werden Profilierungs hierarchische Clustern unter Verwendung. Das Dendrogramm und die Spitzenintensitäten für alle Fraktionen werden in einem wärme Karte visualisiert, erleichtert die Identifizierung Proteinen in Wechselwirkung zu treten, die auf unterschiedliche Proteinkomplexen gehören (Abbildung 8).
Wir nutzten diese Strategie , um die Interaktionspartner von MTA2 zu analysieren, eine Kernuntereinheit der NuRD Chromatin - Komplex 20 Umbau. Wie in 9 gezeigt ist , zeigte das Migrationsprofil von MTA2 zwei Peaks unterschiedlicher Intensitäten zwischen 700 kDa und 1,2 MDa, mit der unteren Massenspitze höher Fülle anzeigt. Andere NuRD Kern - Untereinheiten, einschließlich MTA1 / 3, HDAC1 / 2 und MBD3 identisches Trennungsmuster zeigten, wenn auch Peaks , das für einige der Untereinheiten, nämlich CHD4, Gatad2a / b und Rbbp4 / 7, hatten die inverse Häufigkeitsverteilung (9A, B). Im Gegensatz dazu das Profil von Cdk2ap1, ein Regulationsfaktor, der den Komplex zu NuRD Wnt Genpromotoren rekrutiert 23, dargestellt nur die höheren Massenpeak (Abbildung 9C), und so hat Sall4, einen transkriptionalen Repressor, der auch 20 NuRD zu binden , gezeigt worden ist, 24, 25. Somit Fraktionierung von affinitätsgereinigtem MTA2-assoziierte Proteine, die durch blaue native PAGE konnte zwei verschiedene Formen des NuRD Komplex lösen.
ABC-MS ermöglicht auch die Identifizierung von neuen Interaktoren, während sie zu bestimmten Protein Einheiten zuweisen. Durch Untersuchung der Proteine Cluster und Fraktion in einer Wärme Karte dargestellt Intensitäten detektiert wir eine starke Korrelation zwischen Untereinheiten und NuRD WDR5, eine regulatorischen Untereinheit des MLL - Methyltransferase - Komplexes 26 mit WDR5 Anzeige von zwei Migrations Peaks mit dem beide NuRD p koinzidenteaks (Abbildung 8), eine neuartige Wechselwirkung zwischen WDR5 und NuRD hindeutet. Wir bestätigten diese Interaktion durch Co-Immunpräzipitation und Co-Migration in Grßenausschlußchromatographie 20.
Die Auswahl der Abstandsmetrik-Berechnung in dem hierarchischen Clustering wird die Form der Cluster beeinflussen und damit die Zusammenhänge. Wir empfehlen, mit den verschiedenen Metriken experimentieren die beste Lösung mit bestehenden Interaktion Wissen des Proteins oder ein Komplex von Interesse zu erreichen. Im allgemeinen erreicht man die besten Ergebnisse mit der Manhattan (L1) Distanzmaß 20. Pearson - Korrelation oder euklidische Abstandsmetriken sind auch zur Identifizierung von Komplexen basierend auf alternative Fraktionierungstechniken 10 berichtet, 11, 12.
die quantitative MS Daten aus den blauen nativen erhaltenen Fraktionen können auch die Stöchiometrie von Proteinkomplexen zu bestimmen, verwendet werden. Die iBAQ (Intensität basierend absolute Quantifizierung) -Wert liefert ein Maß für die relative Häufigkeit der identifizierten Proteine 27, 28. Die iBAQ Werte für die Protein-Komplex Mitglieder in allen Fraktionen innerhalb einer peak Migrationsprofil sind, dass der Köder-Protein normalisiert relativen Mengen zu erhalten. Die normalisierten iBAQs über eine Profilspitze für ein Protein-Komplex gegeben Element sollten einen horizontalen Trend folgen, und die Trendwerte spiegeln die Stöchiometrie der interagierenden Proteine relativ zu dem Köder-Protein. Für eine detailliertere Darstellung der Stöchiometrie Berechnungen, siehe 20.

Abbildung 7: Schematische Workflow einer Zusammenfassung der ABC-MS strategie. Diese Zahl wird aus 20 modifiziert ist . Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
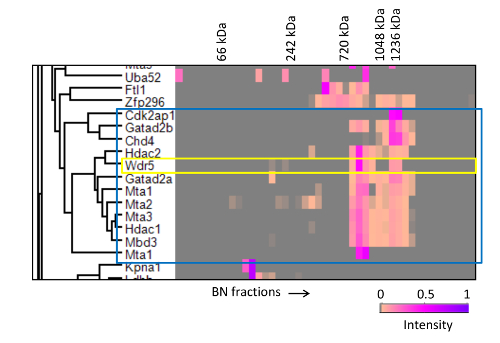
Abbildung 8. Hierarchical Clustering von BN-PAGE Migrationsprofile der MTA2 Interaktoren. MTA2-assoziierte Proteine wurden auf der Grundlage der Ähnlichkeit ihrer Migrationsprofile gruppierten. Nur eine Teilmenge der wärme Karte des NuRD Komplex enthält, wird (eingeschlossen in der blauen Box) gezeigt. Das gelbe Feld unterstreicht die starke Korrelation von WDR5 mit dem NuRD Komplex. Die kommentierten Molekulargewichte wurden aus den Wanderungsstrecken von Proteinstandards in demselben Gel laufen geschätzt. Diese Forschung wurde ursprünglich in Molecular and Cellular Proteomics 20 der amerikanischen Gesellschaft für Biochemie und Mo veröffentlicht molekulare Biologie. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
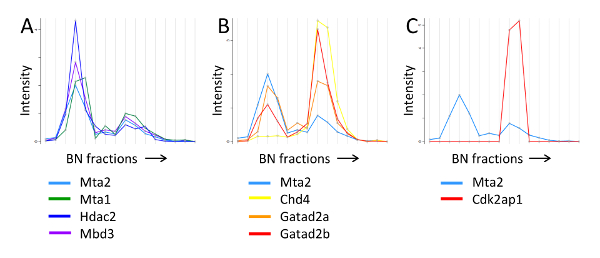
Abbildung 9. BN-PAGE Migrationsprofil von NuRD Untereinheiten und assoziierten Proteinen. A) MTA2 und NuRD Untereinheiten , die in einem ähnlichen Intensitätsmuster. B) MTA2 und NuRD Untereinheiten mit dem inversen Intensitätsmuster. C) MTA2 und Cdk2ap1, die in der höheren Molekulargewicht NuRD Entität nur vorhanden ist. Die kommentierten Molekulargewichte wurden aus dem Migrationsprofil der Proteinstandards geschätzt. Diese Forschung wurde ursprünglich in Molecular and Cellular Proteomics 20 der amerikanischen Gesellschaft für Biochemie und Molekularbiologie veröffentlicht._blank "> Bitte hier klicken, um eine größere Version dieser Figur zu sehen.

Ergänzende Daten: Beispieldatensatz. MaxQuant abgeleitete proteingroups.txt Datei Protein Identifikationen und Quantifizierungswerte von einem ABC-MS-Experiment enthält. Diese Forschung wurde ursprünglich in Molecular and Cellular Proteomics 20 der amerikanischen Gesellschaft für Biochemie und Molekularbiologie veröffentlicht. Bitte klicken Sie hier , um diese Datei herunterzuladen.
Diskussion
Hier beschreiben wir die Verwendung von Affinitätsreinigung gefolgt von blau native Gelelektrophorese in Kombination mit quantitativen Massenspektrometrie Proteinkomplexe aufzulösen. Dieser Ansatz bietet eine Methode eindimensional Protein-Wechselwirkung Listen in funktionelle Proteinkomplexe zu entwirren.
Wir zeigen das Verfahren basiert auf der Verwendung von Epitop-markierten Proteinen. Wenn jedoch eine Zelllinie ein markiertes Protein exprimieren, nicht verfügbar ist, könnte eine Alternative sein, Antikörper gegen das Protein von Interesse zu verwenden, sofern ein Peptid zur Verfügung nativen kompetitive Elution zu erreichen. Die Menge des Ausgangsmaterials und der Menge der Perlen kann in Abhängigkeit von dem Expressionsspiegel des Zielproteins werden müssen modifiziert. Wir führen in der Regel dieses Protokoll mit 2-5 x 10 8 Zellen - Ausgangsmaterial, und dies ist ausreichend , um auch die für Proteine mit niedrigem Expressionsniveau. Die Lysepuffer Zusammensetzung sollte empirisch gewählt werden, um zu erreichen,in der Nähe von vollständiger Auflösung des Köders. Dies kann für einige Arten von Proteinen, insbesondere Chromatin-Bindung oder Membranproteine schwierig sein. Alternative Optionen gehören die Erhöhung die Menge an Salz, vorausgesetzt , daß der Proteinkomplex in Untersuchung in hohem Salz stabil ist, oder unter Verwendung von Ultraschall - Behandlung und / oder Nuklease - Behandlung für Chromatin - Bindungsproteine 20, 29. Im Falle von Membranproteinen, können Waschmittel DDM oder Digitonin Schalt 30 sinnvoll sein. Im Fall von DNA - Bindungsproteinen, ist es sinnvoll , eine Nuklease Benzonase wie während der Reinigungsstufe 20 umfasst. Die vollständige Entfernung von Nukleinsäuren gewährleistet, dass die detektierten Wechselwirkungen zwischen Proteinen auftreten und nicht durch DNA vermittelt.
Ein kritisches Element zu berücksichtigen, wenn mit diesem Ansatz die Stabilität des Komplexes untersucht wird. Das Verfahren ist lang und preservin beinhalten kanng der Proteinkomplex über Nacht. Wir haben gute Erfolge mit zwei Chromatin-Remodeling-Komplexe (D. Bode und M. Pardo, Daten nicht gezeigt) erreicht, aber dies ausgewertet werden soll. Der Affinitätsreinigungsschritt kann bei Bedarf gekürzt werden.
Alternative Techniken, die native Fraktionierung ermöglichen, wie Grßenausschlußchromatographie, sind seit mehr als 50 Jahren zu charakterisieren Proteinkomplexe weit verbreitet. Wir und andere haben gezeigt , dass die Auflösung der blauen native PAGE auf , dass 20, 31, 32, 33 unter Verwendung von Grßenausschlußchromatographie erreicht überlegen ist. Ein weiterer Vorteil der blauen native PAGE ist, dass es nicht Chromatographie-Systeme erfordert, die teuer sind, sondern nutzt Protein elektrophoretische Geräte, die in Laboratorien weit verbreitet ist. In Bezug auf die Hands-on-Zeit, diese Methode beinhaltet nicht mehr Arbeit als die traditionelle GELC-MS / MS einNSATZ oder offline chromatographische Fraktionierung. Da jedoch die meisten Fraktionierungsverfahren tun, hat es eine Grenze für ihre Auflösung. Komplexe, die sehr homogen oder in der Nähe in Masse und Form sind, können über die Auflösung von blau native PAGE angeboten werden, und somit das Protokoll wie hier berichtete nicht sein könnte allgemein erfolgreich bei der Lösung verschiedene Komplexe mit gemeinsamen Untereinheiten. Erfreulicherweise haben wir bei der Trennung von zwei sehr ähnliche tetramere Komplexe teilen drei Untereinheiten (M. Pardo, Manuskript in Vorbereitung) erfolgreich.
Da Massenspektrometrie zunehmend empfindlich geworden ist, auch kleinste Mengen an unspezifischer Interaktoren und Verunreinigungen können in AP-MS Proben nachgewiesen werden. Der hier vorgestellte Ansatz könnte bei der Unterscheidung von echten Interaktionspartner von Hintergrundbelastung in der Affinitätsreinigung helfen, indem die Aufmerksamkeit auf Proteine mit Migrationsspitzen zu konzentrieren, die als bei höheren Molekulargewicht mit Ködern Migration Spitzen zusammenfallen, dass diedie monomeren Proteine.
Mehrere Gruppen haben Fraktionierungstechniken durch Protein - Korrelationsprofilierungs gefolgt verwendet , um Protein - Komplexe auf Zell Maßstab ohne die Notwendigkeit einer vorherigen Isolierung 10, 11, 12, 34 abzugrenzen. Dies kann jedoch dazu führen, substöchiometrischen Wechselwirkungen zu erkennen. Der Einbau eines Anreicherungsschritt durch Affinitätsreinigung kann helfen, diese zu überwinden. Der hier beschriebene Ansatz sollte für die Erkundung der Topologie von Proteinkomplexen und Entschlüsselung der mehrere Komplexe ein bestimmtes Protein an innerhalb der gleichen zellulären Kontext dauert in der Regel nützlich sein. Die Strategie ist einfach und zugänglich für Laboratorien, die nicht teuer chromatographische Fraktionierung Ausrüstung können Proteinkomplexe zu lösen.
Offenlegungen
The authors have nothing to disclose.
Danksagungen
This work was funded by the Wellcome Trust (WT098051).
Materialien
| Name | Company | Catalog Number | Comments |
| Protein G Dynabeads | Life Technologies | 10003D | |
| FLAG antibody M2 | Sigma-Aldrich | F1804 | |
| Tween-20, protein grade, 10% solution | Millipore | 655206 | |
| Dimethyl pimelimidate | Sigma-Aldrich | 80490 | |
| 0.2 M Triethanolamine buffer, pH 8.2 | Sigma-Aldrich | T0449 | |
| 3x FLAG peptide | Sigma-Aldrich | F4799 | |
| Vivaspin 5K NMWCO, PES | Sartorius | VS0112 | |
| NativePAGE 3-12% Bis-Tris gel | Life Technologies | BN1001 | |
| 20x NativePAGE Running Buffer | Life Technologies | BN2001 | |
| 20x NativePAGE Cathode Additive | Life Technologies | BN2002 | |
| 4x NativePAGE sample loading buffer | Life Technologies | BN2003 | |
| NativeMARK | Life Technologies | LC0725 | |
| NativePAGE 5% G-250 sample additive | Life Technologies | BN2004 | |
| Nunc conical bottom 96-well plate, polypropylene | Thermo | 249944 | |
| Multiscreen HTS 96-well filtration system | Thermo | MSDVN6550 | |
| Nunc 96-well cap natural | Thermo | 276002 | |
| TCEP (Tris(2-carboxyethyl)phosphine hydrochloride) | Sigma-Aldrich | 646547 | |
| Iodoacetamide | Sigma-Aldrich | I1149 | |
| Acetonitrile, HPLC grade | Fisher Scientific | A/0627/17 | |
| Trypsin, sequencing grade | Roche | 11418475001 | |
| MaxQuant (software) | N.A. | N.A. | http://www.biochem.mpg.de/5111795/maxquant |
| Perseus (software) | N.A. | N.A. | http://www.biochem.mpg.de/5111810/perseus |
Referenzen
- Goudreault, M., et al. A PP2A phosphatase high density interaction network identifies a novel striatin-interacting phosphatase and kinase complex linked to the cerebral cavernous malformation 3 (CCM3) protein. Mol Cell Proteomics. 8 (1), 157-171 (2009).
- Leitner, A., et al. Probing native protein structures by chemical cross-linking, mass spectrometry, and bioinformatics. Mol Cell Proteomics. 9 (8), 1634-1649 (2010).
- Rappsilber, J. The beginning of a beautiful friendship: cross-linking/mass spectrometry and modelling of proteins and multi-protein complexes. J Struct Biol. 173 (3), 530-540 (2011).
- Sinz, A. Chemical cross-linking and mass spectrometry to map three-dimensional protein structures and protein-protein interactions. Mass Spectrom Rev. 25 (4), 663-682 (2006).
- Walzthoeni, T., Leitner, A., Stengel, F., Aebersold, R. Mass spectrometry supported determination of protein complex structure. Curr Opin Struct Biol. 23 (2), 252-260 (2013).
- Kaake, R. M., et al. A new in vivo cross-linking mass spectrometry platform to define protein-protein interactions in living cells. Mol Cell Proteomics. 13 (12), 3533-3543 (2014).
- Kao, A., et al. Development of a novel cross-linking strategy for fast and accurate identification of cross-linked peptides of protein complexes. Mol Cell Proteomics. 10 (1), (2011).
- Hughes, M. A., Langlais, C., Cain, K., MacFarlane, M. Isolation, characterisation and reconstitution of cell death signalling complexes. Methods. 61 (2), 98-104 (2013).
- Lim, S., Zou, Y., Friedman, E. The transcriptional activator Mirk/Dyrk1B is sequestered by p38alpha/beta MAP kinase. J Biol Chem. 277 (51), 49438-49445 (2002).
- Havugimana, P. C., et al. A census of human soluble protein complexes. Cell. 150 (5), 1068-1081 (2012).
- Kirkwood, K. J., Ahmad, Y., Larance, M., Lamond, A. I. Characterization of native protein complexes and protein isoform variation using size-fractionation-based quantitative proteomics. Mol Cell Proteomics. 12 (12), 3851-3873 (2013).
- Kristensen, A. R., Gsponer, J., Foster, L. J. A high-throughput approach for measuring temporal changes in the interactome. Nat Methods. 9 (9), 907-909 (2012).
- Liu, F., Heck, A. J. Interrogating the architecture of protein assemblies and protein interaction networks by cross-linking mass spectrometry. Curr Opin Struct Biol. 35, 100-108 (2015).
- Wittig, I., Schagger, H. Native electrophoretic techniques to identify protein-protein interactions. Proteomics. 9 (23), 5214-5223 (2009).
- Heide, H., et al. Complexome profiling identifies TMEM126B as a component of the mitochondrial complex I assembly complex. Cell Metab. 16 (4), 538-549 (2012).
- Wessels, H. J., et al. Analysis of 953 human proteins from a mitochondrial HEK293 fraction by complexome profiling. PLoS One. 8 (7), 68340 (2013).
- Wessels, H. J., et al. LC-MS/MS as an alternative for SDS-PAGE in blue native analysis of protein complexes. Proteomics. 9 (17), 4221-4228 (2009).
- Remmerie, N., et al. Unraveling tobacco BY-2 protein complexes with BN PAGE/LC-MS/MS and clustering methods. J Proteomics. 74 (8), 1201-1217 (2011).
- Sessler, N., Krug, K., Nordheim, A., Mordmuller, B., Macek, B. Analysis of the Plasmodium falciparum proteasome using Blue Native PAGE and label-free quantitative mass spectrometry. Amino Acids. 43 (3), 1119-1129 (2012).
- Bode, D., Yu, L., Tate, P., Pardo, M., Choudhary, J. Characterization of Two Distinct Nucleosome Remodeling and Deacetylase (NuRD) Complex Assemblies in Embryonic Stem Cells. Mol Cell Proteomics. 15 (3), 878-891 (2016).
- Cox, J., Mann, M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nat Biotechnol. 26 (12), 1367-1372 (2008).
- Cox, J., et al. A practical guide to the MaxQuant computational platform for SILAC-based quantitative proteomics. Nat Protoc. 4 (5), 698-705 (2009).
- Kim, J. J., et al. A novel regulatory factor recruits the nucleosome remodeling complex to wingless integrated (Wnt) signaling gene promoters in mouse embryonic stem cells. J Biol Chem. 287 (49), 41103-41117 (2012).
- Lauberth, S. M., Rauchman, M. A conserved 12-amino acid motif in Sall1 recruits the nucleosome remodeling and deacetylase corepressor complex. J Biol Chem. 281 (33), 23922-23931 (2006).
- Miller, A., et al. Sall4 controls differentiation of pluripotent cells independently of the Nucleosome Remodelling and Deacetylation (NuRD) complex. Development. 143 (17), 3074-3084 (2016).
- Dharmarajan, V., Lee, J. H., Patel, A., Skalnik, D. G., Cosgrove, M. S. Structural basis for WDR5 interaction (Win) motif recognition in human SET1 family histone methyltransferases. J Biol Chem. 287 (33), 27275-27289 (2012).
- Schwanhausser, B., et al. Global quantification of mammalian gene expression control. Nature. 473 (7347), 337-342 (2011).
- Smits, A. H., Jansen, P. W., Poser, I., Hyman, A. A., Vermeulen, M. Stoichiometry of chromatin-associated protein complexes revealed by label-free quantitative mass spectrometry-based proteomics. Nucleic Acids Res. 41 (1), 28 (2013).
- Lambert, J. P., Tucholska, M., Pawson, T., Gingras, A. C. Incorporating DNA shearing in standard affinity purification allows simultaneous identification of both soluble and chromatin-bound interaction partners. J Proteomics. 100, 55-59 (2014).
- Wittig, I., Braun, H. P., Schagger, H. Blue native PAGE. Nat Protoc. 1 (1), 418-428 (2006).
- Schagger, H., Cramer, W. A., von Jagow, G. Analysis of molecular masses and oligomeric states of protein complexes by blue native electrophoresis and isolation of membrane protein complexes by two-dimensional native electrophoresis. Anal Biochem. 217 (2), 220-230 (1994).
- Schagger, H., von Jagow, G. Blue native electrophoresis for isolation of membrane protein complexes in enzymatically active form. Anal Biochem. 199 (2), 223-231 (1991).
- Camacho-Carvajal, M. M., Wollscheid, B., Aebersold, R., Steimle, V., Schamel, W. W. Two-dimensional Blue native/SDS gel electrophoresis of multi-protein complexes from whole cellular lysates: a proteomics approach. Mol Cell Proteomics. 3 (2), 176-182 (2004).
- Liu, F., Rijkers, D. T., Post, H., Heck, A. J. Proteome-wide profiling of protein assemblies by cross-linking mass spectrometry. Nat Methods. 12 (12), 1179-1184 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten