Transformação de células de E. coli usando um protocolo adaptado de cloreto de cálcio
Visão Geral
Fonte: Natalia Martin1, Andrew J. Van Alst1, Rhiannon M. LeVeque1, e Victor J. DiRita1
1 Departamento de Microbiologia e Genética Molecular, Universidade Estadual de Michigan
As bactérias têm a capacidade de trocar material genético (Ácido Desoxiribonucleico, DNA) em um processo conhecido como transferência de genes horizontais. A incorporação do DNA exógeno fornece um mecanismo pelo qual as bactérias podem adquirir novos traços genéticos que lhes permitem adaptar-se às mudanças nas condições ambientais, como a presença de antibióticos ou anticorpos (1) ou moléculas encontradas em habitats naturais (2). Existem três mecanismos de transferência genética horizontal: transformação, transdução e conjugação (3). Aqui vamos focar na transformação, na capacidade das bactérias de tirar DNA livre do meio ambiente. Em laboratório, o processo de transformação tem quatro etapas gerais: 1) Preparação de células competentes, 2) Incubação de células competentes com DNA, 3) Recuperação de células e 4) Revestimento das células para crescimento dos transformadores (Figura 1).
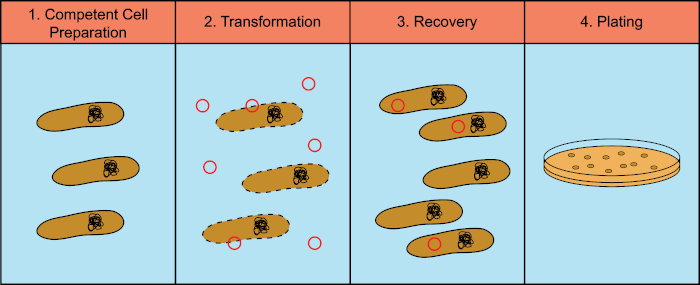
Figura 1: Etapas gerais do processo de transformação. O processo de transformação tem quatro etapas gerais: 1) Preparação de células competentes, 2) Incubação com DNA, 3) Recuperação das células e 4) Células de chapeamento para crescimento dos transformadores.
Para que a transformação ocorra, a bactéria receptora deve estar em um estado conhecido como competência. Algumas bactérias têm a capacidade de se tornarem naturalmente competentes em resposta a certas condições ambientais. No entanto, muitas outras bactérias não se tornam competentes naturalmente, ou as condições para esse processo ainda são desconhecidas. A capacidade de introduzir DNA em bactérias tem uma gama de aplicações de pesquisa: gerar múltiplas cópias de uma molécula de DNA de interesse, expressar grande quantidade de proteínas, como componente em procedimentos de clonagem, entre outros. Devido ao valor da transformação para a biologia molecular, existem vários protocolos que visam tornar as células artificialmente competentes quando as condições de competência natural são desconhecidas. Dois métodos principais são utilizados para preparar células artificialmente competentes: 1) através do tratamento químico das células e 2) expondo as células a pulsos elétricos (eletroporação). O primeiro usa diferentes produtos químicos dependendo do procedimento para criar atração entre o DNA e a superfície celular, enquanto o segundo usa campos elétricos para gerar poros na membrana celular bacteriana através da qual moléculas de DNA podem entrar. A abordagem mais eficiente para a competência química é a incubação com cátions divalent, mais notavelmente cálcio (Ca2+) (4,5) Competência induzida por cálcio é o procedimento que será descrito aqui (6). Este método é usado principalmente para a transformação de bactérias Gram-negativas, e esse será o foco deste protocolo.
O procedimento de transformação química envolve uma série de etapas em que as células são expostas a ceações para induzir a competência química. Essas etapas são posteriormente seguidas por uma mudança de temperatura - choque térmico - que favorece a absorção de DNA estranho pela célula competente (7). Os envelopes de células bacterianas são carregados negativamente. Em bactérias gram-negativas como escherichia coli,a membrana externa é carregada negativamente devido à presença de lipopólise (LPS) (8). Isso resulta em repulsa das moléculas de DNA igualmente carregadas negativamente. Na indução de competência química, íons de cálcio carregados positivamente neutralizam essa repulsão de carga permitindo a absorção de DNA na superfície celular (9). O tratamento de cálcio e a incubação com DNA são feitos no gelo. Posteriormente, é realizada uma incubação a temperaturas mais altas (42°C), o choque térmico. Esse desequilíbrio de temperatura favorece ainda mais a absorção de DNA. As células bacterianas precisam estar na fase de crescimento exponencial médio para suportar o tratamento de choque térmico; em outros estágios de crescimento, as células bacterianas são muito sensíveis ao calor, resultando em perda de viabilidade, o que diminui significativamente a eficiência da transformação.
Diferentes fontes de DNA podem ser usadas para transformação. Tipicamente, plasmídeos, pequenas moléculas circulares de DNA de dupla laga, são usados para transformação na maioria dos procedimentos laboratoriais em E. coli. Para que os plasmídeos sejam mantidos na célula bacteriana após a transformação, eles precisam conter uma origem da replicação. Isso permite que eles sejam replicados na célula bacteriana independentemente do cromossomo bacteriano. Nem todas as células bacterianas são transformadas durante o procedimento de transformação. Assim, a transformação produz uma mistura de células transformadas e células não transformadas. Para distinguir entre essas duas populações, é utilizado um método de seleção para identificar as células que adquiriram o plasmídeo. Plasmídeos geralmente contêm marcadores selecionáveis, que são genes codificando um traço que confere uma vantagem para o crescimento (ou seja, resistência a um antibiótico ou químico ou resgate de uma auxotrofia de crescimento). Após a transformação, as células bacterianas são banhadas em mídias seletivas, o que só permite o crescimento das células transformadas. No caso de células transformadas com uma resistência plasmid conferenciando a um dado antibiótico, a mídia seletiva será a mídia de crescimento contendo esse antibiótico. Vários métodos diferentes podem ser usados para confirmar que as colônias cultivadas nas mídias seletivas são transformadores (ou seja, incorporaram o plasmídeo). Por exemplo, plasmídeos podem ser recuperados dessas células usando métodos de preparação plasmídeos (10) e digeridos para confirmar o tamanho plasmídeo. Alternativamente, a COLÔNIA PCR pode ser usada para confirmar a presença do plasmídeo de interesse (11).
O objetivo deste experimento é preparar células quimicamente competentes E. coli DH5α, utilizando uma adaptação do procedimento de cloreto de cálcio (12), e transformá-las com o pUC19 plasmídeo para determinar a eficiência da transformação. A cepa E. coli DH5α é uma cepa comumente usada em aplicações de biologia molecular. Devido ao seu genótipo, especificamente o recA1 e o endA1,esta cepa permite maior estabilidade da inserção e melhorar a qualidade do DNA plasmídeo nas preparações subsequentes. Uma vez que a eficiência de transformação diminui com o aumento do tamanho do DNA, o plasmídeo pUC19 foi usado neste protocolo por causa de seu pequeno tamanho (2686 bp) (ver https://www.mobitec.com/cms/products/bio/04_vector_sys/standard_cloning_vectors.html para um mapa vetorial). pUC19 confere resistência à ampicilina e, portanto, este foi o antibiótico utilizado para seleção.
Procedimento
Este protocolo descreve a preparação e transformação do competente E. coli DH5α utilizando uma adaptação do procedimento de cloreto de cálcio (12).
1. Configuração
-
Equipamento
- Espectrofotômetro
- Centrífuga de Sorval (ou equivalente)
- Centrífuga benchtop
- Bloco de calor ou banho de água
- Orbital Shaker
- Incubadora Estacionária
- Bandeja de fundição de gel
- Bem pentes
- Fonte de tensão
- Caixa de gel
- Fonte de luz UV
- Microondas
-
Soluções e reagentes
- Caldo luria-bertani (LB) (10 g de hidrolise enzimática de caixa, extrato de levedura de 5 g e cloreto de sódio de 5 g em 1000 mL de H2O)
- Caldo super ideal com repressão catabólica (SOC): (2% (w/v) triptona, extrato de levedura de 0,5% (w/v), 10 mM NaCl, 2,5 mM KCl, 10 mM MgCl2,10 mM MgSO4e 20 mM de glicose)
- CaCl2-MgCl2 (80 mM MgCl2, 20 mM CaCl2) solução.
- Solução M CaCl2 (se as células serão transformadas imediatamente) ou solução de 0,1M CaCl2 contendo 10% (v/v) glicerol (se as células serão congeladas para uso futuro).
- Placas de ágar LB
- Placas seletivas de ágar LB (para este experimento, uma vez que o plasmid usou resistência à ampicilina, foram utilizadas placas de ágar LB contendo ampicillin 100 μg/mL)
- E. coli Tensão DH5α
- DNA plasmid pUC19 (100 pg/ μl)
- QiAprep Spin Miniprep Kit (Qiagen)
- Hind Enzima de restrição III
- 1 kb mais escada de DNA
- Ponto de fusão baixo Agarose
- Tampão TAE 1X (Base Tris de 40 mM, ácido acético de 20 mM e 1mM EDTA)
- Brometo de ethidium (10mg/mL)
-
Notas gerais de segurança
E. coli DH5α é classificado como Biossegurança Nível 1 (BSL1). Micróbios nesta categoria representam pouca ou nenhuma ameaça de infecção em adultos saudáveis. No entanto, é necessária uma manipulação cuidadosa do microrganismo.
IMPORTANTE todas as etapas deste protocolo precisam ser realizadas utilizando técnicas assépticas e em temperaturas de gelo ou 4°C, a menos que indicado.
2. Protocolo
- A partir de um estoque congelado de E. coli DH5α (congelado em 20% de glicerol de uma cultura durante a noite cultivada em LB) listram bactérias para isolamento em uma placa de ágar LB. Incubar a 37°C durante a noite (16-20 horas).
- Inocular uma única colônia em 3 mL de caldo LB em um tubo. Cresça tremendo a 210 rpm a 37°C durante a noite (16-20 horas).
- Meça o OD600 da cultura da noite para o dia. Use a cultura da noite para inocular 100 mL de caldo LB em um frasco de 1 litro para um OD600 =0,01. Incubar a cultura tremendo vigorosamente (210 rpm) a 37°C monitorando OD600 no espectrofotômetro a cada 15-20 min, até que a cultura atinja OD600=0,35 (aproximadamente 3 horas).
NOTA: Para que a transformação seja eficiente, as células bacterianas precisam estar em fase de crescimento exponencial médio. O número máximo de células precisa ser de 108 células/mL, o que para a maioria das cepas de E. coli corresponde a OD600 =0,4. O uso do espectrômetro permite medir o OD600, que permite determinar que as células estão no estágio de crescimento adequado. Se este protocolo for usado para outras cepas de bactérias, a calibração para determinar o número de colônias formando unidades em valores específicos de OD600 será necessária para determinar essa correlação. - Transfira os 50 mL da cultura para cada uma das 2 garrafas de centrífugas de polipropileno geladas. Coloque as garrafas no gelo por 20 minutos para esfriar.
- Recupere as células por centrifugação a 2700g (4100 rpm em um rotor Sorval GSA) por 10 min a 4°C.
- Remova o supernatante. Escorra os últimos traços da mídia colocando a garrafa de cabeça para baixo em um bloco ou papel toalha.
- Resuspenda cada pelota bacteriana em 30 mL de uma solução gelada CaCl2-MgCl2 (80 mM MgCl2, 20 mM CaCl2) solução gelada. Primeiro adicione 5 mL da solução, gire cuidadosamente até que a pelota tenha dissolvido completamente e, em seguida, adicione os 25 mL restantes da solução.
- Repita o passo 2.4.
- Repita o passo 2.5.
- Se as células competentes forem diretamente transformadas, resuspenncie cada pelota bacteriana em 2 mL de uma solução gelada cacl2 (0,1 M) girando os tubos cuidadosamente. Se a pelota não ficar ressuspendida com este método, resuspend por tubos suavemente para cima e para baixo (evitando a formação de bolhas).
Alternativamente, as células competentes podem ser congeladas e armazenadas para uso posterior. Para preparar estoques congelados de células competentes, resuspense a pelota em 2 ml de uma solução 0.1M CaCl2 contendo 10% (v/v) glicerol. Esta solução precisa ser gelada. Suspensão de células de aliquot em tubos de polipropileno gelado de 1,5 mL (160 μl por tubo). Congele as células competentes imediatamente em um banho seco de gelo/etanol. Transfira os tubos para um congelador de -70°C. - Para transformar as células tratadas com CaCl2,transfira 50 μl de células competentes para cada um dos tubos de polipropileno de 2,5 ml. Adicione os 1 μl (100 pg) de DNA plasmídeo pUC19 a um dos tubos e deixe o segundo tubo sem DNA (controle negativo). Misture suavemente (evite a formação de bolhas). Incubar por 30 minutos no gelo.
NOTA: Não mais de 50 ng de DNA em um volume de 10 μL ou menos deve ser usado na transformação. - Transfira os tubos para o bloco de calor e incubar a 42°C por 45 s exatamente.
NOTA: O choque térmico é um passo crítico. Não exceda a temperatura ou o tempo de incubação. - Transferência prontamente de tubos para gelo. Incubar por 2 min.
- Adicione 950 μL de mídia SOC e incubar os tubos por 1 hora a 37°C para permitir que as bactérias se recuperem e expressem o marcador resistente a antibióticos codificado no plasmídeo.
- Diluir 10 μL da suspensão celular em 1000 μL em SOC (diluição de 1/100) e 100 μL da suspensão celular em 1000 μL em SOC (diluição de 1/10). Placa 100 μl das diluições, bem como o controle, em placas seletivas, e espalhar usando uma espátula. Normalmente, o revestimento de 100 μL de uma diluição de 1/100 e 1/10 produzirá número suficiente de unidades formadoras de colônias (cfu) por placa. Idealmente, esse número deve variar entre 30-300 cfu para que haja colônias suficientes, mas separadas umas das outras. No entanto, o número de cfu dependerá da eficiência de transformação (ver Seção de Análise de Dados e Resultados).
- Incubar as placas a 37°C. Colônias transformadas devem aparecer em 12-16 horas (essa faixa dependerá da cepa celular e método de seleção). Nenhuma colônia deve crescer no controle negativo.
- Conte a cfu/placa obtida para a transformação (Tabela 1).
- Para verificar se serão realizados os transformadores do plasmídeo pUC19, será realizada uma preparação plasmida e posterior digestão. Para isso, inocular uma única colônia em 3 ml de caldo LB em um tubo. Cresça tremendo a 210 rpm a 37°C durante a noite (16-20 horas).
- Prepare uma preparação plasmida usando o Kit Miniprep QIAprep Spin, de acordo com as instruções do fabricante.
- Digerir os 1 μg de pUC19 purificado com a enzima de restrição HindIII a 37°C por 1 hora.
NOTA: Qualquer enzima que corte no local de clonagem múltipla pUC19 pode ser usada para esta etapa.
| Componente | Quantidade |
| Tampão de digestão de restrição de 10X | 2,5 μl |
| Plasmid pUC19 | 1 μg |
| Hind III | 1 μl |
| H2O | 20,5 μl (até 25 μl) |
- Execute uma escada de peso molecular, DNA pUC19 digerido e a mesma quantidade de DNA pUC19 não digerido em um gel de 1% de agarose contendo 1 μg/mL brometo de etídio por 1 hora a 95 V.
NOTA: o tempo e a tensão variam dependendo do equipamento utilizado. - Visualize o gel sob a luz UV. Compare o tamanho do DNA pUC19 digerido e não digerido(Figura 2) (ver Seção de Análise de Dados e Resultados).
Prossiga com as etapas necessárias para verificar a transformação de acordo com o objetivo de cada experimento de transformação em particular.

Figura 2: Digestão do DNA plasmídeo recuperado das células DH5α transformadas. O DNA plasmídeo foi recuperado de células DH5α transformadas, digeridas com HindIII, executadas em um gel de 1% de agarose e visualizadas com uma fonte UV (passos 2,19 a 2,22).
3. Análise e Resultados de Dados
Para calcular a eficiência da transformação, um indicador de quão bem as células ocuparam o DNA extracelular, as colônias obtidas na transformação precisam ser contadas:
| Diluição | Cfu |
| 1/100 | 34 |
| 1/10 | 246 |
Tabela 1: Unidades formadoras de colônias (cfu) contaram a partir de experimento de transformação.
A eficiência de transformação (TE) é uma medida do número de cfu resultante da transformação de 1 μg de plasmídeo em um determinado volume de células competentes. Muitos parâmetros afetam a eficiência de transformação: tamanho plasmídeo, genótipo celular, estágio de crescimento durante a preparação de competências, métodos de transformação, etc.). Ao calcular o TE é importante considerar qual diluição (se houver) foi realizada antes do emplacamento e incorporá-la no cálculo do número total de cfu. A eficiência de transformação (TE) é calculada com a seguinte equação:

Primeiro divida a cfu pelo μg de DNA, neste exemplo 0,0001μg. Em seguida, divida o resultado pelo fator de diluição. Neste exemplo, foi utilizada uma diluição de 1/10 e 100μL de uma solução de 1 ml (diluição: 1/10 × 100 μL /1000 μL=0,01).

Resultados
Embora o TE dependa de muitos fatores, preparações celulares não comerciais e competentes, como esta, normalmente produzem de 10a 107 transformadores por micrograma de plasmídeos. Portanto, esta preparação, com um TE = 2,46 x 108 cfu/μg, rendeu um TE muito além da faixa esperada. Protocolos adicionais estão disponíveis para a fabricação de células supercompetente quando são necessárias maiores eficiências de transformação para uma determinada aplicação (13).
A análise da digestão do DNA plasmídeo recuperado das células transformadas indicou que este plasmídeo tem o tamanho esperado de DNA pUC19 (2686 bp).
Aplicação e Resumo
A transformação é um método poderoso para introduzir DNA exógeno em células bacterianas que é fundamental para muitas aplicações de biologia molecular em laboratório. Além disso, desempenha um papel importante na natureza ao permitir que as células bacterianas troquem material genético que poderia resultar em maior variação genética e permitir a aquisição de diferentes traços benéficos para a sobrevivência sob uma ampla gama de condições. Muitas cepas bacterianas codificam os genes necessários para a competência natural. No entanto, as condições em que esses genes são induzidos ainda são desconhecidas. Mais pesquisas são necessárias para determinar essas condições.
Pular para...
Vídeos desta coleção:

Now Playing
Transformação de células de E. coli usando um protocolo adaptado de cloreto de cálcio
Microbiology
87.4K Visualizações

Criando uma coluna de Winogradsky: um método para enriquecer as espécies microbianas em uma amostra de sedimento
Microbiology
130.5K Visualizações

Diluições em série e plaqueamento: enumeração microbiana
Microbiology
318.2K Visualizações

Culturas de enriquecimento: cultivo de micróbios aeróbicos e anaeróbicos em meios seletivos e diferenciais
Microbiology
132.6K Visualizações

Culturas puras e semeadura por esgotamento: isolamento de colônias bacterianas únicas de uma amostra mista
Microbiology
167.0K Visualizações

Sequenciamento de rRNA 16S: uma técnica baseada em PCR para identificar espécies bacterianas
Microbiology
190.7K Visualizações

Curvas de crescimento: gerando curvas de crescimento usando unidades formadoras de colônias e medições de densidade óptica
Microbiology
299.8K Visualizações

Teste de sucetibilidade a antibióticos: testes de epsilômetro para determinar valores de MIC de dois antibióticos e avaliar a sinergia de antibióticos
Microbiology
94.4K Visualizações

Microscopia e Coloração: Coloração de Gram, Cápsula e Endósporo
Microbiology
364.8K Visualizações

Ensaio de placa: um método para determinar o título viral como unidades formadoras de placa (PFU)
Microbiology
187.1K Visualizações

Conjugação: um método para transferir a resistência à ampicilina da E. coli doadora para a receptora
Microbiology
38.6K Visualizações

Transdução fágica: um método para transferir a resistência à ampicilina da E. coli doadora para a receptora
Microbiology
29.4K Visualizações
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados