Transformation des cellules E. coli en utilisant le chlorure de calcium
Vue d'ensemble
Source: Natalia Martin1, Andrew J. Van Alst1, Rhiannon M. LeVeque1, et Victor J. DiRita1
1 Département de microbiologie et de génétique moléculaire, Michigan State University
Les bactéries ont la capacité d'échanger du matériel génétique (acide déoxyribonucléique, ADN) dans un processus connu sous le nom de transfert horizontal de gènes. L'incorporation d'ADN exogène fournit un mécanisme par lequel les bactéries peuvent acquérir de nouveaux traits génétiques qui leur permettent de s'adapter aux conditions environnementales changeantes, telles que la présence d'antibiotiques ou d'anticorps (1) ou de molécules présentes dans les habitats naturels (2). Il existe trois mécanismes de transfert horizontal de gènes : la transformation, la transduction et la conjugaison (3). Ici, nous allons nous concentrer sur la transformation, la capacité des bactéries à prendre l'ADN libre de l'environnement. En laboratoire, le processus de transformation comporte quatre étapes générales : 1) Préparation de cellules compétentes, 2) Incubation de cellules compétentes avec ADN, 3) Récupération des cellules, et 4) Placage des cellules pour la croissance des transformateurs (figure 1).
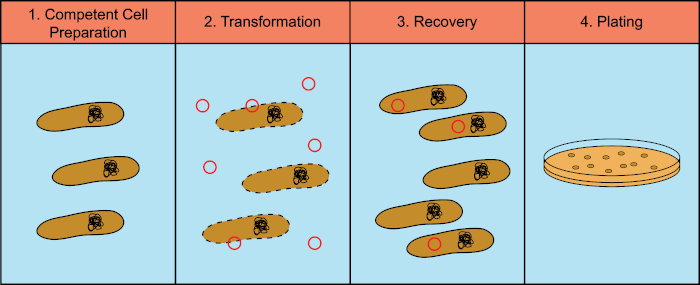
Figure 1 : Étapes générales du processus de transformation. Le processus de transformation comporte quatre étapes générales : 1) Préparation de cellules compétentes, 2) Incubation avec ADN, 3) Récupération des cellules et 4) Cellules de placage pour la croissance des transformateurs.
Pour que la transformation se produise, les bactéries récepteuses doivent être dans un état connu sous le nom de compétence. Certaines bactéries ont la capacité de devenir naturellement compétentes en réponse à certaines conditions environnementales. Cependant, beaucoup d'autres bactéries ne deviennent pas compétentes naturellement, ou les conditions pour ce processus sont encore inconnues. La capacité d'introduire l'ADN dans les bactéries a une gamme d'applications de recherche: pour générer plusieurs copies d'une molécule d'ADN d'intérêt, d'exprimer une grande quantité de protéines, comme un composant dans les procédures de clonage, et d'autres. En raison de la valeur de la transformation à la biologie moléculaire, il existe plusieurs protocoles visant à rendre les cellules artificiellement compétentes lorsque les conditions de compétence naturelle sont inconnues. Deux méthodes principales sont utilisées pour préparer des cellules artificiellement compétentes : 1) par le traitement chimique des cellules et 2) l'exposition des cellules aux impulsions électriques (électroporation). Le premier utilise différents produits chimiques selon la procédure pour créer une attraction entre l'ADN et la surface cellulaire, tandis que le second utilise des champs électriques pour générer des pores dans la membrane cellulaire bactérienne par laquelle les molécules d'ADN peuvent entrer. L'approche la plus efficace pour la compétence chimique est l'incubation avec des cations divalentes, plus particulièrement le calcium (Ca2)(4,5) La compétence induite par le calcium est la procédure qui sera décrite ici (6). Cette méthode est principalement utilisée pour la transformation des bactéries Gram-négatives, et qui sera au centre de ce protocole.
La procédure de transformation chimique implique une série d'étapes dans lesquelles les cellules sont exposées à des cations pour induire la compétence chimique. Ces étapes sont ensuite suivies d'un changement de température - choc thermique - qui favorise l'apport d'ADN étranger par la cellule compétente (7). Les enveloppes de cellules bactériennes sont chargées négativement. Chez les bactéries Gram-négatives comme Escherichia coli, la membrane externe est chargée négativement en raison de la présence de lipopolysaccharide (LPS) (8). Il en résulte la répulsion des molécules d'ADN de même charge négative. Dans l'induction chimique de compétence, les ions de calcium positivement chargés neutralisent cette répulsion de charge permettant l'absorption d'ADN sur la surface de cellules (9). Le traitement du calcium et l'incubation avec de l'ADN se font sur la glace. Par la suite, une incubation à des températures plus élevées (42 oC), le choc thermique, est effectuée. Ce déséquilibre de température favorise encore plus l'apport d'ADN. Les cellules bactériennes doivent être à la phase de croissance mi-exponentielle pour résister au traitement de choc thermique ; dans d'autres stades de croissance, les cellules bactériennes sont trop sensibles à la chaleur, ce qui entraîne une perte de viabilité qui diminue considérablement l'efficacité de la transformation.
Différentes sources d'ADN peuvent être utilisées pour la transformation. Typiquement, les plasmides, petites molécules circulaires d'ADN à double brin, sont utilisés pour la transformation dans la plupart des procédures de laboratoire dans E. coli. Pour que les plasmides soient maintenus dans la cellule bactérienne après la transformation, ils doivent contenir une origine de réplication. Cela leur permet d'être reproduits dans la cellule bactérienne indépendamment du chromosome bactérien. Toutes les cellules bactériennes ne se transforment pas au cours de la procédure de transformation. Ainsi, la transformation donne un mélange de cellules transformées et de cellules non transformées. Pour distinguer entre ces deux populations, une méthode de sélection pour identifier les cellules qui ont acquis le plasmide est utilisée. Les plasmides contiennent habituellement des marqueurs sélectionnables, qui sont des gènes codant un trait qui confère un avantage pour la croissance (c.-à-d. résistance à un antibiotique ou chimique ou sauvetage d'une auotrophie de croissance). Après la transformation, les cellules bactériennes sont plaquées sur des supports sélectifs, ce qui ne permet que la croissance des cellules transformées. Dans le cas des cellules transformées avec un plasmide conférant une résistance à un antibiotique donné, les médias sélectifs seront des supports de croissance contenant cet antibiotique. Plusieurs méthodes différentes peuvent être utilisées pour confirmer que les colonies cultivées dans les milieuis sélectifs sont des transformateurs (c'est-à-dire ont incorporé le plasmide). Par exemple, les plasmides peuvent être récupérés dans ces cellules à l'aide de méthodes de préparation au plasmide (10) et digérés pour confirmer la taille du plasmide. Alternativement, la colonie PCR peut être utilisée pour confirmer la présence du plasmide d'intérêt (11).
L'objectif de cette expérience est de préparer des cellules chimiquement compétentes E. coli DH5MD, en utilisant une adaptation de la procédure de chlorure de calcium (12), et de les transformer avec le plasmide pUC19 pour déterminer l'efficacité de la transformation. La souche E. coli DH5Estest une souche couramment utilisée dans des applications de biologie moléculaire. En raison de son génotype, en particulier le recA1 et endA1, cette souche permet une stabilité accrue de l'insertion et d'améliorer la qualité de l'ADN plasmide dans les préparations ultérieures. Étant donné que l'efficacité de transformation diminue avec l'augmentation de la taille de l'ADN, le plasmide pUC19 a été utilisé dans ce protocole en raison de sa petite taille (2686 bp) (voir https://www.mobitec.com/cms/products/bio/04_vector_sys/standard_cloning_vectors.html pour un carte vectorielle). pUC19 confère une résistance à l'ampicilline et donc, c'était l'antibiotique utilisé pour la sélection.
Procédure
Ce protocole décrit la préparation et la transformation de e. coli DH5compétent compétent à l'aide d'une adaptation de la procédure de chlorure de calcium (12).
1. Mise en place
-
équipement
- Spectrophotomètre
- Sorval Centrifuge (ou équivalent)
- Centrifugeuse benchtop
- Bloc de chaleur ou bain d'eau
- Shaker orbital
- Incubateur stationnaire
- Plateau de coulée de gel
- Peignes de puits
- Source de tension
- Boîte de gel
- Source de lumière UV
- micro-ondes
-
Solutions et réactifs
- Bouillon Luria-Bertani (LB) (10 g d'hydrolysate enzymique de caséine, 5 g d'extrait de levure et 5 g de chlorure de sodium en 1000 ml de H2O)
- Super Optimal bouillon avec répression Catabolite (SOC): (2% (w/v) tryptone, 0.5% (w/v) extrait de levure, 10 mM NaCl, 2.5 mM KCl, 10 mM MgCl2, 10 mM MgSO4, et 20 mM de glucose)
- CaCl2-MgCl2 (80 mM MgCl2, 20 mM CaCl2) solution.
- M CaCl2 solution (si les cellules seront transformées immédiatement) ou 0,1 M CaCl2 solution contenant 10% (v / v) de glycérol (si les cellules seront congelées pour une utilisation future).
- Plaques d'agar LB
- Plaques sélectives lb agar (pour cette expérience, puisque le plasmide utilisé confère une résistance à l'ampicilline, des plaques d'agar LB contenant de l'ampicilline 100 g/mL ont été utilisées)
- Souche E. coli DH5MD
- Plasmid pUC19 ADN (100 pg/ l)
- Kit miniprep QIAprep Spin (Qiagen)
- Hind Hind ENZYME de restriction III
- 1 kb plus échelle d'ADN
- Point de fusion basse Agarose
- 1X tampon TAE (40 mM Base Tris, 20 mM d'acide acétique et 1 mm EDTA)
- Bromure d'ethidium (10mg/mL)
-
Notes générales de sécurité
E. coli DH5MD est classé niveau de biosécurité 1 (BSL1). Les microbes de cette catégorie présentent peu ou pas de menace d'infection chez les adultes en bonne santé. Cependant, une manipulation soigneuse du micro-organisme est nécessaire.
IMPORTANT toutes les étapes de ce protocole doivent être effectuées à l'aide de techniques aseptiques et sur la glace ou des températures de 4 oC, sauf indication contraire.
2. Protocole
- À partir d'un stock congelé d'E. coli DH5MD (congelé à 20 % de glycérol à partir d'une culture de nuit cultivée en LB), les bactéries s'écriapour l'isolement sur une plaque d'agar LB. Incuber à 37oC pendant la nuit (16-20 heures).
- Inoculer une seule colonie en 3 ml de bouillon LB dans un tube. Croissance en secousses à 210 tr/min à 37 oC pendant la nuit (16-20 heures).
- Mesurer l'OD600 de la culture du jour au lendemain. Utilisez la culture de la nuit pour inoculer 100 ml de bouillon LB dans un flacon de 1 litre à un OD600-0,01. Incuber la culture en secouant vigoureusement (210 tr/min) à 37 oC de surveillance OD600 dans le spectrophotomètre toutes les 15-20 min, jusqu'à ce que la culture atteigne OD600-0,35 (environ 3 heures).
REMARQUE : Pour que la transformation soit efficace, les cellules bactériennes doivent être en phase de croissance à mi-exponentielle. Le nombre maximal de cellules doit être de 108 cellules/mL, ce qui correspond pour la plupart des souches d'E. coli à OD600-0,4. L'utilisation du spectrophotomètre permet de mesurer l'OD600, ce qui permet de déterminer que les cellules sont au stade de croissance approprié. Si ce protocole sera utilisé pour d'autres souches de bactéries, l'étalonnage pour déterminer le nombre de colonies formant des unités à des valeurs OD600 spécifiques sera nécessaire pour déterminer cette corrélation. - Transférer les 50 ml de la culture à chacune des 2 bouteilles de centrifugeuse en polypropylène glacée. Placer les bouteilles sur la glace pendant 20 min pour refroidir.
- Récupérer les cellules par centrifugation à 2700g (4100 tr/min dans un rotor Sorval GSA) pendant 10 min à 4oC.
- Retirez le supernatant. Égoutter les dernières traces de supports en plaçant la bouteille à l'envers sur un tampon ou un essuie-tout.
- Resuspendre chaque granule bactérien en 30 ml d'une solution caCl2-MgCl2 (80 mM MgCl 2 , 20 mM CaCl2) solution glacée. D'abord ajouter 5 ml de la solution, tourbillonner soigneusement jusqu'à ce que le granule soit complètement dissous, puis ajouter les 25 ml de solution restantes.
- Répétez l'étape 2.4.
- Répétez l'étape 2.5.
- Si des cellules compétentes doivent être transformées directement, resuspendre chaque granule bactérienne en 2 ml d'une solution de glace CaCl2 (0,1 M) en faisant tourbillonner soigneusement les tubes. Si le granule n'est pas suspendu avec cette méthode, resuspendre en pipetting doucement de haut en bas (en évitant la formation de bulles).
Alternativement, les cellules compétentes peuvent être congelées et stockées pour une utilisation ultérieure. Pour préparer les stocks congelés de cellules compétentes, suspendre la pastille dans 2 ml d'une solution De 0,1 M CaCl2 contenant 10 % (v/v) de glycérol. Cette solution doit être glacée. Suspension des cellules D'Aliquot en tubes de polypropylène de 1,5 ml de glace (160 l par tube). Congelez immédiatement les cellules compétentes dans un bain de glace sèche/éthanol. Transférer les tubes dans un congélateur à -70 oC. - Pour transformer les cellules traitées CaCl2,transférez 50 'l de cellules compétentes à chacune des 2 tubes de polypropylène de 1,5 ml. Ajouter l'ADN plasmide de 1 x l (100 pg) à l'un des tubes et laisser le deuxième tube sans ADN (contrôle négatif). Mélanger délicatement (éviter la formation de bulles). Incuber 30 min sur glace.
REMARQUE : Pas plus de 50 ng d'ADN dans un volume de 10 L ou moins doivent être utilisés dans la transformation. - Transférer les tubes dans le bloc de chaleur et couver à 42oC pendant 45 s exactement.
REMARQUE : Le choc thermique est une étape critique. Ne dépassez pas la température ou le temps d'incubation. - Transférer facilement les tubes sur la glace. Incuber pendant 2 min.
- Ajouter 950 l de milieux SOC et couver les tubes pendant 1 heure à 37oC pour permettre aux bactéries de récupérer et exprimer le marqueur résistant aux antibiotiques encodé dans le plasmide.
- Diluer 10 l de la suspension cellulaire en 1000 l en SOC (1/100 dilution) et 100 oL de la suspension cellulaire en 1000 l l en SOC (1/10 dilution). Plaque 100 'l des dilutions, ainsi que le contrôle, sur les plaques sélectives, et répartis à l'aide d'une spatule. Habituellement, le placage de 100 l d'une dilution de 1/100 et 1/10 donnera un nombre suffisant d'unités de formation de colonies (cfu) par plaque. Idéalement, ce nombre devrait varier entre 30-300 cfu de sorte qu'il y ait assez de colonies mais séparées les unes des autres. Toutefois, le nombre de cfu dépendra de l'efficacité de la transformation (voir la Section analyse des données et résultats).
- Incuber les plaques à 37oC. Les colonies transformées devraient apparaître en 12-16 heures (cette plage dépendra de la souche cellulaire et de la méthode de sélection). Aucune colonie ne devrait se développer dans le contrôle négatif.
- Compter le cfu/plaque obtenu pour la transformation (Tableau 1).
- Pour vérifier les transformateurs hébergent le plasmide pUC19, une préparation plasmide et une digestion ultérieure seront effectuées. À cette fin, inoculer une seule colonie en 3 ml de bouillon LB dans un tube. Croissance en secousses à 210 tr/min à 37 oC pendant la nuit (16-20 heures).
- Préparer une préparation plasmide à l'aide de la trousse QIAprep Spin Miniprep, selon les instructions du fabricant.
- Diger le 1 g de pUC19 purifié avec l'enzyme de restriction HindIII à 37 oC pendant 1 heure.
REMARQUE: Toute enzyme qui coupe dans le site de clonage multiple pUC19 peut être utilisé pour cette étape.
| composant | quantité |
| 10X Tampon de digestion de restriction | 2,5 l |
| Plasmid pUC19 | 1 g |
| Hind Hind Ⅲ | 1 l |
| H2O | 20,5 l (à 25 l) |
- Exécutez une échelle de poids moléculaire, de l'ADN pUC19 digéré et la même quantité d'ADN pUC19 non digéré dans un gel d'agarose de 1 % contenant 1 bromure d'éthidium de g/mL pendant 1 heure à 95 V.
REMARQUE : le temps et la tension varient selon l'équipement utilisé. - Visualisez le gel sous la lumière UV. Comparez la taille de l'ADN pUC19 digéré et non digéré (figure 2) (voir la Section analyse des données et résultats).
Procédez aux étapes nécessaires pour vérifier la transformation en fonction de l'objectif de chaque expérience de transformation particulière.

Figure 2 : Digestion de l'ADN plasmide récupéré à partir de cellules DH5 Transformées. L'ADN de Plasmid a été récupéré à partir de cellules DH5 transformées, digérées avec HindIII, exécutés dans un gel d'agarose de 1% et visualisés avec une source UV (étapes 2.19 à 2.22).
3. Analyse des données et résultats
Pour calculer l'efficacité de transformation, un indicateur de la façon dont les cellules ont pris l'ADN extracellulaire, les colonies obtenues dans la transformation doivent être comptés:
| dilution | Ufc |
| 1/100 | 34 |
| 1/10 | 246 |
Tableau 1 : Unités de formation de colonies (cfu) comptées à partir de l'expérience de transformation.
L'efficacité de transformation (TE) est une mesure du nombre de cfu résultant de la transformation de 1 g de plasmide en un volume donné de cellules compétentes. De nombreux paramètres affectent l'efficacité de la transformation : taille plasmique, génotype cellulaire, stade de croissance lors de la préparation des compétences, méthodes de transformation, etc.). Lors du calcul de l'TE est important de considérer quelle dilution (le cas échéant) a été effectuée avant le placage et de l'intégrer dans le calcul du nombre total de cfu. L'efficacité de transformation (TE) est calculée avec l'équation suivante :

D'abord diviser le cfu par le g d'ADN, dans cet exemple 0,0001g. Divisez ensuite le résultat par le facteur de dilution. Dans cet exemple, une dilution de 1/10 a été utilisée et 100 'L d'une solution de 1 ml a été plaquée (dilution: 1/10 '100 'L /1000 'L '0.01).

Résultats
Bien que l'TE dépende de nombreux facteurs, les préparations cellulaires non commerciales et compétentes, comme celle-ci, donnent normalement de 106 à 107 transformants par microgramme de plasmide. Par conséquent, cette préparation, avec un TE de 2,46 x 108 cfu/g, a donné un TE bien au-delà de la plage prévue. Des protocoles supplémentaires sont disponibles pour fabriquer des cellules supercompétentes lorsque des efficacités de transformation plus élevées sont nécessaires pour une application donnée (13).
L'analyse de la digestion de l'ADN plasmide récupéré dans les cellules transformées a indiqué que ce plasmide a la taille prévue de l'ADN pUC19 (2686 bp).
Applications et Résumé
La transformation est une méthode puissante pour introduire l'ADN exogène dans les cellules bactériennes qui est la clé de nombreuses applications de biologie moléculaire en laboratoire. En outre, il joue un rôle majeur dans la nature en permettant aux cellules bactériennes d'échanger du matériel génétique qui pourrait entraîner une variation génétique accrue et permettre l'acquisition de différents traits bénéfiques pour la survie dans un large éventail de conditions. De nombreuses souches bactériennes codent les gènes nécessaires à la compétence naturelle. Cependant, les conditions dans lesquelles ces gènes sont induits sont encore inconnues. D'autres recherches sont nécessaires pour déterminer ces conditions.
References
- Croucher, N. J. et al. Rapid pneumococcal evolution in response to clinical interventions. Science. 331 (6016):430-434. (2011)
- Borgeaud, S. et al. The type VI secretion system of Vibrio cholerae fosters horizontal gene transfer. Science. 347(6217):63-67. (2015)
- Burmeister, A. R. Horizontal Gene Transfer. Evol Med Public Health. 2015 (1):193-194. (2015)
- Weston A, Brown MG, Perkins HR, Saunders JR, Humphreys GO. Transformation of Escherichia coli with plasmid deoxyribonucleic acid: calcium-induced binding of deoxyribonucleic acid to whole cells and to isolated membrane fractions. J Bacteriol. 145 (2):780-7. (1981)
- Dagert M, Ehrlich SD. Prolonged incubation in calcium chloride improves the competence of Escherichia coli cells. Gene. 6 (1):23-8. (1979)
- Asif A, Mohsin H, Tanvir R, and Rehman Y. Revisiting the Mechanisms Involved in Calcium Chloride Induced Bacterial Transformation. Front Microbiol. 8:2169. (2017)
- Panja S, Aich P, Jana B, Basu T. How does plasmid DNA penetrate cell membranes in artificial transformation process of Escherichia coli? Mol Membr Biol. 25 (5):411-22. (2008)
- Silhavy, TJ, Kahne D, Walker S. The Bacterial Cell Envelope. Cold Spring Harb Perspect Biol. 2 (5): a000414. (2010)
- Panja S, Aich P, Jana B, Basu T. (2008) Plasmid DNA binds to the core oligosaccharide domain of LPS molecules of E. coli cell surface in the CaCl2-mediated transformation process. Biomacromolecules. 9 (9):2501-9.
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Plasmid Purification. JoVE, Cambridge, MA. (2018)
- Bergkessel M and Guthrie C. Colony PCR. Methods in Enzymology. 529: 299-309. (2013)
- Sambrook J and Russell DW. Molecular Cloning A Laboratory Manual. Cold Spring Harbor Laboratory Press, Cold Spring Harbor, NY.Protocol 25 (1.116-118). (2001)
- Wirth R, Friesenegger A, Fiedler S. Transformation of various species of gram-negative bacteria belonging to 11 different genera by electroporation. Molecular & General Genetics. 216 (1): 175-7. (1989)
Tags
Passer à...
Vidéos de cette collection:

Now Playing
Transformation des cellules E. coli en utilisant le chlorure de calcium
Microbiology
86.2K Vues

Création d'une colonne de Winogradsky : une méthode pour enrichir les espèces microbiennes dans un échantillon de sédiments.
Microbiology
128.0K Vues

Dilutions en série et ensemencement des géloses : numération des micro-organismes
Microbiology
313.5K Vues

Cultures enrichies : Cultiver des micro-organismes aérobies et anaérobies dans des milieux sélectifs et différentiels
Microbiology
131.6K Vues

Cultures pures et ensemencement des géloses : isolement des colonies bactériennes pures à partir d'un échantillon mixte
Microbiology
165.5K Vues

Séquençage de l'ARNr 16S : une technique basée sur la PCR pour l'identification d'espèces bactériennes
Microbiology
187.7K Vues

Courbes de croissance : Générer des courbes de croissance en comptant les unités formant colonies (UFC) et en mesurant l'absorbance
Microbiology
292.3K Vues

Tests de sensibilité aux antibiotiques : Utilisation du ETEST pour déterminer la CMI de deux antibiotiques et évaluer la synergie des antibiotiques
Microbiology
93.4K Vues

Microscopie et coloration : Gram, Capsule et endospores.
Microbiology
362.3K Vues

Test de la plaque : méthode de détermination de la charge virale exprimée en unités formant des plaques
Microbiology
185.5K Vues

Conjugaison : méthode de transfert de la résistance à l'ampicilline du donneur à l'hôte E. coli
Microbiology
38.0K Vues

Transduction via bactériophage : méthode de transfert de la résistance à l'ampicilline du donneur au receveur E. coli
Microbiology
28.9K Vues