Method Article
Caracterização de Vesículas Extracelulares a partir de Fluidos Biológicos
Neste Artigo
Resumo
Aqui, demonstramos o método de caracterização de vesículas extracelulares (EVs) coletadas de fluidos biológicos, como lágrimas e saliva, de seres humanos. O scanner usado neste método é capaz de detectar o fenótipo, tamanho e contagem total de partículas de EVs a partir de 1 μL da amostra.
Resumo
As vesículas extracelulares (EVs) são estruturas produzidas a partir de células e participam da comunicação intercelular transportando biomoléculas de uma célula para outra. Foi demonstrado que os EVs viajam distâncias curtas e longas no corpo e são específicos do tecido. Os EVs não são encontrados apenas nos tecidos, mas também podem ser encontrados em praticamente todos os fluidos corporais, como lágrimas, saliva, líquido cefalorraquidiano, sangue, etc. Embora os EVs possam ser coletados de forma não invasiva de lágrimas e saliva, apenas pequenos volumes podem ser coletados por vez, o que pode causar problemas na obtenção de EVs suficientes para analisar proteínas. O scanner discutido neste artigo é um analisador de nanopartículas que fornece uma solução para esse problema, permitindo-nos caracterizar e estudar o fenótipo, tamanho e contagem total de partículas de EVs a partir de apenas 1 μL de fluido biológico. Este protocolo expandirá o conhecimento das VE a partir de pequenos volumes de amostras difíceis de extrair dos pacientes. Isso poderia aumentar o conforto do paciente e potencialmente identificar novos alvos terapêuticos para uma série de doenças e distúrbios.
Introdução
As células se comunicam com as células vizinhas por meio de vários mecanismos de sinalização, incluindo a liberação de vesículas extracelulares (EVs), que desempenham um papel fundamental na comunicação intercelular. Os EVs participam da comunicação intercelular passando carga genética, como DNA, RNA e proteínas, de uma célula para outra 1,2,3,4,5. Atualmente, existem três categorias de EVs: exossomos, microvesículas e corpos apoptóticos, que são caracterizados por seu tamanho. Os exossomos são os menores, com diâmetro de 30-150 nm 6,7,8, e são formados a partir do sistema de membrana endossômica 9,10,11 (Figura 1). As microvesículas são maiores que os exossomos, pois variam de 100 a 1000 nm 12,13,14 e brotam da membrana plasmática 11,12,13 (Figura 1). Os corpos apoptóticos são os maiores dos EVs e variam de 1000-5000 nm 12,14,15, e também brotam da membrana plasmática 12,16,17 (Figura 1). Além do tamanho, as EVs podem ser classificadas com base em outras características biofísicas, que incluem densidade, marcadores moleculares como CD63, CD81, CD9 e também mecanismo de biogênese18. Os EVs podem percorrer distâncias curtas entre células adjacentes e longas distâncias por todo o corpo 19,20,21,22,23. As EVs podem ser encontradas em fluidos biológicos, como sangue 24,25,26, líquido cefalorraquidiano 27,28,29, lágrimas 30,31,32 e saliva 33,34,35, para citar alguns.
Até o momento, a ultracentrifugação é um dos métodos mais conhecidos para isolar EVs de amostras 36,37,38. Este método requer várias rodadas de centrifugações e ultracentrifugação que podem ser realizadas aumentando a velocidade para isolar os EVs das células e detritos celulares. Este método pode ser iniciado com a baixa velocidade com células de pellets que serão seguidas de velocidade média para eliminar vesículas maiores e, finalmente, uma etapa de ultracentrifugação para pellet EVs18. Embora a ultracentrifugação seja considerada o melhor método para isolamento, ainda existem algumas limitações, pois altera a morfologia das VEs 39,40,41. Outra técnica usada para isolar EVs é a citometria de fluxo, que destaca as vantagens da avaliação de vários pontos de tempo e endpoint e da análise de EV único de alto rendimento. No entanto, as limitações da citometria de fluxo incluem, mas não estão limitadas a, entupimento dos poros e sinais fracos. Outra abordagem utilizada é a centrifugação gradiente, que utiliza materiais com diferentes densidades para serem centrifugados com os EVs e permite uma melhor separação dos EVs em comparação com a ultracentrifugação. Embora essa técnica melhore a separação, ela é trabalhosa, demorada e pode levar a uma perda significativa de amostra. Além disso, precipitação e filtração também podem ser usadas para isolar EVs. Ambas as técnicas são simples e rápidas, mas ambas podem levar à contaminação da amostra. Embora existam várias técnicas para isolar EVs, cada técnica tem suas vantagens e limitações, que estão listadas abaixo (Tabela 1) 42,43,44,45,46,47,48,49,50,51,52,53,54,55, 56,57,58,59,60,61,62,63,64,65,66,67.
Uma vez que os EVs tenham sido isolados, ensaios moleculares podem ser realizados para caracterizar os EVs. Western blots são um ensaio comum para procurar a expressão de proteínas de superfície e carga 68,69,70 e a reação em cadeia da polimerase (PCR) é usada para expressão de miRNA 71,72,73 para EVs 74,75,76. Esses ensaios são estabelecidos e podem gerar resultados intrigantes. Uma limitação desses métodos é que eles requerem uma grande quantidade de proteínas ou RNA de EVs para obter uma leiturade 77, o que é um problema para amostras que têm um pequeno volume ou concentração de EV, para começar.
O analisador de nanopartículas discutido neste artigo permite que o usuário supere muitas das limitações declaradas na Tabela 1 e na Tabela 2 78,79,80,81,82,83,84,85,86,87,88,89,90 . Este método não requer a utilização de técnicas de isolamento, o que ajudará a superar o rendimento reduzido dos EVs. Este método também permite ao usuário analisar as proteínas de superfície e carga, contagem total de EV e tamanho de EV a partir de um volume de amostra de apenas 1 μL. Isso é feito usando chips de tetraspanina fornecidos pela empresa que usa um microarray de anticorpos com anticorpos de tetraspanina, CD63, CD81 e CD9, para identificar EVs em uma solução, conforme mostrado na Figura 2. Os anticorpos fluorescentes confirmam a presença de EVs, além de impedir que partículas contaminantes distorçam os resultados.
O objetivo geral dessa técnica é fornecer um método menos demorado para analisar EVs, bem como analisar EVs de um pequeno volume de amostra. O uso deste analisador de nanopartículas permite que os usuários analisem o tamanho, a contagem total de partículas e as proteínas de superfície de apenas 1 μL de uma amostra, o que é ideal para fluidos biológicos, como lágrimas e saliva.
Protocolo
Todos os estudos descritos aderiram à Declaração de Helsinque. O consentimento por escrito foi obtido de cada sujeito antes de ser incluído no estudo. A aprovação do Conselho de Revisão Institucional (IRB) do Hospital Universitário de Aarhus (1-10-72-77-14) e do Instituto Dean McGee (1576837-2) foi recebida de acordo com as diretrizes federais e institucionais. Antes do processamento, todas as amostras de lágrimas e saliva foram desidentificadas. Todos os estudos foram revisados e aprovados pelo Conselho de Revisão Institucional Regional do Norte do Texas (# 2020-030). O protocolo a seguir segue todas as diretrizes e foi aprovado, conforme mencionado acima.
1. Dia 1: Preparação e incubação da amostra
- Colete amostras de fluido biológico, ou seja, lágrimas e saliva de um sujeito ou descongele amostra(s) do freezer.
- Lágrimas: Colete as lágrimas passivamente do menisco lateral usando um tubo capilar de vidro.
- Coloque o tubo capilar de vidro na pálpebra inferior, tomando cuidado para não tocar na córnea.
- Colete um volume de 10 μL de lágrimas em 10 min.
- Use os rasgos imediatamente ou armazene-os a -20 °C.
- Saliva: Colete a saliva da baba passiva em um tubo de microcentrífuga de 1,5 mL
- Permita que o doador acumule saliva em sua boca.
- Peça ao doador para babar no tubo com um funil.
- Use saliva imediatamente ou armazene a -20 °C.
- Lágrimas: Colete as lágrimas passivamente do menisco lateral usando um tubo capilar de vidro.
- Coloque o(s) chip(s) de tetraspanina na placa do lavador de cavacos por 15 min antes de adicionar samples para permitir que atinjam a temperatura ambiente. Ao colocar os cavacos no poço, certifique-se de que os números estejam na parte inferior voltados para cima e que o(s) cavaco(s) não toque(m) nas paredes laterais do(s) poço(s).
- Registre o número de identificação e o número do chip no notebook para acompanhar o(s) chip(s).
- Conecte o barramento serial universal (USB) que acompanha o kit e extraia e salve a pasta com o número do kit associado em uma área designada de sua escolha.
- Pipetar 99 μL de solução B em um tubo de 1,5 mL.
- Adicione 1 μL de fluido biológico no tubo de 1,5 mL preparado na etapa anterior.
- Gire os tubos.
- Pegue 70 μL da solução da etapa 1.6 e pipete no chip de tetraspanina.
- Coloque o filme na placa de lavagem de cavacos para evitar que a amostra evapore.
- Incubar durante 16 h à temperatura ambiente (RT) numa bancada sem vibrações.
2. Dia 2: Lavagem de cavacos
- Ligue a máquina de lavar cavacos.
- Remova o filme da etapa 1.9 da placa.
- Coloque a placa na arruela de cavacos, certificando-se de que ela trave no lugar, e feche a tampa.
NOTA: A placa não se moverá facilmente se travada corretamente no lugar. - Pressione a opção CW-TETRA v0 na arruela de cavacos.
- Selecione a linha inicial e o número de linhas a serem lavadas.
- Pressione Continuar na máquina de lavar cavacos.
- Enquanto o(s) chip(s) está(ão) a ser lavado(s), preparar a solução de bloqueio contendo os anticorpos num tubo do seguinte modo:
- Prepare 300 μL de solução de bloqueio por chip.
- Adicione 0,6 μL ou cada anticorpo por chip (anticorpos de vórtice e spin down antes de adicionar à solução de bloqueio)
- Vórtice a solução de bloqueio + coquetel de anticorpos.
NOTA: Cubra a solução de bloqueio + coquetel de anticorpos da luz para garantir a estabilidade do anticorpo. Isso deve ser feito no dia do uso.
- Quando o lavador de cavacos soar, adicione 250 μL da solução de bloqueio + coquetel de anticorpos em cima do(s) chip(s).
- Feche a tampa e pressione Continuar.
NOTA: A lavadora de cavacos agora fará uma incubação de 1 h junto com várias rodadas adicionais de lavagem e enxágue. - Quando o lavador de cavacos chiar, remova a placa de lavagem de cavacos e mova o(s) cavaco(s) para cima na rampa.
- Coloque a placa de volta na arruela de cavacos, certificando-se de que ela trave no lugar. Feche a tampa e pressione Continuar.
- Quando a lavadora de cavacos concluir o programa, remova a placa e coloque o(s) chip(s) em uma toalha de papel. Cubra o(s) chip(s) da luz.
3. Dia 2: Digitalizando o(s) chip(s)
- Ligue o scanner e abra o software de digitalização.
NOTA: O scanner passará por autoverificações para indicar se há algum erro ou se a digitalização pode ser continuada. - Selecione Salvar pasta e escolha o local para salvar os arquivos.
- Selecione Pasta ChipFile e escolha a pasta salva na etapa 1.4.
- Selecione a opção suspensa para escolher cada cavaco e localize-o na posição do cavaco no mandril. Repita esta etapa para todos os chips que precisam ser digitalizados.
- Ative a fluorescência clicando nos três quadrados abaixo de Posição do chip. Os quadrados ficarão amarelos, azuis e vermelhos, respectivamente.
- Remova a tampa do mandril pressionando a tampa para baixo e puxe-a.
- Coloque o(s) cavaco(s) no mandril, certificando-se de que estejam firmemente presos.
- Coloque a tampa do mandril de volta colocando os pinos nos orifícios, pressione para baixo e deslize a tampa para travá-la no lugar.
- Coloque o mandril no palco, certificando-se de que ele trave no lugar.
- Selecione Digitalizar fichas.
- Selecione OK.
- Assim que o stage estiver totalmente movido para dentro da máquina, feche a porta.
NOTA: Leva aproximadamente 12 minutos para escanear um único chip. - Assim que o programa terminar de escanear o(s) chip(s), abra a porta para ejetar a verificação de estágio para ver se as varreduras foram bem-sucedidas.
- Se as varreduras forem bem-sucedidas, descarte o(s) chip(s) e coloque o próximo conjunto de chips no mandril ou retorne o mandril vazio ao palco. Em seguida, saia do software e desligue a máquina quando terminar.
- Se as verificações não forem bem-sucedidas, observe o código de erro e corrija-o.
4. Processamento de dados
- Abra o software de análise.
- Selecione Dados de pré-digitalização e escolha a pasta que contém a verificação da etapa 3.2 do dia 2.
- Selecione Avançar.
- Rotule o(s) chip(s) na coluna Nome da Amostra com o nome da amostra clicando na célula e digitando a ID da amostra.
- Em seguida, rotule as tetraspaninas na coluna Nome do canal . Vermelho = CD63, Verde = CD81 e Azul = CD9.
- Selecione Avançar.
- Selecione VC alto no grupo Selecionar ponto para inspecionar a caixa suspensa e desativar os VCs altos para cada chip desativando um ou dois pontos.
- Em seguida, selecione High Count e tente desativar os avisos de alta contagem.
NOTA: Algumas amostras podem ter uma contagem muito alta, mas ainda podem ser analisadas. - Selecione Todos os pontos no menu suspenso, selecione Montagem de pontos e verifique visualmente cada chip em busca de deformidades ou arranhões que possam estar presentes.
- Selecione Avançar.
- No canal CD63, defina o mínimo como 300 para se livrar dos glóbulos vermelhos na linha Avg % Incluída; as células amarelas estão bem e prosseguem para o canal CD81. Se a definição do valor mínimo para 300 não alterar a célula vermelha para amarelo ou branco, prossiga para a etapa 4.14 para solução de problemas.
- No canal CD81, defina o mínimo como 300 para se livrar dos glóbulos vermelhos na linha Avg % Incluído; as células amarelas estão bem e prosseguem para o canal CD9. Se a definição do valor mínimo para 300 não alterar a célula vermelha para amarelo ou branco, prossiga para a etapa 4.14 para solução de problemas.
- No canal CD9, defina o mínimo como 400 para se livrar dos glóbulos vermelhos na linha % Média Incluída; As células amarelas estão bem e prossiga para a etapa 4.15. Se a definição do valor mínimo para 400 não alterar a célula vermelha para amarelo ou branco, prossiga para a etapa 4.14 para solução de problemas.
- Solucionando problemas
- Aumente o valor mínimo em 100 unidades.
- Se a etapa 4.14.1 não funcionar, selecione a ficha na seção Escolher fichas para trabalhar e escolha a ficha. Selecione Contagem de partículas e desative o(s) ponto(s) que tem altas contagens de partículas. Selecione Intensidade/Tamanho, selecione Todos os Chips e veja se isso resolve o problema.
- Se a etapa 4.14.2 não funcionar, selecione a ficha na seção Escolher fichas para trabalhar e escolha a ficha. Selecione Single-Chip Cutoffs e aumente o mínimo em 100 unidades. Em seguida, selecione Todas as fichas para ver se a célula vermelha agora está amarela. Se isso não funcionar, repita essa etapa até que a célula % média incluída fique amarela ou branca.
- Selecione Avançar.
- Desmarque o canal de mensagens instantâneas.
- Defina o valor do mapa de calor no canto inferior direito para incluir todas as partículas e selecione Adicionar plotagem ao relatório.
- Ajuste o número máximo na caixa suspensa e insira manualmente um valor.
- Em seguida, selecione o menu suspenso na parte superior central em Capture Probes e selecione CD63.
- Selecione Adicionar plotagem ao relatório para adicionar a contagem total de partículas ao relatório.
- Selecione o menu suspenso em Modo de análise, escolha Colocalização e Adicionar plotagem ao relatório.
- Em seguida, selecione Tamanho, selecione Alterar visualização para Plotagem e selecione Adicionar Plotagem ao Relatório.
- Por fim, selecione Imagens e selecione Adicionar gráfico ao relatório.
- Repita as etapas 4.18 a 4.22 para CD81 e CD9, certificando-se de desmarcar o canal IM todas as vezes.
- Depois que todos os itens forem adicionados ao relatório, selecione Exportar relatório, verifique se ele é salvo no arquivo correto e selecione Pasta.
NOTA: Arquivos e imagens do Excel foram gerados e agora podem ser usados para análise posterior.
Resultados
Ao analisar as manchas nos chips, procure uma concentração ideal de EVs observando EVs marcados com fluorescência nos canais CD63, CD81 e CD9, e o canal MIgG 30,91 deve permanecer preto, pois este é o canal de controle como visto na Figura 3A. CD81 será reduzido para fluidos biológicos. Se não houver fluorescência nos canais CD63, CD81 e CD9, há ausência de EVs (Figura 3B). Tenha cuidado para não saturar demais o chip (Figura 3C). Isso tornará difícil para o software de análise calcular a precisão do tamanho, contagem total de partículas e fenótipos dos EVs. A partir dos pontos de concentração ótima, o software de análise será capaz de medir o tamanho (50-200 nm), contagem total de partículas e expressão de tetraspanina dos EVs (Figura 4) e será exportado para planilhas individuais para posterior análise. A análise da tetraspanina incluirá a colocalização das proteínas de superfície nas EVs. A colocalização será representada com um "/", por exemplo, "CD63/CD81". Isso não significa que CD63 e CD81 estejam combinados; isso significa que tanto o CD63 quanto o CD81 estão localizados na superfície do EV.
Esses resultados fornecerão informações valiosas entre amostras saudáveis e doentes. Seremos capazes de determinar se amostras saudáveis ou amostras doentes produzem mais EVs, EVs maiores ou menores e o fenótipo dos EVs. Qualquer uma ou todas essas características podem desempenhar um papel na biogênese e/ou progressão da doença. Com esses resultados, poderemos ver se há ausência ou aumento dos níveis de tetraspanina, o que pode fornecer informações sobre os processos celulares e a formação de EVs.
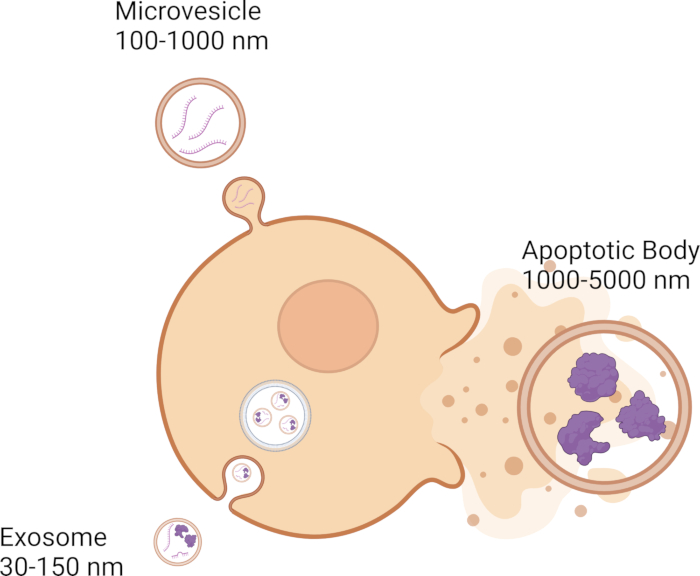
Figura 1: Os tipos e tamanhos dos diferentes EVs. Clique aqui para ver uma versão maior desta figura.

Figura 2: Esquema de como os chips de tetraspanina capturam e detectam EVs. Clique aqui para ver uma versão maior desta figura.
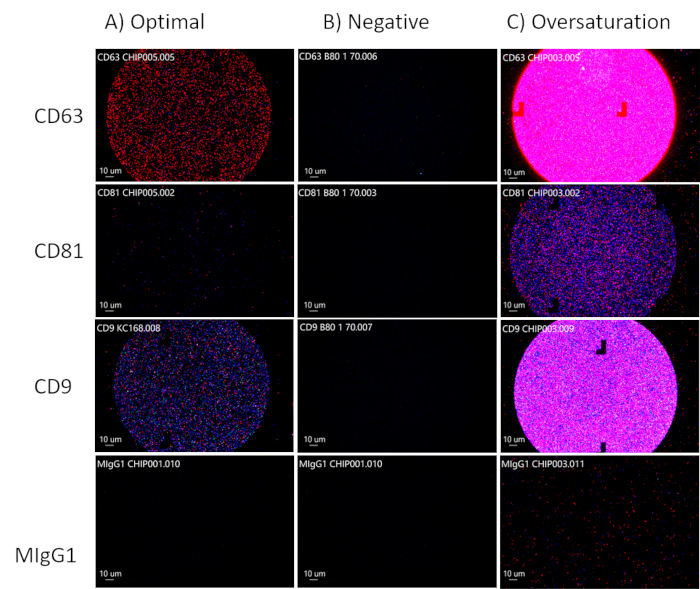
Figura 3: Otimizando as concentrações de EV. (A) Concentração ideal de EVs. (B) Resultado negativo: nenhum EV presente. (C) Supersaturação de EVs. Clique aqui para ver uma versão maior desta figura.

Figura 4: Dados gerados pelo analisador de nanopartículas. (A) Tamanho do diâmetro EV medido em nanômetros. (B) Contagem total de partículas de EVs. (C) Fenótipo co-localizado EV. Clique aqui para ver uma versão maior desta figura.
| Técnica | Vantagens | Limitações |
| Ultracentrífuga | Alta pureza42,43 | Alterações na morfologia EV42,44 |
| Homogeneidade42,45 | Requer grande volume42,44,46 | |
| Funcionalidade42,43 | Caro42,43 | |
| Citometria de fluxo | Avaliar amostras em vários pontos de tempo47 | Sinal fraco48,49 |
| Vários endpoints47,50 | Poros entupidos51 | |
| Análise de EV único de alto rendimento52,53 | ||
| Centrifugação de gradiente de densidade | Produzir amostras altamente puras54 | Trabalho intensivo55,56 |
| Variedade de amostras55,57 | Demorado55,58 | |
| Perda significativa de rendimento54 | ||
| Precipitação | Simples59,60 | Contaminação36,44,61 |
| Rápido61,62 | Não específico63 | |
| Alto rendimento44,59,62 | ||
| pH neutro36 | ||
| Filtração | Simples64 | Armadilha de exossomos65 |
| Rápido64 | Danifica grandes vesículas36 | |
| Barato64 | Torta de filtro66,67 | |
| Entupimento de exossomos65 |
Tabela 1: Vantagens e limitações das técnicas de isolamento de EV. Uma lista das várias maneiras de isolar os VEs, incluindo suas vantagens e limitações.
| Técnica | Vantagens | Limitações |
| Dispersão dinâmica de luz | Meça partículas que variam em tamanho de 1 nm a 6 μM78 | Não é adequado para medir amostras de exossomos complexos com grande faixa de tamanho79 |
| O limite inferior é de 10 nm, adequado para sistemas monodispersos79 | Não é capaz de distinguir proteínas contaminadas de exossomos79 | |
| Microscopia eletrônica de transmissão (MET) | Observar a morfologia dos exossomos79,80 | Preparações complicadas de amostras79 |
| Observe a estrutura interna81 | Não é capaz de distinguir exossomos com base no tamanho e na forma devido a sinais de fluorescência exagerados82 | |
| Análise de rastreamento de nanopartículas | Medir a concentração, o tamanho e a distribuição de tamanho dos exossomos na faixa de 10 nm a 2 μM78 | Não é possível diferenciar os EVs de agregados de proteínas e outros contaminantes83 |
| Preparação e medição rápida de amostras78,84 | Instrumento NTA caro85 | |
| As amostras podem ser recuperadas na forma nativa84 | Sensível a vibrações85 | |
| Western Blot (WBs) | pode analisar qualitativa e quantitativamente proteínas marcadoras79,86 | Complicado e demorado79 |
| Analisando exossomos de meios de cultura de células79 | Os isolados EV podem conter lipoproteínas e outros contaminantes86 | |
| Citometria de fluxo | Maior sensibilidade e imagem de alta resolução para | Demorado e trabalhoso com limite de detecção de 400 nm79,88 |
| distinguir exossomos corados de contenções87 | ||
| Baixa concentração de amostra necessária79 | Sinais ópticos dificultam a precisão e a resolução88 | |
| Exoview | Meça as tetraspaninas (CD9, CD63 e CD81) nos exossomos89 | Não mede tamanhos EV maiores75 |
| Mede proteínas de carga EV90 |
Tabela 2: Vantagens e limitações das técnicas de caracterização de EV.
Discussão
A etapa mais crítica neste protocolo é garantir que uma concentração ideal de EVs seja alcançada. É necessário que haja EVs suficientes presentes para obter uma leitura, mas não muitos EVs que saturem o chip. A melhor maneira de determinar a concentração ideal de EV é fazer uma execução de otimização com 1 μL de amostra e ver se a concentração precisa ser ajustada. Outra etapa crítica é ver se há uma abundância de detritos celulares na amostra, o que pode ser determinado visualizando grandes pedaços nos chips no software de análise. Se a amostra tiver detritos celulares, uma simples centrifugação ou filtragem da amostra deve resolver esse problema.
Uma etapa adicional e crucial é garantir que o(s) chip(s) não toque(m) nas paredes do poço e evitar o contato com a área central do(s) chip(s) ao colocá-los-no na rampa. O scanner lerá a partir do quadrado no centro do chip, por isso é essencial evitar tocar nessa área com a pinça para evitar interromper os EVs.
Este método tem algumas limitações, como ter concentração insuficiente de EVs para detecção pelo instrumento. A concentração pode ser melhorada secando a amostra ou usando um tubo concentrador. Outra limitação é que o software de análise mede apenas EVs na faixa de 50-200 nm, excluindo algumas microvesículas e todos os corpos apoptóticos das medidas de tamanho75.
Existem muitas maneiras de isolar (Tabela 1 42,43,44,45,46,47,48,49,50,51,52, 53,54,55,56,57,58,59,60, 61,62,63,64,65,66,67) e analisar EVs (Tabela 2 78,79,80,81,82,83,84,85, 86,87,88,89,90), e o padrão ouro atual para isolamento EV é a ultracentrifugação 36,37,38 para granular as células para ensaios como Western Blots 68,69,70 e PCR71,72,73,74,75,76. Embora este protocolo funcione bem para amostras grandes 42,44,46, a obtenção de fluidos biológicos pode ser um desafio e, muitas vezes, apenas um pequeno volume de amostras pode ser coletado por vez, o que não é ideal para ultracentrifugação. Em contraste, o uso deste analisador de nanopartículas permite que o usuário gere dados valiosos, como tamanho, contagem total de partículas e fenótipo dos EVs, em apenas 1 μL de amostra, tornando-o ideal para fluidos biológicos. Seremos capazes de expandir o conhecimento das EVs a partir de pequenos volumes de amostras difíceis de extrair, o que pode aumentar o conforto do paciente e possivelmente encontrar potenciais alvos terapêuticos para várias doenças e distúrbios.
Divulgações
Os autores não têm interesses financeiros concorrentes ou outros conflitos de interesse a divulgar.
Agradecimentos
Gostaríamos de agradecer ao NIH pelo financiamento (EY031316 e EY034714). Também gostaríamos de agradecer ao UNTHSC e ao NTERI pelo espaço do laboratório.
Materiais
| Name | Company | Catalog Number | Comments |
| ChipWasher 100 | NanoView | EV-CW100 | Incubates, washes, rinses and dries the tetraspanin chips. This current model is no longer available. Price at time of purchase: $9,995.00 |
| ExoView Analyzer software | NanoView | N/A | Analyzes the chip informations and produces excel files for further analysis. No longer available. |
| ExoView R100 | NanoView | EV-R100 | Used to scan the tetraspanin chips at 3 wavelengths. This current model is no longer available. Price at time of purchase: $110,000.00 |
| ExoView Scanner software | NanoView | N/A | Scans the chips at 3 different wavelengths. No longer available. |
| Human Tetraspanin Kits | Unchained Labs | EV-TETRA-C | Includes 8 tetraspanin chips, Incubation Solution, Blocking Solution, CD63 antibody, CD81 antibody, CD9 antibody, Solution A, Solution B, USB, and plate cover. |
Referências
- Abels, E. R., Breakefield, X. O. Introduction to extracellular vesicles: Biogenesis, RNA cargo selection, content, release, and uptake. Cell Mol Neurobiol. 36 (3), 301-312 (2016).
- Radler, J., Gupta, D., Zickler, A., Andaloussi, S. E. Exploiting the biogenesis of extracellular vesicles for bioengineering and therapeutic cargo loading. Mol Ther. 31 (5), 1231-1250 (2023).
- Gupta, D., Zickler, A. M., El Andaloussi, S. Dosing extracellular vesicles. Adv Drug Deliv Rev. 178, 113961 (2021).
- Dixson, A. C., Dawson, T. R., Di Vizio, D., Weaver, A. M. Context-specific regulation of extracellular vesicle biogenesis and cargo selection. Nat Rev Mol Cell Biol. 24 (7), 454-476 (2023).
- Keshtkar, S., Azarpira, N., Ghahremani, M. H. Mesenchymal stem cell-derived extracellular vesicles: novel frontiers in regenerative medicine. Stem Cell Res Ther. 9 (1), 63 (2018).
- Zhang, H., et al. Exosome-induced regulation in inflammatory bowel disease. Front Immunol. 10, 1464 (2019).
- Barzin, M., et al. Application of plant-derived exosome-like nanoparticles in drug delivery. Pharm Dev Technol. 28 (5), 383-402 (2023).
- Zhou, C., et al. Stem cell-derived exosomes: emerging therapeutic opportunities for wound healing. Stem Cell Res Ther. 14 (1), 107 (2023).
- Kalluri, R., LeBleu, V. S. The biology, function, and biomedical applications of exosomes. Science. 367 (6478), aau6977 (2020).
- Mondal, J., et al. Hybrid exosomes, exosome-like nanovesicles and engineered exosomes for therapeutic applications. J Control Release. 353, 1127-1149 (2023).
- van Niel, G., D'Angelo, G., Raposo, G. Shedding light on the cell biology of extracellular vesicles. Nat Rev Mol Cell Biol. 19 (4), 213-228 (2018).
- Paul, N., Sultana, Z., Fisher, J. J., Maiti, K., Smith, R. Extracellular vesicles- crucial players in human pregnancy. Placenta. 140, 30-38 (2023).
- Zaldivia, M. T. K., McFadyen, J. D., Lim, B., Wang, X., Peter, K. Platelet-derived microvesicles in cardiovascular diseases. Front Cardiovasc Med. 4, 74 (2017).
- Tamasi, V., Nemeth, K., Csala, M. Role of extracellular vesicles in liver diseases. Life (Basel). 13 (5), 1117 (2023).
- Fu, Y., et al. Emerging understanding of apoptosis in mediating mesenchymal stem cell therapy. Cell Death Dis. 12 (6), 596 (2021).
- Santavanond, J. P., Rutter, S. F., Atkin-Smith, G. K., Poon, I. K. H. Apoptotic bodies: Mechanism of formation, isolation and functional relevance. Subcell Biochem. 97, 61-88 (2021).
- Yu, L., et al. Apoptotic bodies: bioactive treasure left behind by the dying cells with robust diagnostic and therapeutic application potentials. J Nanobiotechnology. 21 (1), 218 (2023).
- Momen-Heravi, F., Getting, S. J., Moschos, S. A. Extracellular vesicles and their nucleic acids for biomarker discovery. Pharmacol Ther. 192, 170-187 (2018).
- Liu, Y. J., Wang, C. A review of the regulatory mechanisms of extracellular vesicles-mediated intercellular communication. Cell Commun Signal. 21 (1), 77 (2023).
- Grange, C., Bussolati, B. Extracellular vesicles in kidney disease. Nat Rev Nephrol. 18 (8), 499-513 (2022).
- Cocucci, E., Meldolesi, J. Ectosomes and exosomes: shedding the confusion between extracellular vesicles. Trends Cell Biol. 25 (6), 364-372 (2015).
- Iannotta, D. A. A., Kijas, A. W., Rowan, A. E., Wolfram, J. Entry and exit of extracellular vesicles to and from the blood circulation. Nat Nanotechnol. 19 (1), 13-20 (2024).
- Saint-Pol, J., Gosselet, F., Duban-Deweer, S., Pottiez, G., Karamanos, Y. Targeting and crossing the blood-brain barrier with extracellular vesicles. Cells. 9 (4), 851 (2020).
- Alberro, A., Iparraguirre, L., Fernandes, A., Otaegui, D. Extracellular vesicles in blood: Sources, effects, and applications. Int J Mol Sci. 22 (15), 8163 (2021).
- Nieuwland, R., Siljander, P. R. A beginner's guide to study extracellular vesicles in human blood plasma and serum. J Extracell Vesicles. 13 (1), e12400 (2024).
- Thangaraju, K., Neerukonda, S. N., Katneni, U., Buehler, P. W. Extracellular vesicles from red blood cells and their evolving roles in health, coagulopathy and therapy. Int J Mol Sci. 22 (1), 153 (2020).
- Li, C., et al. Cerebrospinal fluid-derived extracellular vesicles after spinal cord injury promote vascular regeneration via PI3K/AKT signaling pathway. J Orthop Translat. 39, 124-134 (2023).
- Deng, Y., et al. Phosphoproteome analysis of cerebrospinal fluid extracellular vesicles in primary central nervous system lymphoma. Analyst. 148 (15), 3594-3602 (2023).
- Hirschberg, Y., et al. Proteomic comparison between non-purified cerebrospinal fluid and cerebrospinal fluid-derived extracellular vesicles from patients with Alzheimer's, Parkinson's and Lewy body dementia. J Extracell Vesicles. 12 (12), e12383 (2023).
- Hefley, B. S., et al. Revealing the presence of tear extracellular vesicles in Keratoconus. Exp Eye Res. 224, 109242 (2022).
- Cross, T., et al. RNA profiles of tear fluid extracellular vesicles in patients with dry eye-related symptoms. Int J Mol Sci. 24 (20), 15390 (2023).
- Ma, H., et al. Metabolic signatures of tear extracellular vesicles caused by herpes simplex keratitis. Ocul Surf. 31, 21-30 (2024).
- Han, P., Bartold, P. M., Ivanovski, S. The emerging role of small extracellular vesicles in saliva and gingival crevicular fluid as diagnostics for periodontitis. J Periodontal Res. 57 (1), 219-231 (2022).
- Han, P., Li, X., Wei, W., Ivanovski, S. Saliva diagnosis using small extracellular vesicles and salivaomics. Methods Mol Biol. 2588, 25-39 (2023).
- Reseco, L., et al. Characterization of extracellular vesicles from human saliva: Effects of age and isolation techniques. Cells. 13 (1), 95 (2024).
- Tiwari, S., Kumar, V., Randhawa, S., Verma, S. K. Preparation and characterization of extracellular vesicles. Am J Reprod Immunol. 85 (2), e13367 (2021).
- Tomiyama, E., Fujita, K., Nonomura, N. Urinary extracellular vesicles: Ultracentrifugation method. Methods Mol Biol. 2292, 173-181 (2021).
- Xu, K., Jin, Y., Li, Y., Huang, Y., Zhao, R. Recent progress of exosome isolation and peptide recognition-guided strategies for exosome research. Front Chem. 10, 844124 (2022).
- Brennan, K., et al. A comparison of methods for the isolation and separation of extracellular vesicles from protein and lipid particles in human serum. Sci Rep. 10 (1), 1039 (2020).
- Gao, J., et al. Recent developments in isolating methods for exosomes. Front Bioeng Biotechnol. 10, 1100892 (2022).
- Mol, E. A., Goumans, M. J., Doevendans, P. A., Sluijter, J. P. G., Vader, P. Higher functionality of extracellular vesicles isolated using size-exclusion chromatography compared to ultracentrifugation. Nanomedicine. 13 (6), 2061-2065 (2017).
- Ansari, F. J., et al. Comparison of the efficiency of ultrafiltration, precipitation, and ultracentrifugation methods for exosome isolation. Biochem Biophys Rep. 38, 101668 (2024).
- Patel, G. K., et al. Comparative analysis of exosome isolation methods using culture supernatant for optimum yield, purity and downstream applications. Sci Rep. 9 (1), 5335 (2019).
- Stam, J., Bartel, S., Bischoff, R., Wolters, J. C. Isolation of extracellular vesicles with combined enrichment methods. J Chromatogr B Analyt Technol Biomed Life Sci. 1169, 122604 (2021).
- Le Roy, A., et al. AUC and small-angle scattering for membrane proteins. Methods Enzymol. 562, 257-286 (2015).
- Chhoy, P., Brown, C. W., Amante, J. J., Mercurio, A. M. Protocol for the separation of extracellular vesicles by ultracentrifugation from in vitro cell culture models. STAR Protoc. 2 (1), 100303 (2021).
- McDorman, K. S., Chan, C., Rojko, J., Satterwhite, C. M., Morrison, J. P. . Chapter 7 - Special Techniques in Toxicologic Pathology. Haschek and Rousseaux's Handbook of Toxicologic Pathology (Third Edition). , (2013).
- Woud, W. W., et al. An imaging flow cytometry-based methodology for the analysis of single extracellular vesicles in unprocessed human plasma. Commun Biol. 5 (1), 633 (2022).
- Gul, B., Syed, F., Khan, S., Iqbal, A., Ahmad, I. Characterization of extracellular vesicles by flow cytometry: Challenges and promises. Micron. 161, 103341 (2022).
- Black, C. B., Duensing, T. D., Trinkle, L. S., Dunlay, R. T. Cell-based screening using high-throughput flow cytometry. Assay Drug Dev Technol. 9 (1), 13-20 (2011).
- Safford, H. R., Bischel, H. N. Flow cytometry applications in water treatment, distribution, and reuse: A review. Water Res. 151, 110-133 (2019).
- Kobayashi, H., et al. Precise analysis of single small extracellular vesicles using flow cytometry. Sci Rep. 14 (1), 7465 (2024).
- Welsh, J. A., et al. A compendium of single extracellular vesicle flow cytometry. J Extracell Vesicles. 12 (2), e12299 (2023).
- Carnino, J. M., Lee, H. Extracellular vesicles in respiratory disease. Adv Clin Chem. 108, 105-127 (2022).
- Sun, Y., Sethu, P. Low-stress microfluidic density-gradient centrifugation for blood cell sorting. Biomed Microdevices. 20 (3), 77 (2018).
- Strachan, B. C., Xia, H., Voros, E., Gifford, S. C., Shevkoplyas, S. S. Improved expansion of T cells in culture when isolated with an equipment-free, high-throughput, flow-through microfluidic module versus traditional density gradient centrifugation. Cytotherapy. 21 (2), 234-245 (2019).
- Malvezzi, H., Sharma, R., Agarwal, A., Abuzenadah, A. M., Abu-Elmagd, M. Sperm quality after density gradient centrifugation with three commercially available media: a controlled trial. Reprod Biol Endocrinol. 12, 121 (2014).
- Sun, Y., Sethu, P. Microfluidic adaptation of density-gradient centrifugation for isolation of particles and cells. Bioengineering (Basel). 4 (3), 67 (2017).
- Karttunen, J., et al. Precipitation-based extracellular vesicle isolation from rat plasma co-precipitate vesicle-free microRNAs. J Extracell Vesicles. 8 (1), 1555410 (2019).
- De Sousa, K. P., et al. Isolation and characterization of extracellular vesicles and future directions in diagnosis and therapy. Wiley Interdiscip Rev Nanomed Nanobiotechnol. 15 (1), e1835 (2023).
- Sidhom, K., Obi, P. O., Saleem, A. A review of exosomal isolation methods: Is size exclusion chromatography the best option. Int J Mol Sci. 21 (18), 6466 (2020).
- Coughlan, C., et al. Exosome isolation by ultracentrifugation and precipitation and techniques for downstream analyses. Curr Protoc Cell Biol. 88 (1), e110 (2020).
- Konoshenko, M. Y., et al. Isolation of extracellular vesicles from biological fluids via the aggregation-precipitation approach for downstream miRNAs detection. Diagnostics (Basel). 11 (3), 384 (2021).
- Drozdz, A., et al. Low-vacuum filtration as an alternative extracellular vesicle concentration method: A comparison with ultracentrifugation and differential centrifugation. Pharmaceutics. 12 (9), 872 (2020).
- Akbar, A., Malekian, F., Baghban, N., Kodam, S. P., Ullah, M. Methodologies to isolate and purify clinical grade extracellular vesicles for medical applications. Cells. 11 (2), 186 (2022).
- Chen, J., et al. Review on strategies and technologies for exosome isolation and purification. Front Bioeng Biotechnol. 9, 811971 (2021).
- Helling, A., et al. Investigation of microbial cell deformability by filter cake compressibility using ultrafiltration membranes. Colloids Surf B Biointerfaces. 185, 110626 (2020).
- Bagci, C., et al. Overview of extracellular vesicle characterization techniques and introduction to combined reflectance and fluorescence confocal microscopy to distinguish extracellular vesicle subpopulations. Neurophotonics. 9 (2), 021903 (2022).
- Thery, C., et al. Minimal information for studies of extracellular vesicles 2018 (MISEV2018): a position statement of the International Society for Extracellular Vesicles and update of the MISEV2014 guidelines. J Extracell Vesicles. 7 (1), 1535750 (2018).
- van Maanen, J. C., et al. A combined western and bead-based multiplex platform to characterize extracellular vesicles. Tissue Eng Part C Methods. 29 (11), 493-504 (2023).
- Xu, D., et al. MicroRNAs in extracellular vesicles: Sorting mechanisms, diagnostic value, isolation, and detection technology. Front Bioeng Biotechnol. 10, 948959 (2022).
- Lee, H., He, X., Le, T., Carnino, J. M., Jin, Y. Single-step RT-qPCR for detection of extracellular vesicle microRNAs in vivo: a time- and cost-effective method. Am J Physiol Lung Cell Mol Physiol. 318 (4), L742-L749 (2020).
- Kim, J. A., et al. Small RNA sequencing of circulating small extracellular vesicles microRNAs in patients with amyotrophic lateral sclerosis. Sci Rep. 13 (1), 5528 (2023).
- Fan, Y., et al. Differential proteomics argues against a general role for CD9, CD81 or CD63 in the sorting of proteins into extracellular vesicles. J Extracell Vesicles. 12 (8), e12352 (2023).
- Silva, A. M., et al. Quantification of protein cargo loading into engineered extracellular vesicles at single-vesicle and single-molecule resolution. J Extracell Vesicles. 10 (10), e12130 (2021).
- Salunkhe, S., Basak Dheeraj, M., Chitkara, D., Mittal, A. Surface functionalization of exosomes for target-specific delivery and in vivo imaging, tracking: Strategies and significance. J Control Release. 326, 599-614 (2020).
- Silva, A. K. A., et al. Development of extracellular vesicle-based medicinal products: A position paper of the group "Extracellular Vesicle translatiOn to clinicaL perspectiVEs - EVOLVE France". Adv Drug Deliv Rev. 179, 114001 (2021).
- Gurunathan, S., Kang, M. H., Jeyaraj, M., Qasim, M., Kim, J. H. Review of the isolation, characterization, biological function, and multifarious therapeutic approaches of exosomes. Cells. 8 (4), 307 (2019).
- Zhang, Y., et al. Exosome: A review of its classification, isolation techniques, storage, diagnostic and targeted therapy applications. Int J Nanomedicine. 15, 6917-6934 (2020).
- Colombo, M., Raposo, G., Thery, C. Biogenesis, secretion, and intercellular interactions of exosomes and other extracellular vesicles. Annu Rev Cell Dev Biol. 30, 255-289 (2014).
- Pisitkun, T., Shen, R. F., Knepper, M. A. Identification and proteomic profiling of exosomes in human urine. Proc Natl Acad Sci U S A. 101 (36), 13368-13373 (2004).
- Zaborowski, M. P., Balaj, L., Breakefield, X. O., Lai, C. P. Extracellular vesicles: Composition, biological relevance, and methods of study. Bioscience. 65 (8), 783-797 (2015).
- Kowkabany, G., Bao, Y. Nanoparticle tracking analysis: An effective tool to characterize extracellular vesicles. Molecules. 29 (19), 4672 (2024).
- Li, H. Metastatic characteristics of SY86B human gastric carcinoma in athymic nude mice. Zhonghua Zhong Liu Za Zhi. 10 (6), 421-423 (1988).
- Zhao, Z., Wijerathne, H., Godwin, A. K., Soper, S. A. Isolation and analysis methods of extracellular vesicles (EVs). Extracell Vesicles Circ Nucl Acids. 2, 80-103 (2021).
- Gandham, S., et al. Technologies and standardization in research on extracellular vesicles. Trends Biotechnol. 38 (10), 1066-1098 (2020).
- van der Vlist, E. J., Nolte-'t Hoen, E. N., Stoorvogel, W., Arkesteijn, G. J., Wauben, M. H. Fluorescent labeling of nano-sized vesicles released by cells and subsequent quantitative and qualitative analysis by high-resolution flow cytometry. Nat Protoc. 7 (7), 1311-1326 (2012).
- Pospichalova, V., et al. Simplified protocol for flow cytometry analysis of fluorescently labeled exosomes and microvesicles using dedicated flow cytometer. J Extracell Vesicles. 4, 25530 (2015).
- Breitwieser, K., et al. Detailed characterization of small extracellular vesicles from different cell types based on tetraspanin composition by ExoView R100 platform. Int J Mol Sci. 23 (15), 8544 (2022).
- An, H. J., Cho, H. K., Song, D. H., Kee, C. Quantitative analysis of exosomes in the aqueous humor of Korean patients with pseudoexfoliation glaucoma. Sci Rep. 12 (1), 12875 (2022).
- Hefley, B. S., McKay, T. B., Hutcheon, A. E. K., Ciolino, J. B., Karamichos, D. Corneal epithelial-stromal constructs to study differences associated with diabetes mellitus. Exp Eye Res. 248, 110100 (2024).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados