Method Article
Charakterisierung extrazellulärer Vesikel aus biologischen Flüssigkeiten
In diesem Artikel
Zusammenfassung
Hier demonstrieren wir die Charakterisierungsmethode für extrazelluläre Vesikel (EVs), die aus biologischen Flüssigkeiten wie Tränen und Speichel von menschlichen Probanden gewonnen wurden. Der bei dieser Methode verwendete Scanner ist in der Lage, den Phänotyp, die Größe und die Gesamtpartikelzahl von EVs aus 1 μl der Probe zu erkennen.
Zusammenfassung
Extrazelluläre Vesikel (EVs) sind Strukturen, die aus Zellen hergestellt werden und an der interzellulären Kommunikation teilnehmen, indem sie Biomoleküle von einer Zelle zur anderen transportieren. Es hat sich gezeigt, dass Elektrofahrzeuge kurze und weite Strecken im Körper zurücklegen und gewebespezifisch sind. EVs kommen nicht nur im Gewebe vor, sondern können auch in praktisch allen Körperflüssigkeiten wie Tränen, Speichel, Gehirn-Rückenmarksflüssigkeit, Blut usw. gefunden werden. Obwohl EVs nicht-invasiv aus Tränen und Speichel gewonnen werden können, können nur kleine Mengen auf einmal gesammelt werden, was zu Problemen bei der Gewinnung von genügend EVs für die Analyse von Proteinen führen kann. Der in diesem Artikel besprochene Scanner ist ein Nanopartikel-Analysator, der eine Lösung für dieses Problem bietet und es uns ermöglicht, den Phänotyp, die Größe und die Gesamtpartikelzahl von EVs aus nur 1 μl biologischer Flüssigkeit zu charakterisieren und zu untersuchen. Dieses Protokoll wird das Wissen über EVs aus kleinen Probenmengen erweitern, die nur schwer aus Patienten extrahiert werden können. Dies könnte den Patientenkomfort verbessern und möglicherweise neue therapeutische Ziele für eine Reihe von Krankheiten und Störungen identifizieren.
Einleitung
Zellen kommunizieren mit Nachbarzellen über verschiedene Signalmechanismen, einschließlich der Freisetzung von extrazellulären Vesikeln (EVs), die eine Schlüsselrolle bei der interzellulären Kommunikation spielen. EVs nehmen an der interzellulären Kommunikation teil, indem sie genetische Fracht wie DNA, RNA und Proteine von einer Zelle zur anderen weitergeben 1,2,3,4,5. Derzeit gibt es drei Kategorien von EVs: Exosomen, Mikrovesikel und apoptotische Körper, die sich durch ihre Größe auszeichnen. Die Exosomen sind mit einem Durchmesser von 30-150 nm 6,7,8 die kleinsten und werden aus dem endosomalen Membransystem 9,10,11 gebildet (Abbildung 1). Mikrovesikel sind größer als Exosomen, da sie von 100 bis 1000 nmreichen 12,13,14 und Knospen von der Plasmamembran 11,12,13 (Abbildung 1). Apoptotische Körper sind die größten der EVs und reichen von 1000 bis 5000 nm 12,14,15, und sie knospen auch von der Plasmamembran ab 12,16,17 (Abbildung 1). Abgesehen von der Größe können EVs auf der Grundlage anderer biophysikalischer Merkmale klassifiziert werden, zu denen die Dichte, molekulare Marker wie CD63, CD81, CD9 und auch der Biogenese-Mechanismusgehören 18. Elektrofahrzeuge können kurze Strecken zwischen benachbarten Zellen und lange Strecken im ganzen Körper zurücklegen 19,20,21,22,23. EVs können in biologischen Flüssigkeiten wie Blut 24,25,26, zerebraler Rückenmarksflüssigkeit 27,28,29, Tränen 30,31,32 und Speichel 33,34,35 gefunden werden, um nur einige zu nennen.
Bis heute ist die Ultrazentrifugation eine der bekanntesten Methoden zur Isolierung von EVs aus Proben 36,37,38. Diese Methode erfordert mehrere Zentrifugations- und Ultrazentrifugationsrunden, die durch Erhöhen der Geschwindigkeit zur Isolierung der EVs von Zellen und Zellresten durchgeführt werden können. Diese Methode kann mit der niedrigen Geschwindigkeit mit Pelletzellen begonnen werden, gefolgt von einer mittleren Geschwindigkeit zur Eliminierung größerer Vesikel und schließlich einem Ultrazentrifugationsschritt zur Pelletierung von EVs18. Obwohl die Ultrazentrifugation als die beste Methode zur Isolierung gilt, gibt es immer noch einige Einschränkungen, da sie die Morphologie der EVsverändert 39,40,41. Eine weitere Technik zur Isolierung von EVs ist die Durchflusszytometrie, die die Vorteile der Bewertung mehrerer Zeitpunkte und Endpunkte sowie der Hochdurchsatz-Einzel-EV-Analyse hervorhebt. Zu den Einschränkungen der Durchflusszytometrie gehören jedoch unter anderem das Verstopfen der Poren und schwache Signale. Ein weiterer Ansatz ist die Gradientenzentrifugation, bei der Materialien mit unterschiedlichen Dichten mit den EVs zentrifugiert werden und die im Vergleich zur Ultrazentrifugation eine bessere Trennung der EVs ermöglicht. Obwohl diese Technik die Trennung verbessert, ist sie arbeitsintensiv, zeitaufwändig und kann zu erheblichen Probenverlusten führen. Darüber hinaus können Fällung und Filtration auch zur Isolierung von Elektrofahrzeugen verwendet werden. Beide Techniken sind einfach und schnell, aber beide können zu einer Kontamination der Probe führen. Es gibt zwar mehrere Techniken zur Isolierung von Elektrofahrzeugen, aber jede Technik hat ihre Vor- und Nachteile, die unten aufgeführt sind (Tabelle 1): 42,43,44,45,46,47,48,49,50,51,52,53,54, 55, 56,57,58,59,60,61,62,63,64,65,66,67.
Sobald die EVs isoliert sind, können molekulare Assays durchgeführt werden, um die EVs zu charakterisieren. Western Blots sind ein gebräuchlicher Assay zur Suche nach Oberflächen- und Frachtproteinexpression 68,69,70 und Polymerase-Kettenreaktion (PCR) wird für die miRNA-Expression 71,72,73 für EVs 74,75,76 verwendet. Diese Assays sind etabliert und können faszinierende Ergebnisse liefern. Eine Einschränkung dieser Methoden besteht darin, dass sie eine große Menge an Proteinen oder RNA aus EVs benötigen, um einen Messwertvon 77 zu erhalten, was zunächst für Proben mit einem kleinen Volumen oder einer kleinen EV-Konzentration ein Problem darstellt.
Der in diesem Dokument besprochene Nanopartikel-Analysator ermöglicht es dem Benutzer, viele der in Tabelle 1 und Tabelle 2 genannten Einschränkungen zu überwinden 78,79,80,81,82,83,84,85,86,87,88,89,90 . Diese Methode erfordert keine Isolationstechniken, die dazu beitragen, den geringeren Ertrag von Elektrofahrzeugen zu überwinden. Diese Methode ermöglicht es dem Benutzer auch, die Oberflächen- und Frachtproteine, die Gesamt-EV-Anzahl und die EV-Größe ab einem Probenvolumen von nur 1 μl zu analysieren. Dies geschieht durch die Verwendung von Tetraspanin-Chips, die vom Unternehmen zur Verfügung gestellt werden und einen Antikörper-Microarray mit den Tetraspanin-Antikörpern CD63, CD81 und CD9 verwenden, um EVs in einer Lösung zu identifizieren, wie in Abbildung 2 gezeigt. Fluoreszierende Antikörper bestätigen das Vorhandensein von Elektrofahrzeugen und verhindern, dass kontaminierende Partikel die Ergebnisse verzerren.
Das übergeordnete Ziel dieser Technik ist es, eine weniger zeitaufwändige Methode zur Analyse von EVs sowie zur Analyse von EVs aus einem kleinen Probenvolumen bereitzustellen. Die Verwendung dieses Nanopartikel-Analysators ermöglicht es den Benutzern, die Größe, die Gesamtpartikelzahl und die Oberflächenproteine bereits ab 1 μl einer Probe zu analysieren, was ideal für biologische Flüssigkeiten wie Tränen und Speichel ist.
Protokoll
Alle beschriebenen Studien hielten sich an die Deklaration von Helsinki. Von jedem Probanden wurde eine schriftliche Zustimmung eingeholt, bevor er in die Studie aufgenommen wurde. Die Genehmigung des Institutional Review Board (IRB) durch das Universitätskrankenhaus Aarhus (1-10-72-77-14) und das Dean McGee Institute (1576837-2) wurde gemäß den föderalen und institutionellen Richtlinien erhalten. Vor der Verarbeitung wurden alle Tränen- und Speichelproben anonymisiert. Alle Studien wurden vom North Texas Regional Institutional Review Board (#2020-030) überprüft und genehmigt. Das folgende Protokoll hält sich an alle Richtlinien und wurde, wie oben erwähnt, genehmigt.
1. Tag 1: Probenvorbereitung und Inkubation
- Entnehmen Sie Proben biologischer Flüssigkeiten, d. h. Tränen und Speichel von einer oder mehreren Probanden oder tauen Sie Proben aus dem Gefrierschrank auf.
- Tränen: Sammeln Sie die Tränen passiv aus dem Außenmeniskus mit einem Kapillarrohr aus Glas.
- Legen Sie das Glaskapillarrohr auf das untere Augenlid und achten Sie darauf, die Hornhaut nicht zu berühren.
- Sammeln Sie ein Volumen von 10 μL Tränen innerhalb von 10 min.
- Die Tränen sofort verwerten oder bei -20 °C lagern.
- Speichel: Sammeln Sie Speichel aus passivem Speichel in einem 1,5-ml-Mikrozentrifugenröhrchen
- Erlauben Sie dem Spender, Speichel in seinem Mund zu sammeln.
- Bitten Sie den Spender, in das Röhrchen mit einem Trichter zu sabbern.
- Speichel sofort verwenden oder bei -20 °C lagern.
- Tränen: Sammeln Sie die Tränen passiv aus dem Außenmeniskus mit einem Kapillarrohr aus Glas.
- Legen Sie den/die Tetraspanin-Chip (die Tetraspanin-Chips) für 15 Minuten in die Chipwaschplatte, bevor Sie Proben hinzufügen, damit sie Raumtemperatur erreichen können. Achten Sie beim Platzieren der Chips in der Vertiefung darauf, dass die Zahlen auf der Unterseite nach oben zeigen und dass die Chips die Seitenwände der Vertiefung(en) nicht berühren.
- Notieren Sie die ID-Nummer und die Chipnummer im Notebook, um den Überblick über den/die Chip(s) zu behalten.
- Schließen Sie den Universal Serial Bus (USB) an, der mit dem Kit geliefert wird, und extrahieren Sie den Ordner mit der zugehörigen Kit-Nummer, und speichern Sie ihn in einem bestimmten Bereich Ihrer Wahl.
- Pipettieren Sie 99 μl Lösung B in ein 1,5-ml-Röhrchen.
- Geben Sie 1 μl biologische Flüssigkeit in das im vorherigen Schritt vorbereitete 1,5-ml-Röhrchen.
- Drehen Sie die Röhren herunter.
- Nehmen Sie 70 μl der Lösung aus Schritt 1.6 und pipettieren Sie sie auf den Tetraspanin-Chip.
- Legen Sie die Folie auf die Chipwaschplatte, um ein Verdampfen der Probe zu verhindern.
- 16 Stunden bei Raumtemperatur (RT) auf einer vibrationsfreien Bank inkubieren.
2. Tag 2: Spänewaschen
- Schalten Sie die Chipswaschmaschine ein.
- Entfernen Sie die Folie aus Schritt 1.9 von der Platte.
- Legen Sie die Platte in die Chipwaschanlage, stellen Sie sicher, dass sie einrastet, und schließen Sie den Deckel.
HINWEIS: Die Platte lässt sich nicht leicht bewegen, wenn sie korrekt eingerastet ist. - Drücken Sie die Option CW-TETRA v0 an der Chipwaschanlage.
- Wählen Sie die Startreihe und die Anzahl der zu waschenden Reihen aus.
- Drücken Sie auf der Chipswaschmaschine auf Weiter .
- Während der/die Chip(s) gewaschen werden, bereiten Sie die Blockierungslösung, die die Antikörper enthält, in einem Röhrchen wie folgt vor:
- Bereiten Sie 300 μl Blockierungslösung pro Chip vor.
- Fügen Sie 0,6 μl oder jeden Antikörper pro Chip hinzu (Vortex- und Spin-Down-Antikörper, bevor Sie sie zur Blockierungslösung hinzufügen)
- Wirbeln Sie die Blockierungslösung + den Antikörper-Cocktail auf.
HINWEIS: Decken Sie die Blockierungslösung + den Antikörpercocktail vor Licht ab, um die Stabilität des Antikörpers zu gewährleisten. Dies muss am Tag der Nutzung erfolgen.
- Wenn die Chipscheibe zwitschert, fügen Sie 250 μl der Blockierungslösung + des Antikörpercocktails auf den/die Chip(s) hinzu.
- Schließen Sie den Deckel und drücken Sie Weiter.
HINWEIS: Die Chipwaschmaschine führt nun eine 1-stündige Inkubation durch, zusammen mit mehreren zusätzlichen Wasch- und Spülrunden. - Wenn die Spänewaschanlage piept, entfernen Sie die Spänewaschplatte und schieben Sie den/die Späne(n) die Rampe hinauf.
- Setzen Sie die Platte wieder in die Spänescheibe ein und stellen Sie sicher, dass sie einrastet. Schließen Sie den Deckel und drücken Sie Weiter.
- Wenn die Chipwaschmaschine das Programm abgeschlossen hat, entfernen Sie die Platte und legen Sie den/die Chip(s) auf ein Papiertuch. Decken Sie den/die Chip(s) vor dem Licht ab.
3. Tag 2: Scannen des/der Chips/Chips
- Schalten Sie den Scanner ein und öffnen Sie die Scansoftware.
HINWEIS: Der Scanner wird einer Selbstprüfung unterzogen, um festzustellen, ob Fehler vorliegen oder ob der Scanvorgang fortgesetzt werden kann. - Wählen Sie Ordner speichern und wählen Sie den Speicherort für die Dateien aus.
- Wählen Sie ChipFile Folder und wählen Sie den in Schritt 1.4 gespeicherten Ordner aus.
- Wählen Sie die Dropdown-Option aus, um jeden Chip auszuwählen und ihn in der Spanposition auf dem Spannfutter zu platzieren. Wiederholen Sie diesen Schritt für alle Chips, die gescannt werden müssen.
- Schalten Sie die Fluoreszenz ein, indem Sie auf die drei Quadrate unter "Chip-Position" klicken. Quadrate werden gelb, blau bzw. rot.
- Entfernen Sie den Futterdeckel, indem Sie den Deckel nach unten drücken und ziehen.
- Platzieren Sie den/die Späne(n) auf dem Spannfutter und stellen Sie sicher, dass sie fest sitzen.
- Setzen Sie den Futterdeckel wieder auf, indem Sie die Stifte in die Löcher stecken, drücken Sie den Deckel nach unten und schieben Sie ihn, um ihn zu verriegeln.
- Stellen Sie das Bohrfutter auf die Bühne und stellen Sie sicher, dass es einrastet.
- Wählen Sie Scan Chips aus.
- Wählen Sie OK aus.
- Sobald der Tisch vollständig in die Maschine eingefahren ist, schließen Sie die Tür.
HINWEIS: Das Scannen eines einzelnen Chips dauert ca. 12 Minuten. - Sobald das Programm mit dem Scannen der Chips fertig ist, öffnen Sie die Tür, um die Stufenprüfung auszuwerfen, um zu überprüfen, ob die Scans erfolgreich waren.
- Wenn die Scans erfolgreich waren, entsorgen Sie den/die Chip(s) und platzieren Sie den nächsten Satz Chips auf dem Spannfutter oder stellen Sie das leere Spannfutter wieder auf die Bühne. Beenden Sie dann die Software und schalten Sie das Gerät aus, wenn Sie fertig sind.
- Wenn die Scans nicht erfolgreich waren, sehen Sie sich den Fehlercode an, und korrigieren Sie den Fehler.
4. Datenverarbeitung
- Öffnen Sie die Analysesoftware.
- Wählen Sie Prescan Data und dann den Ordner aus, in dem sich der Scan von Tag 2, Schritt 3.2 befindet.
- Wählen Sie Weiter aus.
- Beschriften Sie den/die Chip(s) in der Spalte Probenname mit dem Namen der Probe, indem Sie auf die Zelle klicken und die Proben-ID eingeben.
- Beschriften Sie als Nächstes die Tetraspanine in der Spalte Kanalname . Rot = CD63, Grün = CD81 und Blau = CD9.
- Wählen Sie Weiter aus.
- Wählen Sie in der Gruppe Spot auswählen die Option Hoher CV aus, um das Dropdown-Feld zu überprüfen und die hohen CVs für jeden Chip zu deaktivieren, indem Sie einen oder zwei Spots deaktivieren.
- Wählen Sie als Nächstes Hohe Anzahl aus, und versuchen Sie, die Warnungen mit hoher Anzahl zu deaktivieren.
HINWEIS: Einige Proben haben möglicherweise eine zu hohe Anzahl, können aber dennoch analysiert werden. - Wählen Sie Alle Spots aus dem Dropdown-Menü, wählen Sie Spot-Montage und überprüfen Sie jeden Chip visuell auf eventuell vorhandene Verformungen oder Kratzer.
- Wählen Sie Weiter aus.
- Legen Sie im CD63-Kanal den Mindestwert auf 300 fest, um die roten Blutkörperchen in der Zeile "Durchschnittlich % eingeschlossen" zu entfernen. gelbe Zellen sind in Ordnung und wandern zum CD81-Kanal. Wenn das Setzen des Minimalwerts auf 300 die rote Zelle nicht in gelb oder weiß ändert, fahren Sie mit Schritt 4.14 zur Fehlerbehebung fort.
- Legen Sie im CD81-Kanal den Mindestwert auf 300 fest, um die roten Blutkörperchen in der Zeile "Durchschnittlich % eingeschlossen" zu entfernen. gelbe Zellen sind in Ordnung und gehen zum CD9-Kanal über. Wenn das Setzen des Minimalwerts auf 300 die rote Zelle nicht in gelb oder weiß ändert, fahren Sie mit Schritt 4.14 zur Fehlerbehebung fort.
- Legen Sie im CD9-Kanal den Mindestwert auf 400 fest, um rote Blutkörperchen in der Zeile "Durchschnittlich % eingeschlossen" zu entfernen. Gelbe Zellen sind in Ordnung, und fahren Sie mit Schritt 4.15 fort. Wenn das Setzen des Minimalwerts auf 400 die rote Zelle nicht in gelb oder weiß ändert, fahren Sie mit Schritt 4.14 zur Fehlerbehebung fort.
- Fehlerbehebung
- Erhöhen Sie den Mindestwert um 100 Einheiten.
- Wenn Schritt 4.14.1 nicht funktioniert hat, wählen Sie den Chip im Abschnitt Zu bearbeitende Chips auswählen aus und wählen Sie den Chip aus. Wählen Sie "Partikelanzahl" und deaktivieren Sie die Punkte mit einer hohen Partikelanzahl. Wählen Sie Intensität/Größe aus, wählen Sie Alle Chips aus und prüfen Sie, ob das Problem dadurch behoben wird.
- Wenn Schritt 4.14.2 nicht funktioniert hat, wählen Sie den Chip im Abschnitt Zu bearbeitende Chips auswählen aus und wählen Sie den Chip aus. Wählen Sie Single-Chip-Cutoffs und erhöhen Sie den Mindestwert um 100 Einheiten. Wählen Sie dann Alle Chips , um zu sehen, ob die rote Zelle jetzt gelb ist. Wenn dies nicht funktioniert, wiederholen Sie diesen Schritt, bis die Zelle "Durchschnittlich % eingeschlossen" jetzt gelb oder weiß ist.
- Wählen Sie Weiter aus.
- Deaktivieren Sie den IM-Kanal.
- Legen Sie den Heatmap-Wert in der unteren rechten Ecke so fest, dass er alle Partikel einschließt, und wählen Sie Plot zum Bericht hinzufügen aus.
- Passen Sie die maximale Anzahl im Dropdown-Feld an und geben Sie manuell einen Wert ein.
- Wählen Sie als Nächstes das Dropdown-Menü oben in der Mitte unter Capture Probes aus, und wählen Sie CD63 aus.
- Wählen Sie Plot zum Bericht hinzufügen aus, um dem Bericht die Gesamtpartikelanzahl hinzuzufügen.
- Wählen Sie das Dropdown-Menü unter Analysemodus aus, wählen Sie Kolokalisierung und dann Diagramm zum Bericht hinzufügen aus.
- Wählen Sie als Nächstes Größe aus, wählen Sie Ansicht in Diagramm ändern aus, und wählen Sie Diagramm zum Bericht hinzufügen aus.
- Wählen Sie abschließend Bilder und dann Plot zum Bericht hinzufügen aus.
- Wiederholen Sie die Schritte 4.18 bis 4.22 für CD81 und CD9 und stellen Sie sicher, dass Sie den IM-Kanal jedes Mal abwählen.
- Nachdem Sie dem Bericht alle Elemente hinzugefügt haben, wählen Sie Bericht exportieren aus, stellen Sie sicher, dass er in der richtigen Datei gespeichert wird, und wählen Sie Ordner aus.
HINWEIS: Excel-Dateien und Bilder wurden generiert und können nun für weitere Analysen verwendet werden.
Ergebnisse
Bei der Analyse der Flecken auf den Chips ist eine optimale Konzentration von EVs zu erreichen, indem fluoreszenzmarkierte EVs in den Kanälen CD63, CD81 und CD9 beobachtet werden, wobei der MIgG-Kanal 30,91 schwarz bleiben sollte, da dies der Kontrollkanal ist, wie in Abbildung 3A zu sehen ist. CD81 wird für biologische Flüssigkeiten reduziert. Wenn in den Kanälen CD63, CD81 und CD9 keine Fluoreszenz vorhanden ist, fehlen EVs (Abbildung 3B). Achten Sie darauf, den Chip nicht zu übersättigen (Abbildung 3C). Dies wird es für die Analysesoftware schwierig machen, die Genauigkeit der Größe, der Gesamtpartikelzahl und der Phänotypen der EVs zu berechnen. Ausgehend von den Punkten mit der optimalen Konzentration kann die Analysesoftware die Größe (50-200 nm), die Gesamtpartikelzahl und die Tetraspanin-Expression der EVs messen (Abbildung 4) und zur weiteren Analyse in einzelne Tabellenkalkulationen exportiert werden. Die Tetraspanin-Analyse wird die Kolokalisation der Oberflächenproteine auf den EVs beinhalten. Die Kolokalisierung wird mit einem "/" dargestellt, z. B. "CD63/CD81". Dies bedeutet nicht, dass CD63 und CD81 kombiniert werden; Das bedeutet, dass sich sowohl CD63 als auch CD81 auf der Oberfläche des Elektrofahrzeugs befinden.
Diese Ergebnisse werden wertvolle Erkenntnisse zwischen gesunden und kranken Proben liefern. Wir werden in der Lage sein festzustellen, ob gesunde oder kranke Proben mehr EVs, größere oder kleinere EVs produzieren und welchen Phänotyp die EVs haben. Jedes oder alle diese Merkmale könnten eine Rolle bei der Biogenese und/oder dem Fortschreiten der Krankheit spielen. Mit diesen Ergebnissen werden wir in der Lage sein, zu sehen, ob es ein Fehlen oder einen Anstieg der Tetraspanin-Spiegel gibt, was Aufschluss über Zellprozesse und die Bildung von EVs geben kann.
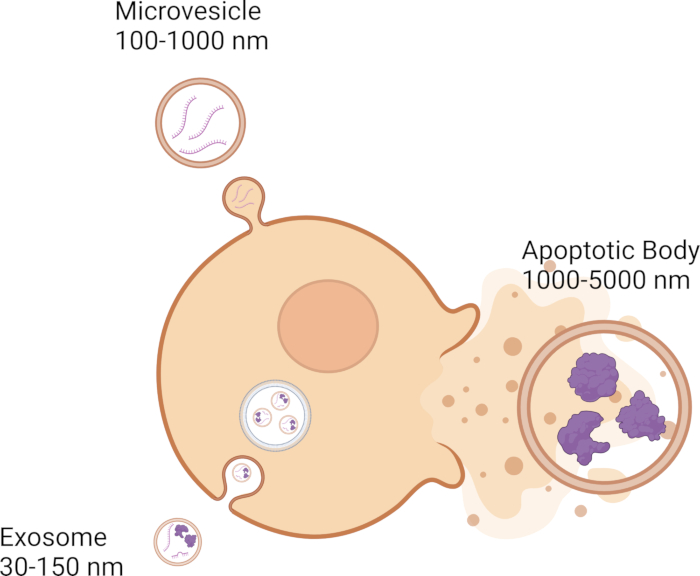
Abbildung 1: Die Typen und Größen der verschiedenen Elektrofahrzeuge. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Schematische Darstellung der Art und Weise, wie die Tetraspanin-Chips EVs erfassen und detektieren. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
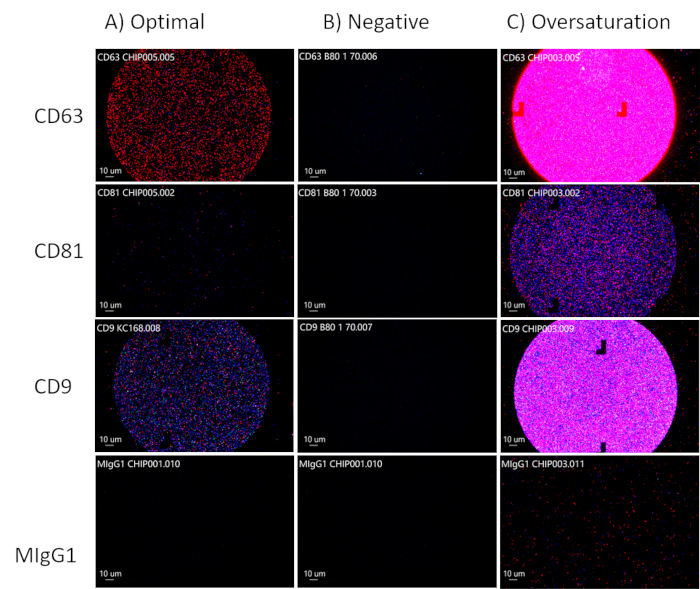
Abbildung 3: Optimierung der EV-Konzentrationen. (A) Optimale Konzentration von EVs. (B) Negatives Ergebnis: keine Elektrofahrzeuge vorhanden. (C) Übersättigung mit Elektrofahrzeugen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Vom Nanopartikel-Analysator erzeugte Daten. (A) EV-Durchmessergröße, gemessen in Nanometern. (B) Gesamtpartikelzahl von Elektrofahrzeugen. (C) EV-kolokalisierter Phänotyp. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Technik | Vorteile | Begrenzungen |
| Ultrazentrifuge | Hohe Reinheit42,43 | Veränderungen der EV-Morphologie42,44 |
| Homogenität42,45 | Erfordert großes Volumen42,44,46 | |
| Funktionalität42,43 | teuer42,43 | |
| Durchflusszytometrie | Auswertung von Proben zu mehreren Zeitpunkten47 | Schwaches Signal48,49 |
| Mehrere Endpunkte47,50 | Verstopfte Poren51 | |
| Einzel-EV-Analyse mit hohem Durchsatz52,53 | ||
| Dichtegradienten-Zentrifugation | Herstellung hochreiner Proben54 | Arbeitsintensiv55,56 |
| Vielzahl von Proben55,57 | zeitaufwändig55,58 | |
| Erheblicher Ertragsverlust54 | ||
| Niederschlag | Einfach59,60 | Verschmutzung36,44,61 |
| Schnell61,62 | Nicht spezifisch63 | |
| Hohe Rendite44,59,62 | ||
| Neutraler pH-Wert36 | ||
| Filtrierung | Einfach64 | Fallen-Exosomen65 |
| Schnell64 | Schädigt große Vesikel36 | |
| Preiswert64 | Kuchen filtern66,67 | |
| Verstopfung von Exosomen65 |
Tabelle 1: Vorteile und Grenzen von EV-Isolationstechniken. Eine Liste der verschiedenen Möglichkeiten zur Isolierung von Elektrofahrzeugen, einschließlich ihrer Vor- und Nachteile.
| Technik | Vorteile | Begrenzungen |
| Dynamische Lichtstreuung | Messung von Partikeln mit einer Größe von 1 nm bis 6 μM78 | Nicht geeignet für die Messung komplexer Exosomenproben mit großem Größenbereich79 |
| Die untere Grenze liegt bei 10 nm, geeignet für monodisperse Systeme79 | Nicht in der Lage, kontaminierte Proteine von Exosomen zu unterscheiden79 | |
| Transmissionselektronenmikroskopie (TEM) | Morphologie der Exosomenbeobachten 79,80 | Komplizierte Probenvorbereitung79 |
| Innere Strukturbeachten 81 | Nicht in der Lage, Exosomen anhand von Größe und Form zu unterscheiden, da die Fluoreszenzsignale übertrieben sind82 | |
| Analyse der Nanopartikel-Verfolgung | Messung von Konzentration, Größe und Größenverteilung von Exosomen im Bereich von 10 nm bis 2 μM78 | Kann die EVs nicht von Proteinaggregaten und anderen Kontaminanten unterscheiden83 |
| Schnelle Probenvorbereitung und -messung78,84 | Teures NTA-Instrument85 | |
| Proben können in nativer Form zurückgewonnen werden84 | Empfindlich gegen Vibrationen85 | |
| Western-Blot (WBs) | kann Markerproteine qualitativ und quantitativ analysieren79,86 | Kompliziert und zeitaufwändig79 |
| Analyse von Exosomen aus Zellkulturmedien79 | EV-Isolate können Lipoproteine und andere Kontaminanten enthalten86 | |
| Durchflusszytometrie | Höhere Empfindlichkeit und hochauflösende Bildgebung für | Zeit- und aufwändig mit Nachweisgrenze von 400 nm79,88 |
| Unterscheidung von gefärbten Exosomen von Containments87 | ||
| Erforderliche niedrige Probenkonzentration79 | Optische Signale beeinträchtigen die Genauigkeit und Auflösung88 | |
| Exoview (Englisch) | Messung von Tetraspaninen (CD9, CD63 und CD81) an Exosomen89 | Misst keine größeren EV-Größen75 |
| Misst EV-Frachtproteine90 |
Tabelle 2: Vorteile und Grenzen von EV-Charakterisierungstechniken.
Diskussion
Der wichtigste Schritt in diesem Protokoll besteht darin, sicherzustellen, dass eine optimale Konzentration von EVs erreicht wird. Es müssen genügend EVs vorhanden sein, um einen Messwert zu erhalten, aber nicht zu viele EVs, die den Chip übersättigen. Der beste Weg, um die optimale EV-Konzentration zu bestimmen, besteht darin, einen Optimierungslauf mit 1 μl Probe durchzuführen und zu prüfen, ob die Konzentration angepasst werden muss. Ein weiterer kritischer Schritt besteht darin, festzustellen, ob die Probe eine Fülle von Zellresten enthält, die durch Betrachten großer Brocken auf den Chips in der Analysesoftware bestimmt werden können. Wenn die Probe Zellreste enthält, sollte eine einfache Zentrifugation oder Filterung der Probe dieses Problem beheben.
Ein weiterer, entscheidender Schritt besteht darin, sicherzustellen, dass der/die Chip(s) die Wände des Bohrlochs nicht berühren, und den Kontakt mit dem mittleren Bereich des Chips/der Chips zu vermeiden, wenn sie die Rampe hinaufgelegt werden. Der Scanner liest aus dem Quadrat in der Mitte des Chips, daher ist es wichtig, diesen Bereich nicht mit der Pinzette zu berühren, um eine Störung der Elektrofahrzeuge zu vermeiden.
Diese Methode weist einige Einschränkungen auf, wie z. B. eine unzureichende Konzentration von EVs für die Detektion durch das Instrument. Die Konzentration kann entweder durch Trocknen der Probe oder durch Verwendung eines Konzentratorröhrchens verbessert werden. Eine weitere Einschränkung besteht darin, dass die Analysesoftware nur EVs im Bereich von 50-200 nm misst und einige Mikrovesikel und alle apoptotischen Körper von den Größenmessungen75 ausschließt.
Es gibt viele Möglichkeiten zu isolieren (Tabelle 1 42,43,44,45,46,47,48,49,50,51,52, 53,54,55,56,57,58,59,60, 61,62,63,64,65,66,67) und EVs analysieren (Tabelle 2 78,79,80,81,82,83,84,85, 86,87,88,89,90), und der aktuelle Goldstandard für die EV-Isolierung ist die Ultrazentrifugation 36,37,38, um die Zellen für Assays wie Western Blots 68,69,70 und PCR71,72,73,74 zu pelletieren,75,76. Während dieses Protokoll für große Proben gut funktioniert 42,44,46, kann die Gewinnung biologischer Flüssigkeiten eine Herausforderung darstellen, und oft kann nur ein kleines Volumen von Proben auf einmal entnommen werden, was für die Ultrazentrifugation nicht ideal ist. Im Gegensatz dazu ermöglicht die Verwendung dieses Nanopartikel-Analysators dem Benutzer, wertvolle Daten wie Größe, Gesamtpartikelzahl und Phänotyp der EVs in nur 1 μl Probe zu generieren, was ihn ideal für biologische Flüssigkeiten macht. Wir werden in der Lage sein, das Wissen über EVs aus kleinen Mengen von Proben, die schwer zu extrahieren sind, zu erweitern, was den Patientenkomfort erhöhen und möglicherweise potenzielle therapeutische Ziele für verschiedene Krankheiten und Störungen finden kann.
Offenlegungen
Die Autoren haben keine konkurrierenden finanziellen Interessen oder andere Interessenkonflikte offenzulegen.
Danksagungen
Wir danken den NIH für die Finanzierung (EY031316 und EY034714). Wir möchten uns auch bei UNTHSC und NTERI für den Laborraum bedanken.
Materialien
| Name | Company | Catalog Number | Comments |
| ChipWasher 100 | NanoView | EV-CW100 | Incubates, washes, rinses and dries the tetraspanin chips. This current model is no longer available. Price at time of purchase: $9,995.00 |
| ExoView Analyzer software | NanoView | N/A | Analyzes the chip informations and produces excel files for further analysis. No longer available. |
| ExoView R100 | NanoView | EV-R100 | Used to scan the tetraspanin chips at 3 wavelengths. This current model is no longer available. Price at time of purchase: $110,000.00 |
| ExoView Scanner software | NanoView | N/A | Scans the chips at 3 different wavelengths. No longer available. |
| Human Tetraspanin Kits | Unchained Labs | EV-TETRA-C | Includes 8 tetraspanin chips, Incubation Solution, Blocking Solution, CD63 antibody, CD81 antibody, CD9 antibody, Solution A, Solution B, USB, and plate cover. |
Referenzen
- Abels, E. R., Breakefield, X. O. Introduction to extracellular vesicles: Biogenesis, RNA cargo selection, content, release, and uptake. Cell Mol Neurobiol. 36 (3), 301-312 (2016).
- Radler, J., Gupta, D., Zickler, A., Andaloussi, S. E. Exploiting the biogenesis of extracellular vesicles for bioengineering and therapeutic cargo loading. Mol Ther. 31 (5), 1231-1250 (2023).
- Gupta, D., Zickler, A. M., El Andaloussi, S. Dosing extracellular vesicles. Adv Drug Deliv Rev. 178, 113961 (2021).
- Dixson, A. C., Dawson, T. R., Di Vizio, D., Weaver, A. M. Context-specific regulation of extracellular vesicle biogenesis and cargo selection. Nat Rev Mol Cell Biol. 24 (7), 454-476 (2023).
- Keshtkar, S., Azarpira, N., Ghahremani, M. H. Mesenchymal stem cell-derived extracellular vesicles: novel frontiers in regenerative medicine. Stem Cell Res Ther. 9 (1), 63 (2018).
- Zhang, H., et al. Exosome-induced regulation in inflammatory bowel disease. Front Immunol. 10, 1464 (2019).
- Barzin, M., et al. Application of plant-derived exosome-like nanoparticles in drug delivery. Pharm Dev Technol. 28 (5), 383-402 (2023).
- Zhou, C., et al. Stem cell-derived exosomes: emerging therapeutic opportunities for wound healing. Stem Cell Res Ther. 14 (1), 107 (2023).
- Kalluri, R., LeBleu, V. S. The biology, function, and biomedical applications of exosomes. Science. 367 (6478), aau6977 (2020).
- Mondal, J., et al. Hybrid exosomes, exosome-like nanovesicles and engineered exosomes for therapeutic applications. J Control Release. 353, 1127-1149 (2023).
- van Niel, G., D'Angelo, G., Raposo, G. Shedding light on the cell biology of extracellular vesicles. Nat Rev Mol Cell Biol. 19 (4), 213-228 (2018).
- Paul, N., Sultana, Z., Fisher, J. J., Maiti, K., Smith, R. Extracellular vesicles- crucial players in human pregnancy. Placenta. 140, 30-38 (2023).
- Zaldivia, M. T. K., McFadyen, J. D., Lim, B., Wang, X., Peter, K. Platelet-derived microvesicles in cardiovascular diseases. Front Cardiovasc Med. 4, 74 (2017).
- Tamasi, V., Nemeth, K., Csala, M. Role of extracellular vesicles in liver diseases. Life (Basel). 13 (5), 1117 (2023).
- Fu, Y., et al. Emerging understanding of apoptosis in mediating mesenchymal stem cell therapy. Cell Death Dis. 12 (6), 596 (2021).
- Santavanond, J. P., Rutter, S. F., Atkin-Smith, G. K., Poon, I. K. H. Apoptotic bodies: Mechanism of formation, isolation and functional relevance. Subcell Biochem. 97, 61-88 (2021).
- Yu, L., et al. Apoptotic bodies: bioactive treasure left behind by the dying cells with robust diagnostic and therapeutic application potentials. J Nanobiotechnology. 21 (1), 218 (2023).
- Momen-Heravi, F., Getting, S. J., Moschos, S. A. Extracellular vesicles and their nucleic acids for biomarker discovery. Pharmacol Ther. 192, 170-187 (2018).
- Liu, Y. J., Wang, C. A review of the regulatory mechanisms of extracellular vesicles-mediated intercellular communication. Cell Commun Signal. 21 (1), 77 (2023).
- Grange, C., Bussolati, B. Extracellular vesicles in kidney disease. Nat Rev Nephrol. 18 (8), 499-513 (2022).
- Cocucci, E., Meldolesi, J. Ectosomes and exosomes: shedding the confusion between extracellular vesicles. Trends Cell Biol. 25 (6), 364-372 (2015).
- Iannotta, D. A. A., Kijas, A. W., Rowan, A. E., Wolfram, J. Entry and exit of extracellular vesicles to and from the blood circulation. Nat Nanotechnol. 19 (1), 13-20 (2024).
- Saint-Pol, J., Gosselet, F., Duban-Deweer, S., Pottiez, G., Karamanos, Y. Targeting and crossing the blood-brain barrier with extracellular vesicles. Cells. 9 (4), 851 (2020).
- Alberro, A., Iparraguirre, L., Fernandes, A., Otaegui, D. Extracellular vesicles in blood: Sources, effects, and applications. Int J Mol Sci. 22 (15), 8163 (2021).
- Nieuwland, R., Siljander, P. R. A beginner's guide to study extracellular vesicles in human blood plasma and serum. J Extracell Vesicles. 13 (1), e12400 (2024).
- Thangaraju, K., Neerukonda, S. N., Katneni, U., Buehler, P. W. Extracellular vesicles from red blood cells and their evolving roles in health, coagulopathy and therapy. Int J Mol Sci. 22 (1), 153 (2020).
- Li, C., et al. Cerebrospinal fluid-derived extracellular vesicles after spinal cord injury promote vascular regeneration via PI3K/AKT signaling pathway. J Orthop Translat. 39, 124-134 (2023).
- Deng, Y., et al. Phosphoproteome analysis of cerebrospinal fluid extracellular vesicles in primary central nervous system lymphoma. Analyst. 148 (15), 3594-3602 (2023).
- Hirschberg, Y., et al. Proteomic comparison between non-purified cerebrospinal fluid and cerebrospinal fluid-derived extracellular vesicles from patients with Alzheimer's, Parkinson's and Lewy body dementia. J Extracell Vesicles. 12 (12), e12383 (2023).
- Hefley, B. S., et al. Revealing the presence of tear extracellular vesicles in Keratoconus. Exp Eye Res. 224, 109242 (2022).
- Cross, T., et al. RNA profiles of tear fluid extracellular vesicles in patients with dry eye-related symptoms. Int J Mol Sci. 24 (20), 15390 (2023).
- Ma, H., et al. Metabolic signatures of tear extracellular vesicles caused by herpes simplex keratitis. Ocul Surf. 31, 21-30 (2024).
- Han, P., Bartold, P. M., Ivanovski, S. The emerging role of small extracellular vesicles in saliva and gingival crevicular fluid as diagnostics for periodontitis. J Periodontal Res. 57 (1), 219-231 (2022).
- Han, P., Li, X., Wei, W., Ivanovski, S. Saliva diagnosis using small extracellular vesicles and salivaomics. Methods Mol Biol. 2588, 25-39 (2023).
- Reseco, L., et al. Characterization of extracellular vesicles from human saliva: Effects of age and isolation techniques. Cells. 13 (1), 95 (2024).
- Tiwari, S., Kumar, V., Randhawa, S., Verma, S. K. Preparation and characterization of extracellular vesicles. Am J Reprod Immunol. 85 (2), e13367 (2021).
- Tomiyama, E., Fujita, K., Nonomura, N. Urinary extracellular vesicles: Ultracentrifugation method. Methods Mol Biol. 2292, 173-181 (2021).
- Xu, K., Jin, Y., Li, Y., Huang, Y., Zhao, R. Recent progress of exosome isolation and peptide recognition-guided strategies for exosome research. Front Chem. 10, 844124 (2022).
- Brennan, K., et al. A comparison of methods for the isolation and separation of extracellular vesicles from protein and lipid particles in human serum. Sci Rep. 10 (1), 1039 (2020).
- Gao, J., et al. Recent developments in isolating methods for exosomes. Front Bioeng Biotechnol. 10, 1100892 (2022).
- Mol, E. A., Goumans, M. J., Doevendans, P. A., Sluijter, J. P. G., Vader, P. Higher functionality of extracellular vesicles isolated using size-exclusion chromatography compared to ultracentrifugation. Nanomedicine. 13 (6), 2061-2065 (2017).
- Ansari, F. J., et al. Comparison of the efficiency of ultrafiltration, precipitation, and ultracentrifugation methods for exosome isolation. Biochem Biophys Rep. 38, 101668 (2024).
- Patel, G. K., et al. Comparative analysis of exosome isolation methods using culture supernatant for optimum yield, purity and downstream applications. Sci Rep. 9 (1), 5335 (2019).
- Stam, J., Bartel, S., Bischoff, R., Wolters, J. C. Isolation of extracellular vesicles with combined enrichment methods. J Chromatogr B Analyt Technol Biomed Life Sci. 1169, 122604 (2021).
- Le Roy, A., et al. AUC and small-angle scattering for membrane proteins. Methods Enzymol. 562, 257-286 (2015).
- Chhoy, P., Brown, C. W., Amante, J. J., Mercurio, A. M. Protocol for the separation of extracellular vesicles by ultracentrifugation from in vitro cell culture models. STAR Protoc. 2 (1), 100303 (2021).
- McDorman, K. S., Chan, C., Rojko, J., Satterwhite, C. M., Morrison, J. P. . Chapter 7 - Special Techniques in Toxicologic Pathology. Haschek and Rousseaux's Handbook of Toxicologic Pathology (Third Edition). , (2013).
- Woud, W. W., et al. An imaging flow cytometry-based methodology for the analysis of single extracellular vesicles in unprocessed human plasma. Commun Biol. 5 (1), 633 (2022).
- Gul, B., Syed, F., Khan, S., Iqbal, A., Ahmad, I. Characterization of extracellular vesicles by flow cytometry: Challenges and promises. Micron. 161, 103341 (2022).
- Black, C. B., Duensing, T. D., Trinkle, L. S., Dunlay, R. T. Cell-based screening using high-throughput flow cytometry. Assay Drug Dev Technol. 9 (1), 13-20 (2011).
- Safford, H. R., Bischel, H. N. Flow cytometry applications in water treatment, distribution, and reuse: A review. Water Res. 151, 110-133 (2019).
- Kobayashi, H., et al. Precise analysis of single small extracellular vesicles using flow cytometry. Sci Rep. 14 (1), 7465 (2024).
- Welsh, J. A., et al. A compendium of single extracellular vesicle flow cytometry. J Extracell Vesicles. 12 (2), e12299 (2023).
- Carnino, J. M., Lee, H. Extracellular vesicles in respiratory disease. Adv Clin Chem. 108, 105-127 (2022).
- Sun, Y., Sethu, P. Low-stress microfluidic density-gradient centrifugation for blood cell sorting. Biomed Microdevices. 20 (3), 77 (2018).
- Strachan, B. C., Xia, H., Voros, E., Gifford, S. C., Shevkoplyas, S. S. Improved expansion of T cells in culture when isolated with an equipment-free, high-throughput, flow-through microfluidic module versus traditional density gradient centrifugation. Cytotherapy. 21 (2), 234-245 (2019).
- Malvezzi, H., Sharma, R., Agarwal, A., Abuzenadah, A. M., Abu-Elmagd, M. Sperm quality after density gradient centrifugation with three commercially available media: a controlled trial. Reprod Biol Endocrinol. 12, 121 (2014).
- Sun, Y., Sethu, P. Microfluidic adaptation of density-gradient centrifugation for isolation of particles and cells. Bioengineering (Basel). 4 (3), 67 (2017).
- Karttunen, J., et al. Precipitation-based extracellular vesicle isolation from rat plasma co-precipitate vesicle-free microRNAs. J Extracell Vesicles. 8 (1), 1555410 (2019).
- De Sousa, K. P., et al. Isolation and characterization of extracellular vesicles and future directions in diagnosis and therapy. Wiley Interdiscip Rev Nanomed Nanobiotechnol. 15 (1), e1835 (2023).
- Sidhom, K., Obi, P. O., Saleem, A. A review of exosomal isolation methods: Is size exclusion chromatography the best option. Int J Mol Sci. 21 (18), 6466 (2020).
- Coughlan, C., et al. Exosome isolation by ultracentrifugation and precipitation and techniques for downstream analyses. Curr Protoc Cell Biol. 88 (1), e110 (2020).
- Konoshenko, M. Y., et al. Isolation of extracellular vesicles from biological fluids via the aggregation-precipitation approach for downstream miRNAs detection. Diagnostics (Basel). 11 (3), 384 (2021).
- Drozdz, A., et al. Low-vacuum filtration as an alternative extracellular vesicle concentration method: A comparison with ultracentrifugation and differential centrifugation. Pharmaceutics. 12 (9), 872 (2020).
- Akbar, A., Malekian, F., Baghban, N., Kodam, S. P., Ullah, M. Methodologies to isolate and purify clinical grade extracellular vesicles for medical applications. Cells. 11 (2), 186 (2022).
- Chen, J., et al. Review on strategies and technologies for exosome isolation and purification. Front Bioeng Biotechnol. 9, 811971 (2021).
- Helling, A., et al. Investigation of microbial cell deformability by filter cake compressibility using ultrafiltration membranes. Colloids Surf B Biointerfaces. 185, 110626 (2020).
- Bagci, C., et al. Overview of extracellular vesicle characterization techniques and introduction to combined reflectance and fluorescence confocal microscopy to distinguish extracellular vesicle subpopulations. Neurophotonics. 9 (2), 021903 (2022).
- Thery, C., et al. Minimal information for studies of extracellular vesicles 2018 (MISEV2018): a position statement of the International Society for Extracellular Vesicles and update of the MISEV2014 guidelines. J Extracell Vesicles. 7 (1), 1535750 (2018).
- van Maanen, J. C., et al. A combined western and bead-based multiplex platform to characterize extracellular vesicles. Tissue Eng Part C Methods. 29 (11), 493-504 (2023).
- Xu, D., et al. MicroRNAs in extracellular vesicles: Sorting mechanisms, diagnostic value, isolation, and detection technology. Front Bioeng Biotechnol. 10, 948959 (2022).
- Lee, H., He, X., Le, T., Carnino, J. M., Jin, Y. Single-step RT-qPCR for detection of extracellular vesicle microRNAs in vivo: a time- and cost-effective method. Am J Physiol Lung Cell Mol Physiol. 318 (4), L742-L749 (2020).
- Kim, J. A., et al. Small RNA sequencing of circulating small extracellular vesicles microRNAs in patients with amyotrophic lateral sclerosis. Sci Rep. 13 (1), 5528 (2023).
- Fan, Y., et al. Differential proteomics argues against a general role for CD9, CD81 or CD63 in the sorting of proteins into extracellular vesicles. J Extracell Vesicles. 12 (8), e12352 (2023).
- Silva, A. M., et al. Quantification of protein cargo loading into engineered extracellular vesicles at single-vesicle and single-molecule resolution. J Extracell Vesicles. 10 (10), e12130 (2021).
- Salunkhe, S., Basak Dheeraj, M., Chitkara, D., Mittal, A. Surface functionalization of exosomes for target-specific delivery and in vivo imaging, tracking: Strategies and significance. J Control Release. 326, 599-614 (2020).
- Silva, A. K. A., et al. Development of extracellular vesicle-based medicinal products: A position paper of the group "Extracellular Vesicle translatiOn to clinicaL perspectiVEs - EVOLVE France". Adv Drug Deliv Rev. 179, 114001 (2021).
- Gurunathan, S., Kang, M. H., Jeyaraj, M., Qasim, M., Kim, J. H. Review of the isolation, characterization, biological function, and multifarious therapeutic approaches of exosomes. Cells. 8 (4), 307 (2019).
- Zhang, Y., et al. Exosome: A review of its classification, isolation techniques, storage, diagnostic and targeted therapy applications. Int J Nanomedicine. 15, 6917-6934 (2020).
- Colombo, M., Raposo, G., Thery, C. Biogenesis, secretion, and intercellular interactions of exosomes and other extracellular vesicles. Annu Rev Cell Dev Biol. 30, 255-289 (2014).
- Pisitkun, T., Shen, R. F., Knepper, M. A. Identification and proteomic profiling of exosomes in human urine. Proc Natl Acad Sci U S A. 101 (36), 13368-13373 (2004).
- Zaborowski, M. P., Balaj, L., Breakefield, X. O., Lai, C. P. Extracellular vesicles: Composition, biological relevance, and methods of study. Bioscience. 65 (8), 783-797 (2015).
- Kowkabany, G., Bao, Y. Nanoparticle tracking analysis: An effective tool to characterize extracellular vesicles. Molecules. 29 (19), 4672 (2024).
- Li, H. Metastatic characteristics of SY86B human gastric carcinoma in athymic nude mice. Zhonghua Zhong Liu Za Zhi. 10 (6), 421-423 (1988).
- Zhao, Z., Wijerathne, H., Godwin, A. K., Soper, S. A. Isolation and analysis methods of extracellular vesicles (EVs). Extracell Vesicles Circ Nucl Acids. 2, 80-103 (2021).
- Gandham, S., et al. Technologies and standardization in research on extracellular vesicles. Trends Biotechnol. 38 (10), 1066-1098 (2020).
- van der Vlist, E. J., Nolte-'t Hoen, E. N., Stoorvogel, W., Arkesteijn, G. J., Wauben, M. H. Fluorescent labeling of nano-sized vesicles released by cells and subsequent quantitative and qualitative analysis by high-resolution flow cytometry. Nat Protoc. 7 (7), 1311-1326 (2012).
- Pospichalova, V., et al. Simplified protocol for flow cytometry analysis of fluorescently labeled exosomes and microvesicles using dedicated flow cytometer. J Extracell Vesicles. 4, 25530 (2015).
- Breitwieser, K., et al. Detailed characterization of small extracellular vesicles from different cell types based on tetraspanin composition by ExoView R100 platform. Int J Mol Sci. 23 (15), 8544 (2022).
- An, H. J., Cho, H. K., Song, D. H., Kee, C. Quantitative analysis of exosomes in the aqueous humor of Korean patients with pseudoexfoliation glaucoma. Sci Rep. 12 (1), 12875 (2022).
- Hefley, B. S., McKay, T. B., Hutcheon, A. E. K., Ciolino, J. B., Karamichos, D. Corneal epithelial-stromal constructs to study differences associated with diabetes mellitus. Exp Eye Res. 248, 110100 (2024).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten