Method Article
Células de crista neural craniana tridimensionais in vitro protocolo de diferenciação para ensaio multiplexado
Neste Artigo
Resumo
Apresentamos um protocolo de diferenciação in vitro tridimensional (3D) gerando neuroesferas de tamanho reprodutível para produzir células da crista neural craniana a partir de células-tronco embrionárias de camundongos. Mostramos que essa metodologia reduz a variabilidade em comparação com os protocolos anteriores e como ela pode ser usada para ensaios multiplexados para estudar o desenvolvimento das células da crista neural craniana.
Resumo
Com sua notável capacidade de gerar derivados ectodérmicos e mesenquimais, as células da crista neural craniana (CNCC) têm atraído muito interesse em estudar os mecanismos que regulam as decisões e a plasticidade do destino celular. Originada no neuroepitélio dorsal, essa população de células é transitória e relativamente rara no embrião em desenvolvimento - tornando os testes funcionais, telas genômicas e ensaios bioquímicos difíceis de realizar in vivo. Para superar essas limitações, vários métodos foram desenvolvidos para modelar o desenvolvimento de CNCC in vitro. Os métodos de cultura baseados em neuroesferas (NS) fornecem um microambiente complexo que recapitula o neuroepitélio anterior em desenvolvimento em 3D. Esses sistemas permitem o crescimento de muitos NS na mesma placa para gerar uma grande quantidade de CNCC, mas os NS produzidos apresentam uma alta variabilidade na forma, tamanho e número de CNCC formados - dificultando a realização de ensaios quantitativos. Este protocolo descreve um método reprodutível para gerar NS a partir de células-tronco embrionárias de camundongo (mESC) em um formato de 96 poços. O NS gerado em placas de 96 poços produz células da crista neural craniana (CNCC), que podem ser cultivadas posteriormente. Ao controlar o número de células iniciais, essa abordagem reduz a variabilidade no tamanho e na forma entre os NS e aumenta a reprodutibilidade entre os experimentos. Finalmente, este sistema de cultura é adaptável a várias aplicações e oferece um maior grau de flexibilidade, tornando-o altamente personalizável e adequado para multiplexação de condições experimentais.
Introdução
As células da crista neural craniana (CNCC) são uma população de células-tronco que surge na parte mais anterior do embrião em desenvolvimento, na fronteira entre a placa neural e o ectoderma de superfície1. O CNCC então sofre uma transição epitelial para mesenquimal (EMT), delamina do neuroepitélio e migra dorsoventralmente para vários locais do embrião, onde se diferenciam em uma ampla variedade de tipos de células2. O estudo dessa população celular é de grande interesse, pois possui uma plasticidade notável3 e a capacidade única de se diferenciar em derivados ectodérmicos e mesenquimais, como ossos e cartilagens craniofaciais4. Embora os CNCC sejam relativamente acessíveis no embrião, eles são uma população transitória com baixo número de células, dificultando a realização de estudos mecanísticos sistêmicos in vivo. As linhagens celulares CNCC foram isoladas e caracterizadas nos últimos anos para superar essas limitações. Em particular, a linha celular O9-1 CNCC é uma ótima ferramenta para estudar o desenvolvimento migratório e pós-migratório da crista neural 5,6; no entanto, esta linhagem celular não permite o estudo dos eventos iniciais anteriores à migração que levam à indução e especificação da crista neural. Nesse sentido, houve desenvolvimentos significativos no desenvolvimento de protocolos de diferenciação in vitro para diferenciar CNCC em uma placa por meio do uso de estruturas 3D semelhantes ao neuroepitélio em desenvolvimento chamadas neuroesferas (NS)7,8- obtidas após a diferenciação de colônias de células-tronco embrionárias (ESC). Esses protocolos 3D produzem de forma robusta um grande número de CNCC, permitindo a realização de estudos mecanicistas bioquímicos e genômicos 9,10. Os SN são cultivados em placas de baixa inserção em meio suplementado com N2B27, juntamente com o fator de crescimento de fibroblastos (FGF) e o fator de crescimento epidérmico (EGF)10,11 para estimular a proliferação celular. Esses protocolos são realizados em placas de Petri, cultivando-se numerosos NS na mesma placa. Dentro do NS em crescimento, as células se agregam e continuam a se dividir - atingindo um diâmetro de 100-200 μm na maturidade. Na maturidade (por volta do dia 5), os NS se ligam ao substrato e se diferenciam em CNCC semelhantes aos seus equivalentes in vivo 9,12. Esses CNCC então passam por EMT e delaminam na superfície da placa. Diferenças morfológicas podem ser observadas dependendo do tamanho do SN, pois esferas maiores parecerão mais escuras no núcleo devido à menor disponibilidade de nutrientes e oxigênio, levando as células a sofrerem apoptose13. Embora esse tipo de procedimento gere um grande número de CNCC no ponto final da diferenciação, ele apresenta várias limitações, tornando quase impossível o estudo das várias dinâmicas moleculares que ocorrem durante o processo de diferenciação. Primeiro, o uso de colônias ESC - que variam em tamanho - dificulta o controle do número inicial de células para cada experimento. Isso resulta na geração de NS de várias formas e diâmetros que se desenvolvem de maneira diferente, ativando vias de sinalização específicas, levando à diferenciação celular alterada e, portanto, não formando uma amostra uniforme em um determinado momento. Em segundo lugar, a cultura de vários NS na mesma placa geralmente leva à fusão14 e potencialmente à liberação de moléculas de sinalização que influenciam o microambiente de seus vizinhos e, portanto, seu desenvolvimento. Em conjunto, esses procedimentos geram muita variabilidade entre amostras e experimentos.
Aqui, apresentamos uma estratégia para superar essas dificuldades que geram NS único - capaz de produzir CNCC - agregando ESC de camundongo (mESC) em placas de 96 poços com fundo em U não tratadas com TC. Começar com mESC permite estudar o processo de especificação e os estágios iniciais do desenvolvimento do CNCC em comparação com a partir de linhagens celulares de crista neural já estabelecidas. Este protocolo começa com a desagregação de colônias de mESC para obter uma única suspensão celular, seguida pela semeadura de um número específico de mESC em cada poço de uma placa de 96 poços com fundo em U não tratada com TC. As células são deixadas para agregar por dois dias e, posteriormente, movidas para uma placa de 96 poços de fundo plano não tratada com TC, na qual o NS poderá se ligar ao fundo da placa. Ao controlar o número de células iniciais e o microambiente de cada SN durante o processo de diferenciação, este protocolo reduz a variabilidade da amostra, o que aumenta a reprodutibilidade experimental. Acreditamos que esta será uma plataforma conveniente para projetar experimentos multiplexados, como testar o efeito de diferentes condições de cultura ou realizar telas de perturbação genética.
Protocolo
1. Geração de uma suspensão unicelular a partir de colônias ESC de camundongos
NOTA: Este protocolo é adaptado para o uso de CK35 mESC (uma linha mESC competente para transmissão de linha germinativa, para ter então a opção de desenvolver modelos in vivo 15) cultivada em alimentadores inativados em uma placa de 6 poços tratada com TC revestida com gelatina. Um poço de uma placa de 6 poços tratada com TC deve render aproximadamente 1,5 × 106 mESC, o que é suficiente para o resto do protocolo. Isso pode ser ampliado, se necessário. Ajuste as etapas iniciais de acordo com a cepa ESC escolhida e o método de cultura de manutenção, bem como o meio de cultura adequado. Este protocolo deve ser realizado em condições estéreis. Consulte a Tabela de Materiais para obter detalhes relacionados a todos os materiais, reagentes e equipamentos usados neste protocolo.
- Comece com mESC em uma confluência de 70% a 80% cultivada em meio de cultura mESC. Consulte a Tabela 1 para a composição do meio de cultura mESC usada neste estudo.
NOTA: Não deixe o mESC crescer mais de 80% de confluência, pois eles começarão a se diferenciar e isso afetará o processo de agregação. As colônias devem ser compactas e mostrar uma morfologia saudável (sem rachaduras ou ondas, contraste citoplasmático nuclear distinto). - Prepare o meio de diferenciação CNCC. Consulte a Tabela 1 para a composição do meio de diferenciação CNCC.
NOTA: Uma vez adicionados os fatores de crescimento, o meio pode ser armazenado por até 3 semanas a 4 °C. Certifique-se de que o meio esteja protegido da luz. - Preparar uma solução de colagenase fresca a uma concentração de 2 mg/ml no meio DMEM-Knockout.
NOTA: O uso de colagenase garante que apenas as colônias sejam destacadas e não os alimentadores, pois sua presença nas etapas a seguir interferirá na agregação da SN. Filtre a solução de colagenase antes de usar com um filtro de 0,22 μm. - Aspirar o meio ESC de mESC.
- Adicione suavemente 1 mL de PBS na lateral do poço. Balance o prato suavemente para garantir uma lavagem uniforme.
- Remova o PBS e substitua por 2 mL de solução de colagenase. Incubar a 37 °C durante 30-45 min.
- Verifique a placa sob um microscópio óptico com ampliação de 10x após os primeiros 20 minutos e depois a cada 5 minutos.
- Quando as colônias mostrarem bordas enroladas, bata vigorosamente no lado da placa e as colônias se soltarão.
- Com uma pipeta sorológica de 5 mL, colete as colônias e transfira-as para um tubo cônico de 15 mL.
NOTA: Verifique se há colônias restantes na placa sob um microscópio óptico. Estes podem ser coletados com uma lavagem PBS. - Centrifugar as colónias a 16 × g durante 3 min à temperatura ambiente (RT).
- Aspire o máximo de meio possível do tubo cônico, tomando cuidado para não perturbar as colônias no fundo do tubo.
- Adicione 1 mL de solução de tripsina a 0,05% e incube o tubo a 37 °C por 5 min.
- Dissocie as colônias no tubo pipetando vigorosamente para cima e para baixo, primeiro com um p1000 e depois com uma micropipeta p200.
NOTA: Isso garante a obtenção de uma suspensão de célula única. - Adicione 2 mL de meio de cultura mESC para bloquear a tripsina e centrifugue a 160 × g por 3 min em RT. Remova o sobrenadante e adicione 1 mL de meio de diferenciação CNCC.
- Conte as células usando um dispositivo automatizado de contagem de células ou um sistema padronizado sob o microscópio, seguindo as instruções do fabricante.
- Após a contagem, diluir com meio de diferenciação CNCC suficiente para obter uma concentração de 3000 células vivas por 50 μL.
- Usando uma micropipeta p200, semeie 50 μL da suspensão celular em cada poço de uma placa de 96 poços de fundo em U não tratada com TC e, em seguida, encha o poço até 200 μL com meio de diferenciação CNCC.
NOTA: Ao encher a placa, ressuspenda de vez em quando a suspensão de célula única com uma micropipeta p1000 para obter uma concentração homogênea. - Incubar durante a noite em uma incubadora a 37 °C, 5% CO2.
2. Transferência para uma placa de fundo plano de 96 poços para diferenciação CNCC
- No dia seguinte (dia 1), observe a placa ao microscópio óptico. Certifique-se de que um pequeno aglomerado de células com bordas claras esteja visível na parte inferior de cada poço. Coloque a placa de volta na incubadora durante a noite.
NOTA: Pode haver algumas células ao redor do agregado principal, algumas mortas. Isso não interferirá na agregação NS. - No dia 2, remova lentamente 100 μL de meio de cada poço.
- Ao aspirar meio, tome cuidado para não remover o SN. Para evitar isso, coloque a ponta da pipeta perto da superfície e longe do fundo.
- Corte a ponta de uma micropipeta p200 a cerca de 3-4 mm da ponta. Use-o para aspirar o NS com o meio restante.
NOTA: Para facilitar a captação do NS, pipete suavemente para cima e para baixo algumas vezes antes de aspirar. - Transfira NS e o meio restante para uma placa de fundo plano de 96 poços não tratada com TC e verifique a transferência sob um microscópio óptico e, em seguida, cubra cada novo poço com 100 μL de meio de diferenciação CNCC pré-aquecido. Deixe NS na incubadora a 37 °C, 5% CO2, até o dia 4.
- No dia 4, remova 100 μL de meio de cada poço e substitua-o por 100 μL de meio de diferenciação CNCC pré-aquecido.
NOTA: Para evitar perturbar a fixação NS na parte inferior da placa, aspire lentamente e substitua o meio. - Nos dias 5 e 6, verifique o acessório NS sob o microscópio óptico. Certifique-se de que as células mais leves - delaminadas do NS - comecem a envolver o corpo principal do NS.
- No dia 7, troque o meio conforme descrito em 2.5. Troque o meio com o mesmo procedimento a cada 2 dias até o ponto final do estudo.
3. Passagem e manutenção do CNCC
NOTA: A passagem do CNCC pode ser realizada assim que houver uma quantidade suficiente de células visíveis ao redor do NS. Isso pode ser já no dia 7, pois os pontos de tempo anteriores não fornecem uma quantidade suficiente de CNCC.
- Prepare o meio de manutenção CNCC. Consulte a Tabela 1 para a composição do meio de manutenção CNCC.
- Antes de adicioná-lo ao meio, filtre o BSA após a solubilização através de um filtro de 0,22 μm. Uma vez adicionados os fatores de crescimento, conservar o meio até 3 semanas a 4 °C. Certifique-se de que o meio esteja protegido da luz.
- Prepare fibronectina a 7,5 μg / mL em PBS. Misture vigorosamente.
- Cubra os poços de uma placa de 96 poços não tratada com TC adicionando 100 μL de solução de fibronectina por poço. Deixe revestir sob o capuz por 30 min em RT.
NOTA: Se o CNCC for destinado à coloração por imunofluorescência, coloque uma lamínula de vidro estéril no fundo do poço antes do revestimento. Certifique-se de que a lâmina fique no fundo do poço e não flutue para evitar revestir o lado oposto ao que será usado. Siga as instruções na seção 5 para fixação e montagem do CNCC. - Enquanto isso, na placa de 96 poços de fundo plano não tratada com TC, aspire o máximo possível de meio de diferenciação CNCC dos poços e substitua por 50 μL de accutase. Incubar a 37 °C durante 5 min.
- Após a incubação, adicione 100 μL de meio de manutenção CNCC por poço para extinguir a accutase. Remova a fibronectina dos poços da placa de 96 poços de fundo plano não tratada com TC receptor. Filtrar o CNCC pós-migratório destacado passando-o através de um filtro de 40 μm no topo do poço da placa de 96 poços de fundo plano não tratada com TC receptor.
NOTA: Isso filtrará aglomerados de células ou NS restantes que não foram removidos anteriormente. O CNCC cultivado na mesma condição pode ser agrupado em um tubo de 50 mL para ser semeado no mesmo poço de uma placa de 6 poços tratada com TC. Neste caso, cubra o poço com 1 mL de fibronectina e use 1 mL de meio de manutenção CNCC para extinguir a accutase. - Deixe o CNCC pós-migratório se conectar por 15-30 min a 37 ° C. Descarte o meio e substitua por 100 μL de meio de manutenção CNCC. Troque o meio a cada 2 dias.
NOTA: Adicione suavemente o meio, pois o fluxo rápido induz a diferenciação de CNCC em derivados neurais. Se estiver trabalhando em um formato de 6 poços, use 1 mL de meio de manutenção CNCC.
4. Fixação e montagem NS para imunofluorescência
- No momento desejado, transfira o NS da placa de fundo plano de 96 poços não tratada com TC para um tubo de 2 mL de baixa ligação de DNA cortando a ponta de uma micropipeta p200 e pipete suavemente para cima e para baixo para pegar o NS.
NOTA: Em momentos posteriores (dia 7 em diante), o SN será grande o suficiente para ser visto a olho nu, mas também será mais difícil de destacar. - Deixe o NS repousar no tubo por 3 min em RT, remova o máximo de meio possível e enxágue com 1 mL de PBS frio.
NOTA: Se transferir NS pequenos de pontos de tempo iniciais (antes dos dias 3-4), gire a 16 × g por 3 min para garantir que o NS se acomode no fundo. - Remova o PBS e, em uma capa química, substitua por 2 mL de PFA a 4% em PBS. Incubar por 20 min em RT.
- Inverta o tubo lentamente após adicionar a solução de PFA a 4% e deixe-o descansar durante a fixação na lateral. O objetivo é espalhar ligeiramente o NS para que eles não fiquem juntos nesta etapa.
- Remover a solução de PFA a 4% (na coifa química) e lavar a SN com 1 mL de PBS frio/Tween 0,5%20. Deixe repousar em RT por 3 min. Repita isso 3 vezes no total.
NOTA: NS se acomodará na parte inferior. - Remova PBS / 0,5% Tween20 e adicione 2 mL de PBS / 0,1% Triton X-100. Inverta o tubo e deixe-o descansar de lado. Incubar por 1 h em RT.
- Lave NS 3 vezes em PBS frio/0,5% Tween20, conforme indicado na etapa 4.4.
- Remova PBS / 0,5% Tween20 e bloqueie com 2% BSA em PBS a 4 ° C por um mínimo de 1 h, de preferência durante a noite.
NOTA: As amostras podem ser armazenadas em 2% BSA/PBS por até 1 semana a 4 °C. Proteja da luz. - Preparar a solução de anticorpos primários adicionando a diluição correcta do anticorpo primário a 2% de BSA/PBS num volume final de 500 μL. Para a mistura de anticorpos primários usada neste estudo, consulte a Tabela 2.
- Remova o máximo possível da solução de bloqueio e transfira o NS para um tubo de 0,5 mL cortando a ponta de uma micropipeta p200.
- Adicione a solução de anticorpo primário e pipete para cima e para baixo lentamente para ressuspender o NS. Incubar durante a noite num rotador a 4 °C.
- Lave NS 3 vezes em PBS frio por 5 min em RT.
- Prepare a solução de anticorpos secundários diluindo os anticorpos secundários de escolha em 2% de BSA/PBS e adicionando 1/1000 de DAPI. Para a mistura de anticorpos secundários usada neste estudo, consulte a Tabela 2.
NOTA: Mantenha os tubos protegidos da luz. - Substitua o PBS por 500 μL de solução de anticorpo secundário e envolva o tubo com papel alumínio para protegê-lo da luz. Incubar durante 1 h no rotador à RT.
- Lave NS 3 vezes em PBS frio, conforme indicado na etapa 4.11.
- Remova o máximo de PBS possível sem aspirar o NS, substitua por 50 μL de agente de limpeza e siga as instruções do fabricante.
- Incubar durante a noite em RT, protegido da luz.
- Prepare as câmaras de montagem.
- Em uma lâmina de microscópio, coloque três camadas de fita dupla-face. Use fita transparente e sem fibras para evitar resíduos na câmara de montagem.
- Usando uma navalha, corte uma janela de 3 mm × 8 mm na fita dupla.
NOTA: As dimensões da câmara dependem do ponto de tempo da amostra. Este exemplo é adequado para hospedar 50 μL de meio de montagem, o que é ideal para 10-20 NS únicos em pontos de tempo posteriores (dias 7-9).
- Sob um estereoscópio, transfira cuidadosamente o NS no agente de limpeza para a câmara de montagem, cortando uma ponta de micropipeta p200 de baixa aderência. Use uma ampliação entre 2x e 4x para fornecer um campo de visão de toda a câmara e ampliação suficiente para identificar o SN. Se o estereoscópio for equipado para imagens fluorescentes, trabalhar com um filtro de fluorescência azul facilitará a identificação do SN, pois a coloração DAPI fará com que o NS transparente se destaque.
NOTA: Verifique se o meio forma um leve menisco convexo acima da câmara. Isso garantirá que não haja bolhas na câmara. - Coloque uma lamínula na superfície e pressione levemente nas laterais para que ela adere. Execute esta etapa sob o estereoscópio e verifique se os NS não são empurrados para fora da câmara.
- Conservar a 4 °C protegido da luz até à obtenção de imagens.
5. Fixação e montagem CNCC para imunofluorescência
- Remova o meio de manutenção CNCC dos poços e lave-os com PBS. Adicione PBS suavemente pipetando na lateral do poço.
- Prossiga com a fixação, permeabilização e coloração conforme descrito nas etapas 4.3-4.14.
- Monte as lamínulas em uma lâmina de vidro de microscopia adicionando meio de montagem na lâmina, agarrando e girando a lamínula com o uso de uma pinça para que o CNCC pós-migratório fique de frente para a lâmina de vidro e colocando-a na gota.
- Opcional: Sele a lamínula com esmalte ou outros sistemas de vedação comumente empregados.
- Conservar a 4 °C protegido da luz até à obtenção de imagens.
Resultados
Seguindo o protocolo, as colônias de mESC foram dissociadas e 3000 células foram semeadas em placas de 96 poços não tratadas com TC. No dia 2, os NS agregados foram transferidos para placas de 96 poços de fundo plano não tratadas com TC para permitir que se fixassem. Uma visualização simplificada do protocolo de agregação NS é fornecida na Figura 1A. Os NS foram cultivados até o dia 9 e depois processados para coloração por imunofluorescência. As células que migraram do SN para a placa foram transferidas para lamínulas para imagem e cultivadas até o dia 13 no meio de manutenção CNCC. Paralelamente, o protocolo para geração de NS em placa de Petri foi realizado como referência e ponto de comparação. O objetivo geral do experimento foi reduzir a variabilidade NS-para-NS observada no protocolo realizado na placa de Petri.
Quando cultivado individualmente em placas de 96 poços não tratadas com TC, o NS mostrou variabilidade fenotípica reduzida. Todos os SN começam a se fixar no dia 5, enquanto a proporção de SN aderido foi muito variável e, em geral, menos importante no dia 7 quando cultivada em uma placa de Petri (Figura 1B, C). Além disso, quando cultivada em uma placa de Petri, a fusão NS é consistentemente observada, como pode ser observado no dia 4 (Figura 1B). No dia 9, a delaminação celular extensa de NS pode ser observada em placas de 96 poços não tratadas com TC e condições de cultura de placas de Petri ( Figura 1B , C ). Os NS foram coletados no dia 4 e no dia 7 para comparar a variabilidade de tamanho entre os protocolos. NS cultivado em placa de 96 poços não tratada com TC mostra uma redução significativa na variabilidade de tamanho no dia 4 e no dia 7. Em comparação, quando cultivado em placa de Petri, os NS exibem uma importante variabilidade de tamanho (Figura 2A). Na placa de 96 poços não tratada com TC, o diâmetro do NS do dia 4 varia entre 139 μm e 295 μm e entre 383 μm e 552 μm no dia 7, enquanto na placa de Petri, o diâmetro do NS do dia 4 varia entre 126 μm e 505 μm e de 106 μm a 868 μm no dia 7. NS cultivados em placa de 96 poços não tratados com TC foram coletados todos os dias do dia 2 ao dia 9, e seu crescimento foi medido. O número de células aumenta exponencialmente, de uma média de 1082 células no dia 2 e uma média de 48352 células no dia 9 (Figura 2B), enquanto o tamanho do diâmetro NS segue um crescimento linear, com um diâmetro médio de 136 μm no dia 2 e um diâmetro médio de 570 μm no dia 9 (Figura 2C).
Após a cultura na placa de 96 poços não tratada com TC, NS e células delaminadas foram coradas para os marcadores CNCC AP2α, PAX7 e SOX916, e TWIST1, um marcador EMT16. Uma visualização esquemática do protocolo de coleta, coloração e montagem da amostra é mostrada na Figura 3. Uma imagem representativa da câmara de montagem para análise de imunofluorescência NS é mostrada na Figura 4A. Esta análise teve como objetivo verificar se as NS cultivadas em placas de 96 poços não TC geram CNCC que sofrem EMT e que as células delaminantes apresentam uma identidade CNCC. Tanto o SN quanto o CNCC pós-migratório mostraram extensa expressão de AP2α, SOX9 e TWIST1 nos dias 9 e 13, enquanto o PAX7 - um marcador CNCC pré-migratório16 - está presente apenas no SN e não no CNCC pós-migratório (Figura 4C). Juntos, esses resultados confirmam que o protocolo de cultura NS em placa de 96 poços não tratado com TC gera CNCC que sofre EMT e migra na superfície da placa. O SN do dia 9 e o CNCC pós-migratório do dia 13 também foram corados para o marcador neuronal TUJ117 (Figura 4B) para visualizar derivados neuronais no SN em desenvolvimento.
Por fim, a análise da expressão gênica de RT-qPCR foi realizada em mESC e CNCC pós-migração no dia 13 (Figura 4D). O RNA foi extraído usando Trizol e, em seguida, o RT-qPCR foi realizado usando as instruções do fabricante. No CNCC pós-migratório, a expressão dos marcadores de crista neural Ap2α, Sox9 e Sox1018 é aumentada em 5,42 vezes, 51,07 vezes e 46,75 vezes em comparação com mESC, respectivamente. A expressão dos marcadores progenitores neurais Eya2, Ascl1 e Neurog219 também aumentou no dia 13 do CNCC pós-migratório em 7,78 vezes, 43,92 vezes e 13,83 vezes em comparação com o mESC, respectivamente. Os marcadores mesenquimais/osteoblásticos Mef2c, Alx4 e Ets120mostraram expressão aumentada em CNCC pós-migratória em 168,44 vezes, 156,47 vezes e 41,23 vezes em comparação com mESC, respectivamente (sequências de primers na Tabela 3). Juntos, isso indica que o CNCC pós-migratório gerado neste protocolo não é tendencioso para um destino neuronal ou ectomesênquio.
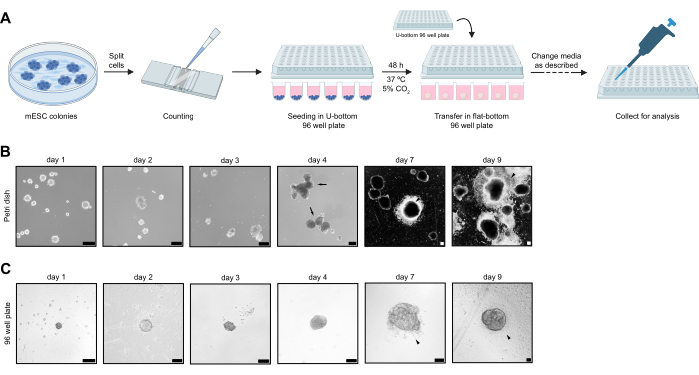
Figura 1: Fluxo de trabalho de agregação de NS e comparação fenotípica com NS cultivado em placa de Petri. (A) Procedimento de fluxo de trabalho de semeadura de mESC e cultura NS. (B) Imagens representativas da diferenciação NS realizadas em uma placa de Petri. SN de diferentes tamanhos pode ser observado desde os primeiros momentos. As setas indicam fusão NS. As pontas das setas brancas mostram células delaminadas da SN. (C) Imagens representativas durante a diferenciação de NS de curso de tempo realizada em placas de 96 poços não tratadas com TC. As pontas das setas pretas mostram células delaminadas da SN. Os dias são indicados na parte superior. As barras de escala representam 100 μm. Clique aqui para ver uma versão maior desta figura.
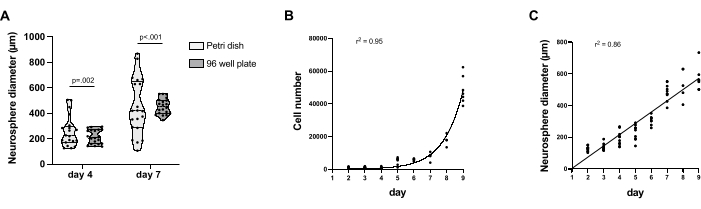
Figura 2: Caracterização do crescimento agregado de NS e comparação com NS cultivado em placa de Petri. (A) Gráfico de violino mostrando a variação do tamanho NS na placa de Petri e no protocolo de cultura em placas de 96 poços não tratado com TC. Foram coletados 20 NS para cada protocolo e momento. Foram realizados testes F para variabilidade de desvio padrão, e os resultados são mostrados nas barras acima do gráfico. (B) Quantificação do número de células NS durante a diferenciação em placas de 96 poços não tratadas com TC. (C) Quantificação da variação do tamanho do diâmetro NS durante a diferenciação em placa de 96 poços não tratada com TC. A contagem de células foi realizada usando o CellProfiler 4.2.5. A geração de gráficos e a análise de regressão foram realizadas usando o Graphpad Prism 9. Para cada ponto de tempo, 5 a 10 NS foram quantificados. Clique aqui para ver uma versão maior desta figura.

Figura 3: Fluxo de trabalho de coleta de NS e análise de imunofluorescência. Esquema representando a coleta de NS, preparação para coloração de imunofluorescência e imagem com intervalos de tempo indicativos para cada etapa indicada na parte superior. Clique aqui para ver uma versão maior desta figura.

Figura 4: NS cultivado em placa de 96 poços não tratada com TC gera CNCC. (A) Imagem representativa da lâmina de imagem (1) e do bisturi utilizado para cortar a câmara (2). (B) Imagens de imunofluorescência de NS no dia 9 (esquerda) e CNCC pós-migratória no dia 13 (direita) mostrando expressão do marcador de neurônio TUJ1. As setas brancas indicam corpos celulares. O quadrado tracejado indica as regiões de zoom mostradas nos painéis do meio. A barra de escala representa 100 μm. (C) Imagens de imunofluorescência de NS no dia 9 (superior) e CNCC pós-migratória no dia 13 (inferior) mostrando expressão do marcador de especificação CNCC AP2α, marcadores CNCC PAX7 e SOX9 e marcador EMT TWIST1. A barra de escala representa 100 μm. (D) Gráficos de barras de colunas comparando a expressão gênica relativa em mESC (em preto) e CNCC pós-migratória (em cinza). A expressão de genes marcadores CNCC (Ap2α, Sox9 e Sox10), neuronal (Eya2, Ascl1, Neurog2) e ectomesênquima (Mef2c, Alx4, Ets1) é comparada à expressão média dos genes housekeeping ActinB, Ywhaz e Tbp. As barras de erro representam os erros padrão calculados a partir de três réplicas técnicas (asterisco = p<0,05, asterisco duplo = p<0,01). Os gráficos foram gerados usando o GraphPad Prism 9. Clique aqui para ver uma versão maior desta figura.
Tabela 1: Composições de meios de cultura. Clique aqui para baixar esta tabela.
Tabela 2: Anticorpos primários e secundários utilizados no estudo. Clique aqui para baixar esta tabela.
Tabela 3: Sequências de primers. Clique aqui para baixar esta tabela.
Discussão
Modelos de diferenciação 3D in vitro permitem analisar interações celulares complexas que podem ser difíceis - ou não - ser observadas em cultura de células 2D. Vários modelos foram desenvolvidos para estudar o desenvolvimento do CNCC in vitro. Estes são geralmente derivados diretamente de colônias ESC 7,21 ou explantes de tecido22,23. Embora esses sistemas tenham se mostrado eficientes na geração de células da crista neural, tais métodos mostram sensibilidade significativa ao método de cultura e ao microambiente, o que leva a diferenças na composição celular e nas propriedades celulares dentro do NS24. Além disso, esses métodos não levam em conta a heterogeneidade inicial do tamanho da colônia ESC e do número de células.
Para resolver essas limitações, este artigo descreve um método para gerar NS único por meio da agregação mESC em placas de 96 poços não tratadas com TC. Este protocolo permite a padronização das condições iniciais, gerando uma suspensão unicelular a partir de colônias de mESC e controlando o número inicial de células no experimento. Isso leva a uma redução na variabilidade do tamanho do diâmetro NS e no número de células durante a diferenciação. Além disso, a geração de um único NS por poço revoga a fusão do NS e evita que o NS vizinho influencie o microambiente, liberando fatores extracelulares que não podem ser controlados. O uso de um formato de placa de 96 poços apresenta vantagens adicionais, como (i) avaliar a influência da condição inicial da cultura, como estados celulares ou número de células; (ii) testar um grande número de condições de cultura em um ambiente padronizado; (iii) realização de experimentos de imagem ao vivo; e (iv) projetar uma tela de perturbação genética de alto rendimento usando silenciamento gênico mediado por siRNA. É importante notar que este protocolo pode ser realizado com linhas ESC de camundongos e humanos 9,25. Juntos, eles permitirão investigar mais a fundo os mecanismos moleculares que controlam a plasticidade do CNCC e as decisões de destino celular.
Uma limitação desse protocolo é o número reduzido de SN produzidos, o que resulta em menos CNCC para a cultura seguinte. Embora o CNCC possa ser amplificado posteriormente10, isso limita o uso desse protocolo para procedimentos que requerem um grande número de células. Uma segunda desvantagem potencial é que a manipulação de NS requer mais destreza, e isso pode levar à perda de amostras - especialmente nos pontos de tempo anteriores - e, assim, reduzir ainda mais o tamanho da amostra. Isso pode ser superado aumentando o número de placas usadas.
Um aspecto crítico do protocolo é a geração de uma suspensão de célula única a partir de mESC. É fundamental que qualquer célula alimentadora usada para cultivar mESC seja removida antes da agregação, pois interfere na agregação e leva a fenótipos inesperados e resultados enganosos.
Em resumo, este método oferece uma maneira reprodutível, escalável e altamente modulável de produzir NS para uso em paralelo com estudos in vivo .
Divulgações
Os autores declaram não haver conflito de interesses.
Agradecimentos
Agradecemos ao Dr. Remi Xavier Coux pelos conselhos sobre o design de primers e experiência em cultura de células. Este trabalho foi apoiado pelo Conselho Europeu de Investigação (ERC Starting Grant 101039995 - REGENECREST) e pela Fondation pour la Recherche Médicale (Amorçage - AJE202205015403).
Materiais
| Name | Company | Catalog Number | Comments |
| 0.22 μm syringe filters | ClearLine | 146560 | |
| 15 mL High-Clarity Polypropylene Conical Tube | Falcon | 352096 | |
| 200 µL ClearLine Plus Low Binding Filter Tips | Dutscher | 713263 | |
| 40 µm filters | Falcon | 352340 | |
| 5 mL Serological pipette | Starstedt | 86.1253.001 | |
| 50 mL High-Clarity Polypropylene Conical Tube | Falcon | 352070 | |
| Accutase | Merck-Sigma | A6964 | |
| Alexa Fluor 488 donkey anti rabbit IgG (H+L) | Thermofisher Scientific | A21206 | |
| Alexa Fluor 594 donkey anti mouse IgG (H+L) | Thermofisher Scientific | A21203 | |
| Alexa Fluor 647 donkey anti goat IgG (H+L) | Thermofisher Scientific | A31571 | |
| Antibiotic-antimycotic solution | Merck-Sigma | A5955 | |
| B27 PLUS supplement | Thermofisher Scientific | 17504044 | |
| Bovine serum albumin (BSA) | Merck-Sigma | A9418 | |
| Chloroform | Carlo Erba | 438601 | |
| Collagenase Type IV | Thermofisher Scientific, Gibco | 17104019 | |
| Costar 6 well clear TC-treated multiple well plates | Corning | 3516 | |
| Cover glasses, round | VWR | 630-2113 | |
| DMEM KnockOut | Thermofisher Scientific | 10829018 | |
| DMEM/F12+Glutamax | Thermofisher Scientific | 10565018 | |
| DMEM high glucose | Merck-Sigma | D0822 | |
| DNA LoBind Tubes, 2 mL | Eppendorf | 30108078 | |
| DNase/RNase-Free Distilled Water | Thermofisher Scientific | 10977-035 | |
| Dulbecco’s Phosphate Buffered Saline (PBS) | Thermofisher Scientific | 14190144 | |
| Eppendorf Safe-Lock Tubes, 0.5 mL | Eppendorf | 30121023 | |
| Eppendorf Safe-Lock Tubes, 2 mL | Eppendorf | 30120094 | |
| ESGRO mLIF Medium Supplement | Merck-Sigma | ESG1107 | |
| Ethanol 70% | Carlo Erba | 528170 | |
| Fetal Bovine Serum | Merck-Sigma | F7524 | |
| Fibronectin | Merck-Sigma | F085-2MG | |
| Fluoromount-G | Invitrogen | 00-4958-02 | |
| Gelatin solution | Merck-Sigma | ES-006-B | |
| GlutaMAX | Thermofisher Scientific | 35050061 | |
| Human EGF | Peprotech | AF-100-15-500UG | |
| Human FGF-basic | Peprotech | 100-18B | |
| Human SOX9 Antibody | R&Dsystems | AF3075 | |
| Insulin from bovine pancreas | Merck-Sigma | I6634 | |
| iScript cDNA Synthesis Kit | Biorad | 1708891 | |
| Mouse Anti-Human AP-2 alpha Monoclonal Antibody, Unconjugated | DSHB | 3B5 | |
| Mouse Anti-Human PAX7 Monoclonal Antibody, Unconjugated | DSHB | PAX7 | |
| N2 supplement | Thermofisher Scientific | 17502048 | |
| Neurobasal Medium | Thermofisher Scientific | 21103049 | |
| Non-Tissue culture treated plate, 96 well, Flat bottom | Falcon | 351172 | |
| Non-Tissue culture treated plate, 96 well, U-bottom | Falcon | 351177 | |
| Paraformaldehyde 16% solution, em grade | Electron Microscopy Sciences | 15710 | |
| Propan-2-ol | Carlo Erba | 415154 | |
| Purified anti-Tubulin β 3 (TUJ1) Antibody | Biolegend | MMS-435P | |
| RapiClear 1.47 | Sunjin Lab | RC147001 | |
| RapiClear 1.52 | Sunjin Lab | RC152001 | |
| Scotch Double Sided 12.7 mm × 22.8 m | Clear fibreless double sided tape | ||
| SensiFAST SYBR No-ROX Kit | Meridian Bioscience | BIO-98020 | |
| Sterile Disposable Surgical Scalpels | Swann-Morton | 05XX | |
| Superfrost Plus Adhesion Microscope Slides | Epredia | J1800AMNZ | |
| Triton X-100 | Thermofisher Scientific | A16046.AP | |
| TRIzol Reagent | FisherScientific | 15596026 | |
| Trypsine-EDTA (0.05%) | Thermofisher Scientific | 25300054 | |
| Tween-20 | Fisher Scientific | 10113103 | |
| TWIST1 Rabbit mAb (IF Formulated) | Cell signaling technology | E7E2G | |
| β-mercaptoethanol | Thermofisher Scientific | 31350010 |
Referências
- Rothstein, M., Bhattacharya, D., Simoes-Costa, M. The molecular basis of neural crest axial identity. Dev Biol. 444, S170-S180 (2018).
- Smeriglio, P., Zalc, A. Cranial neural crest cells contribution to craniofacial bone development and regeneration. Curr Osteoporos Rep. 21 (5), 624-630 (2023).
- Zalc, A., et al. Reactivation of the pluripotency program precedes formation of the cranial neural crest. Science. 371 (6529), eabb4776 (2021).
- Perera, S. N., Kerosuo, L. On the road again: Establishment and maintenance of stemness in the neural crest from embryo to adulthood. Stem Cells. 39 (1), 7-25 (2021).
- Nguyen, B. H., Ishii, M., Maxson, R. E., Wang, J. Culturing and manipulation of O9-1 neural crest cells. J Vis Exp. (140), e58346 (2018).
- Ishii, M., Arias, A. C., Liu, L., Chen, Y. B., Bronner, M. E., Maxson, R. E. A stable cranial neural crest cell line from mouse. Stem Cells Dev. 21 (17), 3069-3080 (2012).
- Rada-Iglesias, A., Bajpai, R., Prescott, S., Brugmann, S. A., Swigut, T., Wysocka, J. Epigenomic annotation of enhancers predicts transcriptional regulators of human neural crest. Cell Stem Cell. 11 (5), 633-648 (2012).
- Cederquist, G. Y., et al. Specification of positional identity in forebrain organoids. Nat Biotechnol. 37 (4), 436-444 (2011).
- Rada-Iglesias, A., Bajpai, R., Swigut, T., Brugmann, S. A., Flynn, R. A., Wysocka, J. A unique chromatin signature uncovers early developmental enhancers in humans. Nature. 470 (7333), 279-283 (2011).
- Prescott, S. L., et al. Enhancer divergence and cis-regulatory evolution in the human and chimp neural crest. Cell. 163 (1), 68-83 (2015).
- Chaddah, R., Arntfield, M., Runciman, S., Clarke, L., van der Kooy, D. Clonal neural stem cells from human embryonic stem cell colonies. J Neurosci. 32 (23), 7771-7781 (2012).
- Bajpai, R., et al. CHD7 cooperates with PBAF to control multipotent neural crest formation. Nature. 463 (7283), 958-962 (2010).
- Sipahi, R., Zupanc, G. K. Stochastic cellular automata model of neurosphere growth: Roles of proliferative potential, contact inhibition, cell death, and phagocytosis. J Theor Biol. 445, 151-165 (2018).
- Mori, H., et al. Effect of neurosphere size on the growth rate of human neural stem/progenitor cells. J Neurosci Res. 84 (8), 1682-1691 (2006).
- Kress, C., Vandormael-Pournin, S., Baldacci, P., Cohen-Tannoudji, M., Babinet, C. Nonpermissiveness for mouse embryonic stem (ES) cell derivation circumvented by a single backcross to 129/Sv strain: establishment of ES cell lines bearing the Omd conditional lethal mutation. Mamm Genome. 9 (12), 998-1001 (1998).
- Simões-Costa, M., Bronner, M. E. Establishing neural crest identity: a gene regulatory recipe. Development. 142 (2), 242-257 (2015).
- Memberg, S. P., Hall, A. K. Dividing neuron precursors express neuron-specific tubulin. J Neurobiol. 27 (1), 26-43 (1995).
- Kim, C. N., Shin, D., Wang, A., Nowakowski, T. J. Spatiotemporal molecular dynamics of the developing human thalamus. Science. 382, eadf9941 (2023).
- Grimaldi, A., Comai, G., Mella, S., Tajbakhsh, S. Identification of bipotent progenitors that give rise to myogenic and connective tissues in mouse. ELife. 11, e70235 (2022).
- To, K., et al. A multiomic atlas of human early skeletal development. Nature. 635 (8039), 657-667 (2024).
- Bajpai, R., et al. Molecular stages of rapid and uniform neuralization of human embryonic stem cells. Cell Death Differ. 16 (6), 807-825 (2009).
- Kerosuo, L., Nie, S., Bajpai, R., Bronner, M. E. Crestospheres, Long-term maintenance of multipotent, premigratory neural crest stem cells. Stem Cell Reports. 5 (4), 499-507 (2015).
- Abe, S., Yamaguchi, S., Sato, Y., Harada, K. Sphere-derived multipotent progenitor cells obtained from human oral mucosa are enriched in neural crest cells. Stem Cells Transl Med. 5 (1), 117-128 (2016).
- Jensen, J. B., Parmar, M. Strengths and limitations of the neurosphere culture system. Mol Neurobiol. 34 (3), 153-161 (2006).
- Ziegler, L., Grigoryan, S., Yang, I. H., Thakor, N. V., Goldstein, R. S. Efficient generation of Schwann cells from human embryonic stem cell-derived neurospheres. Stem Cell Rev Rep. 7 (2), 394-403 (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados