Method Article
Visualizando e analisando o espaço químico de bancos de dados de produtos naturais para descoberta de medicamentos
Neste Artigo
Resumo
Aqui, fornecemos uma metodologia que usa diferentes representações moleculares para exibir e analisar o espaço químico de conjuntos de dados de compostos naturais, com foco em aplicações relacionadas à descoberta de medicamentos.
Resumo
O espaço químico é um espaço descritor multidimensional que engloba todas as moléculas possíveis, e pelo menos 1 x 1060 substâncias orgânicas com peso molecular abaixo de 500 Da são consideradas potencialmente relevantes para a descoberta de medicamentos. Os produtos naturais têm sido a principal fonte das novas entidades farmacológicas comercializadas durante os últimos quarenta anos e continuam a ser uma das fontes mais produtivas para a criação de medicamentos inovadores. As ferramentas computacionais baseadas em quimioinformática aceleram o processo de desenvolvimento de medicamentos para produtos naturais. Métodos que incluem estimativa de bioatividades, perfis de segurança, ADME e medição de semelhança de produtos naturais têm sido usados. Aqui, examinamos os desenvolvimentos recentes em ferramentas quimioinformáticas projetadas para visualizar, caracterizar e expandir o espaço químico de conjuntos de dados de compostos naturais usando várias representações moleculares, criar representações visuais de tais espaços e investigar as relações estrutura-propriedade dentro de espaços químicos. Com ênfase em aplicações de descoberta de medicamentos, avaliamos as bases de dados de código aberto BIOFACQUIM e PeruNPDB como prova de conceito.
Introdução
Os produtos naturais (NPs), que são compostos químicos criados por seres vivos, têm sido utilizados como tratamentos tradicionais há séculos. NPs individuais foram criados como medicamentos na era moderna e explorados com sucesso como compostos principais na descoberta de medicamentos1. Substâncias marinhas, fúngicas, bacterianas, vegetais e endógenas criadas por humanos e animais estão incluídas na categoria de compostos bioativos, assim como venenos e venenos produzidos por vários animais2. Como resultado, por quarenta anos, o número de medicamentos produzidos pelos NPs representou uma fonte significativa de novas substâncias farmacológicas3, enfatizando que os NPs têm sido cruciais no desenvolvimento de novos medicamentos, particularmente para o tratamento de câncer e doenças infecciosas, bem como para outras condições terapêuticas como esclerose múltipla e doenças cardiovasculares4. Além disso, 64,9% dos 185 pequenos compostos autorizados para tratar o câncer entre 1981 e 2019 eram NPs não modificados ou medicamentos sintéticos com farmacoforo NP3.
A quimioinformática, uma interdisciplinaridade bem estabelecida que se baseia no conceito de espaço químico, tem sido usada para analisar e visualizar o espaço químico das qualidades físico-químicas das NPs ligadas a características semelhantes a drogas5. A quimioinformática mostrou um impacto substancial no design e descoberta de medicamentos com base em NPs6. O espaço químico de um grupo de compostos nem sempre é único. Dependerá da coleção de descritores usados para defini-lo, o que significa que estudar o espaço químico de NPs como qualquer outro conjunto de compostos apresenta desafios particulares que repousam na representação molecular7. Esse esforço pode ser abordado usando uma variedade de descritores moleculares e técnicas de visualização de dados. Em contraste, as técnicas mais utilizadas são análise de componentes principais (PCA), árvores de andaimes, mapas auto-organizáveis, mapeamento topográfico generativo (GTM) e uma nova técnica de visualização chamada mapas de árvore (TMAPs)8. Além disso, a coleta, avaliação e disseminação de informações químicas de NP em bancos de dados de compostos é um dos usos da quimioinformática na pesquisa de NP. Em contraste, com a introdução de big data, isso é especialmente pertinente9.
Aqui, os bancos de dados NP de código aberto BIOFACQUIM10 e PeruNPDB11 são usados para descrever o protocolo que busca a visualização e caracterização do espaço químico de conjuntos de dados de compostos naturais usando várias representações moleculares, cria representações visuais de tais espaços e investiga as relações estrutura-propriedade dentro de espaços químicos, com ênfase em aplicações de descoberta de medicamentos.
Protocolo
1. Download e instalação de software
- Torne o diretório deste projeto atualizado. Para acesso conveniente, coloque os executáveis e arquivos neste diretório.
- Instale os pacotes de software necessários após baixá-los.
- Baixe a versão mais recente do software The Osiris DataWarrior (OSIRIS), que pode ser encontrado em https://openmolecules.org/datawarrior/
- Baixe a versão mais recente da plataforma de análise Konstanz Information Miner (KNIME), que pode ser encontrada em https://www.knime.com/
- Baixe a versão mais recente do software The GraphPad Prism, que pode ser encontrado em https://www.graphpad.com/
NOTA: O software Osiris DataWarrior e a plataforma de análise Konstanz Information Miner (KNIME) podem ser usados em um computador pessoal e são gratuitos para uso individual, enquanto o software GraphPad Prism pode ser adquirido em (https://www.graphpad.com/).
2. Construção e curadoria de um banco de dados composto
NOTA: Encontre substâncias e fontes que tenham os dados necessários. O usuário é aconselhado a ter os seguintes detalhes para cada composto em uma planilha.
- Nomeie cada composto. Adicione os nomes de todos os compostos descritos na fonte na primeira coluna de uma planilha.
- Atribua um código interno padronizado se estiver criando uma coleção interna ou atribua um número que identifique exclusivamente esse composto no banco de dados consultado.
- Forneça a entrada da estrutura usando a notação SMILES canônica, que pode ser importada para outras ferramentas de edição molecular.
- Salve o banco de dados idealmente em .csv formato assim que esses dados forem reunidos na planilha.
- Empregue o software OSIRIS para gerar os arquivos de dados de estrutura (SDF), arquivo de dados moleculares (mol) e mol2 do conjunto de dados, que também contêm informações químicas e são interoperáveis com a maioria dos pacotes de software. Para isso, carregue o arquivo .csv clicando no botão Arquivo e, em seguida, no botão Abrir .
- Carregue o conjunto de dados na plataforma de análise KNIME para melhorar a qualidade dos dados e evitar resultados imprecisos. Para isso, carregue o arquivo .sdf ou .mol2 clicando no botão Arquivo e, em seguida, no botão Abrir .
- Garantir uniformidade nas estruturas químicas.
- Examine cada estrutura química para tipos de átomos válidos e verificações de valência. Padronize as estruturas convertendo-as em uma forma tautomérica canônica, kekulizando estruturas aromáticas, padronizando o posicionamento de ligações estéreo e transformando todos os hidrogênios implícitos em hidrogênios explícitos usando o fluxo de trabalho de Padronização de Estruturas Moleculares do KNIME.
- Encontre e elimine duplicatas depois que as moléculas forem padronizadas corretamente, empregando o fluxo de trabalho de Padronização de Estruturas Moleculares do KNIME. Utilize as chaves InChI como uma notação linear para localizar vários estados de protonação e tautômeros.
- Elimine duplicatas.
- Enumere tautômeros e estereoisômeros. Esta etapa é crucial em estudos de triagem virtual, especialmente ao usar métodos de pesquisa, como docking ou filtragem baseada em farmacoforo.
3. Descritores moleculares e análise da diversidade
NOTA: Descritores moleculares, como qualidades físico-químicas, impressões digitais moleculares e andaimes químicos, são as abordagens mais comuns para representar moléculas em aplicações quimioinformáticas. A análise pode ser realizada aqui: http://132.248.103.152:3838/PUMA/. Todas as etapas descritas abaixo são detalhadas no site da PUMA.
- Calcule as seis qualidades físico-químicas mais prevalentes de relevância farmacológica: peso molecular (MW), coeficiente de partição octanol/água (clogP), área de superfície topológica (TPSA), solubilidade aquosa (clogS), número de átomos doadores de ligação H (HBD) e número de átomos aceptores de ligação H (HBA). Consulte o site da PUMA para obter mais informações.
- Calcule as chaves MACCS de 166 bits, a semelhança Tanimoto em pares e as impressões digitais de conectividade estendida de diâmetro 4 (ECFP4), juntamente com outras impressões digitais circulares adequadas para triagem virtual, modelagem de paisagem de atividades e pesquisa de relações estrutura-atividade (SAR).
- Calcule uma estatística de tendência central para cada comparação de pares. Garantir a diversidade no conjunto de dados com uma média ou mediana menor contrária à distância euclidiana ou a qualquer métrica geral de distância.
- Verifique se os valores calculados foram registrados na literatura ou calculados para outros bancos de dados de referência para fins de comparação. Para isso, consulte sites como PubChem ou CHEMBL.
- Gere gráficos de violino para visualização dentro do software GraphPad Prism, exibindo os valores máximo e mínimo.
4. Visualização do espaço químico
NOTA: É possível condensar a maioria dos dados pertinentes em um pequeno número de variáveis usando PCA e outras técnicas de redução de dimensionalidade. As visualizações do espaço químico são, portanto, possíveis.
- Selecione todos os seis descritores para determinar a semelhança ou a distância. Crie a matriz de similaridade (ou distância) de acordo.
- Execute a análise PCA na matriz. Selecione dois ou três componentes principais para plotagem. Considere a proporção de variância capturada por cada componente primário.
- Gere representações de gráfico de dispersão bidimensionais ou tridimensionais para PCA usando o nó Plotly KNIME.
5. Gráficos de diversidade de consenso
NOTA: As representações visuais foram desenvolvidas para resumir algumas características que podem ser usadas para quantificar a variedade. A análise dos gráficos de diversidade de consenso (CDPs)12 pode ser realizada aqui http://132.248.103.152:3838/CDPlots/.
- Crie um gráfico com o número de compostos no banco de dados para determinar o tamanho do ponto de dados. Use a diversidade de impressões digitais moleculares para o eixo x, a diversidade de andaimes para o eixo y, a diversidade baseada nas propriedades físico-químicas para a escala contínua de cores e o número relativo de compostos no conjunto de dados para o tamanho do ponto de dados.
- Gere o gráfico de múltiplas variáveis usando o software GraphPad Prism.
Resultados
Propriedades moleculares e visualização do espaço químico
Todos os compostos nos conjuntos de dados BIOFACQUIM10, PeruNPDB11 e FDA13 tiveram seis propriedades físico-químicas calculadas para eles. Essas qualidades foram então plotadas em gráficos de violino, o que permite ver como as propriedades dos três conjuntos de dados estudados são distribuídas (Figura 1). Os perfis de distribuição dos seis parâmetros físico-químicos de interesse farmacêutico, a saber, peso molecular (MW), coeficiente de partição octanol/água (clogP), área de superfície topológica (TPSA), solubilidade aquosa (clogS), número de átomos doadores de ligação H (HBD) e número de átomos aceitadores de ligação H (HBA), diferem entre os conjuntos de dados. No entanto, os resultados do TPSA demonstraram variações significativas ao comparar os conjuntos de dados BIOFACQUIM e FDA com o PeruNPDB. Usando PCA, a visualização do espaço químico do conjunto de dados foi realizada. No entanto, a análise de PCA visual 3D revela que as moléculas em ambos os conjuntos de dados de NPs se sobrepõem aproximadamente ao espaço químico com a coleção de produtos farmacêuticos aprovados pela FDA. Enquanto em algumas áreas, predominam os produtos químicos do PeruNPDB ou BIOFACQUIM (Figura 2).
Análise de diversidade
Além disso, um CDP baseado em impressões digitais moleculares, andaimes e atributos físico-químicos foi utilizado para avaliar a diversidade dos conjuntos de dados. A diversidade baseada em propriedades dos bancos de dados do PeruNPDB, BIOFAQUIM e FDA foi calculada usando a distância euclidiana das propriedades escalonadas. Além disso, um CDP baseado em impressões digitais moleculares, andaimes e atributos físico-químicos foi utilizado para avaliar a diversidade dos conjuntos de dados. A diversidade baseada em propriedades dos bancos de dados do PeruNPDB, BIOFAQUIM e FDA foi calculada usando a distância euclidiana das propriedades escalonadas. Os valores no gráfico de CD colorido são representados por pontos de dados em uma escala de cores contínua. Tons mais brilhantes denotam maior diversidade, enquanto cores mais escuras denotam menos diversidade. Por último, mas não menos importante, vários tamanhos de pontos são utilizados para mostrar o número relativo de compostos em cada banco de dados, com pontos de dados menores representando bancos de dados com menos moléculas. Como foi descoberto na região onde a maior diversidade de andaimes e impressões digitais deveria estar localizada, os resultados mostraram que os compostos no PeruNPDB tinham a maior diversidade global (Figura 3).
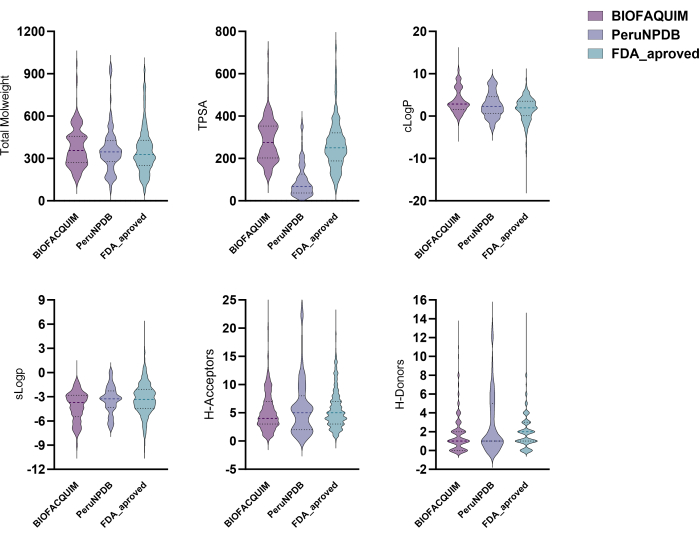
Figura 1: Gráficos de violino para as propriedades físico-químicas. Gráficos de violino para as propriedades físico-químicas dos conjuntos de dados BIOFACQUIM, PeruNPDB e FDA. Clique aqui para ver uma versão maior desta figura.
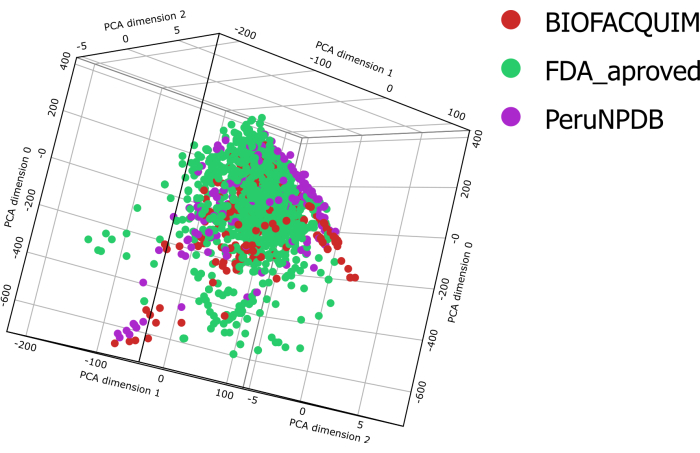
Figura 2: Representação visual do espaço químico. Representação visual dos conjuntos de dados BIOFACQUIM, PeruNPDB e FDA com base nos principais componentes de seis propriedades de relevância farmacêutica. Clique aqui para ver uma versão maior desta figura.

Figura 3: Gráfico de diversidade de consenso. Gráfico de diversidade de consenso comparando a diversidade global dos conjuntos de dados BIOFACQUIM, PeruNPDB e FDA. Clique aqui para ver uma versão maior desta figura.
Discussão
Devido aos seus muitos usos potenciais, como classificação de compostos, seleção de compostos, exploração de ligações estrutura-atividade e navegação por interações estrutura-propriedade, o conceito de espaço químico é hoje amplamente empregado no processo de descoberta e desenvolvimento de medicamentos14. Além disso, a criação de bancos de dados NP é um procedimento fundamental para a realização de diversos estudos computacionais, incluindo o projeto de bibliotecas químicas, caracterização e comparação do espaço químico, o estudo de SAR e triagem virtual, entre outros estudos, em decorrência do aumento da quantidade de informações químicas. Em contraste, o treinamento em algoritmos de inteligência artificial (IA) é outra aplicação crucial. IA refere-se a um grupo de técnicas computacionais que permitem que as máquinas imitem os processos cognitivos humanos, incluindo resolução de problemas e aprendizado com a experiência15,16.
Embora os bancos de dados de compostos químicos, incluindo bancos de dados NP, sejam ferramentas importantes na descoberta de medicamentos, também é viável detectar moléculas de impacto potencial usando uma variedade de técnicas de triagem virtual17. Além disso, vários candidatos a medicamentos foram encontrados em bancos de dados NP que têm potencial para tratar doenças, incluindo doença de coronavírus18, doença de Alzheimer19 e leishmaniose20, entre outras. No entanto, devido às restrições atuais no processamento de "big data", o espaço químico de todas as moléculas potenciais em uma determinada amostra biológica ou ambiental pode ser extremamente grande e quase inexplorado21. Embora não existam técnicas únicas ou universais para representações químicas do espaço, uma maneira amplamente utilizada envolve a criação de matrizes de similaridade que incluem todas as comparações em pares22. A maioria das informações pertinentes pode ser reduzida a um pequeno número de variáveis (embora com perda de informações) usando PCA e outras técnicas de redução de dimensionalidade, permitindo a visualização do espaço químico23.
A diversidade de uma biblioteca química pode ser avaliada de várias maneiras, dependendo em grande parte dos dados que estão sendo examinados e, o mais importante, do objetivo do estudo. A representação molecular é um componente crucial da análise da diversidade, além da medida de diversidade24. Embora os scaffolds químicos e os descritores moleculares sejam as duas abordagens usadas para representar as moléculas com mais frequência na análise quimioinformática, algumas delas têm a desvantagem de serem mais difíceis de compreender25 e não necessariamente identificam as coleções; Por exemplo, é típico que vários compostos tenham perfis de propriedades extremamente comparáveis. Portanto, considerar várias representações de estrutura oferece uma imagem mais abrangente da diversidade de bibliotecas compostas. Essa é a base do conceito de multiverso químico, que pode ser definido como um grupo ou coleção de espaços químicos para um mesmo conjunto de dados, cada um definido por um conjunto de descritores26.
Como os CDPs usam várias representações que podem ser divididas em três ou duas dimensões para analisar a diversidade global de conjuntos de dados compostos usando uma variedade de métricas, eles ajudam a comparar e categorizar bibliotecas químicas12.
Divulgações
Os autores declaram não ter nenhum conflito de interesses.
Agradecimentos
O HLBC e o MACH agradecem o financiamento da Universidad Católica de Santa Maria (subsídios 27499-R-2020, 27574-R-2020, 7309--2020 e 28048-R-2021). A JLMF agradece o financiamento da DGAPA, UNAM, Programa de Apoio a Projetos de Investigação e Inovação Tecnológica (PAPIIT), bolsa nº. IN201321.
Materiais
| Name | Company | Catalog Number | Comments |
| GraphPad Prism | GraphPad Prism | https://www.graphpad.com/ | |
| KNIME platform | KNIME | https://www.knime.com | |
| Osiris DataWarrior (OSIRIS) software | openmolecules.org | https://openmolecules.org/datawarrior/ | |
| PUMA | PUMA: Platform for Unified Molecular Analysis | http://132.248.103.152:3838/PUMA/ |
Referências
- Boufridi, A., Quinn, R. J. Harnessing the properties of natural products. Annu Rev Pharmacol Toxicol. 58, 451-470 (2018).
- Gómez-García, A., et al. Navigating the chemical space and chemical multiverse of a unified Latin American natural product database: LANaPDB. ChemRxiv. , (2023).
- Newman, D. J., Cragg, G. M. Natural products as sources of new drugs over the nearly four decades from 01/1981 to 09/2019. J Nat Prod. 83 (3), 770-803 (2020).
- Atanasov, A. G., Zotchev, S. B., Dirsch, V. M., Supuran, C. T. Natural products in drug discovery: advances and opportunities. Nat Rev Drug Discov. 20 (3), 200-216 (2021).
- Medina-Franco, J. L., Saldívar-González, F. I. Cheminformatics to characterize pharmacologically active natural products. Biomolecules. 10 (11), 1566 (2020).
- Chen, Y., Garcia De Lomana, M., Friedrich, N. O., Kirchmair, J. Characterization of the Chemical Space of Known and Readily Obtainable Natural Products. J Chem Inf Model. 58 (8), 1518-1532 (2018).
- Gaytán-Hernández, D., Chávez-Hernández, A. L., López-López, E., Miranda-Salas, J., Saldívar-González, F. I., Medina-Franco, J. L. Art driven by visual representations of chemical space. ChemRxiv. , (2023).
- Zabolotna, Y., Ertl, P., Horvath, D., Bonachera, F., Marcou, G., Varnek, A. NP Navigator: A new look at the natural product chemical space. Mol Inform. 40 (9), e2100068 (2021).
- Martinez-Mayorga, K., Madariaga-Mazon, A., Medina-Franco, J. L., Maggiora, G. The impact of chemoinformatics on drug discovery in the pharmaceutical industry. Expert Opin Drug Discov. 15 (3), 293-306 (2020).
- Pilón-Jiménez, B., Saldívar-González, F., Díaz-Eufracio, B., Medina-Franco, J. BIOFACQUIM: A Mexican compound database of natural products. Biomolecules. 9 (1), 31 (2019).
- Barazorda-Ccahuana, H. L., et al. PeruNPDB: the Peruvian natural products database for in silico drug screening. Sci Rep. 13 (1), 7577 (2023).
- González-Medina, M., Prieto-Martínez, F. D., Owen, J. R., Medina-Franco, J. L. Consensus diversity plots: a global diversity analysis of chemical libraries. J Cheminform. 8, 63 (2016).
- Irwin, J. J., et al. ZINC20-A free ultralarge-scale chemical database for ligand discovery. J Chem Inf Model. 60 (12), 6065-6073 (2020).
- Naveja, J. J., Medina-Franco, J. L. Finding constellations in chemical space through core analysis. Front Chem. 7, 510 (2019).
- Cavasotto, C. N., Di Filippo, J. I. Artificial intelligence in the early stages of drug discovery. Arch Biochem Biophys. 698, 108730 (2021).
- Rosén, J., Gottfries, J., Muresan, S., Backlund, A., Oprea, T. I. Novel chemical space exploration via natural products. J Med Chem. 52 (7), 1953-1962 (2009).
- Sliwoski, G., Kothiwale, S., Meiler, J., Lowe Jr, E. W. Computational methods in drug discovery. Pharmacol Rev. 66 (1), 334-395 (2014).
- Goyzueta-Mamani, L. D., Barazorda-Ccahuana, H. L., Mena-Ulecia, K., Chávez-Fumagalli, M. A. Antiviral activity of metabolites from Peruvian plants against SARS-CoV-2: An in silico approach. Molecules. 26 (13), 3882 (2021).
- Goyzueta-Mamani, L. D., et al. In silico analysis of metabolites from Peruvian native plants as potential therapeutics against Alzheimer's disease. Molecules. 27 (3), 918 (2022).
- Barazorda-Ccahuana, H. L., et al. Computer-aided drug design approaches applied to screen natural product's structural analogs targeting arginase in Leishmania spp. F1000Research. 12, 93 (2023).
- McGrady, M. Y., Colby, S. M., Nuñez, J. R., Renslow, R. S., Metz, T. O. AI for chemical space gap filling and novel compound generation. arXiv. , (2022).
- Medina-Franco, J., Martinez-Mayorga, K., Giulianotti, M., Houghten, R., Pinilla, C. Visualization of the chemical space in drug discovery. Curr Comput Aided-Drug Des. 4 (4), 322-333 (2008).
- Osolodkin, D. I., Radchenko, E. V., Orlov, A. A., Voronkov, A. E., Palyulin, V. A., Zefirov, N. S. Progress in visual representations of chemical space. Expert Opin Drug Discov. 10 (9), 959-973 (2015).
- Sheridan, R. P., Kearsley, S. K. Why do we need so many chemical similarity search methods. Drug Discov Today. 7 (17), 903-911 (2002).
- Singh, N., Guha, R., Giulianotti, M. A., Pinilla, C., Houghten, R. A., Medina-Franco, J. L. Chemoinformatic analysis of combinatorial libraries, drugs, natural products, and molecular libraries Small Molecule Repository. J Chem Inf Model. 49 (4), 1010-1024 (2009).
- Medina-Franco, J. L., Chávez-Hernández, A. L., López-López, E., Saldívar-González, F. I. Chemical multiverse: An expanded view of chemical space. Mol Inform. 41 (11), e2200116 (2022).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados