Method Article
신약 개발을 위한 천연물 데이터베이스의 화학적 공간 시각화 및 분석
요약
여기에서는 약물 발견과 관련된 응용 분야에 중점을 두고 다양한 분자 표현을 사용하여 천연 화합물 데이터 세트의 화학적 공간을 표시하고 분석하는 방법론을 제공합니다.
초록
화학 공간은 가능한 모든 분자를 둘러싸는 다차원적 설명 공간이며, 분자량이 500Da 미만인 최소 1 x 1060 개의 유기 물질이 약물 발견과 잠재적으로 관련이 있는 것으로 생각됩니다. 천연 제품은 지난 40년 동안 시판된 새로운 약리학적 실체의 주요 공급원이었으며 계속해서 혁신적인 약물 생성을 위한 가장 생산적인 공급원 중 하나였습니다. 화학정보학(Chemoinformatics) 기반 컴퓨팅 도구는 천연 제품의 약물 개발 프로세스를 가속화합니다. 생물 활성, 안전성 프로파일, ADME 및 천연 제품 유사성 측정 추정을 포함한 방법이 사용되었습니다. 여기에서는 다양한 분자 표현을 사용하여 천연 화합물 데이터 세트의 화학적 공간을 시각화, 특성화 및 확장하고, 이러한 공간의 시각적 표현을 생성하고, 화학 공간 내의 구조-특성 관계를 조사하도록 설계된 화학 정보 도구의 최근 개발을 살펴봅니다. 신약 개발 응용 분야에 중점을 두고 오픈 소스 데이터베이스인 BIOFACQUIM 및 PeruNPDB를 개념 증명으로 평가합니다.
서문
생물이 만드는 화합물인 천연물(NP)은 수 세기 동안 전통적인 치료법으로 사용되어 왔습니다. 개별 NP는 현대에 약물로 만들어졌으며 약물 발견에서 선도 화합물로 성공적으로 활용되었습니다1. 인간과 동물이 만들어내는 해양, 곰팡이, 박테리아, 식물, 내인성 물질은 다양한 동물에 의해 생성되는 독과 독극물과 마찬가지로 생리 활성 화합물의 범주에 포함된다2. 그 결과, 40년 동안 NP에 의해 만들어진 약물의 수는 새로운 약리학적 물질의 중요한 원천이 되었으며3, NP는 특히 암 및 전염병 치료뿐만 아니라 다발성 경화증 및 심혈관 질환과 같은 다른 치료 조건4 새로운 약물 개발에 결정적인 역할을 했음을 강조합니다. 또한, 1981년에서 2019년 사이에 암 치료가 승인된 185개의 작은 화합물 중 64.9%는 변형되지 않은 NP 또는 NP 약리3를 함유한 합성 의약품이었습니다.
화학 공간(chemical space)의 개념에 기반을 둔 잘 정립된 학문 간 학문인 화학정보학(Chemoinformatics)은 약물 유사 특성과 관련된 NP의 물리화학적 특성의 화학적 공간을 분석하고 시각화하는 데 사용되어 왔습니다5. 화학정보학(Chemoinformatics)은 NP를 기반으로 한 약물 설계 및 발견에 상당한 영향을 미치는 것으로 나타났습니다6. 화합물 그룹의 화학적 공간이 항상 고유한 것은 아닙니다. 그것은 그것을 정의하는 데 사용되는 설명자의 모음에 따라 달라질 것이며, 이는 NP의 화학적 공간을 다른 화합물 세트와 같이 연구하는 것이 분자 표현7에 의존하는 특별한 도전을 제시한다는 것을 의미합니다. 이러한 노력은 다양한 분자 설명자와 데이터 시각화 기술을 사용하여 접근할 수 있습니다. 대조적으로, 가장 자주 사용되는 기술은 주성분 분석(PCA), 스캐폴드 트리, 자기 조직화 맵, 생성 지형 매핑(GTM) 및 트리 맵(TMAP)이라는 새로운 시각화 기술입니다8. 또한 화합물 데이터베이스에서 NP의 화학 정보를 수집, 평가 및 보급하는 것은 NP 연구에서 화학 정보학의 용도 중 하나입니다. 이와는 대조적으로, 빅 데이터의 도입과 함께 이는 특히 적절합니다9.
여기에서 오픈 소스 NP 데이터베이스 BIOFACQUIM10 및 PeruNPDB11 은 다양한 분자 표현을 사용하여 천연 화합물 데이터 세트의 화학적 공간의 시각화 및 특성화를 검색하고, 이러한 공간의 시각적 표현을 생성하고, 약물 발견 응용 프로그램에 중점을 두고 화학 공간 내의 구조-특성 관계를 조사하는 프로토콜을 설명하는 데 사용됩니다.
프로토콜
1. 소프트웨어 다운로드 및 설치
- 이 프로젝트의 디렉토리를 새로 만듭니다. 편리한 액세스를 위해 실행 파일과 파일을 이 디렉토리에 넣으십시오.
- 필요한 소프트웨어 패키지를 다운로드한 후 설치하십시오.
- https://openmolecules.org/datawarrior/ 에서 찾을 수 있는 최신 버전의 Osiris DataWarrior(OSIRIS) 소프트웨어를 다운로드하십시오.
- 에서 찾을 수 있는 KNIME(Konstanz Information Miner) 분석 플랫폼의 최신 버전을 다운로드하십시오 https://www.knime.com/
- 에서 찾을 수 있는 최신 버전의 GraphPad Prism 소프트웨어를 다운로드하십시오 https://www.graphpad.com/
참고: Osiris DataWarrior 소프트웨어 및 KNIME(Konstanz Information Miner) 분석 플랫폼은 개인용 컴퓨터에서 사용할 수 있으며 개인용으로 무료로 사용할 수 있으며 GraphPad Prism 소프트웨어는 (https://www.graphpad.com/)에서 구입할 수 있습니다.
2. 복합 데이터베이스의 구축 및 큐레이션
참고: 필요한 데이터가 있는 물질 및 출처를 찾으십시오. 사용자는 스프레드시트의 각 화합물에 대해 다음 세부 정보를 갖는 것이 좋습니다.
- 각 화합물의 이름을 지정합니다. 소스에서 설명하는 모든 화합물의 이름을 스프레드시트의 첫 번째 열에 추가합니다.
- 사내 컬렉션을 생성하는 경우 내부의 표준화된 코드를 할당하거나 컨설팅 데이터베이스에서 이 화합물을 고유하게 식별하는 번호를 할당합니다.
- 다른 분자 편집 도구로 가져올 수 있는 표준 SMILES 표기법을 사용하여 구조 입력을 제공합니다.
- 이 데이터가 스프레드시트에 수집되면 데이터베이스를 .csv 형식으로 이상적으로 저장하십시오.
- OSIRIS 소프트웨어를 사용하여 화학 정보를 포함하고 대부분의 소프트웨어 패키지와 상호 운용 가능한 데이터 세트의 구조 데이터 파일(SDF), 분자 데이터 파일(mol) 및 mol2를 생성합니다. 이를 위해 File 버튼을 클릭한 다음 Open 버튼을 클릭하여 .csv 아카이브를 업로드합니다.
- 데이터 세트를 KNIME 분석 플랫폼에 업로드하여 데이터 품질을 개선하고 부정확한 결과를 방지합니다. 이를 위해 파일 버튼을 클릭한 다음 열기 버튼을 클릭하여 .sdf 또는 .mol2 파일을 업로드합니다.
- 화학 구조의 균일성을 보장합니다.
- 유효한 원자 유형 및 원자가 검사에 대해 각 화학 구조를 검사합니다. KNIME의 Standardizing Molecular Structures 워크플로우를 사용하여 표준 tautomeric 형태로 변환하고, 방향족 구조를 kekulizing 하고, 스테레오 결합의 위치를 표준화하고, 모든 암시적 수소를 명시적 수소로 변환하여 구조를 표준화합니다.
- 분자가 올바르게 표준화된 후 KNIME의 Standardizing Molecular Structures 워크플로우를 사용하여 중복을 찾아 제거합니다. InChI 키를 선형 표기법으로 활용하여 다양한 양성자 상태와 tautomers를 찾습니다.
- 중복을 제거합니다.
- tautomers와 stereoisomers를 열거합니다. 이 단계는 가상 스크리닝 연구에서 매우 중요하며, 특히 도킹 또는 약물 기반 필터링과 같은 검색 방법을 사용할 때 중요합니다.
3. 분자 설명자 및 다양성 분석
참고: 물리화학적 특성, 분자 지문 및 화학적 지지체와 같은 분자 설명자는 화학정보학 응용 분야에서 분자를 나타내는 가장 일반적인 접근 방식입니다. 분석은 여기에서 수행할 수 있습니다. http://132.248.103.152:3838/PUMA/. 아래에 설명된 모든 단계는 PUMA 웹 사이트에 자세히 설명되어 있습니다.
- 약리학적 관련성의 가장 일반적인 6가지 물리화학적 특성인 분자량(MW), 옥탄올/물 분배 계수(clogP), 위상 표면적(TPSA), 수성 용해도(clogS), H-결합 공여체 원자(HBD) 및 H-결합 수용체 원자(HBA) 수를 계산합니다. 자세한 내용은 PUMA 웹 사이트를 참조하십시오.
- 가상 스크리닝, 활동 경관 모델링 및 구조-활동 관계(SAR) 연구에 적합한 다른 원형 지문과 함께 166비트 MACCS 키, 쌍별 Tanimoto 유사성 및 ECFP4(Extended Connectivity fingerprints of diameter 4)를 계산합니다.
- 각 쌍별 비교에 대한 중심 경향 통계량을 계산합니다. 유클리드 거리 또는 일반 거리 메트릭과 달리 더 작은 평균 또는 중앙값으로 데이터 세트의 다양성을 보장합니다.
- 계산된 값이 문헌에 기록되었는지 또는 비교를 위해 다른 참조 데이터베이스에서 계산되었는지 확인합니다. 이를 위해 PubChem 또는 CHEMBL과 같은 웹 사이트를 참조하십시오.
- GraphPad Prism 소프트웨어 내에서 시각화를 위한 바이올린 플롯을 생성하여 최대값과 최소값을 표시합니다.
4. 화학 공간의 시각화
참고: PCA 및 기타 차원 축소 기술을 사용하여 대부분의 관련 데이터를 소수의 변수로 압축할 수 있습니다. 따라서 화학 공간의 시각화가 가능합니다.
- 6개의 설명자를 모두 선택하여 유사성 또는 거리를 확인합니다. 그에 따라 유사성(또는 거리) 행렬을 만듭니다.
- 매트릭스에 대한 PCA 분석을 수행합니다. 플로팅할 두 개 또는 세 개의 주요 구성요소를 선택합니다. 각 주성분에 의해 포착된 분산의 비율을 고려하십시오.
- Plotly KNIME 노드를 사용하여 PCA에 대한 2차원 또는 3차원 산점도 표현을 생성합니다.
5. 합의 다양성 플롯
참고: 시각적 표현은 다양성을 정량화하는 데 사용할 수 있는 몇 가지 특성을 요약하기 위해 개발되었습니다. 컨센서스 다양성 플롯(CDP)12 분석은 여기에서 http://132.248.103.152:3838/CDPlots/ 수행할 수 있습니다.
- 데이터베이스에 있는 화합물의 개수가 포함된 플롯을 만들어 데이터 점 크기를 확인합니다. x축에는 분자 지문의 다양성을, y축에는 스캐폴드의 다양성을, 색상 연속 스케일에는 물리화학적 특성에 따른 다양성을, 데이터 포인트 크기는 데이터 세트에 있는 화합물의 상대적 수를 사용합니다.
- GraphPad Prism을 사용하여 다변수 플롯을 생성합니다.
결과
화학 공간의 분자 특성 및 시각화
BIOFACQUIM10, PeruNPDB11 및 FDA13 데이터 세트의 모든 화합물은 6가지 물리화학적 특성이 계산되었습니다. 그런 다음 이러한 특성을 바이올린 플롯에 표시하여 연구된 세 가지 데이터 세트의 속성이 어떻게 분포되어 있는지 확인할 수 있습니다(그림 1). 분자량(MW), 옥탄올/물 분배 계수(clogP), 토폴로지 표면적(TPSA), 수성 용해도(clogS), H-결합 공여체 원자(HBD) 및 H-결합 수용체 원자(HBA)의 수, 즉 약학적 관심 분야의 6가지 물리화학적 매개변수의 분포 프로필은 데이터 세트마다 다릅니다. 그러나 TPSA 결과는 BIOFACQUIM 및 FDA 데이터 세트를 PeruNPDB와 비교할 때 상당한 차이를 보여주었습니다. PCA를 사용하여 데이터 세트의 화학 공간 시각화를 수행했습니다. 그러나 3D 시각적 PCA 분석에 따르면 NP의 두 데이터 세트에 있는 분자는 FDA의 승인된 의약품 컬렉션과 화학적 공간과 거의 겹칩니다. 일부 지역에서는 PeruNPDB 또는 BIOFACQUIM의 화학 물질이 우세합니다(그림 2).
다양성 분석
또한 분자 지문, 골격 및 물리화학적 속성을 기반으로 하는 CDP를 사용하여 데이터 세트의 다양성을 평가했습니다. PeruNPDB, BIOFAQUIM 및 FDA 데이터베이스의 속성 기반 다양성은 스케일링된 속성의 유클리드 거리를 사용하여 계산되었습니다. 또한 분자 지문, 골격 및 물리화학적 속성을 기반으로 하는 CDP를 사용하여 데이터 세트의 다양성을 평가했습니다. PeruNPDB, BIOFAQUIM 및 FDA 데이터베이스의 속성 기반 다양성은 스케일링된 속성의 유클리드 거리를 사용하여 계산되었습니다. 컬러 CD 플롯의 값은 연속 컬러 스케일의 데이터 포인트로 표시됩니다. 밝은 색조는 더 큰 다양성을 나타내고 더 어두운 색상은 다양성이 적다는 것을 나타냅니다. 마지막으로, 각 데이터베이스에 있는 화합물의 상대적인 수를 표시하기 위해 다양한 포인트 크기가 사용되며, 더 작은 데이터 포인트는 더 적은 분자가 있는 데이터베이스를 나타냅니다. 골격과 지문의 다양성이 가장 높아야 하는 지역에서 발견되었기 때문에 연구 결과는 PeruNPDB의 화합물이 전 세계적으로 가장 큰 다양성을 가지고 있음을 보여주었습니다(그림 3).
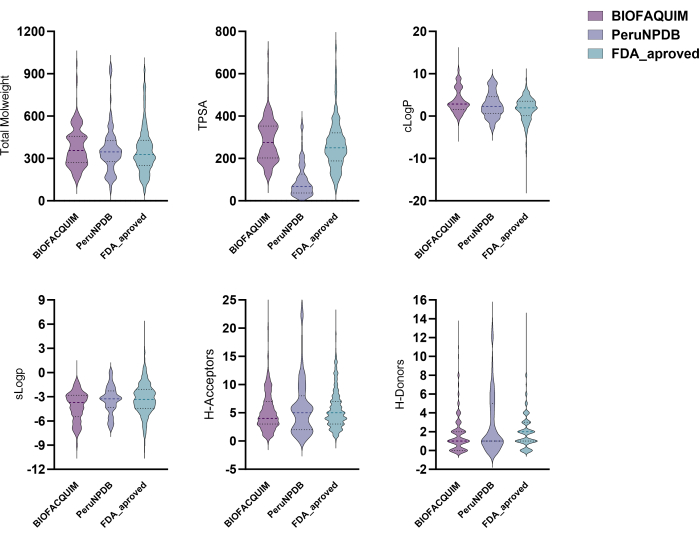
그림 1: 물리화학적 특성에 대한 바이올린 플롯. 바이올린은 BIOFACQUIM, PeruNPDB 및 FDA 데이터 세트의 물리화학적 특성에 대한 플롯입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
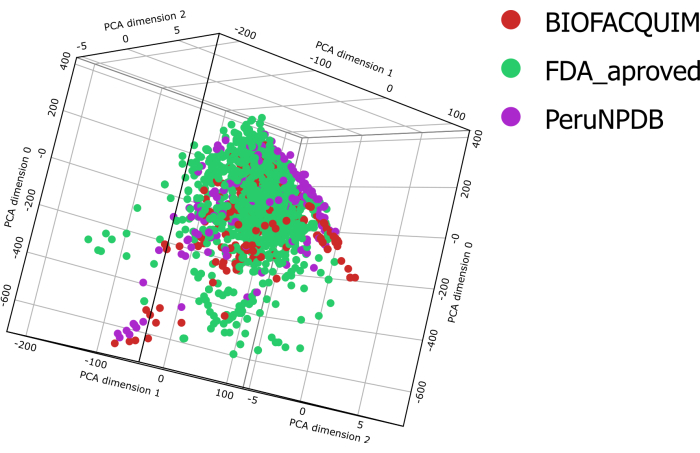
그림 2: 화학 공간의 시각적 표현. 제약 관련성이 있는 6가지 속성의 주요 구성 요소를 기반으로 BIOFACQUIM, PeruNPDB 및 FDA 데이터 세트를 시각적으로 표현합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 3: 컨센서스 다양성 플롯. BIOFACQUIM, PeruNPDB 및 FDA 데이터 세트의 글로벌 다양성을 비교하는 합의 다양성 플롯. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
화합물 분류, 화합물 선택, 구조-활성 연결 탐색, 구조-특성 상호 작용 탐색과 같은 많은 잠재적 용도로 인해 화학 공간의 개념은 오늘날 약물 발견 및 개발 과정에서 널리 사용되고 있습니다14. 또한 NP 데이터베이스의 생성은 화학 정보의 양 증가로 인한 화학 라이브러리 설계, 화학 공간의 특성화 및 비교, SAR 연구, 가상 스크리닝 등 다양한 컴퓨터 연구를 수행하기 위한 기본 절차입니다. 반면, 인공 지능(AI) 알고리즘에 대한 교육은 또 다른 중요한 응용 분야입니다. 인공지능(AI)은 기계가 문제 해결 및 경험을 통한 학습을 포함하여 인간의 인지 과정을 모방할 수 있도록 하는 일련의 계산 기술을 말합니다15,16.
NP 데이터베이스를 포함한 화합물 데이터베이스는 신약 개발에 중요한 도구이지만, 다양한 가상 스크리닝 기법을 사용하여 잠재적인 히트 분자를 검출하는 것도 가능합니다17. 또한 NP 데이터베이스에서 코로나바이러스 질병18, 알츠하이머병19, 리슈마니아증20 등 질병을 치료할 수 있는 잠재력이 있는 여러 약물 후보가 발견되었습니다. 그러나 "빅 데이터" 처리에 대한 현재의 제약으로 인해 특정 생물학적 또는 환경적 샘플에 있는 모든 잠재적 분자의 화학적 공간은 매우 클 수 있으며 대부분 탐사되지 않을 수 있습니다21. 화학적 공간 표현에 대한 고유하거나 보편적인 기술은 없지만, 널리 사용되는 한 가지 방법은 모든 쌍별 비교를 포함하는 유사성 행렬을 만드는 것입니다22. 관련 정보의 대다수는 PCA 및 다른 차원 축소 기술을 사용하여 소수의 변수(정보를 잃었음에도 불구하고)로 감소될 수 있으며, 이는 화학 공간(23)의 시각화를 가능하게 한다.
화학 라이브러리의 다양성은 주로 조사되는 데이터와 가장 중요한 연구의 목표에 따라 다양한 방법으로 평가할 수 있습니다. 분자 표상은 다양성 측정(diversity measure)24 외에도 다양성 분석의 중요한 구성 요소이다. 화학 스캐폴드(chemical scaffolds)와 분자 설명자(molecular descriptors)는 화학정보학 분석에서 가장 빈번하게 분자를 나타내는 데 사용되는 두 가지 접근 방식이지만, 그 중 일부는25를 이해하기가 더 어렵고 컬렉션을 반드시 식별하지는 않는다는 단점이 있습니다. 예를 들어, 다양한 화합물이 매우 유사한 특성 프로파일을 갖는 것이 일반적입니다. 따라서 다양한 구조 표현을 고려하면 복합 라이브러리의 다양성에 대한 보다 포괄적인 그림을 얻을 수 있습니다. 이것은 화학적 다중우주(chemical multiverse) 개념의 기초이며, 이는 동일한 데이터 세트에 대한 화학적 공간의 그룹 또는 집합으로 정의되며, 각각은 설명자세트(26)에 의해 정의된다.
CDP는 다양한 메트릭을 사용하여 화합물 데이터 세트의 전체 다양성을 분석하기 위해 3차원 또는 2차원으로 나눌 수 있는 여러 표현을 사용하기 때문에 화학 라이브러리를 비교하고 분류하는 데 도움이 됩니다12.
공개
저자는 이해 상충이 없음을 선언합니다.
감사의 말
HLBC와 MACH는 Universidad Catolica de Santa Maria(보조금 27499-R-2020, 27574-R-2020, 7309-CU-2020 및 28048-R-2021)의 자금 지원에 감사드립니다. JLMF는 DGAPA, UNAM, Programa de Apoyo a Proyectos de Investigación e Innovación Tecnológica (PAPIIT)의 자금 지원에 감사드립니다. IN201321.
자료
| Name | Company | Catalog Number | Comments |
| GraphPad Prism | GraphPad Prism | https://www.graphpad.com/ | |
| KNIME platform | KNIME | https://www.knime.com | |
| Osiris DataWarrior (OSIRIS) software | openmolecules.org | https://openmolecules.org/datawarrior/ | |
| PUMA | PUMA: Platform for Unified Molecular Analysis | http://132.248.103.152:3838/PUMA/ |
참고문헌
- Boufridi, A., Quinn, R. J. Harnessing the properties of natural products. Annu Rev Pharmacol Toxicol. 58, 451-470 (2018).
- Gómez-García, A., et al. Navigating the chemical space and chemical multiverse of a unified Latin American natural product database: LANaPDB. ChemRxiv. , (2023).
- Newman, D. J., Cragg, G. M. Natural products as sources of new drugs over the nearly four decades from 01/1981 to 09/2019. J Nat Prod. 83 (3), 770-803 (2020).
- Atanasov, A. G., Zotchev, S. B., Dirsch, V. M., Supuran, C. T. Natural products in drug discovery: advances and opportunities. Nat Rev Drug Discov. 20 (3), 200-216 (2021).
- Medina-Franco, J. L., Saldívar-González, F. I. Cheminformatics to characterize pharmacologically active natural products. Biomolecules. 10 (11), 1566 (2020).
- Chen, Y., Garcia De Lomana, M., Friedrich, N. O., Kirchmair, J. Characterization of the Chemical Space of Known and Readily Obtainable Natural Products. J Chem Inf Model. 58 (8), 1518-1532 (2018).
- Gaytán-Hernández, D., Chávez-Hernández, A. L., López-López, E., Miranda-Salas, J., Saldívar-González, F. I., Medina-Franco, J. L. Art driven by visual representations of chemical space. ChemRxiv. , (2023).
- Zabolotna, Y., Ertl, P., Horvath, D., Bonachera, F., Marcou, G., Varnek, A. NP Navigator: A new look at the natural product chemical space. Mol Inform. 40 (9), e2100068 (2021).
- Martinez-Mayorga, K., Madariaga-Mazon, A., Medina-Franco, J. L., Maggiora, G. The impact of chemoinformatics on drug discovery in the pharmaceutical industry. Expert Opin Drug Discov. 15 (3), 293-306 (2020).
- Pilón-Jiménez, B., Saldívar-González, F., Díaz-Eufracio, B., Medina-Franco, J. BIOFACQUIM: A Mexican compound database of natural products. Biomolecules. 9 (1), 31 (2019).
- Barazorda-Ccahuana, H. L., et al. PeruNPDB: the Peruvian natural products database for in silico drug screening. Sci Rep. 13 (1), 7577 (2023).
- González-Medina, M., Prieto-Martínez, F. D., Owen, J. R., Medina-Franco, J. L. Consensus diversity plots: a global diversity analysis of chemical libraries. J Cheminform. 8, 63 (2016).
- Irwin, J. J., et al. ZINC20-A free ultralarge-scale chemical database for ligand discovery. J Chem Inf Model. 60 (12), 6065-6073 (2020).
- Naveja, J. J., Medina-Franco, J. L. Finding constellations in chemical space through core analysis. Front Chem. 7, 510 (2019).
- Cavasotto, C. N., Di Filippo, J. I. Artificial intelligence in the early stages of drug discovery. Arch Biochem Biophys. 698, 108730 (2021).
- Rosén, J., Gottfries, J., Muresan, S., Backlund, A., Oprea, T. I. Novel chemical space exploration via natural products. J Med Chem. 52 (7), 1953-1962 (2009).
- Sliwoski, G., Kothiwale, S., Meiler, J., Lowe Jr, E. W. Computational methods in drug discovery. Pharmacol Rev. 66 (1), 334-395 (2014).
- Goyzueta-Mamani, L. D., Barazorda-Ccahuana, H. L., Mena-Ulecia, K., Chávez-Fumagalli, M. A. Antiviral activity of metabolites from Peruvian plants against SARS-CoV-2: An in silico approach. Molecules. 26 (13), 3882 (2021).
- Goyzueta-Mamani, L. D., et al. In silico analysis of metabolites from Peruvian native plants as potential therapeutics against Alzheimer's disease. Molecules. 27 (3), 918 (2022).
- Barazorda-Ccahuana, H. L., et al. Computer-aided drug design approaches applied to screen natural product's structural analogs targeting arginase in Leishmania spp. F1000Research. 12, 93 (2023).
- McGrady, M. Y., Colby, S. M., Nuñez, J. R., Renslow, R. S., Metz, T. O. AI for chemical space gap filling and novel compound generation. arXiv. , (2022).
- Medina-Franco, J., Martinez-Mayorga, K., Giulianotti, M., Houghten, R., Pinilla, C. Visualization of the chemical space in drug discovery. Curr Comput Aided-Drug Des. 4 (4), 322-333 (2008).
- Osolodkin, D. I., Radchenko, E. V., Orlov, A. A., Voronkov, A. E., Palyulin, V. A., Zefirov, N. S. Progress in visual representations of chemical space. Expert Opin Drug Discov. 10 (9), 959-973 (2015).
- Sheridan, R. P., Kearsley, S. K. Why do we need so many chemical similarity search methods. Drug Discov Today. 7 (17), 903-911 (2002).
- Singh, N., Guha, R., Giulianotti, M. A., Pinilla, C., Houghten, R. A., Medina-Franco, J. L. Chemoinformatic analysis of combinatorial libraries, drugs, natural products, and molecular libraries Small Molecule Repository. J Chem Inf Model. 49 (4), 1010-1024 (2009).
- Medina-Franco, J. L., Chávez-Hernández, A. L., López-López, E., Saldívar-González, F. I. Chemical multiverse: An expanded view of chemical space. Mol Inform. 41 (11), e2200116 (2022).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유
당사 웹 사이트에서는 사용자의 경험을 향상시키기 위해 쿠키를 사용합니다.
당사 웹 사이트를 계속 사용하거나 '계속'을 클릭하는 것은 당사 쿠키 수락에 동의하는 것을 의미합니다.