Method Article
Identificação e análise de progenitores miogênicos in vivo durante lesão muscular esquelética aguda por citometria de massa de célula única de alta dimensão
Neste Artigo
Resumo
O protocolo aqui apresentado permite a identificação e análise de alta dimensão de células-tronco musculares e progenitoras por citometria de massa de célula única e sua purificação por FACS para estudos aprofundados de sua função. Essa abordagem pode ser aplicada para estudar a dinâmica de regeneração em modelos de doenças e testar a eficácia de intervenções farmacológicas.
Resumo
A regeneração do músculo esquelético é um processo dinâmico impulsionado por células-tronco musculares adultas e sua progênie. Principalmente quiescentes em um estado estacionário, as células-tronco musculares adultas são ativadas após a lesão muscular. Após a ativação, eles proliferam e a maior parte de sua progênie se diferencia para gerar células musculares competentes para fusão, enquanto o restante se auto-renova para reabastecer o pool de células-tronco. Embora a identidade das células-tronco musculares tenha sido definida há mais de uma década, com base na co-expressão de marcadores de superfície celular, os progenitores miogênicos foram identificados apenas recentemente usando abordagens unicelulares de alta dimensão. Aqui, apresentamos um método de citometria de massa de célula única (citometria por tempo de voo [CyTOF]) para analisar células-tronco e células progenitoras na lesão muscular aguda para resolver a dinâmica celular e molecular que se desdobra durante a regeneração muscular. Essa abordagem é baseada na detecção simultânea de novos marcadores de superfície celular e fatores-chave de transcrição miogênica cuja expressão dinâmica permite a identificação de células-tronco ativadas e populações de células progenitoras que representam marcos da miogênese. É importante ressaltar que uma estratégia de classificação baseada na detecção dos marcadores de superfície celular CD9 e CD104 é descrita, permitindo o isolamento prospectivo de células-tronco musculares e células progenitoras usando classificação de células ativadas por fluorescência (FACS) para estudos aprofundados de sua função. As células progenitoras musculares fornecem um elo perdido crítico para estudar o controle do destino das células-tronco musculares, identificar novos alvos terapêuticos para doenças musculares e desenvolver aplicações de terapia celular para medicina regenerativa. A abordagem apresentada aqui pode ser aplicada para estudar células-tronco musculares e progenitoras in vivo em resposta a perturbações, como intervenções farmacológicas direcionadas a vias de sinalização específicas. Também pode ser usado para investigar a dinâmica das células-tronco musculares e progenitoras em modelos animais de doenças musculares, avançando nossa compreensão das doenças com células-tronco e acelerando o desenvolvimento de terapias.
Introdução
O músculo esquelético constitui o maior tecido em massa do corpo e regula múltiplas funções, da visão à respiração, da postura ao movimento, bem como o metabolismo1. Portanto, manter a integridade e a função do músculo esquelético é fundamental para a saúde. O tecido muscular esquelético, que consiste em feixes compactados de miofibras multinucleadas cercadas por uma complexa rede de nervos e vasos sanguíneos, exibe notável potencial regenerativo 1,2.
Os principais impulsionadores da regeneração do músculo esquelético são as células-tronco musculares adultas (MuSCs). Também conhecidas como células satélites, devido à sua localização anatômica única adjacente à membrana plasmática da miofibra e abaixo da lâmina basal, foram identificadas pela primeira vez em 19613. Os MuSCs expressam um marcador molecular único, o fator de transcrição pareado box 7 (Pax7)4. Principalmente quiescentes em adultos saudáveis, eles são ativados após lesão muscular e proliferam para dar origem a descendentes que (i) se diferenciam em células musculares competentes para fusão que formarão novas miofibras para reparar danos musculares ou (ii) se auto-renovam para reabastecer o pool de células-tronco5.
No nível celular e molecular, o processo de regeneração é bastante dinâmico e envolve transições de estado celular, caracterizadas pela expressão coordenada de fatores-chave de transcrição miogênicos, também conhecidos como fatores reguladores miogênicos (MRFs)6,7. Estudos anteriores de desenvolvimento in vivo, experimentos de rastreamento de linhagem e trabalho de cultura de células usando mioblastos mostraram que a expressão sequencial desses fatores de transcrição impulsiona a miogênese, com o fator miogênico 5 (Myf5) sendo expresso na ativação, a diferenciação miogênica 1 (MyoD1) marcando o compromisso com o programa miogênico e a expressão de miogenina (MyoG) marcando a diferenciação 8,9,10,11, 12,13,14. Apesar desse conhecimento e da descoberta de marcadores de superfície celular para purificar MuSCs, faltam estratégias e ferramentas para identificar e isolar populações discretas ao longo da via de diferenciação miogênica e resolver uma progressão miogênica in vivo 15,16,17,18.
Aqui, apresentamos um novo método, baseado em pesquisas publicadas recentemente, que permite a identificação de células-tronco e progenitoras no músculo esquelético e a análise de sua dinâmica celular, molecular e de proliferação no contexto da lesão muscular aguda19. Essa abordagem se baseia na citometria de massa de célula única (também conhecida como Citometria por Tempo de Voo [CyTOF]) para detectar simultaneamente os principais marcadores de superfície celular (integrina α7, CD9, CD44, CD98 e CD104), fatores de transcrição miogênica intracelular (Pax7, Myf5, MyoD e MyoG) e um análogo de nucleosídeo (5-Iodo-2′-desoxiuridina, IdU), para monitorar células na fase S19,20, 21,22,23. Além disso, o protocolo apresenta uma estratégia baseada na detecção de dois marcadores de superfície celular, CD9 e CD104, para purificar essas populações celulares por classificação celular ativada por fluorescência (FACS), permitindo assim futuros estudos aprofundados de sua função no contexto de lesões e doenças musculares. Embora os mioblastos primários tenham sido amplamente utilizados no passado para estudar os estágios finais da diferenciação miogênica in vitro, não se sabe se eles recapitulam o estado molecular das células progenitoras musculares encontradas in vivo24 , 25 , 26 , 27 , 28 , 29 , 30. A produção de mioblastos é trabalhosa e demorada, e o estado molecular dessa cultura primária muda rapidamente após a passagem31. Assim, progenitores miogênicos recém-isolados e purificados com este método fornecerão um sistema mais fisiológico para estudar a miogênese e o efeito de manipulações genéticas ou farmacológicas ex vivo.
O protocolo aqui apresentado pode ser aplicado para abordar uma variedade de questões de pesquisa, por exemplo, para estudar a dinâmica do compartimento miogênico in vivo em modelos animais de doenças musculares, em resposta a manipulações genéticas agudas ou em intervenções farmacológicas, aprofundando assim nossa compreensão da disfunção das células-tronco musculares em diferentes contextos biológicos e facilitando o desenvolvimento de novas intervenções terapêuticas.
Protocolo
Os procedimentos em animais foram aprovados pela inspeção dinamarquesa de experimentos com animais (protocolo # 2022-15-0201-01293) e os experimentos foram realizados em conformidade com as diretrizes institucionais da Universidade de Aarhus. A analgesia (buprenorfina) é fornecida na água potável 24 horas antes da lesão para que os camundongos se adaptem ao sabor. O fornecimento de buprenorfina na água potável é continuado por 24 horas após a lesão. Juntamente com uma injeção subcutânea (s.c.) de buprenorfina no momento da lesão muscular aguda, a buprenorfina na água potável após a injeção de notexina aliviará a dor associada à lesão. Embora seja recomendado administrar uma injeção s.c. de buprenorfina no momento da lesão muscular aguda, seguida de buprenorfina na água de beber, a buprenorfina na água de beber antes da lesão é opcional. No entanto, os pesquisadores devem seguir os padrões e diretrizes de bem-estar animal estabelecidos pela agência reguladora competente.
NOTA: Para experimentos de citometria de massa de célula única (CyTOF) de músculos lesionados dos membros posteriores, comece na seção 1: Analgesia em água 24 h antes da lesão muscular até 24 h após a lesão. Para a classificação do tronco muscular e das células progenitoras de camundongos não lesionados, execute as seções 5 e 6: Eutanásia + Dissecção e dissociação do músculo esquelético e continue na seção 11: Coloração com anticorpos conjugados com fluoróforo para FACS. Uma visão geral da configuração experimental e do protocolo é mostrada na Figura 1.
1. Analgesia em água 24 h antes da lesão muscular até 24 h após a lesão
- Em um frasco escuro ou embrulhado em papel alumínio, adicione 3 mL de buprenorfina (0,3 mg / mL) e encha até 100 mL com água filtrada para atingir uma concentração final de 0,009 mg / mL 24 h antes da lesão. Anexe à gaiola do mouse.
- Remova o bebedouro 24 h após a lesão e reconecte a gaiola do mouse ao sistema de válvula de bebida.
2. Preparando-se para o procedimento de lesão aguda
NOTA: Use etanol 70% para desinfetar a bancada de trabalho, a configuração do cone do nariz e a caixa de indução.
- Prepare um tubo de 1,5 mL com notexina diluída em PBS (5 μg / mL). Dilua a buprenorfina (0,3 mg / mL) em solução salina estéril a 0,9% para 0,015 mg / mL em um tubo de 1,5 mL à prova de luz. Mantenha no gelo. Prepare seringas de insulina para injeções de buprenorfina (28 G, 0,5 mL) e notexina (29 G, 0,3 mL).
- Configure unidades anestésicas à base de isoflurano:
- Para anestesia em caixa de indução, use isoflurano a 3% com fluxo de 1,5 L/min (50% O2, 50% ar atmosférico).
- Para manutenção utilizando uma configuração de cone nasal, use isoflurano a 1,5% com um fluxo de 0,6 L/min (50% O2, 50% de ar atmosférico).
3. Lesão aguda por injeção de notexina
CUIDADO: A notoxina tem atividade fosfolipase A2 e é o principal componente do veneno da cobra-tigre australiana (Notechis scutatus), com um LD50 intravenoso de 5–17 mg de notoxina / kg em camundongos32,33. No presente protocolo, o músculo tibial anterior (TA) de cada membro posterior é injetado com 10 μL de 5 mg/mL de notexina, e o músculo gastrocnêmio (GA) de cada membro posterior é injetado duas vezes (uma vez em cada cabeça do músculo) com 15 μL de 5 mg/mL de notexina. É importante realizar as injeções intramusculares (i.m.) corretamente para limitar os danos e inspecionar frequentemente os animais injetados para garantir o mínimo de dor.
- Anestesiar o mouse na caixa de indução (isoflurano a 3%). Quando o camundongo estiver inconsciente, abaixe o nível de isoflurano para 1,5% na caixa de indução.
- Pese e marque individualmente os camundongos de acordo com os procedimentos aprovados pela instituição. Sob a configuração do cone do nariz, raspe os membros posteriores com um aparador e transfira de volta para a caixa de indução.
- Calcule o volume de buprenorfina para cada camundongo:
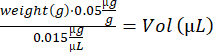
- Misture a solução de notexina pipetando para cima e para baixo. Prepare 2 seringas de insulina (29 G, 0,3 mL) carregadas com 10 μL de solução de notexina (5 μg / mL) para injeções de TA e 4 seringas de insulina carregadas com 15 μL de solução de notexina (5 μg / mL) para injeção de GA.
NOTA: O músculo tibial anterior (TA) está localizado no lado anterior da perna do camundongo, estendendo-se do joelho ao tornozelo. O músculo gastrocnêmio (GA) é um músculo de duas cabeças localizado na parte de trás da perna, superficial ao sóleo. Ele vai de suas duas cabeças (medial e lateral) logo acima do joelho até o calcanhar, estendendo-se por um total de três articulações (joelho, tornozelo e articulações subtalares). - Realizar injeções intramusculares
- Injeção de TA (2 injeções no total)
- Coloque o mouse em decúbito dorsal sob a configuração do cone do nariz e desinfete o local da injeção com lenços umedecidos com álcool estéril (etanol 70% ou isopropanol). Insira a agulha, chanfrada para baixo, na barriga do TA (distal ao meio da barriga) em um ângulo de 30°. Avance a agulha ao longo do músculo, movendo-se paralelamente à tíbia, para alcançar o meio da barriga do TA. Injete a notexina lenta e continuamente, deixando a agulha no lugar por 10 s antes de retirá-la. Não insira a agulha muito fundo inicialmente (para evitar a injeção de notexina abaixo do TA no músculo extensor longo dos dedos) e não muito proximalmente ao avançar a agulha (para evitar injetar muito perto do joelho).
- Injeção de GA (4 injeções no total)
- Insira a agulha, chanfrada para baixo, em um ângulo de ~ 45 ° no meio da barriga da cabeça lateral do GA. Injete a notexina lenta e continuamente, deixando a agulha no lugar por 10 s antes de retirá-la. Gire o membro posterior e injete notexina na cabeça medial do músculo GA como acima. Deve-se tomar cuidado para não inserir a agulha muito profundamente.
- Injeção de TA (2 injeções no total)
- Vire o mouse e injete buprenorfina s.c. usando uma seringa de insulina (28 G, 0,5 mL). Transfira o mouse para uma gaiola de recuperação vazia em uma placa de aquecimento. Apenas metade do fundo da gaiola deve ser colocada sobre a placa de aquecimento para permitir que o mouse termorregule durante a recuperação34. Repita as etapas 3.3 a 3.6 para os mouses restantes.
- Quando o mouse na gaiola de recuperação estiver totalmente acordado, mova-o de volta para a gaiola original. Devolva-o ao estábulo e complemente-o com ração molhada. Remova o frasco de buprenorfina 24 h após a injeção.
- Monitore os camundongos por 6 h após a injeção de notexina e depois a cada 12 h por 2 dias quanto a sinais de dor, mobilidade prejudicada e diminuição do consumo de alimentos35,36.
4. Injeção de 5-Iodo-2'-desoxiuridina
CUIDADO: Suspeita-se que o 5-iodo-2'-desoxiuridina (IdU) cause defeitos genéticos e prejudique a fertilidade ou o feto. Leia a ficha de dados de segurança (SDS) antes de manusear. Equipamento de proteção individual deve ser usado durante o manuseio. Use uma capela ao pesar o pó IdU. Os materiais que estiveram em contato com o IdU devem ser descartados de acordo com os regulamentos de segurança locais.
NOTA: A marcação de IdU in vivo é usada para monitorar a divisão celular durante o curso do tempo de lesão porque o IdU, um análogo da timidina iodada, é incorporado ao DNA das células na fase S. A IdU é injetada por via intraperitoneal (i.p.) a 20 mg/kg de peso corporal 8 h antes do sacrifício do camundongo.
- Utilizar um sonicador a 37 °C para preparar uma solução-mãe de 2 mg/ml de IdU em PBS estéril. IdU é sensível à luz; use no mesmo dia ou congele a -20 °C por até 3 meses. Se estiver usando IdU congelado: descongele, vórtice, centrifugue a 10.000 x g por 30 s e use o sobrenadante para injeções abaixo.
- Anestesiar o mouse na caixa de indução (isoflurano a 3%). Observe a marcação, pese os camundongos e calcule o volume da solução IdU:

- Execute a injeção intravenosa usando uma seringa de insulina (28 G, 0,5 mL) e transfira o mouse para uma gaiola de recuperação vazia em uma placa de aquecimento. Quando acordado, mova o mouse de volta para sua gaiola original. Repita as etapas 4.2 a 4.3 para os mouses restantes e devolva-os ao estábulo.
5. Eutanásia
NOTA: Consulte a Tabela 1 para obter receitas de buffer. Prepare o meio de lavagem (mistura de nutrientes F-10 (presunto), soro de cavalo a 10%, 1x caneta / estreptococo) e filtre através de uma membrana de polietersulfona (PES) em um recipiente de poliestireno. Prepare o tampão de dissociação (meio de lavagem suplementado com 650 U / mL de colagenase, tipo II) e mantenha no gelo. As medições de citometria de massa CyTOF são muito sensíveis a contaminantes. Por esse motivo, é essencial usar reagentes do mais alto grau analítico para o processamento de amostras. Para evitar a contaminação por metais, é altamente recomendável usar plásticos estéreis e copos novos que nunca foram lavados com detergente, pois muitos sabões de laboratório contêm altos níveis de bário. Recomenda-se o uso de água duplamente filtrada, destilada e deionizada para a preparação do reagente. A solução salina tamponada com fosfato (PBS) é preparada internamente. Dilua os estoques 10x para 1x e filtre o PBS 1x com filtros de 0,2 μm. Filtre o PBS 1x novamente no início de cada experimento. As ferramentas de dissecação não devem ser limpas com detergente devido à presença de bário.
- Prepare a balança, o recipiente e a unidade anestésica à base de isoflurano com a caixa de indução. Desinfete as ferramentas de dissecação apropriadas.
- Prepare e marque tubos de 50 mL para cada camundongo com 5 mL de tampão de dissociação e uma placa de Petri. Mantenha o tampão de dissociação no gelo.
- Anestesiar o camundongo na caixa de indução (isoflurano a 3%, aumentar para 5% quando inconsciente) e realizar a luxação cervical.
6. Dissecção e dissociação do músculo esquelético
- Disseque os músculos TA e GA de ambos os membros posteriores e transfira para a tampa de uma placa de Petri. Use uma tesoura para cortar o tecido em uma pasta picada (~ 1 mm3 peças). Transfira para um tubo de 50 mL contendo 5 mL de tampão de dissociação gelado. Mantenha no gelo.
- Repita o procedimento de eutanásia (etapa 5.3) e o procedimento de dissecção e dissociação do músculo esquelético (etapa 6.1) até que todos os camundongos tenham sido processados.
- Pré-aqueça todos os tubos de amostra em banho-maria a 37 °C (2 min) e incube por 45 min em rotação em uma incubadora (37 °C).
- Vortex e lave adicionando 10 mL de meio de lavagem. Centrifugue (380 x g, 10 min, temperatura ambiente [RT]) e aspire a 4 mL.
- Adicione 0,5 mL de colagenase (1000 U/mL) e 0,5 mL de dispase (11 U/mL). Vórtice e incubar por 20 min em rotação a 37 °C (por exemplo, em uma incubadora). Centrifugue (380 x g, 1 min, RT) e ressuspenda com uma pipeta de 5 mL.
- Adicione filtros de células de 40 μm a tubos de 50 mL e pré-umedeça com 5 mL de meio de lavagem. Aspire e ejete a suspensão celular 10 vezes usando uma seringa de 5 mL com uma agulha de 20 G. Coe através do filtro de células pré-umedecido de 40 μm. Lave o tubo de 50 ml com um meio de lavagem de 10 ml, transfira-o para o filtro de células de 40 μm e centrifugue (380 x g, 10 min, 4 °C) a suspensão celular.
7. Coloração viva/morta com fixação de cisplatina e paraformaldeído
CUIDADO: A cisplatina e o paraformaldeído (PFA) são cancerígenos. Leia a FISPQ antes de manusear. O paraformaldeído (PFA; 16%) é um irritante para a pele, os olhos e as vias respiratórias. Use equipamento de proteção individual e manuseie essas substâncias sob uma hotte. Durante a fixação das células, a concentração final de PFA será de 1,6%. Medidas de proteção corretas devem ser tomadas e os resíduos devem ser manuseados de acordo com os regulamentos locais.
NOTA: Prepare DMEM sem soro frio (4 ° C) e quente (37 ° C). Prepare o DMEM suplementado com 10% de FBS, filtre através de uma membrana PES em um recipiente de poliestireno e mantenha no gelo. Prepare PBS e meios de coloração celular (CSM; PBS, 0,5% BSA, 0,02% azida de sódio) em um frasco de vidro dedicado a CyTOF e filtre através de uma membrana PES. O CSM pode ser armazenado a 4 °C por até 6 meses.
- Aspire o sobrenadante, sacuda o pellet e ressuspenda em 1 mL de DMEM sem soro frio. Conte as células e ajuste a densidade celular para 1 x 106 células/mL em DMEM sem soro.
- Adicione estoque de cisplatina (25 mM) a uma concentração final de 25 μM. Vórtice 10 s e incube por exatamente 1 min (a reação é muito sensível ao tempo). Extinguir a reação com DMEM gelado + 10% FBS (3 vezes o volume da amostra) e mantê-lo no gelo. Centrifugar (380 x g, 10 min, 4 °C), aspirar o sobrenadante e ressuspender completamente o sedimento (até 10 x 106 células/ml em CSM). Filtrar a suspensão através de um filtro de células de 35 μm.
- Fixe a suspensão celular com solução estoque de PFA filtrada (16%) e pipete para cima e para baixo (execute sob uma capela de exaustão) para atingir a concentração final de PFA (1,6%) (por exemplo, adicione 100 μL de PFA a 16% a 900 μL de suspensão celular). Vortex por 30 s, incubar por 10 min em gelo e lavar duas vezes com 2 mL de CSM por centrifugação (800 x g, 5 min, RT).
NOTA: Neste ponto, as amostras podem ser congeladas em gelo seco e armazenadas a -80 °C ou usadas diretamente para coloração de anticorpos. Se as células forem congeladas, transfira as células para tubos de polipropileno de 5 mL, pois o poliestireno pode rachar em baixas temperaturas. Se células recém-fixadas forem usadas para coloração, prossiga para a etapa 8.2.
8. Coloração com anticorpos conjugados com metal
CUIDADO: O metanol (MeOH) é altamente inflamável e corrosivo para o trato respiratório. Leia a FISPQ antes de manusear. Use equipamento de proteção individual e manuseie esta substância sob um exaustor. Manuseie os resíduos de acordo com os regulamentos locais.
NOTA: A lista de anticorpos (Ab) direcionados a marcadores de superfície e marcadores intracelulares pode ser encontrada na Tabela 2.
Conjugação de anticorpos: A maioria dos anticorpos usados neste protocolo foram conjugados internamente porque não estavam disponíveis comercialmente. Protocolos para conjugação metálica de anticorpos foram publicados anteriormente, e os kits de conjugação estão agora disponíveis comercialmente37,38. A imunoglobulina tipo G (IgG) é compatível com os protocolos de conjugação disponíveis. É de grande importância que a formulação de anticorpos usada para conjugação de metais esteja livre de proteínas transportadoras contendo cisteína (por exemplo, albumina de soro bovino (BSA)), que podem afetar a eficiência da conjugação competindo pelos grupos maleimida livres do polímero e podem interferir na quantificação do anticorpo conjugado com metal. O teor de cisteína da gelatina é muito menor do que o da BSA. No entanto, recomenda-se que, se a formulação de anticorpos contiver proteínas transportadoras, essas proteínas sejam removidas antes da conjugação. Agora é possível solicitar anticorpos livres de BSA e gelatina do fabricante. Conservantes de moléculas pequenas (por exemplo, azida sódica, glicerol e trealose) são compatíveis com protocolos de conjugação de metais37,38.
Titulação de anticorpos: Após cada conjugação de metais, os anticorpos devem ser titulados para determinar a concentração ideal de anticorpos que fornece a relação sinal-ruído máxima. Para titulação de anticorpos, execute uma diluição serial dupla de 6 etapas e core ambas as amostras conhecidas por expressar (por exemplo, células musculares, controles positivos) e não (controles negativos) a proteína de interesse 19,21,37,38.
Prepare uma nova solução de trabalho Cell-ID Intercalator-Ir (estoque = 500 μM; solução intercalator-ir) diluindo o estoque a 0,1 μM em PBS / 1,6% PFA.
- Descongele as amostras congeladas por 5 min em RT e centrifugue (800 x g, 1 min, RT). Lavar com 2 mL de CSM por centrifugação (800 x g, 5 min, RT).
- Prepare uma mistura de coloração de anticorpos de superfície (Ab) 2,5x em CSM. Aspirar o sobrenadante a ~60 μL e ressuspender completamente o sedimento. Pinte as células adicionando 40 μL de mistura de coloração Ab superficial e incubando-a por 1 h em RT.
- Agite as amostras a cada 20 minutos para misturar. Lavar duas vezes com 1 ml de CSM por centrifugação (800 x g, 5 min, RT). Aspirar o sobrenadante e sacudir o projétil.
NOTA: Se nenhuma coloração intracelular for necessária, a permeabilização do metanol não é necessária. No entanto, se apenas a coloração da superfície for realizada, a solução intercalator-ir deve ser diluída em um tampão contendo um agente permeabilizante (por exemplo, Maxpar fix e tampão permanente) para penetrar na membrana nuclear. Prossiga para a etapa 8.5 e dilua o Cell-ID Intercalator-Ir em um tampão contendo um agente permeabilizante e 1.6% de PFA. Caso contrário, continue com a coloração intracelular abaixo.
CUIDADO: O irídio é perigoso e devem ser tomadas precauções para um manuseio seguro. O irídio é inflamável e irritante para os olhos/pele. Evite criar e respirar poeira ou fumaça. Evite o contato com a pele e os olhos. No entanto, a mistura de solução intercalator-ir é fornecida na concentração de <1% em água, que é considerada não perigosa de acordo com o sistema globalmente harmonizado (GHS) de classificação e rotulagem de produtos químicos. Leia o SDS da Standard Biotools antes de manusear. Equipamento de proteção individual deve ser usado durante o manuseio. Os materiais que estiveram em contato com a solução intercalator-ir devem ser descartados de acordo com os regulamentos de segurança locais. - Para permeabilizar as células, adicione 0,5 mL de MeOH gelado gota a gota durante o vórtice. Incubar durante 15 min em gelo sob uma hotte. Centrifugue (800 x g, 5 min, RT) e depois lave duas vezes com 1 mL de CSM por centrifugação (800 x g, 5 min, RT). Após a última lavagem, aspirar o sobrenadante a ~60 μL e ressuspender completamente o sedimento.
- Prepare uma mistura de coloração de Ab intracelular 2.5x em CSM. Pinte as células adicionando 40 μL de mistura de coloração Ab intracelular e incubando por 1 h em RT. Agite as amostras a cada 20 minutos para misturar.
- Lavar duas vezes com 1 ml de CSM por centrifugação (800 x g, 5 min, RT). Aspirar o sobrenadante e sacudir o projétil. Ressuspender as amostras em 0,5 mL de solução de intercalador-irídio (Tabela 1) e vórtice. Incubar as amostras durante 1 h em RT ou durante a noite (O/N) a 4 °C (ver nota abaixo).
NOTA: As amostras podem ser armazenadas em solução intercalator-irídio a 4 °C por até 48 h. O protocolo de coloração utilizado neste estudo foi desenvolvido com base no trabalho pioneiro do laboratório de Nolan{C}23. Ele difere dos protocolos da Standard Biotools, pois (i) tampões preparados internamente são usados para lavagem e coloração, (ii) as células são fixadas antes da coloração da superfície e (iii) as células são permeabilizadas com metanol para coloração intracelular com anticorpos para fatores de transcrição ou moléculas de sinalização. Os pesquisadores que desenvolvem um novo protocolo devem testar minuciosamente a compatibilidade de seu painel de anticorpos com o metanol como agente de permeabilização. Ao adicionar novos anticorpos a um painel, recomenda-se testar a especificidade do anticorpo por citometria de fluxo usando amostras de controle positivo e negativo antes da conjugação e titulação do metal.
9. Preparação da amostra para carregamento no citômetro de massa
NOTA: Os pellets de células ficam muito soltos quando em tampão CAS (Tabela de Materiais). Durante as lavagens com tampão CAS, não aspire ao ressecamento. Em vez disso, mantenha um volume residual conforme descrito abaixo.
- Vórtice e centrifugação (800 x g, 10 min, RT) as amostras. Despeje o sobrenadante (manuseie como resíduo de PFA) e o vórtice.
- Lavar com 1 ml de CSM por centrifugação (800 x g, 10 min, RT). Aspirar o sobrenadante e sacudir o projétil.
- Lavar com 1 ml de tampão CAS por centrifugação (800 x g, 10 min, RT). Aspire a ~ 200 μL. Vórtice e adicione 1 mL de tampão CAS. Retirar alíquota de 5 μL para contagem de células. Centrifugue (800 x g, 10 min, RT) e aspire cuidadosamente a ≤50 μL. Não perturbe o pellet.
- Ressuspenda o pellet a 1–2 x 106 células/mL em tampão CAS e adicione esferas de calibração (1x; Tabela de Materiais) a uma concentração final de 0,1x (por exemplo, adicione 100 μL de grânulos de calibração 1x a 900 μL de suspensão celular).
- Carregue a amostra no citômetro de massa e colete dados usando uma taxa de fluxo de 400–500 células/s.
- Realizar a normalização de dados de arquivos FCS usando o software CyTOF ou ferramentas de normalização desenvolvidas anteriormente39.
NOTA: A operação dos citômetros de massa CyTOF é específica do instrumento40,41. Recomenda-se consultar o manual do usuário CyTOF antes da operação. Os grânulos de calibração são padrões de grânulos de normalização de poliestireno embutidos em metal contendo concentrações conhecidas dos isótopos metálicos cério (140/142Ce), európio (151/153Eu), hólmio (165Ho) e lutécio (175/176Lu). As esferas de calibração permitem o controle da sensibilidade da máquina, que pode variar ao longo do tempo, principalmente devido ao acúmulo de material biológico e variações na ionização do plasma ao longo do tempo.
10. Análise de dados CyTOF
NOTA: Para análise downstream, os arquivos FCS normalizados podem ser analisados localmente ou carregados em soluções de software baseadas em nuvem, como Cytobank, Cell Engine, OMIQ ou FCS Express42.
- Concatene arquivos FCS individuais para cada exemplo em um único arquivo, se necessário.
- Identifique células individuais por meio de eventos positivos do intercalador de irídio, o que permite a discriminação de células nucleadas únicas de detritos ou dupletos.
- Portão para células vivas selecionando eventos negativos de cisplatina. A cisplatina liga-se covalentemente às proteínas celulares e marca as células moribundas e mortas com membranas comprometidas em maior extensão do que as células vivas43.
- Gate na população de interesse, por exemplo, o compartimento miogênico (Live / CD45- / CD31- / Sca1- / α7integrina + / CD9 +) (Figura 2A) e quantifique a proporção relativa de células-tronco e progenitoras. Essa abordagem requer conhecimento prévio da superfície celular ou expressão de marcadores intracelulares para definir populações individuais.
- Para realizar análises de alta dimensão que permitirão a identificação de subconjuntos de células raras anteriormente não reconhecidos dentro de uma população complexa, exportar a população de interesse e usar algoritmos de agrupamento que foram desenvolvidos especificamente para análise de dados CyTOF44.
NOTA: Trabalhos anteriores empregaram o algoritmo X-shift, que usa estimativa ponderada da densidade do vizinho mais próximo (kNN-DE) em espaço de alta dimensão para realizar agrupamento não supervisionado com base em parâmetros definidos19. O X-shift demonstrou ser altamente eficaz na identificação de populações de células raras45. - Para análise de X-shift, baixe o pacote de software de vórtice (da página do GitHub do laboratório Nolan [https://github.com/nolanlab/vortex]) e Java 64-bit46. As instruções podem ser encontradas aqui: https://github.com/nolanlab/vortex/wiki/Getting-Started.
- Carregue as populações de células exportadas para um banco de dados local e defina os parâmetros de agrupamento. Entre os parâmetros definidos pelo usuário estão (i) os marcadores usados para agrupamento, (ii) o intervalo de valores k (por exemplo, 5 a 150) e (iii) o número de etapas de agrupamento.
NOTA: Trabalhos anteriores usaram, como marcadores de agrupamento, uma combinação de marcadores de superfície conhecidos por serem expressos em células-tronco musculares, novos marcadores de superfície identificados em uma triagem citométrica de fluxo de alto rendimento de células musculares e mioblastos e fatores de transcrição miogênicos (TFs) conhecidos por definir diferentes estágios da miogênese. Essa abordagem permitiu a identificação de dois marcadores de superfície celular, CD9 e CD104, cujo padrão de co-expressão distingue populações de células progenitoras anteriormente não reconhecidas19. A escolha cuidadosa dos marcadores de agrupamento permitirá que o pesquisador responda a perguntas específicas de pesquisa. Por exemplo, se a identificação e análise ao longo do tempo de subconjuntos de células em divisão for desejada, recomenda-se incluir IdU como um dos marcadores de agrupamento. O X-shift pode realizar clustering em vários valores k e identificar automaticamente, calculando o "ponto de cotovelo", o valor k que resulta no número ideal de cluster, evitando assim subclustering ou fragmentação excessiva. Recomenda-se calcular o ponto de cotovelo e usar a análise de agrupamento definida pelo valor ideal de k para visualização de dados e aplicativos downstream. - Para visualizar as relações espaciais entre as populações de células dentro dos aglomerados X-shift, execute um layout direcionado por força, que gerará um mapa 2D onde a distância entre os aglomerados indica sua semelhança na expressão do marcador no espaço fenotípico de alta dimensão. Ao colorir o mapa usando um marcador de cada vez, é possível descobrir novas populações de células e acompanhar sua dinâmica. Recomenda-se a quantificação de acompanhamento por gating manual em software, como Cytobank e Cell Engine.
- Realize análises de mapa de calor para quantificar a expressão dinâmica de várias proteínas de superfície e intracelulares ao longo do curso do tempo e revelar novas tendências.
NOTA: Ao realizar um curso de tempo de lesão, é possível agrupar dados de todos os pontos de tempo para gerar um mapa de regeneração. Além disso, é possível dissecar esse mapa em pontos de tempo individuais para acompanhar a dinâmica celular e molecular da regeneração ao longo do tempo19.
11. Coloração com anticorpos conjugados com fluoróforo para FACS
NOTA: As células usadas para controles de cor única não corados e controles de fluorescência menos um (FMO) podem se originar do conjunto TA e GA de um mouse extra, se disponível. Alternativamente, o quadríceps (músculo anterior superior da coxa) pode ser dissecado e digerido em uma suspensão de célula única, seguindo o mesmo procedimento do conjunto TA+GA acima e usado para controles. Prepare o tampão FACS (PBS, soro de cabra a 2,5%, EDTA 2 mM), filtre através de uma membrana PES em um recipiente de poliestireno e mantenha no gelo. O buffer FACS pode ser armazenado a 4 °C por até 1 mês. Uma lista de anticorpos usados para FACS pode ser encontrada na Tabela 3.
- Prepare uma mistura de linhagem. Manchar cada controle em 50 μL (contendo aprox. 3–5 x 105 células). Calcule a quantidade de mistura de linhagem necessária. Faça 20% a 30% de excesso de volume. A concentração de estoque de anticorpos usados para a mistura de linhagens é de 0,2 mg/mL.
- Em um tubo de 0,5 mL, adicione anti-CD45 APC-Cy7, anti-CD31 APC-Cy7, anti-Sca1 APC-Cy7 e anti-CD11b APC-Cy7 para atingir concentrações finais de 1 μg/mL, 2,5 μg/mL, 2,5 μg/mL e 0,63 μg/mL, respectivamente.
- Prepare a mistura para todas as manchas. Manchar cada amostra totalmente corada em 500 μL (contendo aprox. 3–5 x 106 células). Faça 10% de excesso de volume de mistura de todas as manchas.
- Em um tubo de 1,5 mL, adicione anticorpos de mistura de linhagem como acima + anti-α7 integrina PE, anti-CD9 APC e anti-CD104 FITC para atingir concentrações finais de 2 μg/mL, 1,2 μg/mL e 3 μg/mL, respectivamente.
- Prepare o tampão FACS + DAPI: Para 10 mL de tampão FACS, adicione 1 μL de DAPI (estoque = 1 mg/mL) para atingir uma concentração final de 100 ng/mL de DAPI.
CUIDADO: DAPI (dicloridrato de 4',6-diamidino-2-fenilindol) é classificado como um possível irritante cutâneo e respiratório. No entanto, a solução de DAPI na concentração de <1% é considerada não perigosa, de acordo com o GHS. Leia a FISPQ antes de manusear. Equipamento de proteção individual deve ser usado durante o manuseio. Os materiais que estiveram em contato com o DAPI devem ser descartados de acordo com os regulamentos de segurança locais. - Ressuspender células da seção 6: Dissecção e dissociação do músculo esquelético:
- Para todas as amostras coradas, ressuspenda os conjuntos TA e GA de um único camundongo em 500 μL de tampão FACS e transfira-o para tubos de 15 mL para coloração.
- Para controles, use conjuntos TA e GA ou conjuntos de quadríceps. Se estiver usando 1 mouse para controles, ressuspenda as células em 750 μL de tampão FACS. Se vários camundongos, ressuspenda cada conjunto TA e GA ou quadríceps em 500 μL de tampão FACS e combine os conjuntos em uma única amostra de 1 mL retirando uma fração de cada amostra. Adicione 50 μL de células de controle (3–5 x 105 células) a tubos de polipropileno de 5 mL para coloração.
- Manchar conforme descrito na Tabela 4 por 45 min a 4 ° C no escuro.
- Lave todas as amostras coradas adicionando 5 mL de tampão FACS. Lave os controles adicionando 1 mL de tampão FACS e centrífuga (380 x g, 10 min, 4 °C).
- Ressuspenda todas as amostras coradas em 1 mL de tampão FACS + DAPI. Ressuspenda os controles em 300 μL de buffer FACS com ou sem DAPI.
- Manter as células a 4 °C na obscuridade até serem classificadas num citómetro de fluxo com 4 lasers (405 nm, 488 nm, 561 nm, 633 nm).
- Execute os controles e amostras no classificador e crie portões de classificação usando o software relevante. Para controles sem coloração/cor única e controles FMO, registre 1 x 104 e 0,3–1 x 105 eventos, respectivamente. Para amostras totalmente coradas, registre até 1 x 106 eventos. Classifique todas as amostras coradas, ou quantas forem necessárias, dependendo dos requisitos do ensaio a jusante. Para o citómetro de fluxo utilizado aqui (FACSAria III), classificar em modo de pureza utilizando um bico de 70 mm.
- Consulte a Figura 4 para obter a estratégia de gating.
- Use os controles sem coloração e de cor única para definir a tensão para todos os detectores.
- Use os controles de cor única para configurar a matriz de compensação.
- Use os controles FMO para estabelecer portões de classificação.
- Populações distintas de células-tronco musculares e progenitoras são classificadas em tampão FACS gelado.
- Centrifugar (380 x g, 10 min, 4 °C) as populações celulares. Ressuspenda no buffer apropriado, conte e continue com a análise downstream.
Resultados
Aqui, apresentamos uma visão geral da configuração experimental para usar essa abordagem combinada, que inclui (i) análise CyTOF de alta dimensão de um curso de tempo de lesão aguda por injeção de notexina para estudar a dinâmica celular e molecular de células-tronco e progenitoras no músculo esquelético (Figura 1, esquema superior); e (ii) FACS de células-tronco e progenitoras usando dois marcadores de superfície celular, CD9 e CD104, para isolar essas populações e realizar estudos aprofundados de sua função (Figura 1, esquema inferior). Camundongos C57BL/6 fêmeas de 8 a 10 semanas de idade foram usados nos experimentos descritos abaixo.
Análise CyTOF do músculo esquelético para identificação de células-tronco e progenitoras e estudo de sua dinâmica celular e molecular durante o curso da lesão aguda
Reanalisamos conjuntos de dados CyTOF publicamente disponíveis e publicados anteriormente de um curso de tempo de lesão aguda por injeção de notexina, onde amostras de músculo esquelético foram coradas com um painel de 21 anticorpos compatíveis com CyTOF conjugados com metal19 (Figura 2 e Figura 3). Aqui apresentamos a estratégia de gating hierárquico para a identificação de células-tronco e progenitoras in vivo no músculo esquelético (Figura 2A). Essa abordagem identifica uma sequência de 4 populações, células-tronco (SC) e populações progenitoras 1-3 (P1, P2, P3). As populações progenitoras P1 e P2 correspondem a células progenitoras cada vez mais maduras. P3 foi previamente identificado, mas não caracterizado devido à sua baixa abundância.
Primeiro, identificamos as células por meio de eventos duplos positivos Ir191 / Ir193, o que nos permite discriminar células uninucleadas de detritos ou dublês devido às propriedades intercalantes de ácidos nucleicos desse reagente47. Em seguida, identificamos células vivas por meio de eventos negativos à cisplatina, uma vez que a cisplatina é uma substância química que se liga covalentemente a proteínas celulares e rotula células com membranas celulares comprometidas (células mortas e mortas) em maior extensão do que as células vivas43. Enriquecemos ainda mais as células miogênicas excluindo células imunes (CD45+, CD11b+), células endoteliais (CD31+) e células mesenquimais (Sca1+) e vinculando-se a α7 integrina+/CD9+ células miogênicas16,19. As células-tronco e progenitoras são então identificadas visualizando a expressão de CD9 e CD104 dentro da porta celular miogênica α7 integrina + / CD9 + usando um gráfico de pontos biaxiais CD9 (eixo y) por CD104 (eixo x). As células-tronco (CT) expressam níveis intermediários de CD9 e não expressam CD104 (quadrante inferior esquerdo); As células progenitoras P1 expressam altos níveis de CD9 e não expressam CD104 (quadrante superior esquerdo); As células progenitoras P2 expressam altos níveis de CD9 e CD104 (quadrante superior direito), as células progenitoras P3 expressam níveis intermediários de CD9 e níveis intermediários a altos de CD104 (quadrante inferior direito) (Figura 2A). O painel CyTOF usado no experimento aqui apresentado incluiu anticorpos para marcadores de superfície celular, fatores de transcrição miogênicos intracelulares e moléculas de sinalização (Figura 2B).
Para visualizar a expressão dos fatores de transcrição miogênicos dentro das diferentes populações de células-tronco e progenitoras, mostramos um gráfico de pontos biaxiais CD9 por CD104 representativo de uma amostra de músculo não lesado, colorido na terceira dimensão pela expressão de Pax7, Myf5, MyoD e MyoG, bem como CD9 e CD104 para destacar sua expressão diferencial dentro das diferentes populações (Figura 2C).
Para visualizar a dinâmica celular, molecular e de proliferação de populações de células-tronco e progenitoras durante o curso temporal da lesão aguda, mostramos gráficos de pontos biaxiais CD9 por CD104 representativos em diferentes pontos de tempo de lesão (Dia 0, Dia 3, Dia 6), coloridos na terceira dimensão pela incorporação de IdU (esquerda), que permitem a identificação de células na fase S, ou expressão de MyoD (à direita), um fator de transcrição que é regulado positivamente em células-tronco ativadas que entraram no ciclo celular (Figura 3A). Conforme descrito anteriormente, a população de células-tronco (CT) aumenta proporcionalmente no dia 3 pós-lesão (seta vermelha), sugerindo expansão, o que é corroborado por um grande aumento na incorporação de IdU, indicando proliferação celular (Figura 3A, esquerda). Esse aumento na incorporação de IdU é acompanhado pelo aumento da expressão de MyoD (Figura 3A, à direita).
Dada a definição molecular bem estabelecida de células-tronco musculares ativadas (MuSCs; altamente proliferativas eMyoD altas), a análise CyTOF de alta dimensão de células musculares investigando a expressão de marcadores de superfície celular conhecidos e novos, fatores de transcrição miogênicos e marcadores da fase S, permitiu a identificação de uma assinatura de células-tronco ativadas com base apenas em marcadores de superfície celular19, 48,49,50. Essa assinatura, que se baseia na co-expressão dos marcadores de superfície celular CD98 e CD44, agora permite a identificação e o isolamento prospectivo desse subconjunto MuSC transitório no dia 3 pós-lesão, abrindo as portas para estudos de auto-renovação de células-tronco que antes eram inviáveis19. Para visualizar o subconjunto de células-tronco ativadas, mostramos gráficos de pontos biaxiais CD98 (eixo y) representativos por CD44 (eixo x) da população SC em diferentes pontos de tempo de lesão (Dia 0, Dia 3, Dia 6), coloridos na terceira dimensão pela incorporação de IdU (esquerda) ou expressão de MyoD (direita). As células-tronco ativadas, definidas pela co-expressão de altos níveis de marcadores de superfície celular CD98 e CD44, bem como alta expressão de MyoD e alta incorporação de IdU, são indicadas por uma seta vermelha (Figura 3B). Esses dados destacam o alto estado proliferativo e o fenótipo ativado dos MuSCs no dia 3 pós-lesão.
Purificação de células-tronco e progenitoras por FACS
A estratégia de gating usada para isolar populações de células-tronco musculares e progenitoras por FACS é mostrada na Figura 4. O portão principal é usado para excluir detritos. A porta singlets permite a exclusão de gibões51. A porta de células vivas exclui células mortas e mortas que se coram com DAPI, e a porta de linhagem negativa exclui células imunes (CD45+/CD11b+), células endoteliais (CD31+) e células mesenquimais (Sca1+), que são coradas com anticorpos individuais marcados com o mesmo fluoróforo APC-Cy7. As células dentro do compartimento miogênico são controladas com base na expressão da integrina α7 e níveis intermediários a altos de expressão de CD9. As populações de células-tronco musculares e progenitoras são identificadas com base na expressão diferencial de CD9 e CD104 (Figura 4A), e sua proporção relativa é quantificada (Figura 4B). Cada porta é estabelecida usando controles FMO apropriados, que levam em consideração o sinal de fundo gerado pela combinação dos anticorpos conjugados com fluoróforo usados nesse controle (Figura 4C). A população SC é definida com base na expressão de níveis intermediários de CD9 e na falta de expressão de CD104 (CD9int/CD104-). Enquanto P1 e P2 expressam altos níveis de CD9, CD104 não é expresso em células progenitoras P1 (CD9alto / CD104-), mas é altamente expresso em células progenitoras P2 (CD9alto / CD104alto).

Figura 1: Abordagem combinada para analisar e purificar células-tronco musculares e progenitoras no contexto de lesão muscular aguda. Esquema de protocolo. (Esquema superior) Investigação da dinâmica das células-tronco e progenitoras por análise CyTOF de alta dimensão do músculo esquelético no contexto de lesão aguda. A configuração experimental do curso do tempo de lesão requer injeção intramuscular (i.m.) de notexina no músculo tibial anterior (TA) e gastrocnêmio (GA) de camundongos 6 (dia -6) ou 3 (dia -3) dias antes da coleta do tecido. A injeção de IdU é realizada por via intraperitoneal (i.p.) 8 h antes (-8 h) da colheita do tecido para detectar células em proliferação. Os músculos TA e GA são dissecados e o tecido é dissociado por uma combinação de dissociação mecânica e digestão enzimática. As células são então incubadas com cisplatina para detectar células mortas e mortas, fixadas com paraformaldeído para preservar a expressão do marcador, coradas com anticorpos conjugados com metal e permeabilizadas com metanol para coloração intracelular. As células são finalmente analisadas no instrumento CyTOF. Após a análise do CyTOF e a aquisição de eventos, os dados são normalizados, concatenados e armazenados usando servidores como Cytobank ou Cell Engine42. Os dados podem ser analisados usando (i) gráficos biaxiais tradicionais e (ii) algoritmos de agrupamento de alta dimensão, como X-shift, um algoritmo baseado em k-vizinho mais próximo47. Os aglomerados de células podem ser visualizados por visualização de layout direcionada à força de célula única, conforme descrito anteriormente19. (Esquema inferior) FACS de células-tronco e progenitoras. Os músculos TA e GA de camundongos não lesionados (homeostase) são dissecados e dissociados como acima. As células são coradas com anticorpos para marcadores de linhagem (CD45, CD11b, CD31 e Sca1), marcadores de superfície celular usados para definir populações de células-tronco musculares e células progenitoras (integrina α7 , CD9 e CD104) e DAPI para determinar a viabilidade. As células são analisadas no instrumento FACS para estabelecer a estratégia de classificação e classificadas em populações distintas de células-tronco musculares e progenitoras com base na expressão diferencial dos marcadores de superfície celular CD9 e CD104. Clique aqui para ver uma versão maior desta figura.
Figura 2: Identificação de células-tronco e progenitoras usando citometria de massa de célula única (CyTOF). (A) Estratégia de gating CyTOF para identificar células-tronco musculares e progenitoras. As células são diferenciadas de detritos e dupletos por coloração com um intercalador de ácido nucleico catiônico (Cell-ID Intercalator-Ir) e gating em eventos duplos positivos Ir191 / Ir193. As células vivas são então identificadas com base na falta de coloração com cisplatina, que rotula células com membranas celulares comprometidas (células mortas e mortas) em maior extensão do que as células vivas. Células imunes (CD45+ e CD11b+), células endoteliais (CD31+) e células mesenquimais (Sca1+) são excluídas com base na expressão dos respectivos marcadores. O compartimento miogênico é identificado pela co-expressão de integrina α7 e CD9. As células-tronco e progenitoras são diferenciadas pela expressão diferencial de CD9 e CD104 em um gráfico de pontos biaxiais CD9 (eixo y) por CD104 (eixo x) de α7 integrina + / CD9 + . (B) Resumo do painel de marcadores CyTOF exibindo anticorpos para marcadores de superfície celular, fatores de transcrição intracelular, moléculas de sinalização fosforiladas e reagentes para coloração de DNA (Cell-ID Intercalator-Ir), coloração de viabilidade (cisplatina) e proliferação celular (IdU). (C) Gráficos de pontos biaxiais CD9 representativos (eixo y) por CD104 (eixo x) do compartimento miogênico (α7 células integrina + / CD9 + ) de camundongos não lesionados coloridos pela expressão dos fatores de transcrição Pax7, Myf5, MyoD e MyoG e os marcadores de superfície celular CD9 e CD104. A expressão dos marcadores individuais nas populações de caule e progenitoras revela uma progressão miogênica. Clique aqui para ver uma versão maior desta figura.

Figura 3: Resolvendo a dinâmica celular e molecular do tronco muscular e das células progenitoras durante um curso de tempo de lesão aguda. (A) Gráficos de pontos biaxiais CD9 representativos (eixo y) por CD104 (eixo x) do compartimento miogênico (α7 células integrina + / CD9 + ) durante o curso do tempo de lesão (Dia 0, Dia 3, Dia 6), coloridos pela incorporação de IdU (painéis esquerdos) e expressão de MyoD (painéis direitos). A expressão dos marcadores individuais nas populações de caule e progenitoras revela uma progressão miogênica. A seta vermelha indica a expansão da população de células-tronco (SC) no dia 3 pós-lesão. (B) (Esquerda) A população de células-tronco musculares (SC) é destacada em vermelho em um esquema que descreve um gráfico biaxial CD9 (eixo y) por CD104 (eixo x). (Direita) Gráficos de pontos biaxiais representativos de CD98 (eixo y) por CD44 (eixo x) da população de células-tronco (SC) durante o curso do tempo de lesão (Dia 0, Dia 3, Dia 6), coloridos pela incorporação de IdU (painéis esquerdos) e expressão de MyoD (painéis direitos). A co-expressão dos marcadores de superfície celular CD98 e CD44 identifica um subconjunto de células-tronco ativadas no dia 3 pós-lesão (seta vermelha), que é marcado por alta incorporação de IdU (painéis esquerdos) e aumento da expressão de MyoD (painéis direitos). Clique aqui para ver uma versão maior desta figura.
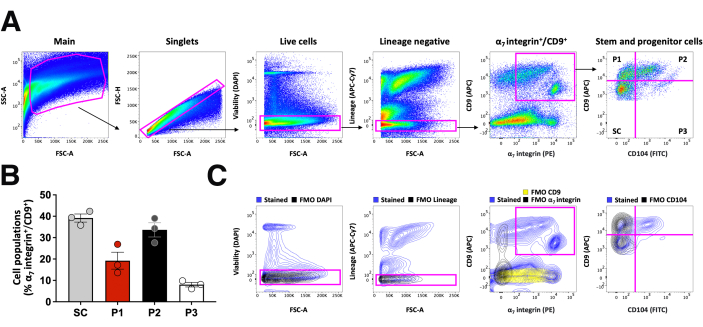
Figura 4: Classificação de células-tronco e progenitoras para ensaios a jusante usando uma estratégia única definida pelos marcadores de superfície celular CD9 e CD104. (A) Estratégia de gating hierárquica usada para identificar células-tronco e progenitoras durante uma classificação. O portão principal, baseado no FSC e SSC, exclui detritos. A porta singlets baseada em FSC-Area e FSC-Height é usada para excluir dublês. A porta de linhagem é usada para excluir células imunes (CD45+ e CD11b+), células endoteliais (CD31+) e células mesenquimais (Sca1+), coradas com anticorpos conjugados ao mesmo fluoróforo, APC-Cy7. A porta integrina+/CD9+ do α7 é usada para definir as células dentro do compartimento miogênico. As células-tronco e progenitoras dentro do compartimento miogênico são distinguidas com base na expressão diferencial dos marcadores de superfície celular CD9 e CD104. (B) Quantificação de células-tronco e progenitoras como uma fração de α7 células integrina + / CD9 +. (C) Os controles de fluorescência menos um (FMO) são usados para estabelecer portões positivos para as diferentes populações na estratégia de gating hierárquico (A). Clique aqui para ver uma versão maior desta figura.
Tabela 1: Tabela de receitas de buffer. Tipo de tampão, conteúdo e descrição da preparação. Clique aqui para baixar esta tabela.
Tabela 2: Anticorpos e reagentes usados para análise CyTOF. Veja também a Figura 2B. A maioria dos anticorpos usados para CyTOF foi conjugada internamente com os metais indicados. Clique aqui para baixar esta tabela.
Tabela 3: Anticorpos usados para FACS de células-tronco e progenitoras. Clique aqui para baixar esta tabela.
Tabela 4: Esquema de coloração usado para FACS. As amostras são categorizadas em controles de cor não corados, com coloração única (único), controles FMO (FMO) e amostras totalmente coradas. A concentração de anticorpos usada no experimento atual é fornecida. É importante notar que a adição de DAPI é realizada durante a ressuspensão celular final. Clique aqui para baixar esta tabela.
Discussão
A regeneração do músculo esquelético é um processo dinâmico que depende da função das células-tronco adultas. Embora estudos anteriores tenham se concentrado no papel das células-tronco musculares durante a regeneração, sua progênie in vivo foi pouco estudada, principalmente devido à falta de ferramentas para identificar e isolar essas populações de células 15,16,17,18. Aqui, apresentamos um método para identificar e isolar simultaneamente células-tronco musculares e células progenitoras in vivo no músculo esquelético e resolver sua dinâmica celular e molecular no contexto de lesão muscular aguda19.
A abordagem apresentada aqui, que combina análise CyTOF de alta dimensão com classificação celular, fornece ferramentas para obter insights mecanicistas sobre como as transições de estado celular impulsionam a regeneração eficaz do tecido e revelam alterações na função das células-tronco durante o envelhecimento e doenças musculares52. Mudanças na proporção relativa das diferentes populações em modelos genéticos de camundongos ou na intervenção farmacológica podem fornecer insights mecanicistas sobre o processo de miogênese e elucidar o papel de moléculas individuais e vias de sinalização a jusante na auto-renovação ou diferenciação de células-tronco. Tais mudanças precisam ser cuidadosamente avaliadas porque podem resultar de alterações na quiescência, sobrevivência celular, tempo de progressão das células-tronco para as células progenitoras ou acúmulo de um subconjunto sobre outro devido a um bloqueio de diferenciação, que é característico das células cancerígenas 53,54,55,56. O poder da análise CyTOF é a capacidade de detectar até 40 proteínas diferentes de superfície celular e intracelulares simultaneamente em uma única célula, permitindo o estudo do status do ciclo celular, danos ao DNA e modificações pós-traducionais, para citar alguns21. É importante ressaltar que a análise CyTOF permite a fácil detecção de IdU sem a necessidade de coloração intracelular, o que pode afetar a detecção de antígenos de superfície19,20. Este método, portanto, oferece a oportunidade de distinguir diferentes mecanismos pelos quais a função das células-tronco é modulada por lesão ou doença, pois permite a investigação da função celular e alterações moleculares no tecido muscular recém-isolado, como sobrevivência celular, status do ciclo celular, sinalização intracelular e modificações epigenéticas, por detecção simultânea baseada em anticorpos de múltiplas proteínas57. A análise molecular e funcional das células-tronco musculares e sua progênie pode, portanto, ser realizada em uma variedade de contextos biológicos ou tratamentos farmacológicos. É importante ressaltar que a capacidade de purificar simultaneamente células-tronco e progenitoras permite estudos funcionais aprofundados de suas propriedades ex-vivo.
Este protocolo contém etapas críticas que devem ser executadas com planejamento cuidadoso para garantir dados de alta qualidade e reprodutibilidade dos resultados. A injeção de Notexin deve ser realizada com alta precisão para obter resultados reprodutíveis. Além disso, para fins de reprodutibilidade, é importante usar o mesmo lote de notexina e testar a potência da toxina antes de cada experimento58. Além da injeção de uma miotoxina (notexina ou cardiotoxina), o dano muscular esquelético agudo pode ser induzido por lesões mecânicas (contração excêntrica), químicas (por exemplo, cloreto de bário (BaCl2)) e congelantes 58,59,60,61,62,63,64,65. Deve-se notar que a injeção de BaCl2 não é um método de lesão ideal para experimentos com CyTOF porque pode introduzir altos níveis de bário em algumas das amostras, o que pode (i) levar ao transbordamento de sinal em canais adjacentes, (ii) gerar óxidos em canais M + 16 e (iii) danificar o detector CyTOF a longo prazo66. A lesão miotóxica geralmente é preferida porque é minimamente invasiva, não requer cirurgia aberta (por exemplo, lesão por congelamento) e pode ser aplicada a vários músculos. A dissociação do músculo esquelético, que emprega digestão mecânica e enzimática em uma suspensão unicelular, também é uma etapa crítica que, se não for realizada de acordo com o protocolo, pode levar à digestão insuficiente ou excessiva do tecido muscular, afetando o rendimento. Os principais fatores no processo de dissociação são a pureza e a atividade das enzimas, a extensão do corte do tecido e o tempo de incubação 18,67.
É importante ressaltar que, para a análise CyTOF, a preparação e o processamento cuidadosos da amostra são essenciais para obter dados de alta qualidade. O procedimento de coloração para determinar a viabilidade, que emprega a coloração com cisplatina, é extremamente sensível ao tempo e precisa ser concluído em 1 minuto43. Uma suspensão de célula única de alta qualidade é fundamental para evitar a formação de agregados durante a fixação e o processamento a jusante. A permeabilização também é uma etapa crítica, e o metanol, o reagente de permeabilização aqui utilizado, pode não ser compatível com a detecção de proteínas intracelulares não descritas neste protocolo 19,23,68,69,70,71,72. Por esse motivo, é importante testar vários reagentes de permeabilização ao adicionar ao painel novos anticorpos para detectar proteínas intracelulares.
As limitações da análise CyTOF incluem (i) a incapacidade de analisar simultaneamente e isolar prospectivamente populações de células; (ii) a necessidade de desenvolver e testar painéis de anticorpos conjugados com metais internos, uma vez que alguns dos anticorpos usados neste protocolo não estão disponíveis comercialmente em formato conjugado com metais (Tabela 2); (iii) o alto custo da instrumentação e dos reagentes 19,66,73. Entre suas diversas vantagens estão (i) a capacidade de analisar simultaneamente até 40 marcadores, o que possibilita estudos aprofundados de amostras biológicas raras; ii) o baixo nível de fundo devido à utilização de elementos de terras raras para conjugação de anticorpos; (iii) a sobreposição mínima entre os canais, devido à alta precisão de resolução de massa; e (iv) a facilidade de investigar a proliferação celular por incorporação de IdU sem a necessidade de coloração intracelular74.
Portanto, esta tecnologia oferece a oportunidade de desenvolver o painel de anticorpos apresentado aqui (por exemplo, incluindo anticorpos para marcadores intracelulares adicionais) para estudar as propriedades moleculares das células-tronco e progenitoras 20,23,75. Ao desenvolver um painel CyTOF atualizado para detectar proteínas adicionais de superfície e/ou intracelulares, é importante, para obter dados de alta qualidade, determinar a especificidade do anticorpo e investigar a reatividade cruzada, que pode ser específica do tecido. Portanto, a adição de novos anticorpos ao painel requer testes extensivos por citometria de fluxo usando amostras de tecido muscular positivas (por exemplo, superexpressão) e negativas (por exemplo, knock out)19,76. Além disso, para maximizar o sinal e minimizar o transbordamento do sinal, é importante escolher isótopos metálicos na faixa de alta sensibilidade (153-176 Da) para a detecção de alvos de baixa abundância, levando em consideração o potencial de oxidação de isótopos mais leves que devem ser escolhidos para detecção de alvos de alta abundância. Finalmente, uma vez que os anticorpos tenham sido testados e conjugados com o metal, é essencial realizar uma titulação para identificar a concentração de anticorpos que fornece a relação sinal/ruído ideal21,66. Embora a construção de um painel CyTOF possa ser demorada, uma vez estabelecido, um novo painel CyTOF pode fornecer insights sem precedentes.
Uma alternativa à análise CyTOF, para reduzir custos e contornar a necessidade do instrumento CyTOF e da conjugação interna de anticorpos, envolverá o projeto de vários painéis menores de anticorpos conjugados com fluoróforo disponíveis comercialmente para realizar a análise tradicional de citometria de fluxo em menor escala. No entanto, tal abordagem não seria adequada para analisar amostras preciosas com disponibilidade limitada (por exemplo, animais raros knock-out ou transgênicos).
Ao realizar citometria de fluxo multiparamétrica ou classificação de células, é fundamental incluir controles de cor única para estabelecer uma matriz de compensação e controles FMO para estabelecer o fundo para populações individuais e / ou subconjuntos 77,78,79 (Figura 4C). Os controles FMO, que exibem, além da autofluorescência, o sinal de fundo gerado por todos os outros anticorpos conjugados com fluoróforo, são essenciais para estabelecer portas positivas que definem as populações de células em uma estratégia de gating hierárquica. Experimentos de classificação destinados a purificar várias populações devem incluir portas não sobrepostas, e verificações de pureza devem ser realizadas para garantir o enriquecimento das populações de interesse80. A classificação celular de populações de células-tronco e progenitoras usando os marcadores de superfície celular CD9 e CD104 pode ser usada para realizar estudos aprofundados desses subconjuntos ex-vivo, como (i) RNA-seq ou análise baseada em proteínas em resposta a perturbações (por exemplo, manipulações genéticas, intervenções farmacológicas); (ii) exames de drogas para identificar alvos que preservam a estaminalidade (por exemplo, para melhorar a regeneração - terapia com células-tronco) ou induzem a diferenciação (por exemplo, para suprimir o crescimento de células cancerígenas); e (iii) ensaios funcionais (por exemplo, transplante de células após manipulações genéticas ex-vivo ou intervenções farmacológicas).
Em resumo, a abordagem aqui apresentada combina análise CyTOF e classificação celular para permitir estudos in vivo da dinâmica celular e molecular de células-tronco musculares e progenitoras em resposta a manipulações genéticas ou intervenções farmacológicas, bem como estudos destinados a investigar a resposta de populações de células isoladas prospectivamente em cultura. Essa abordagem pode ser usada para desenvolver estratégias de triagem de medicamentos destinadas a identificar compostos que empurram as células para a diferenciação para atingir as células cancerígenas ou aumentar a estaminalidade para aplicações de terapia com células-tronco. Essa plataforma ex-vivo seria uma ferramenta poderosa para obter uma compreensão profunda da função das células-tronco e desenvolver intervenções terapêuticas direcionadas. Finalmente, a capacidade de identificar e isolar prospectivamente a população de células-tronco ativadas em resposta à lesão, com base apenas na expressão de marcadores de superfície celular (CD98alto/CD44alto), pode abrir a porta para estudos que antes não eram viáveis, como estudos destinados a entender o retorno das células-tronco à quiescência ou descobrir os defeitos moleculares que prejudicam a função regenerativa das células-tronco musculares no envelhecimento, e doenças musculares.
Divulgações
Os autores declaram não haver conflito de interesses.
Agradecimentos
Agradecemos aos membros do FACS Core Facility no Departamento de Biomedicina da Universidade de Aarhus pelo apoio técnico. Agradecemos a Alexander Schmitz, gerente da Unidade de Citometria de Massa do Departamento de Biomedicina, pela discussão e apoio. Ilustrações científicas foram criadas usando Biorender.com. Este trabalho foi financiado por uma bolsa inicial da Aarhus Universitets Forskningsfond (AUFF) e uma bolsa do pacote inicial (0071113) da Fundação Novo Nordisk para a EP
Materiais
| Name | Company | Catalog Number | Comments |
| 15 mL centrifuge tube | Fisher Scientific | 07-200-886 | |
| 20 G needle | KDM | KD-fine 900123 | |
| 28 G, 0.5 mL insulin syringe | BD | 329461 | |
| 29 G, 0.3 mL insulin syringe | BD | 324702 | |
| 3 mL syringes | Terumo medical | MDSS03SE | |
| 40 µm cell strainers | Fisher Scientific | 11587522 | |
| 5 mL polypropylene tubes | Fisher Scientific | 352002 | |
| 5 mL polystyrene test tubes with 35 µm cell strainer | Falcon | 352235 | |
| 5 mL syringes | Terumo medical | SS05LE1 | |
| 50 mL centrifuge tube | Fisher Scientific | 05-539-13 | |
| 5-Iodo-2-deoxyuridine (IdU) | Merck | I7125-5g | |
| anti-CD104 FITC (clone: 346-11A) | Biolegend | 123605 | Stock = 0.5 mg/mL |
| anti-CD11b APC-Cy7 (Clone: M1/70) | Biolegend | 101226 | Stock = 0.2 mg/mL |
| anti-CD31 APC-Cy7 (clone: 390) | Biolegend | 102440 | Stock = 0.2 mg/mL |
| anti-CD45 APC-Cy7 (Clone: 30-F11) | Biolegend | 103116 | Stock = 0.2 mg/mL |
| anti-CD9 APC (clone: KMC8) | ThermoFisher Scientific | 17-0091-82 | Stock = 0.2 mg/mL |
| anti-Sca1 (Ly6A/E) APC-Cy7 (clone: D7) | Biolegend | 108126 | Stock = 0.2 mg/mL |
| anti-α7 integrin PE (clone: R2F2)) | UBC AbLab | 67-0010-05 | Stock = 1 mg/mL |
| BD FACS Aria III (4 laser) instrument | BD Biosciences | N/A | 405, 488, 561, and 633 nm laser |
| Bovine Serum Albumin | Sigma Aldrich | A7030-50G | |
| Buprenorphine 0.3 mg/mL | Ceva | Vnr 054594 | |
| CD104 (Clone: 346-11A) | BD Biosciences | 553745 | Dy162; In-house conjugated |
| CD106/VCAM-1 (Clone: 429 MVCAM.A) | Biolegend | 105701 | Er170; In-house conjugated |
| CD11b (Clone: M1/70) | BD Biosciences | 553308 | Nd148; In-house conjugated |
| CD29/Integrin β1 (Clone: 9EG7) | BD Biosciences | 553715 | Tm169; In-house conjugated |
| CD31 (Clone: MEC 13.3) | BD Biosciences | 557355 | Sm154; In-house conjugated |
| CD34 (Clone: RAM34) | BD Biosciences | 551387 | Lu175; In-house conjugated |
| CD44 (Clone: IM7) | BD Biosciences | 550538 | Yb171; In-house conjugated |
| CD45 (Clone: MEC 30-F11) | BD Biosciences | 550539 | Sm147; In-house conjugated |
| CD9 (Clone: KMC8) | Thermo Fisher Scientific | 14-0091-85 | Yb174; In-house conjugated |
| CD90.2/Thy1.2 (Clone: 30-H12) | BD Biosciences | 553009 | Nd144; In-house conjugated |
| CD98 (Clone: H202-141) | BD Biosciences | 557479 | Pr141; In-house conjugated |
| Cell Acquisition Solution/Maxpar CAS-buffer | Standard Biotools | 201240 | |
| Cell-ID Intercalator-Iridium | Standard Biotools | 201192B | cationic nucleic acid intercalator |
| Cisplatin | Merck | P4394 | Pt195 |
| Cisplatin (cis-Diammineplatinum(II) dichloride) | Merck | P4394 | |
| Clear 1.5 mL tube | Fisher Scientific | 11926955 | |
| Collagenase, Type II | Worthington Biochemical Corporation | LS004177 | |
| Counting chamber | Merck | BR718620-1EA | |
| CXCR4/SDF1 (Clone: 2B11/CXCR4 ) | BD Biosciences | 551852 | Gd158; In-house conjugated |
| DAPI (1 mg/mL) | BD Biosciences | 564907 | |
| Dark 1.5 mL tube | Fisher Scientific | 15386548 | |
| Dispase II | Thermo Fisher Scientific | 17105041 | |
| Dissection Scissors | Fine Science Tools | 14568-09 | |
| DMEM (low glucose, with pyruvate) | Thermo Fisher Scientific | 11885-092 | |
| EDTA (Ethylenediaminetetraacetic acid disodium salt) | Merck | E5134 | Na2EDTA-2H20 |
| EQ Four Element Calibration Beads (EQ beads) | Standard Biotools | 201078 | Calibration beads |
| Fetal Bovine Serum, qualified, Brazil origin | Thermo Fisher Scientific | 10270106 | |
| Forceps Dumont #5SF | Fine Science Tools | 11252-00 | |
| Forceps Dumont #7 | Hounisen.com | 1606.3350 | |
| Goat serum | Thermo Fisher Scientific | 16210-072 | |
| Helios CyTOF system | Standard Biotools | N/A | |
| Horse Serum, heat inactivated, New Zealand origin | Thermo Fisher Scientific | 26-050-088 | |
| IdU | Merck | I7125 | I127 |
| Iridium-Intercalator | Standard Biotools | 201240 | Ir191/193 |
| Isoflurane/Attane Vet | ScanVet | Vnr 055226 | |
| Methanol | Fisher Scientific | M/3900/17 | |
| Myf5 (Clone: C-20) | Santa Cruz Biotechnology | Sc-302 | Yb173; In-house conjugated |
| MyoD (Clone: 5.8A) | BD Biosciences | 554130 | Dy164; In-house conjugated |
| MyoG (Clone: F5D) | BD Biosciences | 556358 | Gd160; In-house conjugated |
| Nalgene Rapid-Flow Sterile Disposable Bottle Top 0.20 μM PES Filters | Thermo Fisher Scientific | 595-4520 | |
| Notexin | Latoxan | L8104 | Resuspend to 50 µg/ml in sterile PBS. Keep stocks (e.g. 50 µl) at -20 °C |
| Nutrient mixture F-10 (Ham's) | Thermo Fisher Scientific | 31550031 | |
| pAkt (Clone: D9E) | Standard Biotools | 3152005A | Sm152 |
| Pax7 (Clone: PAX7) | Santa Cruz Biotechnology | Sc-81648 | Eu153; In-house conjugated |
| Penicillin-Streptomycin (10,000 U/mL) (Pen/Strep) | Thermo Fisher Scientific | 15140122 | |
| PES Filter Units 0.20 μM | Fisher Scientific | 15913307 | |
| PES Syringe Filter | Fisher Scientific | 15206869 | |
| Petri dish | Sarstedt | 82.1472.001 | |
| PFA 16% EM grade | MP Biomedicals | 219998320 | |
| Potassium chloride (KCl) | Fisher Scientific | 10375810 | |
| Potassium phosphate, monobasic, anhydrous (KH2PO4) | Fisher Scientific | 10573181 | |
| pRb (Clone: J112-906) | Standard Biotools | 3166011A | Er166 |
| pS6 kinase (Clone: N7-548) | Standard Biotools | 3172008A | Yb172 |
| Sca-1 (Clone: E13-161.7) | BD Biosciences | 553333 | Nd142; In-house conjugated |
| Sodium Azide | Sigma Aldrich | S2002 | |
| Sodium chloride (NaCl) | Fisher Scientific | 10553515 | |
| Sodium phosphate, dibasic, heptahydrate (Na2HPO4-6H2O) | Merck | S9390 | |
| Sterile saline solution 0.9% | Fresenius | B306414/02 | |
| α7 integrin (Clone: 3C12) | MBL international | K0046-3 | Ho165; In-house conjugated |
Referências
- Mukund, K., Subramaniam, S. Skeletal muscle: A review of molecular structure and function in health and disease. Wiley Interdiscip Rev Syst Biol Med. 12 (1), e1462 (2020).
- Feige, P., Brun, C. E., Ritso, M., Rudnicki, M. A. Orienting muscle stem cells for regeneration in homeostasis, aging, and disease. Cell Stem Cell. 23 (5), 653-664 (2018).
- Mauro, A. Satellite cell of skeletal muscle fibers. J Biophys Biochem Cytol. 9 (2), 493-495 (1961).
- Seale, P., et al. Pax7 is required for the specification of myogenic satellite cells. Cell. 102 (6), 777-786 (2000).
- Fuchs, E., Blau, H. M. Tissue stem cells: Architects of their niches. Cell Stem Cell. 27 (4), 532-556 (2020).
- Hernández-hernández, J. M., et al. The myogenic regulatory factors, determinants of muscle development, cell identity and regeneration. Semin Cell Dev Biol. 72, 10-18 (2017).
- Zammit, P. S. Function of the myogenic regulatory factors Myf5, MyoD, Myogenin and MRF4 in skeletal muscle, satellite cells and regenerative myogenesis. Semin Cell Dev Biol. 72, 19-32 (2017).
- Sabourin, L. A. The molecular regulation of myogenesis. Clin Genet. 57 (1), 16-25 (2000).
- Cooper, R. N., et al. In vivo satellite cell activation via Myf5 and MyoD in regenerating mouse skeletal muscle. J Cell Sci. 112 (17), 2895-2901 (1999).
- Rudnicki, M. A., Jaenisch, R. The MyoD family of transcription factors and skeletal myogenesis. Bioessays. 17 (3), 203-209 (1995).
- Braun, T., Arnold, H. H. Inactivation of Myf-6 and Myf-5 genes in mice leads to alterations in skeletal muscle development. EMBO J. 14 (6), 1176-1186 (1995).
- Yablonka-Reuveni, Z. Development and postnatal regulation of adult myoblasts. Microsc Res Tech. 30 (5), 366-380 (1995).
- Braun, T., et al. MyoD expression marks the onset of skeletal myogenesis in Myf-5 mutant mice. Development. 120 (11), 3083-3092 (1994).
- Rudnicki, M. A., et al. MyoD or Myf-5 is required for the formation of skeletal muscle. Cell. 75 (7), 1351-1359 (1993).
- Montarras, D., et al. Developmental biology: Direct isolation of satellite cells for skeletal muscle regeneration. Science. 309 (5743), 2064-2067 (2005).
- Sacco, A., Doyonnas, R., Kraft, P., Vitorovic, S., Blau, H. M. Self-renewal and expansion of single transplanted muscle stem cells. Nature. 456 (7221), 502-506 (2008).
- Cerletti, M., et al. Highly efficient, functional engraftment of skeletal muscle stem cells in dystrophic muscles. Cell. 134 (1), 37-47 (2008).
- Liu, L., Cheung, T. H., Charville, G. W., Rando, T. A. Isolation of skeletal muscle stem cells by fluorescence-activated cell sorting. Nat Protoc. 10 (10), 1612-1624 (2015).
- Porpiglia, E., et al. High-resolution myogenic lineage mapping by single-cell mass cytometry. Nat Cell Biol. 19 (5), 558-567 (2017).
- Behbehani, G. K., Bendall, S. C., Clutter, M. R., Fantl, W. J., Nolan, G. P. Single-cell mass cytometry adapted to measurements of the cell cycle. Cytometry Part A. 81 (7), 552-566 (2012).
- Hartmann, F. J., et al. . Mass Cytometry: Methods and Protocols. , (2019).
- Devine, R. D., Behbehani, G. K. Use of the pyrimidine analog, 5-iodo-2'-deoxyuridine (IdU) with cell cycle markers to establish cell cycle phases in a mass cytometry platform. J Vis Exp. (176), e60556 (2021).
- Bendall, S. C., et al. Single-cell mass cytometry of differential immune and drug responses across a human hematopoietic continuum. Science. 332 (6030), 687-696 (2011).
- Nag, A. C., Foster, J. D. Myogenesis in adult mammalian skeletal muscle in vitro. J Anat. 132, 1-18 (1981).
- Le Moigne, A., et al. Characterization of myogenesis from adult satellite cells cultured in vitro). Int J Dev Biol. 34, 171-180 (1990).
- Yablonka-Reuveni, Z. Development and postnatal regulation of adult myoblasts. Microsc Res Tech. 30 (5), 366-380 (1995).
- Chu, C., Cogswell, J., Kohtz, D. S. MyoD functions as a transcriptional repressor in proliferating myoblasts. J Biol Chem. 272 (6), 3145-3148 (1997).
- Shah, B., Hyde-Dunn, J., Jones, G. E. Proliferation of murine myoblasts as measured by bromodeoxyuridine incorporation. Methods in Mol Biol. 75, 349-355 (1997).
- Springer, M. L., Blau, H. M. High-efficiency retroviral infection of primary myoblasts. Somat Cell Mol Genet. 23 (3), 203-209 (1997).
- Rando, T. A., Blau, H. M. Primary mouse myoblast purification, characterization, and transplantation for cell-mediated gene therapy. J Cell Biol. 125 (6), 1275-1287 (1994).
- Springer, M. L., Rando, T. A., Blau, H. M. Gene delivery to muscle. Curr Protoc Hum Genet. , (2002).
- Cull-Candy, S. G., Fohlman, J., Gustavsson, D., Lullmann-Rauch, R., Thesleff, S. The effects of taipoxin and notexin on the function and fine structure of the murine neuromuscular junction. Neuroscience. 1 (3), 175-180 (1976).
- Francis, B., John, T. R., Seebart, C., Kaiser, I. I. New toxins from the venom of the common tiger snake (Notechis scutatus scutatus). Toxicon. 29 (1), 85-96 (1991).
- Navarro, K. L., Huss, M., Smith, J. C., Sharp, P., Marx, J. O., Pacharinsak, C. Mouse Anesthesia: The Art and Science. ILAR Journal. 62, 238-273 (2021).
- Langford, D., Bailey, A., Chanda, M., et al. Coding of facial expressions of pain in the laboratory mouse. Nat Methods. 7, 447-449 (2010).
- Matsumiya, L. C., Sorge, R. E., Sotocinal, S. G., Tabaka, J. M., Wieskopf, J. S., Zaloum, A., King, O. D., Mogil, J. S. Using the Mouse Grimace Scale to reevaluate the efficacy of postoperative analgesics in laboratory mice. J Am Assoc Lab Anim Sci. 2012 (1), 42-49 (2012).
- Gonzalez, V. D., et al. High-grade serous ovarian tumor cells modulate NK cell function to create an immune-tolerant microenvironment. Cell Rep. 36 (9), 109632 (2021).
- Delgado-Gonzalez, A., et al. Measuring trogocytosis between ovarian tumor and natural killer cells. STAR Protoc. 3 (2), 101425 (2022).
- Finck, R., et al. Normalization of mass cytometry data with bead standards. Cytometry Part A. 83 (5), 483-494 (2013).
- Leipold, M. D., Maecker, H. T. Mass cytometry: protocol for daily tuning and running cell samples on a CyTOF mass cytometer. J Vis Exp. (69), e4398 (2012).
- McCarthy, R. L., Duncan, A. D., Barton, M. C. Sample preparation for mass cytometry analysis. J Vis Exp. (122), e54394 (2017).
- Kotecha, N., Krutzik, P. O., Irish, J. M. Web-based analysis and publication of flow cytometry experiments. Curr Protoc Cytom. , (2010).
- Fienberg, H. G., Simonds, E. F., Fantl, W. J., Nolan, G. P., Bodenmiller, B. A platinum-based covalent viability reagent for single-cell mass cytometry. Cytometry Part A. 81 (6), 467-475 (2012).
- Kimball, A. K., et al. A beginner's guide to analyzing and visualizing mass cytometry data. J Immunol. 200 (1), 3-22 (2018).
- Weber, L. M., Robinson, M. D. Comparison of clustering methods for high-dimensional single-cell flow and mass cytometry data. Cytometry Part A. 89 (12), 1084-1096 (2016).
- Samusik, N., Good, Z., Spitzer, M. H., Davis, K. L., Nolan, G. P. Automated mapping of phenotype space with single-cell data. Nat Methods. 13 (6), 493-496 (2016).
- Ornatsky, O. I., et al. Study of cell antigens and intracellular DNA by identification of element-containing labels and metallointercalators using inductively coupled plasma mass spectrometry. Anal Chem. 80 (7), 2539-2547 (2008).
- Relaix, F., et al. Perspectives on skeletal muscle stem cells. Nat Commun. 12 (1), 692 (2021).
- de Morree, A., et al. Staufen1 inhibits MyoD translation to actively maintain muscle stem cell quiescence. Proc Natl Acad Sci U S A. 114 (43), E8996-E9005 (2017).
- Luo, D., et al. Deltex2 represses MyoD expression and inhibits myogenic differentiation by acting as a negative regulator of Jmjd1c. Proc Natl Acad Sci U S A. 114 (15), E3071-E3080 (2017).
- Wersto, R. P., et al. Doublet discrimination in DNA cell-cycle analysis. Cytometry. 46 (5), 296-306 (2001).
- Porpiglia, E., Blau, H. M. Plasticity of muscle stem cells in homeostasis and aging. Curr Opin Genet Dev. 77, 101999 (2022).
- Porpiglia, E., et al. Elevated CD47 is a hallmark of dysfunctional aged muscle stem cells that can be targeted to augment regeneration. Cell Stem Cell. 29 (12), 1653-1668 (2022).
- Brunet, A., Goodell, M. A., Rando, T. A. Ageing and rejuvenation of tissue stem cells and their niches. Nat Rev Mol Cell Biol. 24 (1), 45-62 (2022).
- Danielli, S. G., et al. Single-cell profiling of alveolar rhabdomyosarcoma reveals RAS pathway inhibitors as cell-fate hijackers with therapeutic relevance. Sci Adv. 9 (6), (2023).
- de Morree, A., Rando, T. A. Regulation of adult stem cell quiescence and its functions in the maintenance of tissue integrity. Nat Rev Mol Cell Biol. 24 (5), 334-354 (2023).
- Yucel, N., et al. Glucose metabolism drives histone acetylation landscape transitions that dictate muscle stem cell glucose metabolism drives histone acetylation landscape transitions that dictate muscle stem cell function. Cell Rep. 27 (13), 3939-3955 (2019).
- Tierney, M. T., Sacco, A. Inducing and evaluating skeletal muscle injury by notexin and barium chloride. Methods Mol Biol. 1460, 53-60 (2016).
- Hardy, D., et al. Comparative study of injury models for studying muscle regeneration in mice. PLoS One. 11 (1), e0147198 (2016).
- Call, J. A., Lowe, D. A. Eccentric contraction-induced muscle injury: Reproducible, quantitative, physiological models to impair skeletal muscle's capacity to generate force. Methods Mol Biol. 1460, 3-18 (2016).
- Garry, G. A., Antony, M. L., Garry, D. J. Cardiotoxin Induced Injury and Skeletal Muscle Regeneration. Methods Mol Biol. 1460, 61-71 (2016).
- Le, G., Lowe, D. A., Kyba, M. Freeze injury of the tibialis anterior muscle. Methods Mol Biol. 1460, 33-41 (2016).
- Borok, M., et al. Progressive and coordinated mobilization of the skeletal muscle niche throughout tissue repair revealed by single-cell proteomic analysis. Cells. 10 (4), 744 (2021).
- Petrilli, L. L., et al. High-dimensional single-cell quantitative profiling of skeletal muscle cell population dynamics during regeneration. Cells. 9 (7), 1723 (2020).
- Giordani, L., et al. High-dimensional single-cell cartography reveals novel skeletal muscle-resident cell populations. Mol Cell. 74 (3), 609-621 (2019).
- Hartmann, F. J., et al. Scalable conjugation and characterization of immunoglobulins with stable mass isotope reporters for single-cell mass cytometry analysis. Methods Mol Biol. 1989, 55-81 (2019).
- Frimand, Z., Das Barman, S., Kjær, T. R., Porpiglia, E., de Morrée, A. Isolation of quiescent stem cell populations from individual skeletal muscles. J Vis Exp. (190), e64557 (2022).
- Krutzik, P. O., Nolan, G. P. Intracellular phospho-protein staining techniques for flow cytometry: monitoring single cell signaling events. Cytometry A. 55 (2), 61-70 (2003).
- Bodenmiller, B., et al. Multiplexed mass cytometry profiling of cellular states perturbed by small-molecule regulators. Nat Biotechnol. 30 (9), 858-867 (2012).
- Schulz, K. R., Danna, E. A., Krutzik, P. O., Nolan, G. P. Single-cell phospho-protein analysis by flow cytometry. Curr Protoc Immunol. , 11-20 (2012).
- Krutzik, P. O., Clutter, M. R., Nolan, G. P. Coordinate analysis of murine immune cell surface markers and intracellular phosphoproteins by flow cytometry. J Immunol. 175 (4), 2357-2365 (2005).
- Krutzik, P. O., Irish, J. M., Nolan, G. P., Perez, O. D. Analysis of protein phosphorylation and cellular signaling events by flow cytometry: techniques and clinical applications. Clin Immunol. 110 (3), 206-221 (2004).
- Han, G., Spitzer, M. H., Bendall, S. C., Fantl, W. J., Nolan, G. P. Metal-isotope-tagged monoclonal antibodies for high-dimensional mass cytometry. Nat Protoc. 13 (10), 2121-2148 (2018).
- Chevrier, S., et al. Compensation of signal spillover in suspension and imaging mass cytometry. Cell Syst. 6 (5), 612-620 (2018).
- Bjornson, Z. B., Nolan, G. P., Fantl, W. J. Single-cell mass cytometry for analysis of immune system functional states. Curr Opin Immunol. 25 (4), 484-494 (2013).
- Kalina, T., Lundsten, K., Engel, P. Relevance of antibody validation for flow cytometry. Cytometry A. 97 (2), 126-136 (2020).
- Baumgarth, N., Roederer, M. A practical approach to multicolor flow cytometry for immunophenotyping. J Immunol Methods. 243 (1-2), 77-97 (2000).
- Roederer, M. Spectral compensation for flow cytometry: visualization artifacts, limitations, and caveats. Cytometry. 45 (3), 194-205 (2001).
- Tung, J. W., Parks, D. R., Moore, W. A., Herzenberg, L. A., Herzenberg, L. A. New approaches to fluorescence compensation and visualization of FACS data. Clin Immunol. 110 (3), 277-283 (2004).
- Cossarizza, A., et al. Guidelines for the use of flow cytometry and cell sorting in immunological studies (third edition). Eur J Immunol. 51 (12), 2708-3145 (2021).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados