Method Article
Entendendo o impacto de bacteriófagos de clima temperado sobre seus lisógenos através de transcriptômica
Neste Artigo
Resumo
Esse protocolo permite revelar o impacto dos prófagos em seus hospedeiros. As culturas bacterianas são sincronizadas usando condições que melhor suportam o estado lisogênico, limitando a indução espontânea. A RT-qPCR distingue inequivocamente os genes restritos ao prófago e aqueles desacoplados do controle do fago daqueles que são expressos durante o ciclo de replicação lítica.
Resumo
Fagos temperados são encontrados integrados como prófagos na maioria dos genomas bacterianos. Alguns prófagos são crípticos e fixados no cromossomo bacteriano, mas outros são ativos e podem ser desencadeados de forma replicativa espontaneamente ou por exposição a fatores indutores. Os prófagos são comumente associados à capacidade de conferir a produção de toxinas ou outras características associadas à virulência em sua célula hospedeira. Estudos mais recentes mostraram que eles podem desempenhar um papel muito maior na alteração da fisiologia de seus hospedeiros. A técnica aqui descrita nos permitiu investigar como os prófagos afetam a expressão gênica na bactéria oportunista Pseudomonas aeruginosa.
Neste trabalho, o crescimento da cepa selvagem de P. aeruginosa PAO1 foi comparado com o de lisógenos isogênicos portadores de diferentes combinações de prófagos da cepa epidêmica de Liverpool (LES) LESB58. Em uma cultura de lisogênio, uma proporção de células bacterianas estará apoiando a replicação de bacteriófagos líticos (indução espontânea) com um alto nível de expressão por célula de genes de fagos tardios, como aqueles associados à montagem de partículas de fago, mascarando assim a expressão gênica de baixo nível associada à expressão gênica restrita a lisogênio. O impacto da indução espontânea pode, portanto, obscurecer a expressão gênica de prófagos em uma população lisógena.
Experimentos de perfil de crescimento foram usados para identificar indução espontânea, que foi mínima durante a fase inicial de crescimento exponencial. Este estudo relata como preparar culturas de amostra durante a fase inicial de crescimento exponencial e como estabelecer controles adequados apesar do baixo número de células. Esses protocolos garantem a comparação confiável e reprodutível de bactérias selvagens e lisogênicas sob várias condições, melhorando assim o perfil transcriptômico de genomas de prófagos e auxiliando na identificação de funções de prófagos previamente não reconhecidas.
Introdução
Recentemente, a fagoterapia para combater a resistência antimicrobiana1 e a edição gênica baseada em CRISPR-Cas2 geraram um interesse renovado na pesquisa de bacteriófagos. Novamente, os avanços da biotecnologia têm possibilitado o aprofundamento da investigação das interações entre bactérias e fagos3. No entanto, o uso terapêutico do fago ("fagoterapia") é dificultado pela preocupação com a atuação dos fagos como elementos genéticos móveis com capacidade de transferir horizontalmente genes de virulência e resistência4. A extensão da "matéria escura"5 (genes com funções desconhecidas) é ao mesmo tempo preocupante e sedutora. A matéria escura é considerada uma lacuna em nossa compreensão da biologia dos fagos e um recurso em grande parte inexplorado para ferramentas moleculares e potenciais novas terapêuticas6. O desenvolvimento de técnicas de sequenciamento de alto rendimento, juntamente com a melhoria da anotação gênica 7,8,9 e novos algoritmos de dobramento de peptídeos10, está melhorando a detecção, descrição e predição funcional de genes de fagos. No entanto, a ciência ainda está longe de validar as funções gênicas da maioria dos fagos em cultura ou no mundo real.
O sequenciamento de RNA (RNA-Seq) pode mapear globalmente a expressão gênica durante a infecção fagogênica e tem melhorado significativamente a compreensão dos elementos fagos e bacterianos envolvidos nos ciclos lítico e lisogênico11,12. Durante os processos lisogênicos, genomas de fagos temperados são integrados ao DNA bacteriano para se tornarem prófagos13. Experimentos de perfil de expressão gênica global podem ser usados para identificar genes restritos a prófagos que são codificados em genomas de fagos temperados, mas expressos apenas durante o estado lisogênico11. Tais genes não codificam proteínas estruturais do fago e não estão envolvidos em nenhum processo de infecção por fagos. O RNA-Seq pode ser usado para identificar os genes que têm maior probabilidade de influenciar a biologia do hospedeiro bacteriano, seja induzindo um ganho de função ou regulando os genes bacterianos existentes, permitindo muitas vezes que as bactérias se adaptem a ambientes em mudança. Portanto, a capacidade dos prófagos de atuarem como marionetes microbianas, controlando uma série de funções bacterianas, poderia ser estudada.
Existem duas grandes barreiras para a análise efetiva da expressão gênica restrita a prófagos. Em primeiro lugar, a disponibilidade de hosts suscetíveis é uma questão fundamental. Por definição, os prófagos já estão incorporados em seu genoma específico do hospedeiro, por isso é desafiador encontrar um hospedeiro selvagem suscetível para comparar a expressão gênica global na presença e ausência do prófago. Isso pode ser conseguido através da infecção de novo de outro hospedeiro suscetível ou da deleção do prófago do isolado selvagem original, sem interromper o resto do genoma do hospedeiro. A segunda barreira reside na natureza heterogênea das populações lisogênicas. Alguns prófagos degradam-se através de mutação ou recombinação para se tornarem "crípticos", o que significa que estão fixados em um local específico do genoma bacteriano. No entanto, outros prófagos são "ativos" e podem ser induzidos a um ciclo lítico replicativo espontaneamente ou após exposição a fatores indutores. Em muitas culturas lisogênicas, a taxa de indução espontânea significa que uma proporção das células bacterianas está sempre em fase de replicação lítica do fago14,15,16. Um alto nível de expressão de genes de fagos tardios nessas populações mascara o baixo nível de expressão gênica associada à expressão gênica restrita a lisogênio11,17. A proporção de lisógenos submetidos à indução espontânea de prófagos pode variar com o estado de crescimento, condições de crescimento ou outros gatilhos. Portanto, para estudar os impactos dos prófagos sobre o lisógeno, os eventos de indução espontânea de prófagos devem ser minimizados ao máximo, otimizando as condições de crescimento para favorecer o estado lisogênico.
Este estudo relata o trabalho preparatório realizado para investigar a influência de um conjunto de prófagos coabitantes da cepa epidêmica de Liverpool (LES) de Pseudomonas aeruginosa. Profagos ativos foram induzidos e isolados do LES e usados para infectar a cepa modelo de P. aeruginosa, PAO116,18,19. Os genomas inteiros da cepa selvagem de P. aeruginosa, PAO1, e seu lisogênio, PAO1Φ2, foram sequenciados (a uma profundidade de cobertura de 30x) para garantir a identidade da cepa selvagem e confirmar que o lisogênio era isogênico. O LES tem sido associado ao aumento da morbidade e mortalidade em pacientes com fibrose cística, e fagos do LES 19 têm sido sugeridos para auxiliar na adaptação ao ambiente pulmonar da fibrose cística16,19,20. Apesar das fortes evidências de que esses prófagos afetam a biologia de seu hospedeiro20,21, a maioria de suas funções gênicas ainda não foi caracterizada, e os mecanismos específicos de interação são pouco compreendidos. Uma abordagem transcriptômica pode descobrir empiricamente as funções do gene do prófago em um fundo de hospedeiro controlado. Uma vez que a indução espontânea pode afetar o perfil de expressão, este artigo descreve como otimizar as condições de crescimento para favorecer o estado lisogênico. Essa sincronização de culturas pode ser validada por PCR em tempo real para quantificar os níveis de expressão de marcadores genéticos chave que estão associados a estágios cruciais da replicação de fagos do LES na PAO1. A mesma abordagem foi usada anteriormente para identificar as funções restritas a prófagos de Shiga-toxigênicos que afetam a motilidade, resistência ácida e resistência antimicrobiana em Escherichia coli11,17,21,22.
Protocolo
1. Crie um host indicador selecionável (Figura 1)
NOTA: Os lisados de cultura de fagos podem conter células contaminantes do hospedeiro bacteriano original. Ter uma cepa indicadora resistente a antibióticos permite a discriminação entre a cepa indicadora e o hospedeiro bacteriano original do prófago. O uso de uma cepa indicadora selecionável permite a enumeração precisa das partículas infectantes do fago sem a necessidade de centrifugação ou etapas de filtração para remover o fago das células lisógenas seguindo as etapas de amplificação do fago. A cepa do host indicador selecionável também reduz o tempo e o número de etapas para a enumeração de fagos para que várias condições possam ser testadas simultaneamente.
- Identificar uma cepa hospedeira indicadora adequada suscetível à infecção lítica e lisogênica pelo fago temperado de interesse. A cepa de laboratório de P. aeruginosa PAO118,20 foi utilizada e é suscetível aos três fagos do LES (LESΦ2, LESΦ3 e LESΦ4).
- Escolha um agente seletivo adequado (rifampicina foi usada aqui) e realize um ensaio de diluição em caldo para determinar a concentração inibitória mínima (CIM) para o hospedeiro indicador (16 μg·mL−1 é a CIM para PAO1)23,24.
- Expor sequencialmente as culturas do hospedeiro indicador a concentrações crescentes do agente seletivo em caldo lisogênico (LB), começando abaixo da CIM (neste caso 5 μg·mL−1), por 18–24 h, com agitação e a 37 °C.
- Transfira a cultura que cresce na concentração mais alta na proporção de 1:100 (inóculo/meio) para concentrações duas vezes maiores do agente seletivo (18–24 h de cada vez) até que a CIM tenha sido aumentada suficientemente. A PAO1 tornou-se uma cepa resistente à rifampicina (PAO1-RifR) a 300 μg·mL−1 de rifampicina.
2. Enumeração temporal direta da indução espontânea (Figura 2)
- Estabelecer culturas iniciais durante a noite do lisogênio (por exemplo, lisogênio de P. aeruginosa PAO1 que abriga fagos do LES) e do hospedeiro indicador (PAO1-RifR) inoculando uma única colônia em 5 mL de LB e incubar a 37 °C com agitação a 180 rpm (18–24 h).
- Estabelecer culturas de lisogênio fresco e de hospedeiro indicador inoculando as culturas noturnas em 100 mL de LB na proporção de 1:100 e incubar a 37 °C com agitação (180 rpm).
- Monitorar o crescimento lisogênico medindo o OD600 e a contagem viável usando a técnica de Miles Misra25. Para isso, coletar uma amostra de 1 mL de cada cultura lisógena a cada hora a partir do ponto de inoculação por 8 h.
- Diluir a amostra em série imediatamente após a colheita, adicionando 100 μL da amostra em 900 μL do respectivo meio. Vórtice bem na velocidade máxima, descarte a ponta a cada diluição e continue a série de diluição de 10−1 a 10−9.
- Colocar 10 μL das diluições necessárias em triplicata numa placa de ágar LB, deixar secar e incubar a 37 °C durante 18–24 horas.
- Para calcular o número de células bacterianas viáveis, encontre uma diluição com colônias facilmente contáveis. Conte o número de colônias em cada ponto e use a seguinte fórmula:

NOTA: À medida que a cultura lisógena cresce, partículas ativas de fagos serão produzidas por indução espontânea. A produção de fagos infectantes significa que o transcriptoma da população lisógena está agora contaminado com a expressão gênica associada ao ciclo de replicação lítica a partir do sinal de replicação lítica do fago e da resposta correspondente da célula hospedeira. Assim, é importante identificar o estágio de crescimento em que a proporção de células lisogênicas para partículas livres de fagos infectantes é maior, a fim de limitar o máximo possível de ruído de transcrição de fundo (gerado pelo transcriptoma lítico do fago) no conjunto de dados.
- Para enumerar as partículas infectantes de fagos em cada amostra temporal, inocular 5 mL de ágar bacteriológico 0,4% estéril em LB (ágar superior) com 100 μL de hospedeiro indicador de fase média exponencial (OD600: 0,4–0,5; neste caso, PAO1-RifR) na presença de um agente seletivo apropriado (50 μg·mL−1 de rifampicina neste caso, já que a CIM do hospedeiro PAO1 é de apenas 16 μg·mL−1; ver Tabela de Materiais, linha 8 e linha 9)
- Colocar 10 μL da mesma diluição em série (ver passo 2.2.2) na camada superior de ágar inoculada e deixar secar antes de incubar a 37 °C durante 18–24 horas.
- Para calcular as partículas infectantes de fago, encontre uma diluição com placas facilmente contáveis. Conte o número de placas em cada ponto.

- Encontrar o tempo/condição para o qual a indução espontânea por UFC (unidade formadora de colônia) é mínima para as etapas experimentais posteriores.
3. Preparação de culturas de lisogênio não induzidas e induzidas para extração de RNA (Figura 3)
- Estabelecer uma cultura noturna fresca inoculando uma única colônia de lisogênio em 5 mL de LB e incubar a 37 °C com agitação (180 rpm) por 18–24 h.
- Subcultura: a cultura noturna em 80 mL de LB na proporção de 1:100 em oito frascos de 250 mL.
- Rotular o primeiro frasco como "não induzido" e os demais como "induzido", juntamente com os momentos em que cada amostra deve ser colhida (i.e., "t induzido = 0", "t induzido = 10 min", "t induzido = 20 min", etc.; Gráfico 3).
- Após 90 min de incubação, quando o OD600 estiver entre 0,1–0,2, ou no momento da indução espontânea mínima (ver discussão), adicionar 4 μL de ácido acético glacial a 1% (v/v) ao frasco não induzido (Figura 3).
NOTA: Como o agente indutor neste trabalho foi feito usando ácido acético glacial a 1% como solvente, a mesma quantidade de solvente foi adicionada isoladamente como etapa de controle. Controles alternativos podem ser considerados dependendo da preparação de diferentes indutores. - Adicionar a cultura de 80 ml do balão não induzido a 720 ml de LB estéril e adicionar imediatamente a solução de paragem (fenol gelado a 5% [v/v], pH 4,3, etanol a 95 % [v/v]) utilizando um volume de 20% do volume de cultura (160 ml) e incubar no gelo durante um mínimo de 30 minutos e não mais de 2 horas para estabilizar os transcritos de ARN12, 26,27. Esta é a amostra não induzida.
- Induzir as culturas restantes em sete frascos de 250 mL (Figura 3) com a CIM de um agente indutor apropriado (neste caso, norfloxacina 25 mg·mL−1, preparada em ácido acético glacial a 1% [p/v], usada na concentração final de 1 μg·mL−1), misturar bem e incubar a 37 °C e com agitação a 180 rpm por 1 h.
NOTA: Esta etapa forçará a cultura lisógena a um estado mais coordenado de replicação lítica. A maioria das células da cultura começará a sofrer produção lítica de partículas infectantes de fago. - Permitir que as células se recuperem adicionando 80 mL de cultura do frasco induzido a 720 mL de LB estéril, o que efetivamente dilui o agente indutor. Colher as células bacterianas de cada balão de 10 em 10 min, do tempo 0 até 1 h, adicionando uma solução stop, tal como referido no passo 3.5.
NOTA: A solução stop estabiliza o RNA por até 2 h. No entanto, para melhorar a estabilidade da amostra, execute todas as etapas adicionais a 4 °C. - Colher por centrifugação a 10.000 x g durante 15 min a 4 °C o mais rapidamente possível, não excedendo 2 h após o tratamento para evitar a degradação do ARN.
- Descarte o sobrenadante e ressuspenda suavemente os pellets bacterianos no líquido residual usando uma pipeta automática ajustável antes de transferir cada amostra para um tubo de microfuga de 1,5 mL.
- Centrifugar os tubos de microfuga em alta velocidade (13.000 x g) em um microfuge a 4 °C por 1 min e descartar o sobrenadante residual.
- Congele os pellets mergulhando cada tubo de microfuga selado em nitrogênio líquido. Isso ajudará a lise eficiente das células para extração de RNA.
- Adicionar TRIzol (1 mL) a cada pastilha congelada e homogeneizar a suspensão por pipetagem (não vórtice). Conservar a −80 °C até estar pronto para efectuar a extracção de ARN para todas as amostras.
Observação : o protocolo pode ser pausado neste ponto. - Repita as etapas 3.1–3.13 com três réplicas biológicas.
4. Isolamento de RNA de culturas de lisogênio não induzidas e induzidas
CRÍTICO: Todas essas etapas devem ser realizadas em ambiente livre de RNase28. As bancadas devem ser limpas com NaClO a 10% ou inativadores proprietários de RNase. O material de laboratório deve ser tratado com inibidores de RNase, como o tratamento com DEPC, e água livre de nuclease deve ser usada em todas as reações.
- Descongelar os pellets congelados tratados com TRIzol da etapa 3.12 no gelo e adicionar 400 μL de clorofórmio de grau de biologia molecular.
- Agite bem os frascos para injetáveis por inversão durante 10 s para completar a lise de todas as células ( não vórtice). Em seguida, incubar à temperatura ambiente (21 °C) por 2 a 5 min.
- Separe a camada aquosa da mistura TRIzol/clorofórmio por centrifugação usando um microfuge refrigerado de mesa a 4 °C e 13.000 x g por 15 min.
- Coletar a fase aquosa (~ 500 μL, camada superior) usando uma pipeta de 1.000 μL, tomando cuidado para não perturbar a interfase ou fase orgânica (camada inferior). Transfira para um novo tubo de microfuga de 1,5 mL.
- Adicionar 450 μL de isopropanol de grau de biologia molecular à fase aquosa separada, misturar bem por inversão ( não vórtice) e incubar à temperatura ambiente (21 °C) durante 30 minutos.
- Recuperar o RNA por centrifugação usando uma centrífuga refrigerada a 4 °C e 13.000 x g por 30 min.
- Eliminar o sobrenadante sem perturbar o pellet de ARN e lavar o pellet duas vezes com 800 μL de etanol a 70 % preparado com água isenta de nucleases (não pipetar para cima e para baixo). Garanta a estabilidade do pellet de RNA repetindo a etapa de centrifugação por 5 min após cada lavagem.
- Descarte o etanol e seque o pellet ao ar.
NOTA: Aspirar o etanol ao redor do pellet cuidadosamente usando uma microponta de 10 μL e secar o pellet invertendo o tubo em papel mata-borrão limpo. A pastilha de RNA deve ficar incolor e as bordas devem aparecer babadas e visíveis. Secar muito pouco pode deixar etanol residual que pode afetar os processos a jusante, e secar demais o pellet pode dificultar a ressuspensão. - Ressuspender o RNA em água livre de nucleases (50 μL) incubando a 65 °C em um termoagitador com mistura intermitente (a cada 30 s) por um total de 3–5 min.
CRÍTICO: O grupo 2'-OH do RNA é capaz de catalisar a autoclivagem de fitas de RNA a uma alta temperatura acima de 65 °C e um pH elevado. Temperaturas abaixo de 65 °C retardarão a ressuspensão do DNA residual, limitando assim a quantidade de DNA que deve ser digerida em um estágio posterior com a digestão da DNase I. Assim, manter a temperatura em 65 °C é fundamental para obter as melhores amostras.
NOTA: O protocolo pode ser pausado neste ponto, e as amostras podem ser armazenadas a -80 °C.
5. Remoção de DNA contaminante do RNA pelo tratamento com DNase
- Para remover o DNA contaminante do RNA total antes da síntese de cDNA da primeira fita, adicione um volume de 0,1 de tampão DNase 10x e 1 μL da enzima DNase a 10 μg de RNA total. Misture o tubo suavemente e incube a 37 °C durante 30 minutos.
- Ressuspender o reagente de inativação da DNase e adicionar um mínimo de 2 μL ou um volume de 10% do volume total da reação. Misturar bem e incubar as amostras durante 5 min à temperatura ambiente (21 °C) durante a redispersão do reagente de inactivação da DNase.
- Pellet os reagentes DNase por centrifugação usando uma microcentrífuga de mesa a 10.000 × g por 1,5 min.
- Transfira o sobrenadante que contém o RNA para um tubo fresco sem perturbar o pellet.
NOTA: Verifique a qualidade do RNA usando um espectrofotômetro UV em escala de 1 μL e analisador computacional de ácido nucleico de base microfluídica de acordo com as instruções do fabricante; o RNA total purificado pode ser armazenado a -80 °C. Para qRT-PCR, RNA poderia ser usado diretamente neste ponto. Para processos a jusante mais sensíveis, como o sequenciamento de RNA, que exigem uma qualidade de amostra rigorosa, uma relação A260/230 de ε 2,0 deve ser alcançada para prosseguir. - Completar o volume da solução de RNA livre de DNA para 500 μL usando água livre de nucleases.
- Adicionar 50 μL de acetato de sódio 3 M isento de nuclease (pH 5,3) e 495 μL de isopropanol. Misture bem e incube à temperatura ambiente durante 30 minutos.
NOTA: Esta etapa precipitará o RNA. - Recuperar o RNA por centrifugação a 13.000 x g e 4 °C por 30 min.
- Lavar o pellet de RNA três vezes com etanol gelado a 70%, centrifugando as amostras a 13.000 x g e 4 °C por 5 min após cada lavagem para remover completamente os sais.
- Verificar a qualidade do RNA usando um espectrofotômetro UV em escala de 1 μL e analisador computacional de ácido nucleico de base microfluídica, de acordo com as instruções do fabricante; o RNA total purificado pode ser armazenado a -80 °C.
NOTA: O guia29 foi utilizado para atingir os padrões de qualidade do RNA. Se a razão A260/230 for <2,0, repita as etapas 5,5 a 5,9.
6. Análise qualitativa e quantitativa do RNA livre de DNase
- Validar a eficiência do tratamento com DNase para cada amostra realizando uma PCR quantitativa usando iniciadores de 16S rRNA (Tabela 2) com 1 μg de RNA total e confirmar que nenhum produto de amplificação é produzido.
NOTA: Os primers ideais para avaliar a contaminação por gDNA seriam primers projetados para recozimento em junções íntron-éxon ou regiões reguladoras em procariotos ou em sítios transcricionalmente inativos30,31. - Determine o número de integridade do RNA (RIN) usando um analisador computacional de ácido nucleico baseado em microfluídica de acordo com as instruções do fabricante.
NOTA: Amostras que mostram um RIN ≥ 9 devem ser usadas para a síntese da primeira fita. As amostras que apresentam um RIM < 9 devem ser descartadas e as etapas de isolamento (1.1–5.4) devem ser repetidas. - Quantifique a concentração total de RNA usando o kit de ensaio de RNA HS e um fluorímetro de acordo com as instruções do fabricante.
7. Síntese de cDNA de primeira fita
- Preparar uma mistura de primers de ARN para cada amostra misturando 1 μg de ARN total com 1 μL de hexâmeros aleatórios (50 ng·μL−1) e 1 μL de mistura de 10 mM dNTP. Em seguida, ajuste o volume total para 10 μL usando água livre de nucleases.
- Incubar a reação a 65 °C por 5 min e colocar no gelo por 1 min.
- Preparar uma mistura de síntese de cDNA para cada amostra adicionando 2 μL de tampão 10x RT; 4 μL de 25 mM MgCl2; 2 μL de TDT 0,1 M; 1 μL de inibidor de RNase (40 U·μL−1); e 1 μL do reagente de transcrição reversa (200 U·μL−1) na ordem indicada.
- Adicionar a mistura de síntese de cDNA à mistura RNA/primer. Misture delicadamente e centrifugue brevemente as amostras para coletar os componentes no fundo do tubo.
- Primer a mistura incubando as amostras por 10 min a 25 °C, seguido de 50 min a 50 °C. Terminar as reacções incubando a 85 °C durante 5 minutos e arrefecer no gelo.
- Adicionar 1 μL de RNase H a cada tubo e incubar a 37 °C durante 20 minutos para remover o ARN do híbrido DNA:ARN.
- Finalmente, diluir a reação de síntese de cDNA para um volume total de 80 μL e armazená-la a −80 °C até uso posterior.
Observação : o protocolo pode ser pausado neste ponto.
8. Curva padrão e (q)-PCR quantitativa para determinar os níveis de expressão de genes marcadores que indicam diferentes estágios de replicação de fagos
- Identificar um conjunto de genes-alvo que possam atuar como marcadores para cada estágio de replicação do fago de interesse. Em nosso caso, estes foram os detalhados na Tabela 2.
- Amplificar cada um dos genes-alvo do DNA genômico do molde usando primers relevantes e usando PCR com as seguintes condições de amplificação: desnaturação inicial a 95 °C por 2 min; desnaturação a 95 °C por 30 s; recozimento na temperatura ótima de recozimento dependendo dos primers (58 °C foi usado aqui) por 30 s; extensão a 72°C por 1 min; e extensão final a 72 °C por 5 min.
- Purifice cada amplicon usando um kit de purificação PCR e clone-os em um vetor de clonagem TA de acordo com as instruções do fabricante. Verifique a sequência de cada produto clonado por sequenciamento Sanger.
Observação : o protocolo pode ser pausado neste ponto. - Calcule o número de cópias para plasmídeos individuais usando a seguinte equação20:
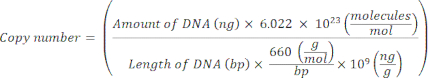
- Preparar um modelo padrão para cada gene marcador diluindo em série o DNA plasmidial de 109 cópias/μL a 10 2 cópias/μL em H2O estéril livre de nuclease de grau molecular.
- Realizar PCR quantitativo de acordo com as instruções do fabricante para o sistema de qPCR preferido com 1 μL de cDNA (a partir do passo 7.7) para cada amostra em triplicata, juntamente com os respectivos padrões plasmidiais em triplicata; realizar a PCR em uma placa de 96 poços para cada alvo.
- Plote o número de cópias de DNA Log (eixo x) versus o limiar do ciclo (eixo y, Ct) e use uma plataforma apropriada, como Excel ou R, para executar um cálculo de regressão linear para exibir o coeficiente de determinação (R2) e uma equação linear.
NOTA: O coeficiente de determinação deve ser superior a 0,98. - Estimar o número de cópias para cada alvo utilizando a equação linear (y = mx + b) derivada da regressão linear (passo 8.7), onde y é o Ct estimado; x é o número da cópia do DNA logarítmico; m é a inclinação da reta, que define a mudança no Ct em relação ao número de cópias do DNA; e b é o intercepto do eixo y que representa a Ct estimada para uma cópia de DNA32.
- Para cada gene marcador calcular a eficiência da amplificação da PCR (E) utilizando os parâmetros da regressão linear da curva padrão e a seguinte equação, onde m é a inclinação derivada dos passos 8.7 e 8.8:

- Valide todos os primers em termos de sua eficiência percentual usando a seguinte equação:
NOTA: A eficiência deve estar na faixa de 90% a 110%.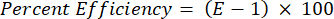
- Calcular o número absoluto de cópias do ADN utilizando a seguinte fórmula:
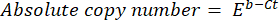
onde Ct (passo 8.8) é o limiar do ciclo, b é o intercepto (passo 8.8), m é a inclinação (passo 8.8) e E é a eficiência da amplificação por PCR (passo 8.9).
CRÍTICO: Ao comparar a amplificação de dois ou mais alvos por q-PCR, a eficiência da PCR deve ser calculada para cada alvo, a fim de comparar os números absolutos de cópias de DNA. - Neste estudo, os genes 16S rRNA, proC e rpoD foram usados como controles internos gerais, e gyrB foi usado como controle de indução33,34,35.
NOTA: Ao escolher controles internos a partir dos dados seq RNA, é melhor selecionar controles internos que não alteram nos níveis de expressão para as condições testadas. A consideração cuidadosa de controles apropriados é sempre importante para a interpretação significativa dos resultados.
Resultados
Neste trabalho, a enumeração temporal direta da produção de fagos a partir de uma cultura lisógena PAO1 LESΦ2 cultivada sob condições não indutoras foi usada para determinar o impacto da indução espontânea de LESΦ2. A densidade de fagos estava em seu ponto mais baixo com uma média de ~2,61 x 106 unidades formadoras de placa (UFP)·mL−1 2 h após o subcultivo em meio fresco durante a fase exponencial inicial de crescimento, sugerindo que a lisogenia foi o estado dominante. O título de LESΦ2 aumentou rapidamente para uma média de ~2,4 x 108 UFP·mL−1 dentro de 4 h e atingiu a maior densidade após 6 h (média de ~5,83 x 109 UFP·mL−1; Gráfico 4).
Mínima indução espontânea foi observada durante a fase logarítmica inicial do crescimento lisogênico (após 2 h). No entanto, a presença mensurável de fagos no meio de cultura foi o resultado de muitos eventos anteriores, incluindo os seguintes: o empacotamento de ácidos nucléicos em cabeças de proteínas, a montagem de proteínas em partículas de fago e a expressão de genes de fagos tardios, genes de fagos de estágio médio e genes de fagos reguladores iniciais. Foi importante capturar as células infectadas antes da expressão dos eventos de replicação associados ao fago; Assim, 90 min foi escolhido para deixar a cultura crescer antes da indução. Para capturar o perfil de expressão gênica da PAO1, amostras de lisogênio LESΦ2 de uma cultura foram colhidas pré-indução e pós-indução durante um período de 90 min, conforme mencionado na etapa 3.4. Este ponto de tempo de 90 minutos é bem antes de altos níveis de indução espontânea do prófago residente serem detectados pelo ensaio de placa do passo 2.3.2. Como a densidade celular bacteriana foi baixa durante o crescimento exponencial inicial, os volumes de cultura foram escalados até 800 mL para garantir amplo material para os estudos de expressão gênica. As amostras foram coletadas da cultura não induzida e induzidas a cada 10 min, e o RNA foi extraído para mapear o perfil de expressão dos principais marcadores de lisogenia e replicação lítica durante o crescimento bacteriano. O RNA total foi purificado e validado para a ausência de DNA genômico usando ensaios de qPCR direcionados ao gene 16S rRNA (passo 6.1). As amostras que atingiram um RIN ≥ 9 passaram pelo controle de qualidade e foram convertidas em cDNA.
O genoma LESΦ2 anotado foi examinado para identificar genes que são atores bem conhecidos nos ciclos de replicação lisogênica e lítica de fagos temperados. Esses genes identificados foram então usados para validar a qRT-PCR para o perfil de expressão dos genes restritos ao ciclo lisonômeo e associados ao ciclo lítico de culturas induzidas e não induzidas. Quantificamos o número absoluto de cópias do DNA e realizamos o teste dos postos sinalizados de Wilcoxon usando R36 para comparar os níveis de expressão em culturas não induzidas e induzidas (Figura 5). Um aumento acentuado na expressão do gene cro (um marcador precoce de replicação lítica) de ~2,31 x 109 cópias em culturas não induzidas para ~3,02 x 1011 cópias 30 min após a indução (teste de Wilcoxon signed-rank: p < 0,01) foi observado. Similarmente, as proteínas O e P, que são marcadores de estágio médio da replicação lítica (e são previstas para estarem envolvidas na replicação do genoma do fago), também mostraram upregulation significativo de ~1,74 x 108 para ~1,25 x 10 10 cópias (teste de Wilcoxon signed-rank: p < 0,01) e de ~ 6,05 x 102 para ~5,68 x10 5 cópias (Wilcoxon signed-rank test: p < 0,01), respectivamente. Finalmente, os genes estruturais associados à cauda foram utilizados como marcadores tardios do ciclo de replicação lítica. Novamente, observamos um aumento significativo na expressão de ~2,31 x 106 cópias em culturas não induzidas para ~4,38 x 108 cópias 30 min após a indução (teste dos postos sinalizados de Wilcoxon: p < 0,01). Assim, os dados quantitativos de RT-PCR confirmaram que a expressão gênica de genes marcadores bem estabelecidos para replicação lítica seguiu a tendência esperada, com os marcadores precoce, médio e tardio apresentando expressão diferencial de múltiplas vezes na ordem predita (Figura 5). Uma vez que a expressão dos marcadores para replicação lítica foi upregulated 30 min pós-recuperação, este é considerado como um ponto de tempo representativo apropriado para estudar a paisagem transcriptômica de fagos temperados ativos e seus hospedeiros bacterianos durante o ciclo lítico.
Observamos alguma expressão de genes líticos em condições não induzidas, confirmando que alguma indução espontânea sempre ocorre, mesmo em culturas otimizadas nas quais os números de lisógenos são representados com a maior proporção de UFC/UFP liberada na fase logarítmica precoce. Isso significa que sempre haverá algum nível de "ruído" nos dados transcriptômicos, o que reforça a importância de controles cuidadosamente preparados, incluindo culturas induzidas e não induzidas. A escolha apropriada dos genes de controle interno para determinar as mudanças de dobra na expressão depende do exame cuidadoso dos dados transcriptômicos para identificar genes que são expressos no mesmo nível nas amostras não induzidas e induzidas. Nossos resultados preliminares sugerem que a rpoD foi o gene controle mais confiável testado e teve a expressão mais estável (~1,71 x 10 5 cópias antes da indução e ~3,33 x 105 cópias 30 min após a indução; Wilcoxon signed rank test: p = 0,3594) em comparação com os genes 16S rRNA ou proC (Figura 5). A variabilidade da expressão dos controles internos levou à mensuração dos números absolutos de transcritos. O exame futuro dos dados transcriptômicos apoiará a escolha de controles internos apropriados para posterior validação.
O gene cI foi utilizado em nosso exercício de perfil genético, por ser um marcador bem reconhecido de lisogenia. Em comparação com os marcadores para replicação lítica, a expressão do gene cI foi relativamente estável (Figura 5), mas o número de cópias desse gene foi reconfortantemente alto nas culturas não induzidas em comparação com as dos marcadores para replicação lítica. Esses dados estão de acordo com os baixos números de UFP nas mesmas amostras, confirmando que a alta expressão de repressores foi associada a menores níveis de produção de fago. Os dados aqui relatados demonstram que a expressão do transcrito cI para este fago em particular não é significativamente diminuída pós-indução, como visto nos fagos Stx11,17. A atividade repressora é normalmente controlada em ambos os níveis transcricional e pós-traducional, de modo que o gene repressor pode ser transcrito, mas a proteína resultante é imediatamente submetida à autoclivagem. Experimentos adicionais são necessários para validar controles transcricionais e pós-traducionais. Além disso, a partir de nossa curva padrão, o limite mínimo de detecção de qPCR parece ser de ~102 cópias.
Juntos, nossos resultados de ensaios de placa e qRT-PCR validam nossa estratégia para cultura e preparação de amostras de RNA para gerar uma entrada bem controlada para experimentos de RNA-Seq. As culturas não induzidas na fase exponencial precoce exibiram baixos níveis de indução espontânea e expressão gênica lítica, sugerindo a dominância da lisogenia. Em contraste, as culturas isoladas 30 min após a indução mostraram aumentos significativos na expressão de genes marcadores que indicam a dominância da replicação lítica.
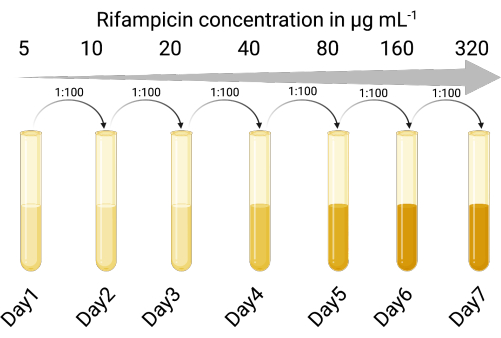
Figura 1: O protocolo para criar o host indicador resistente à rifampicina (Criado com BioRender.com). Clique aqui para ver uma versão maior desta figura.

Figura 2: Desenho experimental para enumeração da UFP e UFC de um lisogênio da mesma amostra. (Criado com BioRender.com) Clique aqui para ver uma versão maior desta figura.

Figura 3: Planejamento experimental para amostragem de culturas induzidas e não induzidas para isolamento de RNA. (Criado com BioRender.com) Clique aqui para ver uma versão maior desta figura.

Figura 4: Enumeração temporal da indução espontânea. Enumeração temporal da produção espontânea de prófagos do LES utilizando a UFP do lisogênio PAO1 Φ2 com a UFC concorrente, n = 8 (duas réplicas biológicas e quatro técnicas); As barras de erro representam o desvio padrão. Os pontos vermelho-escuros indicam o UFC·mL−1 em LB; os pontos azuis escuros indicam a UFP·mL−1 em LB. A liberação espontânea do fago infectante φ2 pelos lisógenos está no nível mais baixo mensurável em 2 h após a inoculação. Clique aqui para ver uma versão maior desta figura.

Figura 5: Número absoluto de cópias dos genes marcadores alvo. O número absoluto de cópias de genes marcadores de fago confirma os padrões de expressão preditos, derivados por RT-qPCR, de genes que se espera que desempenhem papéis importantes na lisogenia e nos ciclos líticos. Os pontos representam três réplicas biológicas e três técnicas (n = 9). (A) A caixa vermelha representa o marcador de lisogenia, cI; (B) o verde representa o marcador lítico inicial, cro; (C,D) azul representa os marcadores líticos médios, genes de replicação de DNA; (E) magenta representa o marcador lítico tardio, genes estruturais da cauda; (F-H) cinza representa os marcadores do hospedeiro que foram usados como controles internos, e (I) branco representa o DNA girase B, que foi usado como controle de indução. As linhas horizontais sólidas mostram a mediana da distribuição. Clique aqui para ver uma versão maior desta figura.
Tabela 1: Primers desenhados neste estudo. As sequências de primers específicos para os genes marcadores e controles internos usados neste estudo são fornecidas, juntamente com suas IDs de acesso NCBI correspondentes. Clique aqui para baixar esta tabela.
Tabela 2: Eficiência dos primers utilizados neste estudo calculada usando a curva padrão da qPCR. Clique aqui para baixar esta tabela.
Discussão
A criação de um hospedeiro indicador selecionável, previamente utilizado em ensaios de placa para quantificar com maior precisão a indução espontânea de fagos Stx de E. coli MC106137,38,39, foi descrita aqui para o fago de P. aeruginosa LESΦ2. Essa intervenção tem o benefício adicional de reduzir as etapas e o tempo de processamento da amostra, permitindo a avaliação simultânea das taxas de indução espontânea em múltiplas condições de cultura. Há risco de gerar outras mutações durante a criação de variantes resistentes à rifampicina40; No entanto, neste trabalho, a cepa evoluída foi utilizada apenas como hospedeiro indicador para a enumeração de placas de culturas de interesse e não foi incluída na análise transcriptômica. Enquanto a cepa indicadora selecionável permanecer igualmente suscetível à infecção pelo fago de interesse, não há preocupação com outras mutações adquiridas. No entanto, não foram detectadas diferenças nos perfis de polimorfismo no comprimento dos fragmentos de restrição pela análise por eletroforese em gel de campo de pulso (PFGE) da PAO1WT e PAO1RIF (dados não mostrados).
Ao escolher células hospedeiras, é raro encontrar uma cepa indicadora que ainda não abriga prófagos. Como exemplo, a PAO1 abriga o prófago filamentoso Pf4. Os controles experimentais para este estudo foram projetados para poder examinar diretamente a expressão gênica de fagos específicos (neste caso, o prófago LES 2) e os efeitos deste fago sobre a expressão gênica bacteriana. Na comparação dos transcritos da PAO1 portadores do prófago LES 2 e sem o prófago 2 do LES (tanto o lisógeno quanto o não lisógeno carregam o Pf4 endógeno), que servem como controles internos para excluir o impacto da Pf4 no hospedeiro. Além disso, tem sido demonstrado que a Pf4 geralmente não causa lise em sua célula hospedeira41 e, portanto, não é capaz de confundir os resultados desses experimentos.
Está bem estabelecido que o controle de qualidade cuidadoso é crucial na preparação de amostras para a produção de dados ômicos significativos42. No entanto, como descritoanteriormente11, a caracterização cuidadosa da atividade profágica na preparação de culturas lisógenas para tais estudos é raramente realizada. Aqui, detalhamos nossos protocolos sistemáticos para preparar um conjunto bem controlado e otimizado de culturas para estudos transcriptômicos para melhor explorar as interações entre bactérias e fagos temperados. A sincronicidade da população foi controlada trazendo-se a cultura por pelo menos quatro duplicações antes de tratá-la com o antibiótico indutor norfloxacina. Ao determinar a CIM de norfloxacina para a cepa no estudo, pudemos garantir que a concentração do agente indutor estivesse um pouco acima da CIM para o tratamento de "indução". As células tratadas foram então diluídas 1:10 para diminuir a concentração de norfloxacina abaixo da CIM após 1 h de tratamento, a fim de permitir que as células se recuperassem e completassem o processo de replicação do fago, terminando na lise da célula e na liberação da progênie infectante do fago. As células só entram no ciclo de replicação lítica após o estímulo de indução quando a concentração de norfloxacina é trazida abaixo da CIM durante o período de recuperação. Nesse caso, ultrapassar 1 μg·mL−1 de norfloxacina significa que a droga não pôde ser efetivamente diluída abaixo da CIM, já que a CIM da norfloxacina para PAO1 é de 0,19 μg·mL−1. O nível de diluição do indutor deve ser equilibrado com a necessidade de recuperação de lisogênio e a retenção da densidade de cultura para a coleta do RNA. Os dados discutidos aqui demonstram que é possível sincronizar culturas para criar amostras nas quais a lisogenia domina, reduzindo assim o ruído da indução espontânea e permitindo a detecção de verdadeiras alterações na expressão gênica impulsionadas pela lisogenia. Uma vez que o estado lisogênico é predominante na fase exponencial inicial do crescimento, quando a densidade celular bacteriana é baixa, sugerimos a ampliação das culturas para coletar RNA suficiente para estudos subsequentes de expressão gênica, como RNA-Seq.
O uso de norfloxacina como agente indutor para forçar culturas ao ciclo lítico é bem relatado43,44; no entanto, isso também afetará a expressão de outros genes bacterianos no processo45,46. Para mitigar isso, bibliotecas de RNA de culturas selvagens de controle cultivadas sob as mesmas condições indutoras e não indutoras devem ser incluídas em experimentos de RNA-Seq. O uso de controles internos e genes marcadores chave para validar os estágios de replicação do fago por qRT-PCR também é crucial para comparações precisas. O perfil quantitativo de RT-PCR não pode ser interpretado comparando o número absoluto de transcritos para cada gene em vários momentos; é a forma do perfil que importa. Primeiro, apenas uma pequena região no transcrito de qualquer gene foi amostrada, portanto, se é um elemento de vida curta ou longa é desconhecido27. Certamente, o mapeamento RNA-Seq de transcritos mostra que a densidade dos dados de mapeamento varia significativamente ao longo do comprimento de um gene. Em segundo lugar, é a forma do perfil de expressão gênica que deve ser interpretada para um gene marcador associado ao ciclo lítico ou ao estilo de vida lisogênico ou mesmo desacoplado dos circuitos reguladores do fago11. A indução espontânea é um problema real em cultura lisógena e sempre resultará na expressão de genes associados ao ciclo lítico. No entanto, o perfilamento mostra que os genes associados ao ciclo de replicação lítica são suprimidos em sua expressão pré-indução (pelo menos duas dobras logarítmicas) e up-regulated pós-indução.
As análises transcriptômicas previamente conduzidas das interações de fagos de Stx com E. coli suportam uma compreensão completa dos genes de fagos envolvidos na manutenção da lisogenia e no desencadeamento do ciclo lítico11,17. Atualmente, os fagos do LES de P. aeruginosa têm sido anotados, mas suas funções-chave gênicas são menos bem compreendidas. Estudos transcriptômicos permitirão a re-anotação dos prófagos do LES e melhorarão nossa compreensão dos genes envolvidos na lisogenia e no ciclo lítico. A ligação da sequência gênica à função representa um grande desafio no estudo de novos prófagos, o que ressalta ainda mais a necessidade de mais estudos para confirmar as funções dos genes dos fagos para a produção de melhores ferramentas de anotação47. A aplicação mais ampla e a adaptação dos protocolos e medidas extras de controle de qualidade detalhadas neste artigo em vídeo podem ajudar a desvendar várias funções do prófago e, assim, melhorar os pipelines de anotação e transformar nossa compreensão da biologia do fago e da bactéria.
Materiais
| Name | Company | Catalog Number | Comments |
| PAO1 | 6 | ||
| LESB58 | 6 | ||
| LES phages | Induced and purified from LESB58 using Norfloxacin. | This study | |
| Lysogeny Broth (LB) | Merck | 1.10285.500 | |
| LB Agar | Merck | 1.10283.500 | |
| Agar Agar | Fisher | A/1080/53 | |
| Top Agar | 0.4 g Agar Agar+2.5 g LB Broth in 100 mL water; autoclave and use. | - | |
| Rifampicin | Sigma (Stock: 50 mg/mL in Methanol- Mix well and use 0.22µm filter to sterilize and store it in -20°C until use) | R3501 | |
| Glacial Acetic Acid | Fisher 1% (v/v) in water | 10060000 | |
| Norfloxacin | Sigma (Stock: 25 mg/mL of 1% Glacial Acetic Acid-Mix well and use 0.22µm filter to sterilize and store it in -20°C until use;To avoid freeze thaw cycles, store as small aliquotes) | N9890 | |
| Phenol saturated with citrate buffer pH 4.3 | Sigma | P-4682 | |
| Molecular Biology grade Ethanol | Fisher | 16695992 | |
| TRIzol | Invitrogen | 12044977 | |
| Chloroform | Fisher | 11398187 | |
| Isopropanol | Fisher | 17150576 | |
| Nuclease-free H2O | Invitrogen | 10526945 | |
| 10X TURBO DNase | Ambion | AM1907 | |
| Qubit RNA HS, BR Kit | Invitrogen | Q10210 | |
| Agilent RNA 6000 Nano Kit | Agilent | 5067-1511 | |
| SuperScriptIII first strand synthesis kit | Invitrogen | 18080051 | |
| PCR Reagents | Bioline Mytaq Red 2X | BIO-25043 | |
| qPCR Reagents | Sensifast SYBR Hi Rox | BIO-92020 | |
| PCR purification kit | Isolate II PCR and Gel Kit | BIO-52060 | |
| TA cloning kit | TA Cloning Kit, with pCR 2.1 Vector, without competent cells | K202040 | |
| StepOne Real Time PCR system | Thermo Fisher Scientific | 4376600 |
Referências
- Lin, D. M., Koskella, B., Lin, H. C. Phage therapy: An alternative to antibiotics in the age of multi-drug resistance. World Journal of Gastrointestinal Pharmacology and Therapeutics. 8 (3), 162-173 (2017).
- Jiang, W., Marraffini, L. A. CRISPR-Cas: New tools for genetic manipulations from bacterial immunity systems. Annual Review of Microbiology. 69 (1), 209-228 (2015).
- Santos, S. B., Azeredo, J. Bacteriophage-based biotechnological applications. Viruses. 11 (8), 737 (2019).
- Rodríguez-Rubio, L., Jofre, J., Muniesa, M. Is genetic mobilization considered when using bacteriophages in antimicrobial therapy. Antibiotics. 6 (4), 32 (2017).
- Hatfull, G. F. Dark Matter of the biosphere: The amazing world of bacteriophage diversity. Journal of Virology. 89 (16), 8107-8110 (2015).
- Yukgehnaish, K., et al. PhageLeads: Rapid assessment of phage therapeutic suitability using an ensemble Machine Learning approach. Viruses. 14 (2), 342 (2022).
- Seemann, T. Prokka: Rapid prokaryotic genome annotation. Bioinformatics. 30 (14), 2068-2069 (2014).
- Arndt, D., et al. PHASTER: A better, faster version of the PHAST phage search tool. Nucleic Acids Research. 44, W16-W21 (2016).
- Banerjee, S., et al. FINDER: An automated software package to annotate eukaryotic genes from RNA-Seq data and associated protein sequences. BMC Bioinformatics. 22 (1), 205 (2021).
- Jumper, J., et al. Highly accurate protein structure prediction with AlphaFold. Nature. 596 (7873), 583-589 (2021).
- Veses-Garcia, M., et al. Transcriptomic analysis of Shiga-toxigenic bacteriophage carriage reveals a profound regulatory effect on acid resistance in Escherichia coli. Applied and Environmental Microbiology. 81 (23), 8118-8125 (2015).
- Owen, S. V., et al. A window into lysogeny: revealing temperate phage biology with transcriptomics. Microbial Genomics. 6 (2), e000330 (2020).
- Davies, E. V., Winstanley, C., Fothergill, J. L., James, C. E. The role of temperate bacteriophages in bacterial infection. FEMS Microbiology Letters. 363 (5), 015 (2016).
- Livny, J., Friedman, D. I. Characterizing spontaneous induction of Stx encoding phages using a selectable reporter system. Molecular Microbiology. 51 (6), 1691-1704 (2004).
- Fogg, P. C. M., et al. Identification of multiple integration sites for Stx-phage Φ24B in the Escherichia coli genome, description of a novel integrase and evidence for a functional anti-repressor. Microbiology. 153 (12), 4098-4110 (2007).
- James, C. E., et al. Differential infection properties of three inducible prophages from an epidemic strain of Pseudomonas aeruginosa. BMC Microbiology. 12, 216 (2012).
- Riley, L. M., et al. Identification of genes expressed in cultures of E. coli lysogens carrying the Shiga toxin-encoding prophage Φ24B. BMC Microbiology. 12 (1), 42 (2012).
- Stover, C. K., et al. Complete genome sequence of Pseudomonas aeruginosa PAO1, an opportunistic pathogen. Nature. 406 (6799), 959-964 (2000).
- Winstanley, C., et al. Newly introduced genomic prophage islands are critical determinants of in vivo competitiveness in the Liverpool Epidemic Strain of Pseudomonas aeruginosa. Genome Research. 19 (1), 12-23 (2009).
- Davies, E. V., et al. Temperate phages enhance pathogen fitness in chronic lung infection. The ISME Journal. 10 (10), 2553-2555 (2016).
- Allison, H. E. Stx-phages: drivers and mediators of the evolution of STEC and STEC-like pathogens. Future Microbiology. 2 (2), 165-174 (2007).
- Allison, H. E., et al. Immunity profiles of wild-type and recombinant Shiga-like toxin-encoding bacteriophages and characterization of novel double lysogens. Infection and Immunity. 71 (6), 3409-3418 (2003).
- Mori, N., et al. A peptide based on homologous sequences of the β-barrel assembly machinery component BamD potentiates antibiotic susceptibility of Pseudomonas aeruginosa. Journal of Antimicrobial Chemotherapy. 67 (9), 2173-2181 (2012).
- Chojnacki, M., et al. A novel, broad-spectrum antimicrobial combination for the treatment of Pseudomonas aeruginosa corneal infections. Antimicrobial Agents and Chemotherapy. 63 (10), e00777 (2019).
- Miles, A. A., Misra, S. S., Irwin, J. O. The estimation of the bactericidal power of the blood. Epidemiology & Infection. 38 (6), 732-749 (1938).
- Srikumar, S., et al. RNA-seq brings new insights to the intra-macrophage transcriptome of Salmonella Typhimurium. PLoS Pathogens. 11 (11), e1005262 (2015).
- Kröger, C., et al. The transcriptional landscape and small RNAs of Salmonella enterica serovar Typhimurium. Proceedings of the National Academy of Sciences of the United States of America. 109 (20), E1277-E1286 (2012).
- Green, M. R., Sambrook, J. How to win the battle with RNase. Cold Spring Harbor Protocols. (2), (2019).
- A practical guide to analyzing nucleic acid concentration and purity with microvolume spectrophotometers. New England BioLabs Inc Available from: https://www.neb.com/-/media/nebus/files/application-notes/technote_mvs_analysis_of_nucleic_acid_concentration_and_purity.pdf?rev=c24cea043416420d84fb6bf7b554dbbb (2019)
- Saunders, N. A., Lee, M. A. . Real-Time PCR: Advanced Technologies and Applications. , (2013).
- Bustin, S. A. . A-Z of Quantitative PCR. , (2004).
- Ruijter, J. M., et al. Efficiency correction is required for accurate quantitative PCR analysis and reporting. Clinical Chemistry. 67 (6), 829-842 (2021).
- Fothergill, J. L., Neill, D. R., Loman, N., Winstanley, C., Kadioglu, A. Pseudomonas aeruginosa adaptation in the nasopharyngeal reservoir leads to migration and persistence in the lungs. Nature Communications. 5 (1), 4780 (2014).
- Huang, J., et al. Temperature-dependent expression of phzM and its regulatory genes lasI and ptsP in rhizosphere isolate Pseudomonas sp. strain M18. Applied and Environmental Microbiology. 75 (20), 6568-6580 (2009).
- Savli, H., et al. Expression stability of six housekeeping genes: a proposal for resistance gene quantification studies of Pseudomonas aeruginosa by real-time quantitative RT-PCR. Journal of Medical Microbiology. 52 (5), 403-408 (2003).
- . rstatix: Pipe-Friendly Framework for Basic Statistical Tests Available from: https://CRAN.R-project.org/package=rstatix (2022)
- McDonald, J. E., et al. High-throughput method for rapid induction of prophages from lysogens and its application in the study of Shiga toxin-encoding Escherichia coli strains. Applied and Environmental Microbiology. 76 (7), 2360-2365 (2010).
- Smith, D. L., et al. Short-tailed Stx phages exploit the conserved YaeT protein to disseminate Shiga toxin genes among Enterobacteria. Journal of Bacteriology. 189 (20), 7223-7233 (2007).
- James, C. E., et al. Lytic and lysogenic infection of diverse Escherichia coli and Shigella strains with a verocytotoxigenic bacteriophage. Applied and Environmental Microbiology. 67 (9), 4335-4337 (2001).
- Rees, V. E., et al. Characterization of hypermutator Pseudomonas aeruginosa isolates from patients with cystic fibrosis in Australia. Antimicrobial Agents and Chemotherapy. 63 (4), e02538 (2019).
- Li, Y., et al. Excisionase in Pf filamentous prophage controls lysis-lysogeny decision-making in Pseudomonas aeruginosa. Molecular Microbiology. 111 (2), 495-513 (2019).
- Van Kampen, A. H. C., Moerland, P. D. Taking bioinformatics to systems medicine. Systems Medicine. 1386, 17-41 (2016).
- Matsushiro, A., Sato, K., Miyamoto, H., Yamamura, T., Honda, T. Induction of prophages of enterohemorrhagic Escherichia coli O157:H7 with norfloxacin. Journal of Bacteriology. 181 (7), 2257-2260 (1999).
- James, C. E., et al. Lytic activity by temperate phages of Pseudomonas aeruginosa in long-term cystic fibrosis chronic lung infections. The ISME Journal. 9 (6), 1391-1398 (2015).
- Shaw, K. J., et al. Comparison of the changes in global gene expression of Escherichia coli induced by four bactericidal agents. Microbial Physiology. 5 (2), 105-122 (2003).
- Long, H., et al. Antibiotic treatment enhances the genome-wide mutation rate of target cells. Proceedings of the National Academy of Sciences of the United States of America. 113 (18), E2498-E2505 (2016).
- González-Tortuero, E., et al. VIGA: A sensitive, precise and automatic de novo VIral Genome Annotator. bioRxiv. , (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados