Method Article
Comprendere l'impatto dei batteriofagi temperati sui loro lisogeni attraverso la trascrittomica
In questo articolo
Riepilogo
Questo protocollo consente di rivelare l'impatto dei profagi sui loro ospiti. Le colture batteriche sono sincronizzate utilizzando le condizioni che meglio supportano lo stato lisogenico, limitando l'induzione spontanea. La RT-qPCR distingue inequivocabilmente i geni ristretti dai profagi e quelli disaccoppiati dal controllo fagico da quelli espressi durante il ciclo di replicazione litica.
Abstract
I fagi temperati si trovano integrati come profagi nella maggior parte dei genomi batterici. Alcuni profagi sono criptici e fissati nel cromosoma batterico, ma altri sono attivi e possono essere innescati in una forma replicativa spontaneamente o per esposizione a fattori induttori. I profagi sono comunemente associati alla capacità di conferire la produzione di tossine o altri tratti associati alla virulenza sulla loro cellula ospite. Studi più recenti hanno dimostrato che possono svolgere un ruolo molto più importante nell'alterare la fisiologia dei loro ospiti. La tecnica qui descritta ci ha permesso di studiare come i profagi influenzano l'espressione genica nel batterio opportunista Pseudomonas aeruginosa.
In questo lavoro, la crescita del ceppo wild-type PAO1 di P. aeruginosa è stata confrontata con quella dei lisogeni isogenici che trasportano diverse combinazioni di profagi dal ceppo epidemico di Liverpool (LES) LESB58. In una coltura di lisogena, una parte delle cellule batteriche supporterà la replicazione dei batteriofagi litici (induzione spontanea) con un alto livello di espressione per cellula di geni fagici tardivi, come quelli associati all'assemblaggio di particelle fagiche, mascherando così l'espressione genica di basso livello associata all'espressione genica ristretta al lisogeno. L'impatto dell'induzione spontanea può quindi oscurare l'espressione genica dei profagi in una popolazione lisogena.
Gli esperimenti di profilazione della crescita sono stati utilizzati per identificare l'induzione spontanea, che era minima durante la prima fase di crescita esponenziale. Questo studio illustra come preparare le colture campione durante la fase iniziale di crescita esponenziale e come impostare controlli adeguati nonostante il basso numero di cellule. Questi protocolli garantiscono il confronto affidabile e riproducibile di batteri wild-type e lisogeni in varie condizioni, migliorando così il profilo trascrittomico dei genomi dei profagi e aiutando nell'identificazione di funzioni profagiche precedentemente non riconosciute.
Introduzione
Recentemente, la terapia fagica per affrontare la resistenza antimicrobica1 e l'editing genetico basato su CRISPR-Cas2 hanno generato un rinnovato interesse per la ricerca sui batteriofagi. Ancora una volta, i progressi della biotecnologia hanno permesso di studiare più a fondo le interazioni tra batteri e fagi3. Tuttavia, l'uso terapeutico dei fagi ("terapia fagica") è ostacolato dalle preoccupazioni sui fagi che agiscono come elementi genetici mobili con la capacità di trasferire orizzontalmente geni di virulenza e resistenza4. La distesa di "materia oscura"5 (geni con funzioni sconosciute) è sia preoccupante che allettante. La materia oscura è considerata una lacuna nella nostra comprensione della biologia dei fagi e una risorsa in gran parte non sfruttata per gli strumenti molecolari e le potenziali nuove terapie. Lo sviluppo di tecniche di sequenziamento ad alto rendimento, insieme a una migliore annotazione genica 7,8,9 e a nuovi algoritmi di ripiegamento peptidico10, sta migliorando il rilevamento, la descrizione e la predizione funzionale dei geni fagici. Tuttavia, la scienza è ancora lontana dal convalidare le funzioni genetiche della maggior parte dei fagi in coltura o nel mondo reale.
Il sequenziamento dell'RNA (RNA-Seq) è in grado di mappare globalmente l'espressione genica durante l'infezione fagica e ha migliorato significativamente la comprensione degli elementi fagici e batterici coinvolti nei cicli litici e lisogenici11,12. Durante i processi lisogenici, i genomi dei fagi temperati vengono integrati nel DNA batterico per diventare profagi13. Gli esperimenti di profilazione dell'espressione genica globale possono essere utilizzati per identificare i geni ristretti dai profagi che sono codificati sui genomi dei fagi temperati ma espressi solo durante lo stato lisogenico11. Tali geni non codificano proteine strutturali dei fagi e non sono coinvolti in alcun processo di infezione dei fagi. RNA-Seq può essere utilizzato per identificare quei geni che hanno maggiori probabilità di influenzare la biologia dell'ospite batterico, inducendo un guadagno di funzione o regolando i geni batterici esistenti, consentendo così spesso ai batteri di adattarsi ai cambiamenti ambientali. Pertanto, potrebbe essere studiata la capacità dei profagi di agire come burattinai microbici, controllando una serie di funzioni batteriche.
Ci sono due principali ostacoli all'analisi efficace dell'espressione genica limitata ai profagi. In primo luogo, la disponibilità di host suscettibili è una questione chiave. Per definizione, i profagi sono già incorporati nel loro genoma specifico dell'ospite, quindi è difficile trovare un ospite wild-type suscettibile per confrontare l'espressione genica globale in presenza e in assenza del profago. Ciò può essere ottenuto attraverso l'infezione de novo di un altro ospite suscettibile o la delezione del profago dall'isolato wild-type originale, senza interrompere il resto del genoma dell'ospite. La seconda barriera risiede nella natura eterogenea delle popolazioni lisogeniche. Alcuni profagi si degradano attraverso la mutazione o la ricombinazione per diventare "criptici", il che significa che sono fissati in una posizione specifica del genoma batterico. Tuttavia, altri profagi sono "attivi" e possono essere indotti in un ciclo litico replicativo spontaneamente o dopo l'esposizione a fattori induttori. In molte colture lisogeniche, il tasso di induzione spontanea significa che una parte delle cellule batteriche è sempre sottoposta a replicazione fagica litica14,15,16. Un alto livello di espressione dei geni fagici tardivi in queste popolazioni maschera l'espressione genica di basso livello associata all'espressione genica ristretta al lisogeno11,17. La proporzione di lisogeni sottoposti a induzione spontanea dei profagi può variare in base allo stato di crescita, alle condizioni di crescita o ad altri fattori scatenanti. Pertanto, per studiare gli impatti dei profagi sul lisosogeno, gli eventi spontanei di induzione dei profagi devono essere minimizzati il più possibile ottimizzando le condizioni di crescita per favorire lo stato lisogenico.
Questo studio riporta il lavoro preparatorio svolto per studiare l'influenza di una serie di profagi conviventi del ceppo epidemico di Liverpool (LES) di Pseudomonas aeruginosa. I profagi attivi sono stati indotti e isolati da LES e utilizzati per infettare il ceppo ospite modello di P. aeruginosa, PAO116,18,19. L'intero genoma del ceppo wild-type di P. aeruginosa, PAO1, e del suo lisosogeno, PAO1Φ2, è stato sequenziato (ad una profondità di copertura 30x) per garantire l'identità del ceppo wild-type e per confermare che il lisogeno era isogenico. Il LES è stato associato ad un aumento della morbilità e della mortalità nei pazienti con fibrosi cistica e i fagi LES 19 sono stati suggeriti per aiutare l'adattamento all'ambiente polmonare della fibrosi cistica16,19,20. Nonostante la forte evidenza che questi profagi influenzano la biologia del loro ospite20,21, la maggior parte delle loro funzioni geniche devono ancora essere caratterizzate e i meccanismi specifici di interazione sono poco compresi. Un approccio trascrittomico può scoprire empiricamente le funzioni del gene profago in un contesto ospite controllato. Poiché l'induzione spontanea può influenzare i profili di espressione, questo articolo descrive come ottimizzare le condizioni di crescita per favorire lo stato lisogenico. Tale sincronizzazione delle colture può essere convalidata mediante PCR in tempo reale per quantificare i livelli di espressione di marcatori genetici chiave associati a fasi cruciali della replicazione dei fagi LES in PAO1. Lo stesso approccio è stato utilizzato in precedenza per identificare le funzioni profagiche dei fagi tossigeni Shiga che influenzano la motilità, la resistenza agli acidi e la resistenza antimicrobica in Escherichia coli11,17,21,22.
Protocollo
1. Creare un host indicatore selezionabile (Figura 1)
NOTA: I lisati di coltura fagica possono contenere cellule contaminanti dell'ospite batterico originale. Avere un ceppo indicatore resistente agli antibiotici consente la discriminazione tra il ceppo indicatore e l'ospite batterico originale del profago. L'utilizzo di un ceppo indicatore selezionabile consente l'enumerazione accurata delle particelle fagiche infettive senza richiedere fasi di centrifugazione o filtrazione per rimuovere il fago dalle cellule lisogene dopo le fasi di amplificazione dei fagi. Il ceppo ospite indicatore selezionabile riduce anche il tempo e il numero di passaggi per l'enumerazione dei fagi in modo che più condizioni possano essere sperimentate contemporaneamente.
- Identificare un ceppo ospite indicatore adatto suscettibile all'infezione litica e lisogena da parte del fago temperato di interesse. È stato utilizzato il ceppo da laboratorio di P. aeruginosa PAO118,20 ed è suscettibile ai tre fagi LES (LESΦ2, LESΦ3 e LESΦ4).
- Scegliere un agente selettivo adatto (in questo caso è stata utilizzata la rifampicina) ed eseguire un saggio di diluizione del brodo per determinare la concentrazione minima inibitoria (MIC) per l'ospite indicatore (16 μg·mL−1 è il MIC per PAO1)23,24.
- Esporre in sequenza le colture dell'ospite indicatore a concentrazioni crescenti dell'agente selettivo nel brodo di lisogenia (LB), a partire da sotto la MIC (in questo caso 5 μg·mL−1), per 18-24 ore, con agitazione, e a 37 °C.
- Trasferire la coltura che cresce alla massima concentrazione in un rapporto di 1:100 (inoculo e terreno) in concentrazioni raddoppiate dell'agente selettivo (18-24 ore ogni volta) fino a quando la MIC non è stata sufficientemente aumentata. PAO1 è diventato un ceppo resistente alla rifampicina (PAO1-RifR) a 300 μg·mL−1 di rifampicina.
2. Enumerazione diretta temporale dell'induzione spontanea (Figura 2)
- Preparare colture starter notturne sia del lisogeno (ad esempio, P. aeruginosa PAO1 lysogen che ospita fagi LES) che dell'ospite indicatore (PAO1-RifR) inoculando una singola colonia in 5 mL di LB e incubare a 37 °C con agitazione a 180 rpm (18-24 h).
- Preparare le colture lisogene fresche e le colture ospiti indicatrici inoculando le colture notturne in 100 mL di LB in un rapporto di 1:100 e incubare a 37 °C con agitazione (180 giri/min).
- Monitorare la crescita del lisogeno misurando l'OD600 e la conta vitale utilizzando la tecnica di Miles Misra25. Per fare ciò, raccogliere un campione di 1 mL da ciascuna coltura di lisogeno ogni ora dal punto di inoculazione per 8 ore.
- Diluire in serie il campione subito dopo il prelievo aggiungendo 100 μL del campione in 900 μL del rispettivo terreno. Vorticare bene alla massima velocità, scartare la punta ad ogni diluizione e continuare la serie di diluizione da 10−1 a 10−9.
- Versare 10 μL delle diluizioni richieste in triplice copia su una piastra di agar LB, lasciare asciugare e incubare a 37 °C per 18-24 ore.
- Per calcolare il numero di cellule batteriche vitali, trovare una diluizione con colonie facilmente numerabili. Contare il numero di colonie in ogni punto, quindi utilizzare la seguente formula:

NOTA: Man mano che la coltura di lisogeno cresce, le particelle fagiche attive saranno prodotte per induzione spontanea. La produzione di fagi infettivi significa che il trascrittoma della popolazione lisogena è ora contaminato dall'espressione genica associata al ciclo di replicazione litica dal segnale di replicazione del fago litico e dalla corrispondente risposta della cellula ospite. È quindi importante identificare la fase di crescita in cui la proporzione di cellule lisogene rispetto alle particelle fagiche infettive libere è più alta, al fine di limitare il più possibile il rumore di trascrizione di fondo (generato dal trascrittoma del fago litico) nel set di dati.
- Per enumerare le particelle fagiche infettive in ciascun campione temporale, inoculare 5 mL di agar batteriologico sterile allo 0,4% in LB (agar superiore) con 100 μL di ospite indicatore di fase esponenziale medio (OD600: 0,4-0,5; in questo caso, PAO1-RifR) in presenza di un agente selettivo appropriato (50 μg·mL−1 rifampicina in questo caso, poiché la MIC dell'ospite PAO1 è di soli 16 μg·mL−1; vedi Tabella dei materiali, riga 8 e riga 9)
- Versare 10 μL della stessa diluizione seriale (vedere fase 2.2.2) sullo strato superiore di agar inoculato e lasciare asciugare prima dell'incubazione a 37 °C per 18-24 ore.
- Per calcolare le particelle fagiche infettive, trovare una diluizione con placche facilmente numerabili. Conta il numero di placche in ogni punto.

- Trovare il tempo/condizione per il quale l'induzione spontanea per CFU (unità formante colonia) è minima per le ulteriori fasi sperimentali.
3. Preparazione di colture di lisogena non indotta e indotta per l'estrazione dell'RNA (Figura 3)
- Preparare una nuova coltura durante la notte inoculando una singola colonia di lisogeno in 5 mL di LB e incubare a 37 °C con agitazione (180 giri/min) per 18-24 ore.
- Subcoltura la coltura notturna in 80 mL di LB in un rapporto di 1:100 in otto palloni da 250 mL.
- Etichettare il primo matraccio come "non indotta" e gli altri come "indotta", insieme ai punti temporali in cui ciascun campione deve essere prelevato (ad esempio, "t indotta = 0", "t indotta = 10 min", "t indotta = 20 min", ecc.; Figura 3).
- Dopo 90 minuti di incubazione, quando l'OD600 è compreso tra 0,1 e 0,2, o al momento dell'induzione spontanea minima (vedere la discussione), aggiungere 4 μL di acido acetico glaciale all'1% (v/v) al matraccio non indotto (Figura 3).
NOTA: Poiché l'agente induttore in questo lavoro è stato prodotto utilizzando acido acetico glaciale all'1% come solvente, la stessa quantità di solvente è stata aggiunta da sola come fase di controllo. Possono essere presi in considerazione controlli alternativi a seconda della preparazione di diversi induttori. - Aggiungere la coltura da 80 mL dal matraccio non indotto a 720 mL di LB sterile e aggiungere immediatamente la soluzione di arresto (fenolo ghiacciato al 5% [v/v], pH 4,3, etanolo al 95% [v/v]) utilizzando un volume pari al 20% del volume di coltura (160 mL) e incubare su ghiaccio per un minimo di 30 minuti e non più di 2 ore per stabilizzare i trascritti dell'RNA12, 26,27. Questo è il campione non indotto.
- Indurre le colture rimanenti in sette matracci da 250 mL (Figura 3) con la MIC di un agente induttore appropriato (in questo caso, 25 mg·mL−1 di norfloxacina, preparata in acido acetico glaciale all'1% [p/v], utilizzata a una concentrazione finale di 1 μg·mL−1), mescolare bene e incubare a 37 °C e agitando a 180 giri/min per 1 ora.
NOTA: questo passaggio forzerà la coltura lisogena in uno stato più coordinato di replicazione litica. La maggior parte delle cellule della coltura inizierà a subire la produzione litica di particelle fagiche infettive. - Lasciare che le cellule si riprendano aggiungendo 80 mL di coltura dal matraccio indotto a 720 mL di LB sterile, che diluisce efficacemente l'agente induttore. Prelevare le cellule batteriche da ciascun matraccio ogni 10 minuti dal tempo 0 a 1 ora aggiungendo una soluzione di arresto, come indicato al punto 3.5.
NOTA: La soluzione di arresto stabilizza l'RNA per un massimo di 2 ore. Tuttavia, per migliorare la stabilità del campione, eseguire tutti gli ulteriori passaggi a 4 °C. - Raccogliere per centrifugazione a 10.000 x g per 15 minuti a 4 °C il prima possibile, non oltre 2 ore dopo il trattamento per evitare la degradazione dell'RNA.
- Scartare il surnatante e risospendere delicatamente i pellet batterici nel liquido residuo utilizzando una pipetta automatica regolabile prima di trasferire ciascun campione in una provetta da microfugo da 1,5 mL.
- Centrifugare le provette del microfugo ad alta velocità (13.000 x g) in un microfugo a 4 °C per 1 minuto ed eliminare il surnatante residuo.
- Congelare i pellet immergendo ogni tubo di microfusione sigillato nell'azoto liquido. Ciò aiuterà l'efficiente lisi delle cellule per l'estrazione dell'RNA.
- Aggiungere TRIzol (1 mL) a ciascun pellet congelato e omogeneizzare la sospensione mediante pipettaggio (non vorticare). Conservare a -80 °C fino al momento di eseguire l'estrazione dell'RNA per tutti i campioni.
NOTA: A questo punto il protocollo può essere messo in pausa. - Ripetere i passaggi da 3.1 a 3.13 con tre repliche biologiche.
4. Isolamento di RNA da colture lisogene non indotte e indotte
CRITICO: Tutti questi passaggi devono essere eseguiti in un ambiente privo di RNasi28. I banchi di lavoro devono essere puliti con NaClO al 10% o inattivatori proprietari di RNasi. Il materiale di laboratorio deve essere trattato con inibitori della RNasi come il trattamento DEPC e in tutte le reazioni deve essere utilizzata acqua priva di nucleasi.
- Scongelare i pellet congelati trattati con TRIzol dalla fase 3.12 su ghiaccio e aggiungere 400 μL di cloroformio di grado biologico molecolare.
- Agitare bene i flaconcini mediante inversione per 10 s per completare la lisi di tutte le cellule ( non vorticare). Quindi, incubare a temperatura ambiente (21 °C) per 2-5 minuti.
- Separare lo strato acquoso dalla miscela TRIzol/cloroformio mediante centrifugazione utilizzando un microfugo da tavolo refrigerato a 4 °C e 13.000 x g per 15 min.
- Raccogliere la fase acquosa (~ 500 μL, strato superiore) utilizzando una pipetta da 1.000 μL, facendo attenzione a non disturbare l'interfase o la fase organica (strato inferiore). Trasferire in una nuova provetta da 1,5 mL.
- Aggiungere 450 μL di isopropanolo di grado biologico molecolare alla fase acquosa separata, mescolare bene per inversione ( non vorticare) e incubare a temperatura ambiente (21 °C) per 30 minuti.
- Recuperare l'RNA mediante centrifugazione utilizzando una centrifuga refrigerata a 4 °C e 13.000 x g per 30 min.
- Scartare il surnatante senza disturbare il pellet di RNA e lavare il pellet due volte con 800 μL di etanolo al 70 % preparato con acqua priva di nucleasi (non pipettare su e giù). Garantire la stabilità del pellet di RNA ripetendo la fase di centrifugazione per 5 minuti dopo ogni lavaggio.
- Scartare l'etanolo e asciugare il pellet all'aria.
NOTA: Aspirare con cautela l'etanolo intorno al pellet utilizzando una micropunta da 10 μL e asciugare il pellet capovolgendo la provetta su carta assorbente pulita. Il pellet di RNA dovrebbe diventare incolore e i bordi dovrebbero apparire increspati e visibili. Un'essiccazione insufficiente può lasciare etanolo residuo che può influire sui processi a valle e l'essiccazione eccessiva del pellet può rendere difficile la risospensione. - Risospendere l'RNA in acqua priva di nucleasi (50 μL) incubando a 65 °C su un termo-shaker con miscelazione intermittente (ogni 30 s) per un totale di 3-5 min.
CRITICO: Il gruppo 2'-OH dell'RNA è in grado di catalizzare l'autoclivaggio dei filamenti di RNA ad una temperatura elevata superiore a 65 °C e ad un pH elevato. Temperature inferiori a 65 °C ritardano la risospensione del DNA residuo, limitando così la quantità di DNA che deve essere digerita in una fase successiva con la digestione della DNasi I. Pertanto, mantenere la temperatura a 65 °C è fondamentale per ottenere i migliori campioni.
NOTA: A questo punto il protocollo può essere messo in pausa e i campioni possono essere conservati a -80 °C.
5. Rimozione del DNA contaminante dall'RNA mediante trattamento con DNasi
- Per rimuovere il DNA contaminante dall'RNA totale prima della sintesi del cDNA del primo filamento, aggiungere un volume 0,1 di tampone DNasi 10x e 1 μL di enzima DNasi a 10 μg di RNA totale. Mescolare delicatamente la provetta e incubare a 37 °C per 30 min.
- Risospendere il reagente di inattivazione della DNasi e aggiungere un minimo di 2 μL o un volume del 10% del volume totale di reazione. Mescolare bene e incubare i campioni per 5 minuti a temperatura ambiente (21 °C) durante la ridispersione del reagente di inattivazione della DNasi.
- Pellet i reagenti DNasi mediante centrifugazione utilizzando una microcentrifuga da tavolo a 10.000 × g per 1,5 min.
- Trasferire il surnatante contenente l'RNA in una provetta fresca senza disturbare il pellet.
NOTA: Controllare la qualità dell'RNA utilizzando uno spettrofotometro UV in scala 1 μL e un analizzatore computerizzato di acidi nucleici basato su microfluidica secondo le istruzioni del produttore; l'RNA totale purificato può essere conservato a −80 °C. Per la qRT-PCR, l'RNA potrebbe essere utilizzato direttamente a questo punto. Per i processi a valle più sensibili, come il sequenziamento dell'RNA, che richiedono una rigorosa qualità del campione, è necessario raggiungere un rapporto A260/230 di ε 2,0 per procedere ulteriormente. - Portare il volume della soluzione di RNA privo di DNA a 500 μL utilizzando acqua priva di nucleasi.
- Aggiungere 50 μL di acetato di sodio 3 M senza nucleasi (pH 5,3) e 495 μL di isopropanolo. Mescolare bene e incubare a temperatura ambiente per 30 minuti.
NOTA: Questo passaggio farà precipitare l'RNA. - Recuperare l'RNA mediante centrifugazione a 13.000 x g e 4 °C per 30 min.
- Lavare il pellet di RNA tre volte con etanolo ghiacciato al 70% centrifugando i campioni a 13.000 x g e 4 °C per 5 minuti dopo ogni lavaggio per rimuovere completamente i sali.
- Controllare la qualità dell'RNA utilizzando uno spettrofotometro UV in scala 1 μL e un analizzatore computerizzato di acidi nucleici basato sulla microfluidica secondo le istruzioni del produttore; l'RNA totale purificato può essere conservato a −80 °C.
NOTA: La guida29 è stata utilizzata per raggiungere gli standard di qualità dell'RNA. Se il rapporto A260/230 è <2,0, ripetere i passaggi da 5,5 a 5,9.
6. Analisi qualitativa e quantitativa dell'RNA DNase-free
- Convalidare l'efficienza del trattamento con DNasi per ciascun campione eseguendo una PCR quantitativa utilizzando primer di rRNA 16S (Tabella 2) con 1 μg di RNA totale e confermare che non viene prodotto alcun prodotto di amplificazione.
NOTA: I primer ideali per valutare la contaminazione da gDNA sono i primer progettati per ricuocere le giunzioni introne-esone o le regioni regolatorie nei procarioti o nei siti trascrizionalmente inattivi30,31. - Determinare il numero di integrità dell'RNA (RIN) utilizzando un analizzatore computerizzato di acidi nucleici basato sulla microfluidica secondo le istruzioni del produttore.
NOTA: I campioni che mostrano un RIN ≥ 9 devono essere utilizzati per la sintesi del primo filamento. I campioni che mostrano un RIN < 9 devono essere scartati e le fasi di isolamento (1,1-5,4) devono essere ripetute. - Quantificare la concentrazione totale di RNA utilizzando il kit del test HS RNA e un fluorimetro secondo le istruzioni del produttore.
7. Sintesi del cDNA del primo filamento
- Preparare una miscela di primer di RNA per ciascun campione mescolando 1 μg di RNA totale con 1 μL di esameri casuali (50 ng·μL−1) e 1 μL di miscela di dNTP da 10 mM. Quindi, regolare il volume totale a 10 μL utilizzando acqua priva di nucleasi.
- Incubare la reazione a 65 °C per 5 minuti e porre in ghiaccio per 1 minuto.
- Preparare una miscela di sintesi di cDNA per ciascun campione aggiungendo 2 μL di tampone RT 10x; 4 μL di 25 mM di MgCl2; 2 μL di DTT 0,1 M; 1 μL di inibitore della RNasi (40 U·μL−1); e 1 μL del reagente di trascrizione inversa (200 U·μL−1) nell'ordine indicato.
- Aggiungere la miscela di sintesi del cDNA alla miscela RNA/primer. Mescolare delicatamente e centrifugare brevemente i campioni per raccogliere i componenti sul fondo della provetta.
- Preparare la miscela incubando i campioni per 10 minuti a 25 °C, seguiti da 50 minuti a 50 °C. Terminare le reazioni incubando a 85 °C per 5 minuti e raffreddare su ghiaccio.
- Aggiungere 1 μL di RNasi H a ciascuna provetta e incubare a 37 °C per 20 minuti per rimuovere l'RNA dall'ibrido DNA:RNA.
- Infine, diluire la reazione di sintesi del cDNA a un volume totale di 80 μL e conservarla a -80 °C fino a un ulteriore utilizzo.
NOTA: A questo punto il protocollo può essere messo in pausa.
8. Curva standard e (q)-PCR quantitativa per determinare i livelli di espressione dei geni marcatori che indicano diversi stadi di replicazione dei fagi
- Identificare un insieme di geni bersaglio che possono fungere da marcatori per ogni fase di replicazione del fago di interesse. Nel nostro caso, questi erano quelli dettagliati nella Tabella 2.
- Amplificare ciascuno dei geni bersaglio dal DNA genomico stampo utilizzando primer pertinenti e utilizzando la PCR con le seguenti condizioni di amplificazione: denaturazione iniziale a 95 °C per 2 min; denaturazione a 95 °C per 30 s; ricottura alla temperatura di ricottura ottimale a seconda dei primer (in questo caso è stata utilizzata una temperatura di 58 °C) per 30 s; prolunga a 72°C per 1 min; e prolungamento finale a 72 °C per 5 min.
- Purificare ogni amplicone utilizzando un kit di purificazione PCR e clonarli in un vettore di clonazione TA secondo le istruzioni del produttore. Verificare la sequenza di ciascun prodotto clonato mediante sequenziamento Sanger.
NOTA: A questo punto il protocollo può essere messo in pausa. - Calcolare il numero di copie per i singoli plasmidi utilizzando la seguente equazione20:
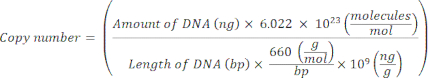
- Preparare un modello standard per ciascun gene marcatore diluendo in serie il DNA plasmidico da 109 copie/μL a 10 2 copie/μL in H2 O sterile senza nucleasi di grado molecolare.
- Eseguire la PCR quantitativa secondo le istruzioni del produttore per il sistema qPCR preferito con 1 μL di cDNA (dal punto 7.7) per ciascun campione in triplice copia, insieme ai rispettivi standard plasmidici in triplice; eseguire la PCR in una piastra a 96 pozzetti per ciascun target.
- Tracciare il numero di copie del DNA logaritmico (asse x) rispetto alla soglia del ciclo (asse y, Ct) e utilizzare una piattaforma appropriata come Excel o R per eseguire un calcolo di regressione lineare per visualizzare il coefficiente di determinazione (R2) e un'equazione lineare.
NOTA: Il coefficiente di determinazione deve essere superiore a 0,98. - Stimare il numero di copie per ogni bersaglio utilizzando l'equazione lineare (y = mx + b) derivata dalla regressione lineare (passo 8.7), dove y è il Ct stimato; x è il numero di copia del DNA logaritmico; m è la pendenza della retta, che definisce la variazione del Ct rispetto al numero di copie del DNA; e b è l'intercetta dell'asse y che rappresenta la Ct stimata per una copiadi DNA 32.
- Per ogni gene marcatore calcolare l'efficienza dell'amplificazione della PCR (E) utilizzando i parametri della regressione lineare della curva standard e la seguente equazione, dove m è la pendenza derivata dal passo 8.7 e dal passo 8.8:

- Convalidare tutti i primer in termini di efficienza percentuale utilizzando la seguente equazione:
NOTA: L'efficienza deve essere compresa tra il 90% e il 110%.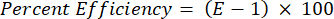
- Calcola il numero assoluto di copie del DNA usando la seguente formula:
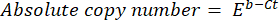
dove Ct (passo 8.8) è la soglia del ciclo, b è l'intercetta (passo 8.8), m è la pendenza (passo 8.8) ed E è l'efficienza dell'amplificazione della PCR (passo 8.9).
CRITICO: Quando si confronta l'amplificazione di due o più bersagli mediante q-PCR, l'efficienza della PCR deve essere calcolata per ciascun bersaglio al fine di confrontare il numero assoluto di copie del DNA. - In questo studio, i geni 16S rRNA, proC e rpoD sono stati utilizzati come controlli interni generali e gyrB è stato utilizzato come controllo di induzione33,34,35.
NOTA: Quando si scelgono i controlli interni dai dati seq dell'RNA, è meglio selezionare i controlli interni che non cambiano nei livelli di espressione per le condizioni testate. Un'attenta considerazione dei controlli appropriati è sempre importante per un'interpretazione significativa dei risultati.
Risultati
In questo lavoro, l'enumerazione temporale diretta della produzione di fagi da una coltura di lisogeno PAO1 LESΦ2 coltivata in condizioni non inducenti è stata utilizzata per determinare l'impatto dell'induzione spontanea di LESΦ2. La densità dei fagi era al suo punto più basso con una media di ~2,61 x 106 unità formanti placca (PFU)·mL−1 2 h dopo la subcoltura in terreno fresco durante la prima fase esponenziale di crescita, suggerendo che la lisogenia era lo stato dominante. Il titolo LESΦ2 è aumentato rapidamente fino a una media di ~2,4 x 108 PFU·mL−1 entro 4 ore e ha raggiunto la massima densità dopo 6 ore (media di ~5,83 x 109 PFU·mL−1; Figura 4).
Un'induzione spontanea minima è stata osservata durante la fase logaritmica iniziale della crescita del lisogeno (dopo 2 ore). Tuttavia, la presenza misurabile di fagi nel terreno di coltura era il risultato di molti eventi precedenti, tra cui i seguenti: l'impacchettamento degli acidi nucleici in teste proteiche, l'assemblaggio di proteine in particelle fagiche e l'espressione di geni fagici tardivi, geni fagici in stadio intermedio e geni fagici regolatori precoci. Era importante catturare le cellule infette prima dell'espressione degli eventi di replicazione associati ai fagi; Quindi, 90 min è stato scelto per far crescere la coltura prima dell'induzione. Per catturare il profilo di espressione genica della PAO1, i campioni di lisogeno LESΦ2 da una coltura sono stati raccolti prima e dopo l'induzione per un periodo di 90 minuti, come indicato nel punto 3.4. Questo lasso di tempo di 90 minuti è ben prima che alti livelli di induzione spontanea del profago residente vengano rilevati dal test della placca dal punto 2.3.2. Poiché la densità delle cellule batteriche era bassa durante la crescita esponenziale iniziale, i volumi di coltura sono stati scalati fino a 800 mL per garantire un ampio materiale per gli studi di espressione genica. I campioni sono stati raccolti dalla coltura non indotta e dalle colture indotte ogni 10 minuti e l'RNA è stato estratto per mappare il profilo di espressione dei marcatori chiave per la lisogenia e la replicazione litica durante la crescita batterica. L'RNA totale è stato purificato e convalidato per l'assenza di DNA genomico utilizzando saggi qPCR mirati al gene rRNA 16S (fase 6.1). I campioni che hanno raggiunto un RIN ≥ 9 hanno superato il controllo di qualità e sono stati convertiti in cDNA.
Il genoma LESΦ2 annotato è stato esaminato per identificare i geni che sono ben noti nei cicli di replicazione lisogenica e litica dei fagi temperati. Questi geni identificati sono stati poi utilizzati per convalidare la qRT-PCR per il profilo di espressione dei geni a ciclo ristretto del lisogeno e dei geni associati al ciclo litico da colture indotte e non indotte. Abbiamo quantificato il numero assoluto di copie del DNA e condotto un test di Wilcoxon signed-rank utilizzando R36 per confrontare i livelli di espressione in colture non indotte e indotte (Figura 5). È stato osservato un marcato aumento dell'espressione del gene cro (un marcatore precoce di replicazione litica) da ~ 2,31 x 109 copie in colture non indotte a ~ 3,02 x 1011 copie 30 minuti dopo l'induzione (test di Wilcoxon signed-rank: p < 0,01). Allo stesso modo, le proteine O e le proteine P, che sono marcatori di fase intermedia della replicazione litica (e si prevede che siano coinvolte nella replicazione del genoma del fago), hanno anche mostrato una significativa sovraregolazione da ~ 1,74 x 108 a ~ 1,25 x 10 10 copie (test di Wilcoxon signed-rank: p < 0,01) e da ~ 6,05 x 102 a ~ 5,68 x10 5 copie (test di Wilcoxon signed-rank: p < 0,01), rispettivamente. Infine, i geni strutturali associati alla coda sono stati utilizzati come marcatori tardivi del ciclo di replicazione litica. Ancora una volta, abbiamo osservato un aumento significativo dell'espressione da ~ 2,31 x 106 copie in colture non indotte a ~ 4,38 x 108 copie 30 minuti dopo l'induzione (test di Wilcoxon signed-rank: p < 0,01). Pertanto, i dati quantitativi della RT-PCR hanno confermato che l'espressione genica di geni marcatori ben consolidati per la replicazione litica ha seguito la tendenza prevista, con i marcatori precoci, medi e tardivi che hanno mostrato un'espressione differenziale multipla nell'ordine previsto (Figura 5). Poiché l'espressione dei marcatori per la replicazione litica è stata sovraregolata 30 minuti dopo il recupero, questo è considerato un punto temporale rappresentativo appropriato per lo studio del panorama trascrittomico dei fagi temperati attivi e dei loro ospiti batterici durante il ciclo litico.
Abbiamo osservato una certa espressione di geni litici in condizioni non indotte, confermando che si verifica sempre una certa induzione spontanea, anche in colture ottimizzate in cui i numeri di lisogeni sono rappresentati con il più alto rapporto tra CFU e PFU rilasciata nella fase logaritmica iniziale. Ciò significa che ci sarà sempre un certo livello di "rumore" nei dati di trascrittomica, il che rafforza l'importanza di controlli accuratamente preparati, comprese le colture indotte e non indotte. La scelta appropriata dei geni di controllo interno per determinare i cambiamenti di espressione si basa sull'attento esame dei dati trascrittomici per identificare i geni che sono espressi allo stesso livello sia nei campioni non indotti che in quelli indotti. I nostri risultati preliminari suggeriscono che rpoD era il gene di controllo più affidabile testato e aveva l'espressione più stabile (~1,71 x 10 5 copie prima dell'induzione e ~3,33 x 105 copie 30 minuti dopo l'induzione; Test di Wilcoxon con segno: p = 0,3594) rispetto ai geni 16S rRNA o proC (Figura 5). La variabilità dell'espressione dei controlli interni ha portato alla misurazione del numero assoluto di trascrizioni. L'esame futuro dei dati trascrittomici supporterà la scelta di appropriati controlli interni per un'ulteriore convalida.
Il gene cI è stato utilizzato nel nostro esercizio di profilazione genica, in quanto è un marcatore ben noto di lisogenia. Rispetto ai marcatori per la replicazione litica, l'espressione del gene cI era relativamente stabile (Figura 5), ma il numero di copie di questo gene era rassicurante nelle colture non indotte rispetto a quelle dei marcatori per la replicazione litica. Questi dati sono in accordo con i bassi numeri di PFU negli stessi campioni, confermando così che un'elevata espressione del repressore era associata a livelli più bassi di produzione di fagi. I dati qui riportati dimostrano che l'espressione del trascritto cI per questo particolare fago non è significativamente sottoregolata dopo l'induzione, come si vede nei fagi Stx11,17. L'attività del repressore è normalmente controllata sia a livello trascrizionale che post-traduzionale, quindi il gene repressore può essere trascritto, ma la proteina risultante è immediatamente soggetta ad autoclivaggio. Sono necessarie ulteriori sperimentazioni per convalidare i controlli trascrizionali e post-traduzionali. Inoltre, dalla nostra curva standard, il limite minimo di rilevamento della qPCR sembra essere ~102 copie.
Insieme, i nostri risultati dei saggi di placca e qRT-PCR convalidano la nostra strategia per la coltura e la preparazione del campione di RNA per generare un input ben controllato per gli esperimenti di RNA-Seq. Le colture non indotte nella fase esponenziale precoce hanno mostrato bassi livelli di induzione spontanea e di espressione genica litica, suggerendo la dominanza della lisogenia. Al contrario, le colture isolate 30 minuti dopo l'induzione hanno mostrato aumenti significativi nell'espressione dei geni marcatori che indicano la dominanza della replicazione litica.
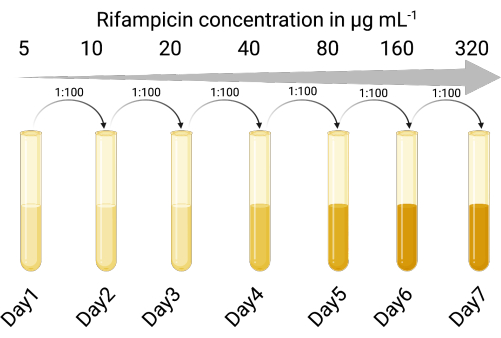
Figura 1: Il protocollo per la creazione dell'host indicatore resistente alla rifampicina (creato con BioRender.com). Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Il disegno sperimentale per l'enumerazione della PFU e della CFU di un lisogeno dello stesso campione. (Creato con BioRender.com) Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: Il disegno sperimentale per il campionamento di colture indotte e non indotte per l'isolamento dell'RNA. (Creato con BioRender.com) Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 4: Enumerazione temporale dell'induzione spontanea. Enumerazione temporale della produzione spontanea di profago LES utilizzando la PFU del lisogeno PAO1 Φ2 con la CFU concomitante, n = 8 (due repliche biologiche e quattro repliche tecniche); Le barre di errore rappresentano la deviazione standard. I punti rosso scuro indicano CFU·mL−1 in LB; i punti blu scuro indicano PFU·mL−1 in LB. Il rilascio spontaneo del fago infettivo φ2 da parte dei lisogeni è al livello più basso misurabile a 2 ore dopo l'inoculazione. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 5: Numero assoluto di copie dei geni marcatori bersaglio. Il numero assoluto di copie dei geni marcatori fagici conferma i modelli di espressione previsti, derivati utilizzando la RT-qPCR, di geni che dovrebbero svolgere ruoli importanti nella lisogenia e nei cicli litici. I punti rappresentano sia tre repliche biologiche che tre repliche tecniche (n = 9). (A) Il riquadro rosso rappresenta il marcatore di lisogenia, cI; (B) il verde rappresenta il marcatore litico precoce, cro; (C,D) il blu rappresenta i marcatori litici medi, i geni di replicazione del DNA; (E) il magenta rappresenta il marcatore litico tardivo, i geni strutturali della coda; (F-H) il grigio rappresenta i marcatori dell'ospite che sono stati utilizzati come controlli interni e (I) il bianco rappresenta la DNA girasi B, che è stata utilizzata come controllo di induzione. Le linee orizzontali continue mostrano la mediana della distribuzione. Fare clic qui per visualizzare una versione più grande di questa figura.
Tabella 1: Primer progettati in questo studio. Vengono fornite le sequenze di primer specifici per i geni marcatori e i controlli interni utilizzati in questo studio, insieme ai corrispondenti ID di accesso all'NCBI. Clicca qui per scaricare questa tabella.
Tabella 2: Efficienza dei primer utilizzati in questo studio calcolata utilizzando la curva standard qPCR. Clicca qui per scaricare questa tabella.
Discussione
La creazione di un ospite indicatore selezionabile, precedentemente utilizzato nei saggi di placca per quantificare in modo più accurato l'induzione spontanea del fago Stx da E. coli MC1061 37,38,39, è stata descritta qui per il fago LESΦ2 di P. aeruginosa. Questo intervento ha l'ulteriore vantaggio di ridurre le fasi e i tempi di elaborazione del campione, consentendo così la valutazione simultanea dei tassi di induzione spontanea in più condizioni di coltura. C'è il rischio di generare altre mutazioni durante la creazione di varianti resistenti alla rifampicina40; Tuttavia, in questo lavoro, il ceppo evoluto è stato utilizzato solo come ospite indicatore per l'enumerazione di placche da colture di interesse e non è stato incluso nell'analisi trascrittomica. Finché il ceppo indicatore selezionabile rimane ugualmente suscettibile all'infezione da parte del fago di interesse, non c'è preoccupazione per altre mutazioni acquisite. Ciononostante, non sono state rilevate differenze nei profili di polimorfismo della lunghezza del frammento di restrizione mediante l'analisi dell'elettroforesi su gel a campo pulsato (PFGE) di PAO1WT e PAO1RIF (dati non mostrati).
Quando si scelgono le cellule ospiti, è raro trovare un ceppo indicatore che non ospiti già profagi. Ad esempio, PAO1 ospita il profago filamentoso Pf4. I controlli sperimentali per questo studio sono stati progettati per essere in grado di esaminare direttamente l'espressione genica di fagi specifici (in questo caso, il profago LES 2) e gli effetti che questo fago ha sull'espressione genica batterica. Nel confronto dei trascritti di PAO1 portatori del profago LES 2 e mancanti del profago LES 2 (sia il lisogeno che il non lisogeno portano il Pf4 endogeno), che fungono da controlli interni per escludere l'impatto di Pf4 sull'ospite. Inoltre, è stato dimostrato che Pf4 di solito non causa lisi nella sua cellula ospite41 e non è, quindi, in grado di confondere i risultati di questi esperimenti.
È assodato che un attento controllo di qualità è fondamentale nella preparazione dei campioni per produrre dati omici significativi42. Tuttavia, come descritto in precedenza11, l'attenta caratterizzazione dell'attività dei profagi nella preparazione di colture di lisogeni per tali studi viene eseguita raramente. Qui, descriviamo in dettaglio i nostri protocolli sistematici per la preparazione di un set di colture ben controllato e ottimizzato per gli studi trascrittomici per esplorare meglio le interazioni tra batteri e fagi temperati. La sincronicità della popolazione è stata controllata sottoponendo la coltura ad almeno quattro raddoppiamenti prima di trattarla con l'antibiotico induttore norfloxacina. Determinando la MIC di norfloxacina per il ceppo nello studio, abbiamo potuto garantire che la concentrazione dell'agente induttore fosse appena al di sopra della MIC per il trattamento di "induzione". Le cellule trattate sono state quindi diluite 1:10 per abbassare la concentrazione di norfloxacina al di sotto della MIC dopo il trattamento di 1 ora al fine di consentire alle cellule di recuperare e completare il processo di replicazione dei fagi, terminando con la lisi della cellula e il rilascio della progenie fagica infettiva. Le cellule entrano nel ciclo di replicazione litica dopo lo stimolo di induzione solo una volta che la concentrazione di norfloxacina è stata portata al di sotto della MIC durante il periodo di recupero. In questo caso, superare 1 μg·mL−1 di norfloxacina significa che il farmaco non può essere efficacemente diluito al di sotto del MIC, poiché la MIC per la norfloxacina per PAO1 è di 0,19 μg·mL−1. Il livello di diluizione dell'induttore deve essere bilanciato con la necessità di recupero del lisogeno e il mantenimento della densità di coltura per la raccolta dell'RNA. I dati qui discussi dimostrano che è possibile sincronizzare le colture per creare campioni in cui domina la lisogenia, riducendo così il rumore dell'induzione spontanea e consentendo il rilevamento di veri cambiamenti guidati dalla lisogenia nell'espressione genica. Poiché lo stato lisogenico è predominante nella fase esponenziale iniziale della crescita, quando la densità cellulare batterica è bassa, suggeriamo di aumentare le colture per raccogliere abbastanza RNA per i successivi studi di espressione genica come RNA-Seq.
L'uso della norfloxacina come agente induttore per forzare le colture nel ciclo litico è ben segnalato43,44; Tuttavia, questo influenzerà anche l'espressione di altri geni batterici nel processo45,46. Per mitigare questo problema, le librerie di RNA provenienti da colture wild-type di controllo coltivate nelle stesse condizioni di induzione e non induzione dovrebbero essere incluse negli esperimenti di RNA-Seq. Anche l'uso di controlli interni e geni marcatori chiave per convalidare le fasi della replicazione dei fagi mediante qRT-PCR è fondamentale per confronti accurati. La profilazione quantitativa della RT-PCR non può essere interpretata confrontando il numero assoluto di trascritti per ciascun gene in vari punti temporali; È la forma del profilo che conta. In primo luogo, è stata campionata solo una piccola regione nel trascritto per qualsiasi gene, quindi nonsi sa se si tratti di un elemento di breve o più lunga durata. Certamente, la mappatura RNA-Seq dei trascritti mostra che la densità dei dati di mappatura varia significativamente sulla lunghezza di un gene. In secondo luogo, è la forma del profilo di espressione genica che dovrebbe essere interpretata per un gene marcatore associato al ciclo litico o allo stile di vita lisogenico o addirittura disaccoppiato dai circuiti regolatori fagici11. L'induzione spontanea è un problema reale nella coltura del lisogeno e si tradurrà sempre nell'espressione di geni associati al ciclo litico. Tuttavia, la profilazione mostra che i geni associati al ciclo di replicazione litica sono soppressi nella loro pre-induzione di espressione (almeno due pieghe logaritmiche) e post-induzione up-regolata.
Le analisi trascrittomiche condotte in precedenza sulle interazioni dei fagi Stx con E. coli supportano una comprensione approfondita dei geni fagici coinvolti nel mantenimento della lisogenia e nell'innesco del ciclo litico11,17. Attualmente, i fagi LES di P. aeruginosa sono stati annotati, ma le loro funzioni geniche chiave sono meno ben comprese. Gli studi trascrittomici consentiranno la ri-annotazione dei profagi LES e miglioreranno la nostra comprensione dei geni coinvolti nella lisogenia e nel ciclo litico. Collegare la sequenza genica alla funzione rappresenta una sfida importante nello studio di nuovi profagi, il che evidenzia ulteriormente la necessità di ulteriori studi per confermare le funzioni del gene fago per la produzione di migliori strumenti di annotazione47. L'applicazione e l'adattamento più ampi dei protocolli e delle misure di controllo della qualità aggiuntive descritte in questo articolo video potrebbero aiutare a svelare varie funzioni dei profagi e, quindi, a migliorare le pipeline di annotazione e a trasformare la nostra comprensione della biologia dei fagi e dei batteri.
Materiali
| Name | Company | Catalog Number | Comments |
| PAO1 | 6 | ||
| LESB58 | 6 | ||
| LES phages | Induced and purified from LESB58 using Norfloxacin. | This study | |
| Lysogeny Broth (LB) | Merck | 1.10285.500 | |
| LB Agar | Merck | 1.10283.500 | |
| Agar Agar | Fisher | A/1080/53 | |
| Top Agar | 0.4 g Agar Agar+2.5 g LB Broth in 100 mL water; autoclave and use. | - | |
| Rifampicin | Sigma (Stock: 50 mg/mL in Methanol- Mix well and use 0.22µm filter to sterilize and store it in -20°C until use) | R3501 | |
| Glacial Acetic Acid | Fisher 1% (v/v) in water | 10060000 | |
| Norfloxacin | Sigma (Stock: 25 mg/mL of 1% Glacial Acetic Acid-Mix well and use 0.22µm filter to sterilize and store it in -20°C until use;To avoid freeze thaw cycles, store as small aliquotes) | N9890 | |
| Phenol saturated with citrate buffer pH 4.3 | Sigma | P-4682 | |
| Molecular Biology grade Ethanol | Fisher | 16695992 | |
| TRIzol | Invitrogen | 12044977 | |
| Chloroform | Fisher | 11398187 | |
| Isopropanol | Fisher | 17150576 | |
| Nuclease-free H2O | Invitrogen | 10526945 | |
| 10X TURBO DNase | Ambion | AM1907 | |
| Qubit RNA HS, BR Kit | Invitrogen | Q10210 | |
| Agilent RNA 6000 Nano Kit | Agilent | 5067-1511 | |
| SuperScriptIII first strand synthesis kit | Invitrogen | 18080051 | |
| PCR Reagents | Bioline Mytaq Red 2X | BIO-25043 | |
| qPCR Reagents | Sensifast SYBR Hi Rox | BIO-92020 | |
| PCR purification kit | Isolate II PCR and Gel Kit | BIO-52060 | |
| TA cloning kit | TA Cloning Kit, with pCR 2.1 Vector, without competent cells | K202040 | |
| StepOne Real Time PCR system | Thermo Fisher Scientific | 4376600 |
Riferimenti
- Lin, D. M., Koskella, B., Lin, H. C. Phage therapy: An alternative to antibiotics in the age of multi-drug resistance. World Journal of Gastrointestinal Pharmacology and Therapeutics. 8 (3), 162-173 (2017).
- Jiang, W., Marraffini, L. A. CRISPR-Cas: New tools for genetic manipulations from bacterial immunity systems. Annual Review of Microbiology. 69 (1), 209-228 (2015).
- Santos, S. B., Azeredo, J. Bacteriophage-based biotechnological applications. Viruses. 11 (8), 737 (2019).
- Rodríguez-Rubio, L., Jofre, J., Muniesa, M. Is genetic mobilization considered when using bacteriophages in antimicrobial therapy. Antibiotics. 6 (4), 32 (2017).
- Hatfull, G. F. Dark Matter of the biosphere: The amazing world of bacteriophage diversity. Journal of Virology. 89 (16), 8107-8110 (2015).
- Yukgehnaish, K., et al. PhageLeads: Rapid assessment of phage therapeutic suitability using an ensemble Machine Learning approach. Viruses. 14 (2), 342 (2022).
- Seemann, T. Prokka: Rapid prokaryotic genome annotation. Bioinformatics. 30 (14), 2068-2069 (2014).
- Arndt, D., et al. PHASTER: A better, faster version of the PHAST phage search tool. Nucleic Acids Research. 44, W16-W21 (2016).
- Banerjee, S., et al. FINDER: An automated software package to annotate eukaryotic genes from RNA-Seq data and associated protein sequences. BMC Bioinformatics. 22 (1), 205 (2021).
- Jumper, J., et al. Highly accurate protein structure prediction with AlphaFold. Nature. 596 (7873), 583-589 (2021).
- Veses-Garcia, M., et al. Transcriptomic analysis of Shiga-toxigenic bacteriophage carriage reveals a profound regulatory effect on acid resistance in Escherichia coli. Applied and Environmental Microbiology. 81 (23), 8118-8125 (2015).
- Owen, S. V., et al. A window into lysogeny: revealing temperate phage biology with transcriptomics. Microbial Genomics. 6 (2), e000330 (2020).
- Davies, E. V., Winstanley, C., Fothergill, J. L., James, C. E. The role of temperate bacteriophages in bacterial infection. FEMS Microbiology Letters. 363 (5), 015 (2016).
- Livny, J., Friedman, D. I. Characterizing spontaneous induction of Stx encoding phages using a selectable reporter system. Molecular Microbiology. 51 (6), 1691-1704 (2004).
- Fogg, P. C. M., et al. Identification of multiple integration sites for Stx-phage Φ24B in the Escherichia coli genome, description of a novel integrase and evidence for a functional anti-repressor. Microbiology. 153 (12), 4098-4110 (2007).
- James, C. E., et al. Differential infection properties of three inducible prophages from an epidemic strain of Pseudomonas aeruginosa. BMC Microbiology. 12, 216 (2012).
- Riley, L. M., et al. Identification of genes expressed in cultures of E. coli lysogens carrying the Shiga toxin-encoding prophage Φ24B. BMC Microbiology. 12 (1), 42 (2012).
- Stover, C. K., et al. Complete genome sequence of Pseudomonas aeruginosa PAO1, an opportunistic pathogen. Nature. 406 (6799), 959-964 (2000).
- Winstanley, C., et al. Newly introduced genomic prophage islands are critical determinants of in vivo competitiveness in the Liverpool Epidemic Strain of Pseudomonas aeruginosa. Genome Research. 19 (1), 12-23 (2009).
- Davies, E. V., et al. Temperate phages enhance pathogen fitness in chronic lung infection. The ISME Journal. 10 (10), 2553-2555 (2016).
- Allison, H. E. Stx-phages: drivers and mediators of the evolution of STEC and STEC-like pathogens. Future Microbiology. 2 (2), 165-174 (2007).
- Allison, H. E., et al. Immunity profiles of wild-type and recombinant Shiga-like toxin-encoding bacteriophages and characterization of novel double lysogens. Infection and Immunity. 71 (6), 3409-3418 (2003).
- Mori, N., et al. A peptide based on homologous sequences of the β-barrel assembly machinery component BamD potentiates antibiotic susceptibility of Pseudomonas aeruginosa. Journal of Antimicrobial Chemotherapy. 67 (9), 2173-2181 (2012).
- Chojnacki, M., et al. A novel, broad-spectrum antimicrobial combination for the treatment of Pseudomonas aeruginosa corneal infections. Antimicrobial Agents and Chemotherapy. 63 (10), e00777 (2019).
- Miles, A. A., Misra, S. S., Irwin, J. O. The estimation of the bactericidal power of the blood. Epidemiology & Infection. 38 (6), 732-749 (1938).
- Srikumar, S., et al. RNA-seq brings new insights to the intra-macrophage transcriptome of Salmonella Typhimurium. PLoS Pathogens. 11 (11), e1005262 (2015).
- Kröger, C., et al. The transcriptional landscape and small RNAs of Salmonella enterica serovar Typhimurium. Proceedings of the National Academy of Sciences of the United States of America. 109 (20), E1277-E1286 (2012).
- Green, M. R., Sambrook, J. How to win the battle with RNase. Cold Spring Harbor Protocols. (2), (2019).
- A practical guide to analyzing nucleic acid concentration and purity with microvolume spectrophotometers. New England BioLabs Inc Available from: https://www.neb.com/-/media/nebus/files/application-notes/technote_mvs_analysis_of_nucleic_acid_concentration_and_purity.pdf?rev=c24cea043416420d84fb6bf7b554dbbb (2019)
- Saunders, N. A., Lee, M. A. . Real-Time PCR: Advanced Technologies and Applications. , (2013).
- Bustin, S. A. . A-Z of Quantitative PCR. , (2004).
- Ruijter, J. M., et al. Efficiency correction is required for accurate quantitative PCR analysis and reporting. Clinical Chemistry. 67 (6), 829-842 (2021).
- Fothergill, J. L., Neill, D. R., Loman, N., Winstanley, C., Kadioglu, A. Pseudomonas aeruginosa adaptation in the nasopharyngeal reservoir leads to migration and persistence in the lungs. Nature Communications. 5 (1), 4780 (2014).
- Huang, J., et al. Temperature-dependent expression of phzM and its regulatory genes lasI and ptsP in rhizosphere isolate Pseudomonas sp. strain M18. Applied and Environmental Microbiology. 75 (20), 6568-6580 (2009).
- Savli, H., et al. Expression stability of six housekeeping genes: a proposal for resistance gene quantification studies of Pseudomonas aeruginosa by real-time quantitative RT-PCR. Journal of Medical Microbiology. 52 (5), 403-408 (2003).
- . rstatix: Pipe-Friendly Framework for Basic Statistical Tests Available from: https://CRAN.R-project.org/package=rstatix (2022)
- McDonald, J. E., et al. High-throughput method for rapid induction of prophages from lysogens and its application in the study of Shiga toxin-encoding Escherichia coli strains. Applied and Environmental Microbiology. 76 (7), 2360-2365 (2010).
- Smith, D. L., et al. Short-tailed Stx phages exploit the conserved YaeT protein to disseminate Shiga toxin genes among Enterobacteria. Journal of Bacteriology. 189 (20), 7223-7233 (2007).
- James, C. E., et al. Lytic and lysogenic infection of diverse Escherichia coli and Shigella strains with a verocytotoxigenic bacteriophage. Applied and Environmental Microbiology. 67 (9), 4335-4337 (2001).
- Rees, V. E., et al. Characterization of hypermutator Pseudomonas aeruginosa isolates from patients with cystic fibrosis in Australia. Antimicrobial Agents and Chemotherapy. 63 (4), e02538 (2019).
- Li, Y., et al. Excisionase in Pf filamentous prophage controls lysis-lysogeny decision-making in Pseudomonas aeruginosa. Molecular Microbiology. 111 (2), 495-513 (2019).
- Van Kampen, A. H. C., Moerland, P. D. Taking bioinformatics to systems medicine. Systems Medicine. 1386, 17-41 (2016).
- Matsushiro, A., Sato, K., Miyamoto, H., Yamamura, T., Honda, T. Induction of prophages of enterohemorrhagic Escherichia coli O157:H7 with norfloxacin. Journal of Bacteriology. 181 (7), 2257-2260 (1999).
- James, C. E., et al. Lytic activity by temperate phages of Pseudomonas aeruginosa in long-term cystic fibrosis chronic lung infections. The ISME Journal. 9 (6), 1391-1398 (2015).
- Shaw, K. J., et al. Comparison of the changes in global gene expression of Escherichia coli induced by four bactericidal agents. Microbial Physiology. 5 (2), 105-122 (2003).
- Long, H., et al. Antibiotic treatment enhances the genome-wide mutation rate of target cells. Proceedings of the National Academy of Sciences of the United States of America. 113 (18), E2498-E2505 (2016).
- González-Tortuero, E., et al. VIGA: A sensitive, precise and automatic de novo VIral Genome Annotator. bioRxiv. , (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon