Method Article
Um Protocolo de Avaliação Funcional da Whole-Protein saturação mutagênese Bibliotecas Utilizando-High Throughput Sequencing
Neste Artigo
Resumo
Nós apresentamos um protocolo para a avaliação funcional de bibliotecas de um único site saturação mutagênese abrangentes de proteínas utilizando sequenciamento de alto rendimento. É importante ressaltar que essa abordagem usa pares de primers ortogonais multiplex construção da biblioteca e sequenciamento. Os resultados representativos utilizando TEM-1 β-lactamase seleccionado a uma dosagem clinicamente relevante de ampicilina são fornecidos.
Resumo
Site-directed mutagenesis has long been used as a method to interrogate protein structure, function and evolution. Recent advances in massively-parallel sequencing technology have opened up the possibility of assessing the functional or fitness effects of large numbers of mutations simultaneously. Here, we present a protocol for experimentally determining the effects of all possible single amino acid mutations in a protein of interest utilizing high-throughput sequencing technology, using the 263 amino acid antibiotic resistance enzyme TEM-1 β-lactamase as an example. In this approach, a whole-protein saturation mutagenesis library is constructed by site-directed mutagenic PCR, randomizing each position individually to all possible amino acids. The library is then transformed into bacteria, and selected for the ability to confer resistance to β-lactam antibiotics. The fitness effect of each mutation is then determined by deep sequencing of the library before and after selection. Importantly, this protocol introduces methods which maximize sequencing read depth and permit the simultaneous selection of the entire mutation library, by mixing adjacent positions into groups of length accommodated by high-throughput sequencing read length and utilizing orthogonal primers to barcode each group. Representative results using this protocol are provided by assessing the fitness effects of all single amino acid mutations in TEM-1 at a clinically relevant dosage of ampicillin. The method should be easily extendable to other proteins for which a high-throughput selection assay is in place.
Introdução
A mutagénese foi longa empregues no laboratório para estudar as propriedades de sistemas biológicos e a sua evolução, e para produzir proteínas mutantes ou organismos com funções melhoradas ou novos. Embora as primeiras abordagens invocado métodos que produzem mutações aleatórias em organismos, o advento da tecnologia de DNA recombinante permitiu aos investigadores a introduzir alterações seleccione a ADN de um modo específico do local, isto é., Mutagénese dirigida ao local 1,2. Com as técnicas actuais, tipicamente utilizando oligonucleótidos mutagénicos numa reacção em cadeia da polimerase (PCR), é relativamente fácil de criar e avaliar um pequeno número de mutações (por exemplo., Mutações pontuais) num dado gene 3,4. É muito mais difícil no entanto, quando as abordagens de meta, por exemplo, a mutações criação e avaliação de todos os possíveis single-site (ou de ordem superior).
Embora muito tenha sido aprendido com os primeiros estudos que tentam avaliar um grande número de mutations em genes, as técnicas utilizadas foram muitas vezes trabalhosa, por exemplo exigindo a avaliação de cada mutação de forma independente usando um absurdo supressor estirpes 5-7, ou foram limitados na sua capacidade quantitativa devido à baixa profundidade sequenciação de Sanger seqüenciamento 8. As técnicas utilizadas nestes estudos têm sido largamente suplantado por métodos que utilizam high-throughput tecnologia de sequenciação 9-12. Estas abordagens conceptualmente simples implicam a criação de uma biblioteca que compreende um grande número de mutações, sujeitando a biblioteca para uma tela ou selecção para a função, e depois profunda-sequenciação (isto é., Da ordem dos> 10 6 sequenciação lê) a biblioteca obtida antes e após selecção. Desta forma, os efeitos fenotípicos ou aptidão de um grande número de mutações, representada como a alteração na frequência de cada população mutante, pode ser avaliada simultaneamente e quantitativamente mais.
Nós já introduziu uma simp(., ou seja, de toda a proteína bibliotecas de mutagénese de saturação) le abordagem para avaliar bibliotecas de todas as mutações de um único aminoácido em proteínas possíveis, aplicáveis aos genes com um comprimento maior que o comprimento ler sequenciação 11,13: Em primeiro lugar, cada posição de aminoácido é randomizado pelo Site-Directed PCR mutagênico. Durante este processo, o gene é dividido em grupos compostos de posições contíguas com um comprimento total acomodados pela plataforma de sequenciação. Os produtos de PCR mutagénica para cada grupo são, então, combinados, e cada grupo submetido independentemente a selecção e sequenciação de alto rendimento. Ao manter uma correspondência entre a localização de mutações na sequência e do comprimento de sequenciação de leitura, esta abordagem tem a vantagem de maximizar a profundidade de sequenciação: enquanto um poderia simplesmente sequenciar tais bibliotecas em janelas curtas sem divisão em grupos (por exemplo, uma espingarda padrão. abordagem de sequenciação), mais leituras obtida seria do tipo selvagem e assim a maioria de rendimento desperdiçado sequenciação (por exemplo, para um conjunto de proteínas biblioteca de mutagénese de saturação de uma proteína de 500 amino ácido sequenciado em 100 aminoácidos (300 pb) janelas, no mínimo 80% de leituras vão ser a sequência de tipo selvagem).
Aqui, um protocolo que utiliza é apresentado sequenciação de alto rendimento para a avaliação funcional de bibliotecas de mutagénese de saturação total em proteínas, utilizando a abordagem acima (descrito na Figura 1). Importante, introduz-se o uso de iniciadores ortogonais no processo de clonagem da biblioteca de código de barras para cada grupo de sequência, o que lhes permite ser multiplexados em uma biblioteca, simultaneamente submetido a rastreio ou selecção, e depois de-multiplexados para a sequenciação de profundidade. Uma vez que os grupos de sequência não são submetidas a selecção de forma independente, o que reduz a carga de trabalho e assegura que cada mutação experimenta o mesmo nível de selecção. TEM-1 β-lactamase, uma enzima que confere resistência de alto nível aoantibióticos β-lactâmicos (por exemplo., ampicilina) em bactérias é usado como um sistema modelo 14-16. Um protocolo é descrito para a avaliação de uma biblioteca de mutagénese de saturação todo-proteína de TEM-1 em E. coli sob selecção com um nível de soro aproximada para uma dose clínica de ampicilina (50 ug / ml) 17,18.
Protocolo
Nota: Consulte a Figura 1 para descrição do protocolo. Vários passos e reagentes no protocolo exigem medidas de segurança (indicados com "CUIDADO"). Consultar fichas de segurança antes de usar. Todos os passos do protocolo são realizadas à TA, a menos que outra indicação.
1. Prepare Meios de Cultura e placas
- Preparar e esterilizar em autoclave de 1 L de água purificada, a 100 ml de Super Broth óptima (SOB; Tabela 1), 1 L de meio de Luria-Bertani (LB; Tabela 2) e 1 L de LB-agar (Tabela 3). Preparar separadamente e esterilizar três frascos de cultura contendo cada um 1 L de LB.
Nota: Durante todo o protocolo de "água" refere-se a autoclave esterilizado água purificada; SOB, LB-agar LB e referem-se às soluções esterilizadas por autoclave. - Preparar um estoque de 12 mg / ml de Tet por dissolução de 0,12 g de cloridrato de tetraciclina em 10 ml de etanol a 70%. Esterilizar utilizando um filtro de 0,2 um e armazenara 4 ° C protegida da luz.
- Fresco LB-agar a 50 ° C e em seguida adicionar 1 ml de estoque de Tet (concentração final de 12 ug / ml Tet). Despeje em placas de Petri e arrefecer à RT protegido da luz. Armazenar a 4 ° C protegida da luz.
2. Construção do Todo-gene saturação mutagênese Biblioteca
Nota: Os primers; PCRs concluída, digestões de restrição e ligaduras; e As amostras de ADN purificado pode ser armazenado a -20 ° C.
- Projetando primers de mutagênese
- Para mutagenizar cada posição de aminoácido a todos os aminoácidos possíveis, conceber um par de iniciadores de mutagénese complementares (com sentido / anti-sentido para a frente e / reverso) para cada posição de aminoácido com as seguintes directrizes:
- Substituir o codão correspondente ao aminoácido a ser mutagenizado por NNS (onde N é uma mistura de todos os quatro bases de nucleótidos e S é uma mistura de citosina (C) e a guanina (G)) e no centro do primer, flanqueada por aproximadamente 15 nucleótidos de cada lado.
- Assegure-se que a 5 'e 3' terminam em C ou G e que a temperatura de fusão (Tm) é de aproximadamente 70 ° C 3. Use o NNS_PrimerDesign.m roteiro computacional (Veja arquivo de código Suplementar) para projetar primers de mutagênese NNS de acordo com essas diretrizes.
- iniciadores da ordem de uma fonte comercial. Para facilidade de utilização, eles têm sintetizado no formato de placa de 96 cavidades e pré-diluído em água a 50 uM, com um conjunto de placas contendo os iniciadores de mutagénese de sentido e anti-sentido mais os iniciadores.
- Encha uma bacia pipeta com água e usar uma pipeta de canais múltiplos para transferir 95 ul a 263 ao longo de três poços de placas de 96 poços. Dilui-se a iniciadores de 20 vezes para 2,5 ^ M, utilizando uma pipeta de canais múltiplos para transferir 5 ul a partir das placas de 96-poços contendo os iniciadores de sentido mutagénese para os poços das placas cognatos contendo água.
- Repetirprotocolo passo 2.1.3 para diluir os primers antisense mutagénese.
- Para mutagenizar cada posição de aminoácido a todos os aminoácidos possíveis, conceber um par de iniciadores de mutagénese complementares (com sentido / anti-sentido para a frente e / reverso) para cada posição de aminoácido com as seguintes directrizes:
- Síntese de NNS sub-bibliotecas para cada posição de aminoácido por PCR de dois passos de mutagénese dirigida
- Execute as PCRs mutagénicos primeira rodada. Para cada iniciador de mutagénese, prepare uma reação PCR de 25 ul usando plasmídeo pBR322_AvrII como molde e iniciadores AatII_F ou AvrII_R (iniciadores sentido de mutagénese emparelhados com o iniciador AvrII_R, e iniciadores de mutagénese antisense emparelhados com o iniciador AatII_F; total de 526 PCRs). Veja a Tabela de Materiais para sequências AatII_F e AvrII_R.
- Prepara-se uma PCR "master mix" através da adição dos reagentes da Tabela 4 para um tubo cónico de 15 ml. Transfira para uma bacia pipeta. Usar uma pipeta de canais múltiplos para transferir 15 ul a 263 ao longo de três poços de placas de 96 poços de PCR. Usar uma pipeta de canais múltiplos para transferir 10 ul a partir das placas de 96-poços contendo os iniciadores de mutagénese sentido diluído aos poços cognatos in as placas de PCR.
- Cobrir cada placa de PCR com um vedante de placa de 96 poços. Centrifugar a 200 xg durante 2 min.
- Transferir as placas de PCR para termociclador e executar o seguinte programa: 98 ° C durante 30 seg; 20 ciclos: 98 ° C durante 10 seg, 55 ° C durante 20 seg, 72 ° C durante 1 min; 72 ° C durante 2 min; manter a 4 ° C.
- Repita os passos protocolo 2.2.1.1 - 2.2.1.3 para as placas de 96-poços contendo os iniciadores de mutagénese antisense diluído.
- Execute as PCRs mutagénicos de segunda ordem. Para cada posição de aminoácido, preparar um 25 ul de reacção de PCR utilizando iniciadores e AatII_F AvrII_R, e a mistura e diluiu-se da primeira volta de PCR produtos mutagénicos como um molde (total de 263 PCRs).
- Encha uma bacia pipeta com água e usar uma pipeta de canais múltiplos para transferir 198 ul a 263 ao longo de três poços de placas de 96 poços.
- Combinar e diluir 100 vezes os produtos PCR mutagénica para cada posição de aminoácidos utilizando uma pipeta de canais múltiplos paraprimeiro 1 ml de transferência as placas de 96 poços de PCR contendo os produtos de PCR resultantes a partir dos iniciadores de sentido mutagénese para os poços das placas cognatos contendo água. Em seguida, repetir a transferência para os produtos de PCR resultantes a partir dos iniciadores anti-sentido de mutagénese.
- Prepara-se uma PCR "master mix" através da adição dos reagentes do Quadro 5 para um tubo cónico de 15 ml. Transfira para uma bacia pipeta. Usar uma pipeta de canais múltiplos para transferir 24 ul a 263 ao longo de três poços de placas de 96 poços de PCR. Usar uma pipeta de canais múltiplos para transferir 1 ml a partir das placas de 96 poços contendo a mistura e diluiu-se em primeiro lugar rodada produtos PCR mutagénica para os poços nas placas cognatos de PCR.
- Cobrir cada placa de PCR com um vedante de placa de 96 poços. Centrifuga-se a aproximadamente 200 xg durante 2 min. placas de transferência para termociclador e executar o mesmo programa como na etapa protocolo 2.2.1.3.
- Analisar os resultados dos PCRs mutagénicos de segunda ordem por eletroforese em gel.Certifique-se de que todos os produtos são do tamanho correto e ausência de contaminação dos produtos.
- Adicionar 2 ml de gel de corante de carga 2x para uma bacia de pipeta e, em seguida, utilizar uma pipeta de canais múltiplos para transferir 6 ul a 263 ao longo de três poços de placas de 96 poços. Usar uma pipeta de canais múltiplos para transferir 6 ul a partir das placas de 96 poços de PCR contendo os produtos de segunda ordem PCR mutagénica para os poços cognatos das placas de 96 poços contendo corante.
- Preparar um gel de agarose a 1,5% com 0,2 ug / ml de brometo de etídio (CUIDADO).
- escada de DNA de carga no primeiro e último pistas de cada linha. Em seguida, use uma pipeta multicanal para carregar 10 ul de amostras do passo protocolo 2.2.3.1.
- Submeter o gel a 100 V durante 40 min e a imagem num transiluminador UV.
- Repita os passos protocolo 2.2.3.2 - 2.2.3.4 até que todas as amostras são analisadas.
- Medir com precisão a concentração de cada produto de PCR NNS sub-biblioteca utilizando um reagente de quantificação ADNcd.
- Transferircerca de 15 ml de tampão de EB para uma bacia de pipeta. Usar uma pipeta de canais múltiplos para transferir 49 ul a 263 ao longo de três poços de 96 poços, placas de ensaio de fundo transparente com paredes negras-.
- Usar uma pipeta de canais múltiplos para transferir 1 uL de cada produto de PCR de segundo passo (passo protocolo 2.2.2) aos poços cognatos de as placas de ensaio de 96 poços.
- Prepara-se uma curva padrão de concentração de DNA por diluição de DNA do fago lambda a 2 ng / ul em 300 ul de tampão de EB e, em seguida, fazer dez diluições de duas vezes (para o total de 11 concentrações). Transferir 50 uL aos primeiros onze colunas de uma linha de uma das placas de ensaio de 96 poços a partir do passo anterior, que não contém amostra; para a coluna XII adicionar 50 ul de tampão de EB (branco de reagente).
- Prepare o reagente quantificação dsDNA adicionando 75 ul de reagente (ver Tabela de Materiais) para um tubo de 15 ml, em seguida, adicione 15 ml de tampão EB. Misture invertendo tubo e, em seguida, transferir para uma bacia pipeta. Proteger reagente da luz.
- Usar uma pipeta de canais múltiplos para transferir 50 ul de reagente preparada a quantificação dsDNA a cada poço das placas de ensaio. Misturar por pipetagem cima e para baixo. Incubar as placas à temperatura ambiente durante 5 minutos ao abrigo da luz.
- Medir a fluorescência de cada amostra utilizando um leitor de microplacas e comprimentos de onda de excitação de fluoresceína padrão (485 nm, emissão a 520 nm; 0,1 seg).
- Subtrair o valor da fluorescência do branco de reagente a partir de todas as amostras. Gerar uma curva padrão a partir das medições de fluorescência de amostras de fago lambda. Calcula-se a concentração de cada amostra utilizando as respectivas medições de fluorescência e a curva padrão.
- Execute as PCRs mutagénicos primeira rodada. Para cada iniciador de mutagénese, prepare uma reação PCR de 25 ul usando plasmídeo pBR322_AvrII como molde e iniciadores AatII_F ou AvrII_R (iniciadores sentido de mutagénese emparelhados com o iniciador AvrII_R, e iniciadores de mutagénese antisense emparelhados com o iniciador AatII_F; total de 526 PCRs). Veja a Tabela de Materiais para sequências AatII_F e AvrII_R.
- Clonagem de NNS sub-bibliotecas em vetores de seleção
- Misture 100 ng de cada produto PCR NNS sub-biblioteca em cinco grupos sub-biblioteca NNS. Seguindo as instruções dos fabricantes, limpar amostras utilizando um kit de purificação de DNA e, em seguida, medir a concentração usando um dsreagente de quantificação de ADN.
Nota: Cada grupo é composto de cerca de 53 posições de aminoácidos contíguos espaçadas ao longo da sequência MET-1 (grupos NNS sub-biblioteca 1-5 são compostos de posições 26-78, 79-132, 133-183, 184-236, e 237-290, respectivamente; numeração de acordo com Ambler et al, 19).. - Criar vectores de clonagem para cada grupo sub-biblioteca NNS.
- Preparar cinco PCRs 100 ul de acordo com a Tabela 6, utilizando os iniciadores e AvrII_F AatII_OP1_R - AatII_OP5_R, e plasmídeos pBR322_OP1-5 como molde (AatII_OP1_R emparelhado com pBR322_OP1, etc).
- Transferência para o termociclador e executar o seguinte programa: 98 ° C durante 30 seg; 25 ciclos: 98 ° C durante 10 seg, 55 ° C durante 20 seg, 72 ° C durante 1,5 min; 72 ° C durante 2 min; manter a 4 ° C. Veja T capazes de Materiais para sequências de AatII_R e AvrII_F.
- Preparar um gel de agarose a 1% com 0,2 ug / ml de brometo de etídio (CUIDADO).
- Adicionar 20 ul de gel de corante 6x carregamento de cada amostra de PCR. Coloque a primeira faixa do gel com escada de ADN; carregar todo o volume de cada amostra, a ignorar pelo menos um poço entre as amostras.
- Executar gel a 100 V durante 50 min.
- Visualize gel usando um iluminador UV de comprimento de onda (CUIDADO). fatias especiais de consumo contendo o produto de PCR de ~ 3500 pb; transferir para tubos de microcentrífuga separados. Fatias de gel pode ser armazenada à temperatura de -20 ° C.
- Seguindo as instruções dos fabricantes, purificar amostras utilizando um kit de extracção de gel e concentração medida usando um reagente de quantificação ADNcd.
- Para ambos os grupos NNS sub-biblioteca (protocolo da etapa 2.3.1) e vetores de clonagem (protocolo da etapa 2.3.2), criado restrição digere com enzimas Aatll e AvrII de acordo com a T capazes 7. Incubar a 37 ° C durante 1 h. Seguindo as instruções dos fabricantes, limpar amostras utilizando um kit de purificação de DNA e, em seguida, medir a concentração usando um quantita dsDNAreagente ção.
- Defina-se reacções de ligação seguinte Tabela 8 para cada grupo de sub-biblioteca NNS digerido por restrição com cognato digerido por restrição vector de clonagem (NNS grupo sub-biblioteca com uma pBR322_OP1, etc). Incubar à temperatura ambiente durante 1 h. Limpar as reacções utilizando um kit de purificação de ADN de acordo com as instruções do fabricante; eluir o ADN com 20 ul de água.
- Transformar a totalidade das reacções de ligação purificados em E. biblioteca eficiente células de E. coli por electroporação.
- Thaw E. electrocompetentes coli e, em seguida células de lugar e reacções de ligação purificados no gelo.
- Transferem-se 10 ul de células descongeladas para cada reacção de ligação purificada e, em seguida, transferir a cuvete de electroporação. Electroporate a 1,8 kV.
- Recuperar células por ressuspensão em 1 ml SOB. Incubar durante 1 hora a 37 ° C.
- Ressuspender 10 mL de cada cultura em 990 ul de recuperação LB; espalhar 100 ul em placas de LB-agar containing 12 ug / ml de Tet. Incubar as placas de S / N (~ 16 horas) a 37 ° C.
- Para cada cultura recuperação, preparar uma garrafa de cultura de 250 ml com 50 ml de LB e 50 estoque Tet ul. Transferir ao balão restantes ~ 1 ml da cultura de recuperação. Incubar S / N (~ 16 horas) a 37 ° C com agitação vigorosa (~ 200 rpm).
- Contar o número de colónias em cada placa. Calcular o número de transformantes bem sucedidos quanto
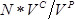 , Onde
, Onde  é o número de colónias,
é o número de colónias, 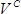 é o volume da cultura de recuperação (1000 uL) e
é o volume da cultura de recuperação (1000 uL) e 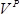 é o volume da cultura de recuperação plaqueadas (1 ul).
é o volume da cultura de recuperação plaqueadas (1 ul).
Nota: Para assegurar a cobertura completa de todas as mutações, como regra geral o número de transformantes bem sucedidos devem ser ≥100 vezes em relação ao número de mutações esperados. Cada sub-biblioteca NNS tem ~ 53 posições, de modo que o número esperado de mutações é de 53 posições × 32 códons / position ≈ 1,7 × 10 3; para se obter um tamanho de biblioteca ≥100 vezes (≥1.7 × 10 5) deve haver ≥170 colónias em cada placa. - De acordo com as instruções do fabricante, isolar o DNA de plasmídeo a partir de culturas, utilizando um kit de purificação de plasmídeo e, em seguida, medir as concentrações utilizando um reagente de quantificação ADNcd. Misture 100 ng de cada plasmídeo. Isso cria o conjunto de proteínas biblioteca final de saturação mutagênese.
- Misture 100 ng de cada produto PCR NNS sub-biblioteca em cinco grupos sub-biblioteca NNS. Seguindo as instruções dos fabricantes, limpar amostras utilizando um kit de purificação de DNA e, em seguida, medir a concentração usando um dsreagente de quantificação de ADN.
3. Seleção do TEM-1 Whole-proteína saturação mutagênese Biblioteca para a resistência aos antibióticos
- Preparação da cultura de pré-selecção.
- Dilui-se o plasmídeo a partir do passo 2.3.7 Protocolo para 0,5 ng / jil em água e transferir 20 ul a um tubo de microcentrífuga. Executar a transformação, recuperação, chapeamento e O crescimento / N, como descrito anteriormente no passo 2.3.5 protocolo, com excepção de transferência de 1 ul da cultura de recuperação de SOB a 999 ul LB.
- Contar o número de colónias. Para assegurar a cobertura completa de todas as mutações deve haver ≥100 colónias, indicando ≥10 6 transformantes bem sucedidos (100 × 263 × 32 posições de codões / posição Å 10 6).
- Medir a concentração da cultura de 50 ml de O / N.
- Prepara-se uma placa de LB por adição de 1 ml de LB para uma cuvete espectrofotómetro. Medida OD600 num espectrofotómetro.
- Dilui-se a / N cultura de 10 vezes por ressuspensão O 100 uL em 900 uL LB. Medir a OD 600. Subtrair a leitura OD600 da peça em bruto e multiplicar por 10 para dar o OD600 da cultura D / N.
- Pré-aquecer as três frascos de cultura a partir do passo 1.1 Protocolo para ~ 30 min a 37 ° C. Dilui-se a cultura D / N para DO600 = 0,1 e adicionar 1 ml para um frasco (OD600 final = 0,001). To seu é o "cultura pré-selecção".
- Incubar a "cultura pré-selecção", a 37 ° C com agitação vigorosa (200 rpm). monitorizar periodicamente crescimento medindo a OD600 como no passo protocolo 3.1.3 (não é necessário diluir a cultura 10 vezes) até DO600 = 0,1 (~ 2,5 h).
- Transferir 100 mL da cultura de pré-selecção a dois tubos cónicos de 50 ml. Centrifugar a 4.000 xg durante 6 minutos a 4 ° C. Remover a maior parte sobrenadante e combinam em um único tubo cônico de 15. Repita centrifugação e remover todo o sobrenadante. Armazenar a -20 ° C.
- A selecção para resistência à ampicilina.
- Embora a cultura de pré-selecção é a incubação, preparar um 50 mg / ml de Amp estoque em água por dissolução de 0,5 g de ampicilina de sódio em 10 ml de água. Esterilizar utilizando um filtro de 0,2 um e armazenar a 4 ° C.
- Para os outros dois frascos, Tet adicionar a uma concentração final de 12 ug / mL e um volume da pré-selecção de cultura, tais THA ta OD600 final = 0,001. Para um frasco, adicionar 1 ml de Amp, para uma concentração final de 50 ug / ml - esta é a "cultura selecção".
- Incubam-se as culturas a 37 ° C com agitação vigorosa (200 rpm). Monitorizar o crescimento da cultura para as quais não ampicilina foi adicionada no passo anterior, até DO600 = 0,1 (~ 2,5 h). Neste momento, também medir a DO600 da cultura selecção.
- Divida a OD600 da cultura seleção em 0,1 e multiplique por 100 ml. Transferir este volume (~ 400 ml) para tubos de 50 mL cónicos e centrifugar a 4000 x g durante 6 min a 4 ° C. Remover a maior parte sobrenadante e combinam em um único tubo cônico de 15. Repita centrifugação e remover todo o sobrenadante.
- De acordo com as instruções do fabricante, isolamento de DNA de pré-selecção (protocolo passo 3.1.6) e seleção (protocolo passo 3.2.4) pellets de células de cultura e, em seguida, medir as concentrações usando um reagente quantificação dsDNA.
- Preparação de amostras para a sequenciação de alto rendimento
- Prepare 25 PCRs ul para de-multiplex os grupos sub-biblioteca NNS com primers ortogonais
- Preparar um master mix de PCR de acordo com a Tabela 9; transferir 23 ul de dez tubos de PCR.
- Adicionar 1 ml de 0,5 ng de DNA plasmídeo / mL purificado a partir da cultura de pré-selecção de PCR tubos 1 - 5 e selecção a partir da cultura para tubos 6-10.
- Misture 50 ul de 50 mM a frente primers ortogonais OP1_F - OP5_F com a respectiva inversa OP1_R primers ortogonais - OP5_R. Na mesma ordem, a transferência de 1 ml para tubos de PCR 1 - 5 e 6 - 10. Ver Tabela de Materiais para sequências de OP1_F - OP5_F e OP1_R - OP5_R.
- Transfira para os tubos de PCR termociclador. Executar o seguinte programa: 98 ° C durante 30 seg; 20 ciclos: 98 ° C durante 10 seg, 55 °; C durante 20 seg, 72 ° C durante 1,5 min; 72 ° C durante 2 min; manter a 4 ° C.
- Preparar 25 ul PCR para isolar cada um dos grupos de sub-biblioteca NNS
- Diluir 100 vezes os PCRs dez a partir do passo 4.1.1.4 protocolo transferindo 1 ml de cada para tubos de PCR e separado adicionando 99 uL de água. Mix, em seguida, pipeta 99 ul e descarte.
- Misture 50 ul de 50 mM iniciadores forward Group1_F - Group5_F com o respectivo Group1_R primers reverso - Group5_R. Na mesma ordem, de transferência de 1 ml para tubos PCR 1 - 5 e 6 - 10. Ver Tabela de Materiais para sequências de Group1_F - Group5_F e Group1_R - Group5_R.
- Prepara-se uma mistura principal de PCR de acordo com o quadro 9, a transferência de 23 uL a cada tubo de PCR. Transfira para os tubos de PCR termociclador; executar o mesmo programa como na etapa protocolo 2.2.1.3.
- Realizar as finais 25 PCRs ul para adicionar sequências de indexação
- dilute 100 vezes os PCRs dez a partir do passo 4.1.2.3 protocolo transferindo 1 ml de cada para tubos de PCR e separado adicionando 99 uL de água. Mix, em seguida, pipeta 99 ul e descarte.
- Prepara-se uma mistura principal de PCR de acordo com o quadro 9, a transferência de 23 uL a cada tubo de PCR.
- Para tubos com template provenientes de NNS sub-biblioteca grupos 1-5, transferir 0,5 ul por tubo iniciadores directos 501_F - 505_F respectivamente. Para tubos com modelo resultante das culturas pré-selecção e selecção, transferir 0,5 mL por tubo primers 701_R e 702_R inversa, respectivamente. Veja a Tabela de Materiais para sequências de 501_F - 505_F e 701_R e 702_R.
- Transferir os tubos de PCR para o termociclador e executar o programa a partir do passo 2.2.1.3.
- Misture e purificar amostras
- medir as concentrações utilizando um reagente de quantificação dsDNA de acordo com as instruções do fabricante. Misturar 100 ng de cada produto de PCR intoa único tubo de microcentrífuga.
- Preparar um gel de agarose a 2% com 0,2 ug / ml de brometo de etídio (CUIDADO).
- Adicionar 6x corante de carga de gel para a amostra de produtos de PCR misturado. Coloque a primeira faixa do gel com escada de ADN; carregar todo o volume da amostra.
- Executar gel a 100 V durante 50 min. Visualize gel usando um iluminador UV de comprimento de onda (CUIDADO). fatia especial de consumo contendo o produto de PCR de ~ 360 pb; transferir para o tubo de microcentrífuga. fatia de gel pode ser armazenada à temperatura de -20 ° C.
- Seguindo as instruções dos fabricantes, purificar amostra usando um kit de extracção de gel e medir a concentração usando um reagente quantificação dsDNA. Esta é a amostra final para o sequenciamento de alto rendimento.
- Seqüência em uma plataforma de sequenciamento de alto rendimento (ver Tabela de Materiais para a plataforma utilizada neste protocolo).
- Calcular a concentração da amostra em nM como
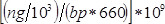 , Onde
, Onde 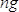 é a concentração da amostra em ng / mL e
é a concentração da amostra em ng / mL e  é o comprimento da sequência do ADN da amostra (~ 360 pb). Diluir a amostra com 4 nM em tampão de EB.
é o comprimento da sequência do ADN da amostra (~ 360 pb). Diluir a amostra com 4 nM em tampão de EB. - Siga as instruções do fabricante para desnaturar amostra e diluir a 9:00 em tampão de hibridação.
- Carga 600 uL de amostra num cartucho de reagente. Sequência seguindo as instruções do fabricante e as instruções na tela.
- Calcular a concentração da amostra em nM como
- Prepare 25 PCRs ul para de-multiplex os grupos sub-biblioteca NNS com primers ortogonais
- A análise dos dados de sequenciação
- DOWNLOAD FLASH (Ajuste Rápido comprimento de curto lê) 20, coloque na pasta junto com os arquivos fastq.gz obtidos a partir de sequenciação.
- Use flash para unir os arquivos fastq.gz correspondentes à-end emparelhado lê para cada par de índices (frente e verso lê para cada grupo sub-biblioteca NNS para as culturas pré-selecção e selecção).
- Abra o Prompt de Comando e altere o diretório para a pasta na etapa protocolo 4.2.1. Junte-se a cada par de leituras usando o comando: Flash , onde mates1.fastq.gz e mates2.fastq.gz são os arquivos que contenham a frente e lê inversa, respectivamente.
- Depois de entrar para cada par de leituras, coloque o arquivo de saída out.extendedFrags.fastq em pastas separadas para os resultados dos pré-selecção ou de seleção de culturas. Renomeie o arquivo de saída out.extendedFrags.fastq acordo com o grupo sub-biblioteca NNS que lhe corresponde (ie., 1.fastq, 2.fastq, etc.).
- Execute o NNS_DataAnalyzer.m roteiro computacional (Veja arquivo de código complementar) de cada pasta para calcular as contagens para cada mutação de um único aminoácido, e as contagens para o tipo selvagem, para cada grupo de sub-biblioteca NNS.
- Calcular o efeito de fitness
 de cada mutação
de cada mutação  em cada posição
em cada posição  como a base dez logaritmo da razão das contagens obtidas na selecção (
como a base dez logaritmo da razão das contagens obtidas na selecção (  ) Versus a pré-selecção (
) Versus a pré-selecção ( 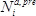 ) Condição, em relação ao tipo selvagem:
) Condição, em relação ao tipo selvagem:  .
.
Resultados
O mapa do plasmídeo para as cinco plasmídeos pBR322 contendo locais de iniciação modificados ortogonais (pBR322_OP1 - pBR322_OP5) é mostrado na Figura 2A. Para testar se os iniciadores são específicos ortogonais, os PCRs foram realizadas utilizando cada par de iniciadores de individualmente ortogonais, juntamente com todos os cinco plasmídeos pBR322_OP1-5, ou com todos os plasmídeos menos o plasmídeo combinando o par de iniciadores ortogonal. O produto correcto só foi obtida quando o plasmídeo correspondente foi incluído, e nenhum produto de qualquer tamanho foi obtido na sua ausência (Figura 2B).
Uma experiência representativa foi realizada seguindo o protocolo descrito no presente texto (Figura 1). Na sequência de processamento (seção de protocolo 4.2), 6,2 × 10 6 lê a partir da condição de pré-selecção e 6,3 × 10 6 lê a partir do 50 ug / ml conditi selecção ampicilinaforam obtidos em. As contagens obtidas para cada amino ácido mutação da condição pré-selecção exibir uma característica log-normal de distribuição (Figura 3A) 13. Pelo menos uma contagem a partir da sequenciação da cultura de pré-selecção por 98,9% de mutações (58 não tinham contagens) e maior do que 100 contagens para 91,2% (465 tinham menos de 100 contagens) foram obtidos. A Figura 3B ilustra o efeito da aptidão relativa () para cada mutação em cada posição de TEM-1; a distribuição dos é mostrado na Figura 3C. Sob selecção com 50 ug / ml de ampicilina, a maioria das mutações têm um efeito de fitness neutro ou quase neutro (≈ 0, correspondendo a pixels brancos, na Figura 3B e do grande pico na Figura 3C). Uma pequena fração de mutações nesta concentração ter efeitos substanciais sobre a aptidão (<< 0, correspondendo a pixels azuis na Figura 3B e a cauda esquerda na Figura 3C); exp ectedly, que incluem mutações dentro dos resíduos no local ativos altamente conservadas (S70, K73, S130, D131, N132, K234 e G236) 13,14. Em contraste, algumas mutações aumentar consideravelmente a aptidão sobre o que de TEM-1 (>> 0, correspondendo a pixels vermelhos na Figura 3B), como poderia ser esperado uma vez que TEM-1 é altamente eficiente na hidrólise de ampicilina ( 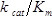 ≈ 10 7 M -1 s -1) 14.
≈ 10 7 M -1 s -1) 14.
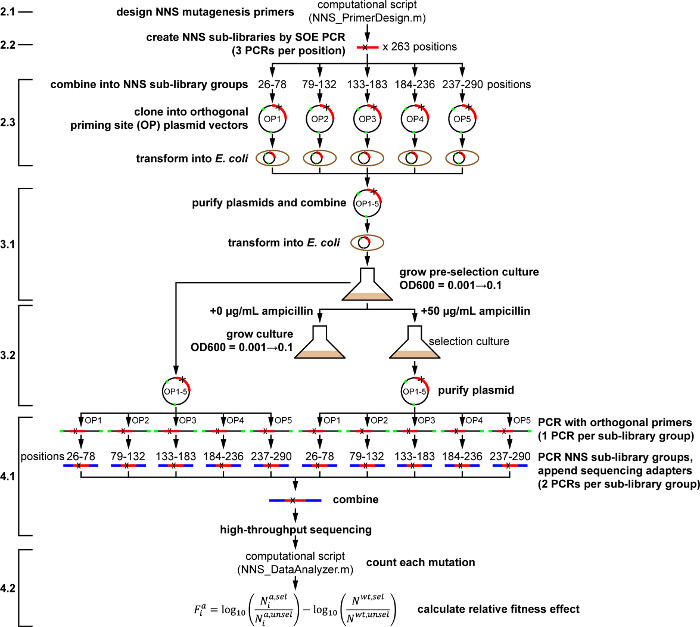
Figura 1. Esboço de Whole-proteína saturação mutagênese Protocolo para TEM-1 β-lactamase em Seleção Ampicilina. Ações, conforme descrito no protocolo são exibidas em negrito. etapas do protocolo numeradas são mostrados à esquerda, por referência ao texto principal.k "> Por favor clique aqui para ver uma versão maior desta figura.

Figura 2. Validação de Orthogonal Priming site plasmídeo vetores. Mapa (A) O plasmídeo para as cinco plasmídeos pBR322 contendo locais de iniciação modificados ortogonais (pBR322_OP1 - pBR322_OP5). Localização e direção dos locais de priming ortogonais são indicados. Localizações de vários sítios de restrição são rotulados; o todo-proteína biblioteca de mutagénese de saturação TEM-1 (que inclui todo o gene TEM-1 e o promotor) é clonado em-entre os sítios de restrição Aatll e AvrII. Abreviaturas: gene de resistência à tetraciclina tet, ori origem da replicação (B) Cada par de iniciadores ortogonais (OP1-OP5) foi testado numa PCR contendo todos os cinco plasmídeos pBR322_OP1-5 (+), ou com todos os plasmídeos menos o respectivo plasmídeo (. ˗). É mostrado um brometo de etídiogel de agarose corado (1% w / v) carregado com cada reacção de PCR, o tamanho separados por electroforese. O tamanho esperado do produto é 1628 pb; a primeira faixa é uma escada de DNA, tamanhos de normas relevantes são indicados. Por favor clique aqui para ver uma versão maior desta figura.

Figura 3. Resultados da TEM-1 β-lactamase Experiment Saturação Whole-proteína. (A) Histograma mostrando a distribuição das contagens para cada mutação de aminoácidos obtida a partir da sequenciação de alto rendimento da biblioteca a partir da condição de pré-selecção. Para simplificar, mutações com zero contagens (53 mutações) são mostrados como tendo uma contagem de um. Efeitos (B) Academia de todas as mutações de um único aminoácido na TEM-1 sob selecção com 50 ug / ml de ampicilina. É mostrada a dadosmatriz que contém o efeito de fitness relativa () representado por colorimetria com o azul efeito deletério representando, vermelho um efeito positivo e branco nenhum efeito de fitness relativamente ao tipo selvagem. As mutações para os quais não há contagens foram obtidas a partir da cultura de pré-selecção são de cor preta. As linhas representam as posições ao longo da sequência primária e colunas indicam mutação de um dos vinte aminoácidos ou codão de paragem (indicado pelo código de uma letra, é * codão de terminação); a estrutura secundária de TEM-1 é indicado em motivos altamente conservadas para a esquerda e vários dentro do local activo está indicado à direita. (C) Histograma mostrando a distribuição dos efeitos relativos de fitness. São mostrados os resultados para essas mutações com> 100 contagens obtidas de sequenciar a cultura pré-selecção. Por favor clique aqui para ver uma versão maior desta figura.
| Reagente | Massa ou volume | Comente |
| tiptona | 2 g | |
| Extracto de levedura | 0,5 g | |
| O cloreto de sódio | 0,06 g | |
| Cloreto de potássio | 0,02 g | |
| Sulfato de magnésio | 0,24 g | |
| Água purificada | 100 ml |
Tabela 1. Super Optimal Caldo (SOB). Nomes de reagentes e quantidades utilizadas na preparação de 100 ml SOB (Protocolo passo 1.1).
| Reagente | Massa ou Volume | Comente |
| tiptona | 10 g | |
| Extracto de levedura | 5 g | |
| O cloreto de sódio | 10 g | |
| Água purificada | 1? L |
Tabela 2. Luria-Bertani (LB). Nomes de reagentes e quantidades utilizadas na preparação de 1 L LB (passo protocolo 1.1).
| Reagente | Massa ou volume | Comente |
| tiptona | 10 g | |
| Extracto de levedura | 5 g | |
| O cloreto de sódio | 10 g | |
| agar | 15 g | |
| Água purificada | 1? L |
Nomes de reagentes Tabela 3. LB-agar. E as quantidades utilizadas na preparação de LB-agar (protocolo passo 1.1).
| Reagente | Volume | Comente |
| tampão 5x PCR | 1.450 l | |
| aditivo de PCR | 1.450 l | |
| dNTP 2 mM | 725 ul | 2 mM de cada nucleótido |
| AatII_F 50 uM ou AvrII_R iniciador | 145 ul | |
| 1 ng / uL do plasmídeo pBR322_AvrII | 145 ul | |
| 2 unidades / ul de polimerase de ADN | 72,5 ul | |
| água | 363 ul |
Tabela 4. primeira rodada mutagênicos PCR Master Mix. Nomes de reagentes e quantidades para preparar a mistura principal para a primeira rodada mutagênico PCR (passo protocolo 2.2.1). quantidade total é suficiente para 290 25 reações ul.
| Reagente | Volume | Comente |
| tampão 5x PCR | 1.450 l | |
| aditivo de PCR | 1.450 l | |
| dNTP 2 mM | 725 ul | 2 mM de cada nucleótido |
| 50 uM de iniciadores AatII_F | 145 ul | |
| 50 uM de iniciadores AvrII_R | 145 ul | |
| 2 unidades / ul de polimerase de ADN | 72,5 ul | |
| água | 2,973 ul |
Tabela 5. Segunda-round mutagênicos PCR Master Mix. Nomes de reagentes e quantidades para preparar a mistura principal para a segunda rodada mutagênico PCR (passo protocolo 2.2.2). quantidade total é suficiente para 290 25 reações ul.
| Reagente | Volume | Comente |
| tampão 5x PCR | 20 ul | |
| aditivo de PCR | 20 ul | |
| dNTP 2 mM | 10 ul | 2 mM de cada nucleótido |
| 50 uM de iniciadores AvrII_F | 2 ul | |
| 50 uM AatII_OP1_R - AatII_OP5_R iniciador | 2 ul | um iniciador por reacção, emparelhado com respectivo plasmídeo |
| 1 ng / uL do plasmídeo pBR322_OP1-5 | 2 ul | um plasmídeo por reacção |
| 2 unidades / ul de polimerase de ADN | 1 ul | |
| água | 43 ul |
Tabela 6. Vector de clonagem PCR. Nomes de reagentes e quantidades para a preparação da PCR para fazer os vectores de clonagem (protocolo passo 2.3.2).
| Reagente | Volume | Comente |
| 10x tampão de enzima de restrição | 5 ul | |
| 4 unidades / ul AvrII | 2,5 ul | |
| 20 unidades / ul Aatll | 0,5 ul | |
| DNA para digestão de restrição | de volume para 500 ng | |
| água | ao volume total de 50 ul |
Tabela 7. Restrição Resumos. Nomes de reagentes e quantidades para as digestões de restrição de vetores de clonagem e grupos sub-biblioteca NNS (protocolo passo 2.3.3).
| Reagente | Volume | Comente |
| de tampão ligase 10x ADN T4 | 5 ul | |
| DNA grupo purificada digerido por restrição NNS sub-biblioteca | de volume para 48 ng | |
| ADN vector de clonagem, purificado, digerido por restrição | de volume para 52 ng | |
| 400 unidades / ul de DNA ligase de T4 | 1 ul | |
| água | ao volume total de 20 ul |
Tabela 8. As ligações nomes de reagentes e quantidades para ligadura de vetores de clonagem com grupos sub-biblioteca NNS de restrição digerida em um 1:. Vector 3: inserir razão molar (protocolo passo 2.3.4).
| Reagente | Volume | Comente |
| tampão 5x PCR | 55 ul | |
| aditivo de PCR | 55 ul | |
| dNTP 2 mM | 27,5 ul | 2 mM de cada nucleótido |
| 2 unidades / ul de polimerase de ADN | 2,75 mL | |
| água | 113 ul |
Tabela 9. Os reagentes de PCR para a preparação de amostras para seqüenciamento de alta capacidade. Nomes de reagentes e quantidades para a preparação de misturas principais de PCR utilizados para de-multiplexing com primers ortogonais (4.1.1), isolando grupos sub-biblioteca NNS (protocolo da etapa 4.1.2) e a adição de sequências de indexação (protocolo de passo 4.1.3). quantidade total é suficiente para 11 25 reações ul.
Discussão
Aqui é um protocolo descrito para realizar a avaliação funcional de bibliotecas de mutagénese de saturação total em proteínas, usando a tecnologia de sequenciação de alto rendimento. Um aspecto importante do método é o uso de iniciadores ortogonais durante o processo de clonagem. Resumidamente, cada posição de aminoácido é ao acaso por PCR mutagénica, e misturados entre si em grupos de posições em que o comprimento de sequência combinada é acomodada por sequenciação de alto rendimento. Estes grupos são clonados em vectores plasmídicos contendo locais de iniciação de síntese de pares ortogonais, misturados em conjunto e sujeitos a selecção, então desmultiplexado utilizando os iniciadores ortogonais, e, subsequentemente, sequenciado profunda. Uma vez que mutações estão confinados dentro do limite de comprimento de sequenciação ler, esta abordagem maximiza o número de leituras útil contendo mutações de genes de tamanho maior do que o comprimento ler sequenciação. Além disso, esta técnica permite a selecção simultânea ou "um-lote" de toda a mutatiobiblioteca final, reduzindo a carga de trabalho, bem como a possibilidade de que as mutações experimentar diferentes níveis de selecção. Praticamente, os passos críticos no protocolo largamente organização preocupação: durante o processo de clonagem (passo protocolo 2.3) deve-se assegurar a mistura correcta de produtos de PCR mutagénica em grupos e a sua subsequente clonagem no vector de sítio de iniciação correcta ortogonal; durante a preparação da amostra para sequenciação (protocolo passo 4.1), os iniciadores ortogonais correctas, bem como iniciadores para isolar cada um dos grupos NNS sub-biblioteca e adicionar sequências de indexação, deve ser utilizado.
Os três principais etapas do protocolo de construção de bibliotecas -, de selecção e de sequenciação - podem ser modificadas em vários aspectos. Durante uma construção da biblioteca poderia introduzir mutações usando uma variedade de técnicas, por exemplo, por PCR propensa a erro, ou através da construção do gene utilizando oligonucleótidos sintetizados através da dopagem em uma pequena fracção de nucleótidos alternativas 21. Pode-se construir a biblioteca para incluir mutações duplas ou de ordem mais elevada em segmentos da proteína (isto é, grupos NNS sub-biblioteca), ou em alternativas genótipo fundos 22. É importante ressaltar que todas as modificações para a etapa de construção da biblioteca no entanto deve satisfazer o critério de que a correspondência entre a localização de mutações na sequência e a sequência lida comprimento é mantida. Por conseguinte, este critério exclui a aplicação do protocolo no sentido de estudos abrangentes de mutações múltiplas através de uma proteína. Modificações na segunda parte do protocolo incluem condições de selecção alternativos: (. Por exemplo, a temperatura, os níveis de nutrientes) diferentes tipos β-lactâmicos (ou uma combinação) e concentrações, condições de stress externos, tipo de hospedeiro (por exemplo, diferentes tipos de bactérias), ou diferentes tempos de amostragem (horas ou dias). Por exemplo, em trabalhos anteriores que examinaram os efeitos da aptidão de todas as mutações ácidos aminados únicana TEM-1 sob diferentes concentrações de ampicilina, e sob a cefalosporina de terceira geração cefotaxima 13. Com relação à terceira etapa do protocolo, no momento, não recomendamos desviar a escolha da plataforma de sequenciamento usada aqui (ver Tabela de Materiais). Enquanto sequenciamento ler comprimentos são, de facto atualmente mais em outras plataformas, o número de leituras obtidas é actualmente muito inferior; Em geral, a precisão com que pode ser determinado o efeito de uma mutação é proporcional ao número de leituras obtidas (ver equação no passo protocolo 4.2.5).
Em grande parte, por simplicidade, o protocolo usa TEM-1 β-lactamase como um sistema modelo, no entanto, a metodologia aqui descrita pode ser estendida a outros sistemas para os quais uma selecção de alto rendimento ou um ensaio de rastreio é em lugar. A construção de tais ensaios é, contudo, frequentemente não trivial: Em primeiro lugar, deve ser estabelecida uma estratégia para a compartimentalização do gene (mutação) e proteína em conjunto, Por exemplo, dentro de uma célula, gota de líquido (como numa plataforma de microfluidos), ou por exibição em fagos. Em segundo lugar, e mais importante, uma ligação quantitativa entre a função da proteína e um fenótipo seleccionável, ou adequação, deve ser estabelecida. Para as enzimas envolvidas no metabolismo ou resistência a antibióticos, a capacidade das células para crescer em nutriente de abandono ou meios de antibióticos é muitas vezes uma função directa da actividade enzimática. Uma abordagem mais sintética pode ser utilizado em outros sistemas, por exemplo através da ligação proteína-proteína de afinidade de ligação ao gene repórter (por ex., Proteína fluorescente) expressão em bactérias ou leveduras 11,23, ou utilizando um substrato de enzima f luorogénico em um sistema de microfluidos 24 . Por fim, um tal ensaio deve ser escalável, para resolver o tamanho de uma biblioteca de mutagénese todo-proteína.
Em resumo, uma abordagem de alto rendimento à base de sequenciamento para a avaliação funcional de bibliotecas saturação mutagênese todo-proteína é described aqui. Central à abordagem é a construção da biblioteca de mutagénese dentro de segmentos ao longo do gene, e a utilização de códigos de barras de iniciadores ortogonais para etiquetar cada segmento para multiplexagem e de desmultiplexagem a biblioteca. Prevemos que este protocolo pode ser facilmente aplicada a outras proteínas para o qual foi desenvolvida uma seleção de alto rendimento adequado ou tela.
Divulgações
The authors declare they have no competing financial interests
Agradecimentos
R.R. acknowledges support from the National Institutes of Health (RO1EY018720-05), the Robert A. Welch Foundation (I-1366), and the Green Center for Systems Biology.
Materiais
| Name | Company | Catalog Number | Comments |
| Typtone | Research Products Intl. Corp. | T60060-1000.0 | |
| Yeast extract | Research Products Intl. Corp. | Y20020-500.0 | |
| Sodium chloride | Fisher Scientific | BP358-212 | |
| Potassium chloride | Sigma-Aldrich | P9333-500G | |
| Magnesium sulfate | Sigma-Aldrich | M7506-500G | |
| Agar | Fisher Scientific | BP1423-500 | |
| Tetracycline hydrochloride | Sigma-Aldrich | T7660-5G | |
| Petri plates | Corning | 351029 | |
| MATLAB | Mathworks | http://www.mathworks.com/products/matlab/ | |
| Oligonucleotide primers | Integrated DNA Technologies | https://www.idtdna.com/pages/products/dna-rna/custom-dna-oligos | 25 nmol scale, standard desalting |
| pBR322_AvrII | available upon request | pBR322 plasmid modified to contain AvrII restriction site downstream of the TEM-1 gene | |
| pBR322_OP1 – pBR322_OP5 | available upon request | five modified pBR322 plasmids each containing a pair of orthogonal priming sites | |
| Q5 high-fidelity DNA polymerase | New England Biolabs | M0491L | includes 5x PCR buffer and PCR additive (GC enhancer) |
| 15 ml conical tube | Corning | 430025 | |
| Multichannel pipettes (Eppendorf ResearchPlus) | Eppendorf | ||
| PCR plate, 96 well | Fisher Scientific | 14230232 | |
| 96 well plate seal | Excel Scientific | F-96-100 | |
| Veriti 96-well thermal cycler | Applied Biosystems | 4375786 | |
| 6x gel loading dye | New England Biolabs | B7024S | |
| Agarose | Research Products Intl. Corp. | 20090-500.0 | |
| Ethidium bromide | Bio-Rad | 161-0433 | |
| UV transilluminator (FOTO/Analyst ImageTech) | Fotodyne Inc. | http://www.fotodyne.com/content/ImageTech_gel_documentation | |
| EB buffer | Qiagen | 19086 | |
| 96-well black-walled, clear bottom assay plates | Corning | 3651 | |
| Lambda phage DNA | New England Biolabs | N3011S | |
| PicoGreen dsDNA reagent | Invitrogen | P7581 | dsDNA quantitation reagent, used in protocol step 2.2.4 |
| Victor 3 V microplate reader | PerkinElmer | ||
| DNA purification kit | Zymo Research | D4003 | |
| Microcentrifuge tubes | Corning | 3621 | |
| Long-wavelength UV illuminator | Fisher Scientific | FBUVLS-80 | |
| Agarose gel DNA extraction buffer | Zymo Research | D4001-1-100 | |
| AatII | New England Biolabs | R0117S | |
| AvrII | New England Biolabs | R0174L | |
| T4 DNA ligase | New England Biolabs | M0202S | |
| EVB100 electrocompetent E. coli | Avidity | EVB100 | |
| Electroporator (E. coli Pulser) | Bio-Rad | 1652102 | |
| Electroporation cuvettes | Bio-Rad | 165-2089 | |
| Spectrophotometer (Ultrospec 3100 pro) | Amersham Biosciences | 80211237 | |
| 50 ml conical tubes | Corning | 430828 | |
| Plasmid purification kit | Macherey-Nagel | 740588.25 | |
| 8 well PCR strip tubes | Axygen | 321-10-551 | |
| Qubit dsDNA HS assay kit | Invitrogen | Q32854 | dsDNA quantitation reagent |
| Qubit assay tubes | Invitrogen | Q32856 | |
| Qubit fluorometer | Invitrogen | Q32866 | |
| Ampicillin sodium salt | Akron Biotechnology | 50824296 | |
| MiSeq reagent kit v2 (500 cycles) | Illumina | MS-102-2003 | |
| MiSeq desktop sequencer | Illumina | http://www.illumina.com/systems/miseq.html | alternatively, one could sequence on Illumina HiSeq platform |
| FLASh software | John Hopkins University - open source | http://ccb.jhu.edu/software/FLASH/ | software to merge paired-end reads from next-generation sequencing data |
| AatII_F | GATAATAATGGTTTCTTAGACG TCAGGTGGC | ||
| AvrII_R | CTTCACCTAGGTCCTTTTAAAT TAAAAATGAAG | ||
| AvrII_F | CTTCATTTTTAATTTAAAAGGA CCTAGGTGAAG | ||
| AatII_OP1_R | ACCTGACGTCCGTATTTCAAC TGTCCGGTCTAAGAAACCATT ATTATCATGACATTAAC | ||
| AatII_OP2_R | ACCTGACGTCCGCTCACGGA GTGTACTAATTAAGAAACCATT ATTATCATGACATTAAC | ||
| AatII_OP3_R | ACCTGACGTCGTACGTCTGA ACTTGGGACTTAAGAAACCA TTATTATCATGACATTAAC | ||
| AatII_OP4_R | ACCTGACGTCCCGTTCTCGAT ACCAAGTGATAAGAAACCATT ATTATCATGACATTAAC | ||
| AatII_OP5_R | ACCTGACGTCGTCCGTCGGA GTAACAATCTTAAGAAACCAT TATTATCATGACATTAAC | ||
| OP1_F | GACCGGACAGTTGAAATACG | ||
| OP1_R | CGACGTACAGGACAATTTCC | ||
| OP2_F | ATTAGTACACTCCGTGAGCG | ||
| OP2_R | AGTATTAGGCGTCAAGGTCC | ||
| OP3_F | AGTCCCAAGTTCAGACGTAC | ||
| OP3_R | GAAAAGTCCCAATGAGTGCC | ||
| OP4_F | TCACTTGGTATCGAGAACGG | ||
| OP4_R | TATCACGGAAGGACTCAACG | ||
| OP5_F | AGATTGTTACTCCGACGGAC | ||
| OP5_R | TATAACAGGCTGCTGAGACC | ||
| Group1_F | ACACTCTTTCCCTACACGAC GCTCTTCCGATCTNNNNNGC ATTTTGCCTACCGGTTTTTGC | ||
| Group1_R | GTGACTGGAGTTCAGACGTG TGCTCTTCCGATCTNNNNNTC TTGCCCGGCGTCAAC | ||
| Group2_F | ACACTCTTTCCCTACACGAC GCTCTTCCGATCTNNNNNGA ACGTTTTCCAATGATGAGCAC | ||
| Group2_R | GTGACTGGAGTTCAGACGTG TGCTCTTCCGATCTNNNNNGT CCTCCGATCGTTGTCAGAAG | ||
| Group3_F | ACACTCTTTCCCTACACGAC GCTCTTCCGATCTNNNNNAG TAAGAGAATTATGCAGTGCTGCC | ||
| Group3_R | GTGACTGGAGTTCAGACGTG TGCTCTTCCGATCTNNNNNTC GCCAGTTAATAGTTTGCGC | ||
| Group4_F | ACACTCTTTCCCTACACGAC GCTCTTCCGATCTNNNNNCC AAACGACGAGCGTGACAC | ||
| Group4_R | GTGACTGGAGTTCAGACGTG TGCTCTTCCGATCTNNNNNGC AATGATACCGCGAGACCC | ||
| Group5_F | ACACTCTTTCCCTACACGAC GCTCTTCCGATCTNNNNNCG GCTGGCTGGTTTATTGC | ||
| Group5_R | GTGACTGGAGTTCAGACGTG TGCTCTTCCGATCTNNNNNTAT ATGAGTAAACTTGGTCTGACAG | ||
| 501_F | AATGATACGGCGACCACCGA GATCTACACTATAGCCTACAC TCTTTCCCTACACGAC | ||
| 502_F | AATGATACGGCGACCACCGA GATCTACACATAGAGGCACA CTCTTTCCCTACACGAC | ||
| 503_F | AATGATACGGCGACCACCGA GATCTACACCCTATCCTACAC TCTTTCCCTACACGAC | ||
| 504_F | AATGATACGGCGACCACCGA GATCTACACGGCTCTGAACA CTCTTTCCCTACACGAC | ||
| 505_F | AATGATACGGCGACCACCGA GATCTACACAGGCGAAGACA CTCTTTCCCTACACGAC | ||
| 701_R | CAAGCAGAAGACGGCATAC GAGATCGAGTAATGTGACTG GAGTTCAGACGTG | ||
| 702_R | CAAGCAGAAGACGGCATAC GAGATTCTCCGGAGTGACTG GAGTTCAGACGTG |
Referências
- Hutchison, C. A., et al. Mutagenesis at a specific position in a DNA sequence. J Biol Chem. 253 (18), 6551-6560 (1978).
- Mullis, K. B., Faloona, F. A. Specific synthesis of DNA in vitro via a polymerase-catalyzed chain reaction. Methods Enzymol. 155, 335-350 (1987).
- Papworth, C., Bauer, J. C., Braman, J., Wright, D. A. Site-directed mutagenesis in one day with >80% efficiency. Strategies. 9 (3), 3-4 (1996).
- Higuchi, R., Krummel, B., Saiki, R. K. A general method of in vitro preparation and specific mutagenesis of DNA fragments: study of protein and DNA interactions. Nucleic Acids Res. 16 (15), 7351-7367 (1988).
- Rennell, D., Bouvier, S. E., Hardy, L. W., Poteete, A. R. Systematic mutation of bacteriophage T4 lysozyme. J Mol Biol. 222 (1), 67-88 (1991).
- Markiewicz, P., Kleina, L. G., Cruz, C., Ehret, S., Miller, J. H. Genetic studies of the lac repressor. XIV. Analysis of 4000 altered Escherichia coli lac repressors reveals essential and non-essential residues, as well as 'spacers' which do not require a specific sequence. J Mol Biol. 240 (5), 421-433 (1994).
- Kleina, L. G., Miller, J. H. Genetic studies of the lac repressor. XIII. Extensive amino acid replacements generated by the use of natural and synthetic nonsense suppressors. J Mol Biol. 212 (2), 295-318 (1990).
- Huang, W., Petrosino, J., Hirsch, M., Shenkin, P. S., Palzkill, T. Amino acid sequence determinants of beta-lactamase structure and activity. J Mol Biol. 258 (4), 688-703 (1996).
- Fowler, D. M., et al. High-resolution mapping of protein sequence-function relationships. Nat Methods. 7 (9), 741-746 (2010).
- Hietpas, R. T., Jensen, J. D., Bolon, D. N. Experimental illumination of a fitness landscape. Proc Natl Acad Sci U S A. 108 (19), 7896-7901 (2011).
- McLaughlin, R. N., Poelwijk, F. J., Raman, A., Gosal, W. S., Ranganathan, R. The spatial architecture of protein function and adaptation. Nature. 491 (7422), 138-142 (2012).
- Deng, Z., et al. Deep sequencing of systematic combinatorial libraries reveals beta-lactamase sequence constraints at high resolution. J Mol Biol. 424 (3-4), 150-167 (2012).
- Stiffler, M. A., Hekstra, D. R., Ranganathan, R. Evolvability as a Function of Purifying Selection in TEM-1 beta-Lactamase. Cell. 160 (5), 882-892 (2015).
- Matagne, A., Lamotte-Brasseur, J., Frere, J. M. Catalytic properties of class A beta-lactamases: efficiency and diversity. Biochem J. 330 (Pt2), 581-598 (1998).
- Salverda, M. L., De Visser, J. A., Barlow, M. Natural evolution of TEM-1 beta-lactamase: experimental reconstruction and clinical relevance. FEMS Microbiol Rev. 34 (6), 1015-1036 (2010).
- Weinreich, D. M., Delaney, N. F., Depristo, M. A., Hartl, D. L. Darwinian evolution can follow only very few mutational paths to fitter proteins. Science. 312 (5770), 111-114 (2006).
- Stewart, S. M., Fisher, M., Young, J. E., Lutz, W. Ampicillin levels in sputum, serum, and saliva. Thorax. 25 (3), 304-311 (1970).
- Giachetto, G., et al. Ampicillin and penicillin concentration in serum and pleural fluid of hospitalized children with community-acquired pneumonia. Pediatr Infect Dis J. 23 (7), 625-629 (2004).
- Ambler, R. P., et al. A standard numbering scheme for the class A beta-lactamases. Biochem J. 276 (Pt 1), 269-270 (1991).
- Magoc, T., Salzberg, S. L. FLASH: fast length adjustment of short reads to improve genome assemblies). Bioinformatics. 27 (21), 2957-2963 (2011).
- Melamed, D., Young, D. L., Gamble, C. E., Miller, C. R., Fields, S. Deep mutational scanning of an RRM domain of the Saccharomyces cerevisiae poly(A)-binding protein. RNA. 19 (11), 1537-1551 (2013).
- Bank, C., Hietpas, R. T., Jensen, J. D., Bolon, D. N. A systematic survey of an intragenic epistatic landscape. Mol Biol Evol. 32 (1), 229-238 (2015).
- Dove, S. L., Joung, J. K., Hochschild, A. Activation of prokaryotic transcription through arbitrary protein-protein contacts. Nature. 386 (6625), 627-630 (1997).
- Romero, P. A., Tran, T. M., Abate, A. R. Dissecting enzyme function with microfluidic-based deep mutational scanning. Proc Natl Acad Sci U S A. 112 (23), 7159-7164 (2015).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados